É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Monitoramento de mudanças na estrutura Equilibrium RNA por "peroxidativo" e "oxidativo" Footprinting Radical Hidroxila
Neste Artigo
Resumo
Este protocolo descreve como quantificar o Mg (II) dependente da formação de RNA estrutura terciária através de dois métodos de pegada radical hidroxila.
Resumo
Moléculas de RNA desempenham um papel essencial na biologia. Além de transmitir a informação genética, RNA pode dobrar em exclusivo estruturas terciárias cumprindo um papel biológico específico como ligante regulador, ou catalisador. Informações sobre a formação de contato superior é essencial para compreender a função das moléculas de RNA. Radicais hidroxila (OH •) são sondas única da estrutura dos ácidos nucléicos, devido à sua alta reatividade e tamanho pequeno. 1 Quando utilizado como uma sonda pegada, os radicais hidroxila mapa da superfície acessível solvente do backbone fosfodiéster do DNA e RNA 1 2 com tão fina como a resolução de nucleotídeo único. Footprinting radical hidroxila pode ser usado para identificar os nucleotídeos dentro de uma superfície de contato intermolecular, por exemplo, DNA-proteína-1 e RNA-proteína complexos. Equilibrium 3 e 4 transições cinética pode ser determinada através da realização de pegada radical hidroxila em função de um solutina variável ou tempo, respectivamente. Uma característica fundamental da pegada é que a exposição limitada à sonda (por exemplo, 'single-hit cinética ") resulta na amostragem uniforme de cada nucleotídeo do polímero. 5
Neste artigo de vídeo, usamos o domínio P4-P6 da ribozima Tetrahymena para ilustrar RNA preparação de amostras e determinação de um (II) mediada por Mg isotermas de dobradura. Descrevemos a utilização do protocolo pegada bem conhecido radical hidroxila que requer H 2 O 2 (nós chamamos este protocolo "peroxidativo ') e um valor, mas não muito conhecido alternativa, que usa naturalmente O 2 dissolvido (chamamos isso de" o protocolo oxidativo "). Uma visão geral da redução de dados, transformação e procedimentos de análise é apresentado.
Protocolo
1. Preparação dos Reagentes Footprinting
- Preparar um tampão de reação 10x, contendo 100 mM cacodilato de sódio, 1 mM EDTA, e 1 M KCl. Ajustar o pH para 7,4. Filtrar o tampão 0,2 mM usando um dispositivo de filtro de acetato (Nalgene). Observação: não pipeta RNA diretamente em tampão 10x.
- Prepare a mistura de reacção de titulação para cada reação, como indicado na Tabela 1. O volume da mistura de titulação (1x buffer e Mg (II) na concentração desejada) deve ser de 90 mL, antes de adicionar 10μl de RNA em tampão 1x.
- Preparar um tampão de digestão RNAse T1 contendo Uréia 6.63M, citrato de sódio 20mM, EDTA 1mM, 0,25 mcg / mL tRNA, cyanol xileno 0,025% e 0,025% azul de bromofenol. Esse buffer pode ser armazenado a 4 ° C por até 6 meses.
- A reação Fenton requer preparada soluções aquosas de 250 mM Fe (NH 4) 2 (SO 4) 2, 250 mM EDTA e 500 mM ascorbato de sódio. As concentrações mencionado no capítulo 10,5 são ideais para experimentos de equilíbrio em tampão cacodilato de sódio, como que não limpam radicais e diversas concentrações de íons divalentes.
- Reagentes para a reação footprinting peroxidativo (1.5.1) e oxidativo (1.5.2).
- A reação Fenton peroxidativo requer preparada soluções aquosas de 100 mM solução Fe-EDTA, e 50 mM ascorbato de sódio e 1,5% H 2 O 2. A solução Fe-EDTA é preparada pela mistura Fe (NH 4) 2 (SO 4) 2 com EDTA a ter 1,1 vezes mais excesso de EDTA de ferro.
- Pouco antes de iniciar a reação de clivagem oxidativa (3.4.2), criar a mistura de reacção de Fenton, combinando 2 l da Fe (NH 4) 2 (SO 4) 2 (250 mM), 2,2 mL de EDTA (250 mM), 62,5 mL ascorbato de sódio (500 mM), 22,5 mL tampão de reação 10x e 135,5 mL de H 2 O para alcançar a concentração final na mistura de reação footprinting (3.4.2) de 0.10 mM, 0,11 mM e 6,61 mM, respectivamente.
2. Preparação para o RNA Experiment Footprinting
- RNA podem ser produzidos pela norma in vitro a transcrição dos modelos de DNA 6 ou, se menor de 50 nucleotídeos, ser adquirido (por exemplo, Integrated DNA Technologies, www.idtdna.com). Siga os procedimentos de limpeza recomendado laboratório para manter a integridade da amostra de RNA durante as experiências footprinting. 7
- Desfosforilação 8 de RNA transcrito in vitro é necessário antes de 32 P rotulagem do terminal 5 'do RNA. 10 pmol de RNA são finais marcados por kinasing. 9 A solução deve ser nublado após a inativação da fosfatase sucesso.
- Purificar RNA marcado radioativamente usando eletroforese em gel desnaturante. O RNA é recuperada por dois subseqüentes gel extrações utilizando acetato de sódio 0,3 M (pH 5,5). RNA é precipitado através da mistura de 0,5 ml de solução aquosa com 1 ml de etanol e posteriorincubação por 1 minuto em gelo seco. Rotação da amostra a 4 ° C, 12500 rpm para 0,5 hr. Desprezar o sobrenadante e lavar RNA pellet com etanol 70%. Remover o sobrenadante após a centrifugação adicional e seco pellet no vácuo.
- Dissolver a 32 amostras P-rotulados RNA em 330 ul de 1x 30 mL Buffer.Pipet da solução de RNA em um tubo de reação, precipitado com etanol e seco RNA no vácuo; manter este tubo para gerar a escada de referência (passo 3.5). Desnaturar a solução tamponada de RNA por aquecimento a 95 ° C por 2 min. Arrefecer as amostras à temperatura ambiente por 15 min e dar um giro rápido para derrubar o condensados nas tampas dos tubos na solução.
3. O Experimento Footprinting
- O pente eletroforese determina o número máximo de pontos de dados por gel analítica. Usamos a 30 comb bem. A escada de referência ocupa 2 poços e um controle uncleaved. Prepare 27 tubos de reação. O Mg final (II) concentrations devem ser uniformemente espaçados em uma escala logarítmica que mede ordens de magnitude em torno do ponto médio de titulação.
- Separadamente, incube a RNA eo Mg (II) de soluções a 50 ° C por 5 min. Misture 10 ml de solução de RNA com 90 mL da correspondente quantidade de Mg (II) em 1x buffer para chegar a um volume final de 100 l. Incubar as soluções por 30 min a 50 ° C.
- Deixe as soluções equilibrar a 25 ° C por 1 hora ao dobrar ocorre.
- Peroxidativo (3.4.1) ou oxidativo (3.4.2) reação pegada radical hidroxila.
- Iniciar a reação peroxidativo colocando gotas de 2 l Fe-EDTA (100 mM), 2 mL de sódio L-ascorbato (50 mM) e 2 mL H 2 O 2 (1,5%) separados uns dos outros na parte superior interna da reação tubo contendo a solução de RNA e iniciar a reação footprinting pela mistura vigorosa. Parar a reacção após 15 segundos, adicionando 300 mL de etanol frio absoluto. Ligue os tubos de 3-5 vezes. Lavar precipitado, esecar o pellet como no passo 2.4.
- Iniciar o footprintingreaction hidroxila oxidativo radical pela adição de 5 mL da mistura preparada reação de Fenton (1,6). Incubar por 30 min a 25 ° C. Adicione 300 ml de etanol absoluto frio para saciar a reação e misture girando os tubos de 3-5 vezes. Precipitado, lavar e secar o pellet, conforme indicado no passo 2.4.
- Gerar uma RNase T1 amostra de referência digerir acordo com os procedimentos padrão de 10 usando a solução preparada na etapa 1.3.
- Dissolver pellets de RNA em 8 mL de gel loading dye II (Ambion) e RNA é ressuspendido confirmar usando o contador Geiger.
- Prepare uma desnaturação, gel de poliacrilamida 8% seqüenciamento de acordo com protocolos padrão. 11 amostras de carga, incluindo duas referências e um controle. Separar fragmentos de RNA de 60 W - 75 W por 2,5 hr. Expor gel seco a uma tela de fósforo de armazenamento durante a noite. Varredura tela de fósforo com um sistema de imagem para autoradiografia filmless, por exemplo,. Tempestade 865 (GE Healthcare) ou Typhoon (GE Healthcare). Transferência de arquivos de imagem gel para o seu computador.
4. Análise de Dados
- A reatividade radical hidroxila de cada nucleotídeo é determinada pela quantificação da intensidade de cada banda no gel analítica. Baixar, instalar e abrir SAFA, um fácil de usar software de código aberto para instalação de banda única e quantificação. 12, 13 Siga as instruções do manual do usuário. Resumidamente, a seqüência de RNA de carga como. Txt seguido da foto como gel. Gel arquivo. Ajustar intensidades banda, define as pistas, escolha uma pista de âncora, e realizar um alinhamento gel. Atribuição de faixas de nucleotídeos ocorre em referência a RNase escada digestão T1. Quantificar as integrais banda e usar o recurso de normalização / colorplot para normalizar e atribuir resíduos potencialmente invariantes. Salvar a saída como um arquivo txt..
- SAFA gera uma planilha contendo colunas representando pistas sobre o gel e linhas representando os emgrados densidade de banda das bandas individuais correspondentes ao fragmento de RNA. Primeiro, identificar as zonas de protecção que apresentam uma notável mudança na acessibilidade do solvente através da comparação do perfil de proteção derivado da amostra não Mg (II) com o perfil do ponto final de concentração Mg (II) (50 mM em nosso exemplo). Quanto menor o valor, mais protegido do nucleotídeo é contra o ataque dos radicais hidroxila e vice-versa.
- Criar isotermas dobrar da banda normalizado integrais de indivíduo ou grupos de nucleotídeos em relação Mg concentração (II). As isotermas são individualmente escalado para saturação fracional por fi = L + (U - L) •
 onde f denota a densidade integrada da banda (s) que está sendo analisado, L e U representam os limites inferior e superior para a transição e
onde f denota a densidade integrada da banda (s) que está sendo analisado, L e U representam os limites inferior e superior para a transição e 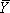 é a saturação fracionada. 14 Os dados estão aptos usando um l não-linearleste programa de análise de quadrados (Usamos tanto Origem (Origin) ou GraphPad (GraphPad Software, Inc.).) com a equação de Hill (1),
é a saturação fracionada. 14 Os dados estão aptos usando um l não-linearleste programa de análise de quadrados (Usamos tanto Origem (Origin) ou GraphPad (GraphPad Software, Inc.).) com a equação de Hill (1), 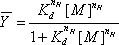
onde K d é o equilíbrio constante de dissociação, [M] corresponde à concentração da variável que media a reação de dobramento, Mg (II), neste exemplo, e n H é o coeficiente de Hill. Este procedimento escalas a transição para a saturação fracionada, determina o seu ponto médio e fornece um teste fenomenológica de saber se é uma transição sigmoidal. Isotermas derivadas de seqüenciamento géis (Figura 3A), que foram analisadas por SAFA (Figura 3B) e ajuste como descrito acima são mostrados na Figura 3C. As isotermas de saturação fracional mostrado na Figura 3 foram gerados pela escala em uma planilha os valores gerados pela SAFA contra os melhores se encaixam limites superior e inferior (U e L), utilizando a fração equação = valor / (U - L) - L / (U - L).
5. Resultados representativos:
A Figura 3 mostra resultados representativos da P4-P6 RNA • experimentos footprinting OH. A imagem gel (à esquerda) indica que uma clivagem de fundo) é mínimo (faixa direita), b) atribuição de único nucleotídeo é possível devido a bandas bem definidas na pista T1 (cliva RNase T1 em cada G), e c) o radical hidroxila induzida fragmentação do RNA é bem acima do fundo. A transição de baixa para alta Mg (II) é afiliado com a diminuição da densidade de banda integrado de individuais e grupos de faixas indicando a formação de RNA estrutura terciária. Individuais ou grupos de nucleotídeos contíguos cuja clivagem alterações concomitantemente são referidos como uma "proteção". As proteções • OH característica do Mg (II) mediada dobramento da RNA P4-P6 perto correspondem às regiões inacessíveis solvente da molécula observada em sua estrutura cristalina. 15
Dobrado moléculas RNA c um tem extensões diferentes (isto é, o quão perto a fundo a banda declínio densidades) de proteção de contato superior que não têm sido correlacionados com fenômeno estrutural ou dinâmico clara. Assim, algumas moléculas de RNA vai mostrar bem definidas transições estruturais, como mostrado na Figura 3, e alguns não. Intensidades banda são quantificados e normalizados pela SAFA análise 12 (Figura 3B). A saída é uma trama 'térmico' visualizando o relativo grau de protecção contra o • OH. A transição de cores descreve a mudança de acessibilidade em cima disso Mg (II). Branco para vermelho ou azul mostra nucleotídeos mais acessível ou mais protegidos, respectivamente. Cada grau de sombreamento é afiliado com um valor numérico que pode ser plotado como uma curva de proteção (Figura 3C) e analisados por um modelo de ligação, tais como a equação de Hill (1). O equilíbrio constante de dissociação afiliada à Proteção 153-155 é aproximadamente o dobro do valor correspondente de Proteção 163-164.
ONTEÚDO ">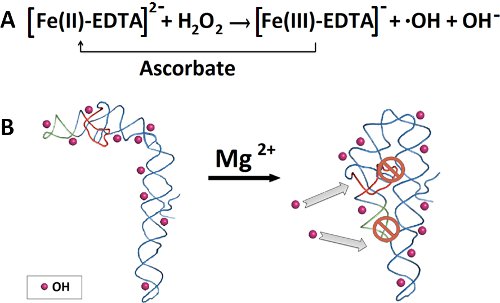
Figura 1. Produção de radicais hidroxila e P4-P6 dobrar RNA. A) Fe (II) catalisa a geração de radicais hidroxila a partir de peróxido de hidrogênio. Ascorbato reduz Fe volta (III) a ferro. B) Na ausência de Mg (II), nenhuma estrutura P4-P6 terciário é formada, permitindo que os radicais hidroxila para penetrar e se unir todas as posições backbone acessível. Além do Mg (II) inicia dobrar de P4-P6, permitindo apenas a espinha dorsal do solvente acessíveis a ser clivada pelos radicais hidroxila.
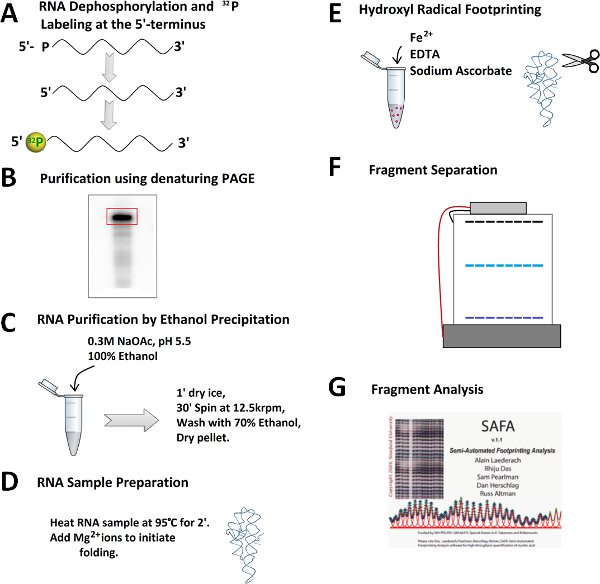
Figura 2. Esboço para uma experiência pegada radical hidroxila. A Dephophorylation) e 32 P rotulagem 5'-final da RNA. B) Purificação de 32 P-rotulados RNA em gel de poliacrilamida desnaturante. C) Excisão da banda de RNA, extração de RNA subseqüentes, e precipitação de etanol. D Prefolding) edobramento de RNA. E) A adição de recém-preparada mistura de reação Fenton para gerar os radicais hidroxila. F) separação fragmento de RNA por eletroforese desnaturante em gel de poliacrilamida. G) A quantificação de fragmentos de RNA por software SAFA.
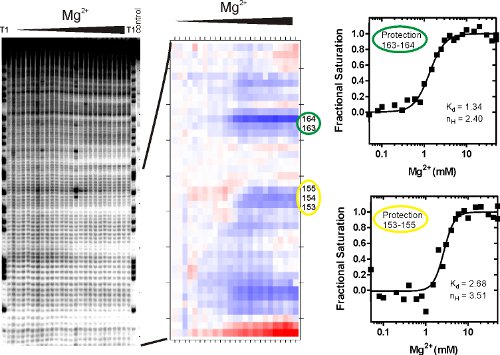
Figura 3. Análise de fragmentos de RNA após a reação peroxidativo footprinting Fenton. (A) O RNA foi exposto a radicais hidroxila e os produtos de dissociação foram resolvidas usando eletroforese em gel de poliacrilamida desnaturante (PAGE). A imagem mostra produtos de dissociação peroxidativo com o aumento da concentração de Mg (II). Pistas de referência e controle são rotulados. Este arquivo de imagem é a entrada para o programa SAFA, que quantitates o volume de cada banda. (B) O enredo "térmico" gerado pela SAFA. (C) A planilha de valores relativos a densidade de banda integrado para a saída número de nucleotídeos de SAFA é analisada por um modelo de vinculação, como o equati Colinaem (1). Regiões de proteção foram escolhidos de acordo com o aumento da densidade de banda integral como uma função da concentração de Mg (II). A d K é a concentração de Mg (II) em que metade do RNA é dobrada no local está sendo monitorado. O coeficiente de Hill, n H, é uma medida da inclinação da curva que dá informações sobre a cooperatividade de ligação e oferece um menor estimativa para o número de íons de magnésio envolvidos na dobragem deste site particular.
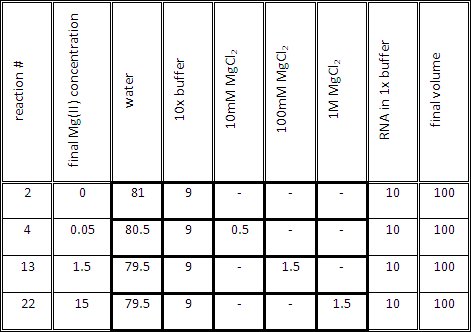
Tabela 1. Volumes Representante para a geração de amostras de RNA contendo diferentes Mg (II) as concentrações.
Discussão
Footprinting radical hidroxila é uma ferramenta valiosa para avaliar a área de superfície acessível solvente de ácidos nucléicos. Formação qualitativa e quantitativa da estrutura terciária 14 pode ser seguido em função de parâmetros como tipo e concentração de íons, pH, temperatura, proteínas de ligação ou dobrar co-fatores. A combinação irresistível de um protocolo para a frente e de baixo custo e acessibilidade resultante de solvente e informações dobrar em um nível de nucleotídeo ?...
Divulgações
Não há conflitos de interesse declarados.
Agradecimentos
Este trabalho foi financiado por doações do Instituto Nacional de Saúde RO1-GM085130 e National Science Foundation MCB0929394. Agradecemos ao Dr. Marion Schmidt por sua hospitalidade e por nos permitir filme em seu laboratório.
Materiais
| Name | Company | Catalog Number | Comments |
| Name | Company | Cat# | |
| Sodium Cacodylate(Caution! Toxic) | Sigma-Aldrich | C4945-25g | |
| EDTA (0.5 M) | Ambion | AM9260G | |
| DEPC treated water | Ambion | AM9915G | |
| Sodium Acetate (3 M) | Ambion | AM9740 | |
| MgCl2 (1 M) | Ambion | AM9530G | |
| Urea | Ambion | AM9902 | |
| Sodium Citrate | Sigma-Aldrich | W302600 | |
| tRNA | Sigma-Aldrich | R-7876 | |
| Sodium-L-ascorbate | Sigma-Aldrich | A7631-25g | |
| Fe(NH4)2(SO4)2 . 6 H2O | Sigma-Aldrich | F1543-500g | |
| RNase T1 | Fermentas | EN0541 | |
| Hydrogen Peroxide (30%) | Sigma-Aldrich | 349887 |
Referências
- Tullius, T. D., Dombroski, B. A. Hydroxyl radical "footprinting": high-resolution information about DNA-protein contacts and application to lambda repressor and Cro protein. Proc. Natl. Acad. Sci. U.S.A. 83, 5469-5473 (1986).
- Celander, D. W., Cech, T. R. Iron(II)-ethylenediaminetetraacetic acid catalyzed cleavage of RNA and DNA oligonucleotides: similar reactivity toward single- and double-stranded forms. Biochemistry. 29, 1355-1361 (1990).
- Celander, D. W., Cech, T. R. Visualizing the higher order folding of a catalytic RNA molecule. Science. 251, 401-407 (1991).
- Sclavi, B., Sullivan, M., Chance, M. R., Brenowitz, M., Woodson, S. A. RNA folding at millisecond intervals by synchrotron hydroxyl radical footprinting. Science. 279, 1940-1943 (1998).
- Tullius, T. D., Dombroski, B. A., Churchill, M., Kam, L. Hydroxyl radical footprinting: a high-resolution method for mapping protein-DNA contacts. Methods. Enzym. 155, 537-558 (1987).
- Milligan, J. F., Groebe, D. R., Witherell, G. W., Uhlenbeck, O. C. Oligoribonucleotide synthesis using T7 RNA polymerase and synthetic DNA templates. Nucleic. Acids. Res. 15, 8783-8798 (1987).
- Schlatterer, J. C., Brenowitz, M. Complementing global measures of RNA folding with local reports of backbone solvent accessibility by time resolved hydroxyl radical footprinting. Methods. 49, 142-147 (2009).
- Sambrook, J., Russell, D. W. . Molecular Cloning: A Laboratory Manual. , (2001).
- Zaug, A. J., Grosshans, C. A., Cech, T. R. Sequence-specific endoribonuclease activity of the Tetrahymena ribozyme: enhanced cleavage of certain oligonucleotide substrates that form mismatched ribozyme-substrate complexes. Biochemistry. 27, 8924-8931 (1988).
- Knapp, G. Enzymatic approaches to probing of RNA secondary and tertiary structure. Methods. Enzym. 180, 192-212 (1989).
- Slatko, B. E., Albright, L. M. Denaturing gel electrophoresis for sequencing. Curr. Protoc. Mol. Biol. Chapter 7, Unit 7.6-Unit 7.6 (2001).
- Das, R., Laederach, A., Pearlman, S. M., Herschlag, D., Altman, R. B. SAFA: semi-automated footprinting analysis software for high-throughput quantification of nucleic acid footprinting experiments. RNA. 11, 344-354 (2005).
- Simmons, K., Martin, J. S., Shcherbakova, I., Laederach, A. Rapid quantification and analysis of kinetic *OH radical footprinting data using SAFA. Methods. Enzym. 468, 47-66 (2009).
- Uchida, T., He, Q., Ralston, C. Y., Brenowitz, M., Chance, M. R. Linkage of monovalent and divalent ion binding in the folding of the P4-P6 domain of the Tetrahymena ribozyme. Biochemistry. 41, 5799-5806 (2002).
- Cate, J. H., Gooding, A. R., Podell, E., Zhou, K., Golden, B. L., Kundrot, C. E., Cech, T. R., Doudna, J. A. Crystal structure of a group I ribozyme domain: principles of RNA packing. Science. 273, 1678-1685 (1996).
- Petri, V., Brenowitz, M. Quantitative nucleic acids footprinting: thermodynamic and kinetic approaches. Curr. Opin. Biotechnol. 8, 36-44 (1997).
- Takamoto, K., Das, R., He, Q., Doniach, S., Brenowitz, M., Herschlag, D., Chance, M. R. Principles of RNA compaction: insights from the equilibrium folding pathway of the P4-P6 RNA domain in monovalent cations. J. Mol. Biol. 343, 1195-1206 (2004).
- Brenowitz, M., Senear, D. F., Shea, M. A., Ackers, G. K. Quantitative DNase footprint titration: a method for studying protein-DNA interactions. Methods. Enzym. 130, 132-181 (1986).
- Shcherbakova, I., Mitra, S. Hydroxyl-radical footprinting to probe equilibrium changes in RNA tertiary structure. Methods. Enzym. 468, 31-46 (2009).
- Howlett, G. J., Minton, A. P., Rivas, G. Analytical ultracentrifugation for the study of protein association and assembly. Curr. Opinion. Chem. Biol. 10, 430-436 (2006).
- McGinnis, J. L., Duncan, C. D., Weeks, K. M. High-throughput SHAPE and hydroxyl radical analysis of RNA structure and ribonucleoprotein assembly. Methods. Enzym. 468, 67-89 (2009).
- Jonikas, M. A., Radmer, R. J., Laederach, A., Das, R., Pearlman, S., Herschlag, D., Altman, R. B. Coarse-grained modeling of large RNA molecules with knowledge-based potentials and structural filters. RNA. 15, 189-199 (2009).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados