このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
"過酸化"と"酸化"ヒドロキシルラジカルフットプリントにより、RNAの構造における均衡の変化を監視する
要約
このプロトコルは、Mg(II)依存性ヒドロキシルラジカルフットプリンティングの2つの方法でRNA三次構造の形成を定量化する方法について説明します。
要約
RNA分子は、生物学において重要な役割を果たしている。遺伝情報を伝達することに加えて、RNAはレギュレータ、バインダーや触媒などの特定の生物学的役割を果たすユニークな立体構造に折りたたまれることができる。三次接触の形成についての情報は、RNA分子の機能を理解することが不可欠です。ヒドロキシルラジカル(•OH)は、その高い反応性と小さなサイズのため、核酸の構造のユニークなプローブである。1フットのプローブとして使用する場合は、ヒドロキシルラジカルがDNAを1とを有するRNA 2のホスホジエステルバックボーンの溶媒接触可能表面をマッピング一塩基の解像度と同じくらい素晴らしい。ヒドロキシルラジカルフットプリンティングはDNA -タンパク質1とRNA -タンパク質複合体で、例えば、分子間の接触面内のヌクレオチドを識別するために使用することができます。平衡3と4の遷移運動は、solutiの関数としてヒドロキシルラジカルフットプリンティングを行うことによって決定することができます変数または時間で、それぞれ。フットプリントの主な特徴は、プローブにその限られた暴露(例えば、"シングルヒット動力学")ポリマーの各ヌクレオチドの均一サンプリングの結果です。5
このビデオの記事では、我々は、RNAサンプルの調製およびMg(II)を介したフォールディング等温線の測定を説明するために、 テトラヒメナのリボザイムのP4 - P6のドメインを使用してください。我々は、H 2 O 2(我々はこの"過酸化"プロトコルと呼ぶ)と自然に溶存O 2を使用している貴重な、しかし広く知られていない、別のを(我々が呼ぶこの"必要とするよく知られているヒドロキシルラジカルフットプリンティングプロトコルの使用を説明し酸化'プロトコル)。データの削減、変換および分析手順の概要が提示される。
プロトコル
1。フットプリントの試薬の調製
- 100mMのカコジル酸ナトリウム、1mMのEDTA、および1 M KClを含む10 ×反応バッファーを準備します。 7.4にpHを調整する。 0.2μMのアセテートフィルターデバイス(Nalgene)を使用してバッファをフィルタリングする。備考:10 ×バッファに直接ピペットのRNAをしないでください。
- 表1に示すように、各反応の滴定の反応ミックスを調製する。滴定のミックスの音量(所望の濃度で1 ×バッファーとMg(II))が1 ×バッファー中のRNAの10μlを添加する前に、90μlのはずです。
- 6.63M尿素、20mMのクエン酸ナトリウム、1mMのEDTA、0.25μg/μLのtRNAは、0.025%キシレンシアノール、および0.025%ブロモフェノールブルーを含むリボヌクレアーゼT1消化バッファーを準備します。このバッファは、最大6ヶ月までは4℃で保存することができます。
- フェントン反応は250mmのFe(NH 4)2(SO 4)2、250mMのEDTA、500 mMのL -アスコルビン酸ナトリウムの新しく調製した水溶液を必要とします。濃度は、第1章で述べた0.5はそのようなラジカルや多様な二価イオンの濃度を清掃しませんカコジル酸ナトリウムなどのバッファーで平衡実験に最適です。
- 過酸化(1.5.1)および酸化(1.5.2)フット反応の試薬。
- 過酸化フェントン反応は、100 mMのFe - EDTA溶液、及び50mMナトリウムL -アスコルビン酸および1.5%H 2 O 2の新しく調製した水溶液を必要とします。のFe - EDTA溶液は鉄以上のEDTAの1.1倍過剰を持っているEDTAとの混合のFe(NH 4)2(SO 4)2によって調製される。
- ちょうど酸化的開裂反応(3.4.2)を開始する前に、液のFeの2μlの(NH 4)2(SO 4)2(250 mM)を、EDTA 2.2μL(250 mM)で、62.5を組み合わせることにより、フェントン反応ミックスを作成するL -アスコルビン酸ナトリウム(500 mM)で、22.5μlの10 ×反応バッファーと0のフットプリント反応混合物(3.4.2)での最終濃度に到達するH 2 Oの135.5μL。それぞれ10 mMの、0.11 mMの、および6.61 mMの、。
2。フットプリントの実験用RNAの調製
- RNAはDNAテンプレート6または、短い50以上のヌクレオチドであれば、購入できる(統合されたDNA技術で例えば、www.idtdna.com)のin vitro転写規格により製造することができる。フットプリント実験中にRNAのサンプルの整合性を維持するために推奨実験室の清浄度の手順に従ってください。7
- in vitroで転写されたRNAの脱リン酸化8は前のRNAの5'末端の32 P標識に必要です。 RNAの10 pmolのは、末端標識kinasingによるものである。9は、解決策はホスファターゼの成功不活化後曇りになるはず。
- 変性ゲル電気泳動を用いて放射標識RNAを精製する。 RNAは、0.3 M酢酸ナトリウム(pH 5.5)を使用して、続く2つのゲル抽出によって回収される。 RNAは1 mlのエタノールとそれに続くと0.5ミリリットルの水溶液を混合して沈殿させ、ドライアイス上で1分間インキュベーション。 4℃、0.5時間12500 rpmでサンプルをスピン。上清を捨て、70%エタノールでRNAペレットを洗浄。追加の遠心分離後上清を除去し、真空中でペレットを乾燥させます。
- 反応チューブにRNA溶液を、エタノールおよび真空中で乾燥したRNAと沈殿物の1X Buffer.Pipet30μlの330 ULで32 P標識したRNAサンプルを溶かし、リファレンスラダー(ステップ3.5)生成するためにこのチューブを保持。 95℃で加熱することによってバッファリングされたRNA溶液を変性℃で2分間。 15分間室温に試料を冷却し、戻って溶液中にチューブの蓋に凝縮をダウンさせるクイックスピンを与える。
3。フットプリント実験
- 電気泳動の櫛は、分析ゲルあたりのデータポイントの上限数を決定します。我々は30も櫛を使用してください。リファレンスラダーは2井戸と未切断の制御1つを占有。 27反応チューブを準備します。最後のマグネシウム(II)concentratiアドオンは、均等に滴定の中間点の周りに、桁違いにまたがる対数スケールで等間隔する必要があります。
- これとは別に、5分間50℃RNAおよびMg(II)ソリューション℃でインキュベートする。 100μlの最終容量に達するまで1 ×バッファー中のMgの対応する量(II)の90μlのRNA溶液の10μlを混ぜる。 50℃で30分のためのソリューションをインキュベート℃に
- 折りたたみが行われている間ソリューションは、25℃で1時間平衡化してみましょう。
- 過酸化(3.4.1)または酸化(3.4.2)ヒドロキシルラジカルフットプリンティング反応。
- 2μlの鉄- EDTA(100 mm)の液滴を配置することにより、過酸化反応を開始、2μlのL -アスコルビン酸ナトリウム(50mMの)と2μlのH 2 O 2(1.5%)は反応の最内側で互いに分離RNA溶液と激しく混合することによりフットプリント反応を開始するを含むチューブ。 300μlの冷無水エタノールを添加することにより15秒後に反応を停止します。チューブを3-5回回し。沈殿、洗浄しステップ2.4のようにペレットを乾燥させます。
- 作りたてのフェントン反応ミックス(1.6)の5μlを添加して酸化的ヒドロキシルラジカルfootprintingreactionを開始します。 25℃で30分間インキュベートするチューブを3-5回回して反応ミックスをクエンチするために冷無水エタノールを300μlを追加。ステップ2.4で示したように沈殿物は、ペレットを洗浄し、乾燥させます。
- ステップ1.3で調製した溶液を使用して標準的な手順10によればリボヌクレアーゼT1ダイジェストのリファレンスサンプルを生成する。
- 8μlのゲルローディング色素II(Ambion社)と確認RNAでRNAペレットを溶解は、ガイガーカウンターを用いて再懸濁されている。
- 11ロードサンプルつの参照や制御を含む標準的なプロトコールに従って変性、8%ポリアクリルアミドシーケンシングゲルを準備します。 60 Wで別々のRNA断片 - 2.5時間で75 W。一晩貯蔵燐光体スクリーンに乾燥ゲルを公開。たとえば、フィルムレスオートラジオグラフィー用のイメージングシステムと蛍光面をスキャン。ストーム865(GEヘルスケア)や台風(GEヘルスケア)。お使いのコンピュータにゲルのイメージファイルを転送します。
4。データ解析
- 各ヌクレオチドのヒドロキシルラジカル反応性は、分析用ゲル電気泳動で各バンドの強度を定量することにより決定されます。ダウンロード、インストール、およびオープンSAFA、シングルバンドのフィッティングと定量化のためのオープンソースソフトウェアを使いやすい。12、13のユーザマニュアルの指示に従ってください。簡単に言えば、。ゲルのファイルとしてゲルの画像が続きます。txtファイルとしてRNA配列を読み込みます。バンドの強度を調整する、レーンを定義する、アンカーのレーンを選択し、ゲルの位置合わせを行います。ヌクレオチドのバンドの割り当ては、RNase T1の消化ラダーへの参照で発生します。バンドの積分を定量化し、潜在的に不変残基を正規化して割り当てる正規化/ colorplot機能を使用してください。 。txtファイルとして出力を保存します。
- SAFAはでを表すゲル上のレーンと行を表す列を含むスプレッドシートを出力するRNA断片に対応する個々のバンドのバンドの密度をtegrated。最初に、エンドポイントのMg(II)濃度(この例での50 mM)のプロファイルを持つマグネシウム(II)のサンプルがないから派生した保護プロファイルを比較することにより、溶媒露出度の顕著な変化を表示する保護サイトを識別する。値が小さいほど、より多くのヌクレオチドは、ヒドロキシルラジカルおよびその逆からの攻撃に対して、保護された。
- 個々の又はMg(II)濃度対ヌクレオチドのグループの正規化されたバンドの積分から折りたたみ等温線を作成します。等温線は、個別にFI = L +(U - L)による飽和度にスケーリングされます•
 fが分析されているバンド(s)の統合された密度を示す場所、LとUは、移行する下限値と上限値を表し、
fが分析されているバンド(s)の統合された密度を示す場所、LとUは、移行する下限値と上限値を表し、 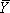 飽和度は14のデータがフィット非線形lを使用しているイーストスクエア解析プログラム(我々は、Origin(OriginLabの)またはグラフパッド(グラフパッドソフトウェア社)のいずれかを使用する。)ヒルの方程式(1)に、
飽和度は14のデータがフィット非線形lを使用しているイーストスクエア解析プログラム(我々は、Origin(OriginLabの)またはグラフパッド(グラフパッドソフトウェア社)のいずれかを使用する。)ヒルの方程式(1)に、 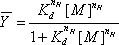
K dが平衡である解離定数、[M]フォールディング反応を媒介する変数、この例のMg(II)の濃度に相当し、n Hはヒル係数である。この手順のスケール飽和度への移行は、その中間点を決定し、遷移がシグモイドであるかどうかの現象学的テストを提供します。 SAFA(図3B)によって分析し、上記のようにフィットされたシーケンシングゲル(図3A)から派生した等温線を図3Cに示されています。図3に示すように、飽和度の等温線は、スプレッドシートのスケーリングによって生成された数式の小数部を使用して最高のフィット上限値と下限値(UとL)に対してSAFAによって生成された値= 値 /(U - L) - L /(U - L)。
5。代表的な結果:
図3は、P4 - P6 RNA•OHフットプリント実験から代表的な結果を示しています。ゲルイメージ(左))、バックグラウンドの切断が最小限であること(右車線を示す)、b)単一ヌクレオチドの割り当てが原因T1車線(各GでのRNase T1を切断)で明確に定義されたバンドに可能であり、c)ヒドロキシルラジカルRNAの誘導の断片化は、よくバックグラウンドを超えています。高Mg(II)へのローからの移行は、個人およびRNA立体構造の形成を示すバンドのグループの統合されたバンドの密度を下げると提携しています。シングルまたはその開裂の変化付随して"保護"と呼ばれる連続したヌクレオチドのグループ。密接にその結晶構造に見られる分子の溶媒にアクセスできない領域に対応するマグネシウム(II)の特徴•OHの保護は、P4 - P6 RNAの折りたたみを媒介。15
折られたRNA分子はC明確な構造や動的な現象と相関されていない第三の接点保護の非常に異なるエクステント(つまり、どのバンドの密度の減少の背景に近い)がある。したがって、いくつかのRNA分子は、図3に示すように、明確に定義された構造の遷移が表示され、一部はしません。バンド強度は、SAFA 12解析(図3B)により定量化し、正規化されています。出力は、•OHに対する保護の相対的な度合いを可視化する"熱"のプロットです。色の変化は、Mg(II)を添加すると、アクセシビリティの変化を説明します。それぞれ、赤または青のショーよりアクセス以上の保護されたヌクレオチドの白。シェーディングの各程度は、保護の曲線(図3C)としてプロットし、ヒルの式(1)のような結合モデルによって分析することができる数値と提携しています。保護153から155に所属する平衡解離定数は約2倍のその保護163から164の対応する値のです。
ontent">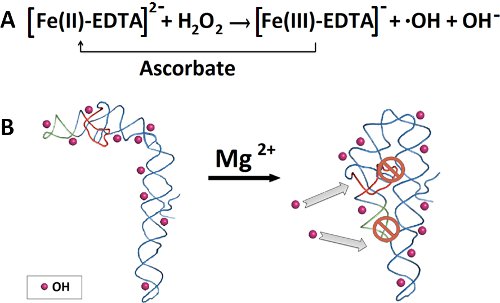
図1。ヒドロキシルラジカル生成とP4 - P6 RNAの折りたたみ。 A)のFe(II)は、過酸化水素からヒドロキシルラジカルの生成を触媒する。アスコルビン酸は第一鉄に鉄(III)バックを低減します。 B)マグネシウム(II)の非存在下で、何P4 - P6三次構造は、ヒドロキシルラジカルは、すべてのアクセス可能なバックボーンの位置を貫通して切断できるように、形成されていない。 Mgの加算(II)は、溶媒のみアクセス可能なバックボーンがヒドロキシルラジカルによって切断できるように、P4 - P6の折り畳みが開始されます。
図2。ヒドロキシルラジカルフットプリンティング実験に概要。 )DephophorylationとRNAの32 P 5'末端ラベリング。 B)変性ポリアクリルアミドゲル上で32 P -ラベルされたRNAの精製。 C)RNAのバンドの切除、その後のRNA抽出、およびエタノール沈殿。 D)PrefoldingとRNAの折りたたみ。 E)新たに調製したフェントン反応混合物の添加は、ヒドロキシルラジカルを生成する。ポリアクリルアミド変性ゲル電気泳動により、F)のRNA断片の分離。 SAFAソフトウェアによるRNA断片のG)定量。
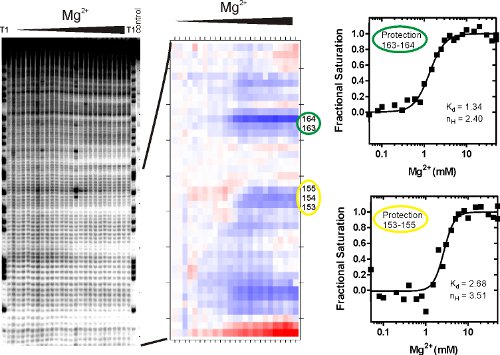
図3。過酸化フェントンフットプリント反応後のRNA断片の解析。 (A)RNAは、ヒドロキシルラジカルにさらされ、切断産物は変性ポリアクリルアミドゲル電気泳動(PAGE)を用いて解決されました。画像は、Mg(II)の濃度の増加と過酸化開裂生成物を示しています。リファレンスとコントロールレーンが表示されています。このイメージファイルは、各バンドの音量をquantitates SAFAプログラムの入力です。 (B)SAFAによって生成される"熱"プロット。 (C)SAFAからヌクレオチド数の出力に統合されたバンドの密度を関連する値のスプレッドシートは、ヒルequatiのような結合モデルによって分析され(1)。保護地域は、Mg(II)濃度の関数としてバンドの積分密度の増加に応じて選ばれた。 K dは、RNAの半分が監視されているサイトで折り畳まれる時にマグネシウム(II)濃度である。ヒル係数、n Hは 、バインディングの共同性に関する情報を提供し、この特定のサイトのフォールディングに関与するマグネシウムイオンの数の下限推定値を提供する曲線の傾きの尺度です。
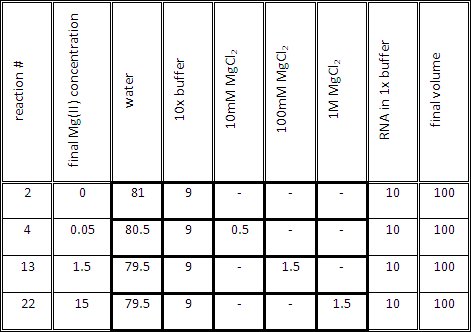
表1。異なるマグネシウム(II)濃度を含むRNAサンプルを生成するための代表的なボリューム。
ディスカッション
ヒドロキシルラジカルフットプリントは、核酸の溶媒接触可能表面積を評価する貴重なツールです。三次構造14の定性的および定量的な形成は、イオンの種類や濃度、pH、温度、結合タンパク質または折りたたみ共同因子としてのパラメータの関数として追跡することができる。まっすぐ進むと安価なプロトコルの強力な組み合わせと単一ヌクレオチドレベルで、その結果、溶媒アク?...
開示事項
利害の衝突は宣言されません。
謝辞
この作品は、健康RO1 - GM085130の国立研究所と国立科学財団MCB0929394からの補助金によって支えられている。私たちは、彼女のもてなしのためにと彼女の研究室でフィルムに私たちを可能にするための博士マリオンシュミットに感謝。
資料
| Name | Company | Catalog Number | Comments |
| Name | Company | Cat# | |
| Sodium Cacodylate(Caution! Toxic) | Sigma-Aldrich | C4945-25g | |
| EDTA (0.5 M) | Ambion | AM9260G | |
| DEPC treated water | Ambion | AM9915G | |
| Sodium Acetate (3 M) | Ambion | AM9740 | |
| MgCl2 (1 M) | Ambion | AM9530G | |
| Urea | Ambion | AM9902 | |
| Sodium Citrate | Sigma-Aldrich | W302600 | |
| tRNA | Sigma-Aldrich | R-7876 | |
| Sodium-L-ascorbate | Sigma-Aldrich | A7631-25g | |
| Fe(NH4)2(SO4)2 . 6 H2O | Sigma-Aldrich | F1543-500g | |
| RNase T1 | Fermentas | EN0541 | |
| Hydrogen Peroxide (30%) | Sigma-Aldrich | 349887 |
参考文献
- Tullius, T. D., Dombroski, B. A. Hydroxyl radical "footprinting": high-resolution information about DNA-protein contacts and application to lambda repressor and Cro protein. Proc. Natl. Acad. Sci. U.S.A. 83, 5469-5473 (1986).
- Celander, D. W., Cech, T. R. Iron(II)-ethylenediaminetetraacetic acid catalyzed cleavage of RNA and DNA oligonucleotides: similar reactivity toward single- and double-stranded forms. Biochemistry. 29, 1355-1361 (1990).
- Celander, D. W., Cech, T. R. Visualizing the higher order folding of a catalytic RNA molecule. Science. 251, 401-407 (1991).
- Sclavi, B., Sullivan, M., Chance, M. R., Brenowitz, M., Woodson, S. A. RNA folding at millisecond intervals by synchrotron hydroxyl radical footprinting. Science. 279, 1940-1943 (1998).
- Tullius, T. D., Dombroski, B. A., Churchill, M., Kam, L. Hydroxyl radical footprinting: a high-resolution method for mapping protein-DNA contacts. Methods. Enzym. 155, 537-558 (1987).
- Milligan, J. F., Groebe, D. R., Witherell, G. W., Uhlenbeck, O. C. Oligoribonucleotide synthesis using T7 RNA polymerase and synthetic DNA templates. Nucleic. Acids. Res. 15, 8783-8798 (1987).
- Schlatterer, J. C., Brenowitz, M. Complementing global measures of RNA folding with local reports of backbone solvent accessibility by time resolved hydroxyl radical footprinting. Methods. 49, 142-147 (2009).
- Sambrook, J., Russell, D. W. . Molecular Cloning: A Laboratory Manual. , (2001).
- Zaug, A. J., Grosshans, C. A., Cech, T. R. Sequence-specific endoribonuclease activity of the Tetrahymena ribozyme: enhanced cleavage of certain oligonucleotide substrates that form mismatched ribozyme-substrate complexes. Biochemistry. 27, 8924-8931 (1988).
- Knapp, G. Enzymatic approaches to probing of RNA secondary and tertiary structure. Methods. Enzym. 180, 192-212 (1989).
- Slatko, B. E., Albright, L. M. Denaturing gel electrophoresis for sequencing. Curr. Protoc. Mol. Biol. Chapter 7, Unit 7.6-Unit 7.6 (2001).
- Das, R., Laederach, A., Pearlman, S. M., Herschlag, D., Altman, R. B. SAFA: semi-automated footprinting analysis software for high-throughput quantification of nucleic acid footprinting experiments. RNA. 11, 344-354 (2005).
- Simmons, K., Martin, J. S., Shcherbakova, I., Laederach, A. Rapid quantification and analysis of kinetic *OH radical footprinting data using SAFA. Methods. Enzym. 468, 47-66 (2009).
- Uchida, T., He, Q., Ralston, C. Y., Brenowitz, M., Chance, M. R. Linkage of monovalent and divalent ion binding in the folding of the P4-P6 domain of the Tetrahymena ribozyme. Biochemistry. 41, 5799-5806 (2002).
- Cate, J. H., Gooding, A. R., Podell, E., Zhou, K., Golden, B. L., Kundrot, C. E., Cech, T. R., Doudna, J. A. Crystal structure of a group I ribozyme domain: principles of RNA packing. Science. 273, 1678-1685 (1996).
- Petri, V., Brenowitz, M. Quantitative nucleic acids footprinting: thermodynamic and kinetic approaches. Curr. Opin. Biotechnol. 8, 36-44 (1997).
- Takamoto, K., Das, R., He, Q., Doniach, S., Brenowitz, M., Herschlag, D., Chance, M. R. Principles of RNA compaction: insights from the equilibrium folding pathway of the P4-P6 RNA domain in monovalent cations. J. Mol. Biol. 343, 1195-1206 (2004).
- Brenowitz, M., Senear, D. F., Shea, M. A., Ackers, G. K. Quantitative DNase footprint titration: a method for studying protein-DNA interactions. Methods. Enzym. 130, 132-181 (1986).
- Shcherbakova, I., Mitra, S. Hydroxyl-radical footprinting to probe equilibrium changes in RNA tertiary structure. Methods. Enzym. 468, 31-46 (2009).
- Howlett, G. J., Minton, A. P., Rivas, G. Analytical ultracentrifugation for the study of protein association and assembly. Curr. Opinion. Chem. Biol. 10, 430-436 (2006).
- McGinnis, J. L., Duncan, C. D., Weeks, K. M. High-throughput SHAPE and hydroxyl radical analysis of RNA structure and ribonucleoprotein assembly. Methods. Enzym. 468, 67-89 (2009).
- Jonikas, M. A., Radmer, R. J., Laederach, A., Das, R., Pearlman, S., Herschlag, D., Altman, R. B. Coarse-grained modeling of large RNA molecules with knowledge-based potentials and structural filters. RNA. 15, 189-199 (2009).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved