需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
哺乳动物链接组蛋白亚型表达分析
摘要
我们描述了一个集检测,分析H1链接组蛋白表达水平。个人H1基因的mRNA定量测定基于随机引物反转录实时PCR其次,而量化的H1组蛋白的蛋白质的高效液相色谱分析实现。
摘要
链接的组蛋白H1结合核小体核心颗粒和DNA连接,促进染色质折叠成较高阶结构。 H1是必不可少的哺乳动物发展1 2-4 体内特定基因的表达调控。之间高度保守的组蛋白H1的连接器组蛋白家族是最异质的群体。在哺乳动物中有11个的H1亚型的差异在发展过程中和在不同类型的细胞调节。这些H1的亚型,包括5躯体H1S(H1A-E),0替换H1,4生殖细胞特定的H1亚型,5 H1x。多个H1亚型在不同的DNA结合的亲和力和染色质压实能力6-9的存在提供了一个额外的染色质功能的调制水平。因此,定量个别H1亚型的mRNA和蛋白质表达分析,为更好地理解更高的监管是必要的为了染色质结构和功能。
在这里,我们描述了一组为分析个别H1亚型的表达水平( 图1),设计实验。各种H1基因变异的基因表达是衡量一套高度敏感和定量逆转录聚合酶链反应(QRT-PCR)检测,这是更快,更准确,更需要少得多的样品与Northern blot分析的替代方法相比。不同于其他大多数细胞mRNA的消息,最组蛋白的基因,包括广大H1基因,基因缺乏长期的polyA的尾巴,但包含茎环结构,在3'非翻译区(UTR)10。因此,cDNA的准备从总RNA反转录使用的寡-dT引物随机引物。每个H1亚型( 见表1)的特异性引物实时荧光PCR检测以获得个别H1亚型的mRNA水平的定量测量第看家基因表达分析作为正常化的控制。
每个H1亚型和核心组蛋白的蛋白质的相对丰度获得通过反相高效液相色谱法(RP-HPLC法)11-13哺乳动物细胞中提取的总组蛋白的分析。高效液相色谱法和洗脱条件,描述了在这里给鼠标的H1亚型的最佳分离。我们通过量化高效液相色谱法剖面,计算个别H1亚型H1的家庭内的相对比例,以及确定在细胞的核小体比H1。
研究方案
1。样品制备和RNA提取
- RNA提取之前,所有工作表面和移液器应该被消灭,用70%乙醇和核糖核酸酶去污解决方案,如核糖核酸酶摧毁,治疗。这种做法降低了RNase污染和RNA降解的机会。戴手套的所有程序。
- 小鼠组织中提取的RNA,从安乐死鼠标利益的解剖器官,组织洗冰冷的磷酸盐缓冲液(PBS:0.13 M氯化钠,5毫米磷酸钠二元亚铁,5毫米磷酸二氢钠亚铁,pH值7.4) 。立即着手中的RNA提取步骤1.4。如果不新鲜组织RNA提取处理,组织样本应该立即在液氮冷冻的单元,并在-80°C保存供以后使用。
- 如果RNA是从贴壁细胞培养提取,吸出培养基,用足量的PBS冲洗,加入Trizol试剂(的体外根到板)和继续加强1.4。对于悬浮细胞生长,收获离心细胞和颗粒细胞。丢弃的媒体,用PBS冲洗简要颗粒,颗粒与离心的细胞。加入Trizol试剂,并继续执行步骤1.4。
- 足够的Trizol试剂获得高质量的RNA是必要的。使用1毫升Trizol试剂提取RNA从50 - 100毫克组织,5 - 10×10 6细胞(悬浮培养)或每3.5厘米的钢板(黏附文化)。同质化的组织与宝创PT2100均质(或同等学历)Trizol试剂。继续从组织样品或细胞中提取RNA根据制造商的手册Trizol试剂(Invitrogen公司)。
- RNA浓度测量使用的NanoDrop 1000(Thermo Scientific的)和凝胶电泳分析RNA的质量。典型的产量范围从1-10微克每毫克或每1×10 6细胞培养的组织5-15微克的RNA的RNA。为了消除从微量基因组DNA,RNA的潜在污染的RNA样品被视为无RNA酶的DNA酶(Sigma公司,AMP-D1)根据制造商的指示。重复RNA浓度测定和凝胶电泳,以确保没有从这种治疗的RNA降解。商店提取的RNA在-80°C。
*注意:使用RNAeasy试剂盒(Qiagen)根据试剂盒说明书也可提取的RNA,或DNA / RNA的试剂盒(Qiagen),如果需要的DNA和RNA。
2。定量逆转录聚合酶链反应(QRT-PCR)
- 总RNA反转录成cDNA的使用上标III第一链合成系统(Invitrogen公司)。由于大多数H1基因的mRNA缺少了聚-A尾巴,它是使用cDNA合成引物随机引物 ,而不是寡-dT的关键。然而,如果也希望在低层次的加尾消息的基因表达分析,混合随机引物和寡-dT寿LD在反转录(RT)的反应,以改善加尾的mRNA逆转录效率。
- 根据制造商的手册执行RT反应。简单地说,
- 在0.5毫升PCR管,5微克总RNA,1μL50 ng /μl的随机引物,1μl10 mM的dNTP混合液的结合起来,并加入DEPC处理过的H 2 O,使反应总体积为10μL。拌匀,静置5分钟,在65°C,1分钟冰孵化。
- 准备10μLcDNA合成组合:2的μl缓冲10xRT,4μL25毫米的MgCl 2的数码地面0.1M,1μl的RNaseOut的(40 U /μL),1μL标三RT(200 U / L,2μL μl)和它添加到RNA /引物混合物。
- 孵育10分钟,在25℃,其次是50°C时50分钟,在85°C 5分钟终止反应。
- 通常每个反应产生100-250纳克/微升cDNA产物。储存在-20 cDNA产物°C或立即进行实时定量PCR(qPCR)。
- 定量PCR可以准确量化目标序列与高效率和可重复性14份。我们选择的qPCR测量用SYBR Green染料,给出了一个荧光信号,只有当它与双链DNA(dsDNA)的intercalates。这种方法虽然不如TaqMan方法14具体,更具成本效益,更容易在实验室中通过,并提供了更多的多功能定量PCR。因此,它是重要的检查,以确保反应效率和特异性扩增曲线( 图2A)及衍生的qPCR产品熔解曲线( 图2B)。
| 组蛋白亚型 | 小鼠组蛋白命名 | 人类组蛋白命名 | ||
| 戈NE名称 | 加入没有。 | 基因名称 | 加入没有。 | |
| 组蛋白H1A | hist1h1a | NM_030609 | HIST1H1A(H1.1) | NM_005325 |
| 组蛋白H1B | hist1h1b | NM_020034 | HIST1H1B(H1.5) | NM_005322 |
| 组蛋白H1C | hist1h1c | NM_015786 | HIST1H1C(H1.2) | NM_005319 |
| 组蛋白H1d | hist1h1d | NM_145713 | HIST1H1D(H1.3) | NM_005320 |
| 组蛋白H1E | hist1h1e | NM_015787 | HIST1H1E(H1.4) | NM_005321 |
| 组蛋白H1° | h1f0 | NM_008197 | H1F0 | NM_005318 |
| 组蛋白H1oo | H1FOO | NM_183811 | H1FOO | NM_153833 |
| 组蛋白H1t | hist1h1t | NM_010377 | HIST1H1T | NM_005323 |
| 组蛋白H1t2 | h1fnt | NM_027304 | H1FNT | NM_181788 |
| 组蛋白H1x | h1fx | NM_198622 | H1FX | NM_006026 |
| 组蛋白Hils1 | hils1 | NM_081792 | HILS1 | AY286318 |
表1。在老鼠和人类的组蛋白H1亚型命名。
- 设计正向和反向PCR引物,具体为每个H1基因( 表1)。由于序列相似性高,特别是在该地区相应的中央球状结构域之间的躯体H1S,关键是要确保不对齐与邻为一个特定的H1亚型的引物设计,进一步H1基因,或交叉扩增其他的H1亚型。同样重要的是要注意,大多数H1基因不含有内含子。因此内含子,跨越通常采用RT-PCR技术,以避免基因污染引物都没有。相反,RNA样品应预先用DNase处理(见1.5),以消除任何微量的基因污染。此外,放疗( - )-qPCR的应平行进行验证缺乏的cDNA样品中的基因污染。
- 也为内部参考基因,其表达之间不改变样品设计引物。看家基因,如3 - 磷酸甘油醛脱氢酶(GAPDH)和β-肌动蛋白基因,往往选择作为参考基因。看家基因的qPCR信号作为正常化的控制。
- 准备每个PCR反应如下(总体积25μL):12.5μl2X IQ SYBR Green超(Bio-Rad公司)(含dNTPs浓度,50 U / ml的iTaq DNA聚合酶,6毫米MgCl 2的 SYBR Green I的20的纳米荧光素),4 ng /μl的基因,1.25微升10纳米的正向/反向引物混合,2μL和9.25 DDH 2μL,混合,在96孔PCR板Microseal。使用Microseal“B”(BIO-RAD)的粘接密封,以确保板盖密封板。点选或简要涡PCR板,旋转下降了一个简短的离心反应混合物。放置在盘MyiQ™实时单色实时PCR检测系统的qPCR(BIO-RAD)。
- 我们用30秒以下的qPCR条件:95℃3分钟10秒,由40个周期为95°C,60°C,20秒,72°C间。检查PCR的效率和Ct(循环阈值)值的扩增曲线( 图2A)。 IQ5光学系统软件2.0版,可以自动设置的门槛线。
- 引物的效率和最佳的cDNA浓度可以测试由标准曲线法,在我的基因组DNA序列稀释采用S qPCR和Ct值绘制对模板DNA量的记录。一个优化的qPCR实验将与引物的特异性和高效率的线性标准曲线,测定系数(R 2)> 0.98。避免引物扩增长度超过200个基点,其中往往有贫困的扩增效率。
- 因为的SYBR Green检测任何双链DNA,这是关键的qPCR,以确保所需的扩增,但没有引物二聚体或污染物,扩增和检测后进行熔解曲线运行。为熔解曲线分析,方案的qPCR仪器升温从55°C至95°C在0.5°C增量数据采集样品。熔解曲线分析MyiQ™实时(BIO-RAD)的默认设置的仪器如下:95℃1分钟,55°C下1分钟,周期为10秒81设定55°C间,融化曲线, +温度0.5°C间(相机在每个周期内收集的数据)。
- 自熔融温度(TM)是双链DNA扩增长度和GC含量的依赖,不同的扩增将有不同的TM(S)。检查的qPCR产品衍生的熔解曲线,以确定所需的扩增特定的熔融温度,以及缺乏噪声扩增峰(图2B)。
- 准备进行统计分析,每个实验的反应式两份或三份。包括QRT-PCR如RT阴性对照,( - )定量PCR(定量PCR与RNA为模板,没有逆转录)和没有基因,RNA或DNA来源的PCR混合物中添加的qPCR。在图2和3的潜在基因组DNA污染(逆转录( - )),放疗( - )-qPCR的可以作为控制。
- qPCR的数据分析与IQ5光学系统软件2.0版(BIO-RAD)。正常化的看家基因(例如GAPDH的,β-肌动蛋白,HPRT基因)的表达,以获得H1基因的相对表达水平的H1亚型基因的表达值。
*所有程序应在冰上或4°C
- 解剖小鼠组织和冰冷的PBS冲洗。 (如果不立即进行萃取,捕捉冻结并存储在步骤1.2中所述的样品。)组织切碎成块刀片。转让百果到Dounce匀浆(二杵)。添加10毫升蔗糖缓冲液(0.3 M蔗糖,氯化钠15毫米,10毫米肝素钠[pH值7.9,2毫米EDTA,PMSF 0.5毫米,完整的迷你蛋白酶抑制剂平板,补充新鲜)每克组织。 10-15杆,均质组织。
- 匀浆转移至15毫升管为30秒(离心模型:Eppendorf公司5810R),在500 rpm的旋转;小心将上清转移到一个新的管(丢弃颗粒组织碎片),并在2000转离心5分钟,沉淀细胞。继续加强3.4。
- 如果组蛋白和染色质是由单层细胞生长的提取,冲洗用PBS,添加的PBS的培养皿中,并收获用离心刮板细胞和颗粒细胞的细胞。对于悬浮液中培养的细胞,颗粒细胞,离心。
- 细胞沉淀悬浮在10毫升蔗糖辅以0.5%NP-40(每克开始组织的金额或10(8)细胞)的缓冲区。样品转移到Dounce匀浆(二杵),并在20分钟内孵化dounce 10招。在这一点上,原子核获得。在显微镜下研究原子核的质量。颗粒原子核在2000转离心5分钟。弃上清。
- 悬浮在3毫升高盐缓冲液(0.35米氯化钾,10毫米的Tris [pH值7.2,5毫米MgCl 2的 0.5毫米PMSF -每次使用前添加新鲜)组织每1克或10(8)细胞的细胞核沉淀。 Dounce均质小(二杵)转移样品均质5-10招。
- 分装到3个Eppendorf管在冰上孵育20分钟(每1毫升),其次是暂停离心10分钟,染色质颗粒14,000 RPM。弃上清。
- 添加0.8毫升0.2 NH 2 SO 4计到每一个染色质颗粒。使用Eppendorf管的杵dounce研磨颗粒,直到颗粒完全分离。在4°C过夜的旋转平台上孵育样品。总组蛋白提取这一步酸处理。
- 在14000转离心10分钟。上清(组蛋白提取物)转移到Eppendorf管(400μL/管)。弃沉淀。新增2.5冰冷乙醇的体积(1毫升)每管......保持样本在-20°C过夜。
- 在14000转离心10分钟总组蛋白沉淀,弃上清。用70%乙醇洗涤沉淀三次,离开板凳上,风干20-30分钟。储存在-80°C干燥的蛋白质或溶于DDH 2他们,并立即进行高效液相色谱分析。干蛋白可以存储在-80℃长达1年。
4。链接组蛋白的高效液相色谱分析
- 组蛋白颗粒悬浮反相柱和高效液相色谱仪的能力而定,建议在DDH 2量。我们使用C18反相柱250×4.6毫米(Vydac)Äktapurifier刚果爱国者联盟900仪器(GE医疗),高效液相色谱分析。我们通常总组蛋白溶解于100μL进行分析DDH 2 0 50-100μg。
- 在5分钟14000转,离心除去不溶性残渣。 Bradford蛋白检测用于确定蛋白质的数量被注入到列。 50-100μg总蛋白注入反相柱高效液相色谱系统。载入中蛋白质过量,应避免防止列堵塞。
- 链接增加乙腈梯度的核心组蛋白和组蛋白的分馏, 表2中列出。
表2。乙腈梯度,随着时间的推移增加。
- 在214 nm处的污水进行监测,高效液相色谱型材(Figu重4)使用Äktapurifier刚果爱国者联盟900(GE医疗)与麒麟5.11软件(GE医疗)进行记录和分析。蛋白质组分,也可以收集分数自动收集器(的压裂-920 - GE公司)作进一步的分析,如SDS-PAGE和质谱。
- H1亚型和H2B各峰的一个214值正常化每个相应的组蛋白肽键的数目。个别的H1组蛋白亚型的H1系列内,以及核小体核心颗粒的H1亚型的比例相对比例,可以计算出从这些归一214值(图5)。
5。代表结果
哺乳动物H1亚型,整体流程图和代表个别组蛋白H1基因的表达分析结果列表于表1, 图1和图2-5所示,分别。图2a显示了典型的扩增曲线H1A使用的qPCR反应基因从小鼠肝和mESCs准备,而图2b显示了相应的扩增衍生熔解曲线。熔解曲线显示在熔融温度(Tm)的特征峰在86°为H1A PCR扩增,缺乏非特定的背景峰,表明H1A定量PCR检测的高特异性。扩增曲线( 图2A)的评价结果显示,每个样品一式三份的qPCR反应了几乎相同的Ct值相一致的信号,表明高重复性。缺乏建立实时扩增( - )-qPCR的反应表明,基因组DNA的污染是不存在,或很少。 H1基因和看家基因,如GAPDH,利用Ct值,每个H1基因的RNA相对表达水平进行了计算。 H1°和H1A基因计算结果的例子所示图3。 H1A mRNA的相对表达水平较高,在mESCs与小鼠肝,而H1°的表达远远高于在肝脏中mESCs。
H1A或H1度在MESC与成人小鼠肝脏的表达差异也是显而易见的,从组蛋白的高效液相色谱法简介( 图4)。分化的具体H1,H1°,在成熟组织的大量积累,占27.2%的成人肝脏中的总H1( 图5A)。相比之下,H1°蛋白质几乎是在未分化的mESCs缺席( 图4B)。 H1A另一方面,是高表达,无论是在mRNA转录和蛋白质,在mESCs( 图3和图4B)。通过高效液相色谱法剖面H1峰的量化,H1的家族内的每个个体的H1亚型的相对比例确定( 图5A)。此外,个别H1亚型值(或总H1每个核小体),可以计算出归一214相应的H1亚型(或总H1的总和)的峰值比一半归214 H2B的一个值( 图5B)。
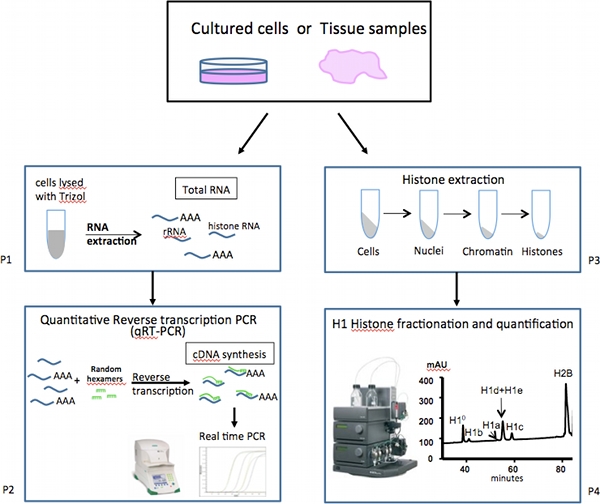
图1。哺乳动物链接组蛋白亚型表达分析的总体方案。
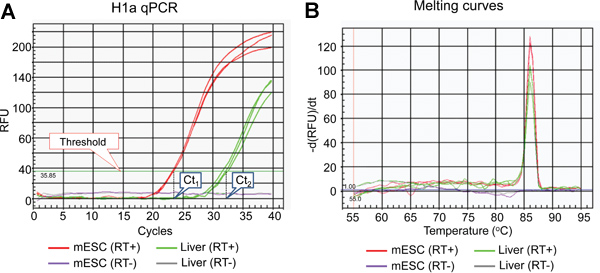
图2。代表H1A定量PCR检测结果。 (一)H1A定量PCR法扩增曲线。门槛线和设置IQ5光学系统软件的Ct值表示。 (二)衍生融化的qPCR产品曲线在(A)所示。
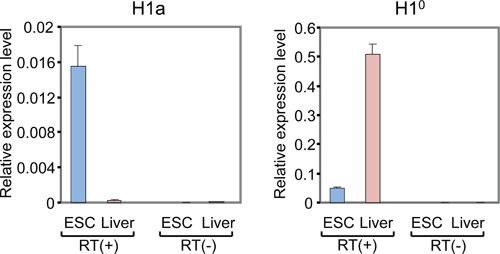
图3。H1A和H1°在mESCs和成人米的mRNA水平的定量RT-PCR分析乌斯肝脏。 Y轴代表H1基因的相对表达水平的参照基因GAPDH。 ( - )用RT qPCR的样品(无逆转录RNA)显示很少或没有信号。
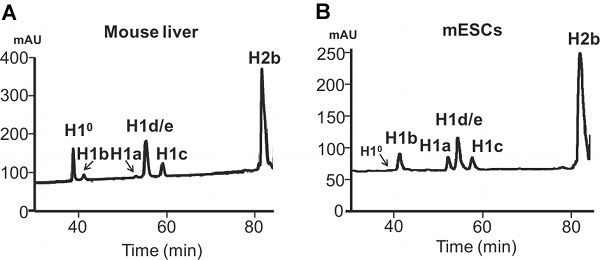
图4。从哺乳动物细胞中提取的组蛋白的高效液相色谱分析。反相高效液相色谱分析,从成年小鼠肝(A)和小鼠胚胎干细胞(二)提取100μg总组蛋白。 X轴:洗脱时间。 Y轴:MAU,毫吸水性单位。
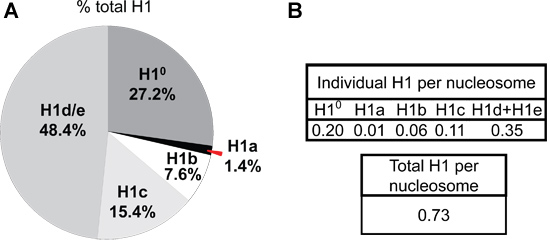
图5。H1亚型组成,每核小体的比率在成年小鼠肝H1。峰面积为每个H1亚型和H2B一个214值计算使用麒麟5.11软件(GE医疗),以及相应的组蛋白中的肽键数量的正常化。归一的总和214所有的H1亚型的值,得到作为总H1的值。每个H1亚型百分比总H1(一),以及H1的比例,核小体(一半归H2B的一个214值表示)(B)在成年小鼠肝从HPLC的文件显示计算在图4A。
讨论
这里提出了一套检测启用链接哺乳动物组蛋白亚型表达水平的综合分析。正确设计的QRT-PCR检测提供从任何哺乳动物组蛋白H1基因的高度敏感和精确的测量RNA的消息。链接组蛋白亚型基因的实时定量PCR检测的重要组成部分,是使用随机引物反转录的cDNA的准备。 mRNA的最组蛋白的基因,包括大多数H1基因,不包含长聚,一个在其他细胞基因的尾巴。因此,传统与寡-dT引物逆转录方法不能有效地将生产?...
披露声明
没有利益冲突的声明。
致谢
支持这项工作是由美国国立卫生研究院授予GM085261和佐治亚癌症联盟杰出学者奖“(银发)。
材料
| Name | Company | Catalog Number | Comments |
| 试剂名称 | 公司 | 目录编号 | |
| 核糖核酸酶摧毁 | 应用生物系统公司 | AM9780 | |
| Trizol试剂 | Invitrogen公司 | 15596-018 | |
| SuperScriptIII | Invitrogen公司 | 18080-051 | |
| 无水乙醇 | Fisher Scientific则 | BP2818-4 | |
| IQ的SYBR Green | BIO-RAD | 170-8880 | |
| 的RNeasy迷你套件 | Qiagen公司 | 74104 | |
| 脱氧核糖核酸我 | 西格玛 | ,AMP-D1 | |
| microseal 96孔PCR板 | BIO-RAD | MSP-9605 | |
| microseal“B”粘接密封 | BIO-RAD | MSB-1001 | |
| 蔗糖 | agros有机物 | AC40594 | |
| 磷酸氢钠亚铁(的Na 2 HPO 4·7H 2) | Fisher Scientific则 | BP332 | |
| 氯化钠(NaCl) | 美国生物分析 | AB01915 | |
| 磷酸二氢钠亚铁(的NaH 2 PO 4·7H2O) | Fisher Scientific则 | BP-330 | |
| 肝素钠 | Fisher Scientific则 | BP310 | |
| 完整的迷你蛋白酶抑制剂鸡尾酒片 | 罗氏应用科学部 | 11836153001 | |
| EDTA的 | 西格玛 | E - 5134 | |
| phenylmethanesulfonyl氟(PMSF) | 美国生物分析 | AB01620 | |
| nonidet-40(NP-40) | 美国生物分析 | AB01425 | |
| 氯化钾(KCl) | Fisher Scientific则 | BP366 | |
| 三羟甲基氨基甲烷] | 美国生物分析 | AB02000 | |
| 氯化镁(MgCl2) | Fisher Scientific则 | BP214 | |
| 硫酸(H2SO4) | 厂商VWR | VW3648-3 | |
| 氢氧化铵( 氨水 ) | agros有机物 | AC42330 | |
| 。布拉德福德蛋白质分析 | BIO-RAD | 500-0001 | |
| 乙腈 | 电解二氧化锰 | AX0145-1 | |
| 三氟乙酸(TFA) | JTBaker | 9470-01 |
参考文献
- Fan, Y. H1 linker histones are essential for mouse development and affect nucleosome spacing in vivo. Mol. Cell Biol. 23, 4559-4572 (2003).
- Woodcock, C. L., Skoultchi, A. I., Fan, Y. Role of linker histone in chromatin structure and function: H1 stoichiometry and nucleosome repeat length. Chromosome Res. 14, 17-25 (2006).
- Shen, X., Gorovsky, M. A. Linker histone H1 regulates specific gene expression but not global transcription in vivo. Cell. 86, 475-483 (1996).
- Alami, R. Mammalian linker-histone subtypes differentially affect gene expression in vivo. Proc. Natl. Acad. Sci. U.S.A. 100, 5920-5925 (2003).
- Happel, N., Doenecke, D. Histone H1 and its isoforms: contribution to chromatin structure and function. Gene. 431, 1-12 (2009).
- Clausell, J., Happel, N., Hale, T. K., Doenecke, D., Beato, M. Histone H1 subtypes differentially modulate chromatin condensation without preventing ATP-dependent remodeling by SWI/SNF or NURF. PLoS One. 4, 0007243-0007243 (2009).
- Khadake, J. R., Rao, M. R. DNA- chromatin-condensing properties of rat testes H1a and H1t compared to those of rat liver H1bdec; H1t is a poor condenser of chromatin. Biochemistry. 34, 15792-15801 (1995).
- Orrego, M. Differential affinity of mammalian histone H1 somatic subtypes for DNA and chromatin. BMC Biol. 5, 22-22 (2007).
- Th'ng, J. P., Sung, R., Ye, M., Hendzel, M. J. H1 family histones in the nucleus. Control of binding and localization by the C-terminal domain. J. Biol. Chem. 280, 27809-27814 (2005).
- Wang, Z. F., Sirotkin, A. M., Buchold, G. M., Skoultchi, A. I., Marzluff, W. F. The mouse histone H1 genes: gene organization and differential regulation. J. Mol. Biol. 271, 124-138 (1997).
- Brown, D. T., Sittman, D. B. Identification through overexpression and tagging of the variant type of the mouse H1e and H1c genes. J. Biol. Chem. 268, 713-718 (1993).
- Fan, Y., Sirotkin, A., Russell, R. G., Ayala, J., Skoultchi, A. I. Individual somatic H1 subtypes are dispensable for mouse development even in mice lacking the H1(0) replacement subtype. Mol. Cell. Biol. 21, 7933-7943 (2001).
- Fan, Y., Skoultchi, A. I. Genetic analysis of H1 linker histone subtypes and their functions in mice. Methods Enzymol. 377, 85-107 (2004).
- Heid, C. A., Stevens, J., Livak, K. J., Williams, P. M. Real time quantitative PCR. Genome Res. 6, 986-994 (1996).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。