Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Expressionsanalyse von Säugetier-Linker-Histon-Subtypen
In diesem Artikel
Zusammenfassung
Wir beschreiben eine Reihe von Tests zur Expression von H1 Linker Histonen zu analysieren. mRNA einzelner Gene H1 werden quantitativ durch zufällige Primer basieren reverse Transkription durch real-time PCR gefolgt gemessen, während Protein Quantifizierung der H1-Histone durch HPLC-Analyse erreicht wird.
Zusammenfassung
Histon H1 bindet an den Nukleosomen Kernteilchen und Linker-DNA, die Erleichterung Faltung von Chromatin in Struktur höherer Ordnung. H1 ist für Säugetier-Entwicklung ein wesentlicher und reguliert die Genexpression in vivo 2-4. Unter den hoch konservierte Histon-Proteine ist die Familie von H1 Linker Histonen die heterogene Gruppe. Es gibt 11 H1-Subtypen bei Säugetieren, die differentiell während der Entwicklung und in verschiedenen Zelltypen geregelt sind. Diese H1 Subtypen umfassen 5 somatischen H1s (H1a-e), der Ersatz H1 0, 4 Keimzellen spezifische H1 Subtypen und H1x 5. Das Vorhandensein von mehreren H1 Subtypen, die DNA-Bindungsaffinität und Chromatin Verdichtung Fähigkeit unterscheiden 6-9 bietet eine zusätzliche Modulation der Chromatinstruktur Funktion. Somit ist die quantitative Analyse der Expression einzelnen H1 Subtypen, sowohl von mRNA und Proteinen, zum besseren Verständnis der Regulation der höheren erforderlichenchromosomaler Struktur und Funktion.
Hier beschreiben wir eine Reihe von Tests zur Analyse der Expression von einzelnen H1 Subtypen (1) ausgebildet ist. mRNA-Expression von verschiedenen Genen H1 Variante wird durch eine Reihe von hochempfindlichen und quantitative RT-PCR (qRT-PCR)-Assays, die schneller, genauer und erfordern viel weniger Proben im Vergleich zu der alternativen Ansatz der Northern-Blot-Analyse gemessen. Anders als die meisten anderen zellulären mRNA-Einträge, fehlt mRNAs für die meisten Histone, einschließlich der Mehrheit der Gene H1, eine lange polyA-Schwanz, aber auch eine Stamm-Schleifen-Struktur am 3'-untranslatierten Region (UTR) 10. Daher werden cDNAs aus Gesamt-RNA durch reverse Transkription unter Verwendung von Zufallsprimern statt Oligo-dT-Primer hergestellt. Echtzeit PCR mit Primern, die an jedem H1 Subtypen (Tabelle 1) werden durchgeführt, um eine möglichst quantitative Messung der mRNA-Spiegel von einzelnen H1-Subtyp zu erhaltens. Die Expression von Housekeeping-Gene werden als Steuerelemente für Normalisierung analysiert.
Die relative Häufigkeit der Proteine des H1-Subtyp und Histone wird durch Umkehrphasen-Hochleistungs-Flüssigchromatographie (RP-HPLC)-Analyse von Gesamt Histone aus Säugerzellen 11-13 extrahiert wurde. Die HPLC-Methode und Elution hier beschriebenen Bedingungen geben optimale Trennungen von Maus H1-Subtypen. Durch die Quantifizierung der HPLC-Profil, berechnen wir den relativen Anteil der einzelnen Subtypen H1 H1 innerhalb der Familie, sowie Bestimmung der H1 um Nukleosomen-Verhältnis in den Zellen.
Protokoll
1. Probenvorbereitung und RNA-Extraktion
- Bevor die RNA-Extraktion, sollten alle Arbeitsflächen und Pipetten mit 70% Ethanol abgewischt werden und mit RNase Dekontaminationslösung, wie RNase Zap. Diese Praxis verringert die Chancen der RNase-Kontamination und RNA-Abbau. Tragen Sie Handschuhe für alle Verfahren.
- Um RNA aus Maus-Gewebe zu extrahieren, zu sezieren die Orgel von Interesse aus eingeschläfert Maus, und waschen Sie das Gewebe in eiskaltem Phosphat-gepufferte Kochsalzlösung (PBS: 0,13 M NaCl, 5 mM Natriumphosphat dibasisches Heptahydrat, 5 mM Natriumdihydrogenphosphat Heptahydrat, pH 7,4) . Fahren Sie direkt mit Extraktion in Schritt 1,4 RNA. Wenn frische Gewebe ist nicht für die RNA-Extraktion verarbeitet werden, sollten die Gewebeproben in flüssigem Stickstoff eingefroren und sofort bei -80 ° C für eine spätere Verwendung gespeichert werden.
- Wenn RNA aus adhärenten Zellkultur gewonnen werden, ansaugen Kulturmedien, Spülen mit einer ausreichenden Menge PBS, und geben Trizol-Reagenz (Invitrogen) auf die Platte und fahren Sie mit Schritt 1,4. Für Zellen in Suspension wachsen, ernten die Zellen-und Pellet-Zellen durch Zentrifugation. Entsorgen Sie Medien, spülen Sie die Pellets kurz mit PBS und die Zellen mit Pellet Zentrifugation. Fügen Sie Trizolreagenz und fahren Sie mit Schritt 1,4.
- Ausreichende Trizolreagenz ist notwendig für den Erhalt von qualitativ hochwertigen RNA. Verwenden Sie 1 ml Trizol-Reagenz zu extrahieren RNA von 50 - 100 mg Gewebe, 5 bis 10 x 10 6 Zellen (für Suspensionskulturen) oder 3,5 cm pro Platte (für adhärente Kulturen). Homogenisieren des Gewebes in Trizolreagenz mit Polytron PT2100 Homogenisator (oder gleichwertig). Fahren Sie mit RNA aus Gewebeproben oder Zellen extrahieren nach der Anleitung des Herstellers für Trizol Reagenz (Invitrogen).
- RNA-Konzentration wird unter Verwendung Nanodrop 1000 (Thermo Scientific) und RNA-Qualität wird durch Gelelektrophorese analysiert. Die typische Ausbeute im Bereich von 1-10 ug RNA pro mg Gewebe oder 5-15 ug RNA pro 1 x 10 6 kultivierten Zellen. Um das zu eliminiereneine mögliche Kontamination von RNA aus Spurenmenge von genomischer DNA, RNA-Proben mit RNase-freier DNase (Sigma AMP-D1) gemäß den Anweisungen des Herstellers behandelt. Wiederholen Sie die RNA-Konzentration und Entschlossenheit Gelelektrophorese auf keinen Abbau von RNA aus dieser Behandlung zu gewährleisten. Shop extrahierte RNA bei -80 ° C
* Anmerkung: Die RNA kann auch extrahiert unter Verwendung RNAeasy-Kits (Qiagen) nach dem Baukastenprinzip manuelle oder durch DNA / RNA-Kit (Qiagen), wenn sowohl DNA als auch RNA erwünscht sind.
2. Quantitative Reverse Transkription PCR (qRT-PCR)
- Gesamt-RNA wird revers in cDNA unter Verwendung von Superscript III Erststrangsynthese (Invitrogen) transkribiert. Da die meisten der mRNA H1 Gene Poly-A-Schwanz fehlt, ist es wichtig, zufälligen Hexameren statt Oligo-dT als Primer für die cDNA-Synthese eingesetzt werden. Wenn jedoch die Expression von Genen mit polyadenylierte Nachrichten auf einem niedrigen Niveau ebenfalls erwünscht ist, eine Mischung aus zufälligen Hexameren und Oligo-dT should in die reverse Transkription (RT)-Reaktion, um die Effizienz der reversen Transkription polyadenylierte mRNA verbessert werden.
- Führen Sie die RT-Reaktion nach der Anleitung des Herstellers. Kurz gesagt,
- In einem 0,5 ml PCR-Röhrchen, kombinieren 5 ug Gesamt-RNA, 1 ul 50 ng / ul zufällige Hexamere, 1 ul 10 mM dNTP-Mix, und fügen Sie DEPC-behandeltem H 2 O, um die gesamte Reaktionsvolumen als 10 pl machen. Gut mischen und Inkubation für 5 Minuten bei 65 ° C, um 1 Minute Inkubation auf Eis folgten.
- Bereiten 10 ul cDNA Synthesis Mix: 2 ul 10xRT Puffer, 4 ul 25 mM MgCl 2, 2 ul 0,1 M DTT, 1 ul RNaseOut (40 U / ul), 1 ul SuperScript III RT (200 U / ul) und fügen Sie sie / RNA-Primer-Mischung.
- Inkubieren für 10 Minuten bei 25 ° C, gefolgt von 50 Minuten bei 50 ° C, gefolgt und Abbruch der Reaktion bei 85 ° C für 5 Minuten.
- Jede Reaktion liefert typischerweise 100 bis 250 ng / ul cDNA-Produkt. Bewahren cDNA-Produkte bei -20° C oder gehen Sie sofort für quantitative Echtzeit-PCR (qPCR).
- qPCR können genau quantifizieren die Zielsequenz Kopien mit hoher Effizienz und Reproduzierbarkeit 14. Wir wählen qPCR von SYBR Green Farbstoff, der ein Fluoreszenz-Signal nur dann, wenn es mit Doppelstrang-DNA (dsDNA) interkaliert gibt gemessen. Obwohl nicht so spezifisch wie TaqMan-Assay 14, ist dieses Verfahren kostengünstiger, leichter, im Labor erlassen werden, und gibt mehr Flexibilität zu qPCR. Daher ist es wichtig, die Amplifikationsplot (Abbildung 2A) und die Ableitung der Schmelzkurven qPCR Produkt (2B) zu untersuchen, um eine Reaktion Effizienz und Spezifität zu gewährleisten.
| Histon-Subtypen | Maus-Histon-Nomenklatur | Menschliche Histon-Nomenklatur | ||
| Gene Namen | Zugangs-Nr. | Gen-Namen | Zugangs-Nr. | |
| Histone H1a | Hist1h1a | NM_030609 | HIST1H1A (H1.1) | NM_005325 |
| Histone H1b | Hist1h1b | NM_020034 | HIST1H1B (H1.5) | NM_005322 |
| Histone H1c | Hist1h1c | NM_015786 | HIST1H1C (H1.2) | NM_005319 |
| Histone H1D | Hist1h1d | NM_145713 | HIST1H1D (H1.3) | NM_005320 |
| Histone H1e | Hist1h1e | NM_015787 | HIST1H1E (H1.4) | NM_005321 |
| Histon H1 ° | H1f0 | NM_008197 | H1F0 | NM_005318 |
| Histone H1oo | H1Foo | NM_183811 | H1Foo | NM_153833 |
| Histone H1t | Hist1h1t | NM_010377 | HIST1H1T | NM_005323 |
| Histone H1t2 | H1fnt | NM_027304 | H1FNT | NM_181788 |
| Histone H1x | H1fx | NM_198622 | H1FX | NM_006026 |
| Histone HILS1 | HILS1 | NM_081792 | HILS1 | AY286318 |
Tabelle 1. Histon H1-Subtyp Nomenklatur in Maus und Mensch.
- Design-Forward-und Reverse-PCR-Primer spezifisch für jeden H1-Gen (Tabelle 1). Aufgrund der hohen Sequenzähnlichkeit unter somatischen H1s, insbesondere im Bereich entsprechend dem zentralen globulären Domäne, ist es wichtig, sicherzustellen, dass die Primer für eine spezifische H1-Subtyp ausgebildet nicht mit o auszurichtenther H1 Gene, oder Kreuz verstärken anderen Subtypen H1. Es ist auch wichtig zu beachten, dass die meisten H1 Gene nicht Introns enthalten. Damit Intron-überspannenden Primern in der Regel für RT-PCR angenommen, um genomische Kontaminationen zu vermeiden sind nicht verfügbar. Stattdessen sollten RNA-Proben mit DNase (siehe 1.5) vorbehandelt werden, um jegliche Spuren von genomischer Verunreinigungen zu beseitigen. Darüber hinaus RT (-)-qPCR sollte parallel durchgeführt werden, um den Mangel an Verunreinigungen in den genomischen cDNA-Proben zu überprüfen.
- Auch Design-Primer für die interne Referenz Gene, deren Expression sich nicht unter den Proben verändert. Oft Housekeeping-Gene, wie Glycerinaldehyd-3-Phosphat-Dehydrogenase (GAPDH) und Beta-Actin-Gene werden als Referenzwerte Genen ausgewählt ist. qPCR Signale der Housekeeping-Gene dienen als Normalisierung Kontrollen.
- Bereiten Sie jede PCR-Reaktion (Gesamtvolumen 25 ul) wie folgt: 12,5 ul 2x IQ SYBR Green Supermix (Bio-Rad) (enthält dNTPs, 50 U / ml iTaq DNA-Polymerase, 6 mM MgCl 2, SYBR Green I und 20nM Fluorescein), 2 ul 4 ng / ul cDNA, 1,25 ul 10 nM Vorwärts / Rückwärts-Primer-Mix, und 9,25 ul ddH 2 O, und mischen Sie gut in Microseal 96-well PCR-Platte. Verwendung Microseal "B" Adhesive Dichtungen (Bio-Rad), um sicherzustellen, daß die Platte Abdeckung an der Platte wird verschlossen. Tippen Sie auf oder kurz Wirbel der PCR-Platte, und Spin-down der Reaktionsgemische durch eine kurze Zentrifugation. Die Platte wird in MyIQ Single Color Real-Time PCR Detection System (Bio-Rad) für die qPCR.
- Wir verwenden die folgenden qPCR: 95 ° C für 3 Minuten, gefolgt von 40 Zyklen von 95 ° C für 10 Sekunden, gefolgt, 60 ° C für 20 Sekunden, 72 ° C für 30 Sekunden. Untersuchen Sie die Amplifikationskurven (Abbildung 2A) für die PCR-Effizienz und CT (Threshold of cycle) Werte. Die Schwelle Linie kann automatisch durch die IQ5 Optical System Software Version 2.0 eingestellt werden.
- Der Primer und eine optimale Konzentration, die notwendig cDNA kann durch eine Standardkurve Assay getestet werden, bei dem eine serielle Verdünnung von genomischer DNA is für die qPCR-und CT-Werte verwendet werden, gegen log der Template-DNA Menge aufgetragen. Eine optimierte qPCR-Assays mit Primer der hohen Spezifität und Effizienz gibt eine lineare Standardkurve, mit dem Bestimmtheitsmaß (R 2)> 0,98. Vermeiden Sie Primer mit Amplikon-Länge länger als 200 bp, was zu schlechter Amplifikationseffizienz neigen.
- Da SYBR Green erkennt jede dsDNA ist es wichtig, eine Schmelzkurve Lauf nach dem qPCR, um sicherzustellen, dass die gewünschte Amplikon, aber nicht Primerdimere oder Verunreinigungen, verstärkt und detektiert durchzuführen. Für Schmelz-Kurven-Analyse, das Programm qPCR Instrument, um die Proben von 55 ° C bis 95 ° C in 0,5 ° C Schritten mit der Datenerhebung zu heizen. Die Standardeinstellung der Schmelze-Kurve Analyse für MyIQ (Bio-Rad) Instrument ist die folgende: 95 ° C für 1 Minute, 55 ° C für 1 Minute, um 81 Zyklen von 10 Sekunden bei Sollwert 55 ° C gefolgt, Schmelzkurve, + Temp. 0,5 ° C (Kamera sammelt die Daten bei jedem Zyklus).
- Seit Schmelztemperatur (Tm) von dsDNA ist abhängig von Amplikonlänge und GC-Gehalt, werden verschiedene Amplikons unterschiedliche Tm (n). Untersuchen der Derivat Schmelzkurven der qPCR Produkte, um die spezifische Schmelztemperatur des gewünschten Amplikons als auch das Fehlen von Rauschen Amplikon Peaks (2B) zu bestätigen.
- Bereiten Sie doppelte oder dreifache Reaktionen für jedes Assay für die statistische Analyse. Einschließen Negativkontrollen für qRT-PCR, wie beispielsweise RT (-)-qPCR (qPCR mit RNA als Matrize ohne reverse Transkription) und qPCR ohne cDNA, RNA oder DNA-Quelle in der PCR Mischung hinzugefügt. RT (-) (-) in Abbildung 2 und 3 RT ()-qPCR kann als Kontrolle für mögliche Kontaminationen mit genomischer DNA dienen.
- Analysieren qPCR-Daten mit IQ5 Optical System Software Version 2.0 (Bio-Rad). Normalisieren die Expressionswerte der H1-Isoform Genen mit der Expression von Housekeeping-Gen (zB GAPDH, ß-Actin, HPRT), um eine relative Expression von H1 Gene zu erhalten.
* Alle Verfahren sollten auf Eis oder bei 4 ° C durchgeführt werden
- Sezieren Mäusegewebe und spülen Sie ihn mit eiskaltem PBS. (Wenn nicht, um die Extraktion sofort gehen, rasten einfrieren und lagern Sie die Proben wie in Schritt 1.2 beschrieben.) Mince das Gewebe in Stücke mit Rasierklinge. Übertragen auf ein Blatt vor den Mund Dounce Homogenisator (B Stößel). 10 ml Saccharose-Puffer (0,3 M Saccharose, 15 mM NaCl, 10 mM HEPES [pH 7,9], in 2 mM EDTA, 0,5 mM PMSF, Complete Mini Protease Inhibitor Cocktail Tablet, frisch) pro Gramm Gewebe. Homogenisieren des Gewebes mit 10-15 Schlägen.
- Übertragen Sie die Homogenate in ein 15 ml Röhrchen, drehen sich mit 500 Umdrehungen pro Minute für 30 Sekunden (Zentrifuge Modell: Eppendorf 5810R); vorsichtig den Überstand in ein neues Röhrchen (verwerfen das Pellet-Gewebereste) und Zentrifuge bei 2000 rpm für 5 Minuten, zu Pellet-Zellen. Fahren Sie mit 3.4 Schritt.
- Wenn Histone und Chromatin von Zellen in Monolayer gewachsen extrahiert werden sollen, spülenmit PBS, PBS hinzufügen, um der Kulturschale und erntet die Zellen mittels Zellschaber und Pellet-Zellen durch Zentrifugation. Für Zellen in Suspension wachsen, Pellet-Zellen durch Zentrifugation.
- Das Zellpellet in 10 ml Saccharose-Puffer mit 0,5% NP-40 (pro Gramm Ausgangsgewebes Menge oder 10 (8) Zellen) ergänzt. Die Probe in einem Dounce-Homogenisator (B Pistill) und Dounce 10 Hüben in 20 Minuten Inkubation. An diesem Punkt werden Kerne erhalten. Untersuchen Sie die Kerne Qualität unter dem Mikroskop. Pellet die Kerne durch Zentrifugation bei 2000 UpM für 5 Minuten. Überstand verwerfen.
- Resuspendieren Sie das Pellet Kerne in 3 ml Puffer mit hoher Salzkonzentration (0,35 M KCl, 10 mM Tris [pH 7,2], 5 mM MgCl 2, 0,5 mM PMSF - fügen Sie vor jedem Gebrauch frisch) pro 1 g Gewebe oder 10 (8)-Zellen. Die Probe in einem kleinen Dounce-Homogenisator (B Stößel) und homogenisieren mit 5-10 Schläge.
- Aliquot der Suspension in 3 Eppendorf-Röhrchen (1 ml), auf Eis für 20 Minuten inkubieren, gefolgt vonZentrifugation bei 14.000 UpM für 10 Minuten zu pelletieren Chromatin. Überstand verwerfen.
- 0,8 ml 0,2 NH 2 SO 4 zu jedem Pellet Chromatin. Verwenden Sie ein Eppendorf-Röhrchen Pistill Dounce, um das Pellet gut schleifen, bis das Pellet vollständig dissoziiert. Inkubieren der Proben auf einer rotierenden Plattform bei 4 ° C über Nacht. Insgesamt Histone mit diesem Schritt der Säurebehandlung extrahiert.
- Zentrifugieren bei 14.000 rpm für 10 Minuten. Den Überstand (Histon-Extrakte) in zwei Eppendorf-Röhrchen (400 ul / Röhrchen). Entsorgen Sie das Pellet. Zugabe von 2,5 Volumina (1 ml) eiskaltem Ethanol zu jedem Röhrchen .. Halten Sie die Proben bei -20 ° C über Nacht.
- Zentrifuge bei 14.000 rpm für 10 Minuten bis Pellet insgesamt Histonen, den Überstand verwerfen. Waschen des Pellets dreimal mit 70% EtOH, auf der Bank lassen für 20-30 Minuten an der Luft trocknen. Bewahren Sie die getrockneten Proteine bei -80 ° C oder löst sie in ddH 2 O und fahren Sie mit HPLC-Analyse sofort. Getrocknete Proteine können gespeichert werdenbei -80 ° C bis zu 1 Jahr.
4. HPLC-Analyse von Linker-Histone
- Resuspendieren des Pellets in Histon das empfohlene Volumen ddH 2 O abhängig von der Kapazität der Reverse-Phase-Säule und der HPLC-Instrument. Wir verwenden C18-Umkehrphasen-Säule 250 × 4,6 mm (Vydac) und Äktapurifier UPC 900 Instrument (GE Healthcare) zur HPLC-Analyse. Wir lösen in der Regel 50-100 ug von insgesamt Histone in 100 ul ddH 2 0 für die Analyse.
- Zentrifugieren bei 14.000 rpm für 5 Minuten, um unlösliche Rückstände zu entfernen. Bradford-Protein-Assay wird verwendet, um die Menge an Protein auf die Säule injiziert werden zu bestimmen. Inject 50-100 ug Gesamt-Protein in die Umkehrphasensäule auf HPLC-System. Laden einer überschüssigen Menge des Proteins zu vermeiden, um ein Verstopfen der Säule zu verhindern.
- Fraktionierung der Linker Histonen und Histone mit einem steigenden Acetonitril-Gradienten, wie in Tabelle 2 aufgeführt.
Tabelle 2. Erhöhung Acetonitril-Gradienten über die Zeit.
- Der Ablauf wird bei 214 nm überwacht, und die HPLC-Profile (FiguRe 4) erfasst und analysiert mit Hilfe Äktapurifier UPC 900 (GE Healthcare) mit 5,11 UNICORN Software (GE Healthcare). Für eine weitere Analyse, zB SDS-PAGE und Massenspektrometrie - Die Proteinfraktionen können auch mit der Fraktion automatischen Kollektor (GE Frac-920) gesammelt werden.
- Die A 214 Werte der Spitzen der einzelnen H1-Subtyp und H2B werden durch die Anzahl der Peptidbindungen von jeder entsprechenden Histon-Protein normalisiert. Der relative Anteil von H1 Subtypen innerhalb der H1-Familie, wie auch das Verhältnis von H1 Subtypen Nukleosomen Teilchen können aus diesen normierten Werte A 214 (5) berechnet werden.
5. Repräsentative Ergebnisse
Die Liste der Säugetier-Subtypen H1, Gesamt-Ablaufplan und repräsentative Ergebnisse des Ausdrucks Analyse der einzelnen Histon H1 Gene sind in Tabelle 1, Abbildung 1 und Abbildung 2-5 gezeigt.2A zeigt typische Amplifikationskurven der H1a qPCR Reaktionen unter Verwendung von cDNA aus Mausleber und mESCs, während 2B zeigt die abgeleiteten Schmelzkurven der entsprechenden Amplikons. Die Schmelzkurve zeigt einen einzelnen charakteristischen Peak bei Schmelztemperatur (Tm) bei 86 ° C für die PCR-Fragments H1a, und ihm fehlt nicht-spezifischen Hintergrund Peaks, was auf eine hohe Spezifität H1a qPCR Assay. Evaluation der Amplifikationsplot (Abbildung 2A) zeigt, dass die dreifach qPCR Reaktionen von jeder Probe konsistente Signale gab mit fast identischen Ct-Werte, was auf eine hohe Reproduzierbarkeit. Der Mangel an Amplikons Aufbau von RT (-)-qPCR-Reaktionen zeigt an, dass genomische DNA-Kontamination nicht anwesend war, oder minimal. Unter Verwendung der Ct-Werte von H1 Gene und Housekeeping-Gene, wie GAPDH, wurden die relative RNA Expression jedes Gens H1 berechnet. Beispiele von berechneten Ergebnisse für H1 ° und H1a Gene sind in gezeigt Abbildung 3. Die relativen Expressionsniveaus der mRNA H1a höher sind im Vergleich mit der Maus mESCs Leber, während H1 ° Expression viel höher in der Leber als in mESCs.
Der Unterschied in der Expression von H1a oder H1 ° in MESC gegenüber erwachsenen Maus Leber zeigt sich auch HPLC-Profile von Histon-Proteine (Abbildung 4). H1 °, die Differenzierung spezifische H1, wird zu einer großen Menge in reifen Gewebe angesammelt einem Anteil von 27,2% des gesamten H1 bei erwachsenen Leber (Abbildung 5A). Im Gegensatz dazu ist H1 ° Protein fast abwesend in undifferenzierten mESCs (Abbildung 4B). Auf der anderen Seite, wird H1a hoch exprimiert, sowohl in der mRNA-Transkripte und Proteine, mESCs (Abbildung 3 und 4B). Durch Quantifizierung von H1 Peaks in HPLC-Profil, ist der relative Anteil der einzelnen H1-Subtyp innerhalb der H1-Familie bestimmt (5A). Weiterhin können die Werte der einzelnen H1-Subtyp (oderinsgesamt H1) pro Nukleosomen kann durch das Verhältnis der normierten A 214 Spitzenwert der entsprechenden H1-Subtypen (oder die Summe der gesamten H1) auf die Hälfte der normalisierten Werte für A 214 H2B (5B) berechnet werden.
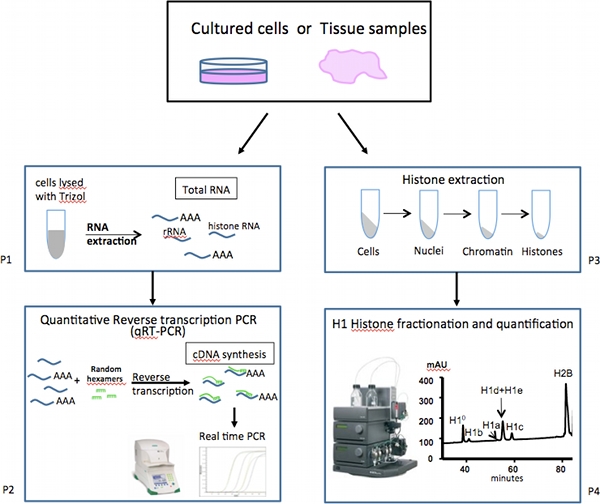
Abbildung 1. Allgemeine Systematik des Expressions-Analyse von Säugetier-Linker-Histon-Subtypen.
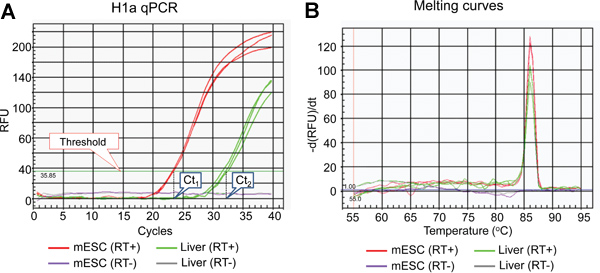
Abbildung 2. Repräsentative Ergebnisse von qPCR-Assays H1a. (A) Amplifikationsplot von H1a qPCR Assay. Die Schwelle Linie und CT-Werte von der IQ5 Optical System-Software gesetzt werden angezeigt. (B) Abwandlung der Schmelze Kurven qPCR Produkte (A) gezeigt.
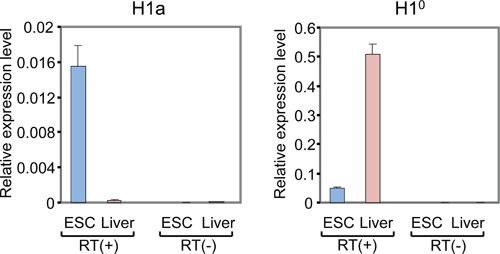
3. QRT-PCR-Analyse der mRNA-Spiegel von H1a und H1 ° mESCs und erwachsenen mouse Leber. Y-Achse stellt relativen Expressionslevel von H1 Gene, dass der Referenz-GAPDH. qPCR mit RT (-) Proben (RNA ohne reverse Transkription) zeigt minimale oder gar keine Signale.
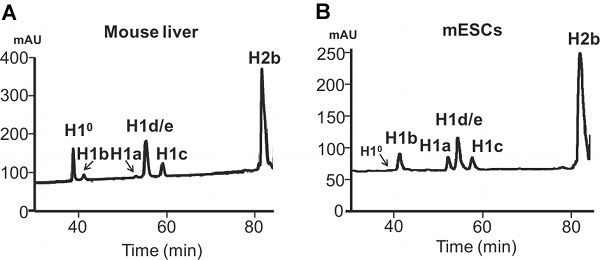
Abbildung 4. HPLC-Analyse von Histonen aus Säugetier-Zellen extrahiert. Umkehrphasen-HPLC-Analyse von 100 ug insgesamt Histone aus adulten Maus Leber (A) und Zellen der Maus (B) extrahiert. X-Achse: Elutionszeit. Y-Achse: Mau, Milli-Einheiten Saugfähigkeit.
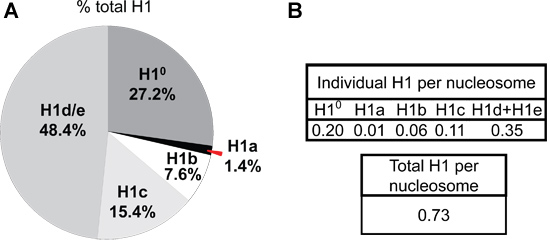
Abbildung 5. H1-Subtyp H1 Zusammensetzung und pro Nukleosom Verhältnisse bei erwachsenen Maus Leber. Die A 214 Werte der Peakfläche für jede Isoform H1 und H2B werden unter Verwendung UNICORN 5,11 Software (GE) und normalisiert durch die Anzahl der Peptidbindungen in der entsprechenden Histon-Protein. Die Summe der normierten A214 Werte aller H1 Subtypen als der Wert des gesamten H1 erhalten. Der Prozentsatz der gesamten H1 für jeden H1-Subtyp (A) sowie das Verhältnis von H 1 bis Nukleosomen (dargestellt durch die Hälfte der normalisierten Werte der A 214 H2B) (B) bei erwachsenen Maus-Leber aus dem HPLC-Profil gezeigten Tabelle berechnet in 4A.
Diskussion
Die Menge der hier vorgestellten Assays ermöglichen eine umfassende Analyse der Expression von Säugetier-Histon-Subtypen. Sorgfältig geplante und qRT-PCR-Assays bieten hochempfindliche und genaue Messungen der RNA-Nachrichten von Säugetier-Histon H1-Gene. Der kritische Teil qRT-PCR-Assays für Histon-Subtyp Gene ist die Herstellung von cDNA unter Verwendung von zufälligen Primers Reverse Transkription. mRNA der meisten Histone, einschließlich der meisten Gene H1, enthalten keine langen Poly-A-Schwanz in anderen ze...
Offenlegungen
Keine Interessenskonflikte erklärt.
Danksagungen
Diese Arbeit wird durch NIH GM085261 und ein Georgien Cancer Coalition Distinguished Scholar Award (zu YF) unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| Name des Reagenzes | Firma | Katalog-Nummer | |
| RNase Zap | Applied Biosystems | AM9780 | |
| Trizolreagenz | Invitrogen | 15596-018 | |
| SuperScriptIII | Invitrogen | 18080-051 | |
| Absolutem Ethanol | Fisher Scientific | BP2818-4 | |
| IQ SYBR Green | Bio-Rad | 170-8880 | |
| RNeasy Mini Kit | Qiagen | 74104 | |
| Ich Deoxyribonuclease | Sigma | AMP-D1 | |
| Microseal 96-well PCR-Platte | Bio-Rad | MSP-9605 | |
| Microseal 'B' Klebedichtungen | Bio-Rad | MSB-1001 | |
| Saccharose | Agros Organics | AC40594 | |
| Dinatriumhydrogenphosphat Heptahydrat (Na 2 HPO 4 · 7H 2 O) | Fisher Scientific | BP332 | |
| Natriumchlorid (NaCl) | Amerikanischen Bioanalytische | AB01915 | |
| Heptahydrat Natriumdihydrogenphosphat (NaH 2 PO 4 · 7H2O) | Fisher Scientific | BP-330 | |
| HEPES | Fisher Scientific | BP310 | |
| Kompletter Mini-Proteinase-Inhibitor Cocktail Tablette | Roche Applied Science | 11836153001 | |
| EDTA | Sigma | E-5134 | |
| Phenylmethansulfonylfluorid (PMSF) | Amerikanischen Bioanalytische | AB01620 | |
| Nonidet-40 (NP-40) | Amerikanischen Bioanalytische | AB01425 | |
| Kaliumchlorid (KCl) | Fisher Scientific | BP366 | |
| Tris [hydroxymethyl aminomethan] | Amerikanischen Bioanalytische | AB02000 | |
| Magnesiumchlorid (MgCl2) | Fisher Scientific | BP214 | |
| Schwefelsäure (H2SO4) | VWR | VW3648-3 | |
| Ammoniumhydroxid (NH 4 OH) | Agros Organics | AC42330 | |
| Bradford-Protein-Assay | Bio-Rad | 500-0001 | |
| Acetonitril | EMD | AX0145-1 | |
| Trifluoressigsäure (TFA) | JTBaker | 9470-01 |
Referenzen
- Fan, Y. H1 linker histones are essential for mouse development and affect nucleosome spacing in vivo. Mol. Cell Biol. 23, 4559-4572 (2003).
- Woodcock, C. L., Skoultchi, A. I., Fan, Y. Role of linker histone in chromatin structure and function: H1 stoichiometry and nucleosome repeat length. Chromosome Res. 14, 17-25 (2006).
- Shen, X., Gorovsky, M. A. Linker histone H1 regulates specific gene expression but not global transcription in vivo. Cell. 86, 475-483 (1996).
- Alami, R. Mammalian linker-histone subtypes differentially affect gene expression in vivo. Proc. Natl. Acad. Sci. U.S.A. 100, 5920-5925 (2003).
- Happel, N., Doenecke, D. Histone H1 and its isoforms: contribution to chromatin structure and function. Gene. 431, 1-12 (2009).
- Clausell, J., Happel, N., Hale, T. K., Doenecke, D., Beato, M. Histone H1 subtypes differentially modulate chromatin condensation without preventing ATP-dependent remodeling by SWI/SNF or NURF. PLoS One. 4, 0007243-0007243 (2009).
- Khadake, J. R., Rao, M. R. DNA- chromatin-condensing properties of rat testes H1a and H1t compared to those of rat liver H1bdec; H1t is a poor condenser of chromatin. Biochemistry. 34, 15792-15801 (1995).
- Orrego, M. Differential affinity of mammalian histone H1 somatic subtypes for DNA and chromatin. BMC Biol. 5, 22-22 (2007).
- Th'ng, J. P., Sung, R., Ye, M., Hendzel, M. J. H1 family histones in the nucleus. Control of binding and localization by the C-terminal domain. J. Biol. Chem. 280, 27809-27814 (2005).
- Wang, Z. F., Sirotkin, A. M., Buchold, G. M., Skoultchi, A. I., Marzluff, W. F. The mouse histone H1 genes: gene organization and differential regulation. J. Mol. Biol. 271, 124-138 (1997).
- Brown, D. T., Sittman, D. B. Identification through overexpression and tagging of the variant type of the mouse H1e and H1c genes. J. Biol. Chem. 268, 713-718 (1993).
- Fan, Y., Sirotkin, A., Russell, R. G., Ayala, J., Skoultchi, A. I. Individual somatic H1 subtypes are dispensable for mouse development even in mice lacking the H1(0) replacement subtype. Mol. Cell. Biol. 21, 7933-7943 (2001).
- Fan, Y., Skoultchi, A. I. Genetic analysis of H1 linker histone subtypes and their functions in mice. Methods Enzymol. 377, 85-107 (2004).
- Heid, C. A., Stevens, J., Livak, K. J., Williams, P. M. Real time quantitative PCR. Genome Res. 6, 986-994 (1996).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten