É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Análise de Expressão de mamíferos Linker-histona Subtipos
Neste Artigo
Resumo
Nós descrevemos um conjunto de ensaios para analisar os níveis de expressão de histonas linker H1. mRNA de genes H1 individuais são quantitativamente medido por acaso a transcrição reversa com base iniciador seguido por PCR em tempo real, enquanto que a quantificação de proteína de histonas H1 é conseguido através da análise por HPLC.
Resumo
Histona H1 Linker liga-se à partícula núcleo nucleossomo e DNA linker, facilitando o dobramento da cromatina em estrutura de ordem superior. H1 é essencial para o desenvolvimento de mamíferos 1 e regula a expressão do gene específica in vivo 2-4. Entre as proteínas altamente conservadas histona, a família de histonas linker H1 é o grupo mais heterogénea. Existem 11 subtipos H1 em mamíferos que são diferencialmente regulados durante o desenvolvimento e em diferentes tipos celulares. Estes subtipos H1 incluem 5 somática H1s (H1a-e), o H1 substituição 0, 4 subtipos de células germinativas H1 específicos, e H1x 5. A presença de subtipos H1 múltiplas que diferem na afinidade de ligação ao ADN ea capacidade de compactação cromatina 6-9 fornece um nível adicional de modulação da função da cromatina. Assim, a análise quantitativa da expressão subtipos H1 individuais, tanto de ARNm e as proteínas, é necessário para melhor compreensão da regulação de maiorestrutura de ordem cromatina e função.
Descrevemos aqui um conjunto de ensaios concebidos para analisar os níveis de expressão dos subtipos H1 individuais (Figura 1). a expressão do mRNA de genes de variantes diferentes H1 é medido por um conjunto de altamente sensíveis e quantitativa transcrição reversa PCR (qRT-PCR), que são mais rápida, mais precisa e requerem amostras muito menos em comparação com a abordagem alternativa de análise de Northern blot. Ao contrário da maioria outras mensagens de ARNm celulares, mRNAs para os genes mais histona, incluindo a maioria dos genes H1, carecem de uma cauda poliA de comprimento, mas contêm uma estrutura de haste loop-na extremidade 3 'região não traduzida (UTR) 10. Portanto, cDNAs são preparados a partir de RNA total por transcrição inversa utilizando iniciadores aleatórios, em vez de oligo-dT iniciadores. Os ensaios em tempo real de PCR com primers específicos para cada subtipos H1 (Tabela 1) são realizados para obter medições altamente quantitativa dos níveis de mRNA de subtipo H1 indivíduos. A expressão de genes de limpeza são analisados como controlos para a normalização.
A abundância relativa de proteínas de cada subtipo H1 e histonas núcleo é obtido através de cromatografia líquida de fase reversa de alto desempenho (RP-HPLC) análise de histonas total extraído de células de mamíferos 11-13. O método de HPLC e as condições de eluição descrito aqui dar separações óptimas de subtipos H1 do rato. Pela quantificação do perfil de HPLC, calcula-se a proporção relativa de subtipos H1 individuais dentro da família H1, bem como determinar a H1 a razão de nucleossomas nas células.
Protocolo
1. Preparação de amostras e de extração de RNA
- Antes da extracção de RNA, todas as superfícies de trabalho e pipetas deve ser limpo com etanol a 70% e tratado com RNase solução de descontaminação, tais como a RNase Zap. Esta prática reduz as possibilidades de RNase contaminação e degradação do ARN. Usar luvas para todos os procedimentos.
- Para extrair o RNA a partir de tecido do rato, dissecar o órgão de interesse a partir do rato sacrificados, e lavar o tecido em fosfato gelada salino tamponado (PBS: 0,13 M de NaCl, 5 mM de fosfato de sódio dibásico hepta-hidratado, 5 mM de sódio di-hidrogenofosfato de hepta-hidrato, pH 7,4) . Proceder de imediato à extração de RNA na etapa 1.4. Se o tecido fresco não é para ser processado para RNA de extracção, as amostras de tecido deve ser encaixe congelados em azoto líquido imediatamente e armazenado a -80 ° C para utilização posterior.
- Se o RNA é para ser extraída a partir de cultura de células aderentes, aspirar meios de cultura, lavar com uma quantidade suficiente de PBS, e adicionar reagente Trizol (Invitrogen) sobre a placa e avançar para o passo 1.4. Para as células cultivadas em suspensão, a colheita das células e as células de pelotas por centrifugação. Descartar meios, lavar os peletes brevemente com PBS, e sedimentar as células com centrifugação. Adicionar Reagente Trizol e avance para o passo 1.4.
- Reagente Trizol suficiente é necessário para a obtenção de RNA de alta qualidade. Use 1 ml de reagente Trizol para extrair RNA 50-100 mg de tecido, 5 - 10 x 10 6 células (por culturas em suspensão) ou por chapa 3,5 cm para culturas aderentes). Homogeneizar o tecido em reagente Trizol com Polytron PT2100 homogeneizador (ou equivalente). Continuar para extrair ARN a partir de amostras de tecidos ou células de acordo com o manual do fabricante para o reagente Trizol (Invitrogen).
- Concentração de RNA é medida usando Nanodrop 1000 (Thermo Scientific) e qualidade de RNA é analisada por electroforese em gel. As gamas de rendimento típicos de 1-10 ug de RNA por mg de tecido ou 5-15 ug de RNA por 1 x 10 6 células em cultura. Para eliminar ocontaminação potencial de RNA a partir quantidade vestigial de ADN genómico, as amostras de RNA são tratadas com RNase ADNase (Sigma AMP-D1) de acordo com as instruções do fabricante. Repita determinação da concentração de RNA e electroforese em gel para garantir que não a degradação do ARN a partir deste tratamento. Loja extraído ARN a -80 ° C.
* Nota: o RNA pode também ser extraído usando o kit RNAeasy (Qiagen) de acordo com o manual do kit, ou por DNA / RNA kit (Qiagen), se ambos ADN e ARN são desejados.
2. Quantitativa reversa PCR de transcrição (qRT-PCR)
- O RNA total é a transcrição reversa em ADNc Superscript III usando o Sistema de Síntese da primeira cadeia (Invitrogen). Uma vez que a maioria dos genes de mRNA de H1 falta de poli-A-caudas, é crítica a utilização hexâmeros aleatórios, em vez de oligo-dT como iniciadores para a síntese de cDNA. No entanto, se a análise da expressão de genes com mensagens poliadenilados em níveis baixos é também desejado, uma mistura de hexâmeros aleatórios e oligo-dT should ser utilizado na reacção de transcrição reversa (RT) para melhorar a eficiência da transcrição reversa de ARNm poliadenilado.
- Realizar a reacção de RT de acordo com o manual do fabricante. Resumidamente,
- Num tubo de 0,5 ml de PCR, combinar 5 ug de RNA total, 1 ul de 50 ng / uL hexâmeros aleatórios, 1 ul de 10 mM de dNTP mistura, e adicionar-tratada com DEPC H 2 O para perfazer o volume total de reacção de 10 uL. Misturar bem e incubar durante 5 minutos a 65 ° C, seguido por uma incubação minutos em gelo.
- Preparar 10 uL da mistura de síntese de cDNA: 2 uL de tampão de 10xRT, 4 uL de 25 mM de MgCl2, 2 ul de 0,1 M de DTT, 1 ul de RNaseOut (40 U / ul), 1 ul de SuperScript III RT (200 U / ul) e adicioná-lo ao RNA mistura / primer.
- Incubar durante 10 minutos a 25 ° C, seguido por 50 minutos a 50 ° C e terminar a reacção a 85 ° C durante 5 minutos.
- Cada reacção tipicamente produz 100-250 ng / uL de produto de cDNA. Armazenar produtos de cDNA a -20° C ou proceder de imediato em tempo real PCR quantitativo (qPCR).
- qPCR pode quantificar com precisão as cópias alvo de seqüência com alta eficiência e reprodutibilidade 14. Nós escolha qPCR medido por SYBR Green corante, o que dá um sinal fluorescente apenas quando se intercala com ADN de cadeia dupla (dsDNA). Apesar de não ser tão específico quanto Taqman ensaio 14, este método é mais rentável, mais fácil de ser adotada no laboratório, e dá mais versatilidade para qPCR. Portanto, é importante para examinar a trama de amplificação (Figura 2A) e as curvas de fusão derivada do produto qPCR (Figura 2B) para garantir a eficiência da reacção e especificidade.
| Subtipos histonas | Nomenclatura do mouse histona | Nomenclatura histona humana | ||
| Genome ne | Não adesão. | Nome Gene | Não adesão. | |
| H1a histona | Hist1h1a | NM_030609 | HIST1H1A (H1.1) | NM_005325 |
| H1b histona | Hist1h1b | NM_020034 | HIST1H1B (H1.5) | NM_005322 |
| H1c histona | Hist1h1c | NM_015786 | HIST1H1C (H1.2) | NM_005319 |
| H1d histona | Hist1h1d | NM_145713 | HIST1H1D (H1.3) | NM_005320 |
| H1E histona | Hist1h1e | NM_015787 | HIST1H1E (H1.4) | NM_005321 |
| Histona H1 ° | H1f0 | NM_008197 | H1F0 | NM_005318 |
| H1oo histona | H1foo | NM_183811 | H1FOO | NM_153833 |
| H1t histona | Hist1h1t | NM_010377 | HIST1H1T | NM_005323 |
| H1t2 histona | H1fnt | NM_027304 | H1FNT | NM_181788 |
| H1x histona | H1fx | NM_198622 | H1FX | NM_006026 |
| Hils1 histonas | Hils1 | NM_081792 | HILS1 | AY286318 |
Tabela 1. Nomenclatura subtipo Histona H1 em rato e humano.
- Desenho para a frente e para trás iniciadores de PCR específicos para cada gene H1 (Tabela 1). Devido à semelhança de sequência elevada entre somática H1s, particularmente na região correspondente ao domínio globular central, é crítico para assegurar que os primers desenhados para um subtipo H1 específico não se alinham com ogenes utras H1, ou transversais amplificar subtipos H1 outros. É também importante notar que a maioria dos genes H1 não contêm intrões. Assim intrão-medindo iniciadores tipicamente adoptadas para RT-PCR para evitar a contaminação genómico não estão disponíveis. Em vez disso, as amostras de RNA deve ser pré-tratados com DNase (ver 1,5) para eliminar qualquer quantidade vestigial de contaminação genómico. Além disso, a RT (-)-qPCR deve ser realizado em paralelo para validar a ausência de contaminação genómico nas amostras de cDNA.
- Também primers para genes de design de referência internos, cuja expressão não são alterados entre as amostras. Muitas vezes, os genes de manutenção, tais como gliceraldeído-3-fosfato desidrogenase (GAPDH) e genes da beta-actina, são escolhidos como genes de referência. sinais qPCR de genes de limpeza servir como controlos de normalização.
- Preparar cada reacção de PCR (volume total 25 ul) da seguinte forma: 12,5 uL de 2x IQ SYBR Green Supermix (Bio-Rad) (dNTPs que contenham, 50 U / ml de DNA polimerase iTaq, 6 mM MgCl2, SYBR Green I e 20nM fluoresceína), 2 uL de 4 ng / uL de cDNA, 1,25 ul de 10 nM para a frente / mistura de iniciador reverso, e 9,25 ul de ddH2O, e misturar bem em Microseal placa de 96 poços de PCR. Use auto-colantes «B» Microseal (Bio-Rad) para assegurar que a placa de cobertura é selada à placa. Toque ou vortex brevemente a placa de PCR, e girar as misturas de reação por uma centrifugação curta. Colocar a placa na MyIQ única cor Sistema de Detecção de PCR em tempo real (Bio-Rad) para qPCR.
- Usamos as condições qPCR seguintes: 95 ° C durante 3 minutos, seguido por 40 ciclos de 95 ° C durante 10 segundos, 60 ° C durante 20 segundos, 72 ° C durante 30 segundos. Examinar as curvas de amplificação (Figura 2A) para PCR eficiência e Ct (Limiar do ciclo) valores. A linha de limite pode ser definido automaticamente pelo sistema óptico IQ5 Versão 2.0 do software.
- A eficiência primer e concentração de cDNA óptima necessária pode ser testada por um ensaio de curva padrão, no qual uma diluição em série de ADN genómico is usado para qPCR e valores Ct são plotados contra registro de quantidade de DNA molde. Um ensaio qPCR otimizado com primers de alta especificidade e eficiência vai dar uma curva linear padrão, com o coeficiente de determinação (R 2)> 0,98. Evitar iniciadores com comprimento maior do que amplicão de 200 pb, que tendem a ter uma eficiência de amplificação pobre.
- Devido SYBR Green detecta qualquer dsDNA, é crítico para realizar um funcionamento curva de fusão na sequência da qPCR para assegurar que o fragmento amplificado desejado, mas não dímeros de iniciadores ou contaminantes, são amplificados e detectados. Para fusão curva-análise do programa, o instrumento qPCR para aquecer as amostras a partir de 55 ° C a 95 ° C em 0,5 ° C com incrementos de recolha de dados. A configuração padrão do material fundido curva-análise para MyIQ (Bio-Rad) instrumento é a seguinte: 95 ° C durante 1 minuto, 55 ° C durante 1 minuto, seguido por 81 ciclos de 10 segundos a ponto de ajuste 55 ° C, derreter curva, + temp 0,5 ° C (câmara recolhe dados em cada ciclo).
- Desde temperatura de fusão (Tm) de dsDNA é dependente do comprimento do fragmento amplificado e conteúdo de GC, amplicons diferentes terão Tm diferente (s). Examinar as curvas de fusão derivados dos produtos qPCR para confirmar a temperatura de fusão específica de amplicões desejados, bem como a falta de picos de ruído amplicon (Figura 2B).
- Prepare duplicado ou triplicado, reações para cada ensaio para análise estatística. Incluir controlos negativos para qRT-PCR, tais como RT (-)-qPCR (qPCR com ARN como molde sem transcrição reversa) e qPCR sem ADNc, ARN ou fonte de DNA adicionada na mistura de PCR. RT (-)-qPCR pode servir como controlo para a contaminação de ADN genómico potencial (RT (-) na Figura 2 e 3).
- Analisar dados qPCR com IQ5 Optical System Software versão 2.0 (Bio-Rad). Normalizar os valores de expressão de genes H1 isoforma com a expressão de gene de limpeza (por exemplo, GAPDH, ß-actina, HPRT) para se obter níveis de expressão relativa de genes H1.
* Todos os procedimentos devem ser realizadas em gelo ou a 4 ° C.
- Dissecar o tecido do mouse e lave-o com PBS gelado. (Se não proceder à extracção imediatamente, encaixe congelar e armazenar as amostras como descrito no passo 1.2.) Picar o tecido em pedaços com lâmina de barbear. Transferir mince para um homogeneizador de Dounce (B pilão). Adicionar 10 ml tampão de sacarose (0,3 M de sacarose, 15 mM de NaCl, 10 mM de HEPES [pH 7,9], 2 mM de EDTA, 0,5 mM de PMSF, Complete Mini Protease Inhibitor Tablet Cocktail, adicionar fresco) por grama de tecido. Homogeneizar o tecido com 10-15 pancadas.
- Transfira os homogenatos para um tubo de 15 ml, centrifugação a 500 rpm durante 30 segundos (modelo de centrífuga: Eppendorf 5810R); transferir cuidadosamente o sobrenadante para um novo tubo (descartar os detritos sedimento de tecido), e centrifugação a 2000 rpm durante 5 minutos, para as células de peletes. Vá para o passo 3.4.
- Se histonas e cromatina são para ser extraída a partir de células cultivadas em monocamada, lavarcom PBS, adicionar PBS para o prato de cultura, e colheita das células utilizando raspador de células, e células de pelotas por centrifugação. Para células cultivadas em suspensão, de aglomerados de células por centrifugação.
- Ressuspender o sedimento de células em 10 ml tampão de sacarose suplementado com 0,5% de NP-40 (por grama de partida quantidade de tecido ou 10 (8) células). Transferir a amostra para um homogeneizador Dounce (B pilão) e Dounce 10 cursos dentro de 20 minutos de incubação. Neste ponto, os núcleos são obtidos. Examine a qualidade núcleos sob um microscópio. Pellet núcleos por centrifugação a 2000 rpm durante 5 minutos. Descartar o sobrenadante.
- Ressuspender o sedimento em núcleos 3 tampão salino ml alta (0,35 M de KCl, 10 mM de Tris [pH 7,2], 5 mM de MgCl2, 0,5 mM de PMSF - adicionar fresco antes de cada utilização) por 1 g de tecido ou 10 (8) das células. Transferir a amostra para um pequeno homogeneizador Dounce (B pilão) e homogeneizar com traços 5-10.
- Alíquota da suspensão em 3 tubos Eppendorf (1 ml cada), incubar em gelo durante 20 minutos, seguido porcentrifugação a 14.000 rpm durante 10 minutos para sedimentar cromatina. Descartar o sobrenadante.
- Adicionar 0,8 ml 0,2 NH 2 SO 4 para cada pellet cromatina. Use um tubo eppendorf Dounce pilão para moer o sedimento bem até a pelete é completamente dissociado. Incubar as amostras sobre uma plataforma rotativa a 4 ° C durante a noite. Histonas totais são extraídos com este passo de tratamento com ácido.
- Centrifugar a 14.000 rpm durante 10 minutos. Transferir o sobrenadante (extractos histona) em dois tubos Eppendorf (400 ul / tubo). Descartar o sedimento. Adicionar 2,5 volumes (1 ml) de etanol gelado a cada tubo .. Manter as amostras a -20 ° C durante a noite.
- Centrifugar a 14.000 rpm durante 10 minutos a histonas pelota totais, descartar o sobrenadante. Lavar o sedimento três vezes com EtOH a 70%, para sair em bancada durante 20-30 minutos para secar ao ar. Armazenar as proteínas secas a -80 ° C ou dissolvê-los em ddH2O e proceder à análise por HPLC imediatamente. Proteínas secas podem ser armazenadasa -80 ° C durante até 1 ano.
4. A análise por HPLC de histonas Linker
- Ressuspender o sedimento histona no volume recomendado de ddH2O, dependendo da capacidade de coluna de fase reversa e do instrumento de HPLC. Usamos coluna de fase reversa C18 250 x 4,6 mm (Vydac) e ÄKTApurifier UPC 900 instrumento (GE Healthcare) para análise por HPLC. Nós tipicamente dissolver 50-100μg de histonas totais em 100 uL de DDQ 2 0 para a análise.
- Centrifugar a 14.000 rpm durante 5 minutos para remover os resíduos insolúveis. Bradford ensaio de proteína é usada para determinar a quantidade de proteína a ser injectada na coluna. Injectar 50-100 ug de proteína total na coluna de fase reversa em sistema de HPLC. A carregar uma quantidade em excesso de proteína deve ser evitado para evitar o entupimento da coluna.
- Fraccionar as histonas linker e histonas do núcleo com um gradiente de acetonitrilo crescente como listado na Tabela 2.
Tabela 2. Aumentando gradiente de acetonitrilo ao longo do tempo.
- O efluente é monitorizado a 214 nm, e os perfis de HPLC (FIGUre 4) são registrados e analisados utilizando ÄKTApurifier UPC 900 (GE Healthcare) com UNICORN 5,11 software (GE Healthcare). As fracções de proteína também pode ser recolhida com a fração de auto colector-(Frac-920 - GE) para posterior análise, por exemplo, espectrometria de SDS-PAGE e de massa.
- As um 214 valores dos picos de cada subtipo H1 e H2B são normalizados pelo número de ligações peptídicas de cada proteína correspondente histona. A proporção relativa de subtipos H1 individuais dentro da família histona H1, bem como a razão entre os subtipos H1 a partículas de núcleo nucleossomas pode ser calculado a partir destes valores normalizados um 214 (Figura 5).
5. Os resultados representativos
A lista de subtipos H1 mamíferos, fluxograma global e os resultados representativos da análise da expressão de genes individuais histona H1 são mostrados na Tabela 1, Figura 1 e Figura 2-5, respectivamente.A Figura 2A mostra curvas típicas de amplificação de reacções de H1A qPCR utilizando ADNc preparada a partir de fígado de rato e mESCs, enquanto que a Figura 2B mostra as curvas de fusão derivadas dos amplicões correspondentes. A curva de fusão exibe um pico único característica, a temperatura de fusão (Tm) a 86 ° C para a PCR H1a amplicão, e carece de picos não-específicos de fundo, o que sugere uma alta especificidade de H1a ensaio qPCR. Avaliação da trama de amplificação (Figura 2A) mostra que as reacções em triplicado qPCR de cada amostra deu sinais consistentes com os valores quase idênticos Ct, sugerindo elevada reprodutibilidade. A falta de amplicões acumulação de RT (-)-qPCR reacções indica que a contaminação de ADN genómico não estava presente, ou mínima. Utilizando os valores de TC de genes H1 e genes de manutenção, tais como GAPDH, os níveis relativos de expressão de RNA de cada gene H1 foram calculados. Exemplos de resultados calculados para H1 ° e genes H1A são mostrados na Figura 3. Os níveis de expressão relativa de ARNm de H1a são mais elevados em comparação com mESCs fígado de rato, enquanto que H1 expressão ° é muito mais elevada no fígado do que em mESCs.
A diferença na expressão de H1a ou ° H1 em MESC versus adulto fígado de rato é também evidente a partir de perfis de HPLC de proteínas histonas (Figura 4). H1 °, o H1 diferenciação específica, é acumulado a uma grande quantidade de tecidos maduros, representando 27,2% do total de H1 no fígado adulto (Figura 5A). Em contraste, a proteína H1 ° é praticamente ausente no mESCs indiferenciadas (Figura 4B). Por outro lado, H1a é altamente expresso, tanto em transcritos de ARNm e proteínas, em mESCs (Figura 3 e 4B). Através da quantificação de picos H1 no perfil de HPLC, a proporção relativa de cada subtipo H1 indivíduo dentro da família H1 é determinada (Figura 5A). Além disso, os valores de subtipo H1 individual (ouH1 total) por nucleossomo pode ser calculada pela relação do valor de pico normalizada Um 214 de subtipos H1 correspondente (ou soma de H1 total) para uma metade da normalizada um 214 valores para H2B (Figura 5B).
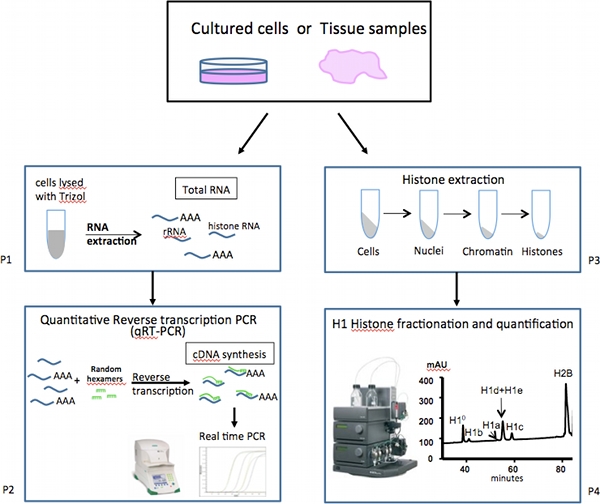
Figura 1. Esquema geral de análise de expressão de mamífero linker-histona subtipos.
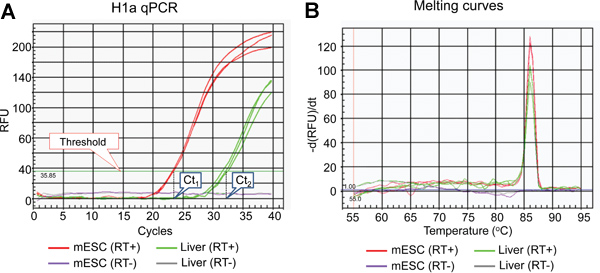
Figura 2. Os resultados representativos de H1a ensaio qPCR. (A) Ampliação da trama H1a ensaio qPCR. A linha de limiar e valores Ct definidos pelo software do sistema óptico IQ5 são indicadas. (B) Derivados derreter-curvas de produtos qPCR mostrado em (A).
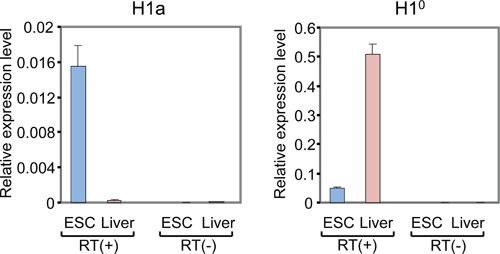
Figura 3. Análise qRT-PCR de níveis de mRNA de H1a e ° H1 em mESCs e m adultoOuse fígado. Eixo Y representa os níveis de expressão relativa dos genes H1 à do gene GAPDH referência. qPCR com RT (-) amostras de RNA (transcrição reversa sem) mostra sinais mínimos ou não.
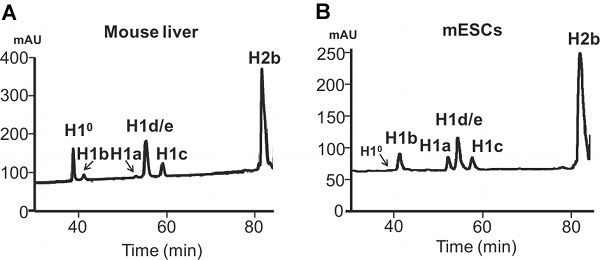
Figura 4. Análise por HPLC de histonas extraídos a partir de células de mamíferos. Inversão de fases de análise por HPLC de 100 ug histonas totais extraídos a partir de fígado de rato adulto (A) e CES de rato (B). Eixo X: tempo de eluição. Eixo Y: mAU, mili-absorção unidades.
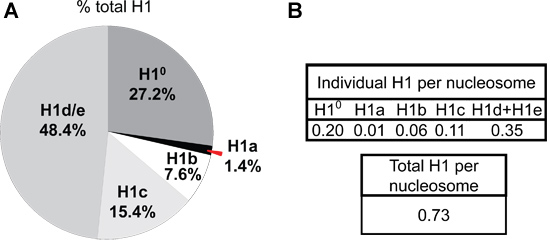
Figura 5. H1 composição subtipo H1 e por razões de nucleossomo no fígado do rato adulto. Os valores de um 214 a área do pico para cada isoforma H1 e H2B são calculados usando o software UNICORN 5,11 (GE Healthcare) e normalizado pelo número de ligações peptídicas presentes na proteína correspondente histona. A soma normalizada A214 valores de todos os subtipos H1 é obtido como o valor para H1 total. A percentagem de H1 total para cada subtipo H1 (A), bem como a proporção de H1 a nucleossomo (representada por uma metade do normalizada um 214 valores de H2B) (B) no fígado do rato adulto são calculados a partir do perfil de HPLC mostrado na Figura 4A.
Discussão
O conjunto de ensaios aqui apresentados permitir uma análise abrangente dos níveis de expressão de mamífero vinculador subtipos histonas. Adequadamente projetados qRT-PCR ensaios fornecem medições altamente sensível e precisa de mensagens de RNA a partir de quaisquer genes de mamíferos histona H1. A parte crítica de qRT-PCR ensaios para linker genes subtipo histona é a preparação de ADNc utilizando o iniciador aleatório baseado transcrição reversa. mRNA de genes mais histona, incluindo genes mais H1, não...
Divulgações
Não há conflitos de interesse declarados.
Agradecimentos
Este trabalho é apoiado pelo NIH concessão GM085261 e um Cancer Coalition Geórgia Prêmio Distinguished Scholar (a FA).
Materiais
| Name | Company | Catalog Number | Comments |
| Nome do reagente | Companhia | Número de catálogo | |
| RNase Zap | Applied Biosystems | AM9780 | |
| Reagente Trizol | Invitrogen | 15596-018 | |
| SuperScriptIII | Invitrogen | 18080-051 | |
| Etanol absoluto | Fisher Scientific | BP2818-4 | |
| QI SYBR Green | Bio-Rad | 170-8880 | |
| Kit Mini RNeasy | Qiagen | 74104 | |
| Desoxirribonuclease I | Sigma | AMP-D1 | |
| Microseal placa de 96 poços de PCR | Bio-Rad | MSP-9605 | |
| Auto-colantes 'B' Microseal | Bio-Rad | MSB-1001 | |
| Sacarose | Organics Agros | AC40594 | |
| Fosfato de sódio dibásico hepta-hidratado (Na 2 HPO 4 · 7H 2 O) | Fisher Scientific | BP332 | |
| Cloreto de sódio (NaCl) | Americana Bioanalytical | AB01915 | |
| Hepta-di-hidrogenofosfato de sódio (NaH 2 PO 4 · 7H2O) | Fisher Scientific | BP-330 | |
| HEPES | Fisher Scientific | BP310 | |
| Completa Mini proteinase comprimido coquetel inibidor | Roche Applied Science | 11836153001 | |
| EDTA | Sigma | E-5134 | |
| Fluoreto de Phenylmethanesulfonyl (PMSF) | Americana Bioanalytical | AB01620 | |
| Nonidet-40 (NP-40) | Americana Bioanalytical | AB01425 | |
| Cloreto de potássio (KCl) | Fisher Scientific | BP366 | |
| Tris [hidroximetil aminometano] | Americana Bioanalytical | AB02000 | |
| Cloreto de magnésio (MgCl2) | Fisher Scientific | BP214 | |
| Ácido sulfúrico (H2SO4) | VWR | VW3648-3 | |
| Hidróxido de amónio (NH4OH) | Organics Agros | AC42330 | |
| Bradford Protein Assay | Bio-Rad | 500-0001 | |
| Acetonitrila | EMD | AX0145-1 | |
| Ácido trifluoroacético (TFA) | JTBaker | 9470-01 |
Referências
- Fan, Y. H1 linker histones are essential for mouse development and affect nucleosome spacing in vivo. Mol. Cell Biol. 23, 4559-4572 (2003).
- Woodcock, C. L., Skoultchi, A. I., Fan, Y. Role of linker histone in chromatin structure and function: H1 stoichiometry and nucleosome repeat length. Chromosome Res. 14, 17-25 (2006).
- Shen, X., Gorovsky, M. A. Linker histone H1 regulates specific gene expression but not global transcription in vivo. Cell. 86, 475-483 (1996).
- Alami, R. Mammalian linker-histone subtypes differentially affect gene expression in vivo. Proc. Natl. Acad. Sci. U.S.A. 100, 5920-5925 (2003).
- Happel, N., Doenecke, D. Histone H1 and its isoforms: contribution to chromatin structure and function. Gene. 431, 1-12 (2009).
- Clausell, J., Happel, N., Hale, T. K., Doenecke, D., Beato, M. Histone H1 subtypes differentially modulate chromatin condensation without preventing ATP-dependent remodeling by SWI/SNF or NURF. PLoS One. 4, 0007243-0007243 (2009).
- Khadake, J. R., Rao, M. R. DNA- chromatin-condensing properties of rat testes H1a and H1t compared to those of rat liver H1bdec; H1t is a poor condenser of chromatin. Biochemistry. 34, 15792-15801 (1995).
- Orrego, M. Differential affinity of mammalian histone H1 somatic subtypes for DNA and chromatin. BMC Biol. 5, 22-22 (2007).
- Th'ng, J. P., Sung, R., Ye, M., Hendzel, M. J. H1 family histones in the nucleus. Control of binding and localization by the C-terminal domain. J. Biol. Chem. 280, 27809-27814 (2005).
- Wang, Z. F., Sirotkin, A. M., Buchold, G. M., Skoultchi, A. I., Marzluff, W. F. The mouse histone H1 genes: gene organization and differential regulation. J. Mol. Biol. 271, 124-138 (1997).
- Brown, D. T., Sittman, D. B. Identification through overexpression and tagging of the variant type of the mouse H1e and H1c genes. J. Biol. Chem. 268, 713-718 (1993).
- Fan, Y., Sirotkin, A., Russell, R. G., Ayala, J., Skoultchi, A. I. Individual somatic H1 subtypes are dispensable for mouse development even in mice lacking the H1(0) replacement subtype. Mol. Cell. Biol. 21, 7933-7943 (2001).
- Fan, Y., Skoultchi, A. I. Genetic analysis of H1 linker histone subtypes and their functions in mice. Methods Enzymol. 377, 85-107 (2004).
- Heid, C. A., Stevens, J., Livak, K. J., Williams, P. M. Real time quantitative PCR. Genome Res. 6, 986-994 (1996).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados