需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
赖氨酸去甲基化酶活性的定量测量中的应用的MassSQUIRM
摘要
我们提出了一个使用MALDI质谱和还原甲基化化学的方法来量化赖氨酸甲基化的变化。
摘要
近日,后生监管机构已发现1-3在许多不同的疾病的关键球员。因此,这些酶的主要目标是为小分子的研究和药物开发4。许多后生监管机构最近才发现,仍然是在被分类的过程。在这些酶的赖氨酸去甲基从组蛋白和其他蛋白质的赖氨酸甲基组。由于这一类酶的小说性质,一些实验已经开发,研究他们的活动。这一直是一个路障,以分类和高吞吐量研究组蛋白去甲基化酶。目前,很少去甲基化酶检测存在。那些确实存在往往在本质上是定性的,不能同时不同赖氨酸甲基化状态之间的辨别(联合国,单,二和三)。质谱是常用来确定脱甲基酶活性,但目前质谱分析没有解决差异甲基化多肽是否不同的电离。差甲基化肽的电离,使甲基化状态比较困难,肯定不定量(图1A)。因此,可以检测不优化为脱甲基酶活性的全面分析。
在这里,我们描述的方法称为MassSQUIRM(质谱定量使用同位素还原甲基)是基于还原甲基氘甲醛强制所有赖氨酸是DI-甲基胺基,从而使它们基本上是相同的化学物质,因此,电离相同(图1B)。唯一的化学还原甲基化的差异,是氢和氘,这并不影响电离电离效率。 MassSQUIRM法是与联合国,单或二甲基赖氨酸去甲基反应产物具体。该法也适用于赖氨酸甲基转移给SA我的反应产物。在这里,我们使用相结合的还原甲基化化学和MALDI质谱测量类LSD1活动,赖氨酸去甲基化酶能够消除DI-和单甲基基团,合成肽基板5。此法是简单,容易服从任何与电离质谱在实验室或通过蛋白质组学设施的实验室。该法〜8倍的动态范围,很容易扩展到板格式5。
研究方案
该协议被修改布莱尔等6。
1。类LSD1去甲基化检测
- 在终体积20μL,结合0.25微克二甲基化组蛋白H3肽脱甲基缓冲区(ARTKme2QTARKSTGGKAPRKQLYK-生物素)(50毫米的Tris-Cl的pH值8.5,50 mM的氯化钾,氯化镁2 5毫米,5%的125纳克重组类LSD1甘油)。此外,执行没有酶肽单独控制。控制是第3和不需要还原的甲基化。
- 在37°C孵育2小时
- 收集肽,8μL,波罗斯R2的20微米微珠添加到每个样品为15分钟,在室温下搅拌。准备中止与1体积的甲醇1体积珠的“波罗斯珠,然后添加5%的甲酸/ 0.2%TFA的10卷。
- 条件2 C 18 ZipTips吸入20μL0.1%TFA的尖端,把它推回去移液器。这样做两次,0.1%T的足总杯,四次,用70%乙腈/ 0.1%TFA的,和4倍,0.1%TFA的再次。
- 条件C 18 ZipTips加载到C 18树脂在ZipTips的背后吸取珠浆和推用移液器体积的两个样本(控制和脱甲基反应)。
- 20μL0.1%TFA的两次洗ZipTips。
- 洗脱在40μL70%乙腈/ 0.1%TFA的。
- 完全用SpeedVac浓缩冻干每个洗脱。
2。还原甲基化
修改本协议的一部分,从布莱尔等。Rayment等。6,7。
- 重新暂停在100μL50 mM磷酸盐缓冲液,pH值7.4的脱甲基反应。
- 要脱甲基反应,添加15毫克/毫升硼烷二甲胺8μL。硼烷二甲胺溶解在50 mM磷酸盐缓冲液,pH值7.4,给予15毫克/毫升原液。这应新鲜。
- 到去甲基反应,添加16μL250毫米氘甲醛。 250毫米的氘甲醛准备稀释的股票,在H 2 0。这应新鲜。
- 在4°C孵育2小时。
- 重复步骤2.2,2.3和2.4。
- 重复步骤2.2。
- 孵育4°C的〜15小时。
- 到去甲基样品,加12.5μL1M的Tris,pH值7.4解渴还原甲基化反应。
3。电离质谱光谱法
- 波罗斯R2的20微米微珠添加到脱甲基反应,并收集ZipTips步骤1.3-1.6所述。
注:重新挂起100μL50 mM磷酸盐缓冲液,pH值7.4的控制和治疗其他样品在3.1步开始。
- 洗脱到电离的样品板使用2μL33%饱和从ZipTips的控制和脱甲基反应样本ð2,5 - 二羟基苯甲酸。准备2,5 - 二羟基苯甲酸33%,使70%的乙腈/ 0.1%TFA的2,5 - 二羟基苯甲酸的饱和溶液,然后稀释至33%,70%乙腈/ 0.1%TFA的饱和。允许洗脱样品干燥和结晶。
- 收集质谱使用的MALDI-prOTOF 1 PerkinElmerSciex质谱仪或提供高分辨率MALDI质谱8,9,10。
- 查看光谱和提取使用PerkinElmerSciex TOFworks质谱仪制造商提供的软件或软件的峰面积。
- 由MS验证可用质谱系统提供串联质谱功能的反应产物。
4。数据分析
- 为了弥补使用重型甲醛(图1B)创建的同位素重叠,确定峰面积(A)的13 C 2和碳同位素的相对论比e来控制样品,没有经过还原甲基化(图2A)的单一同位素峰。
公式1:一个一个12C / 13C2 = R 1
公式2:13C4 / 12C = R 2
这将使你在这些同位素的国家现有的肽比(R 1和R 2)具体的实验和质谱仪。 - 使用下列公式R 1和R 2值量化的脱甲基反应样品的每个修改状态(图2B)在现有的肽的相对量:
公式3:H3K4me2 = 1
4式:H3K4me = 2 - R 1(1)
5式:H3K4的= 3 - R 2(1) - [R 1(2 - R 1(1))]
5。代表结果
使用类LSD1,我们将展示为量化的MassSQUIRM的有效性赖氨酸去甲基化。如在协议文本部分中所述,我们进行了125纳克类LSD1和0.25微克二甲基化组蛋白H3肽(ARTKme2QTARKSTGGKAPRKQLYK-生物素)1去甲基化酶的检测。受到控制和脱甲基反应样品还原甲基化和MALDI质谱。控制反应,没有经过还原甲基表明,这种肽的典型的同位素信封和峰面积为方程1和2(图2A)。为脱甲基反应的质谱峰面积为方程3-5(图2B)提供。使用控制和脱甲基反应的峰面积,我们确定的肽类LSD1去甲基化给下面的反应产物:33.7%二甲基Lys4,单甲基Lys4 42.3%,24%未甲基化的赖氨酸4。参考布莱尔等。类LSD1去甲基化酶活性的全面分析以及抑制剂的研究6。注意变量的变化,如反应时间,酶浓度和底物浓度将允许活动的深入分析。

图1。MassSQUIRM的概述。 (一)从组蛋白H3 N端肽是被联合国(绿色),单(红色),或DI-赖氨酸4甲基化(蓝色)。在每个肽的化学成分的变化,导致差电离量化复杂。 (二)还原甲基化转换所有赖氨酸残基二甲基状态,这会导致所有肽同样电离。使用甲醛还原甲基化反应中的重,允许保留原甲基化的身份。开放各界表明轻甲基化而封闭的圆圈表示沉重的甲基化。 从布莱尔等人,2011年 6 修饰 。
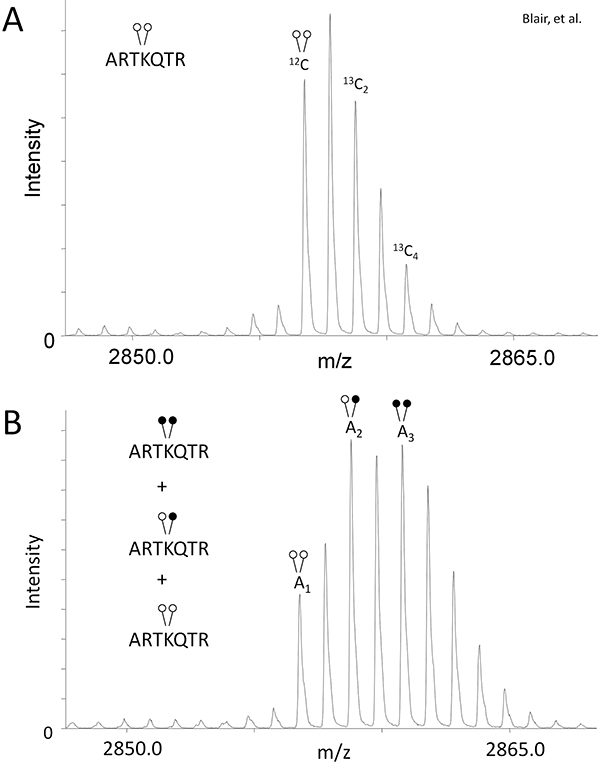
figurE 2。MassSQUIRM可以用来量化差异甲基化肽。 (一)一个DI-甲基化合成蛋白H3胜肽(ARTKme2QTARKSTGGKAPRKQLYK-生物素)分析用质谱和相对单一同位素峰的峰比值分别为R 1和R 2方程1和2中指出。 (二)同一合成肽孵育在37°C与125纳克在两个小时的脱甲基缓冲区类LSD1样品受到MassSQUIRM分析。一个叠嶂的混居的代表在图1B所示的三个不同的甲基化状态。单一同位素峰下面积分别记为1,A 2和A 3。峰面积被用来在方程3-5。开放各界表明轻甲基化而封闭的圆圈表示沉重的甲基化。 从布莱尔等修饰。 2011年 6。
讨论
MassSQUIRM是一种廉价单和二甲基赖氨酸去甲基化酶活性的综合分析和定量的方法。 MassSQUIRM提供定量不仅对产品的反应,但也为中间体。此法可以用来作为一个强大的工具学习类LSD1和其他组蛋白去甲基化酶的机制。这也将是有益的许多新发现PHF8如赖氨酸去甲基酶的分类,可用于某些甲基酶。
披露声明
我们什么都没有透露。
致谢
我们感谢质谱支持UAMS蛋白质组学设施。由NIH资助P20RR015569,P20RR016460和R01DA025755为这个项目提供了资金。
材料
| Name | Company | Catalog Number | Comments |
| 试剂名称 | 公司 | 目录编号 | |
| 类LSD1 | BPS的生物科技公司 | 50100 | |
| H3K4me2-肽素 | 准备中的房子 | 没有 | |
| 波罗斯R2的20微米微珠 | 应用生物系统公司 | 1-1129-06 | |
| C 18 ZipTip | Millipore公司 | ZTC18M | |
| 三氟乙酸(TFA) | 热 | 28904 | |
| 乙腈 | 费舍尔 | A996 | |
| 2,5 - 二羟基苯甲酸 | 西格玛 | 85707 | |
| 硼烷二甲胺 | 西格玛 | 180238 | |
| 同位素重D 2 -甲醛 | 剑桥同位素实验室 | DLM-805-20 | |
| 三 | 费舍尔 | BP154 | |
| 氯化钾 | 费舍尔 | BP366 | |
| MgCl 2的 | 费舍尔 | BP214 | |
| 甘油 | 费舍尔 | 33国集团 | |
| 蚁酸 | Fluka公司 | 06440 | |
| 甲醇 | 费舍尔 | A452 | |
| 钠 - 磷酸 | 费舍尔 | BP329 | |
| SpeedVac浓缩 | 学者 | DNA110 | |
| MALDI - prOTOF质谱仪和TOFworks的软件的 | PerkinElmerSciex | 没有 |
参考文献
- Goldberg, A. D., Allis, C. D., Bernstein, E. Epigenetics: a landscape takes shape. Cell. 128, 635-638 (2007).
- Kouzarides, T. Chromatin modifications and their function. Cell. 128, 693-705 (2007).
- Blair, L. P., Cao, J., Zou, M. R., Sayegh, J., Yan, Q. Epigenetic Regulation by Lysine Demethylase 5 (KDM5) Enzymes in Cancer. Cancers (Basel). 3, 1383-1404 (2011).
- Cole, P. A. Chemical probes for histone-modifying enzymes. Nat. Chem. Biol. 4, 590-597 (2008).
- Shi, Y. Histone demethylation mediated by the nuclear amine oxidase homolog LSD1. Cell. 119, 941-953 (2004).
- Blair, L. P. MassSQUIRM: An assay for quantitative measurement of lysine demethylase activity. Epigenetics. 6, 490-499 (2011).
- Rayment, I. Three-dimensional structure of myosin subfragment-1: a molecular motor. Science. 261, 50-508 (1993).
- Collom, S. L., Jamakhandi, A. P., Tackett, A. J., Radominska-Pandya, A., Miller, G. P. CYP2E1 active site residues in substrate recognition sequence 5 identified by photoaffinity labeling and homology modeling. Arch. Biochem. Biophys. 459, 59-69 (2007).
- Gradolatto, A. Saccharomyces cerevisiae Yta7 Regulates Histone Gene Expression. Genetics. 179, 291-304 (2008).
- Taverna, S. D. Yng1 PHD finger binding to histone H3 trimethylated at lysine 4 promotes NuA3 HAT activity at Lysine 14 of H3 and transcripiton at a subset of targeted ORFs. Mol. Cell. 24, 785-796 (2006).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。