È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Applicazione di MassSQUIRM per le misure quantitative delle attività demetilasi lisina
In questo articolo
Riepilogo
Presentiamo un metodo per utilizzare spettrometria di massa MALDI e chimica metilazione riduttiva per quantificare variazioni di metilazione lisina.
Abstract
Recentemente, i regolatori epigenetici sono stati scoperti come i giocatori chiave per molte diverse malattie 1-3. Come risultato, questi enzimi sono obiettivi primari di studi piccola molecola e sviluppo 4 farmaci. Molti regolatori epigenetici hanno solo recentemente è stato scoperto e sono ancora in fase di classificazione. Tra questi enzimi sono demethylases lisina che rimuovono gruppi metilici da lisine degli istoni ed altre proteine. A causa della natura di questa nuova classe di enzimi, pochi saggi sono stati sviluppati per studiare la loro attività. Questo è stato un blocco stradale sia per la classificazione e lo studio elevato throughput di demethylases istoni. Attualmente, saggi demetilasi esistono pochi. Quelli che esistono tendono ad essere di natura qualitativa e non possono contemporaneamente discernere tra i diversi stati di metilazione della lisina (Nazioni Unite-, mono-, di-e tri-). La spettrometria di massa è comunemente usato per determinare l'attività demetilasi ma gli attuali dosaggi di massa spettrometria di farenon affrontare se peptidi differenzialmente metilati ionizzare in modo diverso. Ionizzazione dei peptidi metilati differenziale rende il confronto stati metilazione difficile e certamente non quantitativa (Figura 1A). Così test disponibili non sono ottimizzati per l'analisi completa delle attività demetilasi.
Qui si descrive un metodo chiamato MassSQUIRM (spettrometria di massa quantificazione utilizzando isotopica metilazione riduttiva) che si basa sulla metilazione riduttiva di gruppi amminici con formaldeide deuterato di costringere tutti lisine essere di-metilato, rendendoli così in sostanza la stessa specie chimiche e quindi ionizzare l' stesso (figura 1B). La differenza chimica soltanto dopo la metilazione riduttiva è idrogeno e deuterio, che non influenza l'efficienza di ionizzazione MALDI. Il dosaggio MassSQUIRM è specifico per i prodotti di reazione demetilasi con un-lisine, mono-o di-metilati. Il saggio è applicabile anche a methyltransferases lisina dando la same prodotti di reazione. Qui, si usa una combinazione chimica di metilazione riduttiva e spettrometria di massa MALDI per misurare l'attività di LSD1, uno demetilasi lisina in grado di rimuovere di-e mono-metil gruppi, su un substrato peptidico sintetico 5. Questa analisi è semplice e facilmente suscettibili di qualsiasi laboratorio con accesso ad uno spettrometro di massa MALDI in laboratorio o attraverso un servizio di proteomica. Il saggio deve ~ 8 volte la gamma dinamica, ed è facilmente scalabile in formato piastra 5.
Protocollo
Questo protocollo è stato modificato da Blair et al. 6.
1. LSD1 demetilazione Assay
- In un volume finale di 20 pl, combinare 125 ng LSD1 ricombinante con 0,25 mg di metil-istone H3 peptide (ARTKme2QTARKSTGGKAPRKQLYK-biotina) in tampone demetilasi (50 mM Tris-Cl pH 8,5, 50 mM KCl, 5 mM MgCl 2, 5% glicerolo). Inoltre, eseguire un controllo solo peptide senza enzima. Il controllo è per la sezione 3 e non necessita di metilazione riduttiva.
- Incubare per 2 ore a 37 ° C.
- Per raccogliere i peptidi, aggiungere 8 microlitri Poros R2 sfere 20 micron a ciascun campione e agitare per 15 minuti a temperatura ambiente. Preparare perline Poros sospendendo 1 volume di perline con 1 volume di metanolo, e quindi aggiungere 10 volumi di 5% di acido formico / 0,2% TFA.
- Condizione due C 18 ZipTips aspirando 20 microlitri di 0.1% TFA nella punta e spingendolo indietro con una pipetta. Fate questo due volte con lo 0,1% TFA, quattro volte con il 70% acetonitrile / 0,1% TFA, e quattro volte di nuovo con 0,1% TFA.
- Caricare i due campioni (controllo e la reazione demetilasi) sulle condizionata C 18 ZipTips pipettando l'impasto tallone dietro la resina C 18 nelle ZipTips e spingendo il volume attraverso con una pipetta.
- Lavare ZipTips due volte con 20 pl di 0,1% TFA.
- Eluire in 40 pl di 70% acetonitrile / 0,1% TFA.
- Liofilizzato ogni eluente completamente con un concentratore SpeedVac.
2. Metilazione riduttiva
Questa porzione del protocollo viene modificato da Blair et al. E Rayment et al. 6,7.
- Risospendere la reazione demetilasi in 100 pl di tampone fosfato 50 mM, pH 7,4.
- Per la reazione demetilasi, aggiungere 8 pl di 15 mg / mL borano dimetilammina. Borano dimetilammina viene sciolto in tampone fosfato 50 mM, pH 7,4, per dare 15 mg / ml di soluzione concentrata.Questo dovrebbe essere fresco.
- Per la reazione demetilasi, aggiungere 16 pl di 250 mM formaldeide deuterato. La 250 formaldeide mM deuterato si prepara diluendo lo stock in H 2 0. Questo dovrebbe essere fresco.
- Incubare a 4 ° C per 2 ore.
- Ripetere i punti 2.2, 2.3 e 2.4.
- Ripetere il punto 2.2.
- Incubare a 4 ° C per circa 15 ore.
- Per il campione demetilasi, aggiungere 12,5 microlitri di 1 M Tris, pH 7,4 per placare la reazione riduttiva metilazione.
3. Spettrometria di massa MALDI
- Aggiungi Poros R2 20 perline micron alla reazione demetilasi, e raccogliere il ZipTips come descritto nei passaggi 1,3-1,6.
Nota: Re-sospendere il controllo in 100 pl di tampone fosfato 50 mM, pH 7,4 e trattare come l'altro campione partendo dal punto 3,1.
- Eluire il controllo e campioni di reazione demetilasi dalle ZipTips su una piastra di campioni utilizzando MALDI 2 microlitri di 33% saturared 2,5-diidrossibenzoico acido. Per preparare il 33% di acido 2,5-diidrossibenzoico, una soluzione satura di acido 2,5-diidrossibenzoico in 70% acetonitrile / 0,1% TFA e quindi diluire al 33% saturo in 70% acetonitrile / 0,1% TFA. Lasciare i campioni eluiti ad asciugare e cristallizzare.
- Raccogliere spettri di massa con un MALDI-prOTOF spettrometro di massa PerkinElmerSciex o disponibili ad alta risoluzione spettrometro di massa MALDI 8,9,10.
- Vedi spettri ed estrarre le aree dei picchi che utilizzano software PerkinElmerSciex TOFworks o software fornito dal produttore spettrometro di massa.
- Verificare prodotti di reazione da MS 2 con un sistema di spettrometria di massa disponibile fornendo funzionalità di spettrometria di massa tandem.
4. Analisi dei dati
- Al fine di compensare la sovrapposizione isotopici creati utilizzando formaldeide pesante (Figura 1B), determinare l'area di picco (A) rapporto di 13 C 2 e 13 C 4 isotopi relative per il picco monoisotopico per il campione di controllo, che non ha subito metilazione riduttiva (Fig. 2A).
Eq 1: A 13C2 / A 12C = r 1
Eq 2: A 13C4 / A 12C = r 2
Questo vi darà il rapporto di peptide esistenti in questi stati isotopica (r 1 e r 2) specifico per il tuo esperimento e spettrometro di massa. - Utilizzare la r 1 r 2 e valori nelle seguenti formule per quantificare la quantità relativa di peptide esistente in ogni stato modificazione del campione reazione demetilasi (Fig. 2B):
Eq 3: H3K4me2 = A 1
Eq 4: H3K4me = A 2 - r 1 (A 1)
Eq 5: H3K4 = A 3 - r 2 (A 1) - [r 1 (A 2 - r 1 (A 1))]
5. Risultati rappresentativi
Utilizzando LSD1, ci mostrano l'efficacia di MassSQUIRM per quantificaredemetilazione lisina. Come descritto nella sezione Testo protocollo, abbiamo eseguito un saggio demetilasi con 125 ng di LSD1 e 0,25 mg di metil-istone H3 peptide (ARTKme2QTARKSTGGKAPRKQLYK-biotina). Il controllo e la reazione demetilasi campioni sono stati sottoposti a metilazione riduttiva e spettrometria di massa MALDI. La reazione di controllo che non ha subito metilazione riduttiva mostrato la busta isotopico tipico per questo peptide e fornito aree di picco di equazioni 1 e 2 (Figura 2A). Lo spettro di massa per la reazione demetilasi fornito aree di picco per 3-5 equazioni (Figura 2B). Utilizzando le aree dei picchi dal controllo e reazione demetilasi, abbiamo determinato che LSD1 demetilato il peptide per dare i seguenti prodotti di reazione: 33,7% di metil-Lys4, 42,3% mono-metil Lys4, e 24% non-metilata Lys 4. Fare riferimento a Blair et al. Per l'analisi completa di LSD1 attività demetilasi nonché studi inibitori 6. Si noti che le variabili cambiano come tempo di reazione, enzimaticaconcentrazione e la concentrazione del substrato consentirà una approfondita analisi delle attività.

Figura 1. Panoramica MassSQUIRM. (A) Un peptide N-terminale da istone H3 viene mostrato come un-(verde), mono-(rosso), o di-(blu) metilato a lisina 4. Variazione della composizione chimica di ciascun peptide porta a ionizzazione differenziale rendere complesso quantificazione. (B) metilazione riduttiva converte tutti i residui di lisina al di-metil stato che fa sì che tutti i peptidi per ionizzare in modo simile. L'uso di formaldeide pesante nella reazione di metilazione riduttiva permette la conservazione dell'identità della metilazione originale. Cerchi aperti indicano metilazione luce, mentre i cerchi chiusi indicano metilazione pesante. Modificato da Blair et al. 2011 6.
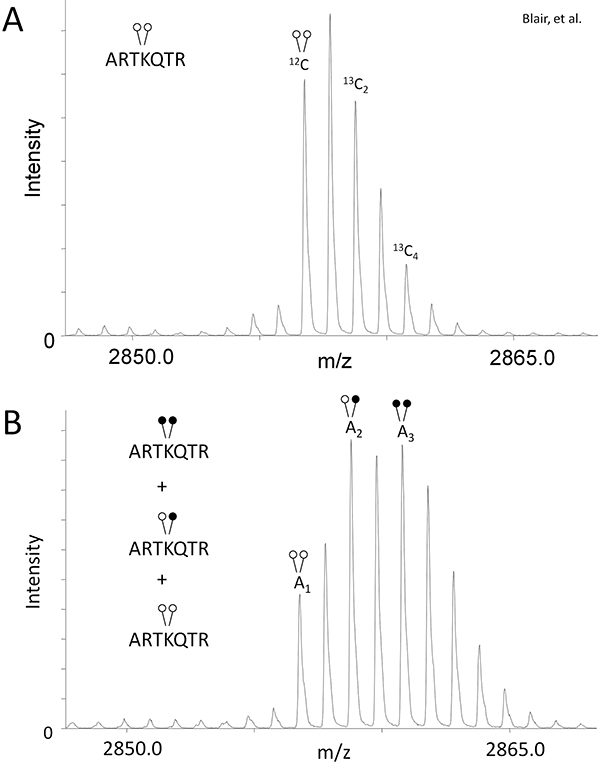
Figure 2. MassSQUIRM può essere utilizzato per quantificare peptidi differenzialmente metilati. (A) di A-metilato sintetico istone H3 peptide (ARTKme2QTARKSTGGKAPRKQLYK-biotina) è stato analizzato mediante spettrometria di massa e rapporti di picco relativa al picco monoisotopico sono state rilevate come r 1 ed r 2 in equazioni 1 e 2. (B) Lo stesso peptide sintetico è stato incubato con 125 ng LSD1 in tampone demetilasi per due ore a 37 ° C. I campioni sono stati poi sottoposti ad analisi MassSQUIRM. Una popolazione mista di sovrapposizione picchi rappresentano tre diversi stati di metilazione come si vede nella figura 1B. Le aree sotto i picchi sono stati notati monoisotopico come A 1, A 2 e A 3. Aree dei picchi sono stati utilizzati nelle equazioni 3-5. Cerchi aperti indicano metilazione luce, mentre i cerchi chiusi indicano metilazione pesante. Modificato da Blair et al. 2011 6.
Discussione
MassSQUIRM è un metodo poco costoso e quantitative per l'analisi complessiva dell'attività di demethylases lisina coinvolti in mono-e di-metilazione. MassSQUIRM quantificazione offre non solo del prodotto della reazione, ma anche per gli intermedi. Questa analisi può essere utilizzato come un potente strumento per studiare il meccanismo di demethylases istone LSD1 e altri. Sarà anche utile per classificare molti enzimi recentemente scoperti demetilasi lisina come PHF8 e potrebbe essere utilizzato per alcuni ...
Divulgazioni
Non abbiamo nulla da rivelare.
Riconoscimenti
Ringraziamo il Fondo UAMS Proteomics supporto spettrometria di massa. Il finanziamento per questo progetto è stato fornito da sovvenzioni NIH P20RR015569, P20RR016460 e R01DA025755.
Materiali
| Name | Company | Catalog Number | Comments |
| Nome del reattivo | Azienda | Numero di catalogo | |
| LSD1 | BPS Biosciences | 50100 | |
| H3K4me2-biotina peptide | preparato in-house | nessuno | |
| POROS R2 20 perline micron | Applied Biosystems | 1-1129-06 | |
| C 18 ZipTip | Millipore | ZTC18M | |
| Acido trifluoroacetico (TFA) | Thermo | 28904 | |
| acetonitrile | Pescatore | A996 | |
| 2,5-diidrossibenzoico acido | Sigma | 85707 | |
| Borano dimetilammina | Sigma | 180238 | |
| isotopicamente pesante d 2-formaldeide | Cambridge Isotope Laboratories | DLM-805-20 | |
| Tris | Pescatore | BP154 | |
| KCl | Pescatore | BP366 | |
| MgCl 2 | Pescatore | BP214 | |
| Glicerina | Pescatore | G33 | |
| Acido formico | Fluka | 06440 | |
| Metanolo | Pescatore | A452 | |
| Na-fosfato | Pescatore | BP329 | |
| SpeedVac Concentrator | Dotto | DNA110 | |
| MALDI-prOTOF Spettrometro di massa e TOFworks software | PerkinElmerSciex | nessuno |
Riferimenti
- Goldberg, A. D., Allis, C. D., Bernstein, E. Epigenetics: a landscape takes shape. Cell. 128, 635-638 (2007).
- Kouzarides, T. Chromatin modifications and their function. Cell. 128, 693-705 (2007).
- Blair, L. P., Cao, J., Zou, M. R., Sayegh, J., Yan, Q. Epigenetic Regulation by Lysine Demethylase 5 (KDM5) Enzymes in Cancer. Cancers (Basel). 3, 1383-1404 (2011).
- Cole, P. A. Chemical probes for histone-modifying enzymes. Nat. Chem. Biol. 4, 590-597 (2008).
- Shi, Y. Histone demethylation mediated by the nuclear amine oxidase homolog LSD1. Cell. 119, 941-953 (2004).
- Blair, L. P. MassSQUIRM: An assay for quantitative measurement of lysine demethylase activity. Epigenetics. 6, 490-499 (2011).
- Rayment, I. Three-dimensional structure of myosin subfragment-1: a molecular motor. Science. 261, 50-508 (1993).
- Collom, S. L., Jamakhandi, A. P., Tackett, A. J., Radominska-Pandya, A., Miller, G. P. CYP2E1 active site residues in substrate recognition sequence 5 identified by photoaffinity labeling and homology modeling. Arch. Biochem. Biophys. 459, 59-69 (2007).
- Gradolatto, A. Saccharomyces cerevisiae Yta7 Regulates Histone Gene Expression. Genetics. 179, 291-304 (2008).
- Taverna, S. D. Yng1 PHD finger binding to histone H3 trimethylated at lysine 4 promotes NuA3 HAT activity at Lysine 14 of H3 and transcripiton at a subset of targeted ORFs. Mol. Cell. 24, 785-796 (2006).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon