Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Применение MassSQUIRM для количественного измерения Лизин активность деметилазы
В этой статье
Резюме
Мы представляем способ использования MALDI масс-спектрометрии и восстановительной химии метилирования количественного изменения в лизин метилирования.
Аннотация
В последнее время эпигенетические регуляторы были обнаружены играть ключевую роль в самых различных заболеваний 1-3. В результате, эти ферменты являются главными целями для небольших исследований молекулы и 4 разработки лекарственных препаратов. Многие эпигенетические регуляторы только недавно были обнаружены и до сих пор в процессе классификации. Среди этих ферментов лизин деметилаз которые удаляют из метильных групп лизина на гистонов и других белков. В связи с новым характером этого класса ферментов, несколько анализов были разработаны с целью изучения их деятельности. Это был дорогой блок как классификация и высокой пропускной способностью исследования гистонов деметилаз. В настоящее время очень мало тестов деметилазы существует. Те, которые действительно существуют, как правило, качественный характер и не может одновременно различать между различными государствами метилирование лизина (не-, моно-, ди-и три-). Масс-спектрометрия широко используется для определения деметилазы деятельности, но текущий масс-спектрометрических анализов делатьне затрагивает ли дифференциально метилированные пептидов ионизируют по-разному. Дифференциальная ионизации метилированных пептидов делает сравнение метилирования государства трудно и, конечно, не количественный (рис. 1А). Таким образом, доступные анализы не были оптимизированы для всестороннего анализа деметилазы деятельности.
Здесь мы опишем метод MassSQUIRM (масс-спектрометрического количественного использовании изотопных восстановительного метилирования), который основан на редуктивных метилирование аминогрупп с дейтерированных формальдегид, чтобы заставить всех лизинов быть ди-метиловым, что делает их по существу и того же вида химической ионизации и, следовательно, же (рис. 1б). Разница лишь в химической после восстановительного метилирования водорода и дейтерия, которая не влияет на эффективность MALDI ионизации. Анализ MassSQUIRM является специфическим для деметилазы продуктов реакции с не-, моно-или ди-метилированных лизина. Анализ также применим к лизин метилтрансферазы давая SAМеня продуктов реакции. Здесь мы используем сочетание восстановительной химии метилирования и MALDI масс-спектрометрии для измерения активности LSD1, лизин деметилазы способен удалять ди-и моно-метильных групп, на синтетической подложке пептид 5. Этот тест является простым и легко поддаются любой лаборатории, имеющие доступ к спектрометр MALDI масс в лаборатории или через средства протеомики. Анализ имеет ~ 8-кратный динамический диапазон и легко масштабируемой, чтобы пластины формата 5.
протокол
Этот протокол изменяется от Блэра и соавт. 6.
1. LSD1 Деметилирование Пробирной
- В конечном объеме 20 мкл, объединить 125 нг рекомбинантного LSD1 с 0,25 мкг ди-метил гистона H3 пептида (ARTKme2QTARKSTGGKAPRKQLYK-биотин) в буфере деметилазы (50 мМ Трис-Cl рН 8,5, 50 мМ KCl, 5 мМ MgCl 2, 5% глицерин). Кроме того, выполнение пептид только контроль без фермента. Управление по разделу 3 и не требует восстановительного метилирования.
- Выдержите в течение 2 часов при температуре 37 ° C.
- Для сбора пептидов, добавить 8 мкл R2 ПОРОС 20 бусин микрон до каждого образца и агитировать за 15 минут при комнатной температуре. Подготовить ПОРОС бисером, приостановив 1 том из бисера с 1 объема метанола, а затем добавить 10 томов 5% муравьиной кислоты / 0,2% TFA.
- Условие две С 18 по ZipTips аспирационных 20 мкл 0,1% TFA в наконечник и толкая его обратно с пипетки. Сделайте это в два раза с 0,1% TFA, в четыре раза с 70% ацетонитрил / 0,1% TFA, и в четыре раза раз с 0,1% TFA.
- Загрузите двух образцов (контроль и деметилазы реакции) на условный C 18 ZipTips с помощью пипетки борт шлама за C 18 смолой ZipTips и нажав объеме через с пипетки.
- Вымойте ZipTips дважды с 20 мкл 0,1% TFA.
- Элюции в 40 мкл 70% ацетонитрил / 0,1% TFA.
- Lyophilize каждый элюента с полностью SpeedVac концентратора.
2. Восстановительное метилирование
Эта часть протокола изменяется от Блэра и соавт. И Рэймент соавт. 6,7.
- Повторно приостановить деметилазы реакцию в 100 мкл 50 мМ фосфатного буфера, рН 7,4.
- Для деметилазы реакции, добавить 8 мкл 15 мг / мл боран диметиламин. Боран диметиламин растворяют в 50 мМ фосфатный буфер, рН 7,4, чтобы дать 15 мг / мл маточного раствора.Это должно быть свежим.
- Для деметилазы реакции, добавить 16 мкл 250 мМ дейтерированных формальдегида. 250 мМ дейтерированного формальдегида готовят путем разбавления акции H 2 0. Это должно быть свежим.
- Инкубировать при температуре 4 ° С в течение 2 часов.
- Повторите шаги 2.2, 2.3 и 2.4.
- Повторите шаг 2.2.
- Инкубировать при температуре 4 ° С в течение ~ 15 часов.
- Для деметилазы образца, добавьте 12,5 мкл 1М Трис, рН 7,4, чтобы утолить восстановительной реакции метилирования.
3. MALDI масс-спектрометрии
- Добавить ПОРОС R2 20 бусин микрон до деметилазы реакции, и собирать на ZipTips как описано в пунктах 1.3-1.6.
Примечание: Re-приостановить контроль в 100 мкл 50 мМ фосфатного буфера, рН 7,4 и лечения, как и другие образцы, начиная с шага 3.1.
- Элюции контроля и деметилазы образцы реакции ZipTips на тарелку MALDI примера с использованием 2 мкл 33% насыщенияг 2,5-диоксибензойной кислоты. Для приготовления 33% 2,5-диоксибензойной кислоты, сделать насыщенный раствор 2,5-диоксибензойной кислоты в 70% ацетонитрил / 0,1% TFA и затем разбавить до 33% насыщенных в 70% ацетонитрил / 0,1% TFA. Позвольте элюированных образцы высохнуть и кристаллизоваться.
- Соберите масс-спектры использования PerkinElmerSciex MALDI-масс-спектрометр prOTOF или доступны с высоким разрешением MALDI масс-спектрометра 8,9,10.
- Вид спектра и извлечь максимум области использования PerkinElmerSciex TOFworks программного обеспечения или программного обеспечения, средств массовой производитель спектрометра.
- Проверка реакции продуктов MS 2 с доступным масс-спектрометрического системы, обеспечивающей тандемной масс-спектрометрического возможности.
4. Анализ данных
- Для того, чтобы компенсировать изотопические перекрытия создаются с помощью тяжелой формальдегид (рис. 1б), определить площадь пика (А) соотношение 13 C 2 и 13 C 4 изотопов релятивистскихе моноизотопный пик для контрольного образца, который не претерпел восстановительного метилирования (рис. 2).
Уравнению 1: 13C2 / 12С = R 1
Уравнение 2: 13C4 / 12С = R 2
Это даст вам отношение пептида, существующие в этих изотопических состояниях (R 1 и R 2), специфичные для вашего эксперимента и масс-спектрометра. - С помощью R 1 и R 2 значения в формулы для количественной оценки относительного количества пептида, существующих в каждой модификации государства в деметилазы пример реакции (рис. 2):
Уравнения 3: H3K4me2 = 1
Уравнения 4: H3K4me = 2 - R 1 (1)
Уравнение 5: H3K4 = 3 - R 2 (1) - [г 1 (2 - R 1 (1))]
5. Представитель Результаты
Использование LSD1, мы покажем эффективность MassSQUIRM для количественноголизин деметилирования. Как описано в разделе текста протокола, мы провели анализ деметилазы 125 нг LSD1 и 0,25 мкг ди-метил гистона H3 пептида (ARTKme2QTARKSTGGKAPRKQLYK-биотин). Контроль и деметилазы образцы реакции подвергались восстановительного метилирования и MALDI масс-спектрометрии. Контроль реакции, которые не прошли восстановительного метилирования показали типичные изотопного конверт для этого пептида и при условии, пик областях для уравнений 1 и 2 (рис. 2A). Масс-спектр для деметилазы реакции при условии площадей пиков для уравнений 3-5 (рис. 2В). Использование площадей пиков от контроля и деметилазы реакции, мы решили, что LSD1 деметилированные пептид дать следующие продукты реакции: 33,7% ди-метил Lys4, 42,3% моно-метил Lys4, а 24% не-метилированных Lys 4. Обратитесь к Блэру и соавт. Для полного анализа LSD1 деметилазы деятельности, а также ингибитор исследования 6. Обратите внимание, что изменения переменных, таких как время реакции, ферментконцентрация и концентрация субстрата позволит углубленный анализ деятельности.

Рисунок 1. MassSQUIRM обзор. (A) N-концевой пептид из гистонов H3 показано как ООН (зеленый), моно-(красный), или ди-(синий) метилированный на лизин 4. Изменение химического состава каждого пептида приводит к дифференциальной ионизации делает количественного комплекса. (B) Восстановительное метилирование преобразует все остатков лизина в ди-метил состояние, которое вызывает все пептиды для ионизации аналогично. Использование тяжелой формальдегида в восстановительной реакции метилирования позволяет сохранение идентичности оригинального метилирования. Открытые кружки света метилирования, а закрытые кружки тяжелой метилирования. Изменения от Блэра и соавт. 2011 6.
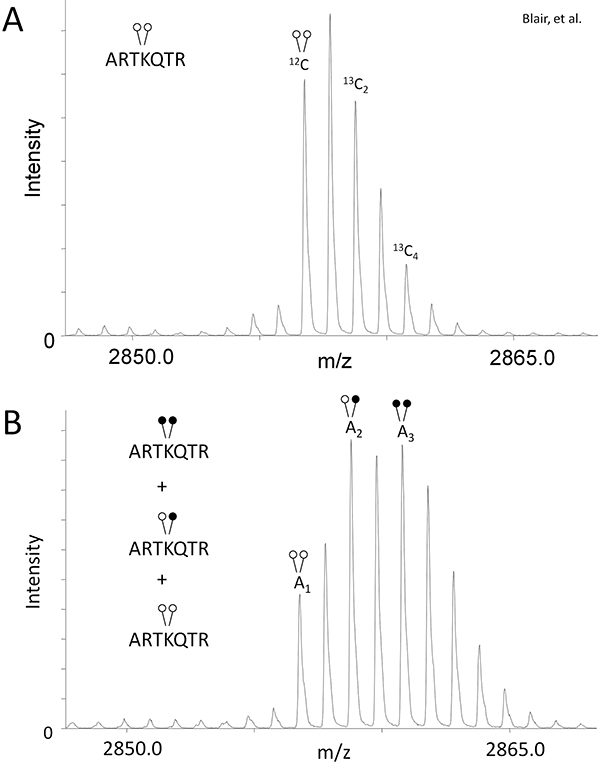
Figurе 2. MassSQUIRM может быть использована для количественного дифференциально метилированных пептидов. (A) ди-метилированных синтетический пептид гистона H3 (ARTKme2QTARKSTGGKAPRKQLYK-биотин) анализировали с помощью масс-спектрометрии и пик отношения по отношению к моноизотопный пик был отмечен как R 1 и R 2 в уравнениях 1 и 2. (Б) то же синтетический пептид инкубировали с 125 нг LSD1 в деметилазы буфера в течение двух часов при температуре 37 ° C. Образцы были подвергнуты MassSQUIRM анализа. Смешанным населением перекрывающихся пиков представляет собой три различные состояния метилирования, как показано на рисунке 1b. Площади под моноизотопный пиков были отмечены как 1, 2 и 3. Пик областях были использованы в уравнениях 3-5. Открытые кружки света метилирования, а закрытые кружки тяжелой метилирования. Изменения от Блэра и соавт. 2011 6.
Обсуждение
MassSQUIRM является недорогим и количественных методов для всестороннего анализа деятельности лизин деметилаз участие в моно-и ди-метилирования. MassSQUIRM предлагает количественного не только продукт реакции, но и для промежуточных. Этот анализ может быть использован как мощный инструмент в ...
Раскрытие информации
Нам нечего раскрывать.
Благодарности
Мы благодарим фонд UAMS протеомики для масс-спектрометрического поддержки. Финансирование этого проекта было предоставлено грантов NIH P20RR015569, P20RR016460 и R01DA025755.
Материалы
| Name | Company | Catalog Number | Comments |
| Название реагента | Компания | Номер по каталогу | |
| LSD1 | БПС Biosciences | 50100 | |
| H3K4me2-биотин пептид | подготовлен в доме | никто | |
| ПОРОС R2 20 микрон бисера | Applied Biosystems | 1-1129-06 | |
| C 18 ZipTip | Millipore | ZTC18M | |
| Трифторуксусной кислоты (ТФК) | Thermo | 28904 | |
| ацетонитрил | Рыбак | A996 | |
| 2,5-диоксибензойной кислоты | Сигма | 85707 | |
| Боран диметиламин | Сигма | 180238 | |
| изотопно тяжелой D 2-формальдегидные | Кембридж изотопных лабораторий | DLM-805-20 | |
| Трис | Рыбак | BP154 | |
| KCl | Рыбак | BP366 | |
| MgCl 2 | Рыбак | BP214 | |
| Глицерин | Рыбак | G33 | |
| Муравьиная кислота | Fluka | 06440 | |
| Метанол | Рыбак | A452 | |
| Na-фосфат | Рыбак | BP329 | |
| SpeedVac концентратор | Ученый | DNA110 | |
| MALDI-масс-спектрометр prOTOF и TOFworks программного обеспечения | PerkinElmerSciex | никто |
Ссылки
- Goldberg, A. D., Allis, C. D., Bernstein, E. Epigenetics: a landscape takes shape. Cell. 128, 635-638 (2007).
- Kouzarides, T. Chromatin modifications and their function. Cell. 128, 693-705 (2007).
- Blair, L. P., Cao, J., Zou, M. R., Sayegh, J., Yan, Q. Epigenetic Regulation by Lysine Demethylase 5 (KDM5) Enzymes in Cancer. Cancers (Basel). 3, 1383-1404 (2011).
- Cole, P. A. Chemical probes for histone-modifying enzymes. Nat. Chem. Biol. 4, 590-597 (2008).
- Shi, Y. Histone demethylation mediated by the nuclear amine oxidase homolog LSD1. Cell. 119, 941-953 (2004).
- Blair, L. P. MassSQUIRM: An assay for quantitative measurement of lysine demethylase activity. Epigenetics. 6, 490-499 (2011).
- Rayment, I. Three-dimensional structure of myosin subfragment-1: a molecular motor. Science. 261, 50-508 (1993).
- Collom, S. L., Jamakhandi, A. P., Tackett, A. J., Radominska-Pandya, A., Miller, G. P. CYP2E1 active site residues in substrate recognition sequence 5 identified by photoaffinity labeling and homology modeling. Arch. Biochem. Biophys. 459, 59-69 (2007).
- Gradolatto, A. Saccharomyces cerevisiae Yta7 Regulates Histone Gene Expression. Genetics. 179, 291-304 (2008).
- Taverna, S. D. Yng1 PHD finger binding to histone H3 trimethylated at lysine 4 promotes NuA3 HAT activity at Lysine 14 of H3 and transcripiton at a subset of targeted ORFs. Mol. Cell. 24, 785-796 (2006).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены