需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
DNA的亲和纯化的芯片(DAP芯片)方法来确定目标基因的细菌双组分调控系统
摘要
这个视频文章介绍了一种体外基于微阵列方法来确定两个组件系统响应监管机构的靶基因和结合位点。
摘要
在体内方法,如芯片芯片是用于确定转录因子基因总体目标行之有效的方法。然而,它们的用途有限的探索细菌双组分调控系统具有未知功能的激活条件。这种系统只调节转录时的独特信号的存在被激活。由于这些信号往往是未知的,这个视频文章中描述的体外基于微阵列方法可用于确定基因目标和响应监管机构的结合位点。在一个测序基因组的任何生物体可以用于任何纯化的调节器将该DNA-亲和纯化的芯片的方法。该协议涉及允许纯化标记的蛋白结合到剪切基因组DNA,然后亲和纯化的蛋白结合的DNA,然后将DNA与杂交到自定义平铺阵列的荧光标记。前述可用于优化测定法SPECIFI步骤Ç监管部门也有所说明。由阵列数据分析所产生的峰被用来预测的结合位点基序,其然后通过实验验证。动机的预测可以进一步用于确定密切相关的物种的直系同源响应调节剂的靶基因。我们证明了该方法的适用性通过确定目标基因和结合位点基序,从而预测功能,用于在环境细菌脱硫寻常 Hildenborough一个sigma54依赖性反应调节DVU3023。
引言
细菌的生存和发展的能力严重依赖于他们如何能够感知并在其环境中的扰动做出响应,而这又取决于它们的信号转导系统。信令系统的细菌进行编码的数量被称为它的"微生物IQ",可以是其环境的两个变异和其检测多个信号的能力并微调其反应1的指示。二组分信号转导系统(TCS)是用于由细菌最普遍的信令系统,并且它们包括感测外部信号,并通过磷酸化的效应反应器(RR)2发送一个组氨酸激酶(HK)的。反射器可以具有多种输出域,因此不同的效应器模式,但最常见的反应是通过DNA结合结构域1的转录调控。感测到的信号和输精管的相应功能吨多数个TCS的仍下落不明。
虽然体内方法,如芯片芯片通常用于测定的转录因子3基因组结合位点,它们只能用于细菌的双组分体系的RR,如果活化条件或信号是已知的。通常情况下,环境因素,激活一个TCS是难以确定比他们的靶基因。 在体外微阵列此处描述的基础的检测,可用于有效和快速地确定该基因的目标和预测的TCS的功能。该测定法利用了一个事实,即反射器能够被磷酸化,从而使用小分子献血者像乙酰磷酸4 体外活化的优势。
在该方法中,命名为DAP芯片用于DNA-亲和纯化的芯片( 图1)中,感兴趣的RR基因的克隆与在大肠杆菌中的His-标签大肠杆菌和纯化后的蛋白标记允许双向第二,以剪切基因组DNA。该蛋白结合的DNA,然后通过亲和纯化富集,富集和输入DNA被扩增,荧光标记,汇集在一起,并杂交到被定制到感兴趣的有机体( 图1)由一个平铺阵列。微阵列实验如有文物,因此额外的步骤来优化检测。一个这样的步骤是试图确定一个目标为RR正在研究利用电泳迁移率变动分析(EMSA)(见工作流在图2中)。然后,下面的结合到基因组DNA和DAP的步骤中,蛋白结合和输入DNA是通过定量PCR检查,以查看是否阳性靶富集的蛋白结合部分相对于输入级分,从而确认为最佳的结合条件RR( 图2)。阵列杂交后,对数据进行分析,发现较高的强度信号的指示的基因位点的峰,其中蛋白h广告的约束。功能可预测的基础上获得的基因目标的路由反射器。靶基因位点被用于预测的结合位点基序,然后将其实验使用EMSAs( 图2)进行验证。功能预测和靶基因的RR然后可以延伸到密切相关,通过扫描这些基因对于相似的结合基序( 图2)进行编码的直向同源物种的RR。民主行动党芯片法可以为TCS那里以前有没有提供了丰富的信息。该方法也可用于任何转录调节子,如果蛋白质可被纯化和DNA结合条件可以被确定,并且针对感兴趣与基因组序列的任何有机体提供。
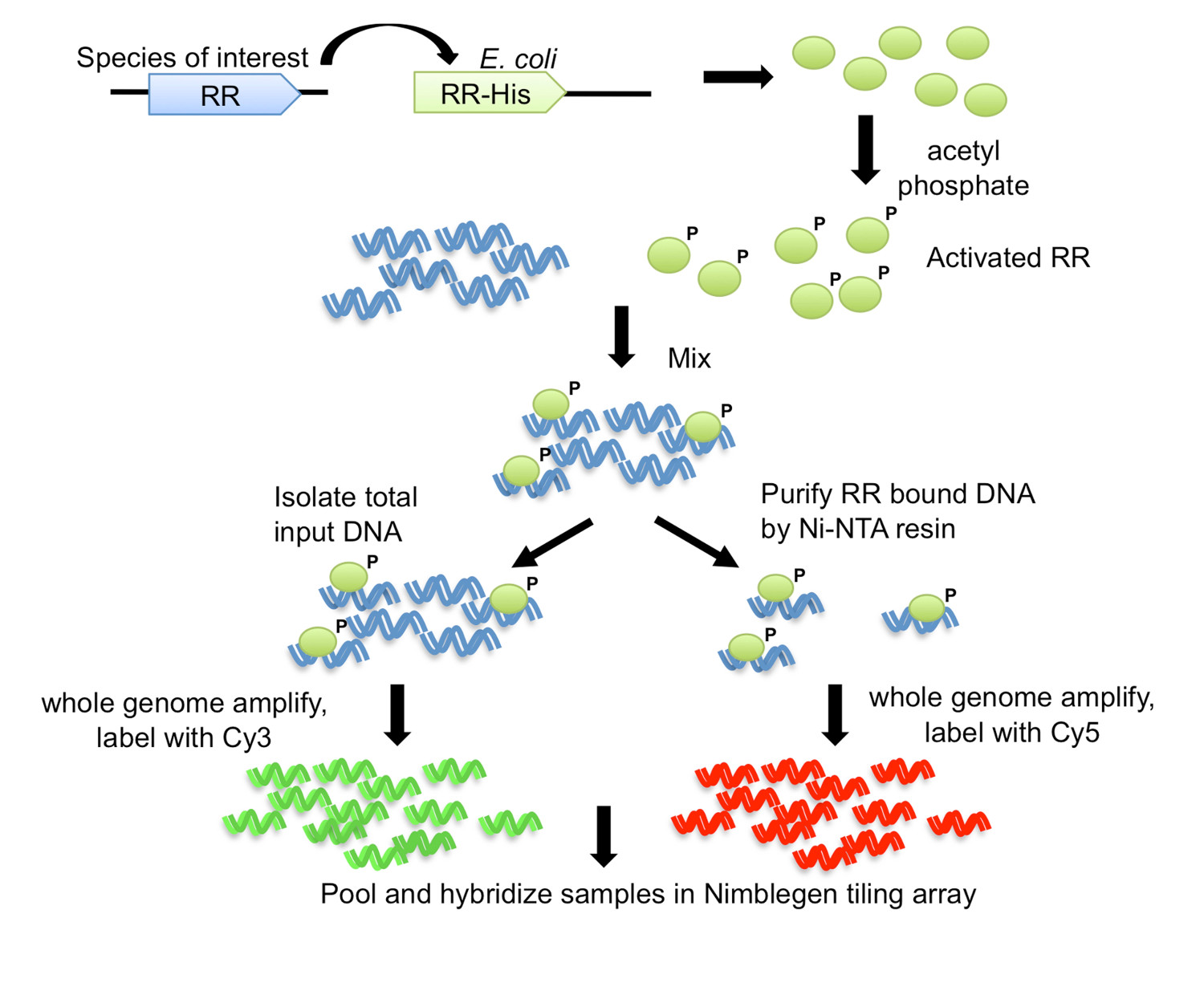
图1的DNA-亲和纯化的芯片(DAP-芯片)战略7。从感兴趣的有机体RR基因被克隆与羧基末端His-标签到大肠杆菌大肠杆菌表达菌株。纯化的His-标记的蛋白是由磷酸与乙磷酸激活,并与剪切基因组DNA混合。结合反应的等分试样保存为输入DNA,而其余的是使用Ni-NTA树脂进行亲和纯化。输入和RR结合的DNA是扩增整个基因组中,并标记有Cy3和Cy5分别。标记的DNA被汇集在一起,并杂交到一个平铺阵列中,然后进行分析以确定该基因的目标。图修改并使用Creative Commons授权从7转载。
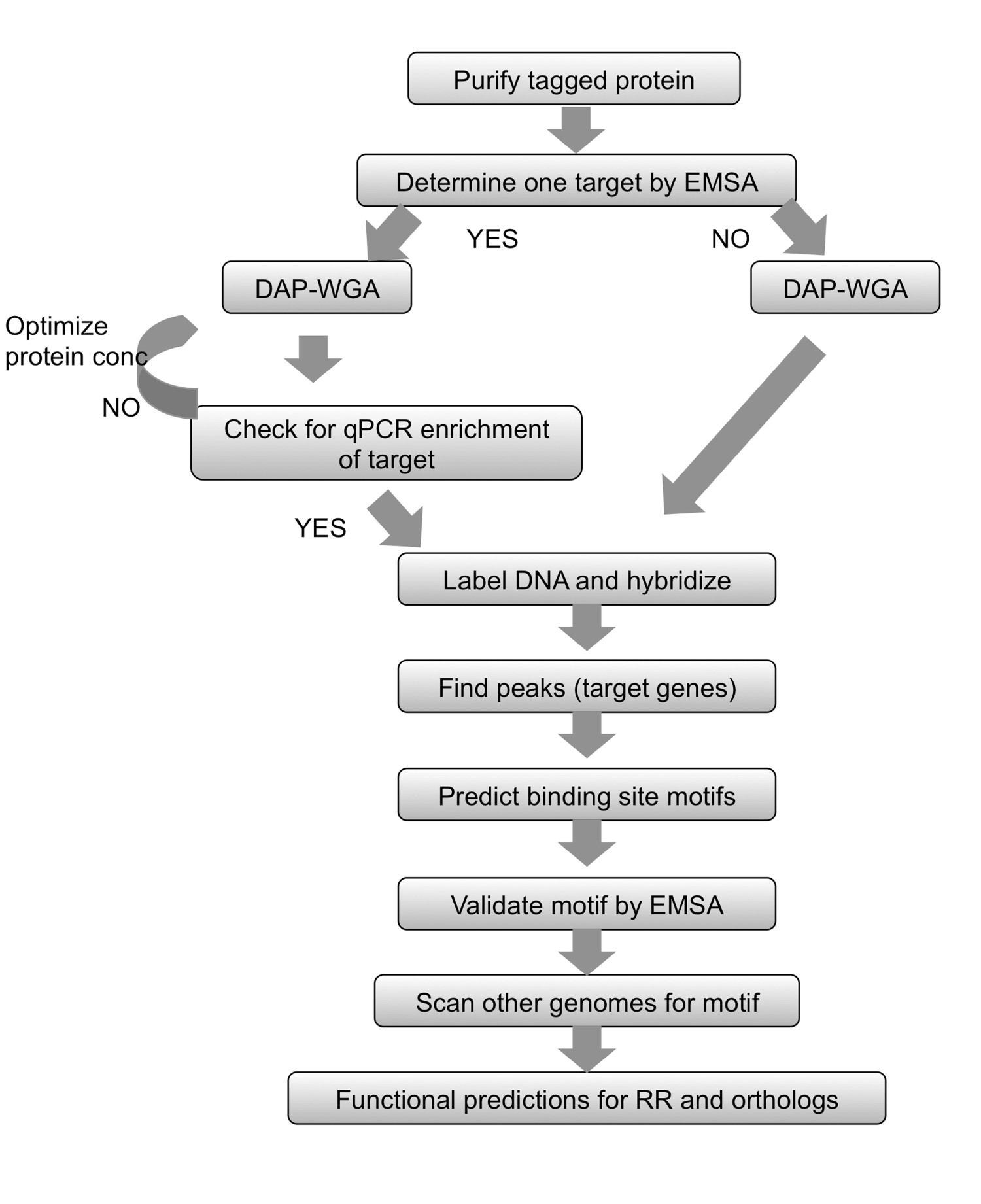
图2总结工作流程。对于任何纯化标签proteiN,利用EMSA确定目标开始。让蛋白结合的基因组DNA,然后DNA的亲和净化(DAP)和全基因组扩增(WGA)的丰富和输入的DNA。如果一个基因靶标是已知的,可使用定量PCR,以确保已知靶标富集的蛋白质结合的级分。如果没有目标可确定,直接进行DNA标记和阵列杂交。如果富集通过qPCR无法观察到的,然后重复蛋白基因组DNA结合,并使用不同的蛋白量DAP-WGA的步骤。利用阵列分析发现峰,并将其映射到目标基因。使用靶基因的上游区域,预测的结合位点基序。实验使用EMSAs验证图案。使用主题来扫描相关品种正在研究编码RR的同源基因的基因组,并预测目标基因在这些物种也是如此。根据所获得的基因目标,RR和其同源基因的生理功能可预测。图修改,并使用广告C转载ommons从7许可。
研究方案
注意:下面的协议是专为测定从细菌脱硫寻常 Hildenborough的RR DVU3023的靶基因。它可以适用于任何感兴趣的其它转录调节子。
1,克隆,纯化RR
- 克隆RR基因,特别是DVU3023,从D。寻常 Hildenborough到大肠杆菌表达载体中,使得该基因是C-端His-标记的和表达是一个T7启动子的控制下。
注意:几个克隆方法可用于与由研究者决定。可替代的亲和标签,也可以使用。 - 变换表达构建到大肠杆菌大肠杆菌 BL21(DE3)表达菌株。
- 长大了1升BL21表达菌株在37℃在对数中期,IPTG加至0.5mM至诱导蛋白表达。继续生长在室温下放置24小时。
- 细胞离心机在5000×g离心10英里n在4°C。将细胞再悬浮于裂解缓冲液(磷酸盐20mM的钠,pH 7.4,500 mM氯化钠,40 mM咪唑,1毫克/毫升溶菌酶,1 Benzonase核酸)。使用法国媒体在4℃裂解细胞
- 离心溶胞产物以15,000×g离心30分钟,在4℃下过滤用0.45μm的注射器过滤器。
- 使用10毫升的洗涤缓冲液(20mM磷酸钠,pH 7.4,500 mM氯化钠,40 mM咪唑)的FPLC仪器洗1毫升的Ni Sepharose柱。
- 加载裂解液上柱。用20ml洗涤缓冲液洗柱。
- 洗脱DVU3023使用0-100%洗脱缓冲液梯度(20mM的磷酸钠,pH 7.4,500 mM氯化钠,500mM咪唑)。
- 加载上脱盐柱洗脱级分,并洗用脱盐缓冲液(20mM磷酸钠pH值7.4,100mM的NaCl)中。
- 在高分子量截留离心过滤器以浓缩蛋白离心机蛋白质样品。添加DTT至0.1毫米和甘油在-20℃下50%和店铺蛋白
注意:纯化方法将需要在研究单独优化每个蛋白质。
2,确定目标基因的RR采用电泳迁移率变动分析(EMSA)
- PCR扩增400 bp的区域的候选靶基因DVU3025的上游,使用D。寻常 Hildenborough基因组DNA作为模板,以及未标记的正向引物和反向5'-生物素标记的引物。
注:提示选择一个候选靶基因:通常的RR绑定自己的基因/操纵子的上游地区,或者他们可以调节近端编码基因。如果RR在其他物种同源基因,看是否有保守在附近的基因。替代的方法包括基于调节子的预测( 例如 ,RegPrecise 5),或预测sigma54-依赖型启动子6为反射器本身是sigma54依赖选择的候选基因。 - 运行PCR产物在1%琼脂糖凝胶上,切出400 Bp大小的产物,并使用凝胶提取纯化试剂盒纯化DNA。
- 混合DVU3023蛋白(0.5皮摩尔)与在10mM Tris盐酸,pH值7.5,50mM的氯化钾,5毫摩尔MgCl 2,1mM的DTT,25%甘油和1微克/毫升的聚dI.dC(非100 fmol的生物素化的DNA底物的在20微升的总体积特异性竞争DNA)。还设置了无蛋白作为对照的反应。孵育反应在室温下在板凳上20分钟。
- 注:这些都是上市及其他反应组分可根据感兴趣的RR被加入标准条件。如果激活需要,加上50毫米乙酰磷酸反应(通常的RR将绑定在体外的DNA无需激活)。
- 在100伏30分钟预运行一个小型预制构件6%聚丙烯酰胺-0.5X TBE的0.5倍TBE缓冲液凝胶。加入5微升的5倍上样缓冲液(0.1%溴酚蓝,0.1%二甲苯青,在1x TBE 30%甘油)就到克结合反应和负载18微升的反应埃尔。在100V 2小时运行。
注意:为了避免过热,这可能会导致该蛋白质-DNA复合物的解离,用运行缓冲液,使得大部分的凝胶是由缓冲绝缘填充外缓冲室中的电泳系统。另外,该凝胶可以在4°C下运行 - 切电荷的尼龙膜对凝胶的大小和浸泡在0.5×TBE为至少十分钟。从盒中取出凝胶。切割任何脊关闭凝胶和夹持浸泡在0.5×TBE 2厚滤纸,凝胶和膜,并放置一个半干印迹装置内部并在20 V 30分钟。
- 将膜商用UV光交联剂仪器内部和时间设置为3分钟。
注意:该膜可以立即继续下一步骤之前被存储在室温下晾干数天。 - 使用市售的化学发光检测试剂盒,它采用链霉亲和辣根的过氧化SE共轭发展印迹按照制造商的说明。
- 图像使用计算机的印迹挂接到一个装有CCD摄像机和查找在DNA底物的流动性的转变在RR中,这表明在RR结合被测试的DNA的存在。
注:蛋白质-DNA转移的特异性可以通过在反应中过量的未标记的竞争DNA,这将消除或减少可见移位进行检测。
3,后基因组DNA-蛋白质结合验证靶向序列捕获
- 基因组DNA-蛋白质结合反应
- 在采用微尖与1秒的9低幅度脉冲,以在两者之间2秒的间隙在1.5 ml微量离心管中剪切基因组DNA到500碱基对的平均尺寸由声波处理100微升基因组DNA(浓度100〜200毫微克/微升)每个。
注意:如果该DNA飞溅到该管的两侧,每3个脉冲后停止旋转的内容。所使用的确切条件将与所使用的超声波仪仪器而有所不同。经剪切的DNA片段的范围可以从100〜1000碱基,但平均大小应在400〜600 bp之间。过多的剪切可能导致更少的完整的结合位点,以及过多或过少的剪切将在下游全基因组扩增步骤影响库制剂。 - 在10mM的Tris-HCl pH为7.5,1mM的DTT,50mM的氯化钾,5毫摩尔MgCl 2,25%甘油和50mM乙酰磷酸混合2-3的剪切基因组DNA微克与RR蛋白(0.5皮摩尔DVU3023的)。孵育反应在25℃下30分钟。转移10微升该反应到1.5ml管中,标号作为输入的DNA。
注意:乙酰磷酸盐加入通过在体外磷酸化来激活蛋白质。磷酸化刺激DNA结合,但许多的RR也体外结合DNA无需激活7。在加入反应蛋白的量将取决于蛋白质制备的活性。在EMSA可用于选择imize这一数额。
- 在采用微尖与1秒的9低幅度脉冲,以在两者之间2秒的间隙在1.5 ml微量离心管中剪切基因组DNA到500碱基对的平均尺寸由声波处理100微升基因组DNA(浓度100〜200毫微克/微升)每个。
- 亲和纯化蛋白结合的DNA
- 加入30微升的Ni-NTA琼脂糖树脂的0.6 ml离心管中。离心100×g离心1分钟在底部收集树脂,并取出上清液。加入100μl洗涤缓冲液(10毫米的Tris-HCl,pH值7.5,5毫米MgCl 2,50毫米氯化钾,25%甘油),轻弹管混合,并离心100×g离心2分钟。除去上清液。
- 加入剩余的90微升的结合反应的对洗涤的Ni-NTA树脂和孵化在旋转摇床上30分钟。
- 离心100×g离心2分钟,然后取出上清液(未结合DNA)。加入100μl洗涤缓冲液,轻弹管混合内容,离心100×g离心2分钟。除去上清液。重复洗涤步骤两次。
- 加35微升洗脱缓冲液(磷酸盐20mM的磷酸钠缓冲液pH为8,500 mM氯化钠,500mM咪唑),以树脂和通过涡旋混合。孵育在板凳上在室温下5分钟。离心机在100×g离心2分钟。将上清液转移至新的1.5mL管中,并标记为蛋白结合的DNA部分。
- 还加35微升的洗脱缓冲液对输入的DNA。
- 净化的输入,并使用PCR纯化试剂盒的蛋白结合的DNA的级分。
- 全基因组扩增的输入和蛋白质结合的DNA样本。
- 加10微升的输入和蛋白结合的DNA来分离PCR管。加入1μl的10倍破碎缓冲液,2微升的文库制备缓冲液,和1μl的库稳定溶液向每个样品。通过涡旋搅拌均匀。热在热循环仪在95℃下进行2分钟,并且冰上冷却。
注意:DNA的起始量应至少为10毫微克,以避免引入的扩增偏差。 - 加入1μl文库制备酶,吹打混匀,孵育在热循环仪在16°C/20分钟,24°C/20分钟,37℃20分钟,75℃/ 5分钟,并保持在4°C。
- 在每个试管中,添加47.5微升水,7.5微升10×扩增预混液和5微升聚合酶。搅拌均匀,热在95℃/ 3分钟,之后20个循环的94°C/15秒,65℃/ 5分钟。保持反应在4°C。
- 使用PCR纯化试剂盒纯化扩增的DNA样品,并测量DNA浓度用分光光度法。
- 加10微升的输入和蛋白结合的DNA来分离PCR管。加入1μl的10倍破碎缓冲液,2微升的文库制备缓冲液,和1μl的库稳定溶液向每个样品。通过涡旋搅拌均匀。热在热循环仪在95℃下进行2分钟,并且冰上冷却。
- 验证蛋白结合的DNA靶向富集使用qPCR
- 设计定量PCR引物扩增200个基点的EMSA验证靶基因(DVU3025)使用任何可自由查看的引物设计软件的上游地区。
- 设置一式三份qPCR反应与每个引物组各DNA模板。准备一个主结构为每个引物组。 1X主混合物含有10微升的2倍的SYBR Green qPCR的组合,0.5μM的每种引物和水,共18微升。分装18μL每孔母液96孔PCR板的。
- Dilut辰扩增和纯化的输入和蛋白质结合的DNA样品,以5纳克/微升的水和作为DNA模板使用。加入2μlDNA模板的孔中。
- 具有超清晰的定量PCR封口膜封板,在离心机降速板在200 XG / 1分钟。将板在实时定量PCR机。循环使用相关联的qPCR软件如下:95℃/ 1分钟,95°C/10秒40个循环,59°C/15秒,70°C/35秒。
- 还设置了一式三份的qPCR反应中,用引物来扩增不相关的基因(阴性对照)的上游区域。
- 计算ΔCŦ减去从输入的DNA蛋白结合的DNA的C T值。计算靶基因的倍浓缩的蛋白质结合的DNA为2ΔCSki。
注意:如果目标上游区域富集在蛋白结合的DNA与输入的DNA,它表明结合反应和亲和纯化分别SUCCessful。进行到步骤4,如果没有观察到靶上游区域的富集,用不同量的蛋白质重复该结合反应(步骤3.1)。
4,标记DNA和阵列杂交
- 标签输入的DNA与Cy5标记Cy3和富集的DNA
OTE:Cy3和Cy5染料是光敏感,应小心,避免光线照射到最低限度。- 将1微克的DNA与40微升Cy3/Cy5-labeled九聚体,并调整音量至80微升水。
- 热在黑暗中(在热循环仪)变性在98℃持续10分钟。冰上2分钟迅速冷却。
- 加入2μlKlenow聚合酶(3'-5'外切- ,50,000单位/毫升),5 mM的dNTPs和8μl水到每个反应,拌匀,并在37℃下2小时在黑暗中(在热循环)。
- 加EDTA至50mM,使反应停止和NaCl溶液至0.5M。
- 样品转移到1.5 ml离心管续癌宁0.9倍体积的异丙醇,孵育在暗处放置10分钟,12,000×g离心10分钟。颗粒应该是粉红色的Cy3标记的DNA和蓝色为Cy5标记的DNA。
- 用80%乙醇(500微升)和12,000×g离心2分钟,洗涤沉淀物。空气干燥颗粒在黑暗5-10分钟。
注:颗粒可以保存在-20℃。 - 重新悬浮颗粒在25μl水。测量DNA浓度分光光度计。
- 一起池6微克每对在黑暗低热离心机在1.5ml管和真空干燥的的Cy3和Cy5-标记的DNA(包括离心分离机盖,如果它是透明的)的。
注:颗粒可被存储于-20℃,直到准备用于杂交。
- 芯片杂交
- 把杂交系统上之前3-4小时内使用,并设定温度为42℃。
- 准备杂交液母液,这样1X解决方案包含11.8微升有杂交2倍zation缓冲液,4.7μL杂交A组份和0.5微升对齐寡。
- 悬浮颗粒在5μl水。加入13微升这样的搭配到样品。涡旋15秒,孵育在95℃下在干燥的浴5分钟。保持样品在42℃的杂交系统,直到准备装载。
- 制备微阵列载玻片,混合器组件,根据制造商的方案。
- 将杂交系统中的混音器,滑动组件。负载16微升样品进入填充口,密封带粘接膜的端口,关闭系统中的混合,并杂交16-20小时,在42℃下
- 制备1X洗涤缓冲液I(250ml)中,二(50毫升)和III(50毫升)稀释10倍的市售缓冲器中的水,并以每添加DTT至1mM。温暖的缓冲I至42°C。
- 滑动混频器滑入一个反汇编工具,以及含有地方温暖缓冲一剥混频器关闭一个培养皿内,在大力SH摄影功能的反汇编工具的手。
- 将滑动到一个容器中,用50ml洗涤缓冲液I和用手剧烈振摇2分钟。
- 转移滑动到第二容器,用50ml洗涤缓冲液II,剧烈摇晃手1分钟。
- 转让滑动到第三容器用50ml洗涤缓冲液III,并用手剧烈振荡15秒。
- 快速涂抹在纸巾上的幻灯片的边缘,并将其放置在玻片架。离心机在200×g离心2分钟以干燥的幻灯片。放置在幻灯片的情况下,用铝箔包起来,并存储在干燥器中。
- 扫描阵列
- 根据仪器的指示将滑动的微阵列扫描仪中。
- 使用扫描仪的软件来设置的波长为532毫微米= Cy3和635毫微米= Cy5标记,以及350和400之间的初始光电倍增管增益。
- 预览幻灯片定位在幻灯片的数组。选择用于扫描的阵列区域。
- 扫描阵列,并调整光电倍增管的设置使得阵列的特点是多为黄色。直方图应该显示叠加红色和绿色的曲线或彼此接近越好。该曲线应结束上述饱和10-5标准计数。
- 分别节省532和635 nm的图像。
- 阵列数据分析
- 导入图像到一个数组中的分析软件。使用该软件,创建两对报告(一)为每个数组(Cy3和Cy5图像)。创建使用对文件缩放的日志2比文件。使用日志2比文件来搜索使用500个基点的滑动窗口峰。地图峰位点的基因目标的上游地区。
- 检查阳性靶(使用EMSA测定和定量PCR,DVU3025在这个例子中证实)会出现在顶部的山峰。
注意:如果正目标不是最佳命中当中,有可能是下consi在RR减额有几个基因靶和复制DAP-芯片可以进行和共同所有的重复的峰,可用于生成目标列表。
5,结合位点的Motif预测和验证
- 检索上游区域(400bp)的序列上的靶基因通过DAP芯片分析生成的。通过微生物对这些序列适用MEME在线(meme.nbcr.net)来预测图案。
注意:增强子结合蛋白,如sigma54依赖性调节器可以绑定几百个碱基对的起始位点的上游。对于其它转录调节,较短的区域,如200 bp的上游就足够了。 - 设计顶部和包含该预测的结合位点基序由任一末端10个碱基侧翼底链DNA寡聚物。责令顶链5'生物素化。
- 在混合比为1:1.5的顶部和底部链的寡核苷酸在10mM Tris盐酸,pH值8.0,1mM的EDTA和50mM NaCl的总反应体积为20μl的PCR管中。热至95℃5分钟,在热循环仪中,随后缓慢冷却至25℃。稀释10倍,作为EMSA野生型双链DNA底物使用。
- 制备的修饰底物使用与上述相同的步骤,但设计的低聚物进行4-6取代的结合基序的保守碱基。
- 成立了结合反应与RR和野生型或修饰底物,并检查使用EMSA如步骤2所述。
- 注意:如果在RR结合野生型衬底,但不能修改一项的预测动机进行了验证。
母题的6。其他相关细菌物种保护
- 选择包含DVU3023的直系同源物的兴趣基因组。获得序列和注释文件从NCBI网站基因组。
- 使用编程语言如Perl编写,将使用来自DAP芯片目标的基序序列建立一个POS脚本银行足球比赛加权矩阵,并使用矩阵得分出现在其他测序的基因组类似的图案。
结果
上述方法用于确定模型中硫酸盐还原菌脱硫寻常 Hildenborough 7的RR的全球性的靶基因。这种微生物具有大量的TCS的超过70的RR为代表,说明各种可能的信号,它检测并响应, 在体内对这些信令系统的功能分析是很难,因为它们的信号来执行,因此其活性的条件是未知的。这里对DAP芯片法是用来确定该靶基因,从而预测可能的功能为代表的RR DVU3023。
DVU3023是?...
讨论
这里所描述的DAP芯片方法成功地用于确定多个路由反射器在脱硫寻常 Hildenborough 7其中之一是这里示出为具有代表性的结果的靶基因。对于RR DVU3023,选择一个候选基因的目标是简单的。 DVU3025位于紧接在RR基因的下游,并在RR和靶基因是保守的几个脱硫弧菌种,另外DVU3025具有预测的sigma54依赖型启动子。在EMSA提供了一种简单的方法来快速测试在RR为结合到候选靶基因,并且还允?...
披露声明
作者没有利益冲突披露。
致谢
我们感谢陈小姐为她准备的视频拍摄帮助和演示该技术。由ENIGMA进行这项工作:生态系统和网络集成与基因和分子组装(http://enigma.lbl.gov),一个科学的对焦区域计划在劳伦斯伯克利国家实验室,通过科学,生物办公室的办公室,并支持环境研究能源的合同号DE-AC02-05CH11231美系,。
材料
| Name | Company | Catalog Number | Comments |
| HisTrapFF column (Ni-Sepharose column) | GE Lfe Sciences, Pittsburgh, PA, USA | 17-5255-01 | |
| Akta explorer (FPLC instrument) | GE Lfe Sciences, Pittsburgh, PA, USA | ||
| HiPrep 26/10 Desalting column | GE Lfe Sciences, Pittsburgh, PA, USA | 17-5087-01 | |
| Qiaquick Gel extraction kit | Qiagen Inc, Valencia, CA, USA | 28704 | |
| Biotin-labeled oligonucleotides | Integrated DNA Technologies | N/A | |
| 6% polyacrylamide-0.5x TBE precast mini DNA retardation gel | Life Technologies, Grand Island, NY, USA | EC63652BOX | Alternately, you can pour your own gel. |
| Nylon membrane | EMD Millipore, Billerica, MA, USA | INYC00010 | |
| Trans-Blot SD Semi-dry electrophoretic transfer cell | Biorad, Hercules, CA, USA | 170-3940 | |
| Extra thick blot paper, 8 x 13.5 cm | Biorad, Hercules, CA, USA | 170-3967 | |
| UV crosslinker Model XL-1000 | Fisher Scientific | 11-992-89 | |
| Nucleic Acid chemiluminescent detection kit (Pierce) | Thermo fisher Scientific, Rockford, IL, USA | 89880 | |
| Ni-NTA agarose resin | Qiagen Inc, Valencia, CA, USA | 30210 | |
| GenomePlex Whole genome amplification kit (Fragmentation buffer, library preparation buffer, library stabilization solution, library preparation enzyme, 10x amplification master mix, WGA polymerase) | Sigma-Aldrich, St. Louis, MO, USA | WGA2-50RXN | |
| Nanodrop ND-1000 | Thermo Scientific, Wilmington, DE, USA | For quantitation of DNA | |
| Perfecta Sybr Green SuperMix, with ROX | Quanta biosciences | 95055-500 | Any Sybr Green PCR mix may be used |
| PlateMax Ultra clear heat sealing film for qPCR | Axygen | ||
| [header] | |||
| 96-well clear low profile PCR microplate | Life Technologies, Grand Island, NY, USA | PCR-96-LP-AB-C | |
| Applied Biosystems StepOne Plus Real time PCR system | Life Technologies, Grand Island, NY, USA | 4376600 | Any real time PCR system may be used |
| Qiaquick PCR purification kit | Qiagen Inc, Valencia, CA, USA | 28104 | Any PCR clean up kit may be used |
| Cy3/Cy5-labeled nonamers | Trilink biotechnologies, San Diego, CA, USA | N46-0001, N46-0002 | |
| Klenow polymerase 50,000 U/ml, 3'-5' exo- | New England Biolabs, Ipswich, MA | M0212M | |
| Hybridization system | Roche-Nimblegen, Madison, WI, USA | N/A | This company no longer makes arrays or related items, so alternate sources such as Agilent or Affymetrix will need to be used. |
| Custom printed microarrays and mixers | Roche-Nimblegen, Madison, WI, USA | N/A | |
| Hybridization kit (2x Hybridization buffer, Hybridization component A, Alignment oligo) | Roche-Nimblegen, Madison, WI, USA | N/A | |
| Wash buffer kit (10x Wash buffer I, II, III, 1 M DTT) | Roche-Nimblegen, Madison, WI, USA | N/A | |
| GenePix 4200A microarray scanner | Molecular Devices, Sunnyvale CA, USA | This model has been replaced by superior ones | |
| GenePix Pro microarray software | Molecular Devices, Sunnyvale CA, USA | ||
| Nimblescan v.2.4, ChIP-chip analysis software | Roche-Nimblegen, Madison, WI, USA | N/A | |
参考文献
- Galperin, M. Y. Diversity of structure and function of response regulator output domains. Curr Opin Microbiol. 13, 150-159 (2010).
- Casino, P., Rubio, V., Marina, A. The mechanism of signal transduction by two-component systems. Curr Opin Struct Biol. 20, 763-771 (2010).
- Aparicio, O., Geisberg, J. V., Struhl, K. Chromatin immunoprecipitation for determining the association of proteins with specific genomic sequences in vivo. Curr Protoc Cell Biol. 17 (17), (2004).
- McCleary, W. R., Stock, J. B. Acetyl phosphate and the activation of two-component response regulators. J Biol Chem. 269, 31567-31572 (1994).
- Novichkov, P. S., et al. RegPrecise web services interface: programmatic access to the transcriptional regulatory interactions in bacteria reconstructed by comparative genomics. Nucleic Acids Res. 40, 604-608 (2012).
- Studholme, D. J., Buck, M., Nixon, T. Identification of potential sigma(N)-dependent promoters in bacterial genomes. Microbiology. 146 (12), 3021-3023 (2000).
- Rajeev, L., et al. Systematic mapping of two component response regulators to gene targets in a model sulfate reducing bacterium. Genome Biol. 12, 99 (2011).
- Crooks, G. E., Hon, G., Chandonia, J. M., Brenner, S. E. WebLogo: a sequence logo generator. Genome Res. 14, 1188-1190 (2004).
- Barbieri, C. M., Mack, T. R., Robinson, V. L., Miller, M. T., Stock, A. M. Regulation of response regulator autophosphorylation through interdomain contacts. J Biol Chem. 285, 32325-32335 (2010).
- Bourret, R. B. Receiver domain structure and function in response regulator proteins. Curr Opin Microbiol. 13, 142-149 (2010).
- Gao, R., Stock, A. M. Molecular strategies for phosphorylation-mediated regulation of response regulator activity. Curr Opin Microbiol. 13, 160-167 (2010).
- Hung, D. C., et al. Oligomerization of the response regulator ComE from Streptococcus mutans is affected by phosphorylation. J Bacteriol. 194, 1127-1135 (2012).
- Ladds, J. C., et al. The response regulator Spo0A from Bacillus subtilis is efficiently phosphorylated in Escherichia coli. FEMS Microbiol Lett. 223, 153-157 (2003).
- Wen, Y., Feng, J., Scott, D. R., Marcus, E. A., Sachs, G. Involvement of the HP0165-HP0166 two-component system in expression of some acidic-pH-upregulated genes of Helicobacter pylori. J Bacteriol. 188, 1750-1761 (2006).
- Liu, X., Noll, D. M., Lieb, J. D., Clarke, N. D. DIP-chip: rapid and accurate determination of DNA-binding specificity. Genome Res. 15, 421-427 (2005).
- Gossett, A. J., Lieb, J. D. DNA Immunoprecipitation (DIP) for the Determination of DNA-Binding Specificity. CSH Protoc. 2008, (2008).
- Mascher, T., et al. The Streptococcus pneumoniae cia regulon: CiaR target sites and transcription profile analysis. J Bacteriol. 185, 60-70 (2003).
- Park, P. J. ChIP-seq: advantages and challenges of a maturing technology. Nat Rev Genet. 10, 669-680 (2009).
- Lefrancois, P., Zheng, W., Snyder, M. ChIP-Seq using high-throughput DNA sequencing for genome-wide identification of transcription factor binding sites. Methods Enzymol. 470, 77-104 (2010).
- Ho, J. W., et al. ChIP-chip versus ChIP-seq: lessons for experimental design and data analysis. BMC Genomics. 12, 134 (2011).
- Liu, X., Lee, C. K., Granek, J. A., Clarke, N. D., Lieb, J. D. Whole-genome comparison of Leu3 binding in vitro and in vivo reveals the importance of nucleosome occupancy in target site selection. Genome Res. 16, 1517-1528 (2006).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。