É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
DNA-afinidade-purificada Método Chip (DAP-chip) para determinar genes alvos para bacteriana Dois Sistemas Regulatórios componentes
Neste Artigo
Resumo
Este artigo vídeo descreve um método de microarray baseado in vitro para determinar os genes alvos e sítios de ligação para dois reguladores de resposta do sistema de componentes.
Resumo
Os métodos in vivo, tais como Chip-chip são técnicas bem estabelecidas usados para determinar genes alvos globais para fatores de transcrição. No entanto, eles são de uso limitado na exploração de dois sistemas de regulação componente bacteriano com condições de ativação descaracterizados. Tais sistemas de regular a transcrição apenas quando activada, na presença de sinais únicos. Uma vez que estes sinais são muitas vezes desconhecidos, o método de microarray com base in vitro descritos neste artigo de vídeo podem ser utilizados para determinar alvos de genes e sítios de ligação para reguladores da resposta. Este método de ADN purificado por afinidade-chip pode ser utilizado para qualquer regulador purificada em qualquer organismo com um genoma sequenciado. O protocolo envolve permitindo que a proteína purificada marcado para se ligar ao DNA genómico cortado e, em seguida, a purificação por afinidade de ADN ligado à proteína, seguido de marcação fluorescente do ADN e hibridação com uma matriz de ladrilhos costume. Precedendo passos que podem ser utilizados para optimizar o ensaio para a especificidadereguladores c também são descritos. Os picos gerados pela análise dos dados de matriz são utilizados para prever motivos sítios de ligação, que são, em seguida, validada experimentalmente. As previsões motivo pode ser ainda utilizado para determinar genes alvos dos reguladores de resposta ortólogos em espécies estreitamente relacionadas. Nós demonstramos a aplicabilidade deste método através da determinação dos genes alvos e motivos do site vinculativo e prevendo, assim, a função de um regulador de resposta DVU3023-dependente sigma54 na bactéria Desulfovibrio vulgaris Hildenborough ambiental.
Introdução
A capacidade das bactérias para sobreviver e prosperar é criticamente dependente da forma como eles são capazes de perceber e responder a perturbações em seus ambientes, e este por sua vez é dependente de seus sistemas de transdução de sinal. O número de sistemas de sinalização uma bactéria codifica tem sido chamado o seu "QI microbiana" e pode ser uma indicação de ambos variabilidade do seu ambiente e da sua capacidade de sentir múltiplos sinais e afinar a sua resposta 1. Dois sistemas de componentes de transdução de sinal (TCS) são os sistemas de sinalização mais prevalentes utilizados por bactérias, e que consistem de uma histidina-quinase (HK), que detecta o sinal externo e transmite através da fosforilação de um regulador de resposta efectora (RR) 2. RRS pode ter uma variedade de domínios de saída e, portanto, diferentes modos efetoras, mas a resposta mais comum é a regulação da transcrição por meio de um DNA domínio 1 vinculativo. Os sinais detectados e as funções correspondentes dos vast maioria dos TCSs permanecem desconhecidos.
Embora métodos in vivo, tais como Chip-chip são habitualmente usadas para a determinação de locais de ligação de factores de transcrição do genoma 3, que apenas pode ser utilizado para RRS sistema de dois componente bacteriano, se são conhecidas as condições de activação ou sinalização. Muitas vezes, os sinais ambientais que ativam a TCS são mais difíceis de determinar que os seus genes alvos. O microarray ensaio in vitro com base aqui descrita pode ser utilizada para determinar rápida e eficazmente os genes alvos e prever as funções de TCS. Este ensaio tira partido do facto de RRS pode ser fosforilada e assim activado in vitro utilizando pequenos doadores molécula como fosfato acetil 4.
Neste método, chamado DAP-chip para ADN de afinidade-purificada de chip (Figura 1), o gene da RR de interesse é clonado com uma cauda de His no E. coli, e uma proteína marcada subsequentemente purificado é permitido biª a cortado DNA genômico. O DNA ligado à proteína é então enriquecida por afinidade-purificação, o ADN enriquecido e entrada são amplificados, marcado por fluorescência, reunidas e hibridado com um conjunto azulejos que é feito para o organismo de interesse (Figura 1). Microarray experiências estão sujeitos a artefactos e, por conseguinte, são empregues passos adicionais para optimizar o ensaio. Um tal passo é a tentativa de determinar um destino para o RR em estudo utilizando ensaios de desvio de mobilidade electroforética (EMSA) (ver fluxo de trabalho na Figura 2). Em seguida, após a ligação ao ADN genómico e as etapas de DAP, o DNA ligado à proteína e de entrada são examinados por qPCR para ver se o alvo positivo é enriquecido na fracção ligada à proteína em relação à fracção de entrada, confirmando, assim, as condições óptimas de ligação para o RR (Figura 2). Após a matriz de hibridação, os dados são analisados para encontrar os picos de intensidade de sinal mais elevado, indicando locus genómico onde a proteína hanúncio vinculada. As funções podem ser previstos para o RR com base nos genes alvos obtidos. Os loci alvo genómicas são usadas para prever motivos sítios de ligação, que são, em seguida, experimentalmente validados usando EMSAs (Figura 2). As previsões funcionais e genes alvos para a RR pode então ser estendido para as espécies que codificam RRS ortólogos por inventariar os genomas de motivos de ligação semelhantes (Figura 2) intimamente relacionados. O método DAP-chip pode fornecer uma riqueza de informações para a TCS onde antes não havia nenhuma. O método também pode ser utilizado para qualquer regulador de transcrição se a proteína pode ser purificada e as condições de ligação de ADN pode ser determinada, e para todo o organismo de interesse com uma sequência de genoma disponível.
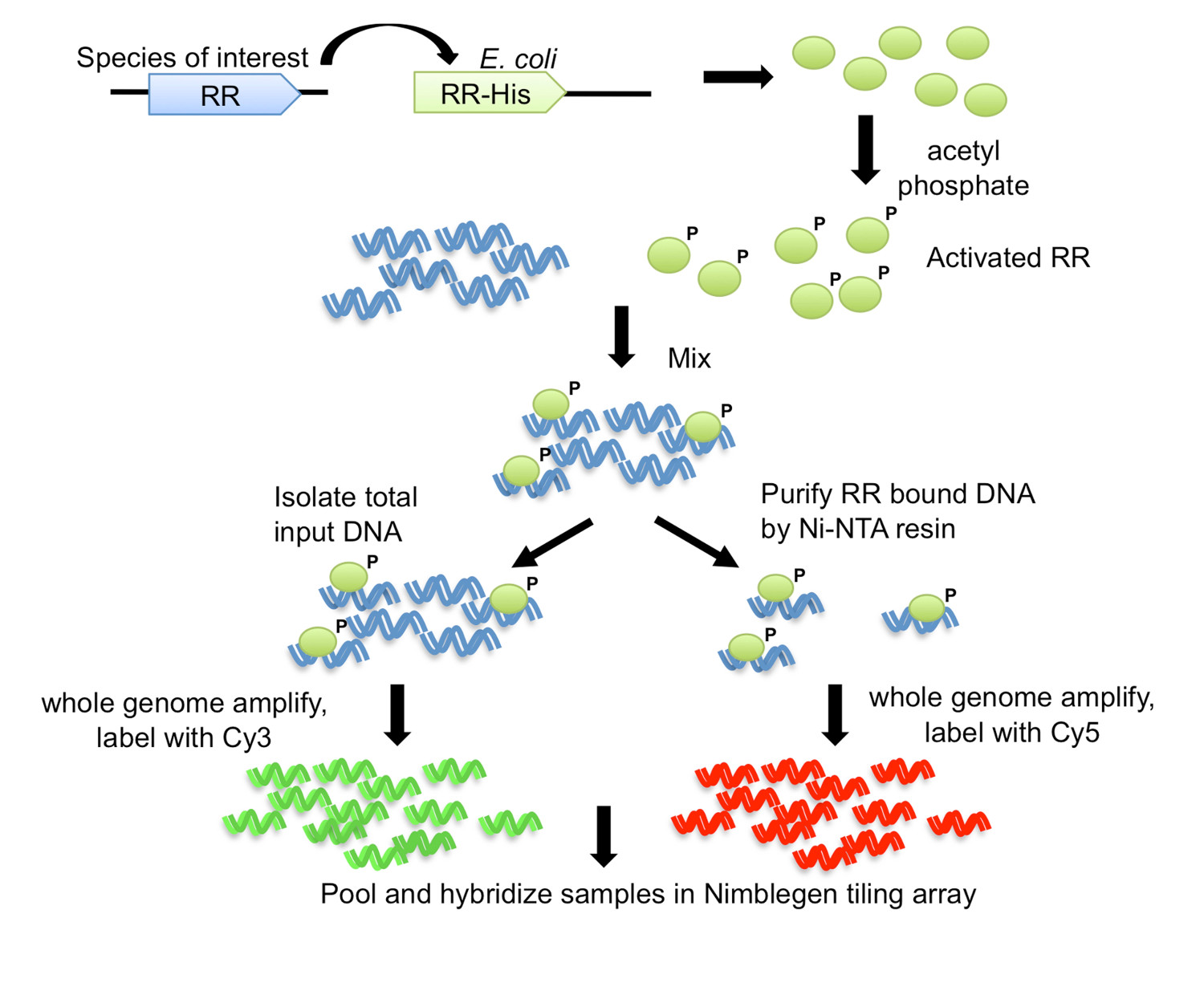
Figura 1. O chip de ADN purificado por afinidade (DAP-estratégia chip) 7. O gene RR a partir do organismo de interesse é clonado com um carboxi-terminal His-tag numa E. tensão expressão coli. Purificado proteína marcada com His é activada por fosforilação com fosfato de acetilo, e misturados com o DNA genómico cortado. Uma aliquota da reacção de ligação é guardado como o ADN de entrada, enquanto o resto é submetido a purificação por afinidade usando uma resina Ni-NTA. A entrada e o DNA ligado a RR são todo o genoma amplificado e marcado com Cy3 e Cy5, respectivamente. O ADN marcado é reunidas e hibridado com uma matriz de ladrilhos, que é então analisado para determinar os alvos do gene. Figura modificada e reimpresso com a licença creative commons de 7.
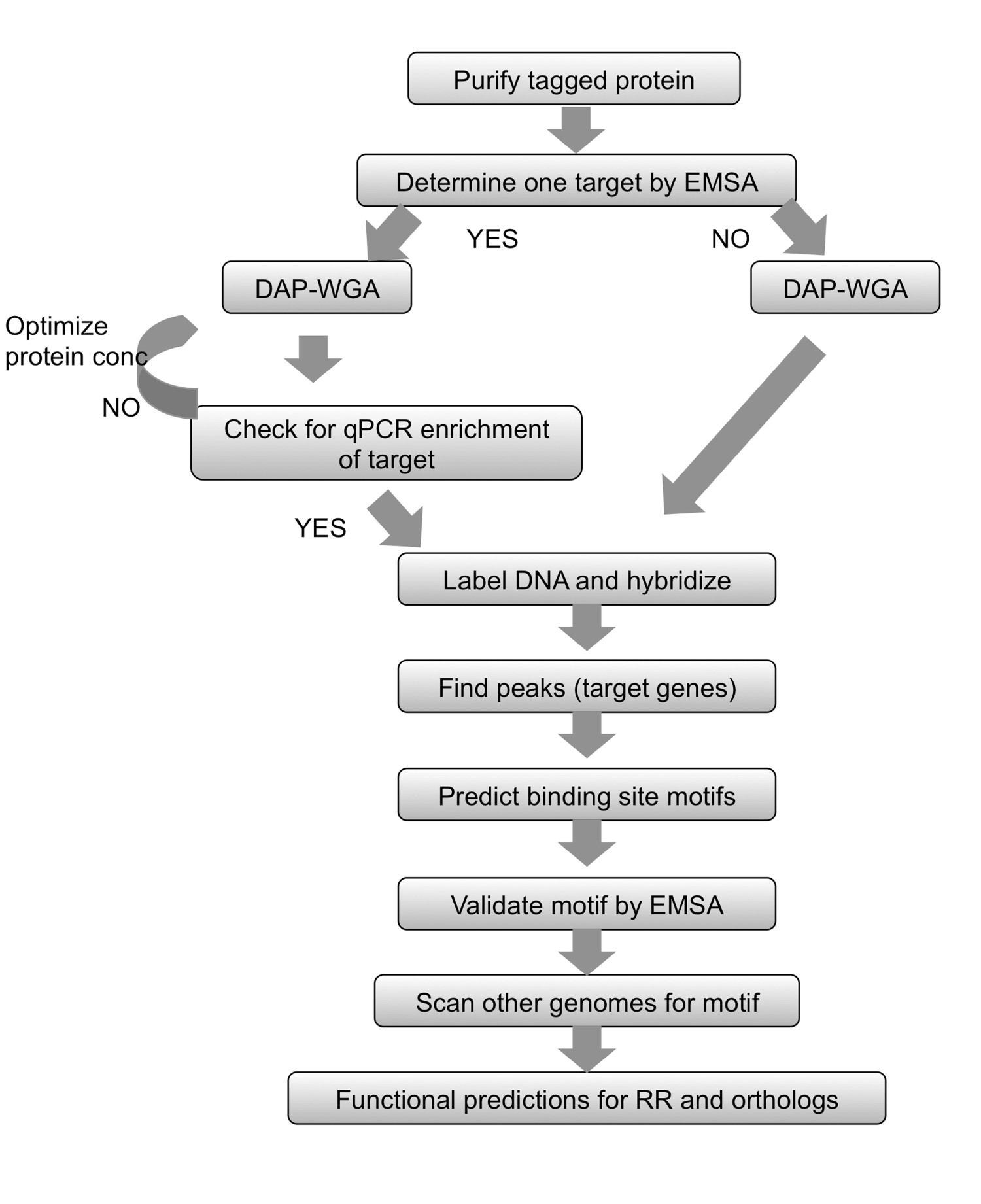
Figura 2. Resumo do fluxo de trabalho. Para qualquer Protei marcado purificadon, começar por determinar um alvo usando EMSA. Permitir proteína para ligar DNA genômico e DNA-afinidade-Purify (DAP) e todo o genoma amplificar (WGA) e do DNA de entrada enriquecido. Se um gene alvo é conhecido, usar qPCR para garantir que o alvo conhecido é enriquecido na fracção ligada a proteína. Se nenhum destino poderia ser determinado, prossiga diretamente para rotulagem de DNA e variedade de hibridização. Se o enriquecimento por qPCR não pôde ser observado, em seguida, repita a ligação proteína-gDNA e passos DAP-WGA usando diferentes quantidades de proteína. Use a análise de matriz para encontrar picos e mapeá-los para atingir genes. Use as regiões a montante de genes alvo de prever motivos local de ligação. Validar os motivos experimentalmente usando EMSAs. Use o motivo para digitalizar os genomas de espécies relacionadas que codificam ortólogos da RR em estudo, e prever genes alvo nas espécies também. Com base nos genes alvos obtidos, a função fisiológica da RR e seus ortólogos pode ser previsto. Figura modificada e reimpresso usando o c criativoommons licença de 7.
Protocolo
Nota: O protocolo a seguir é adaptado para determinação de genes alvos do DVU3023 RR da bactéria Desulfovibrio vulgaris Hildenborough. Ele pode ser adaptado a qualquer outro regulador transcricional de interesse.
1. Clone e Purify RR
- Clonar o gene RR, especificamente DVU3023, de D. vulgaris Hildenborough num vector de expressão de Escherichia coli de modo a que o gene é o C-terminal marcado com His e de expressão está sob o controlo de um promotor de T7.
Nota: Vários métodos de clonagem pode ser utilizado e é determinado pelo investigador. Também podem ser utilizados marcadores de afinidade alternados. - Transformar a construção de expressão em E. coli BL21 (DE3) tensão expressão.
- Cresça 1 L da BL21 expressão tensão a 37 ° C. Na fase mid-log, adicionar IPTG a 0,5 mM para induzir a expressão da proteína. Continuar o crescimento à temperatura ambiente durante 24 horas.
- Células centrifugar a 5000 xg por 10 min a 4 ° C. Ressuspender as células em tampão de lise (fosfato de sódio 20 mM, pH 7,4, 500 mM de NaCl, 40 mM de imidazole, 1 mg / ml de lisozima, 1x benzonase nuclease). Lise das celulas usando uma imprensa francesa, a 4 ° C.
- Centrifugar o lisado a 15.000 x g durante 30 min a 4 ° C. Filtrar através de um filtro de seringa de 0,45 um.
- Lavar uma coluna de 1 ml de Ni Sepharose em um instrumento de FPLC utilizando 10 ml de tampão de lavagem (fosfato de sódio 20 mM, pH 7,4, 500 mM de NaCl, 40 mM de imidazole).
- Carregar lisado na coluna. Lavar coluna com 20 ml de tampão de lavagem.
- Eluir DVU3023 usando um gradiente de 0-100% de tampão de eluição (fosfato de sódio 20 mM, pH 7,4, NaCl 500 mM, imidazol 500 mM).
- Carregar fracções eluídas numa coluna de dessalinização, e lava-se com um tampão de dessalinização (fosfato de sódio 20 mM pH 7,4, NaCl 100 mM).
- Centrífuga amostra de proteína em um filtro de centrífuga de alta molecular de corte de peso para concentrar a proteína. Adicionar DTT a 0,1 mM e glicerol a 50% e armazenar a proteína a -20 ° C.
Nota: terão de ser optimizados individualmente para cada proteína sob estudo métodos de purificação.
2. Determine gene alvo para RR Usando Electrophoretic Mobilidade Mudança Assay (EMSA)
- Amplificar por PCR a região de 400 pb a montante do DVU3025 gene alvo candidato, usando D. ADN genómico vulgaris Hildenborough como molde, e um iniciador não marcado para a frente e um reverso 5'-biotina marcado com iniciador.
Nota: Dicas para escolher um gene alvo candidato: Muitas vezes RRS ligar as regiões a montante do seu próprio gene / operon, ou eles podem regular genes codificados proximal. Se a RR tem ortólogos em outras espécies, olhar para genes que são conservados no bairro. Métodos alternativos incluem genes candidatos que escolhem com base em previsões regulão (por exemplo, RegPrecise 5), ou prevendo promotores dependentes de sigma54 6 RRS para que sejam eles próprios sigma54-dependente. - Executar o produto de PCR num gel de agarose a 1%, cortado a 400 bp produto de tamanho, e purificar o DNA usando uma extração de gel limpar kit.
- Misturar a proteína DVU3023 (0,5 pmol) com 100 fmol de substrato DNA biotinilado em 10 mM Tris-HCl, pH 7,5, KCl 50 mM, MgCl2 5 mM, DTT 1 mM, glicerol a 25% e 1 ug / ml de poli dI.dC (non -específica concorrente ADN) num volume total de 20 ul. Também criou uma reação sem qualquer proteína como um controle. Incubar as reações à temperatura ambiente no banco para 20 min.
- Nota: Estas são as condições padrão enumerados e outros componentes da reacção, podem ser adicionados dependendo do RR de interesse. Se a ativação for desejada, adicionar fosfato de acetil 50 mM para a reação (muitas vezes RRS ligará DNA in vitro, sem ativação).
- Pré-executar um mini 6% de poliacrilamida-0.5x gel TBE pré-moldado em tampão TBE 0,5 x a 100 V por 30 min. Adicionar 5 mL de tampão 5x de carga (0,1% de azul de bromofenol, 0,1% xilenocianol, 30% de glicerol em 1x TBE) para as reações de ligação e de carga de 18 mL das reações sobre o gel. Execute a 100 V durante 2 horas.
Nota: Para evitar o sobreaquecimento, o que pode conduzir a dissociação dos complexos proteína-ADN, encher a câmara tampão externo no sistema de electroforese com tampão de operação de tal forma que a maior parte do gel é isolado por tampão. Em alternativa, o gel pode ser realizada a 4 ° C. - Cortar uma membrana de nylon carregada ao tamanho do gel e mergulhe-o em 0,5 x TBE, durante pelo menos dez minutos. Remover o gel a partir da cassete. Cortar quaisquer saliências fora do gel e o gel de sanduiche e a membrana entre dois papéis de filtro de espessura embebido em TBE 0,5 x, e colocar dentro de um aparelho de transferência semi-seco e à temperatura de 20 V durante 30 min.
- Coloque a membrana dentro de um instrumento de luz UV crosslinker comercial e definir o tempo de 3 min.
Nota: A membrana podem agora ser armazenados secos à temperatura ambiente durante vários dias antes de prosseguir para o próximo passo. - Use um kit de detecção quimioluminescente disponível no mercado que emprega um estreptavidina-cavalo rabanete peroxidaçãosi conjugado para desenvolver a mancha como as instruções do fabricante.
- Imagem da mancha utilizando um computador ligado a uma câmara CCD equipado e procurar uma mudança na mobilidade do substrato de DNA na presença do RR, o que indica que a RR liga o ADN a ser testado.
Nota: A especificidade da transferência de DNA-proteína pode ser testada através da inclusão na reacção de um excesso de DNA competidor não marcado, que deverá eliminar ou reduzir a mudança de vista.
3. Verifique Alvo Enriquecimento após Genomic DNA-binding protein
- Reacção de ligação proteína-ADN genómico
- ADN genómico de cisalhamento até um tamanho médio de 500 pb por sonicaçao de DNA genómico 100 ul (em concentrações de 100-200 ng / ul) num tubo de microcentrífuga de 1,5 ml utilizando uma microponta com 9 baixos impulsos de amplitude de um segundo, com 2 seg lacuna entre cada.
Nota: Se os respingos de DNA para os lados do tubo, girar o conteúdo a cada 3 pulsos. As condições exatas usadasvariará de acordo com o instrumento de ultra-sons utilizado. Os fragmentos de ADN cortados podem variar de 100-1000 pb, mas o tamanho médio deve ser dentro de 400-600 pb. Demasiada corte pode resultar em menos sítios de ligação intactas, e quer muito ou pouco corte afetará preparação biblioteca durante as etapas a jusante inteiras de amplificação do genoma. - Misturar 2-3 ug de DNA genómico cortado com proteína RR (0,5 pmol de DVU3023) em 10 mM Tris-HCl pH 7,5, DTT 1 mM, KCl 50 mM, MgCl2 5 mM, 25% glicerol, e 50 mM de fosfato de acetilo. Incubar as reacções a 25 ° C durante 30 min. Transferir 10 uL desta reacção para um tubo de 1,5 ml e rotular como ADN de entrada.
Nota: fosfato Acetil é adicionada para activar a proteína in vitro por fosforilação. A fosforilação estimula a ligação ao ADN, mas muitos RRS também ligar o ADN in vitro, sem activação 7. A quantidade de proteína adicionada à reacção dependerá de actividade da preparação de proteína. O EMSA pode ser utilizado para optartimize este montante.
- ADN genómico de cisalhamento até um tamanho médio de 500 pb por sonicaçao de DNA genómico 100 ul (em concentrações de 100-200 ng / ul) num tubo de microcentrífuga de 1,5 ml utilizando uma microponta com 9 baixos impulsos de amplitude de um segundo, com 2 seg lacuna entre cada.
- Afinidade purificar o ADN ligado às proteínas
- Adicionar 30 mL de resina de agarose Ni-NTA para um tubo de microcentrífuga de 0,6 ml. Centrifugar a 100 xg durante 1 minuto para recolher a resina na parte inferior, e remover o sobrenadante. Adicionar 100 ul de tampão de lavagem (10 mM Tris-HCl, pH 7,5, MgCl2 5 mM, KCl 50 mM, glicerol a 25%), o movimento do tubo para misturar e centrifugar a 100 xg durante 2 min. Remover o sobrenadante.
- Adicionar os restantes 90 ul da reacção de ligação com a resina de Ni-NTA lavada e incubar num agitador rotativo durante 30 min.
- Centrifugar a 100 xg durante 2 min e remove-se o sobrenadante (o ADN não ligado). Adicionar 100 ul de tampão de lavagem, apertar o tubo para misturar o conteúdo, e centrifugar a 100 xg durante 2 min. Remover o sobrenadante. Repita o passo de lavagem mais duas vezes.
- Adicionar 35 ul de tampão de eluição (tampão fosfato pH 20 mM de sódio 8, NaCl 500 mM, imidazol 500 mM) à resina e misturar por vortex. Incubar no banco a temperatura ambiente durante 5 min. Centrifugar a 100 xg por 2 min. Transferir o sobrenadante para um novo tubo de 1,5 ml e rotular como a fracção de DNA ligada à proteína.
- Também adicionar 35 ul de tampão de eluição para o ADN de entrada.
- Purifica-se a entrada e as fracções de ADN ligado à proteína usando um kit de purificação de PCR.
- Genoma inteiro amplificar a entrada e amostras de DNA ligadas às proteínas.
- Adicionar 10 ul de entrada e de DNA ligado a proteínas para separar tubos PCR. Adicionar 1 mL de tampão de fragmentação de 10x, 2 ul de tampão de preparação da biblioteca, e 1 ul de solução de biblioteca de estabilização para cada amostra. Misture bem em vórtice. Aquecer num termociclador a 95 ° C durante 2 min, e frio no gelo.
Nota: A quantidade inicial de ADN deve ser de pelo menos 10 ng, a fim de evitar a introdução de viés de amplificação. - Adicionar 1 ml de preparação de enzima Biblioteca, misturar por pipetagem, e incubar em termociclador a 16 ° C/20 min, 24 ° C/20 min, 37 ° C durante 20 min, 75 ° C / 5 min, E espera a 4 ° C.
- Para cada tubo, adicionar 47,5 mL de água, 7,5 ul de amplificação 10x mistura principal, e 5 ul de polimerase. Misturar bem e aquece-se a 95 ° C / 3 min, seguido por 20 ciclos de 94 ° C/15 seg, 65 ° C / 5 min. Segurar reacções a 4 ° C.
- Purifica-se as amostras de DNA amplificado usando um kit de purificação PCR e medir a concentração de DNA por espectrofotometria.
- Adicionar 10 ul de entrada e de DNA ligado a proteínas para separar tubos PCR. Adicionar 1 mL de tampão de fragmentação de 10x, 2 ul de tampão de preparação da biblioteca, e 1 ul de solução de biblioteca de estabilização para cada amostra. Misture bem em vórtice. Aquecer num termociclador a 95 ° C durante 2 min, e frio no gelo.
- Verifique alvo enriquecimento no ADN ligada a proteína utilizando qPCR
- Primers Projeto qPCR para amplificar 200 pb da região a montante do gene alvo EMSA-verificada (DVU3025) usando qualquer software de desenho de primers livremente disponível.
- Configure reações de qPCR em triplicado para cada modelo de DNA com cada conjunto de primers. Prepare uma mistura de mestre para cada conjunto de primers. 1x mistura principal contém 10 mL de mistura SYBR Green qPCR 2x, 0,5 uM de cada iniciador e água para um total de 18 ul. Alíquotas de 18 ul da mistura principal por poço de uma placa de 96 poços de PCR.
- Dilute a entrada e amplificados purificados e amostras de DNA ligadas a proteínas de 5 ng / mL com água e usado como molde de ADN. Adicionar 2 l de molde de ADN para os poços.
- Selar a placa com um filme de selagem ultra claro qPCR, spin down placa em uma centrífuga a 200 xg / 1 min. Coloque a placa em uma máquina de qPCR tempo real. Ciclo como se segue, utilizando o software associado qPCR: 95 ° C / 1 min, e 40 ciclos de 95 ° C/10 seg, 59 ° C/15 seg e 70 ° C/35 seg.
- Além disso, definir as reações de qPCR triplicadas com primers para amplificar regiões a montante de um gene relacionado (controle negativo).
- Calcular ôc T subtraindo valor T C do DNA ligada à proteína do que o ADN de entrada. Calcular o enriquecimento de dobragem do gene alvo no ADN ligado à proteína como 2 Ac t.
Nota: Se o alvo a região a montante foi enriquecida com o ADN ligado às proteínas contra o ADN de entrada, que indica que as reacções de ligação e purificação por afinidade foram successful. Vá para a etapa 4. Se o enriquecimento de alvo a região a montante não foi observado, repita as reações de ligação (passo 3.1) com diferentes quantidades de proteína.
4. Labeling DNA e matriz de Hibridização
- Etiqueta DNA de entrada com DNA Cy3 e enriquecido com Cy5
ota: Cy3 e Cy5 corantes são sensíveis à luz e os cuidados devem ser tomados para manter a exposição à luz a um mínimo.- Misture 1 mg de DNA com 40 mL Cy3/Cy5-labeled 9-meros e ajustar o volume para 80 mL com água.
- Calor desnaturar a 98 ° C durante 10 minutos no escuro (num termociclador). Frio rápida no gelo por 2 min.
- Adicionar 2 l de Klenow da polimerase (3'-5'-exo -, 50.000 U / ml), 5 mM dNTPs, e 8 mL de água a cada reacção, misturar bem e incubar a 37 ° C durante 2 horas no escuro (em um termociclador).
- Adicionar EDTA para 50 mM, para parar a reacção e uma solução de NaCl a 0,5 M.
- Amostras de transferência para tubos de 1,5 ml containing 0,9 volume de isopropanol, incubar no escuro durante 10 minutos, e centrifugar a 12000 xg durante 10 min. A pastilha deve ser rosa para DNA Cy3 marcado e azul para DNA Cy5 marcado.
- Lavar sedimento com etanol a 80% (500 mL) e centrifugar a 12000 xg durante 2 min. Pelotas secarem naturalmente durante 5-10 min no escuro.
Nota: Os grânulos podem ser armazenadas a -20 ° C. - Ressuspender o sedimento em 25 mL de água. Medir a concentração de DNA por espectrofotometria.
- Piscina juntos 6 ug cada de ADN Cy3 e Cy5 marcado num tubo de 1,5 ml e seco sob vácuo em uma centrífuga em baixo calor no escuro (cobrir a tampa da centrifugadora, se for transparente).
Nota: Os grânulos podem ser armazenadas a -20 ° C até estar pronto para a hibridação.
- Microarray Hibridização
- Ligue o sistema de hibridização em 3-4 horas antes de usar e definir a temperatura a 42 ° C.
- Prepare uma solução de mistura principal hibridização tal que 1x solução contém 11,8 mL 2x Hybriditampão zação, 4,7 ul de Hibridização de componentes A e 0,5 ul de oligo alinhamento.
- Ressuspender as peletes em 5 mL de água. Adicionar 13 ul desta mistura para a amostra. Vortex durante 15 seg, incubar a 95 ° C em um banho seco de 5 min. Manter as amostras a 42 ° C no sistema de hibridação até estarem prontos para carregamento.
- Prepare o microarray montagem slide-mixer, de acordo com o protocolo do fabricante.
- Coloque o conjunto misturador de slides dentro do sistema de hibridização. Carga de 16 ul de amostra no orifício de enchimento, selar as portas com uma película adesiva, transformar a mistura no no sistema, e hibridar durante 16-20 horas a 42 ° C.
- Prepare 1x tampões de lavagem I (250 ml), II (50 ml) e III (50 ml) por diluição das 10x tampões comercialmente disponíveis em água, e para cada adicionar DTT a 1 mM. Tampão Quente I a 42 ° C.
- Deslize o misturador de slides em uma ferramenta de desmontagem, e coloque dentro de um prato quente contendo Tampão I. Peel a batedeira desligada, enquanto vigorosamente shendo a ferramenta de desmontagem com a mão.
- Coloque o slide em um recipiente com 50 ml de tampão de lavagem I e agitar vigorosamente durante 2 minutos com a mão.
- Transferência de slides para um segundo recipiente com tampão de 50 ml Wash II e agitar vigorosamente durante 1 min com a mão.
- Transferência de slides em um terceiro recipiente com 50 ml de Tampão de lavagem III, e agitar vigorosamente por 15 segundos com a mão.
- Apagar rapidamente as bordas do slide em uma toalha de papel e coloque em um rack slide. Centrifugar a 200 xg durante 2 minutos para secar a corrediça. Coloque dentro de caso slide, envolvê-la com papel alumínio e guarde em um dessecador.
- A digitalização da Matriz
- Colocar a lâmina dentro do scanner de microarray de acordo com as instruções do instrumento.
- Use um software de scanner para definir os comprimentos de onda como 532 nm = Cy3 e 635 nm = Cy5, e os ganhos fotomultiplicadores iniciais entre 350 e 400.
- Pré-visualizar o slide para localizar a matriz no slide. Selecione a região de matriz para a digitalização.
- Digitalizar a matriz e ajustar as configurações de fotomultiplicadores de tal forma que as características da matriz são principalmente amarelo. O histograma que mostra as curvas de vermelhas e verdes, sobreposto ou o mais próximo possível uns dos outros quanto possível. As curvas devem terminar acima dos 10 -5 contagens normalizadas em saturação.
- Salve ambos os 532 e 635 nm imagens separadamente.
- Análise de Dados Matriz
- Importe as imagens em um software de análise de matriz. Usando o software, criar dois relatórios (par. Par) para cada matriz (por Cy3 e Cy5 imagens). Criar log 2 arquivos relação em escala usando os arquivos par. Use log 2 arquivos relação ao procurar picos usando uma janela deslizante de 500 pb. Mapear o pico loci para as regiões a montante de genes alvos.
- Verifique se o alvo positivo (determinado usando EMSA e confirmado por qPCR, DVU3025 neste exemplo) aparece no top picos.
Nota: Se o alvo positivo não está entre os top hits, é possível que a RR em consisideração tem vários genes alvos e replicar DAP-chips pode ser realizado e picos comuns a todas as repetições podem ser utilizados para gerar uma lista de destino.
5. Binding Previsão do Site Motif e Validação
- Recuperar as sequências de regiões a montante (400 pb) para os genes alvos principais como gerado pela análise DAP-chip. Aplicar MEME sobre essas sequências através de micróbios Online (meme.nbcr.net) para prever motivos.
Nota: as proteínas de ligação, tais como os reguladores Enhancer sigma54-dependentes podem ligar-se várias centenas de pares de bases a montante do local de início. Para outros reguladores da transcrição, uma região mais curto, tal como de 200 pb a montante será suficiente. - Projeto superior e oligômeros DNA, fundo que contêm o motivo sítio de ligação previsto ladeado por 10 bases em cada extremidade. Encomende o topo vertente 5 'biotinilado.
- Misturar os oligos de cadeia de topo e de fundo em relação de 1:1,5 em 10 mM Tris-HCl, pH 8,0, EDTA 1 mM e NaCl a 50 mM emum volume total de reacção de 20 ul em tubos PCR. Aquece-se a 95 ° C durante 5 minutos num termociclador, seguido de arrefecimento lento até 25 ° C. Diluir dez vezes e usar como substrato dsDNA tipo selvagem para EMSA.
- Prepare substratos modificados usando os mesmos passos acima, mas oligômeros projeto para levar 4-6 substituições nas bases conservadas do motivo de ligação.
- Defina-se reacções de ligação com a RR e do tipo selvagem ou substratos modificados e analisar usando EMSA, tal como descrito no passo 2.
- Nota: O motivo predito é validado se o RR liga o substrato do tipo selvagem, mas não a uma modificação.
6. Conservação da Motif em Outras bacterianas relacionadas Espécies
- Selecione genomas de interesse que contêm ortólogos de DVU3023. Obter arquivos de seqüência e de anotação dos genomas do site do NCBI.
- Use uma linguagem de programação como Perl para escrever scripts que irão utilizar as seqüências motivo de as metas DAP-chip para construir um position matriz ponderada e usar a matriz para marcar motivos semelhantes presentes em outros genomas seqüenciados.
Resultados
O método acima foi aplicado para determinar os genes alvos globais dos RRS no sulfato de modelo reduzindo bactéria Desulfovibrio vulgaris Hildenborough 7. Este organismo tem um grande número de TCS representado por mais de 70 RRS, indicando a vasta variedade de possíveis sinais que detecta e responde. Análise in vivo sobre as funções destes sistemas de sinalização são difíceis de realizar uma vez que os sinais e, assim, as suas condições de activação são desconhecidos. Aqui o...
Discussão
O método DAP-chip aqui descrito foi utilizado com sucesso para determinar os genes alvos para vários RRS em Desulfovibrio vulgaris Hildenborough 7 dos quais um é mostrado aqui como um resultado representativo. Para RR DVU3023, escolhendo um alvo gene candidato foi simples. DVU3025 está localizado imediatamente a jusante do gene RR, e os genes-alvo e RR são conservadas em diversas espécies Desulfovibrio, e adicionalmente DVU3025 tem um promotor dependente de sigma54 previsto. O EMSA for...
Divulgações
Os autores não têm qualquer conflito de interesse de divulgar.
Agradecimentos
Agradecemos Amy Chen por sua ajuda na preparação para a filmagem do vídeo e para demonstrar a técnica. Este trabalho realizado pelo ENIGMA: Ecossistemas e redes integradas com os genes e Assembléias Molecular (http://enigma.lbl.gov), um programa científico da área de foco no Lawrence Berkeley National Laboratory, foi apoiado pelo Escritório de Ciência, Instituto de Ciências Biológicas e Pesquisa Ambiental, do Departamento de Energia dos EUA sob Contrato n º DE-AC02-05CH11231.
Materiais
| Name | Company | Catalog Number | Comments |
| HisTrapFF column (Ni-Sepharose column) | GE Lfe Sciences, Pittsburgh, PA, USA | 17-5255-01 | |
| Akta explorer (FPLC instrument) | GE Lfe Sciences, Pittsburgh, PA, USA | ||
| HiPrep 26/10 Desalting column | GE Lfe Sciences, Pittsburgh, PA, USA | 17-5087-01 | |
| Qiaquick Gel extraction kit | Qiagen Inc, Valencia, CA, USA | 28704 | |
| Biotin-labeled oligonucleotides | Integrated DNA Technologies | N/A | |
| 6% polyacrylamide-0.5x TBE precast mini DNA retardation gel | Life Technologies, Grand Island, NY, USA | EC63652BOX | Alternately, you can pour your own gel. |
| Nylon membrane | EMD Millipore, Billerica, MA, USA | INYC00010 | |
| Trans-Blot SD Semi-dry electrophoretic transfer cell | Biorad, Hercules, CA, USA | 170-3940 | |
| Extra thick blot paper, 8 x 13.5 cm | Biorad, Hercules, CA, USA | 170-3967 | |
| UV crosslinker Model XL-1000 | Fisher Scientific | 11-992-89 | |
| Nucleic Acid chemiluminescent detection kit (Pierce) | Thermo fisher Scientific, Rockford, IL, USA | 89880 | |
| Ni-NTA agarose resin | Qiagen Inc, Valencia, CA, USA | 30210 | |
| GenomePlex Whole genome amplification kit (Fragmentation buffer, library preparation buffer, library stabilization solution, library preparation enzyme, 10x amplification master mix, WGA polymerase) | Sigma-Aldrich, St. Louis, MO, USA | WGA2-50RXN | |
| Nanodrop ND-1000 | Thermo Scientific, Wilmington, DE, USA | For quantitation of DNA | |
| Perfecta Sybr Green SuperMix, with ROX | Quanta biosciences | 95055-500 | Any Sybr Green PCR mix may be used |
| PlateMax Ultra clear heat sealing film for qPCR | Axygen | ||
| [header] | |||
| 96-well clear low profile PCR microplate | Life Technologies, Grand Island, NY, USA | PCR-96-LP-AB-C | |
| Applied Biosystems StepOne Plus Real time PCR system | Life Technologies, Grand Island, NY, USA | 4376600 | Any real time PCR system may be used |
| Qiaquick PCR purification kit | Qiagen Inc, Valencia, CA, USA | 28104 | Any PCR clean up kit may be used |
| Cy3/Cy5-labeled nonamers | Trilink biotechnologies, San Diego, CA, USA | N46-0001, N46-0002 | |
| Klenow polymerase 50,000 U/ml, 3'-5' exo- | New England Biolabs, Ipswich, MA | M0212M | |
| Hybridization system | Roche-Nimblegen, Madison, WI, USA | N/A | This company no longer makes arrays or related items, so alternate sources such as Agilent or Affymetrix will need to be used. |
| Custom printed microarrays and mixers | Roche-Nimblegen, Madison, WI, USA | N/A | |
| Hybridization kit (2x Hybridization buffer, Hybridization component A, Alignment oligo) | Roche-Nimblegen, Madison, WI, USA | N/A | |
| Wash buffer kit (10x Wash buffer I, II, III, 1 M DTT) | Roche-Nimblegen, Madison, WI, USA | N/A | |
| GenePix 4200A microarray scanner | Molecular Devices, Sunnyvale CA, USA | This model has been replaced by superior ones | |
| GenePix Pro microarray software | Molecular Devices, Sunnyvale CA, USA | ||
| Nimblescan v.2.4, ChIP-chip analysis software | Roche-Nimblegen, Madison, WI, USA | N/A | |
Referências
- Galperin, M. Y. Diversity of structure and function of response regulator output domains. Curr Opin Microbiol. 13, 150-159 (2010).
- Casino, P., Rubio, V., Marina, A. The mechanism of signal transduction by two-component systems. Curr Opin Struct Biol. 20, 763-771 (2010).
- Aparicio, O., Geisberg, J. V., Struhl, K. Chromatin immunoprecipitation for determining the association of proteins with specific genomic sequences in vivo. Curr Protoc Cell Biol. 17 (17), (2004).
- McCleary, W. R., Stock, J. B. Acetyl phosphate and the activation of two-component response regulators. J Biol Chem. 269, 31567-31572 (1994).
- Novichkov, P. S., et al. RegPrecise web services interface: programmatic access to the transcriptional regulatory interactions in bacteria reconstructed by comparative genomics. Nucleic Acids Res. 40, 604-608 (2012).
- Studholme, D. J., Buck, M., Nixon, T. Identification of potential sigma(N)-dependent promoters in bacterial genomes. Microbiology. 146 (12), 3021-3023 (2000).
- Rajeev, L., et al. Systematic mapping of two component response regulators to gene targets in a model sulfate reducing bacterium. Genome Biol. 12, 99 (2011).
- Crooks, G. E., Hon, G., Chandonia, J. M., Brenner, S. E. WebLogo: a sequence logo generator. Genome Res. 14, 1188-1190 (2004).
- Barbieri, C. M., Mack, T. R., Robinson, V. L., Miller, M. T., Stock, A. M. Regulation of response regulator autophosphorylation through interdomain contacts. J Biol Chem. 285, 32325-32335 (2010).
- Bourret, R. B. Receiver domain structure and function in response regulator proteins. Curr Opin Microbiol. 13, 142-149 (2010).
- Gao, R., Stock, A. M. Molecular strategies for phosphorylation-mediated regulation of response regulator activity. Curr Opin Microbiol. 13, 160-167 (2010).
- Hung, D. C., et al. Oligomerization of the response regulator ComE from Streptococcus mutans is affected by phosphorylation. J Bacteriol. 194, 1127-1135 (2012).
- Ladds, J. C., et al. The response regulator Spo0A from Bacillus subtilis is efficiently phosphorylated in Escherichia coli. FEMS Microbiol Lett. 223, 153-157 (2003).
- Wen, Y., Feng, J., Scott, D. R., Marcus, E. A., Sachs, G. Involvement of the HP0165-HP0166 two-component system in expression of some acidic-pH-upregulated genes of Helicobacter pylori. J Bacteriol. 188, 1750-1761 (2006).
- Liu, X., Noll, D. M., Lieb, J. D., Clarke, N. D. DIP-chip: rapid and accurate determination of DNA-binding specificity. Genome Res. 15, 421-427 (2005).
- Gossett, A. J., Lieb, J. D. DNA Immunoprecipitation (DIP) for the Determination of DNA-Binding Specificity. CSH Protoc. 2008, (2008).
- Mascher, T., et al. The Streptococcus pneumoniae cia regulon: CiaR target sites and transcription profile analysis. J Bacteriol. 185, 60-70 (2003).
- Park, P. J. ChIP-seq: advantages and challenges of a maturing technology. Nat Rev Genet. 10, 669-680 (2009).
- Lefrancois, P., Zheng, W., Snyder, M. ChIP-Seq using high-throughput DNA sequencing for genome-wide identification of transcription factor binding sites. Methods Enzymol. 470, 77-104 (2010).
- Ho, J. W., et al. ChIP-chip versus ChIP-seq: lessons for experimental design and data analysis. BMC Genomics. 12, 134 (2011).
- Liu, X., Lee, C. K., Granek, J. A., Clarke, N. D., Lieb, J. D. Whole-genome comparison of Leu3 binding in vitro and in vivo reveals the importance of nucleosome occupancy in target site selection. Genome Res. 16, 1517-1528 (2006).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados