Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Purificado de ADN afinidad Chip (DAP-chip) Método para determinar los objetivos de genes para bacteriana Dos Sistemas Regulatorios componentes
En este artículo
Resumen
Este artículo de vídeo describe un método de microarrays basado en vitro para determinar los objetivos de genes y sitios de unión para dos reguladores de respuesta del sistema de componentes.
Resumen
Los métodos in vivo como chip-chip son técnicas bien establecidas que se utilizan para determinar dianas génicas globales para los factores de transcripción. Sin embargo, son de uso limitado en la exploración de dos sistemas de regulación componente bacteriano con condiciones de activación no caracterizados. Tales sistemas regulan la transcripción sólo cuando se activa en la presencia de señales únicas. Puesto que estas señales son a menudo desconocida, el método in vitro de microarrays basado en describe en este artículo de vídeo se puede utilizar para determinar objetivos de genes y sitios de unión para los reguladores de respuesta. Este método chip purificado-ADN-afinidad puede ser utilizado para cualquier regulador purificado en cualquier organismo con un genoma secuenciado. El protocolo consiste en permitir que la proteína marcada purificada para unirse al ADN genómico cortado y después de purificación por afinidad del ADN a las proteínas, seguido de marcaje fluorescente del ADN y la hibridación con un suelo de baldosas gama personalizado. Precediendo pasos que se pueden usar para optimizar el ensayo para la especificaciónTambién se describen los reguladores c. Los picos generados por el análisis de datos de la matriz se utilizan para predecir motivos sitio de unión, que luego se validan experimentalmente. Los motivo de las predicciones se pueden usar además para determinar objetivos de genes de respuesta de los reguladores ortólogos en especies estrechamente relacionadas. Se demuestra la aplicabilidad de este método mediante la determinación de los objetivos de genes y vinculante motivos sitio y predecir así la función de un regulador de respuesta DVU3023 sigma54 dependiente en la bacteria Desulfovibrio vulgaris ambiental Hildenborough.
Introducción
La capacidad de las bacterias para sobrevivir y prosperar depende críticamente de lo bien que son capaces de percibir y responder a las perturbaciones en sus ambientes, y esto a su vez depende de los sistemas de transducción de señales. El número de sistemas de señalización A codifica bacteria que se ha llamado su "IQ microbiana" y puede ser una indicación de la variabilidad de su medio ambiente y su capacidad de detectar señales múltiples y afinar su respuesta 1. Sistemas de dos componentes de transducción de señales (TCS) son los sistemas de señalización más prevalentes utilizados por las bacterias, y que consisten en una histidina quinasa (HK) que detecta la señal externa y transmite a través de la fosforilación de un regulador de la respuesta efectora (RR) 2. RR puede tener una variedad de dominios de salida y por lo tanto diferentes modos efectoras, pero la respuesta más común es la regulación transcripcional a través de un ADN de dominio de unión 1. Las señales detectadas y las funciones correspondientes de los conductos deferentest mayoría de TCSs siguen siendo desconocidos.
Aunque los métodos in vivo, tales como chip-chip se utilizan rutinariamente para la determinación de sitios de unión de factores de transcripción genómica 3, que sólo se pueden utilizar para bacterianas RR sistema de dos componentes si se conocen las condiciones de activación o señales. A menudo, las señales ambientales que activan una TCS son más difíciles de determinar que sus genes diana. El microarray de ensayo in vitro basado descrito aquí se puede utilizar para determinar eficaz y rápidamente los objetivos de genes y predecir funciones de TCSs. Este ensayo se aprovecha del hecho de que los RR se pueden fosforilar y por lo tanto activan in vitro utilizando pequeñas moléculas donantes como fosfato de acetilo 4.
En este método, llamado DAP-chip para ADN-afinidad-chip purificado (Figura 1), el gen RR de interés se clona con una etiqueta de His en E. coli, y una proteína marcada posteriormente purificado se permite a Bind a esquilada de ADN genómico. El ADN unido a la proteína se enriquecen entonces por purificación por afinidad, el ADN enriquecido y de entrada se amplifican, marcado con fluorescencia, agrupados juntos y se hibridó a una matriz de mosaico que está hecho a medida para el organismo de interés (Figura 1). Microarray experimentos están sujetos a los artefactos, por lo que se emplean pasos adicionales para optimizar el ensayo. Una de esas medidas es tratar de determinar un objetivo para el RR en estudio el uso de ensayos de cambio de movilidad electroforética (EMSA) (véase el flujo de trabajo en la Figura 2). Entonces, después de la unión al ADN genómico y los pasos de DAP, el ADN unido a proteína y de entrada se examinó por qPCR para ver si el objetivo positivo se enriquece en la fracción unida a proteínas en relación con la fracción de entrada, confirmando de este modo las condiciones de unión óptimas para el RR (Figura 2). Después de la gama de hibridación, los datos se analizan para encontrar picos de señal de mayor intensidad que indican loci del genoma en donde la proteína had obligado. Las funciones pueden ser predichos para el RR en base a los objetivos de genes obtenidos. Los loci genómicos de destino se utilizan para predecir motivos sitio de unión, que luego son validados experimentalmente utilizando EMSAs (Figura 2). Las predicciones funcionales y objetivos de genes para el RR pueden entonces extenderse a especies que codifican los RR ortólogos mediante el escaneo de los genomas de los motivos de unión similares (Figura 2), estrechamente relacionado. El método DAP-chip puede proporcionar una gran cantidad de información de un TCS donde antes no había ninguna. El método también puede utilizarse para cualquier regulador transcripcional si la proteína se puede purificar y condiciones de unión de ADN se puede determinar, y para cualquier organismo de interés con una secuencia del genoma disponible.
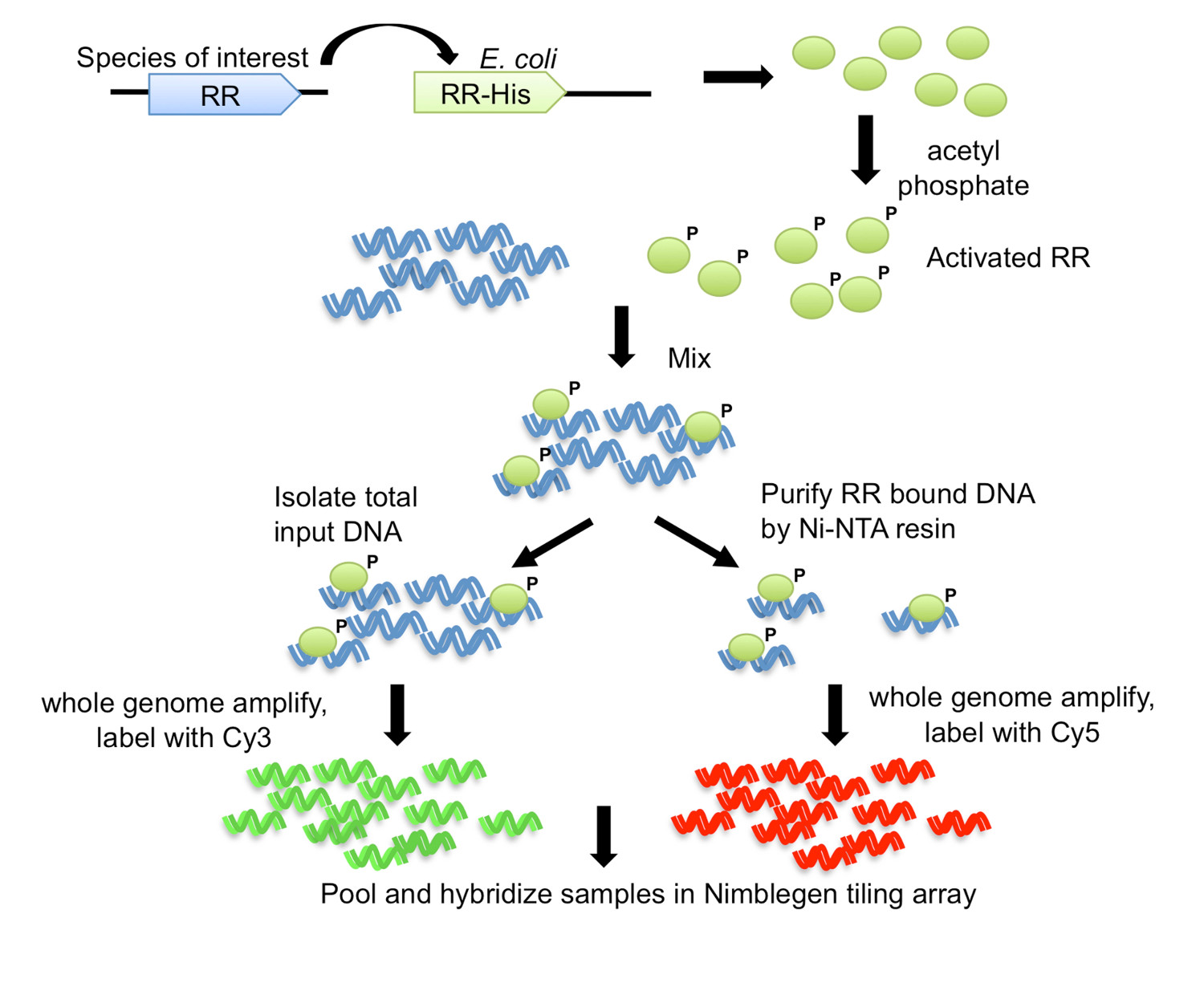
Figura 1. El chip purificado-ADN-afinidad (DAP-chip) estrategia 7. El gen RR a partir del organismo de interés se clona con un carboxi-terminal de His-tag en una E. cepa de expresión coli. Se purificó la proteína marcada con His se activa por fosforilación con fosfato de acetilo, y se mezcla con el ADN genómico cortado. Una alícuota de la reacción de unión se guarda como ADN de entrada, mientras que el resto se sometió a purificación de afinidad utilizando resina de Ni-NTA. La entrada y el ADN RR-unidos se todo el genoma amplificado, y marcado con Cy3 y Cy5, respectivamente. El ADN marcado se agrupen y se hibridó a una matriz de suelo de baldosas, que luego se analizó para determinar los objetivos de genes. Figura modificado y reimpreso con el licencia creative commons de 7.
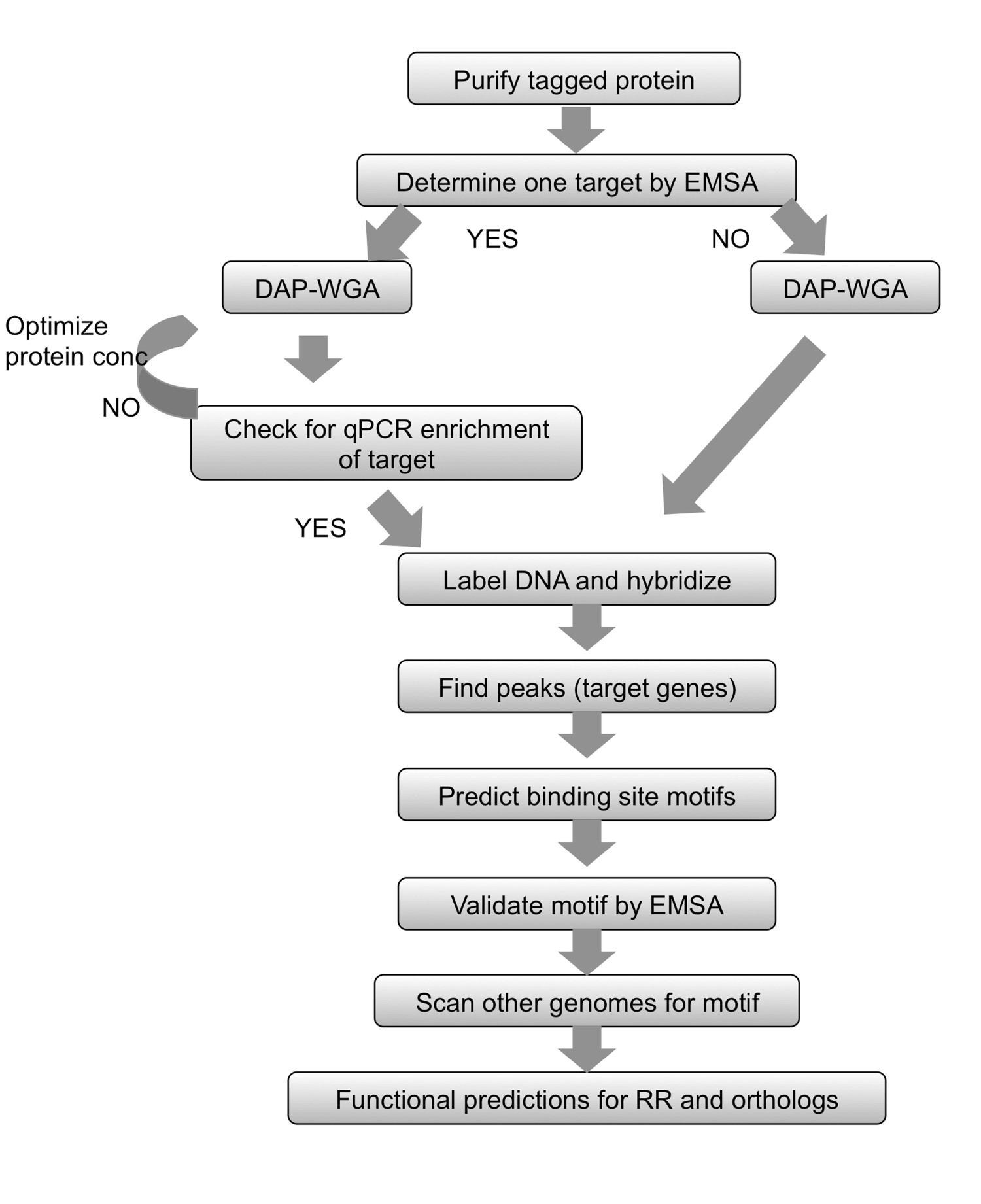
Figura 2. Resumen de flujo de trabajo. Para cualquier protei etiquetados purificadon, comenzar por la determinación de un destino utilizando la EMSA. Permitir proteínas se unen al ADN genómico y ADN-purificar por afinidad (DAP) y la totalidad de su genoma amplificar (WGA) y la entrada de ADN enriquecido. Si se conoce un gen objetivo, utilizar la qPCR para asegurar que el objetivo conocido se enriquece en la fracción unida a proteínas. Si se pudo determinar ningún objetivo, pase directamente al marcaje de ADN y la gama de hibridación. Si no se pudo observar el enriquecimiento por qPCR, a continuación, repetir la proteína de unión-ADNg y pasos DAP-WGA utilizando diferentes cantidades de proteína. Utilice el análisis conjunto para encontrar picos y asignarlos a los genes objetivo. Utilice las regiones aguas arriba de los genes diana de predecir motivos sitio de unión. Validar los motivos experimentalmente utilizando EMSAs. Utilice el motivo para escanear los genomas de las especies relacionadas que codifican ortólogos de la RR en estudio, y predecir determinados genes en estas especies también. Sobre la base de los objetivos de genes obtenidos, la función fisiológica de la RR y sus ortólogos se puede predecir. Figura modificado y reimpreso usando el C creativoommons licencia de 7.
Protocolo
Nota: El protocolo a continuación está diseñado para la determinación de objetivos de genes de la DVU3023 RR de la bacteria Desulfovibrio vulgaris Hildenborough. Se puede adaptar a cualquier otro regulador transcripcional de interés.
1. Clone y Purify RR
- Clonar el gen RR, específicamente DVU3023, de D. vulgar Hildenborough en un vector de expresión de Escherichia coli tal que el gen es C-terminal marcada con His y la expresión es bajo el control de un promotor T7.
Nota: Varios métodos de clonación se pueden utilizar y es determinada por el investigador. Etiquetas de afinidad alternativos también pueden ser utilizados. - Transformar la construcción de expresión en E. coli BL21 (DE3) cepa de expresión.
- Crece 1 L de la cepa de expresión BL21 a 37 ° C. A mediados de la fase de registro, añadir IPTG a 0,5 mM para inducir la expresión de la proteína. Continuar el crecimiento a temperatura ambiente durante 24 h.
- Centrifugar las células a 5000 xg durante 10 millasn a 4 ° C. Resuspender las células en tampón de lisis (fosfato de sodio 20 mM, pH 7,4, NaCl 500 mM, imidazol 40 mM, 1 mg / ml de lisozima, 1x Benzonase nucleasa). Lyse células utilizando una prensa francesa a 4 ° C.
- Lisado Centrifugar a 15.000 xg durante 30 min a 4 ° C. Filtrar utilizando un filtro de jeringa de 0,45 micras.
- Lavar una columna de 1 ml de Ni Sepharose en un instrumento de FPLC usando 10 ml de tampón de lavado (fosfato de sodio 20 mM, pH 7,4, NaCl 500 mM, imidazol 40 mM).
- Cargar lisado en la columna. Lavar la columna con 20 ml de tampón de lavado.
- Eluir DVU3023 usando un gradiente de tampón de elución 0-100% (fosfato de sodio 20 mM, pH 7,4, NaCl 500 mM, imidazol 500 mM).
- Cargar fracciones eluidas en una columna de desalación, y lavar con un tampón de desalado (20 mM de fosfato de sodio de pH 7,4, NaCl 100 mM).
- Centrífuga muestra de proteína en un filtro de centrífuga de alto peso molecular de corte de peso para concentrar la proteína. Añadir TDT a 0,1 mM y glicerol al 50% y la proteína se almacena a -20 ° C.
Nota: tendrán que ser optimizado de forma individual para cada proteína en estudio métodos de purificación.
2. Determinar Target génica para RR Usando movilidad electroforética cambio de ensayo (EMSA)
- Amplificar por PCR la región de 400 pb aguas arriba de la DVU3025 gen diana candidato, utilizando D. vulgar Hildenborough ADN genómico como plantilla, y un cebador no marcado hacia delante y una inverso 5'-biotina marcado con cebador.
Nota: Consejos para seleccionar un gen diana candidato: A menudo los RR se unen las regiones aguas arriba de su propio gen / operón, o pueden regular los genes codificados proximal. Si el RR tiene orthologs en otras especies, buscar los genes que se conservan en el barrio. Los métodos alternativos incluyen la elección de genes candidatos sobre la base de las predicciones regulón (por ejemplo, RegPrecise 5), o que predicen los promotores sigma54 dependiente de 6 para los RR que son en sí mismos sigma54-dependiente. - Ejecute el producto de PCR en un gel de agarosa al 1%, cortar el 400 bproducto de tamaño p, y purificar el ADN usando un gel extracción kit de limpieza.
- Mezclar proteína DVU3023 (0,5 pmol) con 100 fmol de ADN sustrato biotinilado en 10 mM Tris HCl, pH 7,5, KCl 50 mM, MgCl2 5 mM, DTT 1 mM, 25% de glicerol y 1 mg / ml de poli dI.dC (no -específica de ADN competidor) en un volumen total de 20 l. También establecer una reacción sin ninguna proteína como control. Se incuban las reacciones a temperatura ambiente en el banquillo durante 20 minutos.
- Nota: Estas son las condiciones generales que se indican y otros componentes de la reacción se pueden añadir según el RR de interés. Si se desea la activación, añadir acetil fosfato 50 mM a la reacción (a menudo RR se unirá ADN in vitro sin activación).
- Pre-ejecutar un mini 6% de gel de poliacrilamida TBE-0.5x prefabricado en tampón TBE 0,5 x a 100 V durante 30 min. Añadir 5 l de tampón de carga 5x (0,1% azul de bromofenol al 0,1% xilenocianol, 30% de glicerol en 1x TBE) para las reacciones de unión y de carga 18 l de las reacciones en el geléct. Corre a 100 V durante 2 h.
Nota: Con el fin de evitar el sobrecalentamiento, que puede conducir a la disociación de los complejos proteína-DNA, llenar la cámara de tampón exterior en el sistema de electroforesis con tampón de ejecución de tal manera que la mayoría del gel está aislado por tampón. Alternativamente, el gel puede llevar a cabo a 4 ° C. - Cortar una membrana de nylon cargada con el tamaño del gel y sumergirlo en TBE 0.5x durante al menos diez minutos. Retirar el gel de la casete. Cortar algunos cantos el gel y Sandwich del gel y la membrana entre dos papeles de filtro gruesos empapados en TBE 0.5x, y colocar dentro de un aparato de transferencia semiseca y hacerlo funcionar a 20 V durante 30 min.
- Coloque la membrana en el interior de un instrumento de luz UV reticulante comercial y establecer el tiempo de 3 min.
Nota: La membrana puede ahora ser almacenado en seco a temperatura ambiente durante varios días antes de proceder a la siguiente etapa. - Utilice un kit de detección de quimioluminiscencia disponible en el mercado que emplea un rábano peroxidación estreptavidina-caballoSE conjugado para desarrollar la transferencia según las instrucciones del fabricante.
- Imagen de la mancha usando un ordenador conectado a una cámara CCD equipada y buscar un cambio en la movilidad sustrato de ADN en presencia de la RR, lo que indica que el RR se une el ADN se está probando.
Nota: La especificidad del turno de proteína-DNA se puede probar mediante la inclusión en la reacción de un exceso de ADN competidor no marcado, que debe eliminar o disminuir el cambio visto.
3. Verifique Target Enrichment después Genomic DNA-proteína de unión
- Reacción de unión proteína-ADN genómico
- ADN genómico de cizallamiento a un tamaño medio de 500 pb por sonicación de 100 l de ADN genómico (a concentraciones de 100-200 ng / mu l) en un tubo de microcentrífuga de 1,5 ml usando una micropunta con 9 pulsos de baja amplitud de 1 seg, con 2 seg brecha entre cada uno.
Nota: Si las salpicaduras de ADN a los lados del tubo, girar hacia abajo el contenido después de cada 3 pulsos. Las condiciones exactas utilizadasvariará con el instrumento de ultrasonidos utilizado. Los fragmentos de ADN cortados pueden van desde 100-1000 pb, pero el tamaño medio deben estar dentro de 400-600 pb. El exceso de cizallamiento puede resultar en un menor número de sitios de unión intactos, y ya sea demasiado o demasiado poco cizallamiento afectará preparación de la biblioteca durante las etapas de amplificación de todo el genoma aguas abajo. - Mezclar 2-3 g de ADN genómico cortado con la proteína RR (0,5 pmol de DVU3023) en 10 mM de Tris-HCl, pH 7,5, DTT 1 mM, KCl 50 mM, MgCl2 5 mM, 25% de glicerol, y 50 mM de fosfato de acetilo. Incubar las reacciones a 25 º C durante 30 min. Transferencia de 10 l de esta reacción a un tubo de 1,5 ml y se etiqueta como ADN de entrada.
Nota: acetil fosfato se añade para activar la proteína por la fosforilación in vitro. La fosforilación estimula unión al ADN, pero muchas RR también se unen el ADN in vitro sin activación 7. La cantidad de proteína añadida a la reacción dependerá de la actividad de la preparación de proteína. El EMSA se puede utilizar para optarmizar esta cantidad.
- ADN genómico de cizallamiento a un tamaño medio de 500 pb por sonicación de 100 l de ADN genómico (a concentraciones de 100-200 ng / mu l) en un tubo de microcentrífuga de 1,5 ml usando una micropunta con 9 pulsos de baja amplitud de 1 seg, con 2 seg brecha entre cada uno.
- Affinity purificar el ADN unido a proteínas
- Añadir 30 l de resina de agarosa Ni-NTA a un tubo de microcentrífuga de 0,6 ml. Centrifugar a 100 xg durante 1 min para recoger la resina en la parte inferior, y eliminar el sobrenadante. Añadir 100 l de tampón de lavado (Tris-HCl 10, pH 7,5, 5 mM de MgCl 2, 50 mM de KCl, 25% de glicerol), película de la tubo para mezclar y centrifugar a 100 xg durante 2 min. Aspirar el sobrenadante.
- Añadir el resto de los 90 l de la reacción de unión a la resina de Ni-NTA se lavó y se incuba en un agitador rotatorio durante 30 min.
- Centrifugar a 100 xg durante 2 minutos, y retirar el sobrenadante (ADN no unido). Añadir 100 l de tampón de lavado: arrastra el tubo para mezclar el contenido, y se centrifuga a 100 xg durante 2 min. Aspirar el sobrenadante. Repita el paso de lavado dos veces más.
- Añadir 35 l de tampón de elución (pH tampón fosfato de sodio 20 mM de 8, NaCl 500 mM, imidazol 500 mM) a la resina y mezclar mediante agitación. Incubar en el banquillo a temperatura ambiente durante 5 min. Se centrifuga a 100 g durante 2 min. Transferir el sobrenadante a un nuevo tubo de 1,5 ml y se etiqueta como la fracción de ADN unido a proteínas.
- También añadir 35 l de tampón de elución a la entrada de ADN.
- Purificar la entrada y las fracciones de ADN unidas a proteínas utilizando un kit de purificación de PCR.
- Todo el genoma amplificar la entrada y muestras de ADN unidas a proteínas.
- Añadir 10 l de la entrada y el ADN unido a proteínas para separar los tubos de PCR. Añadir 1 l de 10x tampón de fragmentación, 2 l de tampón de preparación de la biblioteca, y 1 l de solución de Biblioteca de estabilización para cada muestra. Mezclar bien con un vórtex. Calentar en un termociclador a 95 ° C durante 2 minutos, y enfriar en hielo.
Nota: La cantidad inicial de ADN debe ser de al menos 10 ng con el fin de evitar la introducción de sesgo de amplificación. - Añadir 1 l de Biblioteca preparación de enzima, mezclar mediante pipeteo, y se incuba en el termociclador a 16 º C/20 min, 24 ° C/20 min, 37 ° C durante 20 min, 75 ° C / 5 minY mantener a 4 ° C.
- A cada tubo, añadir 47,5 l de agua, 7,5 l de 10x mezcla maestra de amplificación, y 5 l de polimerasa. Mezclar bien y se calienta a 95 ° C / 3 min, seguido por 20 ciclos de 94 ° C/15 seg, 65 ° C / 5 min. Mantenga reacciones a 4 ° C.
- Purificar las muestras de ADN amplificados utilizando un kit de purificación de PCR y medir la concentración de ADN espectrofotométricamente.
- Añadir 10 l de la entrada y el ADN unido a proteínas para separar los tubos de PCR. Añadir 1 l de 10x tampón de fragmentación, 2 l de tampón de preparación de la biblioteca, y 1 l de solución de Biblioteca de estabilización para cada muestra. Mezclar bien con un vórtex. Calentar en un termociclador a 95 ° C durante 2 minutos, y enfriar en hielo.
- Verifique el enriquecimiento de destino en el ADN unido a proteínas utilizando qPCR
- Cebadores Diseño qPCR para amplificar 200 pb de la región aguas arriba del gen diana verificado-EMSA (DVU3025) usando cualquier software de diseño de cebadores de libre acceso.
- Configure las reacciones de qPCR por triplicado para cada plantilla de ADN a cada conjunto de cebadores. Preparar una mezcla maestra para cada conjunto de cebadores. 1x mezcla maestra contiene 10 l de mezcla 2x SYBR Green qPCR, 0.5 M de cada cebador y agua hasta un total de 18 l. Alícuotas de 18 l de la mezcla maestra por pocillo de una placa de PCR de 96 pocillos.
- DILUTe la amplifican y entrada purificado y muestras de ADN unidas a proteínas a 5 ng / l con agua y utilizar como plantilla de ADN. Añadir 2 l de plantilla de ADN a los pocillos.
- Sellar la placa con una lámina de sellado qPCR ultra claro, girar hacia abajo la placa en una centrifugadora a 200 xg / 1 min. Colocar la placa en una máquina de qPCR en tiempo real. Ciclo de la siguiente manera usando el software asociado qPCR: 95 ° C / 1 min, y 40 ciclos de 95 º C/10 seg, 59 ° C/15 seg, y 70 ° C/35 seg.
- Establezca también las reacciones de qPCR por triplicado con cebadores para amplificar regiones aguas arriba de un gen no relacionado (control negativo).
- Cálculo de? C T C restando valor T de ADN unido a la proteína de la de la entrada de ADN. Calcular el enriquecimiento pliegue de gen diana en el ADN unido a proteínas como 2? C T.
Nota: Si la región aguas arriba de destino se enriqueció en el ADN unido a la proteína frente a la entrada de ADN, indica que las reacciones de unión y purificación por afinidad fueron successful. Continúe con el paso 4. Si no se observó enriquecimiento de la región aguas arriba de destino, repita las reacciones de unión (paso 3.1) con diferente cantidad de proteína.
4. De marcaje de ADN y Array hibridación
- Etiqueta de ADN de entrada con el ADN Cy3 y enriquecido con Cy5
ota: Cy3 y Cy5 tintes son sensibles a la luz y se debe tener cuidado para evitar la exposición de luz al mínimo.- Mezclar 1 g de ADN con 40 l Cy3/Cy5-labeled 9-dores y ajustar el volumen a 80 l con agua.
- El calor desnaturaliza a 98 ° C durante 10 minutos en la oscuridad (en un termociclador). Enfriamiento rápido en hielo durante 2 min.
- Añadir 2 l de Klenow de la polimerasa (3'-5'-exo -, 50000 U / ml), dNTPs 5 mM, y 8 l de agua a cada reacción, se mezcla bien, y se incuba a 37 ° C durante 2 h en la oscuridad (en un termociclador).
- Añadir EDTA a 50 mM para detener la reacción y la solución de NaCl a 0,5 M.
- Transferir las muestras a tubos de 1,5 ml containing 0,9 volumen de isopropanol, se incuba en la oscuridad durante 10 min, y se centrifuga a 12.000 xg durante 10 min. El sedimento debe ser de color rosa para el ADN marcado con Cy3 y azul para el ADN marcado con Cy5.
- Lavar el precipitado con etanol al 80% (500 l) y se centrifuga a 12.000 xg durante 2 min. Pellets de aire seco durante 5-10 minutos en la oscuridad.
Nota: Los pellets se puede almacenar a -20 ° C. - Resuspender el sedimento en 25 l de agua. Medir la concentración de ADN espectrofotométricamente.
- Piscina juntos 6 mg cada uno de los ADN marcado con Cy5-Cy3 y en un tubo de 1,5 ml y se seca al vacío en una centrifugadora a fuego lento en la oscuridad (cubrir la tapa de la centrífuga si es transparente).
Nota: Los pellets se puede almacenar a -20 ° C hasta que esté listo para la hibridación.
- La hibridación de microarrays
- Encienda el sistema de hibridación en 3-4 horas antes de su uso y ajuste la temperatura a 42 ° C.
- Preparar una mezcla maestra solución de hibridación de tal manera que la solución contiene 11,8 l 1x 2x hibridacióntampón zación, 4,7 l de hibridación componente A, y 0,5 l de oligo alineación.
- Resuspender los pellets en 5 l de agua. Añadir 13 l de esta mezcla a la muestra. Vortex durante 15 segundos, se incuba a 95 ° C en un baño seco durante 5 min. Mantenga las muestras a 42 ° C en el sistema de hibridación hasta que esté listo para la carga.
- Preparar la corredera-mezclador de microarrays, según el protocolo del fabricante.
- Coloque el conjunto de mezcladora de diapositivas dentro del sistema de hibridación. De carga 16 l de muestra en el orificio de llenado, sellar los puertos con una película adhesiva, gire la mezcla en en el sistema, e hibridar durante 16-20 horas a 42 ° C.
- Preparar los tampones de lavado 1x I (250 ml), II (50 ml) y III (50 ml) diluyendo los 10x buffers disponibles comercialmente en el agua, y para cada uno añada la TDT a 1 mM. Buffer Warm I a 42 ° C.
- Deslice el mezclador-slide en una herramienta de desmontaje, y colocar dentro de un recipiente lleno de Buffer cálida I. Peel mezclador off, mientras vigorosamente shaking la herramienta de desmontaje a mano.
- Colocar el portaobjetos en un recipiente con 50 ml de tampón de lavado I y agitar enérgicamente durante 2 minutos por lado.
- Transferencia de diapositivas en un segundo recipiente con tampón 50 ml Gel II y agitar enérgicamente durante 1 min a mano.
- Transferencia de diapositivas en un tercer recipiente con 50 ml de tampón de lavado III, y agitar vigorosamente durante 15 segundos con la mano.
- Borrar rápidamente los bordes del portaobjetos con una toalla de papel, y coloque en una parrilla corrediza. Centrifugar a 200 xg durante 2 minutos para secar la diapositiva. Coloque en caso de diapositiva, envuélvalo en papel de aluminio, y guárdelo en un desecador.
- El escaneo de la matriz
- Colocar el portaobjetos en el escáner de microarrays de acuerdo con las instrucciones del instrumento.
- Utilice un software de escáner para establecer las longitudes de onda como 532 nm = Cy3 y Cy5 = 635 nm, y las ganancias de fotomultiplicadores iniciales entre 350 y 400.
- Obtenga una vista previa de la diapositiva para localizar la matriz en la diapositiva. Seleccione la región de matriz para la exploración.
- Busque en la matriz y ajustar la configuración de fotomultiplicadores de tal manera que las características de la matriz son en su mayoría de color amarillo. El histograma debe mostrar las curvas de rojas y verdes superpuestas o lo más cerca entre sí como sea posible. Las curvas deben terminar por encima de los 10 -5 recuentos normalizados en la saturación.
- Guarde ambos 532 y 635 nm imágenes por separado.
- Análisis de datos de matriz
- Importe las imágenes en un software de análisis de matriz. Usando el software, crear dos informes par (. Par) para cada variedad (para Cy3 y Cy5 imágenes). Crear log 2 archivos proporción a escala utilizando los archivos de par. Uso Entre 2 archivos de relación para buscar picos utilizando una ventana deslizante de 500 pb. Mapa del loci pico a las regiones aguas arriba de los genes diana.
- Compruebe si el objetivo positivo (determinado mediante EMSA y confirmada por qPCR, DVU3025 en este ejemplo) aparece dentro de los mejores picos.
Nota: Si el objetivo positivo no se encuentra entre los mejores éxitos, es posible que el RR bajo consideración tiene varios objetivos de genes y replicar DAP-chips puede llevarse a cabo y los picos comunes a todas las repeticiones se puede utilizar para generar una lista de objetivos.
5. Binding Site Predicción Motif y Validación
- Recuperar secuencias para regiones de aguas arriba (400 pb) para los objetivos de genes superiores a las generadas por análisis de PCD-chip. Aplicar MEME en estas secuencias a través de microbios Online (meme.nbcr.net) para predecir los motivos.
Nota: las proteínas de unión Enhancer tales como reguladores de sigma54-dependientes pueden unirse varios cientos de pares de bases aguas arriba del sitio de inicio. Para otros reguladores transcripcionales, una región más corto tal como 200 pb aguas arriba será suficiente. - Diseño superior y oligómeros de ADN cadena inferior que contienen el motivo del sitio de unión previsto flanqueada por 10 bases en cada extremo. Ordenar la cadena superior 5 'con biotina.
- Mezclar los superiores e inferiores oligos Strand en relación 1:1,5 en Tris HCl 10, pH 8,0, EDTA 1 mM y NaCl 50 mM enun volumen total de reacción de 20 l en tubos de PCR. Calentar a 95 ° C durante 5 min en un termociclador, seguido de enfriamiento lento a 25 ° C. Diluir diez veces y el uso como sustrato dsDNA tipo salvaje para EMSA.
- Preparar sustratos modificados siguiendo los mismos pasos que el anterior, pero oligómeros de diseño para llevar a 4-6 sustituciones en las bases conservadas del motivo de unión.
- Configure reacciones de unión con el RR y el tipo salvaje o sustratos modificados y examinar el uso de la EMSA, como se describe en el paso 2.
- Nota: El motivo predicho se valida si el RR se une el sustrato de tipo salvaje pero no el modificado.
6. Conservación de Motif en otros bacterianas Especies Relacionadas
- Seleccione genomas de interés que contienen orthologs de DVU3023. Obtener la secuencia y anotación de archivos para los genomas de la NCBI sitio.
- Use un lenguaje de programación como Perl para escribir scripts que utilizarán las secuencias de motivos de los objetivos DAP-chip para construir un position matriz ponderada y usar la matriz para anotar motivos similares presentes en otros genomas secuenciados.
Resultados
El método anterior se aplicó para determinar los genes objetivos globales de los RR en el modelo de reducción de sulfato bacteria Desulfovibrio vulgaris Hildenborough 7. Este organismo tiene un gran número de TCSs representado por más de 70 RR, lo que indica la amplia variedad de posibles señales que detecta y responde a. Los análisis in vivo en las funciones de estos sistemas de señalización son difíciles de realizar, ya que sus señales y por lo tanto sus condiciones de activaci?...
Discusión
El método DAP-chip descrito aquí se utilizó con éxito para determinar los objetivos de genes durante varios RR en Desulfovibrio vulgaris Hildenborough 7 de los cuales uno se muestra aquí como un resultado representativo. Para DVU3023 RR, la elección de un objetivo de genes candidatos fue sencillo. DVU3025 está situada inmediatamente aguas abajo del gen RR, y los genes RR y de destino se conservan en varias especies Desulfovibrio, y adicionalmente DVU3025 tiene un promotor sigma54 depe...
Divulgaciones
Los autores no tienen ningún conflicto de intereses a revelar.
Agradecimientos
Damos las gracias a Amy Chen por su ayuda en la preparación para la filmación del video y para la demostración de la técnica. Este trabajo realizado por ENIGMA: Ecosistemas y redes integradas con Genes y Asambleas Molecular (http://enigma.lbl.gov), un programa científico del área de focalización en el Laboratorio Nacional Lawrence Berkeley, con el apoyo de la Oficina de Ciencia, Oficina de Biológica y Investigación del Medio Ambiente, del Departamento de Energía de los EE.UU. bajo el Contrato No. DE-AC02-05CH11231.
Materiales
| Name | Company | Catalog Number | Comments |
| HisTrapFF column (Ni-Sepharose column) | GE Lfe Sciences, Pittsburgh, PA, USA | 17-5255-01 | |
| Akta explorer (FPLC instrument) | GE Lfe Sciences, Pittsburgh, PA, USA | ||
| HiPrep 26/10 Desalting column | GE Lfe Sciences, Pittsburgh, PA, USA | 17-5087-01 | |
| Qiaquick Gel extraction kit | Qiagen Inc, Valencia, CA, USA | 28704 | |
| Biotin-labeled oligonucleotides | Integrated DNA Technologies | N/A | |
| 6% polyacrylamide-0.5x TBE precast mini DNA retardation gel | Life Technologies, Grand Island, NY, USA | EC63652BOX | Alternately, you can pour your own gel. |
| Nylon membrane | EMD Millipore, Billerica, MA, USA | INYC00010 | |
| Trans-Blot SD Semi-dry electrophoretic transfer cell | Biorad, Hercules, CA, USA | 170-3940 | |
| Extra thick blot paper, 8 x 13.5 cm | Biorad, Hercules, CA, USA | 170-3967 | |
| UV crosslinker Model XL-1000 | Fisher Scientific | 11-992-89 | |
| Nucleic Acid chemiluminescent detection kit (Pierce) | Thermo fisher Scientific, Rockford, IL, USA | 89880 | |
| Ni-NTA agarose resin | Qiagen Inc, Valencia, CA, USA | 30210 | |
| GenomePlex Whole genome amplification kit (Fragmentation buffer, library preparation buffer, library stabilization solution, library preparation enzyme, 10x amplification master mix, WGA polymerase) | Sigma-Aldrich, St. Louis, MO, USA | WGA2-50RXN | |
| Nanodrop ND-1000 | Thermo Scientific, Wilmington, DE, USA | For quantitation of DNA | |
| Perfecta Sybr Green SuperMix, with ROX | Quanta biosciences | 95055-500 | Any Sybr Green PCR mix may be used |
| PlateMax Ultra clear heat sealing film for qPCR | Axygen | ||
| [header] | |||
| 96-well clear low profile PCR microplate | Life Technologies, Grand Island, NY, USA | PCR-96-LP-AB-C | |
| Applied Biosystems StepOne Plus Real time PCR system | Life Technologies, Grand Island, NY, USA | 4376600 | Any real time PCR system may be used |
| Qiaquick PCR purification kit | Qiagen Inc, Valencia, CA, USA | 28104 | Any PCR clean up kit may be used |
| Cy3/Cy5-labeled nonamers | Trilink biotechnologies, San Diego, CA, USA | N46-0001, N46-0002 | |
| Klenow polymerase 50,000 U/ml, 3'-5' exo- | New England Biolabs, Ipswich, MA | M0212M | |
| Hybridization system | Roche-Nimblegen, Madison, WI, USA | N/A | This company no longer makes arrays or related items, so alternate sources such as Agilent or Affymetrix will need to be used. |
| Custom printed microarrays and mixers | Roche-Nimblegen, Madison, WI, USA | N/A | |
| Hybridization kit (2x Hybridization buffer, Hybridization component A, Alignment oligo) | Roche-Nimblegen, Madison, WI, USA | N/A | |
| Wash buffer kit (10x Wash buffer I, II, III, 1 M DTT) | Roche-Nimblegen, Madison, WI, USA | N/A | |
| GenePix 4200A microarray scanner | Molecular Devices, Sunnyvale CA, USA | This model has been replaced by superior ones | |
| GenePix Pro microarray software | Molecular Devices, Sunnyvale CA, USA | ||
| Nimblescan v.2.4, ChIP-chip analysis software | Roche-Nimblegen, Madison, WI, USA | N/A | |
Referencias
- Galperin, M. Y. Diversity of structure and function of response regulator output domains. Curr Opin Microbiol. 13, 150-159 (2010).
- Casino, P., Rubio, V., Marina, A. The mechanism of signal transduction by two-component systems. Curr Opin Struct Biol. 20, 763-771 (2010).
- Aparicio, O., Geisberg, J. V., Struhl, K. Chromatin immunoprecipitation for determining the association of proteins with specific genomic sequences in vivo. Curr Protoc Cell Biol. 17 (17), (2004).
- McCleary, W. R., Stock, J. B. Acetyl phosphate and the activation of two-component response regulators. J Biol Chem. 269, 31567-31572 (1994).
- Novichkov, P. S., et al. RegPrecise web services interface: programmatic access to the transcriptional regulatory interactions in bacteria reconstructed by comparative genomics. Nucleic Acids Res. 40, 604-608 (2012).
- Studholme, D. J., Buck, M., Nixon, T. Identification of potential sigma(N)-dependent promoters in bacterial genomes. Microbiology. 146 (12), 3021-3023 (2000).
- Rajeev, L., et al. Systematic mapping of two component response regulators to gene targets in a model sulfate reducing bacterium. Genome Biol. 12, 99 (2011).
- Crooks, G. E., Hon, G., Chandonia, J. M., Brenner, S. E. WebLogo: a sequence logo generator. Genome Res. 14, 1188-1190 (2004).
- Barbieri, C. M., Mack, T. R., Robinson, V. L., Miller, M. T., Stock, A. M. Regulation of response regulator autophosphorylation through interdomain contacts. J Biol Chem. 285, 32325-32335 (2010).
- Bourret, R. B. Receiver domain structure and function in response regulator proteins. Curr Opin Microbiol. 13, 142-149 (2010).
- Gao, R., Stock, A. M. Molecular strategies for phosphorylation-mediated regulation of response regulator activity. Curr Opin Microbiol. 13, 160-167 (2010).
- Hung, D. C., et al. Oligomerization of the response regulator ComE from Streptococcus mutans is affected by phosphorylation. J Bacteriol. 194, 1127-1135 (2012).
- Ladds, J. C., et al. The response regulator Spo0A from Bacillus subtilis is efficiently phosphorylated in Escherichia coli. FEMS Microbiol Lett. 223, 153-157 (2003).
- Wen, Y., Feng, J., Scott, D. R., Marcus, E. A., Sachs, G. Involvement of the HP0165-HP0166 two-component system in expression of some acidic-pH-upregulated genes of Helicobacter pylori. J Bacteriol. 188, 1750-1761 (2006).
- Liu, X., Noll, D. M., Lieb, J. D., Clarke, N. D. DIP-chip: rapid and accurate determination of DNA-binding specificity. Genome Res. 15, 421-427 (2005).
- Gossett, A. J., Lieb, J. D. DNA Immunoprecipitation (DIP) for the Determination of DNA-Binding Specificity. CSH Protoc. 2008, (2008).
- Mascher, T., et al. The Streptococcus pneumoniae cia regulon: CiaR target sites and transcription profile analysis. J Bacteriol. 185, 60-70 (2003).
- Park, P. J. ChIP-seq: advantages and challenges of a maturing technology. Nat Rev Genet. 10, 669-680 (2009).
- Lefrancois, P., Zheng, W., Snyder, M. ChIP-Seq using high-throughput DNA sequencing for genome-wide identification of transcription factor binding sites. Methods Enzymol. 470, 77-104 (2010).
- Ho, J. W., et al. ChIP-chip versus ChIP-seq: lessons for experimental design and data analysis. BMC Genomics. 12, 134 (2011).
- Liu, X., Lee, C. K., Granek, J. A., Clarke, N. D., Lieb, J. D. Whole-genome comparison of Leu3 binding in vitro and in vivo reveals the importance of nucleosome occupancy in target site selection. Genome Res. 16, 1517-1528 (2006).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados