需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
受体动力学通过单分子荧光显微镜的高分辨率时空分析
摘要
This protocol describes how to use total internal reflection fluorescence microscopy to visualize and track single receptors on the surface of living cells and thereby analyze receptor lateral mobility, size of receptor complexes as well as to visualize transient receptor-receptor interactions. This protocol can be extended to other membrane proteins.
摘要
单分子显微术正在成为一个强大的方法来分析信号分子,特别是关于那些方面( 如 。,动力学,不同的国家和人群,短暂的相互作用共存),它们通常隐藏在合奏测量,如行为那些与标准生化或显微镜方法获得。因此,动态事件,如受体 - 受体相互作用,可以实时跟踪与高时空分辨率的活细胞。这个协议描述的基础上以小而明亮的有机荧光团和全内反射荧光(TIRF)显微镜直接可视化单一受体的活细胞的表面上的标签的方法。这种方法允许一个精确定位的受体,测定受体复合物的大小,并捕捉动态事件,如瞬时受体 - 受体相互作用。该协议提供了如何PE的详细说明rform单分子实验,包括样品制备,图像采集和图像分析。作为一个例子,这种方法的应用,分析2的G-蛋白偶联受体, 即 ,,β2 -肾上腺素能受体和γ-氨基丁酸B型(GABA B)受体,报道。该协议可以适于其他膜蛋白和不同的细胞模型,转染方法和标记策略。
引言
位于细胞表面上的受体感知细胞外环境和各种刺激,如气味分子,离子,小神经递质和大蛋白激素的反应。细胞膜的流体性质允许受体和其他膜蛋白的移动。这是为蛋白质复合物和瞬时蛋白质 - 蛋白质相互作用,如那些用于通过受体组装成的功能单元和信号转导到细胞内发生的形成是必不可少的。例如,G-蛋白偶联受体(GPCR),它构成细胞表面受体1的最大家族,已经提出了以形成di-/oligomers,这似乎是参与信号转导的微调,调节和可能有重要的生理和药理后果2-5。
单分子显微镜具有高spatiotemp直接可视化的巨大潜力口服分辨率位于活细胞,包括其相关联的表面上的单个受体的动态行为,以形成二聚体和更高阶的分子复合物6-10。这比标准的生化和显微镜方法,通常报告的分子数千或数百万的平均行为提供了几个优点。
具有足够明亮,耐光荧光蛋白标记是必不可少的单分子显微镜。该协议需要在近期推出SNAP标签11的优势,以共价连接的小而明亮的有机荧光细胞表面受体。 SNAP是从人类的DNA修复酶的O 6 -烷基鸟嘌呤-DNA烷基转移酶,它可以不可逆地标记有荧光团标记的苄基鸟嘌呤(荧光团- BG)衍生物衍生的20 kD蛋白标记。 CLIP,从SNAP派生的进一步工程化标签,可以代替标记荧光基团-Conjugated苄基胞嘧啶衍生物12。
在报道这个手稿的协议说明如何转染和标签SNAP-11标记的受体与小的有机荧光,并使用全内反射荧光(TIRF)显微镜可视化单一受体或受体复合物的活细胞10的表面上。该报告协议结果在细胞外SNAP-标记的细胞表面蛋白10> 90%标记效率。关于如何使用单分子数据来分析受体复合物的大小和流动性,以及捕捉瞬时受体 - 受体相互作用的进一步信息,提供。整个协议的示意流是在图1,作为一个例子,中国仓鼠卵巢(CHO)细胞,SNAP-标签的G-蛋白偶联受体(GPCRs)的转染,然后用荧光团-BG衍生物作为标记以及其应用的量化y和显示器受体di-/oligomerization描述。这个协议可以扩展到其它细胞表面蛋白和荧光标记( 例如 ,,CLIP),以及其他的转染和标记方法。
研究方案
1,样品制备
- 清洗盖玻片
注意:在通风橱中操作。- 用干净的镊子把盖玻片(直径为24毫米)插入分隔各个盖玻片盖玻片持有人。
- 把夹持器与盖玻片到烧杯中,并加入氯仿,直到盖玻片覆盖。用铝箔覆盖烧杯以减少在超声波仪浴1小时,在室温下蒸发,并超声处理。就拿盖玻片架从烧杯,让盖玻片干。
- 重复步骤1.1.2用5M NaOH溶液代替氯仿。
- 取盖玻片夹持器进入一个新的烧杯中,并洗涤3次,用蒸馏水。把清洁的盖玻片的玻璃细胞培养板中填充有100%的乙醇。
- 校准样品的制备
- 在适当的溶剂中溶解的荧光染料。
- 制备荧光染料测距1:10系列稀释下午1时至1纳米的过滤除菌(0.22微米)的水。
- 取存储在100%乙醇清洗盖玻片和洗涤用过滤灭菌的水。现货20微升在一个单独的清洗盖玻片每个荧光染料稀释。让盖玻片下无菌罩干燥。保护盖玻片从光线和灰尘,直到使用。使用这些样本来估计单分子荧光的强度(参见步骤3)。
- 转染
- 培养CHO细胞以1:1的Dulbecco改良的Eagle培养基/营养混合物F-12(DMEM/F12)补充有10%胎牛血清(FBS),100 U / ml青霉素和100微克/毫升链霉素,在37℃,在5%的CO 2 注:使用无酚红的媒体在整个实验中,以尽量减少自体荧光。
- 取清洗盖玻片从100%乙醇溶液中,用无菌磷酸盐缓冲盐水(PBS)冲洗,然后放置1盖玻片到6孔细胞培养PL的各孔中吃了。
- 胰蛋白酶消化,计数和种子的CHO细胞以3×10 5个细胞/孔在含有盖玻片的6孔细胞培养板的密度。让我们在孵化器,以达到大约细胞生长(37℃,5%CO 2)为24小时。 80%汇合时,这是最佳的细胞密度转染。
- 对于每孔,稀2微克所需的质粒DNA( 例如 。,SNAP-标记的β2 -肾上腺素受体 )和6μL脂质体2000中含有500微升OPTIMEM媒体两个独立的管道。在室温下孵育5分钟。
- 从步骤1.3.4结合的解决方案为一体管和组合,以获得转染混合物。在室温下孵育转染混合物20分钟。
- 在潜伏期(1.3.5),取CHO细胞和预热(37℃)的PBS洗两次。取代的PBS用补充有10%FBS的无酚红的DMEM/F12培养基中以1毫升/孔,但没有抗生素。
- 添加整个转染离子混合物(1ml)中从步骤1.3.5滴加到每个孔中,并轻轻摇动平板来回,以确保完全混合。
- 孵育2〜4小时,在37℃,5%CO2,之后立即进行下一个步骤。 注意:这些转染条件进行了优化,以实现受体密度<0.45粒/ 2微米,这是适用于单分子成像。调整可能使用不同的细胞,构造或试剂时是必需的。
- 蛋白标记
- 稀释1微升的荧光团-BG原液在补充有10%FBS的取得1μM的终浓度1毫升DMEM/F12培养基。取转染细胞从孵化器和预热(37℃),PBS洗涤两次。更换PBS以1毫升1μM的荧光团-BG溶液并孵育20分钟,在37℃,5%CO 2的培养箱中。
- 保温后,洗涤细胞3用补充有10%FBS的DMEM/F12培养基次,接着用5分钟温育,每次在37℃下取盖玻片(有标记的细胞),用钳子将其放入成像室。
- 用300μl成像缓冲液洗两次。加入300微升新鲜成像缓冲区并立即着手成像(第2部分)。
2,图像采集
注:使用全内反射荧光(TIRF)显微镜,配有一个油浸高数值孔径的物镜( 例如 ,100X magnification/1.46数值孔径),合适的激光器(例如,405纳米,488纳米,561纳米和645纳米二极管激光器),电子倍增电荷耦合器件(EMCCD)相机,孵化器和一个温度控制形象化单个荧光分子。
- 设置所需的显微镜参数, 即得 。,激光线,TIRF角度(此参数控制着穿透渐逝场的化深度),曝光时间,帧速率和每片10影像数。保持器/孵化器和温度控制总是避免温度漂移和结露。
- 把一滴浸油对显微镜的100X目标。放置在成像腔室与标记的细胞到显微镜的试样保持器,并采用明场照明带来的细胞焦点。
- 切换到全内反射荧光照明。保持激光功率尽可能地低,以允许搜索的期望的细胞,但在同一时间最小化的光漂白。
- 选择所需的细胞和精细调整焦点。设置激光功率的电平,使单个荧光团的可视化。获取图像序列和原始图像序列文件保存为。TIFF。
3,校准(在玻璃和单体/二聚体受体控制单个荧光基团)
- 组装每个校准SAMPLe作为在成像室1.2中所述的方法制备。将每个样品在显微镜上,并选择含有分离良好的受限衍射斑点,漂白在单一步骤中注意样品:这些点代表单个分子的荧光染料。
- 如步骤2中所述收购全内反射荧光图像序列重要提示 :相同的成像参数必须被用于所有的实验,包括那些进行校准。
- 进行检测,并在4.1详细的跟踪分析 - 4.2。提取作为在4.2.6中描述的每个粒子的强度。从这些数据,计算平均值(μ)和单荧光团的强度的标准偏差(σ)。
- 可选:执行对转染的细胞与单体的细胞表面受体( 如 CD86)的同样的分析,N末端 具有标记的SNAP 10的任一个或两个拷贝和标记有荧光团-BG衍生物。福卢瓦特上述用于在玻璃上的单荧光团的步骤。如Calebiro,D. 等人描述的估算标记效率。10
4,图像分析
- 图像序列的准备
- 使用图像处理软件( 如。,ImageJ的 ),裁剪图像。
- 除个别帧作为单独的。tiff图像在一个新的文件夹,显示每个图像帧号上。
- 通过沿细胞轮廓绘制感兴趣区(ROI)的区域,并使用测量工具在ImageJ的或其他软件类似的工具测量单元面积。使用此值除以颗粒的总数在电影由单元表面面积的开头来计算粒子的密度。
- 粒子探测和跟踪
注:使用非商业软件,如U-13的轨道 ,在Matlab工作环境,自动检测并跟踪单个受体颗粒。
注:U 型轨道算法是基于多假设跟踪方法。这种方法通过构建成本矩阵,其中,在一帧给定的粒子对应于一个给定的粒子,在下一帧时,会出现,消失或合并/拆分与/从其他颗粒中的个体的概率被分配链路帧之间的颗粒。 ,全球最小的成本解决方案, 即得 。,一个具有最高概率,最终选定。这也使得跟踪暂时消失的粒子,引起荧光闪烁的典型现象。 U型轨道 (2.1.0)的最新版本具有促进这些分析的执行图形用户界面。- 从MATLAB命令提示符下,键入"movieSelectorGUI"打开影片选择界面。按照说明创建一个新的电影数据库从以前保存的单独的图像开始(见4.1.2)。
- 提供在像素大小为nm,以秒为单位的时间间隔,数值孔径,相机的位深度和荧光团的发射波长,所需颗粒探测和跟踪。保存电影资料库。
- 从电影选择界面,运行分析,选择" 单粒子 "作为对象的类型。将出现一个新的窗口,可以定义用于粒子探测和跟踪的参数。开始使用默认的参数。后来,调整这些参数,如果检测和/或跟踪的质量不理想( 如有些颗粒不检测或轨道是支离破碎) 可选:根据跟踪设置,勾选" 导出跟踪结果矩阵格式 "存储在一个单一的矩阵(字段称为"trackedFeaturedInfo的坐标和各颗粒的振幅")。对于这些参数的详细说明,请参阅U型轨道文档。
- 运行检测算法。该算法自动确定的位置和强度上的每个衍射限制斑( 即 ,单一的受体/受体复合物)通过拟合二维高斯函数的标准偏差等于显微镜的周围局部强度最大值的点扩散函数的背景。然后,运行跟踪算法。存储分析的结果在一个。mat文件。
- 使用"movieViewer"套路,包含在U型轨道包装,或类似的自定义的可视化的轨道,并检查检测和质量跟踪。
- 打开。mat文件以查看所跟踪的粒子在每一帧的位置和幅度( 即 。,强度)。在步骤4.2.4生成的数据被包含在tracksFinal AR的tracksCoordAmpCG字段射线和/或在trackedFeaturedInfo。从粒子的总数计算检测到的粒子密度通过在4.1.3测定细胞表面的面积除以该值。
- 可选:使用粒子坐标随着时间的推移(见4.2.6)来分析受体粒子的运动。计算用Matlab或类似软件的均方位移(MSD)和扩散系数(D)。每个粒子,每一个时间间隔(Δt)的考虑,使用下面的公式计算出的均方位移(MSD):
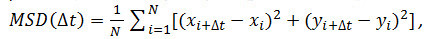
其中Δt是在帧中的时间间隔,N是所分析的步骤数,x和y是粒子的x和y在由索引指示的帧坐标。使用MSD随时间曲线来评估一个给定的粒子的运动的类型:线性关系说明自由扩散( 即 。,布朗运动),一个正曲率( 即 。,曲线看起来像一个抛物线)提出定向运动,负曲率指示局限于运动14。的情况下自由地漫射颗粒,计算出每个粒子由以下方程拟合得到的MSD数据的扩散系数(D):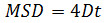
- 颗粒大小与高斯拟合方法计算
注意:一旦校准样品(单荧光团上的玻璃和/或荧光标记的单体受体)的强度分布是已知的,粒子强度的分布进行混合高斯拟合在图像序列的开始,以确定受体复合物的大小( 即 ,,受体每个颗粒的数量)10。利用Matlab或s进行这些分析imilar软件。- 通过平均粒子从第一帧中的强度到框架前的强度的第一个发生变化(在大多数情况下,由于光漂白的减少)计算每个粒子的强度。
- 根据下面的公式进行混合高斯拟合:
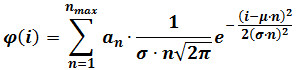
其中,φ(i)为具有强度粒子I的频率,n是该组件数,αn是有助于向分量n的高度的参数,μ和σ的平均值和参考单个荧光强度的标准偏差(计算方法为在步骤3中所述) 注意 :确定吨他部件(正最大值 )对每个图像顺序逐步增加N 最大 ,直到另外的组件的最大数量不不再产生在统计上更好的贴合,作为判断F检验。 - 可选:( 例如 ,一个400帧序列的最后60帧)上获得对电影的最后一个画面的亮度分布进行混合高斯拟合。更换μ和σ与重复步骤4.3.2本拟合后得到的值,提供精致估计这些参数,和。
- 计算混合高斯拟合的各组分的曲线(AUC)下的面积。计算不同的大小(即,单体,二聚体,三聚体等),通过由整个分布的AUC除以各成分的AUC值的受体粒子的相对丰度。
- 可选的:从不同的小区和相应的颗粒密度(如在4.2.6中描述计算),以生成重复的地方具有不同大小颗粒的分布是相关与粒子密度10使用的数据。
- 计算粒度的步骤拟合方法
注:使用步骤拟合分析作为一种替代方法,以确定受体复合物10的大小。依据这种分析是单一荧光团的结果在其瞬间消失的光诱导损伤(光漂白) -包含n个荧光团从而颗粒预期逐步漂白剂产生的强度分布与n个步骤。- 从U型轨道或类似检测/跟踪软件(参见4.2.6)生成的。mat文件提取每个粒子的强度分布。
- 使用步骤拟合算法,如1提出文献。 10,计数的漂白步骤,为每个粒子的数目。
- 可选 :使用结果生成的分布呈现出不同大小的受体粒子的相对丰度和以前的混合高斯拟合的结果描述粒子密度关联起来(见4.3)。
结果
所描述的协议可以被应用到各种不同的膜蛋白。作为一个例子,与β2 -肾上腺素能受体和GABA受体乙获得代表性结果列10。由于从单个分子的荧光信号弱,尽量减少背景荧光是第一关键步骤,成功的结果。因此,要广泛使用清洁的盖玻片( 图2A),以及最大限度地减少样品自发荧光( 例如 ,通过使用无酚红培养基)是很重要的。下一个步骤是确定单个荧?...
讨论
所描述的协议允许的空间布置,流动性和细胞表面受体复合物的大小在单分子水平的分析。相比,使用荧光蛋白,具有小的有机荧光团,它们是明亮,更耐光的标签,具有允许单个受体粒子的扩展可视化的优点。由于非常低的表达水平得以实现(<0.45受体粒子个/μm2),受体和其他膜蛋白的性质可以在密度不超过生理相关的分析。此外,受体刺激激动剂10或其它操作的影响,例如...
披露声明
The authors declare that they have no competing financial interests.
致谢
The development of this protocol was supported by grants from the European Research Council (Advanced Grant TOPAS to M.J.L.) and the Deutsche Forschungsgemeinschaft (Grants CA 1014/1-1 to D.C. and SFB487 to M.J.L.). T.S. was supported by the Alexander von Humboldt Foundation.
材料
| Name | Company | Catalog Number | Comments |
| Chloroform | AppliChem GmbH | A1585 | CAUTION: toxic and irritating substance as well as a possible carcinogen |
| NaOH | Sigma-Aldrich | S8045 | CAUTION: strong base and highly corrosive reagent |
| Absolute ethanol | Sigma-Aldrich | 32205 | |
| Glass coverslip | Marienfeld-Superior | 111640 | 24 mm diameter, 0.13-0.16 mm thickness |
| 0.2 mm sterile filter | Sarstedt | 83.1826.001 | |
| CHO cells | ATCC, USA | ATCC CCL-61 | Chinese hamster ovary cell line |
| 6-well cell culture plate | Nunc | 140675 | |
| DMEM/F-12 medium | GIBCO, Life Technologies | 11039-021 | Phenol-red free medium |
| Fetal bovine serum | Biochrom | S 0115 | |
| Penicillin - streptomycin | Pan Biotech GmbH | P06-07 100 | |
| Trypsin-EDTA | Pan Biotech GmbH | P10-23100 | |
| Lipofectamine 2000 | Invitrogen, Life Technologies | 11668-019 | |
| Opti-MEM I Reduced Serum Medium | Invitrogen, Life Technologies | 31985-047 | |
| Fluorophore-conjugated benzylguanine | New England BioLabs | S9136S | SNAP-Surface Alexa Fluor 647. Make a 1 mM stock solution in DMSO. Store at -20°C. |
| DMSO | AppliChem GmbH | A1584 | |
| Imaging buffer: | 137 mM NaCl, 5.4 mM KCl, 2 mM CaCl2, 1 mM MgCl2, 10 mM HEPES, pH 7.3, sterile-filtered | ||
| NaCl | AppliChem GmbH | A1371 | |
| KCl | AppliChem GmbH | A3582 | |
| CaCl2 | AppliChem GmbH | A2303 | |
| MgCl2 | AppliChem GmbH | A3618 | |
| HEPES | AppliChem GmbH | A3724 | |
| Imaging chamber | Molecular Probes, Life Technologies | A-7816 | Attofluor Cell Chamber, for microscopy |
| TIRF-M | Leica | Model: DMI6000B | |
| TIRF objective | Leica | 11 506 249 | HCX PL Apo 100x/1.46 Oil CORR |
| EM-CCD camera | Roper Scientific | Photometrics Cascade 512B | |
| Temperature controller | Pecon | Tempcontrol 37-2 digital | |
| ImageJ software | NIH, USA | http://rsbweb.nih.gov/ij | |
| u-track software | Laboratory for computational cell biology, Dept. of Cell Biology, Harvard Medical School, USA | http://lccb.hms.harvard.edu/software.html | |
| Matlab software | The MathWorks, USA |
参考文献
- Pierce, K. L., Premont, R. T., Lefkowitz, R. J. Seven-transmembrane receptors. Nat Rev Mol Cell Biol. 3, 639-650 (2002).
- Angers, S., Salahpour, A., Bouvier, M. Dimerization: an emerging concept for G protein-coupled receptor ontogeny and function. Annu Rev Pharmacol Toxicol. 42, 409-435 (2002).
- Ferré, S., et al. Building a new conceptual framework for receptor heteromers. Nat Chem Biol. 5, 131-134 (2009).
- Milligan, G. G. protein-coupled receptor hetero-dimerization: contribution to pharmacology and function. Br J Pharmacol. 158, 5-14 (2009).
- Lohse, M. J. Dimerization in GPCR mobility and signaling. Curr Opin Pharmacol. 10, 53-58 (2010).
- Ulbrich, M. H., Isacoff, E. Y. Subunit counting in membrane-bound proteins. Nat Methods. 4, 319-321 (2007).
- Triller, A., Choquet, D. New concepts in synaptic biology derived from single-molecule imaging. Neuron. 59, 359-374 (2008).
- Hern, J. A., et al. Formation and dissociation of M1 muscarinic receptor dimers seen by total internal reflection fluorescence imaging of single molecules. Proc Natl Acad Sci U S A. 107, 2693-2698 (2010).
- Kasai, R. S., et al. Full characterization of GPCR monomer-dimer dynamic equilibrium by single molecule imaging. J Cell Biol. 192, 463-480 (2011).
- Calebiro, D., et al. Single-molecule analysis of fluorescently labeled G-protein-coupled receptors reveals complexes with distinct dynamics and organization. Proc Natl Acad Sci U S A. 110, 743-748 (2013).
- Keppler, A., et al. A general method for the covalent labeling of fusion proteins with small molecules in vivo. Nat Biotechnol. 21, 86-89 (2003).
- Gautier, A., et al. An engineered protein tag for multiprotein labeling in living cells. Chem Biol. 15, 128-136 (2008).
- Jaqaman, K., et al. Robust single-particle tracking in live-cell time-lapse sequences. Nat Methods. 5, 695-702 (2008).
- Saxton, M. J., Jacobson, K. Single-particle tracking: applications to membrane dynamics. Annu Rev Biophys Biomol Struct. 26, 373-399 (1997).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。