Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Hochauflösende Räumlich-zeitliche Analyse der Rezeptordynamik von Einzelmolekül-Fluoreszenzmikroskopie
In diesem Artikel
Zusammenfassung
This protocol describes how to use total internal reflection fluorescence microscopy to visualize and track single receptors on the surface of living cells and thereby analyze receptor lateral mobility, size of receptor complexes as well as to visualize transient receptor-receptor interactions. This protocol can be extended to other membrane proteins.
Zusammenfassung
Einzelmolekül-Mikroskopie ist als ein leistungsfähiges Konzept, um das Verhalten von Signalmolekülen, insbesondere in Bezug auf jene Aspekte (zB., Kinetik, Koexistenz der verschiedenen Staaten und Bevölkerungen, transiente Wechselwirkungen), die in der Regel in Ensemblemessungen versteckt sind, wie die sich abzeichnen die mit biochemischen Standard oder Mikroskopieverfahren erhalten. So, dynamische Ereignisse, wie Rezeptor-Rezeptor-Wechselwirkungen, kann in Echtzeit in einem lebenden Zelle mit hoher raum-zeitlichen Auflösung verfolgt werden. Dieses Protokoll beschreibt eine Methode, die auf der Markierung mit kleinen und hellen organischen Fluorophore und totale interne Reflexion (TIRF) Mikroskopie einzelner Rezeptoren auf der Oberfläche von lebenden Zellen direkt sichtbar zu machen. Dieser Ansatz erlaubt es, genau zu lokalisieren Rezeptoren, messen Sie die Größe der Rezeptor-Komplexe, und erfassen dynamische Vorgänge, wie transient receptor-Rezeptor-Wechselwirkungen. Das Protokoll enthält eine detaillierte Beschreibung, wie perform eine Einzelmolekül-Experiments, einschließlich Probenvorbereitung, Bildaufnahme und Bildanalyse. Als ein Beispiel der Anwendung dieses Verfahrens auf zwei G-Protein-gekoppelten Rezeptoren zu analysieren, dh., Β 2-adrenergen und γ-Aminobuttersäure Typ B (GABA)-Rezeptor berichtet. Das Protokoll kann auch für andere Membranproteine und verschiedene Zellmodelle, Transfektionsmethoden und Markierungsstrategien anpassen.
Einleitung
Rezeptoren auf der Zelloberfläche zu erfassen und die extrazelluläre Umgebung reagieren auf eine Vielzahl von Stimuli, wie z. B. Duftstoffe, Ionen, Neurotransmittern kleinen und großen Proteinhormone. Die Flüssigkeit Natur der zellulären Membranen ermöglicht Bewegungen von Rezeptoren und anderen Membranproteinen. Dies ist wichtig für die Bildung von Proteinkomplexen und dem Auftreten von transienten Protein-Protein-Wechselwirkungen, wie sie von Rezeptoren verwendet werden, um in Funktionseinheiten zusammenzubauen und zu transduzieren Signale ins Zellinnere. Zum Beispiel G-Protein-gekoppelten Rezeptoren (GPCRs), die die größte Familie von Zelloberflächenrezeptoren 1 ausmachen, sind vorgeschlagen worden, um di-/oligomers, die in der Feinabstimmung Regulierung der Signaltransduktion beteiligt zu sein scheint und bilden könnte wichtige physiologische und pharmakologische Auswirkungen haben 2-5.
Einzelmolekül-Mikroskopie hat das große Potential der Visualisierung direkt mit hoher spatiotempMund Auflösung das dynamische Verhalten der einzelnen Rezeptoren auf der Oberfläche von lebenden Zellen, einschließlich ihrer Verbindung angeordnet ist, um Dimere und höhere Ordnung Molekülkomplexe 6-10 bilden. Dies bietet mehrere Vorteile gegenüber Standard biochemischen und Mikroskopieverfahren, die in der Regel den Durchschnittswert Verhalten von Tausenden oder Millionen von Molekülen.
Proteinmarkierung mit einem ausreichend hell und photo Fluorophor für Einzelmolekül-Mikroskopie von wesentlicher Bedeutung. Dieses Protokoll nutzt die kürzlich eingeführten SNAP-Tag 11 bis kovalent klein und hell organischen Fluorophore Zelloberflächenrezeptoren. SNAP ist ein 20 kD Protein-Tag aus der menschlichen DNA-Reparaturenzym O 6-Alkylguanin-DNA Alkyltransferase, die irreversibel mit fluorophorkonjugierten Benzylguanin (Fluorophor-BG)-Derivate bezeichnet werden kann, abgeleitet. CLIP, eine weitere technisch-Tag aus SNAP abgeleitet, kann stattdessen mit Fluorophor-c gekennzeichnet werdenonjugated benzylcytosine Derivate 12.
Das Protokoll in diesem Manuskript berichtet, erklärt, wie Transfektion und Label-SNAP-11-Rezeptoren getaggt mit kleinen organischen Farbstoffen und benutzen Totalreflexion (TIRF) Mikroskopie einzelne Rezeptoren oder Rezeptor-Komplexe auf der Oberfläche lebender Zellen 10 visualisieren. Die berichteten Ergebnisse im Protokoll> 90% Markierungseffizienz eines extrazellulär SNAP-markierten Zelloberflächenprotein 10. Weitere Informationen darüber, wie Einzelmolekül-Daten verwenden, um die Größe und die Mobilität von Rezeptor-Komplexe zu analysieren, als auch transient receptor-Rezeptor-Wechselwirkungen zu erfassen, bereitgestellt. Eine schematische Ablauf des gesamten Protokolls ist in Fig. 1 angegeben. Als Beispiel die Transfektion von Chinese Hamster Ovary (CHO)-Zellen, die mit SNAP-markierten G-Protein-gekoppelten Rezeptoren (GPCRs) durch Markierung mit einem Fluorophor-BG-Derivat wie folgt sowie ihre Anwendung auf quantify und Monitor-Rezeptor di-/oligomerization beschrieben. Dieses Protokoll kann auch für andere Zelloberflächenproteine und fluoreszierende Markierungen (zB., CLIP), sowie zu anderen Transfektions-und Markierungsverfahren erweitert werden.
Protokoll
1. Probenvorbereitung
- Deckglasreinigung
HINWEIS: Arbeiten unter einer Abzugshaube.- Verwenden Sie saubere Pinzette Deckgläschen (24 mm Durchmesser) in einem Deckglas Halter, die einzelnen Deck trennt platzieren.
- Setzen Sie die Halterung mit Deckgläsern in ein Becherglas, und fügen Chloroform, bis die Deckgläschen abgedeckt. Das Becherglas wird mit Aluminiumfolie, die Verdunstung und beschallen in einem Ultraschallbad für 1 h bei RT reduzieren. Nehmen Sie die Deckhalter aus dem Becherglas und lassen Sie die Deckgläser trocken.
- Wiederholen Sie Schritt 1.1.2 mit 5 M NaOH-Lösung anstelle von Chloroform.
- Nehmen Sie die Deckhalter in eine neue Becherglas gegeben und dreimal mit destilliertem Wasser. Setzen gereinigt Deck in einem Glas-Zellkulturplatte mit 100% Ethanol gefüllt.
- Herstellung der Eichproben
- Auflösen fluoreszierenden Farbstoff in einem geeigneten Lösungsmittel.
- Bereiten Sie eine serielle 01.10 Verdünnung der Fluoreszenzfarbstoff im Bereichvon 01.00 bis 1 nM in Filter sterilisiert (0,22 um) Wasser.
- Nehmen gereinigt Deckgläser in 100% Ethanol gelagert und waschen mit Filter-sterilisiertem Wasser. Punkt 20 ul jeder Fluoreszenzfarbstoff Verdünnung auf einem separaten Deckglas gereinigt. Lassen Sie die Deckgläser trocknen unter einem sterilen Abzug. Schützen Sie die Deckgläser, vor Licht und Staub bis zur Verwendung. Mit diesen Proben, die Intensität der einzelnen fluoreszierenden Molekülen (siehe Schritt 3) zu schätzen.
- Transfektion
- CHO-Zellen in Kultur 1:1 Dulbeccos modifiziertem Eagle Medium / Nährstoffgemisch F-12 (DMEM/F12) mit 10% fötalem Rinderserum (FBS), 100 U / ml Penicillin und 100 ug / ml Streptomycin bei 37 ° C, supplementiert . 5% CO 2 HINWEIS: Phenolrot-freien Medien während des Experiments zu Autofluoreszenz zu minimieren.
- Nehmen gereinigt Deckgläser aus 100% Ethanol-Lösung, waschen Sie sie mit steriler phosphatgepufferter Kochsalzlösung (PBS) und legen Sie ein Deckglas in jedes Well einer 6-Well-Zellkultur plaß.
- Trypsinize, zählen und Samen CHO-Zellen in einer Dichte von 3 x 10 5 Zellen / Well in 6-Well-Zellkulturplatte mit den Deckgläsern. Lassen sich die Zellen (37 ° C, 5% CO 2) wachsen in einem Inkubator für 24 Stunden, um einen max. 80% Konfluenz, die die optimale Zelldichte für die Transfektion ist.
- Für jeden gut verdünnen 2 ug des gewünschten Plasmid-DNA (zB., Β 2-adrenergen Rezeptor-SNAP-tagged) und 6 ul Lipofectamine 2000 in zwei getrennten Röhren mit 500 ul OptiMem Medium. Inkubieren bei Raumtemperatur für 5 min.
- Kombinieren Sie die Lösungen aus Schritt 1.3.4 in ein Rohr und mischen, um eine Transfektion Mischung zu erhalten. Inkubieren der Transfektion bei RT für 20 min.
- Während der Inkubation (1.3.5), nehmen Sie die CHO-Zellen und waschen zweimal mit vorgewärmten (37 ° C) PBS. Ersetzen PBS mit 1 ml / Vertiefung phenolrotfreiem DMEM/F12-Medium mit 10% FBS, aber keine Antibiotika.
- Fügen Sie den gesamten TransfektionIonen-Mischung (1 ml) aus Schritt 1.3.5 tropfenweise in jede Vertiefung und schaukeln die Platte hin und her, um eine vollständige Durchmischung zu gewährleisten.
- Inkubation für 2 bis 4 Stunden bei 37 ° C in 5% CO 2 und gehen unmittelbar danach zum nächsten Schritt. HINWEIS: Die Transfektion Bedingungen wurden optimiert, um Rezeptordichten <0,45 Partikel / &mgr; m 2, die für Einzel zu erzielen Molekül-Bildgebung. Anpassungen könnten bei der Verwendung von verschiedenen Zellen, Konstrukte oder Reagenzien erforderlich.
- Proteinmarkierungs
- 1 &mgr; l der verdünnten Fluorophor-BG-Stammlösung in 1 ml DMEM/F12 Medium mit 10% FBS, um eine Endkonzentration von 1 uM erhalten. Nehmen Sie die transfizierten Zellen aus dem Inkubator und waschen zweimal mit vorgewärmten (37 ° C) PBS. Ersetzen PBS mit 1 ml von 1 uM Fluorophor-BG-Lösung und Inkubation für 20 min bei 37 ° C 5% CO 2 im Brutschrank.
- Nach der Inkubation, Waschen Sie die Zellen dreimals mit DMEM/F12 Medium mit 10% FBS, jedesmal gefolgt von 5 min Inkubation bei 37 ° C. Nehmen Sie ein Deckglas (mit markierten Zellen) mit einer Pinzette und legen Sie sie in einem bildgebenden Kammer.
- Zweimal waschen mit 300 ul Bildpuffer. In 300 ml frisches Bildpuffer und gehen sofort auf Bildgebung (Teil 2).
2. Image Acquisition
HINWEIS: Verwenden Sie eine Totalreflexion (TIRF)-Mikroskop, mit einer Öl-Immersion mit hoher numerischer Apertur Ziel (zB 100X magnification/1.46 numerische Apertur.), Geeignete Laser (zB 405 nm, 488 nm, 561 nm und 645 ausgestattet nm Diodenlaser), ein Elektronenvervielfachungs Charge Coupled Device (EMCCD)-Kamera, ein Inkubator und eine Temperaturregelung, einzelne fluoreszierende Moleküle zu visualisieren.
- Stellen Sie die gewünschten Parameter Mikroskop, dh., Laserlinie, TIRF-Winkel (steuert dieser Parameter die Durchdringungtion Tiefe des evaneszenten Feld), Belichtungszeit, Bildrate und die Anzahl der Bilder pro Film 10. Halten Sie die Heizung / Inkubator-und Temperatursteuerung immer auf Temperaturdriften und Feuchtigkeitskondensation zu vermeiden.
- Geben Sie einen Tropfen Immersionsöl auf dem 100X Objektiv des Mikroskops. Legen Sie die Bildkammer mit den markierten Zellen auf den Probenhalter des Mikroskops, und bringen die Zellen im Fokus mit Hellfeldbeleuchtung.
- Wechseln Sie in den TIRF-Beleuchtung. Halten Laserleistung so gering wie möglich, damit die Suche nach der gewünschten Zelle, aber zur gleichen Zeit minimiert Photobleichen.
- Wählen Sie die gewünschte Zelle und Feineinstellung des Fokus. Stellen Sie die Laserleistung auf einem Niveau, das die Visualisierung einzelner Fluorophore ermöglicht. Erwerben Sie Bildfolge und speichern rohen Bildsequenz-Datei als. Tiff.
3. Kalibrierung (Single Fluorophore auf Glas und Monomer / Dimer-Rezeptor-Controls)
- Montieren Sie jede Kalibrierung sample wie in 1.2 in der Imaging-Kammer beschrieben. Platzieren Sie jede Probe auf dem Mikroskop und wählen Sie die Probe, die gut getrennten beugungsbegrenzten Flecken, die in einem einzigen Schritt zu bleichen HINWEIS:. Diese Spots einzelne Moleküle des Fluoreszenzfarbstoffs zu vertreten.
- Erwerben TIRF Bildsequenzen, wie in Schritt 2 beschrieben. Wichtig: die gleichen Abbildungsparameter muss für alle Experimente, wie die, die für die Kalibrierung verwendet werden.
- Führen Sie die Erkennung und Tracking-Analyse, wie in 4.1 beschrieben - 4.2. Entpacken Sie die Intensität der einzelnen Teilchen wie in 4.2.6 beschrieben. Aus diesen Daten berechnet den Mittelwert (μ) und Standardabweichung (σ) der Intensität der einzelnen Fluorophore.
- Optional führen die gleiche Analyse auf Zellen mit einem monomeren Zelloberflächenrezeptor (. Z. B. CD86) transfiziert, N-terminal mit ein oder zwei Kopien von SNAP 10 markiert und mit dem Fluorophor-BG-Derivat markiert. Follow der oben für die einzelnen Fluorophor auf Glas beschriebenen Verfahren. Schätzen Sie die Markierungseffizienz wie in Calebiro, D. et al. 10.
4. Bildanalyse
- Bildsequenz Vorbereitung
- Verwenden Sie eine Bildverarbeitungssoftware (zB., ImageJ), um die Bilder zu beschneiden.
- Speichern Sie einzelne Bilder als separate. TIFF-Bilder in einem neuen Ordner, was auf jedem Bild die Rahmennummer.
- Messung der Zelloberfläche, indem ein interessierender Bereich (ROI) entlang der Kontur der Zelle und mit dem Messwerkzeug in ImageJ oder ein ähnliches Werkzeug in eine andere Software. Diesen Wert verwenden, um die Partikeldichte, die durch Dividieren der Gesamtzahl der Teilchen am Anfang des Films von der Oberfläche der Zelle zu berechnen.
- Partikeldetektion und-verfolgung
HINWEIS: Verwenden Sie nicht-kommerzielle Software wie U-Bahn 13, die in MatlabUmwelt, automatisch zu erkennen und zu verfolgen einzelnen Rezeptor-Partikel.
HINWEIS: Die u-Track-Algorithmus basiert auf der Hypothese, Multiple-Tracking-Ansatz. Dieser Ansatz verbindet Teilchen zwischen Frames, indem Kosten Matrizen, wobei die einzelnen Wahrscheinlichkeiten, dass ein bestimmtes Teilchen in einem Rahmen entspricht einem bestimmten Teilchen im nächsten Rahmen angezeigt wird, verschwindet oder verschmilzt / Splits mit / von anderen Teilchen zugeordnet sind. Die Lösung, die die Kosten minimiert, global, dh., Die mit der höchsten Wahrscheinlichkeit wird schließlich ausgewählt. Dies ermöglicht auch die Verfolgung eines vorübergehend verschwinden Teilchen, ein typisches Phänomen von Fluorophor Blinken verursacht. Die neueste Version von u-Bahn (2.1.0) hat grafische Benutzerschnittstellen, die die Ausführung dieser Analysen zu erleichtern.- Von der Matlab-Eingabeaufforderung "movieSelectorGUI", um den Film Auswahl Schnittstelle zu öffnen. Folgen Sie den Anweisungen, um eine zu erstellenneue Filmdatenbank ausgehend von den zuvor gespeicherten separate Bilder (siehe 4.1.2).
- Stellen die Pixelgröße in nm, Zeitintervall in Sekunden, numerische Apertur, Kamera Bittiefe und Emissionswellenlänge des Fluorophors zur Partikeldetektion und-verfolgung erforderlich. Speichern Sie die Film-Datenbank.
- Aus dem Film-Auswahl-Schnittstelle, führen Sie die Analyse, die Wahl "single-Teilchen" als Typ der Objekte. Ein neues Fenster wird angezeigt, in dem die für die Partikelerkennung und-verfolgung verwendeten Parameter definiert werden können. Beginnen Sie mit den Standardparametern. Später, stellen Sie diese Parameter, wenn die Qualität der Erkennung und / oder Verfolgung nicht zufriedenstellend ist (zB sind einige Teilchen nicht erkannt oder Tracks sind fragmentiert) Optional:. Unter den Tracking-Einstellungen überprüfen "Export Tracking-Ergebnisse zu Matrix-Format" zu speichern die Koordinaten und die Amplituden aller Teilchen in einer Matrix (Feld "trackedFeaturedInfo"). Für eine detaillierte Beschreibung dieser Parameter, auf die U-Bahn-Dokumentation.
- Führen Sie den Erkennungsalgorithmus. Dieser Algorithmus den Ort und die Intensität über den Hintergrund der einzelnen beugungsbegrenzten Fleck (dh., Einzel-Rezeptoren / Rezeptor-Komplexe), indem eine zweidimensionale Gauß-Funktion mit einer Standardabweichung gleich der Punktbildfunktion des Mikroskops um lokale Intensitätsmaxima automatisch ermitteln . Führen Sie dann den Tracking-Algorithmus. Bewahren Sie die Ergebnisse der Analysen in einem. Matte Datei.
- Verwenden Sie die "movieViewer"-Routine, in der u-Track-Paket enthalten ist, oder ähnliche Gewohnheit eine, um die Titel zu visualisieren und überprüfen die Qualität der Erkennung und Verfolgung.
- Öffnen Sie die. Mat-Datei, um die Position und Amplitude (dh., Intensität) der verfolgten Teilchen bei jedem Rahmen sehen. In Schritt 4.2.4 erzeugten Daten werden in der tracksCoordAmpCG Feld der tracksFinal ar enthaltenray und / oder in trackedFeaturedInfo. Aus der Gesamtzahl der Teilchen detektiert errechnen Teilchendichte Dividieren dieses Werts durch die Zelloberfläche gemessen bei 4.1.3.
- Optional: Verwenden Sie die Partikelkoordinaten über die Zeit (siehe 4.2.6), um die Bewegung der Rezeptor-Partikel zu analysieren. Berechnen Sie die mittlere quadratische Verschiebungen (MSD) und Diffusionskoeffizienten (D) mit Matlab oder ähnliche Software. Zu jedem Teilchen und jedes Zeitintervall (Δ t) betrachtet, die Berechnung der mittleren quadratischen Verschiebung (MSD) mit folgender Formel:
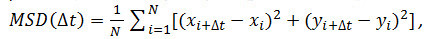
wobei Δ T das Zeitintervall in Rahmen ist, N die Anzahl von Schritten analysiert, x und y des Teilchens x-und y-Koordinaten an der durch den Index angegebenen Rahmen. Verwenden Sie den MSD im Laufe der Zeit Grundstücke, um die Art der Bewegung einer bestimmten Partikel bewerten: Lineare Beziehungenzeigen freie Diffusion (dh., Brownsche Bewegung), eine positive Krümmung (dh., sieht die Kurve wie eine Parabel) deutet gerichtete Bewegung, ist eine negative Krümmung bezeichnend für Bewegung beschränkt 14. Im Falle von frei diffundierenden Teilchen, die Berechnung der Diffusionskoeffizienten (D) von jedem Teilchen durch Anpassung der erhaltenen MSD Daten mit der folgenden Gleichung: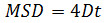
- Berechnung der Partikelgröße mit Gauß-Anpassungsverfahren
HINWEIS: Wenn die Intensitätsverteilung des Kalibrierungsproben (einzelner Fluorophore auf Glas und / oder fluoreszierend markierten monomeren Rezeptoren) bekannt ist, führen eine gemischte Gauß-Fit auf die Verteilung der Partikelintensitäten zu Beginn einer Bildsequenz, die Größe der Rezeptor-Komplexe zu bestimmen, (dh. die Anzahl der Rezeptoren pro Partikel) 10. Führen Sie diese Analysen mit Matlab oder similar Software.- Berechnen der Intensität von jedem Teilchen durch Mittelung der Intensität des Teilchens aus dem ersten Bild zu dem Rahmen vor der ersten Intensitätsänderung aufgetreten ist (in den meisten Fällen eine Abnahme durch Photobleaching).
- Durchführen einer Gaußschen Mischarmatur nach der folgenden Gleichung:
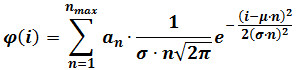
wobei φ (i) die Frequenz von Teilchen mit Intensität i, n die Komponentennummer, α n ein Parameter ist, die der Höhe der Komponente n trägt, μ und σ sind der Mittelwert und der Standardabweichung der Intensität des Referenz einzelner Fluorophore . (berechnet wie in Schritt 3 beschrieben) HINWEIS: Bestimmen Sie ter maximale Anzahl von Komponenten (n max) für jede Bildsequenz von progressiv zunehmenden n max bis die Zugabe einer Komponente ist nicht mehr produzieren eine statistisch bessere Passform, wie durch einen F-Test beurteilt. - Optional: (. ZB die letzten 60 Frames einer Bildfolge 400) Führen Sie eine gemischte Gauß-Sitz auf der Intensitätsverteilung auf den letzten Frames des Films erhalten. Μ und σ Ersetzen mit den nach dieser Armatur erhaltenen Werte, die raffinierte Schätzungen für diese Parameter liefern, und wiederholen Sie Schritt 4.3.2.
- Berechnen der Fläche unter der Kurve (AUC) für jede Komponente der gemischten Gauß Form. Berechnung der relativen Häufigkeit von Rezeptor Teilchen unterschiedlicher Größe (dh Monomer, Dimer, Trimer etc.) durch Dividieren der AUC-Wert von jeder Komponente von der AUC der gesamten Verteilung.
- Optional: Verwendung von Daten aus verschiedenen Zellen und die entsprechenden Partikeldichten (berechnet wie in 4.2.6 beschrieben) auf Flächen, auf denen die Verteilung der Teilchen mit unterschiedlicher Größe mit Partikeldichte 10 korreliert zu erzeugen.
- Berechnung der Partikelgröße mit Schritt Anpassungsverfahren
HINWEIS: Verwenden Sie eine Schritt-für Montage-Analyse als eine alternative Methode, um die Größe der Rezeptor-Komplexe 10 zu bestimmen. Die Grundlage für diese Analyse ist, dass die lichtinduzierte Zerstörung (Photobleaching) eines einzelnen Fluorophors Ergebnisse in seiner augenblicklichen Verschwinden - also Teilchen mit n Fluorophore werden voraussichtlich schrittweise Bleich Herstellung eines Intensitätsprofil mit n Schritten.- Auszug Intensitätsprofile der einzelnen Teilchen aus dem von U-Bahn oder ähnliche Erkennung /-Tracking-Software (siehe 4.2.6) erzeugt. Matte Datei.
- Verwenden einen Schritt Anpassungsalgorithmus, wie der vorgein ref. 10, um die Anzahl der Bleichschritte für jedes Teilchen zählen.
- Optional: Verwenden Sie die Ergebnisse auf Ausschüttungen, die die relative Häufigkeit von Rezeptor-Partikeln unterschiedlicher Größe erzeugen und korrelieren sie mit Partikeldichte, wie zuvor für die Ergebnisse der gemischten Gaußsche Anpassung beschrieben (siehe 4.3).
Ergebnisse
Das beschriebene Protokoll kann auf eine Vielzahl unterschiedlicher Membranproteine verwendet werden. Als Beispiel sind mit β 2-adrenergen Rezeptoren und GABA B erhaltenen Ergebnisse repräsentativ 10 gemeldet. Seit fluoreszierenden Signale von einzelnen Molekülen schwach sind, ist die Minimierung der Hintergrundfluoreszenz der erste entscheidende Schritt zur erfolgreichen Ergebnissen. Daher ist es wichtig, intensiv gereinigt Deckgläser (2A) verwenden sowie Prob...
Diskussion
Die beschriebenen Protokoll ermöglicht die Analyse der räumlichen Anordnung, Mobilität und Größe der Zelloberflächenrezeptor-Komplexen auf Einzelmolekülebene. Verglichen mit der Verwendung von fluoreszierenden Proteinen, Markierung mit kleinen organischen Fluorophoren, die heller und photostabil sind, hat den Vorteil, daß die erweiterte Darstellung der einzelnen Rezeptor Teilchen. Da sehr niedrige Expressionsniveaus erzielt werden (<0,45 Rezeptor Teilchen / &mgr; m 2), die Eigenschaften der Rez...
Offenlegungen
The authors declare that they have no competing financial interests.
Danksagungen
The development of this protocol was supported by grants from the European Research Council (Advanced Grant TOPAS to M.J.L.) and the Deutsche Forschungsgemeinschaft (Grants CA 1014/1-1 to D.C. and SFB487 to M.J.L.). T.S. was supported by the Alexander von Humboldt Foundation.
Materialien
| Name | Company | Catalog Number | Comments |
| Chloroform | AppliChem GmbH | A1585 | CAUTION: toxic and irritating substance as well as a possible carcinogen |
| NaOH | Sigma-Aldrich | S8045 | CAUTION: strong base and highly corrosive reagent |
| Absolute ethanol | Sigma-Aldrich | 32205 | |
| Glass coverslip | Marienfeld-Superior | 111640 | 24 mm diameter, 0.13-0.16 mm thickness |
| 0.2 mm sterile filter | Sarstedt | 83.1826.001 | |
| CHO cells | ATCC, USA | ATCC CCL-61 | Chinese hamster ovary cell line |
| 6-well cell culture plate | Nunc | 140675 | |
| DMEM/F-12 medium | GIBCO, Life Technologies | 11039-021 | Phenol-red free medium |
| Fetal bovine serum | Biochrom | S 0115 | |
| Penicillin - streptomycin | Pan Biotech GmbH | P06-07 100 | |
| Trypsin-EDTA | Pan Biotech GmbH | P10-23100 | |
| Lipofectamine 2000 | Invitrogen, Life Technologies | 11668-019 | |
| Opti-MEM I Reduced Serum Medium | Invitrogen, Life Technologies | 31985-047 | |
| Fluorophore-conjugated benzylguanine | New England BioLabs | S9136S | SNAP-Surface Alexa Fluor 647. Make a 1 mM stock solution in DMSO. Store at -20°C. |
| DMSO | AppliChem GmbH | A1584 | |
| Imaging buffer: | 137 mM NaCl, 5.4 mM KCl, 2 mM CaCl2, 1 mM MgCl2, 10 mM HEPES, pH 7.3, sterile-filtered | ||
| NaCl | AppliChem GmbH | A1371 | |
| KCl | AppliChem GmbH | A3582 | |
| CaCl2 | AppliChem GmbH | A2303 | |
| MgCl2 | AppliChem GmbH | A3618 | |
| HEPES | AppliChem GmbH | A3724 | |
| Imaging chamber | Molecular Probes, Life Technologies | A-7816 | Attofluor Cell Chamber, for microscopy |
| TIRF-M | Leica | Model: DMI6000B | |
| TIRF objective | Leica | 11 506 249 | HCX PL Apo 100x/1.46 Oil CORR |
| EM-CCD camera | Roper Scientific | Photometrics Cascade 512B | |
| Temperature controller | Pecon | Tempcontrol 37-2 digital | |
| ImageJ software | NIH, USA | http://rsbweb.nih.gov/ij | |
| u-track software | Laboratory for computational cell biology, Dept. of Cell Biology, Harvard Medical School, USA | http://lccb.hms.harvard.edu/software.html | |
| Matlab software | The MathWorks, USA |
Referenzen
- Pierce, K. L., Premont, R. T., Lefkowitz, R. J. Seven-transmembrane receptors. Nat Rev Mol Cell Biol. 3, 639-650 (2002).
- Angers, S., Salahpour, A., Bouvier, M. Dimerization: an emerging concept for G protein-coupled receptor ontogeny and function. Annu Rev Pharmacol Toxicol. 42, 409-435 (2002).
- Ferré, S., et al. Building a new conceptual framework for receptor heteromers. Nat Chem Biol. 5, 131-134 (2009).
- Milligan, G. G. protein-coupled receptor hetero-dimerization: contribution to pharmacology and function. Br J Pharmacol. 158, 5-14 (2009).
- Lohse, M. J. Dimerization in GPCR mobility and signaling. Curr Opin Pharmacol. 10, 53-58 (2010).
- Ulbrich, M. H., Isacoff, E. Y. Subunit counting in membrane-bound proteins. Nat Methods. 4, 319-321 (2007).
- Triller, A., Choquet, D. New concepts in synaptic biology derived from single-molecule imaging. Neuron. 59, 359-374 (2008).
- Hern, J. A., et al. Formation and dissociation of M1 muscarinic receptor dimers seen by total internal reflection fluorescence imaging of single molecules. Proc Natl Acad Sci U S A. 107, 2693-2698 (2010).
- Kasai, R. S., et al. Full characterization of GPCR monomer-dimer dynamic equilibrium by single molecule imaging. J Cell Biol. 192, 463-480 (2011).
- Calebiro, D., et al. Single-molecule analysis of fluorescently labeled G-protein-coupled receptors reveals complexes with distinct dynamics and organization. Proc Natl Acad Sci U S A. 110, 743-748 (2013).
- Keppler, A., et al. A general method for the covalent labeling of fusion proteins with small molecules in vivo. Nat Biotechnol. 21, 86-89 (2003).
- Gautier, A., et al. An engineered protein tag for multiprotein labeling in living cells. Chem Biol. 15, 128-136 (2008).
- Jaqaman, K., et al. Robust single-particle tracking in live-cell time-lapse sequences. Nat Methods. 5, 695-702 (2008).
- Saxton, M. J., Jacobson, K. Single-particle tracking: applications to membrane dynamics. Annu Rev Biophys Biomol Struct. 26, 373-399 (1997).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten