Method Article
比率钙显像对单个神经元的行为线虫线虫
摘要
该协议描述了使用基因编码的 Ca2记者记录在行为上的神经活动的变化, 在线虫线虫蠕虫。
摘要
越来越明显的是, 行为动物的神经回路活动与麻醉或固定化动物中所看到的不同。高度敏感, 基因编码的 Ca2 +的荧光记者已经革命性的记录细胞和突触活动使用非侵入性光学方法的行为动物。结合遗传和 optogenetic 技术, 可以确定在不同行为状态下调节细胞和电路活动的分子机制。
在这里, 我们描述的方法为比率 Ca2 +成像的单神经元在自由行为的线虫线虫蠕虫。我们展示了一个简单的安装技术, 轻轻地覆盖生长在标准线虫生长培养基 (NGM) 琼脂块与玻璃片, 允许动物被记录在高分辨率在无限制的运动和行为。利用这种技术, 我们使用敏感的 ca2 + GCaMP5 记录在素雌雄同体特定神经元 (HSNs) 中的胞内 ca2 +中的变化, 因为它们驱动产卵行为。通过共同表达的 mCherry, 一个 Ca2 +不敏感的荧光蛋白, 我们可以跟踪在〜1µm 内的 HSN 的位置, 并纠正因焦点或运动的变化引起的荧光波动。同时, 红外明成像允许行为记录和动物跟踪使用一个机动阶段。通过集成这些显微技术和数据流, 我们可以记录C. 线虫产卵电路中的 Ca2 +活动, 因为它在数十分钟内的非活动和活动行为状态之间进行。
引言
神经科学的中心目标是了解神经元如何在电路中进行交流以推动动物行为。神经回路集成了一系列不同的感官提示, 以改变电路的活动, 从而推动行为变化的动物, 以响应他们的环境。线虫, 线虫,有一个简单的神经系统与302神经元的突触连接已经完全映射1。此外, 基因编码的蛋白质涉及神经是高度保守之间的C. 线虫和哺乳动物2。尽管其神经系统的解剖简单, 它显示了一个复杂的保存行为的剧目提供一个肥沃的平台, 以了解如何神经元调节行为3。
线虫适用于多种方法, 如遗传操作、激光细胞消融、电生理技术以及活体光学成像4、5。最近的研究已经产生了在线虫中主要的神经递质信号系统的详细图谱, 包括胆碱能和 gaba 神经元网络。这些研究, 连同正在进行的研究来映射所有神经元 G 蛋白耦合受体的表达, 把这个模型放在一个独特的位置, 利用高度详细的结构和功能性的神经元连通图来充分理解这些不同的神经递质信号通过不同的感受器官和时间表驾驶不同的方面动物行为。
为了研究任何系统中的动态神经元活动模式, 一个必要的先决条件是开发健壮的方法来记录单个神经元或整个回路在行为过程中的活动。特别重要的是这种光学方法的可, 以可视化前活动驱动突触囊泡融合。快速和高度敏感的细胞内钙2 +的荧光记者, 随着敏感探测器的增加, 已经彻底改变了在清醒时, 活着的动物在行为过程中细胞和突触活动的记录。由于突触电活动的主要结果是调节 ca2 +通道, 因此认为细胞内 ca2 +的变化会忠实地报告行为的相关变化。
在本研究中, 我们提出了一种方法来执行比率 Ca2 +成像在素 HSN 运动神经元, 促进产卵行为在C. 线虫6,7。此方法基于以前的努力, 在C. 线虫中可视化 Ca2 +活动, 并在行为过程中的产卵电路5,8,9,10,11.该方法可以同时关联观察到的细胞/电路活动的变化与产卵事件, 以及动物运动状态的变化。虽然我们使用这种方法来研究成年蠕虫的活动, 但我们的实验室未发表的工作表明, 这种方法也可以推广到第四幼体 (L4) 阶段的幼兽。在不同的电路和行为中起作用的其他C. 线虫神经元的活动很可能与此技术相似。其他最近开发的快速 Ca2 +具有非重叠发射光谱的指示器12,13,14,15,16, optogenetic 工具17, 和基因编码的薄膜电压的光学指标18, 应该允许我们执行穿透性的 ' 全光 ' 研究如何改变神经回路活动驱动不同的行为状态。
研究方案
1. 菌种、培养基和动物的安装
- 在20° c 的标准60毫米线虫生长培养基 (NGM) 上种植线虫蠕虫, OP50 大肠杆菌细菌性食品19。
- 为每个细胞特异的促进剂准备两个质粒: 一、驱动表达 GCaMP5 以记录胞内 Ca2 +, 第二, mCherry 的驱动表达, 以允许比率定量的 GCaMP5 荧光变化和简化对象查找和测量。
注: GCaMP5:mCherry 比率成像纠正 GCaMP5 荧光的波动, 这是由于焦点和动物运动的变化引起的, 而不是细胞内 Ca2 +的实际变化。 - 在 LX1832建兴-1 (ce314), lin-15 (n765ts) X 突变动物的性腺中注入 GCaMP5 和 mCherry 表达质粒, 并恢复非 Muv 的林-15 (+)动物表达 GCaMP5 和mCherry 从高拷贝转基因20,21,22。使用lite-1变种人背景来减少蓝光避光行为23,24。
- 将转基因与染色体结合以减少嵌, 简化复合突变株的生成25。
注: 这里描述的 LX2004 菌株携带一个综合的高拷贝转基因, 表达 GCaMP5 和 mCherry 在 HSNs 从nlp-3启动子 (请参见材料表)26,27,28,29. 发现nlp-3启动子在晚期 L4 和成年动物的 HSNs 中具有强烈的表达力, 而在 HSN 发育或产卵行为方面与其他试验者相比, 没有造成明显的缺陷 (如, 1的,egl-6和unc-86)。这种应变和其他表达 GCaMP5 和 mCherry 在腹线 (VC) 运动神经元, 外阴肌肉, 和 uv1 神经内分泌细胞可从线虫遗传学中心, 他们的建设细节已被描述27。 - 应用 OP50 细菌食品从一个种子 NGM 琼脂板到底部的白金蠕虫采摘, 并使用它转移〜20晚 L4 LX2004 动物, 表达 GCaMP5 和 mCherry 在 HSNs 从nlp-3启动子。确保发展中的外阴出现在一个显微镜, 是一个白色新月包围的明显的黑点。孵育动物在20° c 为 24-40 h。
- 在孵化后, 应用 OP50 到采摘和转移〜3的蠕虫到一个种子选手 NGM 板, 留下少量的食物背后的蠕虫饲料后, 在成像。确保足够的食物, 因为太少的细菌食物会促使蠕虫从盘子的中心游走, 而过多的食物会增加背景荧光并导致缺氧。
- 使用扁平圆铲削减〜20毫米 x 20 毫米块从板块携带的蠕虫和转移块面-向下到中心的清洁 24 mm x 60 mm #1. 5 片 (图 1)。从一侧开始应用, 防止气泡被困在片, 干扰成像。应用22毫米 x 22 mm #1 片到该区块的顶部, 以减少黏附和蒸发。
2. 硬件和仪表安装
- 将上演的, 转基因蠕虫放到一个倒置显微镜的舞台上, 有一个≥0.7 的数字孔径计划-色差20x 目标, 电动 XY 舞台可控与操纵杆, 图像分配器, 荧光过滤器同时 GCaMP5 和mCherry 激发和发射, 荧光和红外明成像相机, 和 triggerable 发光二极管 (LED) 照明系统 (图 2a)。
注: 80/20 分束器将80% 的图像信号发送到荧光相机和20% 的明相机。也可以使用直立显微镜, 但在这种情况下, NGM 块应该放在玻璃滑梯和大片之间。- 使用双目目镜为图像选择蠕虫。当动物被选中时, 将红外线滤镜滑入冷凝器上方。
- 晶体管-晶体管逻辑 (TTL) 触发器
- 在荧光相机上, 从三 TTL 触发输出线中的每一个都附上同轴电缆。将第一个输出连接到 LED 照明系统的 BNC 输入 #3。
- 将第二个输出连接到一个 BNC ' 香蕉 ' 适配器与绿色和棕色电线从8针 GPIO 连接器运行到 GPIO 输入 #3 (pin 4) 和地面 (pin 5) 的红外线相机, 分别。
- 将第三个输出连接到 BNC "香蕉" 适配器, 并在数字采集设备 (图 2B) 中 #8 数字输入和接地线上运行跳线。
- 数字采集设备 (数据采集)
- 通过 USB 电缆将数据采集单片机板 (参见材料表) 连接到 PC。使用标准的 Firmata 协议更新固件 (请参阅材料表), 并配置 USB 端口以57600波特通信。
- 指示灯
- 运行 LED 控制器软件 (请参见材料表)。将触发模式从 "连续" 切换到 "封闭", 并为470和 590 nm led 选择触发通道 "3"。
- 打开并输入 led 电源为每个 led (例如,设置 470 nm 导致20% 和 590 nm 导致 40%)。
- 在盆景中运行串行级通信脚本 "XY 阶段决赛" (请参见材料表)30。单击灰色的 "CsvWriter" 节点, 然后选择一个文件夹以保存记录的 X 和 Y 阶段信息 (图 3)。按工具栏中的绿色箭头来初始化数据采集。
注意: 当荧光相机发送 TTL 信号时, 脚本将开始记录 X 和 Y 阶段的位置。此脚本输出四列数据: 帧数、X 位置 (µm)、Y 位置 (µm) 和帧之间的间隔 (s)。 - 在红外摄像机录制软件 (参见材料表) 下的 "自定义视频模式" 下, 选择 "模式 1" (2 x 2 分; 1024 x 1024 像素) 和 "像素格式原始 8"。在 "触发/频闪" 下, 将触发输入线 (GPIO) 设置为 "3", 极性为 "高", "模式" 为 "14"。切换 "启用" 以停止帧获取, 直到接收到 TTL 触发信号。
- 打开窗口, 单击主照相机视图工具栏上的红色 "记录" 按钮。选择要保存的图像序列的文件夹。选择 "缓冲" 录制模式, 并以 JPEG 格式保存 "图像"。单击 "开始录制" 以初始化获取。
- 荧光相机和图像分配器
注: GCaMP5 和 mCherry 荧光通道必须同时收集, 以确保正确的图像配准比率定量。图像拆分器允许双通道采集 GCaMP5 和 mCherry 荧光到一个传感器上。- 在图像采集软件的 "捕获" 标签中 (请参阅材料表), 将曝光时间设置为10毫秒, 分到 4x, 图像深度为16位。选择一个中心相机子512像素宽256像素高。单击 "显示输出触发器选项" 并将所有触发器设置为 "正"。
注意: 如果10毫秒或更少, 数据采集可以偶尔漏掉 TTL 触发器。 - 确保 "触发器 1" 和 "触发器 2" 被设置为 "曝光", 而 "触发器 3" 设置为 "可编程", 且 "句点" 为25毫秒. 在 "序列" 选项卡下, 选择 "时间推移" 与 "字段 Delay1" 的50毫秒, 以收集图像20赫兹. 选择 "保存到临时缓冲区"。
- 在图像采集软件的 "捕获" 标签中 (请参阅材料表), 将曝光时间设置为10毫秒, 分到 4x, 图像深度为16位。选择一个中心相机子512像素宽256像素高。单击 "显示输出触发器选项" 并将所有触发器设置为 "正"。
- 三通道共焦成像中的 PMT 增益和激光强度
- 设置 PMT 增益, 使背景荧光的水平略高于最小 (黑色水平)。增加 561 nm (绿色) 激光功率, 直到一个饱和的12位或16位像素被观察到在 mCherry 通道的前总站。
- 调整 488 nm (蓝色) 激光强度, 使 GCaMP5 荧光只是可见的前总站以上背景。此低设置可防止在响应强 Ca2 +瞬变时, 荧光增加时 GCaMP5 像素的饱和度。打开共焦针孔所有的方式, 以最大限度地捕捉光线。
3. 比率 Ca2 +成像和行为记录
- 在荧光采集软件的 "序列" 标签下, 单击 "开始" 开始录制。跟踪与游戏杆的蠕虫, 保持细胞 (s) 和在焦点的兴趣突触和在视野的中心 (视)。单击 "直方图" 窗口中的 "统计" 按钮, 以显示每个通道的像素统计信息。
- 调整 LED 电源, 以确保在≥8000计数 (〜4000光电子) 的前总站上有一个最大的单像素 mCherry 荧光, 在背景 (〜100光电子) 上提供12位动态范围。GCaMP5 信号在前总站期间休息 (低) Ca2 +应该是大约〜2500计数-只是在上面可见的背景。
- 记录, 直到一个产卵活动状态达到;通常, 这种情况在野生类型的蠕虫7中每隔20-30 分钟发生一次。保存一个10分钟的子集 (12001 帧), 6000 帧前后的第一个产卵事件 (框架 6001)。一定要保持相同的明图像子集的蠕虫行为和 timepoints 的 XY 阶段位置, 或数据流的精确同步将丢失。
- 使用 ImageJ 和 BioFormats 插件 (请参见材料表) 将图像序列转换为图像细胞仪标准 (. ics) 格式, 以便可以将其导入到比率定量软件中。
4. 图像分割与定量分析
- 将图像序列导入比率定量软件 (请参见材料表)。单击 "工具" 菜单中的 "自动对比度", 并调整每个通道的对比度以建立适当的黑色 (〜 1800) 和白色级别 (1万)。在 "工具" 菜单中选择 "更改颜色..." 以确认 mCherry 和 GCaMP5 通道颜色分配是否正确 (图 4-c)。
- 右键单击 "序列" 标签中的时间序列, 并将其设置为20帧/秒和1.25 µm/像素。
- 从 "工具" 菜单中选择 "比率", 然后为 "通道 a" 和 "mCherry" 选择 "GCaMP5"。单击 "阈值" 旁边的 "计算" 以减去每个图像序列的背景。选择 "应用彩虹查找的比率渠道"。
注意: 对于具有平均基线比率为 0.3 (低 ca2 +) 和最大比率为 2-3 (高 ca2 +) 的记录, 合适的查找表将从 0 (蓝色) 到 1 (红色)。 - 使用 mCherry 通道 (图 4D) 生成强度调制比率通道。强度调制比通道是一幅数以百万计的彩色图像, 其中比例颜色被映射到 mCherry 通道的亮度上。
- 在 "测量" 选项卡下, 通过将 "查找对象使用强度" 工具拖到 "协议" 窗格中, 创建一个对象查找协议。单击 "齿轮" 以设置 mCherry 强度值的窗口并查找对象。
- 选择强度值≥2标准偏差 (SD) 以上的背景 (例如,下限为 2500, 上限为 65535)。确保检测到前总站, 并从 "测量" 菜单中选择 "自动更新反馈"。
注意: 该软件还可以使用相关协议 "SD 强度" 来识别对象的标准偏离背景。然而, 如果一个特别明亮的物体进入视, 它可以戏剧性地改变平均强度在这些 timepoints, 影响强度保险丝用于寻找 HSN。如果使用原始强度值来查找对象, 则不会发生这种可变性。
- 选择强度值≥2标准偏差 (SD) 以上的背景 (例如,下限为 2500, 上限为 65535)。确保检测到前总站, 并从 "测量" 菜单中选择 "自动更新反馈"。
- 如果需要, 添加其他筛选器只针对 mCherry 通道 (大小、最大强度、等), 以排除头部、尾部和肠道荧光等不希望的对象。从 "测量" 菜单中选择 "制作测量项目", 然后选择 "所有 Timepoints"。
注意: 这将执行协议, 将所有度量写入一个逗号分隔的值 (. csv) 文件, 并使用 "文件" 菜单中的 "导出" 命令导出。 - 打开导出的. csv 文件并复制 Timepoint、区域 (µm2)、平均 (比率通道)、质心 X (µm) 和质心 Y (µm) 数据到新的工作表中。导出不带列标题的. csv 文件。比率定量软件可以将感兴趣的单元格分类为每个 timepoint 的一个或多个对象。
- 要重新组合这些对象, 请使用自定义脚本 "AnalzyeGCaMP_2017. m"。
注意: 该脚本还标识了数据中的 Ca2 + (比率) 峰值, 并保存了它们的 timepoints、峰值振幅和峰值宽度的. csv 文件。它还为原始和标注的比率跟踪生成 postscript 文件。使用此信息, 应确定 Ca2 +的瞬时振幅和瞬时间隔。
- 要重新组合这些对象, 请使用自定义脚本 "AnalzyeGCaMP_2017. m"。
- 将每个荧光物体的输出 x 和 y 质心值添加到由盆景的 XY 阶段脚本记录的 x 和 y 值。使用净 XY 位置生成蠕虫移动跟踪, 并计算伴随不同行为状态的单元格位移和速度31。
- 将记录的明图像导入 ImageJ 作为虚拟堆栈。注释产卵事件和其他行为。将这些事件的时间与比率跟踪中的 Ca2 +峰值进行比较。
结果
这里描述的简单安装技术 (图 1) 会对 L4 和成年的C. 线虫的文化环境造成最小的更改, 同时允许在行为动物中通过玻璃片 (图 4) 进行高分辨率记录。LED 光源、舞台控制器和相机曝光的同步允许在20帧/秒的多个流中获取数据, 最多可达到 1 h. 中间放大倍数 (20x), 高数值孔径目标 (0.7-0.8) 提供了良好在产卵行为回路中的突触区域的空间分辨率, 即使有 4 x 4 像素分 (1.25 µm/像素)。同时获取 GCaMP5 和 mCherry 荧光信号 (图 4A, B) 用于生成逐像素的比率通道, 以补偿动物移动和焦点的变化 (图 4D)。在C. 线虫中, HSN 前总站与许多神经元细胞体一样大, 而前 HSN2 +中的变化可以清楚地可视化。红外明相机的大视允许在录制过程中手动跟踪蠕虫 (图 4E)。对于每种动物, 细胞内钙2 +的变化可以与明成像中明显的行为相关, 包括卵子释放和运动改变 (图 4E)。
定量的 HSN Ca2 +和速度证实, 蠕虫改变他们的运动, 因为他们过渡到产卵行为。在产卵活动状态 (图 5A) 之前、期间和之后, 蠕虫速度有显著差异。这不是由成像或跟踪系统所固有的噪音造成的。放大成一个产卵的事件, 我们观察 GCaMP5 荧光 (δ/F) 在产卵事件之前的强烈变化, 而 mCherry 荧光是相对不变的 (图 5B)。GCaMP5:mCherry 比率 (ΔR/R) 的测量变化清楚地显示了在卵子释放之前 (图 5B) 之前的 HSN Ca2 +瞬态〜4秒。与外阴肌肉收缩一致, 明显减慢蠕虫的运动发生, 以卵子释放结束。先前的结果表明, 胆碱能 VC 运动神经元, 这是支配的 HSNs, 显示峰值活动期间强外阴肌肉收缩和在卵子释放8,10,27。我们还表明, optogenetic 激活的 vc 神经元驱动立即减慢运动, 表明 vc 神经元可能被激活的外阴肌肉收缩, 从而减慢运动, 直到收到反馈的卵子释放27.
描述的成像和跟踪系统允许可视化产卵行为的空间组织 (图 5C)。如前所示, 蠕虫在活动状态32之前进入持续的向前运动运行。蠕虫将大部分时间花在琼脂块中心的细菌上觅食。在进入活动状态之前, 蠕虫会从食物中移开, 这与不频繁 HSN 的 Ca2 +瞬态的外观是重合的。HSN 活动然后过渡到爆发射击, 与几个紧密间隔的 HSN Ca2 +瞬态, 维持产卵事件。然后蠕虫经常转过身来, 继续向前移动, 然后在 OP50 细菌附近的起始位置产卵。我们假定本地 O2和/或 CO2浓度的变化可能会影响蠕虫决定产卵的位置33,34。
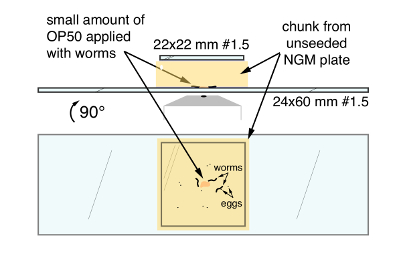
图 1。C. 线虫用于高分辨率成像的产卵电路活动和行为的安装技术.顶部, 从侧面最后的坐骑。底部, 最后的坐骑, 从底部的大片。箭头表明 OP50 细菌食物,线虫蠕虫和鸡蛋, 夹在 NGM 琼脂块和大24毫米 x 60 毫米片。请单击此处查看此图的较大版本.
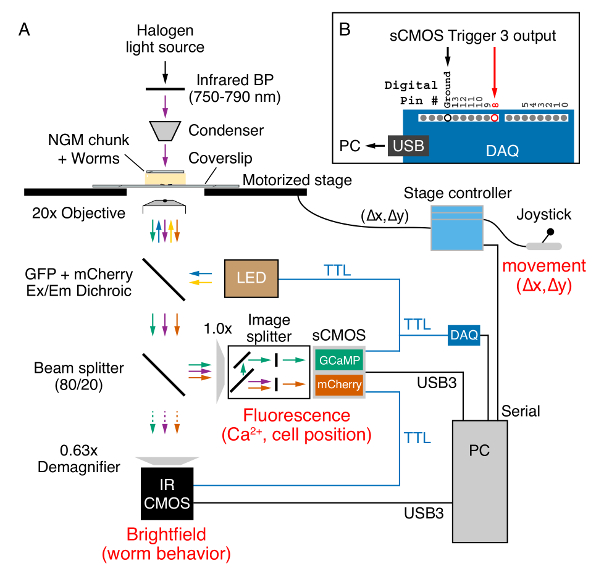
图 2.Widefield 比率 Ca2 +成像和行为记录在一个倒置的荧光显微镜.(A) 蠕虫的位置和行为在明通过 20x (0.8 NA) 计划-色差目标, 使用红外线 (750-790 nm) 光 (紫色箭头) 从卤素灯发出, 通过 NGM 块。在记录过程中, 使用操纵杆和机动舞台控制器来维护视图中的蠕虫。舞台位置 (x、Δy) 由串行端口发送到 PC。GCaMP5 和 mCherry 蛋白表达的蠕虫是兴奋使用 470 nm (蓝色箭头) 和 590 nm (黄色箭头) 发光二极管 (LED)。发射的 GCaMP5 (绿色箭头) 和 mCherry (橙色箭头) 荧光连同红外线通过多波段分色镜 (参见材料表)。80/20 分束器通过 0.63x demagnifer 发送20% 的光, 用于在红外敏感 CMOS 相机 (紫色箭头) 上捕获。剩余的80% 的光通过显微镜的侧面端口发送到一个图像分配器, 将 GCaMP5 和 mCherry 荧光分离到 sCMOS 相机的分开的一半, 同时去除红外线明光。两个摄像头的数据通过 USB3 电缆传输到 PC。来自荧光 sCMOS 相机 (蓝色) 的触发端口用于向 LED 照明系统、红外明 CMOS 摄像头和数字采集设备 (数据采集器) 发送 + 5 v TTL 触发器。(B) 触发器3输出 TTL 信号由数字 pin 的数据采集 #8 并通过 USB 连接发送到 PC。这些数字输入触发了一个 ' xy ' 串行命令从盆景软件脚本 (xy 阶段决赛), 它读取 X 和 Y 阶段的位置为每个 GCaMP5/mCherry 的荧光/红外图像捕获。请单击此处查看此图的较大版本.
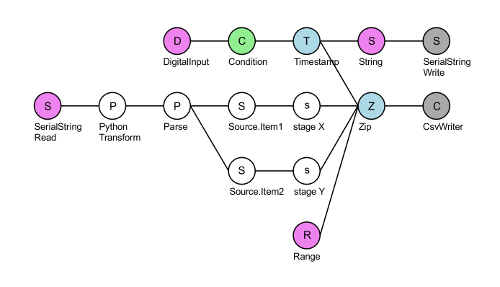
图 3: 盆景系列舞台排版的布局 XY 阶段-决赛.顶部的 DigitalInput 节点 (粉红色) 读取的 TTL 触发器进入 pin #8 的数据采集。对于每个正 TTL 电压 (绿色), 数据采集器会使时间戳 (蓝色), 并通过串行端口 (灰色) 将 "XY?" 字符串 (粉红色) 写入舞台控制器。SerialStringRead 节点 (粉红色) 从舞台控制器读取 X 和 Y 坐标响应。然后将此字符串转换为微米, 并将其分成 X 和 Y 阶段坐标。最后, 这些四流是使用 zip 节点 (蓝色) 和四列. csv 文件编写的: 接收的 TTL 信号的帧计数 (范围节点、粉红色)、X 和 Y 坐标以及后续 timepoints 之间的间隔 (通常为50毫秒记录在20赫兹)。请单击此处查看此图的较大版本.
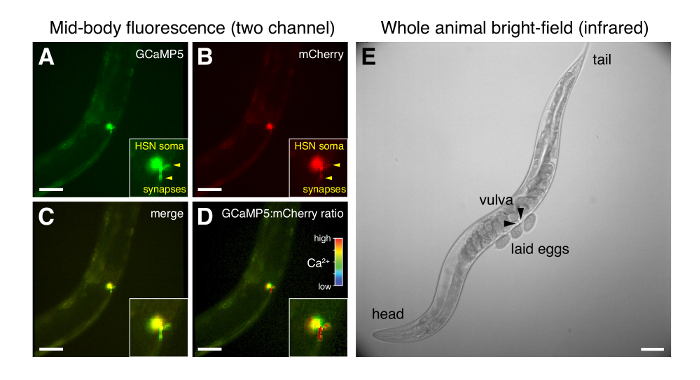
图 4: HSN 荧光和全动物明在产卵行为中的代表性显微.(A-D)同时比率成像的 GCaMP5 和 mCherry 荧光在 HSNs 只是在卵子释放之前。HSN 前总站用箭头表示。(C) GCaMP5 和 mCherry 荧光的合并。(D) 强度调制的 GCaMP5:mCherry 比;高比率 (红色) 表示高胞内 Ca2 +在前总站。(E) 明整个蠕虫病毒的图像后, 鸡蛋已经奠定。箭头指的是的前部和后半部分。所有图像的缩放栏是50µm.请单击此处查看此图的较大版本.

图 5: 记录 HSN Ca2 +和在产卵行为期间的移动速度.(A) 在响应胞内 Ca2 +瞬变 (ΔR/R; red) 以及瞬时蠕虫速度 (µm/秒; 蓝色) 时, GCaMP5:mCherry 比率的变化。产卵事件由箭头表示。(B) HSN GCaMP5 荧光 (绿色; δ/f)、mCherry 荧光 (红色; δ/f)、GCaMP5:mCherry 荧光比 (黑色; ΔR/R) 和虫卵释放瞬间的蠕虫速度 (蓝色) 的痕迹。(C) 在整个10分钟录制过程中产卵和运动行为的空间组织。从 mCherry 荧光记录得到的 HSN 的质心位置, 得到了从 XY 阶段信息中得到的蜗杆轨迹。计时 HSN 瞬变 (红色圈子), 产卵事件 (黑箭头) 和录音的开始和结尾 (绿色和蓝色金刚石) 被表明。缩放条是 1 mm.请单击此处查看此图的较大版本.
讨论
转基因:
由于 mCherry 表达已经通过密码子优化和内含子插入 (大多数 GCaMP 记者没有), 注入〜4x 的数量 GCaMP5-expressing 质粒, 以确保可比水平的 GCaMP5 荧光在强钙2 +瞬态。拯救的林-15 (n765ts)突变, 使转基因动物的简单恢复, 对产卵和其他行为的影响最小,35。转基因应整合, 以确保不同动物之间的统一表达和成像条件25。可选择的标记包括抗菌素耐药性36,37和对温度敏感的 lethals38的抢救也应该工作, 但是, 由于非转基因动物已经死亡, 转基因融合的确认和与呈现可见表型25的标记相比, 纯更难。应避免39, 从而破坏正常移动和降低适应性的显性标记, 如6 (dm)。在lite-1突变体背景中进行图像处理对于减少图像中的蓝色光逸出响应是必不可少的23、40、41。gur-3的其他变体 (与lite-1并行操作) 可能会进一步减少由蓝光42导致的残余行为和生理变化。方便地, gur-3、 lite-1和lin-15都位于染色体 X 的右半部分, 这将简化 Ca2 +成像和 optogenetic 实验的新菌株的构建。
由于曝光时间短, 图像收集 20 Hz, GCaMP5 和 mCherry 信号必须明亮。对于嘈杂的 Ca2 +录音, 最可能的解释是由弱报告器表达式引起的暗淡荧光。推广者也应该是合理的具体区域正在拍摄。由于 HSN 细胞体和突触到外阴肌肉是在身体的中心, 额外的表达从nlp-3启动器的头部和尾部通常是在视野之外, 可以排除使用其他筛选比率定量软件。为了确保在细胞内 Ca2 +的实际变化中观察到的 GCaMP5 荧光的变化, 应独立准备并分析26, 以表达 GFP 而非 GCaMP5 的控制转基因。具有不同 ca2 +灵敏度、动力学和颜色的较新的 ca2记者扩展了可用的图像工具的选择, 但每个新的报告员都应该使用 ca2 +不敏感的荧光变体14 进行验证 ,16。
媒体:
NGM 板的成像应准备高品质的琼脂。低质量的琼脂不会完全溶解在灭菌, 留下小颗粒, 散射光在成像, 并增加背景荧光。分子生物学级琼脂糖可以代替, 但增加的数量应减少, 以保持等效板块的硬度。
与其他安装方法的比较:
以前的方法, 图像活动在产卵行为电路已使用的蠕虫固定与胶水的琼脂糖垫。在这些条件下, 没有减少培养基的活动和产卵行为, 渗透8,10,44,45。最近的证据表明, 一些蠕虫的行为被调整的运动状态, 提出的问题, 细胞活动观察到固定的动物, 看到在自由行为的动物。我们最近已经表明, 产卵电路活动是意外地分期与运动26,27。同样, RIA interneuron 中的隔离 Ca2 +信号在觅食运动中集成了感觉神经元和头部运动神经元的活动,46。排便行为也伴随着运动的精确和定型的变化, 允许动物离开他们的觅食点, 然后驱逐废物47。这些结果表明, 固定化动物的电路活动的简要记录可能与在自由行为动物中获得的基本不同。
尽管直接在片下, 蠕虫的行为类似于在标准 NGM 板上看到的。产卵将继续孵化, 少量的食物沉积允许 L1 幼虫长成成人 (数据没有显示)。大块的琼脂大块大小允许合理的气体交换和抵抗脱水, 虽然太多食物将增加背景荧光。虽然这种方法适用于成年人, 该技术也可以用于图像 L4 动物。然而, 大块和片之间的水层的厚度是这样的, 小的幼虫很难爬行, 而且经常会在一个移动的成人之后被困住。
这种安装技术的一个限制是, 直接机械或化学刺激的精确区域的身体是困难的。在中间体48,49中, 采用阵列光照技术的最新发展, 可以单独激发头部或尾部表达的微生物 opsins, 并分别进行光照和检测 GCaMP/mCherry 荧光。因此, 可以使用 optogenetic 的方法来解决此问题。
与其他 Ca 的比较2 +图像处理方法:
这种方法非常适用于记录细胞质 Ca2 +中的单个、已解析的细胞及其突触区域的变化。实时的, 体积成像的大脑神经元最近已经取得了使用的位置, 细胞细胞核的细胞识别和定量变化的 nucleoplasmic 钙50,51,52。核和突触 Ca2 +之间的关系仍不清楚。我们的数据表明, 最明显的变化, HSN 胞内 Ca2 +发生在前总站远离细胞 soma (图 4D)。HSN 和 VC 神经元的前终点嵌在突触后外阴肌26。不清楚是否会有足够的分辨率在 Z 维度, 即使用旋转圆盘或光片技术将前 Ca2 +信号归咎于特定的细胞, 而不依赖于体细胞钙的细胞识别.使用这里描述的技术, 每10分钟, 双通道记录与 12001 256 x 256 16 位像素 TIFF 图像是〜 4 GB。比率和强度调制比通道将文件大小加倍到 ~ 8 GB。一个典型的实验记录10动物从每一个基因型 (野生类型和实验) 产生近 150 GB 的主要数据, 并要求20小时收集和分析。每 timepoint 10 Z 切片的体积分析需要更多的数据和分析时间, 解释为什么如此少的研究已经完成。
硬件:
我们在双处理器工作站上记录和分析图像序列, 具有高性能 (例如,游戏) 显卡、64 GB RAM 和高性能固态驱动器 (请参阅材料表)。数据应存储在独立磁盘的网络冗余阵列 (RAID) 上, 并在非现场数据中心备份到云上。
我们建议使用高功率准直 led 脉冲荧光激发金属卤化物或汞为基础的光源。市面上有多种可供使用的多色 LED 系统。虽然这些 LED 系统有较高的前期成本, 他们有一个长寿命 (> 2万小时), 可以同时激发四或更多不同的荧光, 并提供精确的时序控制使用的串行和/或 TTL 接口低滞后时间 (10-300 µs 开关时间)。触发可确保在实际收集数据时只照亮样本。我们通常使用 10 ms 曝光每50毫秒 (20% 的税率)。这减少了图像捕获期间的光和运动模糊, 如先前所报告的43。
我们使用 sCMOS 相机的速度和大阵列的小, 敏感的像素。一个 EM CCD 相机是一个更昂贵的替代品, 但 ' 布卢姆 ' 的效果可能导致捕获光电子溢出到相邻像素。新的后照 sCMOS 相机具有相似的光敏性 EM-ccd 在显著降低成本。无论使用哪种传感器, 都必须同时获得 GCaMP5 和 mCherry 通道。连续捕获移动蠕虫将导致不良的注册图像不适合比率定量。使用图像拆分器 (图 2) 或使用两个相同的相机, 可以在分离通道后将双通道成像完成到单个相机上。16位图像的动态范围建议超过8位图像的准确比率定量。对于明的蠕虫行为图像, 我们使用1英寸 4.1 MP 近红外 USB3 相机捕获大 2 x 2 搁置 1024 x 1024 8 位图像序列 JPEG 压缩后。在较新的显微镜模型上提供的较大的视可使成虫在 20x 0.63x demagnification 后可视化, 只有轻微的晕 (图 4E)。
我们建议使用标准的 TTL 电压信号来同步光照和帧捕获。由于在不同的软件程序中潜在的延迟, 我们建议用户配置的荧光相机与触发输出的 TTL 输出驱动所有其他设备的主。这样, 将为每个 Ca2 +测量收集明和阶段位置信息。
在共享共焦设备中通常发现的狭缝或共振点扫描共聚焦显微镜在比率 Ca2 +成像期间也提供了优异的性能。此类仪器可用于捕获两个或多个荧光通道以及明24。在这种情况下, 应将共焦针孔打开至其最大直径, 并应使用光谱探测器来分离 GCaMP5、mCherry 和红外明信号。这最大限度地从厚 (〜20µm) 切片的光收集, 同时仍然允许排斥外聚焦荧光。一个缺点是较小的视和更多的硬件和软件定制的限制。
软件:
大多数制造商都使用专有软件来安装他们的照相机和显微镜, 包括触发输入和输出的配置。在录制过程中, 此软件的功能和性能会有所不同。因为快速移动的蠕虫会对跟踪具有挑战性, 所以在录制过程中的图像显示需要平滑和稳定在20赫兹。为了提高性能, 可以将图像序列临时保存在 RAM 中, 并在实验结束时保存行为相关子集。这些双通道图像序列文件可以转换为开放的图像细胞仪标准格式 (. ics), 导入到比率定量软件。tiff 是一种较新的开源图像格式, 尽管不同的安装可能无法保存大于 4 GB 的 tiff 图像序列。
定量管道的一个关键特征是比率通道的生成, 然后采用 mCherry 荧光的无偏图像分割程序来查找感兴趣的细胞。从每个被发现的对象, 对象大小, XY 质心位置, 和最小, 平均和最大荧光强度值为每个渠道 (包括比率渠道) 被计算。这些值一起用于在记录中的每个 timepoint 定量在胞内 Ca2 +中进行的更改。然后将每个 timepoint 的对象度量导出为. csv 文件以供后续分析。
这里所描述的协议的一个主要限制是依赖于通过拼凑的软件产品以不同的形式移动数据。另外一种刺激是, 有些软件是开源的, 而其他的则是免费的, 而其他的则是封闭的、昂贵的和不均匀的更新。一个主要的改进是使用或开发一个软件 (理想的开源), 它提供相似的性能水平和从获取到分析的易用性。如上所述, 比率分析使完成实验所需的文件大小和时间加倍。生成可以集成到用户自定义框架 (如盆景) 中的摄像头驱动程序将允许实时收集和分析图像和其他数据流, 从而显著提高吞吐量。
未来前景:
虽然我们通常手动跟踪蠕虫的移动, 但在红外亮或暗场记录中检测到的蠕虫质心的跟踪应该允许额外的图像处理节点, 提供舞台位置的闭环调整和自动跟踪 (图 3和未显示的数据)。用这种方法获得的大多数荧光记录是深色的, 缺乏生物学上有趣的数据。传感器或实时后采集图像处理技术, 将图像序列裁剪到相关对象, 可以增加空间分辨率, 加快数据分析管道, 特别是在为每个timepoint 在所有前和突触后细胞的活动中可视化。
披露声明
作者声明, 没有竞争的利益存在。
致谢
这项工作的经费来自一至 KMC (R01 NS086932) 的赠款。LMN 得到了研究院 IMSD 计划 (R25 GM076419) 的资助。这项研究中使用的菌株是由 NIH 研究基础设施项目 (P40 OD010440) 资助的线虫遗传学中心。我们感谢詹姆斯. 贝克和梅森的有益讨论。
材料
| Name | Company | Catalog Number | Comments |
| C. elegans growth, cultivation, and mounting | |||
| Escherichia coli bacterial strain, OP50 | Caenorhabditis Genetic Center | OP50 | Food for C. elegans. Uracil auxotroph. E. coli B. Biosafety Level 1 |
| HSN GCaMP5+mCherry worm strain | Caenorhabditis Genetic Center | LX2004 | Integrated transgene using nlp-3 promoter to drive GCaMP5 and mCherry expression in HSN. Full genotype: vsIs183 [nlp-3p::GCaMP5::nlp-3 3'UTR + nlp-3p::mCherry::nlp-3 3'UTR + lin-15(+)], lite-1(ce314), lin-15(n765ts) X |
| lite-1(ce314), lin-15(n765ts) mutant strain for transgene preparation | author | LX1832 | Strain for recovery of high-copy transgenes after microinjection with pL15EK lin-15(n765ts) rescue plasmid. Also bears the linked lite-1(ce314) mutation which reduces blue-light sensitivity. Available from author by request |
| pL15EK lin-15a/b genomic rescue plasmid | author | pL15EK | Rescue plasmid for recovery of transgenic animals after injection into LX1832 lite-1(ce314), lin-15(n765ts) X strain. Available from author by request |
| pKMC299 plasmid | author | pKMC299 | Plasmid for expression of mCherry in the HSNs from the nlp-3 promoter. Has nlp-3 3' untranslated region |
| pKMC300 plasmid | author | pKMC300 | Plasmid for expression of GCaMP5 in the HSNs from the nlp-3 promoter. Has nlp-3 3' untranslated region |
| Potassium Phosphate Monobasic | Sigma | P8281 | For preparation of NGM plates |
| Potassium Phosphate Dibasic | Sigma | P5655 | For preparation of NGM plates |
| Magnesium Sulfate Heptahydrate | Amresco | 0662 | For preparation of NGM plates |
| Calcium Chloride Dihydrate | Alfa Aesar | 12312 | For preparation of NGM plates |
| Peptone | Becton Dickinson | 211820 | For preparation of NGM plates |
| Sodium Chloride | Amresco | 0241 | For preparation of NGM plates |
| Cholesterol | Alfa Aesar | A11470 | For preparation of NGM plates |
| Agar, Bacteriological Type A, Ultrapure | Affymetrix | 10906 | For preparation of NGM plates |
| 60 mm Petri dishes | VWR | 25384-164 | For preparation of NGM plates |
| 24 x 60 mm micro cover glasses, #1.5 | VWR | 48393-251 | Cover glass through which worms are imaged |
| 22 x 22 mm micro cover glasses, #1 | VWR | 48366-067 | Cover glass that covers the top of the agar chunk |
| Stereomicroscope with transmitted light base | Leica | M50 | Dissecting microscope for worm strain maintenance, staging, and mounting |
| Platinum iridium wire, (80:20), 0.2mm | ALFA AESAR | AA39526-BW | For worm transfer |
| Calcium imaging microscope | |||
| Anti-vibration air table | TMC | 63-544 | Micro-g' Lab Table 30" x 48" anti-vibration table with 4" CleanTop M6 on 25mm top |
| Inverted compound microscope | Zeiss | 431007-9902-000 | Axio Observer.Z1 inverted microscope |
| Sideport L80/R100 (3 position) | Zeiss | 425165-0000-000 | To divert 20% of output to brightfield (CMOS) camera, 80% to fluorescence (sCMOS) camera |
| Tilt Back Illumination Carrier | Zeiss | 423920-0000-000 | For infrared/behavior imaging |
| Lamphousing 12V/100W w/ Collector | Zeiss | 423000-9901-000 | For infrared/behavior imaging |
| Halogen lamp 12V/100W | Zeiss | 380059-1660-000 | For infrared/behavior imaging. White-light LEDs do not emit significant infrared light, so they will not allow brightfield imaging after the infrared bandpass filter |
| 32 mm Infrared bandpass filter (750-790 nm) for Halogen lamp | Zeiss | 447958-9000-000 | BP 750-790; DMR 32mm, for infrared illumination for brightfield and behavior |
| 6-filter Condenser Turret (LD 0.55 H/DIC/Ph), Motorized | Zeiss | 424244-0000-000 | For infrared/behavior imaging |
| Condenser & Shutter | Zeiss | 423921-0000-000 | For infrared/behavior imaging |
| Binocular eyepiece with phototube for infrared CMOS camera | Zeiss | 425536-0000-000 | For infrared/behavior imaging |
| Eyepiece 10x, 23mm | Zeiss | 444036-9000-000 | For worm localization on the agar chunk |
| C-Mount Adapter 2/3" 0.63x demagnifier | Zeiss | 426113-0000-000 | Mount for infrared CMOS camera |
| CMOS camera for infrared brightfield and behavior (1" sensor) | FLIR (formerly Point Grey Research) | GS3-U3-41C6NIR-C | Camera for brightfield imaging |
| USB 3.0 Host Controller Card | FLIR (formerly Point Grey Research) | ACC-01-1202 | Fresco FL1100, 4 Ports |
| 8 pins, 1m GPIO Cable, Hirose HR25 Circular Connector | FLIR (formerly Point Grey Research) | ACC-01-3000 | Cable for TTL triggering. The green wire connects to GPIO3 / Pin 4 and the brown wire connects to Ground / Pin 5 |
| Plan-Apochromat 20x/0.8 WD=0.55 M27 | Zeiss | 420650-9901-000 | Best combination of magnification, numerical aperture, and working distance |
| 6-cube Reflector Turret, Motorized | Zeiss | 424947-0000-000 | For fluorescence imaging |
| Fluorescence Light Train, Motorized | Zeiss | 423607-0000-000 | For fluorescence imaging |
| Fluorescence Shutter | Zeiss | 423625-0000-000 | For fluorescence imaging |
| GFP and mCherry dual excitation and emission filter cube (for microscope) | Zeiss | 489062-9901-000 | FL Filter Set 62 HE BFP+GFP+HcRed for fluorescence imaging |
| LED illumination system | Zeiss | 423052-9501-000 | Triggerable Colibri.2 LED system for fluorescent illumination |
| GFP LED module (470 nm) | Zeiss | 423052-9052-000 | Colibri.2 LED for GFP fluorescence excitation |
| mCherry LED module (590 nm) | Zeiss | 423052-9082-000 | Colibri.2 LED for mCherry fluorescence excitation |
| Iris stop slider for incident-light equipment | Zeiss | 000000-1062-360 | Field aperture iris to limit LED illumination to the camera field of view |
| C-Mount Adapter 1" 1.0x | Zeiss | 426114-0000-000 | Adapter for image-splitter and sCMOS fluorescence camera |
| Image splitter | Hamamatsu | A12801-01 | Gemini W-View, other image splitters may be used, but they may not be optimized for the large sensor size of the sCMOS cameras |
| GFP / mCherry dichroic mirror (image splitter) | Semrock | Di02-R594-25x36 | Splitting GCaMP5 from mCherry and infrared signals |
| GFP emission filter (image splitter) | Semrock | FF01-525/30-25 | Capturing GCaMP5 fluorescence |
| mCherry/ emission filter (image splitter) | Semrock | FF01-647/57-25 | This filter is necessary to exclude the infrared light used for brightfield imaging |
| sCMOS camera for fluorescence (1" sensor) | Hamamatsu | A12802-01 / C11440-22CU | Orca FLASH 4.0 V2. Newer models allow for separate image acquisition settings on separate halves of the sensor, allowing acquisition of two-channel images in combination with an image splitter |
| Motorized XY Stage | Märzhäuser | SCAN IM 130 x 100 | Stage movement; the XY resolution of this stage is 0.2µm per step |
| XY Stage controller with joystick | LUDL | MAC6000, XY joystick | Manual tracking of worms. MAC6000 controller should be connected to the PC through the serial (RS-232) port configured to 115200 baud |
| Digital Acquisition board (DAQ) | Arduino | Uno | Receiving TTL triggers from sCMOS camera. The Uno should be loaded with the standard Firmata package, and the computer USB port configured to 57600 baud |
| BNC Male to BNC Male Cable - 6 ft | Hosa Technology | HOBB6 | BNC connectors for TTL triggering |
| Gold-Plated BNC Male to SMA male coaxial cable (8.8") | uxcell | 608641773651 | To connect the fluorescence camera trigger outputs |
| BNC turn head adapter | Hantek | RRBNCTH21 | BNC to Banana Plug Adapter (4mm) |
| BNC female to female connector | Diageng | 20130530009 | Female to female BNC adapter to connect the BNC output from the camera to the Banana Plug |
| Solderless flexible breadboard jumper wires | Z&T | GK1212827 | To connect the BNC trigger outputs to the Arduiono DAQ. Male to male. |
| High performace workstation | HP | Z820 | Windows 7, 64GB RAM, Dual Xeon processor, solid state C: drive, serial (RS-232) port, multiple PCIe3 slots for ethernet connectivity, USB 3.0 cards, and additional solid state drives |
| M.2 Solid state drive | Samsung | MZ-V5P512BW | High-speed streaming and analysis of image data |
| M.2 Solid state drive adapter for workstations | Lycom | DT-120 | M.2 to PCIe 3.0 4-lane adapter |
| Network attached storage | Synology | DS-2415+ | Imaging data storage and analysis |
| Hard disk drives | Western Digital | WD80EFZX | RED 8 TB, 5400 RPM Class SATA 6 Gb/s 128MB Cache 3.5 Inch. Storage of imaging data (10 drives + 2 drive redundancy) |
| Software | |||
| Fluorescence Acquisition | Hamamatsu | HCImage DIA | Recording of two channel (GCaMP5 and mCherry) fluorescence image sequences at 20 fps |
| Brightfield Acquisition | FLIR (formerly Point Grey Research) | Flycapture | Recording of brightfield JPEG image sequences |
| Stage Serial Port Reader | Bonsai | https://bitbucket.org/horizongir/bonsai | Facilitates tracking of worms during behavior |
| LED controller software | Zeiss | Micro Toolbox Test 2011 | To set up the intensity and trigger inputs for the different LEDs in the Colibri.2 unit |
| ImageJ | NIH | https://imagej.net/Fiji/Downloads | Simple review of image sequences and formatting changes for import into Ratiometric Quantitation software |
| Excel | Microsoft | 2002984-001-000001 | For generating subsets of comma-separated value data from Volocity for MATLAB analysis |
| Peak Finding | MATLAB | R2017a | Script used for Ratio peak feature calculations |
| Ratiometric Quantitation | Perkin Elmer | Volocity 6.3 | Facilitates calculation of ratiometric image channels, image segmentation for object finding, and ratio measurement of found objects |
| Scripts | |||
| XY-stage-final.bonsai | Bonsai | TTL-triggered DAQ and stage position serial port reader | Records X and Y stage position (in microns) when the attached Arduino receives a positive TTL signal from sCMOS camera during frame exposure. Script writes a .csv file with four columns: frame number, X position (microns), Y position (microns), and the time elapsed between frames (typically ~50 msec when recording at 20 fps). X and Y stage position from this output (columns 2 and 3, respectively) are added to the X and Y centroid positions from the AnalyzeGCaMP_2017.m MATLAB script (columns 4 and 5, respectively), to give the final X and Y position of the fluorescent object for the recording. |
参考文献
- White, J. G., Southgate, E., Thomson, J. N., Brenner, S. The structure of the nervous system of the nematode Caenorhabditis elegans. Phil Trans R. Soc. 314, 1-340 (1986).
- Bargmann, C. I. Neurobiology of the Caenorhabditis elegans genome. Science. 282, 2028-2033 (1998).
- Piggott, B. J., Liu, J., Feng, Z., Wescott, S. A., Xu, X. Z. S. The neural circuits and synaptic mechanisms underlying motor initiation in C. elegans. Cell. 147, 922-933 (2011).
- Chung, S. H., Sun, L., Gabel, C. V. In vivo neuronal calcium imaging in C. elegans. J. Vis. Exp. JoVE. , (2013).
- Kerr, R., et al. Optical imaging of calcium transients in neurons and pharyngeal muscle of C. elegans. Neuron. 26, 583-594 (2000).
- Desai, C., Garriga, G., McIntire, S. L., Horvitz, H. R. A genetic pathway for the development of the Caenorhabditis elegans HSN motor neurons. Nature. 336, 638-646 (1988).
- Waggoner, L. E., Zhou, G. T., Schafer, R. W., Schafer, W. R. Control of alternative behavioral states by serotonin in Caenorhabditis elegans. Neuron. 21, 203-214 (1998).
- Zhang, M., et al. A self-regulating feed-forward circuit controlling C. elegans egg-laying behavior. Curr. Biol. CB. 18, 1445-1455 (2008).
- Shyn, S. I., Kerr, R., Schafer, W. R. Serotonin and Go modulate functional states of neurons and muscles controlling C. elegans egg-laying behavior. Curr. Biol. CB. 13, 1910-1915 (2003).
- Zhang, M., Schafer, W. R., Breitling, R. A circuit model of the temporal pattern generator of Caenorhabditis egg-laying behavior. BMC Syst. Biol. 4, 81 (2010).
- Kerr, R. A., Schafer, W. R. Intracellular Ca2+ imaging in C. elegans. Methods Mol. Biol. Clifton NJ. 351, 253-264 (2006).
- Akerboom, J., et al. Optimization of a GCaMP calcium indicator for neural activity imaging. J. Neurosci. Off. J. Soc. Neurosci. 32, 13819-13840 (2012).
- Dana, H., et al. Sensitive red protein calcium indicators for imaging neural activity. eLife. 5, (2016).
- Inoue, M., et al. Rational design of a high-affinity, fast, red calcium indicator R-CaMP2. Nat. Methods. 12, 64-70 (2015).
- Oheim, M., et al. New red-fluorescent calcium indicators for optogenetics, photoactivation and multi-color imaging. Biochim. Biophys. Acta. 1843, 2284-2306 (2014).
- Chen, T. -. W., et al. Ultrasensitive fluorescent proteins for imaging neuronal activity. Nature. 499, 295-300 (2013).
- Kim, C. K., Adhikari, A., Deisseroth, K. Integration of optogenetics with complementary methodologies in systems neuroscience. Nat. Rev. Neurosci. 18, 222-235 (2017).
- Storace, D., et al. Toward Better Genetically Encoded Sensors of Membrane Potential. Trends Neurosci. 39, 277-289 (2016).
- Brenner, S. The genetics of Caenorhabditis elegans. Genetics. 77, 71-94 (1974).
- Mello, C., Fire, A. DNA transformation. Methods Cell Biol. 48, 451-482 (1995).
- Berkowitz, L. A., Knight, A. L., Caldwell, G. A., Caldwell, K. A. Generation of stable transgenic C. elegans using microinjection. J. Vis. Exp. JoVE. , (2008).
- Clark, S. G., Lu, X., Horvitz, H. R. The Caenorhabditis elegans locus lin-15, a negative regulator of a tyrosine kinase signaling pathway, encodes two different proteins. Genetics. 137, 987-997 (1994).
- Edwards, S. L., et al. A novel molecular solution for ultraviolet light detection in Caenorhabditis elegans. PLoS Biol. 6, 198 (2008).
- Thapaliya, E. R., et al. Bioimaging with Macromolecular Probes Incorporating Multiple BODIPY Fluorophores. Bioconjug. Chem. 28, 1519-1528 (2017).
- Mariol, M. -. C., Walter, L., Bellemin, S., Gieseler, K. A rapid protocol for integrating extrachromosomal arrays with high transmission rate into the C. elegans genome. J. Vis. Exp. JoVE. , e50773 (2013).
- Collins, K. M., Koelle, M. R. Postsynaptic ERG Potassium Channels Limit Muscle Excitability to Allow Distinct Egg-Laying Behavior States in Caenorhabditis elegans. J. Neurosci. Off. J. Soc. Neurosci. 33, 761-775 (2013).
- Collins, K. M., et al. Activity of the C. elegans egg-laying behavior circuit is controlled by competing activation and feedback inhibition. eLife. 5, e21126 (2016).
- Li, P., Collins, K. M., Koelle, M. R., Shen, K. LIN-12/Notch signaling instructs postsynaptic muscle arm development by regulating UNC-40/DCC and MADD-2 in Caenorhabditis elegans. eLife. 2, 00378 (2013).
- Banerjee, N., Bhattacharya, R., Gorczyca, M., Collins, K. M., Francis, M. M. Local neuropeptide signaling modulates serotonergic transmission to shape the temporal organization of C. elegans egg-laying behavior. PLoS Genet. 13, 1006697 (2017).
- Lopes, G., et al. Bonsai: an event-based framework for processing and controlling data streams. Front. Neuroinformatics. 9, 7 (2015).
- Flavell, S. W., et al. Serotonin and the neuropeptide PDF initiate and extend opposing behavioral states in C. elegans. Cell. 154, 1023-1035 (2013).
- Hardaker, L. A., Singer, E., Kerr, R., Zhou, G., Schafer, W. R. Serotonin modulates locomotory behavior and coordinates egg-laying and movement in Caenorhabditis elegans. J. Neurobiol. 49, 303-313 (2001).
- Ringstad, N., Horvitz, H. R. FMRFamide neuropeptides and acetylcholine synergistically inhibit egg-laying by C. elegans. Nat. Neurosci. 11, 1168-1176 (2008).
- Hallem, E. A., et al. Receptor-type guanylate cyclase is required for carbon dioxide sensation by Caenorhabditis elegans. Proc. Natl. Acad. Sci. U. S. A. 108, 254-259 (2011).
- Tanis, J. E., Moresco, J. J., Lindquist, R. A., Koelle, M. R. Regulation of serotonin biosynthesis by the G proteins Galphao and Galphaq controls serotonin signaling in Caenorhabditis elegans. Genetics. 178, 157-169 (2008).
- Giordano-Santini, R., et al. An antibiotic selection marker for nematode transgenesis. Nat. Methods. 7, 721-723 (2010).
- Semple, J. I., Garcia-Verdugo, R., Lehner, B. Rapid selection of transgenic C. elegans using antibiotic resistance. Nat. Methods. 7, 725-727 (2010).
- Granato, M., Schnabel, H., Schnabel, R. pha-1, a selectable marker for gene transfer in C. elegans. Nucleic Acids Res. 22, 1762-1763 (1994).
- Mello, C. C., Kramer, J. M., Stinchcomb, D., Ambros, V. Efficient gene transfer in C.elegans: extrachromosomal maintenance and integration of transforming sequences. EMBO J. 10, 3959-3970 (1991).
- Gong, J., et al. The C. elegans Taste Receptor Homolog LITE-1 Is a Photoreceptor. Cell. 167, 1252-1263 (2016).
- Liu, J., et al. C. elegans phototransduction requires a G protein-dependent cGMP pathway and a taste receptor homolog. Nat. Neurosci. 13, 715-722 (2010).
- Bhatla, N., Horvitz, H. R. Light and hydrogen peroxide inhibit C. elegans Feeding through gustatory receptor orthologs and pharyngeal neurons. Neuron. 85, 804-818 (2015).
- Larsch, J., Ventimiglia, D., Bargmann, C. I., Albrecht, D. R. High-throughput imaging of neuronal activity in Caenorhabditis elegans. Proc. Natl. Acad. Sci. U. S. A. 110, 4266-4273 (2013).
- Zang, K. E., Ho, E., Ringstad, N. Inhibitory peptidergic modulation of C. elegans serotonin neurons is gated by T-type calcium channels. eLife. 6, (2017).
- Branicky, R., Miyazaki, H., Strange, K., Schafer, W. R. The voltage-gated anion channels encoded by clh-3 regulate egg laying in C. elegans by modulating motor neuron excitability. J. Neurosci. Off. J. Soc. Neurosci. 34, 764-775 (2014).
- Hendricks, M., Ha, H., Maffey, N., Zhang, Y. Compartmentalized calcium dynamics in a C. elegans interneuron encode head movement. Nature. 487, 99-103 (2012).
- Nagy, S., Huang, Y. -. C., Alkema, M. J., Biron, D. Caenorhabditis elegans exhibit a coupling between the defecation motor program and directed locomotion. Sci. Rep. 5, 17174 (2015).
- Leifer, A. M., Fang-Yen, C., Gershow, M., Alkema, M. J., Samuel, A. D. T. Optogenetic manipulation of neural activity in freely moving Caenorhabditis elegans. Nat. Methods. 8, 147-152 (2011).
- Stirman, J. N., et al. Real-time multimodal optical control of neurons and muscles in freely behaving Caenorhabditis elegans. Nat. Methods. 8, 153-158 (2011).
- Kato, S., et al. Global brain dynamics embed the motor command sequence of Caenorhabditis elegans. Cell. 163, 656-669 (2015).
- Nguyen, J. P., et al. Whole-brain calcium imaging with cellular resolution in freely behaving Caenorhabditis elegans. Proc. Natl. Acad. Sci. U. S. A. 113, 1074-1081 (2016).
- Toyoshima, Y., et al. Accurate Automatic Detection of Densely Distributed Cell Nuclei in 3D Space. PLOS Comput. Biol. 12, 1004970 (2016).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。