Method Article
粗病毒提取中 DNA 的组合分析及对感染叶 RNA 的分析发现新的病毒基因组
摘要
在这里, 我们提出了一种新的方法来识别植物病毒的双链 DNA 基因组。我们使用标准方法从受感染的树叶中提取 DNA 和 RNA, 并进行下一代测序。生物信息学工具将序列组合成重叠, 确定重叠代表病毒基因组并将基因组分配给分类组。
摘要
这种 metagenome 方法是用来识别植物病毒与循环 DNA 基因组及其转录。通常植物 DNA 病毒发生在他们的宿主低效度或不能被机械接种到另一宿主是很难传播, 以达到更大的感染性物质的价。受感染的叶子是地面在一个温和的缓冲与最佳的 pH 值和离子组成建议净化大多数杆状对逆转录。尿素被用来分解病毒和溶解细胞成分的包涵体。差速离心提供了病毒与植物污染物的进一步分离。蛋白酶 K 处理去除衣壳。然后将病毒 DNA 浓缩并用于下一代测序。重叠数据用于装配提交给 NCBI BLASTn 的信息, 以确定生成的数据集中的病毒序列的子集。在平行管道中, rna 是用标准的基于柱的 rna 提取方法从受感染的树叶中分离出来的。然后核糖体损耗被进行, 以丰富的一个子集的 mRNA 和病毒转录。从 rna 测序 (rna 序列) 中提取的组合序列被提交到 NCBI-BLASTn, 以确定此数据集中病毒序列的子集。在我们的研究中, 我们发现两个相关的全长 badnavirus 基因组在两个数据集。这种方法更倾向于另一种常用的方法, 即提取小 RNA 序列的总种群来重建植物病毒基因组序列。这后宏基因组管道恢复与病毒相关的序列, 是复古转录元素插入到植物基因组。这是结合生物化学或分子化验, 以进一步辨别积极的传染性药物。本研究中记录的方法, 恢复序列代表复制病毒可能表明有源性病毒感染。
引言
新兴的植物病害驱使研究人员开发新的工具来识别正确的因果剂。初次报告新的或复发的病毒疾病是基于常见的症状, 如马赛克和畸形的叶子, 静脉清除, 侏儒症, 枯萎, 病变, 坏死, 或其他症状。报告一种新病毒作为一种疾病的致病剂的标准是将其与其他污染病原体分离, 并将其传播到适当的宿主中, 并通过对原寄主物种的健康植物进行接种而重现这种疾病。这种方法的局限性是, 植物病毒的许多属依赖于昆虫或其他传播到合适寄主或返回原寄主物种的媒介。在这种情况下, 寻找合适的向量可以延长, 可能会有困难, 建立实验室殖民地的载体, 并进一步努力, 以制定一个实验性传输协议。如果不能取得成功的实验室传输研究的条件, 那么这项工作就没有达到报告新病毒病的标准。对于在其自然宿主中发生的病毒的低效度, 研究人员必须确定可供传播的替代宿主, 以维持足够的传染性种群进行研究。对于只感染少数植物的病毒物种, 这也可能成为增长的种群文化的障碍1。
近年来, 科学家们更经常使用高通量的宏基因组方法来发现环境中存在的病毒序列, 这可能与已知疾病无关, 但可以分配给分类物种和属。2,3,4. 这种在不同环境中发现和分类遗传物质的方法为描述自然界中的病毒多样性或它们在某种生态系统中的存在提供了一种方法, 但并不一定能证实一个框架, 可以确定一种明显疾病的致病因子。
Badnavirus属属 pararetroviruses 的家族Caulimoviridae 。这些病毒是杆状的形状与圆形双链 DNA 基因组约7至 9 kb。所有 pararetroviruses 通过 RNA 中间体复制。Pararetroviruses 存在作为 episomes 和复制独立植物染色体脱氧核糖核酸5,6。对病毒种群的田间研究表明, 这些病毒种群是遗传复杂的。此外, 通过高通量测序在一系列植物基因组中获得的信息揭示了许多 badnavirus 基因组片段被非法整合事件插入植物基因组的例子。这些内源 badnavirus 序列不一定与感染7、8、9、10、11相关。随后, 利用 episomal 基因组的亚群多样性以及内源序列12、13的发生, 将新的 badnaviruses 作为疾病的致病动因来识别。
虽然没有一个最佳的管道, 以发现新的 pararetrovirus 基因组, 有两种常见的方法, 以确定这些病毒作为致病剂的疾病。一种方法是丰富的小 RNA 序列从受感染的叶子, 然后组装这些序列, 以重建病毒基因组 (s)14,15,16,17。另一种方法是滚动圈放大 (RCA) 放大循环 DNA 病毒基因组18。RCA 的成功取决于叶子的年龄和病毒在选定的组织中的效价。RCA 产品受到限制消化和克隆成质粒, 直接测序19,20,21。
美人蕉黄斑驳病毒(CaYMV) 是一种 badnavirus, 被描述为在美人蕉黄斑驳病的病因, 虽然只有565的 bp 片段的基因组以前被隔离感染 cannas22。一项当代研究发现 CaYMV purpurata (开花姜;CaYMV-美联社)23。本研究的目的是从受感染的芭蕉百合中恢复完整的 badnavirus 基因组序列。我们描述了一种从植物污染物中纯化病毒的协议, 然后将病毒 dna 从这一制备中分离出来, 并准备一个 DNA 库以供使用。这种方法消除了中间分子放大步骤的需要。我们还从受感染植物中分离出 rna-后向的基因, 其中包括 rna 序列, 使用每种核酸制剂进行。组装重叠被发现与Badnavirus分类在两个数据集使用国家生物技术和信息中心 (NCBI) 基本的本地对齐搜索工具核酸 (BLASTn)。我们确定了两个 badnavirus 物种的基因组24。
研究方案
1. 采用标准方法对普通病毒进行差速离心纯化 。25
- 首先, 用200毫升研磨缓冲器 (0.5 米 NaH2华林 4, 0.5 米 Na2HPO4 (pH 7.2), 从患病的植物中切下80-100 克叶子, 并在4摄氏度研磨在一个混合的搅拌器上, 并在上碾磨. 和 0.5% (w/v) Na2如此3)。在这一过程的所有步骤上都要穿上实验室的大衣和手套。
- 然后, 将匀浆 (300 毫升) 转移到1.0 升烧杯中。加入18克尿素和25毫升10% 非离子洗涤剂 (t-10月-C6H4-(CH2)2OH) 匀浆内的化学罩。
注意: 对于这个步骤, 最好戴上安全护目镜和一个简单的个人防护口罩。 - 用磁力搅拌器在遮光罩中简单搅拌, 用箔盖上烧杯。然后将箔盖烧杯转到冷室, 在4摄氏度一夜之间用磁力搅拌器搅拌。
- 将匀浆输送到离心机转子瓶 (250 毫升容器) 和离心机在固定角度转子在 4000 x g 10 分钟在4°c。在一个化学油烟机, 恢复上清和过滤通过4层棉布。
- 将匀浆在38.5 毫升聚丙烯离心机管和离心机中分2.5 小时, 在4摄氏度 4万 x g。通常情况下, 检查管底部是否存在绿色颗粒, 以及沿管长的白色颗粒。倒上清液并保留两粒;把样品放在冰上。
注: 绿色颗粒含有叶绿体、淀粉和其他细胞器。 - 在化学罩里工作, 用橡皮警察把小球分开。并用重悬在1毫升 ddH2O 的每一个转子瓶中的白色颗粒在1-2 小时的过程中, 同时保持悬浮在4°c 过夜, 使材料完全溶解成溶液。离心机悬浮在 6000 x g 和在4°c 为10分钟去除剩余的残骸。
- 离心机集中悬浮在 13.6万 x g 为2小时在4°c 到颗粒病毒。并用重悬1毫升的缓冲颗粒 (50 毫米三盐酸, pH 7.5, 5 毫米氯化镁2)。
注意: 一个可选的步骤是治疗病毒与 DNAse I (10 µg/毫升) 10 分钟37摄氏度, 以消除非 encapsidated DNA,即污染叶绿体和线粒体 dna。然后, DNAse 我加入 EDTA 到1毫米。 - 扰乱病毒与40µL 2 µg/µL 蛋白酶 K 37 °c 15 分钟。
- 在化学罩内工作, 通过有机萃取来恢复病毒 DNA。在提取过程中戴上面罩、手套和实验室大衣, 防止潜在的急性健康影响。将1的苯酚-氯仿-异戊醇 (49:50:1) 量添加到样品中, 并用手在二十年代进行握手. 在室温下离心机在 1.6万 x g 处的5分钟. 去除上水相, 转移到新管。重复此提取两次或多次。将有机相放置在玻璃废料瓶中, 用于适当的机构化学处理26。
- 用乙醇沉淀浓缩 DNA。使用 0.3 M 醋酸钠的最后浓度 (pH 5.2) 和2.5 容量95% 乙醇。将样品在-20 °c 为30-60 分钟和离心机在 1.3万 x g 为10-20 分钟颗粒 DNA26。
- 在实验室的长凳上工作, 并用重悬 DNA 颗粒在1毫升0.1 毫米 TE 缓冲 (pH 8.0)。通过商业凝胶过滤柱 (通常用于聚合酶链反应 (PCR) 清理), 将悬浮液通过滤除, 以消除可能会阻碍其使用的盐和低分子量物质。
- 采用溴溴化染色法对样品进行1% 琼脂糖凝胶电泳分析, 以观察制剂的质量。使用 nanodrop 分光光度计评估 DNA 的质量。
注: 在1.85 和2.0 之间, 样品吸光度在260λ和280λ之间的比率通常表明, 制备是 "清洁" 的杂质, 是理想的质量。 - 使用基于芯片的毛细管电泳仪分析 DNA 的质量 (使用 5 pg 到 10 ng)。
注: 质量输出显示干净的峰值, 代表 DNA 片段分布的大小沿 X 轴。峰值高度表示碎片的丰度。锯齿状峰表示部分退化的碎片或化学污染物。圆曲线代表了 DNA 的涂片, 表明质量低劣。
2. 使用 DNA 和基于乳液的克隆扩增 (emPCR 放大) 的图书馆准备工作
注: 图书馆通常是由一个以客户为导向的服务机构编写的。
- 用喷雾器将 dna 转化为碎片, 以此来剪切 dna (> 200 ng) 的溶液。根据手册的说明, 结扎商业适配器27。
- 根据制造商的说明28,29,30进行 DNA 样品的 emPCR 放大。重复洗涤步骤三次, 每次洗完后, 将珠子在十年代的 minicentrifuge 中颗粒状. 每次洗完后弃上清。
注: 该程序始于捕获珠的准备, 洗涤在与套件提供的商业洗涤缓冲器。emPCR 通常用于模版放大。 - 热变性的 DNA 或 RNA 在95°c 2 分钟, 然后4°c, 直到准备使用。使用2亿分子的 dna/rna 到500万捕获珠在最后的体积30µL. 在 DNA/RNA 样品旁边准备一个模拟样本, 并与核酸样品以及模拟样品一起执行以下步骤。
- 在十年代以最大速度涡流乳化液管进行乳化, 然后将整个含量 (4 毫升) 倒入与平台均质体兼容的塑料搅拌管中。将搅拌管放在平台上, 将乳液混合在2000转5分钟。
- 将100µL 整除数的乳液放入8条带盖管或96井板中。使用制造商推荐的程序28, 将管盖或密封板并进行 emPCR。
注: PCR 完成后, 检查水井, 看看乳液是否完好无损, 然后继续。如果乳液坏了, 把整个井都扔掉。 - 穿上实验室大衣, 在化学罩里工作, 收集扩增的 DNA 珠子 (亚行)。真空吸入油井中的乳液, 并在50毫升管中收集珠子。用100µL 异丙醇冲洗两次水井, 并将冲洗液吸至同50毫升管。
- 涡流收集的乳液和并用重悬亚银与异丙醇的最终体积35毫升。以 930 x g 为5分钟, 将亚行颗粒取出并添加10毫升增强缓冲剂。涡的亚行, 然后清洗通过加入异丙醇到40毫升最终容积离心机, 并在每次清洗后丢弃上清, 并重复洗涤步骤两次。
- 用乙醇代替异丙醇进行最终清洗。添加增强缓冲器到35毫升最终体积, 漩涡, 并颗粒珠在 930 x g 5 分钟. 除去上清, 但留下2毫升的增强缓冲。
- 将悬浮液转移至离心管, 并简要离心机将其颗粒状于亚行。丢弃上清后, 用1毫升的增强缓冲液冲洗亚银颗粒两次。每次清洗后, 离心机并丢弃上清液。
- 为准备 DNA 库珠浓缩, 在珠子上加入1毫升 1 N 氢氧化钠。将亚行涡旋, 室温下孵化2分钟。离心机并丢弃上清液。重复这个洗涤步骤一次。
- 加入1毫升的退火缓冲液, 然后涡旋亚银, 在室温下孵化2分钟。简要地离心和放弃上清。使用100µL 退火缓冲再次重复此步骤。
- 为了将测序底漆退火到 DNA 中, 添加15µL 的序列底漆 A 和15µL 在试剂盒中提供的顺序底漆 B。简单地混合由涡流和放置离心管在一个热量块在65°c 5 分钟. 转移到冰2分钟。
- 用1.0 毫升的退火缓冲液洗涤三次。漩涡为 5 s 和放弃上清每次。
- 在测序之前, 用商业珠子计数器测量珠子的数量。应该至少有50万丰富的珠子。
注: 珠计数器是一种特殊的装置, 用于测量供应的离心管中的珠子。
3. 从受感染的美人蕉叶开始的一般 mRNA 分离和 dsDNA 合成 CaYMV 用 rt-pcr 法检测诊断底漆
- 在以后的所有步骤中, 佩戴实验室大衣和乳胶手套进行个人防护。在实验室的长凳上工作, 从树叶中收集12份样品, 然后将样品浸入液氮中。用珠磨均匀化。使用一个商业工具包, 它提供了一种标准的基于列的方法, 用于总的植物 RNA 隔离。将该试剂盒提供的胍-异硫氰酸酯裂解缓冲液添加到地面样品中, 并于二十年代摇动。
- 根据套件说明, 添加乙醇并彻底混合。将每个匀浆添加到将 RNA 绑定到膜的旋转柱上。洗三次, 将 RNA 洗脱到回收管24。
- 用分光光度计量化 rna, 测量260λ和280λ的吸光度比值. 用1% 琼脂糖凝胶电泳染色溴溴化物, 验证 rna 的完整性。
注: 在1.85 和2.0 之间的吸光度率表明, 制剂的质量是理想的。治疗 RNA 与 DNase I (10 µg/毫升) 为10分钟在37°c。使用商用自旋柱将 RNA 集中在无 RNase 水中31。在继续之前, 池 RNA 样本。 - 使用 rRNA 去除套件去除植物核糖体 RNA。整除磁珠到离心管上, 用 RNase 无水冲洗两次。涡流管整除到并用重悬, 将管子放在磁支架上, 等待液体清除。丢弃上清, 用磁珠泥沙溶液代替。涡并用重悬和添加1µL 的 RNase 抑制剂。
注: 此类试剂盒使用寡核苷酸与杂交的磁性珠子结合。该方法采用标准磁珠分离技术恢复成绩单24。 - 将 500 ng 与1.25 µg 的 RNA、无 RNase 水以及试剂盒提供的反应缓冲器结合起来。将混合物放置在50摄氏度的10分钟。从热中除去, 并在 RNAse 中加入洗涤过的磁性珠子。漩涡简要地和设置在室温下5分钟。
- 放置在磁性支架上, 等待液体清除。将上清液转移到新鲜的离心管。放在冰上。
- 使用基于解决方案的捕获方法来丰富外来体和200的 RNA, 以准备 cDNA 文库。
注: 双链 cDNA 文库通常是由一个以客户为导向的服务机构编写的。 - 使用商业 rna 破碎溶液 (0.136 克 ZnCl2和100毫米三盐酸 pH 值 7.0) 片段 rna。添加2µL 的溶液到18µL 的 RNA (200 ng 总)。旋转管在离心简要地, 放置样品在70°c 三十年代, 并且转移到冰。用2µL 0.5 米 EDTA ph 8.0 和28µL 的10毫米三盐酸 ph 值7.5 来停止反应。
- 将 RNA 与磁性珠子结合在室温下混合10分钟. 使用磁性选矿机收集珠子并丢弃上清。用200µL 70% 乙醇清洗珠三次。每洗一次, 然后在室温下将颗粒状珠干燥3分钟, 并用重悬19µL 10 毫米三盐酸盐 pH 值7.5。
- 将随机引物退火至碎片 RNA, 加热至70°c 10 分钟, 然后将管放在冰上2分钟. 用标准的商业 cDNA 合成试剂盒制备第一链和第二链 cdna。
- 用磁珠选矿机净化双链 cDNA。用800µL 70% 乙醇清洗三次。将每个洗涤和空气干燥的颗粒在室温下3分钟并用重悬在16µL 10 毫米三盐酸 pH 7.5。用磁珠选矿机将珠子与双绞链 cDNA 分离, 这是目前解决的问题。吹打新的200µL PCR 管去除该 cDNA。
- 使用 Taq 聚合酶和由商业图书馆准备工具包提供的 deoxyribonucleotides 混合物进行片段端修复。商业套件提供预稀释的适配器, 以增加到双链 cDNA 的每一端使用商业连接酶在25°c 10 分钟。
4. 从 mRNA 制备的粗病毒制剂和 dsDNA 文库中制备的 DNA 文库的研究
- 使用标准高通量 pyrosequencing 仪器, 并遵循所有推荐的制造商的协议, 以生成直接读数的 DNA 序列。使用商业测序试剂, 包括荧光标记核苷酸。
注: 有关详细信息, 请参阅制造商提供的仪器说明。 - 使用基因组组装软件进行后测序分析, 自动装配读数, 以生成第一组重叠, 平均长度为 < 700 bp. 使用 FastQC 软件在 iPlant/CyVerse 网站上执行质量控制检查原始序列数据32。选择序列与 Phred 分数≥30继续重建更长的序列从更小的序列读24使用映射和扩增子软件。
注: 有关详细信息, 请参阅制造商的指示。 - 使用 MEGABLAST 默认模块以及 Viridplantae (TaxID: 33090) 和病毒 (TaxID: 10239) 将这些组装的重叠提交到 NCBI BLASTn 分析, 作为限制生物体名称33。收集重叠的亚群, 显示出与报告的Badnavirus基因组有很高的相似性。
- 验证表示一个或多个候选全长病毒基因组的联接支架, 正确地生成与标准 badnavirus 基因组具有相同组织的帧内序列。为此, 将候选的全长病毒基因组输入到质粒绘图软件中。然后确认前15核苷酸包括一个 tRNA满足(TGGTATCAGAGCGAG), 这是高度保守的 badnaviruses 之间。在基因组 3 ' 端附近找到潜在的 polyadenylation 信号。注释完整的基因组, 以确定两个小码和一个大的 ORF 编码 polyprotein 的存在。然后使用专家系统门户翻译工具识别 badnavirus ORF1、ORF2 和 ORF3 翻译产品34。
注意: 这个科学软件是免费的, 将产生循环 DNA, 识别所有开放的阅读框架, 并提供立即输出, 以验证序列代表全长循环 DNA 基因组。 - 使用开放源码多序列比较工具, 肌肉和 CLUSTALW, 比较从 DNA 和 RNA 分析获得的病毒基因组35,36。
- 搜索 NCBI 核苷酸数据库, 获取 30 badnavirus 物种的完整基因组序列, 并将其导出为. fasta 格式的文档。上传序列到一个软件, 进行序列的进化遗传分析与病毒基因组序列获得的。使用肌肉37生成多个序列对齐和最大似然树。
5. 从受感染植物的病毒基因组 PCR 扩增对从头测序的质量评估
- 输入新确定的全长 badnavirus 基因组序列 (. fasta 格式) 进入免费在线 Primer3 工具, 以获得 PCR 引物38。确定将产生交错的产品 1,000-1, 500 bp 沿病毒基因组的整个长度的底漆集。发送序列到一个服务设施, 将合成和提供 PCR 引物。
注: 输出标识可接受的底漆对, 具有共同的和可接受的熔化温度和精确的底漆位置沿介绍的序列。 - 在实验室的长凳上工作, 戴着实验室的大衣和手套, 用一种自动的方法将5µg 的 dna 从病毒感染的和健康的控制叶中分离出来, 其中包括标准的顺磁性纤维素粒子, 以分离植物材料39的 dna。在离心管内液氮中冷冻叶片材料 (20-40 毫克), 用珠磨研磨。将样品与溶解缓冲剂结合在离心管中, 并向每个样品添加 RNase a。涡流样品为 10-20 s 和简要地旋转样品去除固体微粒。
注: 顺磁性纤维素颗粒具有较高的 dna 结合能力, 并能分离出纯 dna 的高产量。标准的商用二氧化硅柱方法用于 dna 隔离, 不能有效地从各种各样的植物中提取 dna。因此, 存在着许多方法来修改这些程序, 以提高个别植物物种的效率。选择顺磁性纤维素粒子法, 是因为它能从25多种草本被子植物中获得更多高质量的 DNA40。 - 使用商用试剂盒自动顺磁性 DNA 隔离。将300µL 的核酸酶免费水添加到每个商业试剂盒中, 并将植物裂解液转移到同一墨盒上。将墨盒放在墨盒机架中, 将柱塞放在离洗脱管最近的井中, 并将洗脱缓冲器放入洗脱管内。将墨盒装入自动核酸隔离机, 并运行植物 DNA 隔离协议41,42。
- 进行 pcr 衍生出一套重叠的 pcr 产物。使用5µM 的每向前和反向底漆与35周期的 PCR 放大。使用以下循环条件: 变性在95°c 六十年代, 退火在50°c 四十五年代, 并且引伸在72°c 为1-2 分钟以最后的引伸在72°c 为7-10 分钟. 使用预包装凝胶过滤柱消除盐和低分子量材料作为 在步骤 1.231中。
- 计算3:1 摩尔率的 pcr 产物到载体, 以确定 pcr 产品的数量结扎到 50 ng 线性化 pGEM 质粒43。使用控件插入 DNA 以确定结扎是否有效工作。使用 T4 DNA 连接酶 (3 U/µL) 在4摄氏度进行结扎过夜。然后转化为商业制备的 JM109 能力大肠杆菌细胞。使用控制 100 pg 的未切割质粒 DNA 作为一个积极的控制, 有效的转化。用抗生素和蓝/白选择100µL 转化细胞到 LB 琼脂板上以恢复结扎质粒26。孵化板为16-24 小时在37°c。
注: pGEM 载体有一个 lacZ 基因编码β-乳糖酶。在含有100µg/毫升氨苄西林、0.5 毫米 IPTG、80µg/毫升 5-溴-4-氯-3-indoyl β D galactopyranosidase (X 加仑) 的平板上生长的细菌, 由于β-乳糖酶活性而变蓝。pGEM 质粒以扰乱lacZ基因的方式进行线性化。包含 PCR 产品插入物的菌落扰乱 lacZ 基因, 不代谢 X-加仑。这些殖民地是白色的。因而殖民地与插入可以区分从那些, 不用插入由殖民地的颜色 (白色与蓝色)26。 - 使用标准柱基质粒隔离套件39将 DNA 从三个菌落中分离出来。三系列转化产物质粒。比较每个 DNA 序列与全新组装的病毒基因组产生的。使用 CLUSTALW 对齐序列并确保它们得到适当的排序。
结果
这种改进的病毒纯化方法提供了一种丰富的病毒 dna, 有助于识别两种病毒物种的研究和生物信息学。匀浆后, 离心在 4万 x g 2.5 小时, 有一个绿色颗粒在底部的管和一个白色颗粒沿长度。绿色颗粒被悬浮成一个离心管, 白色颗粒悬浮成两个离心管。采用标准的 CaYMV pcr 诊断底漆进行 pcr, 并检测出可溶性白色颗粒, 而不是绿色颗粒 (图 1A)。通过透射电镜检查了原油制剂的样品, 并观察了杆状粒子长度 124-133 nm (图 1B)。这是在预测的模态长度最 badnaviruses。DNA 从白色和绿色的小球和悬浮分别提取。在图 1C中, 我们从绿色和白色颗粒样本 (1.6 µg dna 的绿色分数和3.1 µg dna 中提取的白分数) 到0.8% 琼脂糖凝胶电泳, 并分析了溴溴化物中的 dna, 对 dna 进行了5µL。染色。绿色分数包含低分子量的 dna, 而白分数产生两个较高的分子量 dna, 以及较低的分子量 dna (图 1C)。图 1C所示的凝胶在 100 V 处运行40分钟, 在3车道的涂抹表明, 应降低凝胶电压以产生更清晰的波段。这些数据表明, 白颗粒是丰富的病毒。从白样本中提取的 dna (0.6 µg/毫升) 浓度较低, 但对其含量有足够的要求, 需要至少10的 dna 才能进行。零碎的 dna 被用来为一个图书馆准备一个库。
同时, 从受感染的美人蕉植物中提取 rna (图 1D) 用于高通量 RNA--对图书馆的准备、重叠、创建和识别病毒基因组序列进行了标准工作流 (图 1E)。比较了以 DNA 和 RNA 为起始材料的输出结果。
我们获得了188626的原始 dna 读取由使用 dna 分离的粗病毒制剂。读取被组装成13269重叠, BLASTn 被用来搜索核苷酸序列的 NCBI 数据集 (使用 Viridplantae TaxID: 33090 和病毒 TaxID: 10239 作为限制有机体) (图 1E)。NCBI-BLASTn 结果显示, 93% 的重叠是细胞序列, 22% 是未知的, 0.3% 是病毒重叠 (图 2A)。大多数被归类为细胞序列的重叠被确定为线粒体或叶绿体 DNA。在病毒重叠的数据集中, 32% 的病毒重叠与Caulimoviridae (不是 Badnavirus 序列) 的成员有关, 其中58% 与Badnavirus 有关.病毒重叠, 29% 是高度相似的 (e < 1 x 10-30) 到 CaYMV 分离 V17 ORF3 基因 (EF189148.1),甘蔗杆状病毒分离巴达维亚 D, 完全基因组 (FJ439817.1) 和香蕉条纹 CA 病毒完整基因组 (KJ013511)。在这个人群中, 有长的重叠, 类似于两个全长的基因组。
高通量 RNA 序列生产153488个被清洗的单独顺序读以平均读的长度 < 500 bp. Contig 装配减少了这到8243重叠。这些被提交给 NCBI-BLASTn (使用 Viridplantae TaxID: 33090 和病毒 TaxID: 10239 作为限制有机体) 和输出放置76% 重叠在一个类别的植物细胞序列, 23% 是未知的, 0.1% 被归类为病毒重叠 (图 2B). 对0.1% 人病毒重叠的人口进行更密切的检查, 确定其中68% 个被分配到Caulimoviridae (图 2B)。三大重叠在这个人口之内被辨认了以高度相似性 (e < 1 X 10-30) 到 CaYMV 分离 V17 ORF3 基因 (EF189148.1),甘蔗杆状病毒分离巴达维亚 D, 完全基因组 (FJ439817.1) 和香蕉条纹CA 病毒完整基因组 (KJ013511)。检查三重叠, 我们手动加入其中的两个, 以产生一个全长病毒基因组。
我们比较了病毒基因组长度重叠的 DNA 和 RNA 测序作为一个相互的支架, 以确认存在两个全长病毒基因组。6966 bp 的一个全长病毒基因组暂定为美人蕉黄斑驳相关病毒1 (CaYMAV-1) (图 3A)。第二个基因组是 7385 bp 和一个变种的 CaYMV 感染purpurata (CaYMV-Ap01) (图 3a)。
最后, PCR 引物被设计为克隆 ~ 1000 bp 片段的每种病毒, 用于差异检测两个基因组在227的美人蕉植物代表九个商业品种。在许多情况下, 个别植物感染了两种病毒。我们提供了 CaYMAV-1 和 CaYMV-Ap01 在12植物中的 rt-pcr 检测的例子。其中三只为 CaYMV-Ap01 阳性, 九例为两种病毒阳性 (图 3B)。

图 1: 病毒核酸制剂和工作流程.(A) 琼脂糖 (1.0%) 凝胶电泳 565 CaYMV 基因组的 bp PCR 片段。两个 PCR 产品检测的样品, 从白色颗粒 (车道 1, 2), 但不在绿色颗粒样品 (车道 3)。正控制 (+) 表示从被感染的植物 DNA 中扩增的 PCR 产物, 该产品使用一种涉及标准顺磁性纤维素粒子的自动方法隔离。车道 L 包含用于测量样品车道中线性 dna 带尺寸的标准的 DNA 阶梯。(B) 通过在被感染的芭蕉叶的粗分馏中回收的白色颗粒中透射电镜观察到的病毒微粒的例子。(C) 琼脂糖 (0.8%) 从绿色 (1 车道) 和白色 (2 车道) 微丸中恢复的 DNA 凝胶电泳, 在 A 组中通过 PCR 检测阳性。2车道旁边的红色和黄色斑点识别出两个高分子量的 DNA 带, 在白色分数中发生。(D) 琼脂糖 (1%) 用柱基 rna 纯化法回收总 RNA 的凝胶电泳。车道 L 包含用于测量样品车道中线性带尺寸的标准的 DNA 阶梯。1-6 车道含有从受感染的芭蕉叶中分离出来的 RNA, 它们被汇集到一个单一的样品中, 用于 ribo 和 RNA (E) 核酸隔离、库制备、测序、contig 组装和病毒基因组的原理管道。发现。请单击此处查看此图的较大版本.
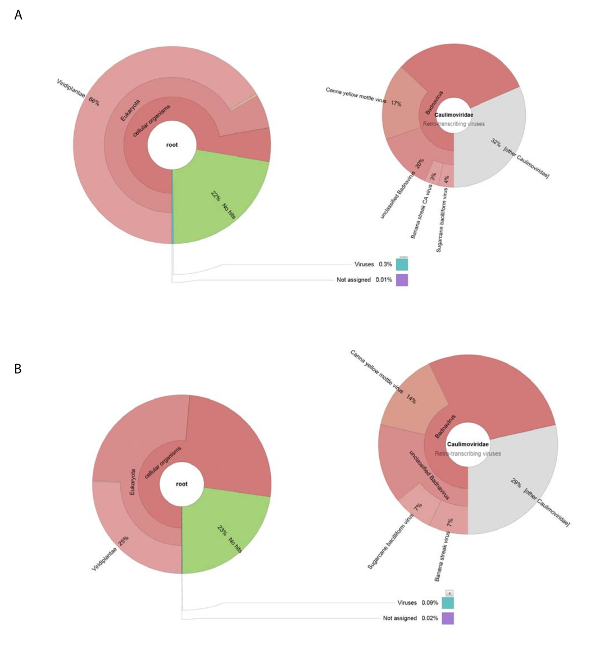
图 2: 克朗图可视化了重叠的分类类别.(A) 左边的图表显示了从粗病毒制剂中收集的重叠的丰度和分类分布。右图描述了与Caulimoviridae家族、 Badnavirus属和三密切相关的物种相关的病毒重叠的比例。(B) 左边的小组显示了基于 RNA 序列的重叠的丰富性, 它们的分类分布。右侧是描述与Caulimoviridae家族、 Badnavirus属和三种密切相关的病毒重叠种群内重叠丰度的图。请单击此处查看此图的较大版本.
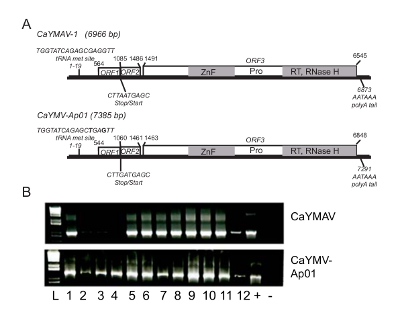
图 3.CaYMAV-1 和 CaYMV-Ap01 基因组的特征.(A)美人蕉黄斑驳副病毒 1 (CaYMAV) 和美人蕉黄斑驳病毒的图解表示, 类似于purpurata (CaYMV-Ap01)分离的基因组。核苷酸位置1-10 被确定为基因组的开始, 并包含一个 tRNA满足anticodon 站点典型的大多数 badnavirus 基因组。开放阅读框架 (ORF) 1 和2翻译的停止和开始位置相邻。这些蛋白质有未知的功能。ORF3 是一种含锌指 (ZnF)、蛋白酶 (Pro)、逆转录酶 (RT) 和 RNAse H 域的 polyprotein。3 ' 多 (A) 信号序列是保守的两个病毒基因组。(B) 采用 rt-pcr 方法对病毒感染的叶子和引物检测 CaYMAV 和 CaYMV-Ap01 的 RNA 进行 RT PCR 分析。在同一人口的12株植物中, 仅有三只感染 CaYMV-Ap01, 而其余的则感染 CaYMAV 和 CaYMV-Ap01。(+) 表示正控制, (-) 指示负控制。这个数字是从 Wijayasekara等复制/修改。24以允许。请单击此处查看此图的较大版本.
讨论
近年来, 在自然环境中研究植物病毒的生物多样性, 包括丰富的病毒样粒子 (vip) 或病毒特异 RNA 或 DNA2,3,44, 已采用多种方法, 45,46 。这些方法其次是生物信息学分析。本研究的目的是寻找一种常见疾病的致病剂在栽培植物。据报道, 这种疾病是一种未知病毒的结果, 它具有非封闭的杆状粒子, 并且只有565的 bp 片段被克隆了47。这些信息足以让先前的研究人员假设将病毒分配给家庭Caulimoviridae中的Badnavirus属。虽然先前的报告假设, 芭蕉百合的美人蕉斑驳病是单一 badnavirus 的结果, 使用本研究概述的 metagenomics 方法, 我们确定这一疾病是由两个暂定 badnavirus 物种24。因此, 使用 metagenome 方法来发现一种疾病的因果动因的力量是, 我们现在可以确定可能有不止一个原因的情况。
我们的方法结合 DNA 和 RNA 测序数据是彻底的, 并表明, 使用两种方法的结果产生了一致的结果, 并确认存在两个相关的病毒。我们使用了一个改良的程序, 以隔离 caulimoviruses, 并产生了一个样本, 丰富的病毒相关的核酸, 并在病毒衣壳内保护。承包了一个服务实验室进行 DNA 测序。在 dna 合成的时序周期中, dna 聚合酶将荧光标记的核苷酸纳入 dna 模板链中, 这是从头测序的基本概念。组装后的重叠被提交到一个生物信息学工作流中, 产生了几个重叠, 被确定为病毒重叠。通过对 ribo 的 rna 制剂的 rna 序列数据的生物信息学分析, 得到了两种病毒基因组10、24、48、49、50的进一步确认。一个有趣的结果是了解到, DNA 和 RNA 序列恢复的序列的数量提供了类似的非病毒和病毒核酸分布。对于 DNA 和 RNA 序列, < 0.5% 序列是病毒起源。在病毒序列的人口之内78-82% 属于家庭Caulimoviridae。通过将组装后的病毒重叠与 DNA 和 RNA 测序进行比较, 证实了两种组装的基因组均发生在两个数据集中。
一个只使用 DNA 测序来识别新病毒基因组的关注是 badnavirus 基因组是一个开放的循环 DNA。我们推测, 序列重叠的不连续性在基因组可能会阻碍基因组组装从重叠。对 DNA 测序结果的初步检验发现两种相似的病毒基因组。我们假设这些基因组要么代表了一个尚未研究的物种的遗传多样性, 要么代表了两个物种共同感染同一植物24。因此, 对 DNA 和 RNA 测序所获得的数据集进行的集体生物信息学分析, 能够证实两个全长基因组的存在。
还有另一份报告提出了一种从植物组织匀浆中提取 vip 和核酸的替代方法, 用于宏基因组研究, 其基础是从花椰菜镶嵌病毒(CaMV; caulimovirus) 中恢复 DNA 的程序.3。该方法确定了非栽培植物中新的 RNA 和 DNA 病毒序列。这项研究中所使用的 caulimovirus 隔离程序的步骤, 以发现一种栽培植物病害的致病剂, 不同于从自然感染植物中提取 vip 的步骤24。两种改进方法的成功表明, caulimovirus 隔离的框架程序可能是宏基因组研究一般植物病毒的一个重要起点。
披露声明
作者没有什么可透露的。
致谢
研究由俄克拉荷马州科技进步中心应用研究计划第二阶段 AR 132-053-2 资助;并由俄克拉荷马州农业部特种作物研究补助金项目。我们感谢鸿锦博士和一所由 NSF (EOS-0132534) 和 NIH (2P20RR016478-04、1P20RR16478-02 和 5P20RR15564-03) 赠款支持的俄勒冈州立的生物信息学核心设施。
材料
| Name | Company | Catalog Number | Comments |
| NaH2PO4 | Sigma-Aldrich St. Louis MO | S5976 | Grinding buffer for virus purification |
| Na2HPO4 | Sigma-Aldrich | S0751 | Grinding buffer for virus purification |
| Na2SO3 | Thermo-Fisher Waltham, MA | 28790 | Grinding buffer for virus purification |
| urea | Thermo-Fisher | PB169-212 | Homogenate extraction |
| Triton X-100 | Sigma-Aldrich | X-100 | Homogenate extraction |
| Cheesecloth | VWR Radnor, PA | 21910-107 | Filter homogenate |
| Tris | Thermo-Fisher | BP152-5 | Pellet resuspension& DNA resuspension buffers |
| MgCl2 | Spectrum, Gardena, CA | M1035 | Pellet resuspension buffer |
| EDTA | Spectrum | E1045 | Stops enzyme reactions |
| Proteinase K | Thermo-Fisher | 25530 | DNA resuspension buffer |
| phenol:chloroform:isoamylalcohol | Sigma-Aldrich | P2069 | Dissolve virion proteins |
| DNAse I | Promega | M6101 | Degrade cellular DNA from extracts |
| 95% ethanol | Sigma-Aldrich | 6B-100 | Virus DNA precipitation |
| Laboratory blender | VWR | 58984-030 | Grind leaf samples |
| Floor model ultracentrifuge &Ti70 rotor | Beckman Coulter, Irving TX | A94471 | Separation of cellular extracts |
| Floor model centrifuge and JA-14 rotor | Beckman Coulter | 369001 | Separation of cellular extracts |
| Magnetic stir plate | VWR | 75876-022 | Mixing urea into samples overnight |
| Rubber policeman | VWR | 470104-462 | Dissolve virus pellet |
| 2100 bioanalyzer Instrument | Agilent Genomics, Santa Clare, CA | G2939BA | Sensitive detection of DNA and RNA quality and quantity |
| 2100 Bioanalyzer RNA-Picochip | 5067-1513 | Microfluidics chip used to move, stain and measure RNA quality in a 2100 Bioanalyzer | |
| 2100 Bioanalyzer DNA-High Sensitive chip | 5067-4626 | Microfluidics chip used to move, stain and measure DNA quality in a 2100 Bioanalyzer | |
| Nanodrop spectrophotometer | Thermo-Fisher | ND-2000 | Analysis of DNA/RNA quality at intermediate steps of procedures |
| Plant total RNA isolation kit | Sigma-Aldrich | STRN50-1KT | Isolate RNA for RNA-seq |
| RNase-free water | VWR | 10128-514 | Resuspension of DNA and RNA for NGS |
| RNA concentrator spin column | Zymo Research, Irvine, CA | R1013 | Prepare RNA for RNA-seq |
| rRNA removal kit | Illumina, San Diego, CA | MRZPL116 | Prepare RNA for RNA-seq |
| DynaMag-2 Magnet | ThermoFisher | 12321D | Prepare RNA for RNA-seq |
| RNA enrichment system | Roche | 7277300001 | Prepare RNA for RNA-seq |
| Agarose | Thermo-Fisher | 16500100 | Gel analysis of DNA/RNA quality at intermediate steps of procedures |
| Ethidium bromide | Thermo-Fisher | 15585011 | Agarose gel staining |
| pGEM-T +JM109 competent cells | Promega, Madison, WI | A3610 | Clone genome fragments |
| pFU Taq polymerase | Promega | M7741 | PCR amplify virus genome |
| dNTPs | Promega | U1511 | PCR amplify virus genome |
| PCR oligonucleotides | IDT, Coralvill, IA | Custom order | PCR amplify virus genome |
| Miniprep DNA purification kit | Promega | A1330 | Plasmid DNA purification prior to sequencing |
| PCR clean-up kit | Promega | A9281 | Prepare PCR products for cloning |
| pDRAW32 software | ACAClone | Computer analysis of circular DNA and motifs | |
| MEGA6.0 software | MEGA | Molecular evolutionary genetics analysis | |
| Primer 3.0 | Simgene.com | ||
| Quant-iT™ RiboGreen™ RNA Assay Kit | Thermo-Fisher | R11490 | Fluorometric determination of RNA quantity |
| GS Junior™ pyrosequencing System | Roche | 5526337001 | Sequencing platform |
| GS Junior Titanium EmPCR Kit (Lib-A) | Roche | 5996520001 | Reagents for emulsion PCR |
| GS Jr EmPCR Bead Recovery Reagents | Roche | 5996490001 | Reagents for emulsion PCR |
| GS Junior EmPCR Reagents (Lib-A) | Roche | 5996538001 | Reagents for emulsion PCR |
| GS Jr EmPCR Oil & Breaking Kit | Roche | 5996511001 | Reagents for emulsion PCR |
| GS Jr Titanium Sequenicing kit* | Roche | 5996554001 | Includes sequencing reagents, enzymes, buffers, and packing beads |
| GS Jr. Titanium Picotiter Plate Kit | Roche | 5996619001 | Sequencing plate with associated reagents and gaskets |
| IKA Turrax mixer | 3646000 | Special mixer used with Turrax Tubes | |
| IKA Turrax Tube (specialized mixer) | 20003213 | Specialized mixing tubes with internal rotor for creating emulsions | |
| GS Nebulizers Kit | Roche | 5160570001 | Nucleic acid size fractionator for use during library preparations |
| GS Junior emPCR Bead Counter | Roche | 05 996 635 001 | Library bead counter |
| GS Junior Bead Deposition Device | Roche | 05 996 473 001 | Holder for Picotiter plate during centrifugation |
| Counterweight & Adaptor for the Bead Deposition Devices | Roche | 05 889 103 001 | Used to balance deposition device with picotiter plate centrifugation |
| GS Junior Software | Roche | 05 996 643 001 | Software suite for controlling the instrument, collecting and analyzing data |
| GS Junior Sequencer Control v. 3.0 | Roche | (Included in item 05 996 643 001 above) | |
| GS Run Processor v. 3.0 | Roche | (Included in item 05 996 643 001 above) | |
| GS De Novo Assembler v. 3.0 | Roche | (Included in item 05 996 643 001 above) | |
| GS Reference Mapper v. 3.0 | Roche | (Included in item 05 996 643 001 above) | |
| GS Amplicon Variant Analyzer v. 3.0 | Roche | (Included in item 05 996 643 001 above) |
参考文献
- Dijkstra, J., Jager, C. P. Practical Plant Virology : Protocols and Exercises. , Springer-Verlag. Berlin Heidelberg. 1 edn (1998).
- Roossinck, M. J. Plant virus metagenomics: biodiversity and ecology. Annu Rev Genet. 46, 359-369 (2012).
- Melcher, U., et al. Evidence for novel viruses by analysis of nucleic acids in virus-like particle fractions from Ambrosia psilostachya. J Virol Methods. 152 (1-2), 49-55 (2008).
- Stobbe, A. H., Schneider, W. L., Hoyt, P. R., Melcher, U. Screening metagenomic data for viruses using the e-probe diagnostic nucleic Acid assay. Phytopathology. 104 (10), 1125-1129 (2014).
- Borah, B. K., et al. Bacilliform DNA-containing plant viruses in the tropics: commonalities within a genetically diverse group. Mol Plant Pathol. 14 (8), 759-771 (2013).
- Bousalem, M., Douzery, E. J., Seal, S. E. Taxonomy, molecular phylogeny and evolution of plant reverse transcribing viruses (family Caulimoviridae) inferred from full-length genome and reverse transcriptase sequences. Arch Virol. 153 (6), 1085-1102 (2008).
- Geering, A. D., et al. Banana contains a diverse array of endogenous badnaviruses. J Gen Virol. 86, Pt 2 511-520 (2005).
- Kunii, M., et al. Reconstruction of putative DNA virus from endogenous rice tungro bacilliform virus-like sequences in the rice genome: implications for integration and evolution. BMC Genomics. 5, 80(2004).
- Laney, A. G., Hassan, M., Tzanetakis, I. E. An integrated badnavirus is prevalent in Figure germplasm. Phytopathology. 102 (12), 1182-1189 (2012).
- Gambley, C. F., Geering, A. D., Steele, V., Thomas, J. E. Identification of viral and non-viral reverse transcribing elements in pineapple (Ananas comosus), including members of two new badnavirus species. Arch Virol. 153 (8), 1599-1604 (2008).
- Gayral, P., et al. A single Banana streak virus integration event in the banana genome as the origin of infectious endogenous pararetrovirus. J Virol. 82 (13), 6697-6710 (2008).
- Lyttle, D. J., Orlovich, D. A., Guy, P. L. Detection and analysis of endogenous badnaviruses in the New Zealand flora. AoB Plants. 2011, 008(2011).
- Le Provost, G., Iskra-Caruana, M. L., Acina, I., Teycheney, P. Y. Improved detection of episomal Banana streak viruses by multiplex immunocapture PCR. J Virol Methods. 137 (1), 7-13 (2006).
- Singh, K., Talla, A., Qiu, W. Small RNA profiling of virus-infected grapevines: evidences for virus infection-associated and variety-specific miRNAs. Funct Integr Genomics. 12 (4), 659-669 (2012).
- Alfson, K. J., Beadles, M. W., Griffiths, A. A new approach to determining whole viral genomic sequences including termini using a single deep sequencing run. J Virol Methods. 208, 1-5 (2014).
- Kreuze, J. F., et al. Complete viral genome sequence and discovery of novel viruses by deep sequencing of small RNAs: a generic method for diagnosis, discovery and sequencing of viruses. Virology. 388 (1), 1-7 (2009).
- Zheng, Y., et al. VirusDetect: An automated pipeline for efficient virus discovery using deep sequencing of small RNAs. Virology. 500, 130-138 (2017).
- James, A. P., Geijskes, R. J., Dale, J. L., Harding, R. M. Molecular characterisation of six badnavirus species associated with leaf streak disease of banana in East Africa. Annals of Applied Biology. 158 (3), 346-353 (2011).
- Baranwal, V. K., Sharma, S. K., Khurana, D., Verma, R. Sequence analysis of shorter than genome length episomal Banana streak OL virus like sequences isolated from banana in India. Virus Genes. 48 (1), 120-127 (2014).
- Sukal, A., Kidanemariam, D., Dale, J., James, A., Harding, R. Characterization of badnaviruses infecting Dioscorea spp. in the Pacific reveals two putative novel species and the first report of dioscorea bacilliform RT virus 2. Virus Res. 238, 29-34 (2017).
- BÖmer, M., Turaki, A. A., Silva, G., Kumar, P. L., Seal, S. E. A sequence-independent strategy for amplification and characterisation of episomal badnavirus sequences reveals three previously uncharacterised yam badnaviruses. Viruses. 8 (7), (2016).
- Momol, M. T., Lockhart, B. E. L., Dankers, H., Adkins, S. Canna yellow mottle virus detected in Canna in Florida. Plant Health Progress. , August 2-4 (2004).
- Zhang, J., et al. Characterization of Canna yellow mottle virus in a new host, Alpinia purpurata, in Hawaii. Phytopathology. 107 (6), 791-799 (2017).
- Wijayasekara, D., et al. Molecular characterization of two badnavirus genomes associated with Canna yellow mottle disease. Virus Res. 243, 19-24 (2018).
- Covey, S. N., Noad, R. J., al-Kaff, N. S., Turner, D. S. Caulimovirus isolation and DNA extraction. Methods Mol Biol. 81, 53-63 (1998).
- Sambrook, J., Fritsch, E. F., Maniatis, T. Molecular cloning: A laboratory manual. 2nd edn. , Cold Spring Harbor Press. (1989).
- Radford, A. D., et al. Application of next-generation sequencing technologies in virology. J Gen Virol. 93, Pt 9 1853-1868 (2012).
- Kanagal-Shamanna, R. Emulsion PCR: Techniques and Applications. Methods Mol Biol. 1392, 33-42 (2016).
- Getts, D. R., et al. Targeted blockade in lethal West Nile virus encephalitis indicates a crucial role for very late antigen (VLA)-4-dependent recruitment of nitric oxide-producing macrophages. J Neuroinflammation. 9, 246(2012).
- van Dijk, E. L., Jaszczyszyn, Y., Thermes, C. Library preparation methods for next-generation sequencing: tone down the bias. Exp Cell Res. 322 (1), 12-20 (2014).
- Gel filtration principles and methods. GE Healthcare. , (2010).
- Goff, S., et al. The iPlant Collaborative: Cyberinfrastructure for Plant Biology. Frontiers in Plant Science. 2, (2011).
- Lin, Z., et al. Next-generation sequencing and bioinformatic approaches to detect and analyze influenza virus in ferrets. J Infect Dev Ctries. 8 (4), 498-509 (2014).
- Artimo, P., et al. ExPASy: SIB bioinformatics resource portal. Nucleic Acids Res. 40, Web Server issue 597-603 (2012).
- Edgar, R. C. MUSCLE: a multiple sequence alignment method with reduced time and space complexity. BMC Bioinformatics. 5, 113(2004).
- Hung, J. H., Weng, Z. Sequence Alignment and Homology Search with BLAST and ClustalW. Cold Spring Harb Protoc. 2016 (11), (2016).
- Sohpal, V. K., Dey, A., Singh, A. MEGA biocentric software for sequence and phylogenetic analysis: a review. Int J Bioinform Res Appl. 6 (3), 230-240 (2010).
- Untergasser, A., et al. Primer3--new capabilities and interfaces. Nucleic Acids Res. 40 (15), 115(2012).
- Dhaliwa, A. DNA extraction and purification. Mater Methods. 3, 191(2013).
- Moeller, J. R., Moehn, N. R., Waller, D. M., Givnish, T. J. Paramagnetic cellulose DNA isolation improves DNA yield and quality among diverse plant taxa. Appl. Plant Sci. 2 (10), (2014).
- Moeller, J. R., et al. Paramagnetic cellulose DNA isolation improves DNA yield and quality among diverse plant taxa. Appl. Plant Sci. 2 (10), (2014).
- Grooms, K. Review: Improved DNA Yield and Quality from Diverse Plant Taxa. , (2015).
- Nishimori, A., et al. In vitro and in vivo antivirus activity of an anti-programmed death-ligand 1 (PD-L1) rat-bovine chimeric antibody against bovine leukemia virus infection. PLoS One. 12 (4), 0174916(2017).
- Rojas, M. R., Gilbertson, R. L. Plant Virus Evolution. Roossinck, M. J. 1, Springer-Verlag. 27-51 (2008).
- Roossinck, M. J. The big unknown: plant virus biodiversity. Curr Opin Virol. 1 (1), 63-67 (2011).
- Roossinck, M. J., Martin, D. P., Roumagnac, P. Plant Virus Metagenomics: Advances in Virus Discovery. Phytopathology. 105 (6), 716-727 (2015).
- Momol, M. T., Lockhart, B. E. L., Dankers, H., Adkins, S. Plant Health Progress. , Online (2004).
- Eni, A., Hughes, J. D., Asiedu, R., Rey, M. Sequence diversity among badnavirus isolates infecting yam (Dioscorea spp.). Archives of Virology. 153 (12), Ghana, Togo, Benin and Nigeria. 2263-2272 (2008).
- Harper, G., et al. The diversity of Banana streak virus isolates in Uganda. Arch Virol. 150 (12), 2407-2420 (2005).
- Muller, E., Sackey, S. Molecular variability analysis of five new complete cacao swollen shoot virus genomic sequences. Arch Virol. 150 (1), 53-66 (2005).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。