需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
可重构 DNA 手风琴架的设计与合成
* 这些作者具有相同的贡献
摘要
我们描述了设计, 模拟, 湿实验室实验的详细协议, 和分析的可重构的 DNA 手风琴架 6 6 网格。
摘要
基于 dna 纳米结构的机械系统或 dna nanomachines, 在纳米到ångström 分辨率的2D 和3D 中产生复杂的纳米尺度运动, 在纳米技术的各个领域, 如分子反应器、药物输送, 都显示出巨大的潜力,和 nanoplasmonic 系统。描述了可重构的 dna 手风琴机架, 它可以集体操纵2D 或3D 纳米元素网络, 在多个阶段响应 dna 输入。该平台有可能增加 DNA nanomachines 可以从几个元素控制到网络规模的元素数量, 并可重新配置多个阶段。
在本协议中, 我们描述了 6 6 网格可重构 DNA 手风琴架的整个实验过程。该协议包括结构的设计规则和仿真程序, 以及用于合成和重构的湿实验室实验。此外, 该协议还包括了透射电镜 (透射电子显微镜) 和焦虑 (荧光共振能量传递) 的结构分析。本协议所涵盖的新颖设计和仿真方法将帮助研究人员使用 DNA 手风琴机架进行进一步的应用。
引言
基于 dna 纳米结构或 dna nanomachines 的机械系统,1,2,3,4,5是独一无二的, 因为它们在纳米的2D 和3D 中产生复杂的纳米尺度运动, 以ångström 决议, 根据各种生物分子刺激2,3,6。通过在这些结构上附加功能材料并控制它们的位置, 这些结构可应用于各个领域。例如, 为分子反应器7, 药物交付8和 nanoplasmonic 系统9,10提出了 DNA nanomachines。
以前, 我们介绍了可重构的 DNA 手风琴架, 它可以操纵2D 或3D 元素11 (图 1A) 的纳米尺度网络。与其他仅控制少数元素的 DNA nanomachines 不同, 该平台可以将周期性地排列2D 或3D 元素的过程集体地操作到不同的阶段。我们预计, 可编程的化学和生物反应网络或分子计算系统可以建立从我们的系统, 通过增加数量的可控元素。dna 手风琴架是一种结构, 其中多个 dna 束网络连接到由单链 dna 组成的关节 (图 1B)。由 dna 束产生的手风琴架由 dna 锁重新配置, 杂交在光束的粘性部分, 根据锁 (锁定状态) 的桥接部分的长度改变光束之间的夹角。此外, 通过通过基于立足点的绞线12、13分离 DNA 锁, 在自由状态形成后添加新锁, 可以证明多步重构。
在本协议中, 我们描述了可重构 DNA 手风琴架的整个设计和合成过程。该协议包括设计, 模拟, 湿实验室实验, 并分析了 6 6 网格的 DNA 手风琴架的合成和重新配置这些。该协议所涵盖的结构是前一项研究的基本模型,11是 65 nm, 大小为65纳米, 由14束组成。在设计和仿真方面, 手风琴架的结构设计不同于传统的 DNA 折纸14、15 (即紧密包装)。因此, 采用传统的方法对设计规则和分子模拟进行了改进。为了演示, 我们用 caDNAno14的改进方法和使用 oxDNA16、17和附加脚本的手风琴机架的仿真来展示设计技术。最后, 介绍了该方法的基本原理和对所配置的手风琴架结构分析的烦恼。
Access restricted. Please log in or start a trial to view this content.
研究方案
1. 设计6由6个 DNA 手风琴架与 caDNAno14
- 下载并安装 caDNAno 2.0 软件14设计一个 DNA 手风琴架 (caDNAno 2.5 也可在 https://github.com/cadnano/cadnano2.5)。打开 caDNAno14并单击正方形工具以添加一个正方形格子的新零件。
- 将手风琴架的每束光束编号, 并在 caDNAno14的左格子面板上绘制 (图 2)。
- 单击铅笔工具并在 caDNAno14的右侧编辑面板上绘制每个光束。每32的 bp, 这是为相邻梁之间的连接断裂光束。将短钉交叉处与接头位置相同。使用插入工具和铅笔工具在 caDNAno14 , 让关节有额外的单链分频器。
- 单击铅笔工具并连接接头。每束有七个关节。
- 使用先前报告的脚手架路由算法11, 生成脚手架分频器将支架合并到单个循环中。不要让脚手架和短链之间的最小绑定域小于 8 bp (图 3)。
- 将未在组件中使用的脚手架放置在折叠机架的另一侧的顶点上, 如图 3所示。
- 单击 "中断" 工具。折断短链是圆形或长于 60 bp。
- 设计 DNA 锁链。
- 单击 "中断" 工具。打破 8 bp 的主要 dna 区域, 使一个粘性部分和删除 8 bp 的主要 dna 区域。6 6 个手风琴架上有18个粘性零件 (图 1)。
- 将在锁链两端的粘滞部分反向互补的序列, 并由桥接区域连接, 由所需长度的聚 T 链组成 (图 1B)。
- 对于重构, 添加 8 bp 的立足点序列在 DNA 锁的末端为绞线位移。所用的立足点序列在表 2中。
- 准备聚一股, 这是反向互补的桥接区域。
- 设计的链, 是反向互补的 DNA 锁的重新配置实验。
- 单击序列工具, 然后单击 "脚手架 DNA"。选择脚手架作为标准 M13mp18. 单击导出工具并以 csv 格式保存序列 (表 1)。
2. 用 oxDNA 模拟结构
- 下载并安装 oxDNA16,17。最新的源代码可在 https://sourceforge.net/projects/oxdna/files/上使用。
- 使用 python 脚本 "cadnano_interface" (oxDNA16、17包中提供), 从 caDNAno14文件中启动配置文件。其用法如下: "python cadnano_interface cadnano_file. json 平方 '。此时将生成拓扑文件和配置文件。
注: 拓扑文件包括结构中有多少股和核苷酸, 以及核苷酸之间骨干-骨干键之间的信息。配置文件包含一般信息, 如 timestep、能量和框大小。研究了核苷酸的位置向量、主干基矢量、正常矢量、速度和角速度等方向信息 (图 4)。 - 从 caDNAno14更改拓扑和配置文件中的信息, 使其反映手风琴机架的真实结构信息。当 caDNAno14中的拓扑和配置文件可视化时, 所有光束都是平行排列的。然而, 手风琴架是一个格子结构, 所以粘结核苷酸之间的距离是很远的模拟 (图 5)。
- 旋转并移动每个光束到所需的晶格结构。配置文件左侧的九列是位置向量、主干基向量和正则向量 (图 4)。要旋转光束, 请使用旋转变换旋转所有位置、主干基和正常矢量。然后通过改变位置向量来移动一个光束, 以找到它, 如图 5所示。
- 使用 oxDNA 包中提供的脚本放松结构 (请参见 $oxDNA 中的示例/示例/RELAX_INITIAL_CONFIGURATION 以了解更多信息)。
- 使用轻松配置文件运行1000万步骤的分子动力学模拟。其用法如下: './oxDNA < 输入 > ' 每5000或10000个步骤保存数据。
- 可视 化
注意: 这些结构是使用 cogli 可视化的。- 下载并安装最新版本的 cogli (https://sourceforge.net/projects/cogli1/)。
- 使用 oxDNA 模拟中的拓扑和配置文件运行 cogli。其用法如下: "./cogli1 < 拓扑文件 > < 配置文件 >"。
- 按b把盒子藏起来。
3. 结构的综合
注: 合成方法适用于以前的15、18号议定书。
- 从寡核苷酸提供商那里购买设计的 DNA 主食。
- 使用无核酸酶的水, 将这些 DNA 主食的浓度调整为100微米。
- 将构成 "自由状态" 结构的每个 DNA 链放入一个管中, 并将每个链的浓度调整为2微米。
- 池 DNA 锁链的长度和数量的锁定点进入管和调整浓度为2微米的每一个链。使用18、9和4个锁定站点。添加聚一股, 这是互补的桥区, 在同一浓度。
- 池链, 是反向互补的 DNA 锁链长度进入管和调整浓度为2微米的每一个链。
- 准备氯化镁2溶液 300 nM 混合 70 ul 的核酸酶水和 30 ul 的1微米氯化镁2解决方案。通过混合 95 ul 的核酸酶水和 5 ul 的100x 三 edta 溶液, 制备5x 三 edta 溶液。
- 添加 2 ul 的主食 dna, 1.1 ul 的氯化镁2溶液, 2 ul 的三 EDTA 溶液, 7.6 ul 的核酸酶水和 7.3 ul 的脚手架 DNA 的浓度是 110 nM, 使 20 ul 的混合股票。将支架 dna 的最终浓度设置为 40 nm, 将 dna 短至 200 nm, 氯化镁2至16毫米, 三 EDTA 溶液为0.5x。
- 迅速加热混合股票溶液在一个热循环仪到80°c 和冷却到60°c 以每°c 4 分钟的速度和冷却从60°c 到4°c 以每°c 的速度40分钟。
4. 结构的净化
注: 在分析前, 所有结构的样品都经过纯化。在这一节中, 我们描述了 PEG 纯化的协议, 这是根据以前的文献19。样品也可以通过凝胶电泳纯化, 如前文15、18所述。
- 准备5米氯化钠和100x 三乙 EDTA。
- 通过混合 150 ul 的 PEG 8000, 500 ul 100x 三 EDTA 和 101 ul 的5米氯化钠和249核酸酶水的 ul, 准备沉淀缓冲。
- 通过混合 5.5 ul 300 nM 氯化镁2溶液从 3.3, 10 ul 5x 三 EDTA 溶液从3.3 和 84.5 ul 的核酸酶水, 准备目标缓冲区。
- 混合 20 ul 的合成结构从3和 20 ul 的沉淀缓冲从4.2 节。然后旋转的混合股票在 16000 x g 在4摄氏度。取出上清液, 从4.3 节中溶解靶缓冲器中的颗粒。
5. 将手风琴架从 "自由状态" 重新配置为 "锁定状态"
- 对构型实验无 DNA 锁的结构进行综合。
- 从3节准备 DNA 锁链。
- 将所需长度的 DNA 锁链的 2 ul 添加到合成结构的 20 ul 中。DNA 锁链的浓度比结构高五倍。
- 将样本孵化0、10、25、50或100分钟, 以查看重新配置的速度。
- 为100分钟孵化, 孵化样品在50°c 30 分钟, 慢慢降温到25°c 的速率0.33 °c/分钟。
- 为50分钟孵化, 孵化样品在50°c 15 分钟, 慢慢降温到25°c 的速率0.66 °c/分钟。
- 为25分钟孵化, 孵化样品在50°c 7.5 分钟, 慢慢降温到25°c 的速率1.32 °c/分钟。
- 为10分钟孵化, 孵化样品在50°c 3 分钟, 慢慢降温到25°c 的速率3.3 °c/分钟。
- 在0分钟的孵化, 储存样本在4摄氏度右后 DNA 锁链的添加。
- 在附加步骤后, 迅速冷却到4摄氏度的样品, 以防止不必要的变性。
6. 将手风琴架从 "锁定状态" 重新配置为 "自由状态"
- 用所需长度的 DNA 锁合成结构实验。
- 准备反向互补链从3节。
- 添加 2 ul 的股, 是反向互补的锁链的期望长度为 20 ul 的合成结构。DNA 锁链的浓度比结构高五倍。
- 孵化样本 0, 12, 60, 120, 240 分钟, 看看如何快速重构发生。
- 为 12, 60, 120, 240 分钟孵化, 迅速加热样品到40°c 和慢慢地冷却下来到20°c 为相应的时间。在分离步骤后, 快速冷却样品到4°c, 以防止不必要的变性。
- 在0分钟的孵化, 存储样品在4摄氏度右后, 反向互补链的补充。
7. TEM 成像
注: TEM 成像协议是根据以前的文献18,20改编的。
- 通过混合 87.5 ul 的无核酸酶水和 12.5 ul 10 米氢氧化钠溶液, 制备1.25 米氢氧化钠溶液。
- 添加 1 ul 1.25 米氢氧化钠溶液, 50 ul 的2% 甲酸铀溶液。
- 涡流解决方案3分钟, 离心机以最大速度3分钟。将纯化样品的 3 ul 存入辉光放电 TEM 网格上3分钟, 用滤纸迅速冲洗出来。
- 将制备的甲酸铀溶液的 7 ul 存入30秒, 用滤纸迅速冲洗。
- 用透射电镜测量手风琴结构的长度和角度。
8. 烦恼分析
- 使用阿托550和阿托647N 染料, Förster 距离为 6.5 nm。用荧光标记的链替换表 1中的主食58和主食117。然后用3节中描述的方法合成荧光标记的链结构。
- 测量纯化样品的浓度。
- 将样品正常化为 10 nM, 并将 50 ul 加载到384微型井。
- 激发样品与捐赠者和受体染料在550毫微米和测量荧光光谱从570毫微米到800毫微米与荧光计。
- 用同样的方法测量供体样本的荧光光谱。
- 激发样品的染料在650毫微米和荧光光谱和测量从670毫微米到800毫微米。这是测量受体的浓度。
- 通过重复相同的实验, 分别合成纯化三个样品, 获得标准偏差。
- 计算的烦恼效率与比率的方法, 如所描述的等式低于21。
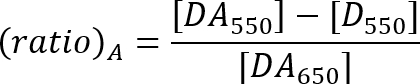
DA550: 受体的峰值荧光强度的样本与捐助者和受体在 550 nm 激发。
D550: 在 550 nm 激发下, 仅供方样品的受体发射范围内的荧光强度。
DA650: 受体的峰值荧光强度的样本与捐助者和受体在 650 nm 激发。
Access restricted. Please log in or start a trial to view this content.
结果
设计的6由 6 DNA 手风琴机架模拟从 oxDNA16,17和结果显示在图 6。仿真结果表明, 该结构在不变形的情况下形成了拟合结构。
图 7中的 TEM 图像是配置结构的图像, 其锁定长度为2、8、13和 20 bp。在图像上, 当锁的长度变长时, 结构的角度 (
Access restricted. Please log in or start a trial to view this content.
讨论
该协议介绍了从设计、仿真、合成和分析基本的 2D DNA 手风琴机架的整个过程。修改后的设计和仿真规则被描述, 因为设计规则不同于标准的 dna 折纸, 因为 dna 手风琴架有额外的核苷酸在分频器为灵活性14,15。由此, 我们期望该协议可以加速各种研究使用 DNA 手风琴架。此外, 所描述的协议也可以应用于其他研究使用 dna 纳米结构, 而不是标准的 dna 折纸。
Access restricted. Please log in or start a trial to view this content.
披露声明
作者没有什么可透露的
致谢
这项研究由科学和信息和通信技术部 (MSIT) (2015K1A4A3047345) 资助的韩国国家研究基金会 (NRF) 在全球研究发展中心的项目中得到部分支持, Nano·通过由科学和信息和通信技术部 (MSIT) 资助的韩国国家研究基金会 (NRF) 提供的材料技术发展方案 (2012M3A7A9671610)。首尔国立大学工程研究所为这项工作提供了研究设施。作者感谢泰青年尹 (生物科学, 首尔国立大学) 关于荧光光谱的烦恼分析。
Access restricted. Please log in or start a trial to view this content.
材料
| Name | Company | Catalog Number | Comments |
| M13mp18 Single-stranded DNA | NEB | N4040s | |
| 1M MgCl2 Solution | Biosesang | M2001 | |
| Tris-EDTA buffer | Biosesang | T2142 | |
| Nuclease-Free Water | Qiagen | 129114 | |
| 5M Sodium Chloride solution | Biosesang | s2007 | |
| PEG 8000 | Sigma Aldrich | 1546605 | |
| 10N NaOH | Biosesang | S2038 | |
| Uranyl formate | Thomas Science | C993L42 | |
| Thermal cycler C1000 | Biorad | ||
| Nanodropic 2000 | Thermo Fisher Scientific | ||
| TEM (LIBRA 120) | Carl Zeiss | ||
| Fluorometer Enspire 2300 | Perkin-Elmer | ||
| Centrifuge | Labogene | LZ-1580 |
参考文献
- Andersen, E. S., et al. Self-assembly of a nanoscale DNA box with a controllable lid. Nature. 459 (7243), 73-76 (2009).
- Cha, T. -G., et al. Design principles of DNA enzyme based walkers: Translocation kinetics and photo-regulation. Journal of the American Chemical Society. 137 (29), 9429-9437 (2015).
- Gerling, T., Wagenbauer, K. F., Neuner, A. M., Dietz, H. Dynamic DNA devices and assemblies formed by shape-complementary, non-base pairing 3D components. Science. 347 (6229), 1446-1452 (2015).
- Pinheiro, A. V., Han, D., Shih, W. M., Yan, H. Challenges and opportunities for structural DNA nanotechnology. Nature nanotechnology. 6 (12), 763-772 (2011).
- Li, J., et al. Exploring the speed limit of toehold exchange with a cartwheeling DNA acrobat. Nature Nanotechnology. 1, (2018).
- Krishnan, Y., Simmel, F. C. Nucleic acid based molecular devices. Angewandte Chemie International Edition. 50 (14), 3124-3156 (2011).
- Liu, M., et al. A DNA tweezer-actuated enzyme nanoreactor. Nature communications. 4, 2127(2013).
- Douglas, S. M., Bachelet, I., Church, G. M. A logic-gated nanorobot for targeted transport of molecular payloads. Science. 335 (6070), New York, N.Y. 831-834 (2012).
- Kuzyk, A., et al. Reconfigurable 3D plasmonic metamolecules. Nature Materials. 13 (9), 862-866 (2014).
- Zhou, C., Duan, X., Liu, N. A plasmonic nanorod that walks on DNA origami. Nature communications. 6, 8102(2015).
- Choi, Y., Choi, H., Lee, A. C., Lee, H., Kwon, S. A Reconfigurable DNA Accordion Rack. Angewandte Chemie International Edition. 57 (11), 2811-2815 (2018).
- Chen, H., et al. Understanding the Mechanical Properties of DNA Origami Tiles and Controlling the Kinetics of their Folding and Unfolding Reconfiguration. Journal of the American Chemical Society. 136 (19), 6995-7005 (2014).
- Han, D., Pal, S., Liu, Y., Yan, H. Folding and cutting DNA into reconfigurable topological nanostructures. Nature Nanotechnology. 5 (10), 712-717 (2010).
- Douglas, S. M., et al. Rapid prototyping of 3D DNA-origami shapes with caDNAno. Nucleic Acids Research. 37 (15), 5001-5006 (2009).
- Castro, C. E., et al. A primer to scaffolded DNA origami. Nature methods. 8 (3), 221-229 (2011).
- Ouldridge, T. E., Louis, A. A., Doye, J. P. K. DNA Nanotweezers Studied with a Coarse-Grained Model of DNA. Physical Review Letters. 104 (17), 178101(2010).
- Snodin, B. E. K., et al. Direct Simulation of the Self-Assembly of a Small DNA Origami. ACS Nano. 10 (2), 1724-1737 (2016).
- Amir, Y., Abu-Horowitz, A., Bachelet, I. Folding and Characterization of a Bio-responsive Robot from DNA Origami. Journal of Visualized Experiments. (106), e51272(2015).
- Stahl, E., Martin, T. G., Praetorius, F., Dietz, H. Facile and Scalable Preparation of Pure and Dense DNA Origami Solutions. Angewandte Chemie International Edition. 53 (47), 12735-12740 (2014).
- Wei, B., Vhudzijena, M. K., Robaszewski, J., Yin, P. Self-assembly of Complex Two-dimensional Shapes from Single-stranded DNA Tiles. Journal of Visualized Experiments. (99), e52486(2015).
- Clegg, R. M. Fluorescence resonance energy transfer and nucleic acids. Methods in enzymology. 211, 353-388 (1992).
- Kopperger, E., et al. A self-assembled nanoscale robotic arm controlled by electric fields. Science. 359 (6373), New York, N.Y. 296-301 (2018).
- Lauback, S., et al. Real-time magnetic actuation of DNA nanodevices via modular integration with stiff micro-levers. Nature Communications. 9 (1), 1446(2018).
Access restricted. Please log in or start a trial to view this content.
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。