È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Progettazione e sintesi di un Rack di fisarmonica di DNA riconfigurabile
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Descriviamo il protocollo dettagliato per la progettazione, la simulazione, gli esperimenti di laboratorio bagnato e analisi per un rack di fisarmonica DNA riconfigurabile di 6 per 6 maglie.
Abstract
Sistemi meccanici basati su nanostrutture di DNA o DNA nanomacchine, che producono movimento complessi su scala nanometrica in 2D e 3D in nanometro ångström risoluzione, mostrano il grande potenziale in vari campi della nanotecnologia come i reattori molecolare, consegna della droga, e sistemi di nanoplasmonic. Il rack di fisarmonica DNA riconfigurabile, che può manipolare collettivamente una rete su scala nanometrica 2D o 3D di elementi, in più fasi in risposta agli ingressi del DNA, è descritto. La piattaforma ha il potenziale per aumentare il numero di elementi che DNA nanomacchine controllabili da pochi elementi per una scala di rete con più fasi di riconfigurazione.
In questo protocollo, descriviamo l'intero processo sperimentale del rack fisarmonica DNA riconfigurabile di 6 per 6 maglie. Il protocollo comprende una procedura di simulazione e regola di progettazione delle strutture e un esperimento di bagnato-laboratorio per sintesi e riconfigurazione. Inoltre, analisi della struttura utilizzando TEM (microscopia elettronica di trasmissione) e FRET (trasferimento di energia di risonanza di fluorescenza) sono incluso nel protocollo. I nuovi metodi di progettazione e simulazione coperti in questo protocollo aiuterà i ricercatori a utilizzare il rack di fisarmonica di DNA per ulteriori applicazioni.
Introduzione
Sistemi meccanici basati su nanostrutture di DNA o DNA nanomacchine1,2,3,4,5 sono unici perché producono movimento complessi su scala nanometrica in 2D e 3D in nanometro a Ångström risoluzione, secondo vari biomolecolari stimoli2,3,6. Associare i materiali funzionali su queste strutture e controllando le loro posizioni, queste strutture possono essere applicate alle varie aree. Per esempio, DNA nanomacchine sono stati proposti per un reattore molecolare7, droga consegna8e nanoplasmonic sistemi9,10.
In precedenza, abbiamo introdotto il riconfigurabile rack fisarmonica del DNA, che può manipolare una rete su scala nanometrica 2D o 3D di elementi11 (Figura 1A). A differenza di altre DNA nanomacchine che controllano solo pochi elementi, la piattaforma può manipolare collettivamente periodicamente disposti elementi 2D o 3D in varie fasi. Prevediamo che una rete di reazione chimica e biologica programmabile o un sistema di calcolo molecolare può essere costruito dal nostro sistema, aumentando il numero di elementi controllabili. La cremagliera della fisarmonica di DNA è una struttura, in cui la rete di fasci multipli di DNA è collegata alle articolazioni composti di DNA single-stranded (Figura 1B). La cremagliera della fisarmonica generata dai fasci del DNA viene riconfigurata da blocchi di DNA, che ibridano a parte adesiva di travi e cambiare l'angolo tra le travi secondo la lunghezza della parte passerella delle serrature (stato bloccato). Inoltre, multi-step riconfigurazione è dimostrato aggiungendo nuove serrature dopo la formazione dello stato libero rimuovendo blocchi di DNA attraverso basati su toehold strand displacement12,13.
In questo protocollo, descriviamo l'intero processo di progettazione e sintesi di rack fisarmonica DNA riconfigurabile. Il protocollo comprende progettazione, simulazione, gli esperimenti di laboratorio bagnato e analisi per la sintesi del DNA della fisarmonica rack di 6 per 6 maglie e una riconfigurazione di questi. La struttura coperta nel protocollo è il modello base della precedente ricerca11 e 65 nm da 65 nm in dimensioni, composto da 14 fasci. In termini di progettazione e simulazione, la progettazione strutturale della cremagliera della fisarmonica è diversa da convenzionale del DNA origami14,15 (cioè, imballato strettamente). Pertanto, la regola di progettazione e simulazione molecolare sono stati modificati dai metodi tradizionali. Per illustrare, mostriamo la tecnica di progettazione utilizzando il metodo modificato di caDNAno14 e la simulazione del rack fisarmonica utilizzando oxDNA16,17 con script aggiuntivi. Infine, sono descritti entrambi i protocolli di TEM e FRET per l'analisi delle strutture configurato rack della fisarmonica.
Access restricted. Please log in or start a trial to view this content.
Protocollo
1. progettazione dei 6 da 6 DNA fisarmonica Rack con caDNAno14
- Scaricare e installare caDNAno 2.0 software14 per progettare un rack di fisarmonica di DNA (caDNAno 2.5 è anche disponibile su https://github.com/cadnano/cadnano2.5). Aprire caDNAno14 e fare clic sullo Strumento di Piazza per aggiungere una nuova parte con un reticolo quadrato.
- Numero ogni fascio della cremagliera della fisarmonica e disegnare sul pannello di sinistra della grata del caDNAno14 (Figura 2).
- Fare clic sullo strumento matita e disegnare ogni fascio sul pannello di destra modifica il caDNAno14. Interruzione di travi ogni 32 bp, che è per giunti tra fasci adiacenti. Posto fiocco crossover nella stessa posizione come le articolazioni. Utilizzare lo Strumento di inserimento e lo Strumento matita in caDNAno14 a lasciare i giunti hanno ulteriori singolo filamento crossover.
- Fare clic sullo Strumento matita e collegare i giunti. Ogni fascio ha sette articolazioni.
- Generare patibolo crossover per unire le impalcature il singolo ciclo utilizzando il patibolo precedentemente segnalato11di algoritmo di routing. Non lasciate il dominio obbligatorio minimo tra le ciocche impalcatura e fiocco sia minore di 8 bp (Figura 3).
- Posizionare i ponteggi che non vengono utilizzati nell'assieme i vertici situati sui lati opposti della cremagliera della fisarmonica, come mostrato nella Figura 3.
- Scegliere il Tool di rompere. Rompere i fili dove fili graffette sono circolari o più di 60 bp.
- Progettare i filamenti di DNA serratura.
- Scegliere il Tool di rompere. Pausa 8 bp di una regione di DNA fiocco per fare una parte appiccicosa ed eliminare 8 bp di una regione di DNA fiocco. Ci sono 18 parti appiccicosi (Figura 1) nel rack fisarmonica 6 per 6.
- Inserire sequenze che sono inversione complementari alle parti appiccicose ad entrambe le estremità dei fili di blocco e collegarli da una regione gettante un ponte, che consiste di poli T fili della lunghezza desiderata (Figura 1B).
- Per la riconfigurazione, aggiungere 8 bp delle sequenze di punto d'appoggio all'estremità del DNA serrature per spostamento del filo. La sequenza di punto d'appoggio utilizzata è nella tabella 2.
- Preparare i fili di poli A cui sono invertire complementari alla regione gettante un ponte.
- Fili di progettazione che sono invertire complementari per i blocchi di DNA per l'esperimento di riconfigurazione.
- Fare clic sullo Strumento di sequenza e fare clic su impalcatura DNA. Scegliere il patibolo come standard M13mp18. fare clic sullo Strumento Esporta e Salva sequenza in formato csv (tabella 1).
2. simulare la struttura con il oxDNA
- Scaricare e installare il oxDNA16,17. L'ultimo codice sorgente è disponibile su https://sourceforge.net/projects/oxdna/files/.
- Rendere i file di configurazione iniziale dal caDNAno14 file utilizzando script python 'cadnano_interface.py', che viene fornito nel pacchetto di17 16,oxDNA. L'utilizzo è come segue: 'python cadnano_interface.py cadnano_file.json sq'. Il file della topologia e il file di configurazione vengono ora generati.
Nota: Il file della topologia include quanti fili e nucleotidi sono nella struttura e informazioni per quanto riguarda la spina dorsale-backbone legami tra nucleotidi. Il file di configurazione contiene informazioni generali quali timestep, energia e formato del contenitore. Informazioni di orientamento come vettore posizione, vettore di spina dorsale-base, vettore normale, velocità e velocità angolare di nucleotidi è anche incluso (Figura 4). - Modificare le informazioni nel file di configurazione e topologia da caDNAno14 per farli riflettere le informazioni reali strutturali della cremagliera della fisarmonica. Tutti i fasci sono disposti in parallelo quando sono visualizzati i file di configurazione e topologia da caDNAno14 . Tuttavia, il rack della fisarmonica è una struttura reticolare così la distanza fra i nucleotidi legati sono lontano per simulazione (Figura 5).
- Ruotare e spostare ogni trave a struttura reticolare desiderata. Le nove colonne sulla sinistra nel file di configurazione sono il vettore posizione, vettore di spina dorsale-base e vettore normale (Figura 4). Per ruotare un fascio, ruota tutto della posizione, spina dorsale-base e normali vettori utilizzando la trasformazione di rotazione. Quindi spostare una trave modificando il vettore posizione per individuarlo come mostrato nella Figura 5.
- Rilassarsi alla struttura utilizzando lo script fornito nel pacchetto oxDNA (vedere l'esempio in $oxDNA/esempi/RELAX_INITIAL_CONFIGURATION per maggiori informazioni).
- Eseguire la simulazione di dinamica molecolare per 10 milioni passaggi utilizzando il file di configurazione rilassato. L'utilizzo è come segue: '. / oxDNA < input >' Salva dati ogni 5000 o 10000 passi.
- Visualizzazione
Nota: Le strutture sono state visualizzate utilizzando cogli.- Scaricare e installare l'ultima versione di cogli (https://sourceforge.net/projects/cogli1/).
- Eseguire la cogli con i file di configurazione e topologia dalla simulazione oxDNA. L'utilizzo è come segue: '. / cogli1 -t < file della topologia >< file di configurazione >'.
- Nascondere la casella premendo b.
3. Sintesi della struttura
Nota: Il metodo di sintesi è adattato dal precedente protocollo15,18.
- Acquistare le graffette di DNA progettate da un provider di oligonucleotidi.
- Regolare la concentrazione di queste graffette di DNA a 100 μM utilizzando acqua priva di nucleasi.
- Piscina ogni filamento di DNA che costituisce una struttura 'stato libero' in una provetta e regolare la concentrazione a 2 μM per ogni filo.
- Piscina del DNA serratura fili di lunghezza e numero di blocco siti nelle provette e regolare la concentrazione a 2 μM per ogni filo. 18, 9 e 4 siti di blocco vengono utilizzati. Aggiungere filamenti di A poli che sono complementari alla regione passerella alla stessa concentrazione.
- Fili di piscina che sono invertire complementare al DNA bloccare fili di lunghezza nelle provette e regolare la concentrazione a 2 μM per ogni filo.
- Preparare MgCl2 soluzione di 300 nM mescolando 70 μL di acqua esente da nucleasi e 30 μL della 1 soluzione μM MgCl2 . Preparare una soluzione di EDTA Tris di 5x con 95 μL di acqua esente da nucleasi e 5 μL della soluzione di EDTA Tris 100 x.
- Aggiungere 2 μL di DNA, 1,1 μL della soluzione di MgCl2 fiocco, 2 μL di soluzione di EDTA Tris, 7,6 μL di acqua esente da nucleasi e 7,3 μL di DNA dell'impalcatura di cui la concentrazione è di 110 nM a fare 20 μL di stock misto. Impostare la concentrazione finale dell'impalcatura del DNA a 40 nM, fiocco del DNA a 200 nM, MgCl2 a 16 mM e la soluzione di EDTA Tris a 0.5 x.
- Rapidamente riscaldare la soluzione stock mista in un termociclatore a 80 ° C e fredda a 60 ° C a una velocità di 4 min per ° C e cool da 60 ° C a 4 ° C a una velocità di 40 min per ° C.
4. purificazione della struttura
Nota: I campioni di tutte le strutture sono stati purificati prima dell'analisi. In questa sezione descriviamo il protocollo di purificazione di PEG, che è adattato da precedente letteratura19. Il campione può essere purificato anche mediante elettroforesi su gel come descritto nella precedente letteratura15,18.
- Preparare 5 M di NaCl e 100 x Tris-EDTA.
- Preparare tampone di precipitazione miscelando 150 μL di PEG 8000, 500 μL di 100 x Tris EDTA e 101 μL di 5 M di NaCl e 249 μL di acqua esente da nucleasi.
- Preparare buffer di destinazione con 5,5 μL di 300 nM MgCl2 soluzione da sezione 3.3, 10 μL di soluzione di EDTA Tris x 5 dalla sezione 3.3 e 84,5 μL di acqua esente da nucleasi.
- Mix 20 μL della struttura sintetizzata dalla sezione 3 e 20 μL di buffer di precipitazione da sezione 4.2. Poi girare lo stock misto a 16000 x g a 4 ° C. Rimuovere il supernatante e dissolva pallina nel buffer di destinazione da sezione 4.3.
5. riconfigurazione del Rack fisarmonica da uno stato di' libero' per un 'stato bloccato'
- Sintetizzare la struttura senza blocchi di DNA per l'esperimento di configurazione.
- Preparare i filamenti di DNA serratura dalla sezione 3.
- Aggiungere 2 μL di filamenti di DNA serratura della lunghezza desiderata in 20 μL della struttura sintetizzata. Concentrazione dei filamenti di DNA serratura è cinque volte maggiore rispetto alla struttura.
- Incubare il campione per 0, 10, 25, 50, o 100 minuti per vedere quanto veloce riconfigurazione si verifica.
- Per l'incubazione di 100 minuti, incubare il campione a 50 ° C per 30 minuti e raffreddare lentamente fino a 25 ° C a una velocità di 0,33 ° C/minuto.
- Per l'incubazione di 50 minuti, incubare il campione a 50 ° C per 15 minuti e raffreddare lentamente fino a 25 ° C a una velocità di 0.66 ° C/minuto.
- Per l'incubazione di 25 minuti, incubare il campione a 50 ° C per 7,5 minuti e raffreddare lentamente fino a 25 ° C a una velocità di 1,32 ° C/minuto.
- Per l'incubazione di 10 minuti, incubare il campione a 50 ° C per 3 minuti e raffreddare lentamente fino a 25 ° C a una velocità di 3,3 ° C/minuto.
- Per l'incubazione di 0 minuti, conservare il campione a 4 ° C a destra dopo l'aggiungono di filamenti di DNA serrature.
- Subito dopo la fase di fissaggio, raffreddare rapidamente il campione a 4 ° C per evitare indesiderata denaturazione.
6. riconfigurazione del Rack fisarmonica da un stato 'bloccato' per uno "stato libero"
- Sintetizzare la struttura con blocchi di DNA della lunghezza desiderata per l'esperimento di configurazione.
- Preparare fili complementari inversione dalla sezione 3.
- Aggiungere 2 μL di fili che sono invertire complementari sui filamenti di blocco della lunghezza desiderata in 20 μL della struttura sintetizzata. Concentrazione dei filamenti di DNA serratura è cinque volte maggiore rispetto alla struttura.
- Incubare il campione per 0, 12, 60, 120, 240 minuti per vedere quanto veloce riconfigurazione si verifica.
- Per le 12, 60, 120, incubazione di 240 minuti, rapidamente riscaldare il campione a 40 ° C e raffreddare lentamente fino a 20 ° C per il tempo corrispondente a ciascuno. Subito dopo la fase di scollegamento, raffreddare rapidamente il campione a 4 ° C per evitare indesiderata denaturazione.
- Per l'incubazione di 0 minuti, conservare campione a 4 ° C a destra dopo invertire fili complementari sono state aggiunte.
7. TEM Imaging
Nota: Protocollo imaging TEM è stato adattato da precedente letteratura18,20.
- Preparare soluzione di NaOH M 1,25 con 87,5 μL di acqua esente da nucleasi e 12,5 μL di soluzione di NaOH 10 M.
- Aggiungere 1 μL di soluzione di NaOH di 1,25 M a 50 μL di soluzione di formiato di uranile al 2%.
- Vortice la soluzione per 3 minuti e centrifugare a massima velocità per 3 minuti. Deposito 3 μL del campione purificato sulla griglia di partenza TEM di Scarica a bagliore per 3 minuti e lavare rapidamente con carta da filtro.
- Deposito 7 μL di soluzione di formiato di uranile preparato per 30 secondi e lavare rapidamente con carta da filtro.
- Misurare la lunghezza e l'angolo della struttura della fisarmonica imaged di TEM.
8. FRET analisi
- Utilizzare Atto 550 e tintura di Atto 647N, per cui la distanza di Förster è 6.5 nm. Sostituire 58 fiocco e fiocco 117 nella tabella 1 con filamenti fluorescente etichettati. Quindi di sintetizzare la struttura con fili fluorescente etichettati secondo il metodo descritto nella sezione 3.
- Misurare la concentrazione del campione purificato.
- Normalizzare il campione a 10 nM e carico 50 μL di 384 micropiastre bene.
- Eccitare il campione con coloranti donatore e accettore a 550 nm e misura dello spettro di fluorescenza da 570 nm a 800 nm con un fluorimetro.
- Misurare lo spettro di fluorescenza del campione solo donatore nello stesso modo.
- Eccitare le tinture del campione a 650 nm e lo spettro di fluorescenza e misura da 670 nm a 800 nm. Si tratta di misurare la concentrazione di acceptor.
- Ottenere le deviazioni standard ripetendo lo stesso esperimento con tre campioni, che vengono sintetizzati e purificati separatamente.
- Calcolare l'efficienza FRET con il rapporto di un metodo come descritto dall'equazione sotto21.
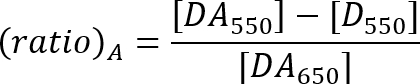
DA550: intensità di fluorescenza di picco accettore del campione con donatore e accettore a eccitazione di 550 nm.
D550: intensità di fluorescenza nell'intervallo di emissione accettore del campione solo donatore a eccitazione di 550 nm.
DA650: intensità di fluorescenza di picco accettore del campione con donatore e accettore a eccitazione di 650 nm.
Access restricted. Please log in or start a trial to view this content.
Risultati
Il rack di fisarmonica del DNA 6 progettato per 6 è simulato dal16,oxDNA17 e i risultati sono mostrati in Figura 6. Dal risultato della simulazione, è stato confermato che la struttura desiderata è costituita senza distorsione della struttura.
Le immagini TEM nella Figura 7 sono immagini di strutture configur...
Access restricted. Please log in or start a trial to view this content.
Discussione
Questo protocollo introduce l'intero processo dalla progettazione, simulazione, sintesi e analisi della cremagliera della fisarmonica DNA 2D base. Le regole di simulazione e Progettazione modificata sono stati descritti perché la regola di progettazione è diverso da quello di origami di DNA standard, in quanto il rack di fisarmonica di DNA ha nucleotidi supplementari presso i crossover per flessibilità14,15. Da questo, ci aspettiamo che il protocollo può acce...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Gli autori non hanno nulla a rivelare
Riconoscimenti
Questa ricerca è stata parzialmente sostenuta dal programma internazionale del centro di sviluppo ricerca attraverso National Research Foundation di Korea(NRF) finanziato dal Ministero della scienza e ICT (MSIT) (2015K1A4A3047345) e Nano· Programma di sviluppo di tecnologia materiale attraverso la National Research Foundation di Corea (NRF) finanziato dal Ministero della scienza e ICT (MSIT) (2012M3A7A9671610). L'Istituto di ricerca ingegneria all'Università nazionale di Seoul fornito di strutture di ricerca per questo lavoro. Autori riconoscono gratitudine verso Yoon Tae-Young (scienze biologiche, Università nazionale di Seul) per quanto riguarda la spettroscopia di fluorescenza per l'analisi FRET.
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| M13mp18 Single-stranded DNA | NEB | N4040s | |
| 1M MgCl2 Solution | Biosesang | M2001 | |
| Tris-EDTA buffer | Biosesang | T2142 | |
| Nuclease-Free Water | Qiagen | 129114 | |
| 5M Sodium Chloride solution | Biosesang | s2007 | |
| PEG 8000 | Sigma Aldrich | 1546605 | |
| 10N NaOH | Biosesang | S2038 | |
| Uranyl formate | Thomas Science | C993L42 | |
| Thermal cycler C1000 | Biorad | ||
| Nanodropic 2000 | Thermo Fisher Scientific | ||
| TEM (LIBRA 120) | Carl Zeiss | ||
| Fluorometer Enspire 2300 | Perkin-Elmer | ||
| Centrifuge | Labogene | LZ-1580 |
Riferimenti
- Andersen, E. S., et al. Self-assembly of a nanoscale DNA box with a controllable lid. Nature. 459 (7243), 73-76 (2009).
- Cha, T. -G., et al. Design principles of DNA enzyme based walkers: Translocation kinetics and photo-regulation. Journal of the American Chemical Society. 137 (29), 9429-9437 (2015).
- Gerling, T., Wagenbauer, K. F., Neuner, A. M., Dietz, H. Dynamic DNA devices and assemblies formed by shape-complementary, non-base pairing 3D components. Science. 347 (6229), 1446-1452 (2015).
- Pinheiro, A. V., Han, D., Shih, W. M., Yan, H. Challenges and opportunities for structural DNA nanotechnology. Nature nanotechnology. 6 (12), 763-772 (2011).
- Li, J., et al. Exploring the speed limit of toehold exchange with a cartwheeling DNA acrobat. Nature Nanotechnology. 1, (2018).
- Krishnan, Y., Simmel, F. C. Nucleic acid based molecular devices. Angewandte Chemie International Edition. 50 (14), 3124-3156 (2011).
- Liu, M., et al. A DNA tweezer-actuated enzyme nanoreactor. Nature communications. 4, 2127(2013).
- Douglas, S. M., Bachelet, I., Church, G. M. A logic-gated nanorobot for targeted transport of molecular payloads. Science. 335 (6070), New York, N.Y. 831-834 (2012).
- Kuzyk, A., et al. Reconfigurable 3D plasmonic metamolecules. Nature Materials. 13 (9), 862-866 (2014).
- Zhou, C., Duan, X., Liu, N. A plasmonic nanorod that walks on DNA origami. Nature communications. 6, 8102(2015).
- Choi, Y., Choi, H., Lee, A. C., Lee, H., Kwon, S. A Reconfigurable DNA Accordion Rack. Angewandte Chemie International Edition. 57 (11), 2811-2815 (2018).
- Chen, H., et al. Understanding the Mechanical Properties of DNA Origami Tiles and Controlling the Kinetics of their Folding and Unfolding Reconfiguration. Journal of the American Chemical Society. 136 (19), 6995-7005 (2014).
- Han, D., Pal, S., Liu, Y., Yan, H. Folding and cutting DNA into reconfigurable topological nanostructures. Nature Nanotechnology. 5 (10), 712-717 (2010).
- Douglas, S. M., et al. Rapid prototyping of 3D DNA-origami shapes with caDNAno. Nucleic Acids Research. 37 (15), 5001-5006 (2009).
- Castro, C. E., et al. A primer to scaffolded DNA origami. Nature methods. 8 (3), 221-229 (2011).
- Ouldridge, T. E., Louis, A. A., Doye, J. P. K. DNA Nanotweezers Studied with a Coarse-Grained Model of DNA. Physical Review Letters. 104 (17), 178101(2010).
- Snodin, B. E. K., et al. Direct Simulation of the Self-Assembly of a Small DNA Origami. ACS Nano. 10 (2), 1724-1737 (2016).
- Amir, Y., Abu-Horowitz, A., Bachelet, I. Folding and Characterization of a Bio-responsive Robot from DNA Origami. Journal of Visualized Experiments. (106), e51272(2015).
- Stahl, E., Martin, T. G., Praetorius, F., Dietz, H. Facile and Scalable Preparation of Pure and Dense DNA Origami Solutions. Angewandte Chemie International Edition. 53 (47), 12735-12740 (2014).
- Wei, B., Vhudzijena, M. K., Robaszewski, J., Yin, P. Self-assembly of Complex Two-dimensional Shapes from Single-stranded DNA Tiles. Journal of Visualized Experiments. (99), e52486(2015).
- Clegg, R. M. Fluorescence resonance energy transfer and nucleic acids. Methods in enzymology. 211, 353-388 (1992).
- Kopperger, E., et al. A self-assembled nanoscale robotic arm controlled by electric fields. Science. 359 (6373), New York, N.Y. 296-301 (2018).
- Lauback, S., et al. Real-time magnetic actuation of DNA nanodevices via modular integration with stiff micro-levers. Nature Communications. 9 (1), 1446(2018).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon