Method Article
利用自动高通量微流控系统对铜绿假单胞菌剪切应力的活细胞分析
* 这些作者具有相同的贡献
摘要
在这里, 我们描述了使用高通量微流体生物反应器结合荧光显微镜来分析剪切应力对表达绿色荧光蛋白的铜绿假单胞菌生物膜的剪切应力影响, 包括仪器建立了生物膜覆盖、生长速率和形态特性的测定。
摘要
采用高通量的体外生物反应器与荧光显微镜结合, 研究了细菌生物膜的生长和形态, 包括铜绿假单胞菌 (铜绿假单胞菌)。在这里, 我们将描述如何利用该系统来研究生长动力学和形态性质, 如表面粗糙度和纹理熵的铜绿假单胞菌菌株 pa01, 表达了增强的绿色荧光蛋白 (pa1-egfp).详细的协议将描述如何生长和种子 pa01-egfp 培养物, 如何设置显微镜和自动运行, 并进行图像分析, 以确定生长速度和形态特性使用各种剪切力控制的微流体装置。本文将提供一个技术的详细说明, 以改进的研究 pa01-egfp 生物膜, 最终可以应用于其他菌株的细菌, 真菌, 或藻类生物膜使用微流体平台。
引言
在这里, 我们将演示一种方法, 以测量剪切应力的影响, 形成荧光假单胞菌铜绿假单胞菌 (铜绿假单胞菌) pa01 生物膜使用自动高吞吐量微流体系统。
生物膜是微生物的群落, 如细菌, 由附着在支撑物上的细胞外聚合物物质组织, 通常存在于液体和固体表面1之间的界面上。这些生物膜群落可以对环境有益, 例如改善供水线路的水质, 以及对顽抗化合物的生物修复 2、3.然而, 生物膜也可能对人类健康造成极大的危害, 并产生不良后果。例如, 医疗设备, 如髋关节和膝关节植入物, 是一种表面, 其中生物膜的积累一直是一个挑战, 并导致严重的医疗并发症4,5。生物膜还可以进入河流和湖泊等天然供水系统, 并渗透供水管道, 导致饮用水中的细菌污染,导致 6、7、8感染。海洋环境中形成的生物膜粘附在船舶和其他人造基板上, 并带来重大的经济和环境问题, 因为摩擦的增加导致燃料消耗增加9、10.抗菌涂料, 如三丁基锡, 已经开发, 以防止这些问题, 但对海洋生物有毒 11.
铜绿假单胞菌是一种革兰氏阴性细菌, 在各种环境和营养条件下具有较高的生长能力。铜绿假单胞菌是社区和医院感染的常见原因, 被发现与严重烧伤等损伤和免疫功能低下的宿主 (如囊性纤维化 (cystic)5,12, 13、艾滋病和癌症患者5,13。铜绿假单胞菌的形成与 cf 联系最为密切, 慢性肺部感染是本病死亡的主要原因5。
本报告中使用了铜绿假单胞菌的参考菌株 pa01, 并进行了转基因, 以表达增强的绿色荧光蛋白 (pa-egfp)。egfp 代表了一种具有更大荧光特性的 gfp 突变形式, 允许使用荧光显微镜14、15、16进行原位生物膜分析。这种类型的荧光分析有利于生物膜的研究, 因为 gfp 不会显著干扰细胞生长和功能17。例如, 与对照细菌17相比, 贴有 gfp 标记的大肠杆菌细胞生长良好, 持续没有受到任何毒性影响.其他报告证实了这一说法 18、19、20。此外, 使用荧光记者, 如 egfp 是快速和简单的, 但只有活细胞将被测量, 因为死细胞很快停止荧光 21.
生物膜可以在各种环境条件下生长, 包括流量不同的环境条件下。例如, 薄膜可以在高剪切应力下生长, 例如在河流中, 高水流条件会导致更大的微生物多样性 22。相反, 池塘或口腔生物膜中的积水的剪切力要低得多。除了流速外, 还有其他影响生物膜粘附的因素, 包括表面粗糙度和疏水性、培养基成分, 甚至细菌细胞表面1,4, 7, 24. 条件也可能引起生物膜空间结构或形态的变化。这包括环境条件, 如运动流体或养分供应梯度所施加的剪切应力, 以及系统中存在的物种、细胞的运动和细胞外存在的特定蛋白质等生物因素聚合物物质 25,26,27。在某些条件下, 生物膜将像草坪一样 (光滑和平坦), 而在其他条件下, 生物膜将是粗糙的, 蓬松的,甚至像蘑菇一样28。虽然生物膜草坪和蘑菇结构之间的质的差异可以从微观图像中清楚地看到, 但要了解薄膜结构与薄膜内生物过程之间的关系, 需要系统和定量描述形态的方法。研究人员建议研究的形态特征包括孔隙度、分形维数、扩散长度、底层微菌落面积、微菌落体积、粗糙度系数和纹理熵29,30.
生物反应器被用于生物膜的研究, 以模拟现实生活条件31。滴流反应器 (dfr) 代表了低剪切环境, 在这种环境中, 介质中的营养物质会缓慢地流经随着时间的推移附着在表面的细胞, 形成具有高细胞密度32的生物膜。cdc 反应器是生物反应器, 通过控制在介质填充罐33内连续旋转的搅拌杆,从而创造高剪切应力流体环境。这类生物反应器设置起来很简单, 但由于样品尺寸相对较低, 介质消费量高, 介质滴流产生的生物危险废物大量, 从125μlp 分钟不等, 用于滴流反应器, 因此范围有限cdc 反应堆的使用量超过 1 mL/min, 需要对大量玻璃器皿和废介质进行高压灭菌 34.在滴水流反应器中, 生物膜在表面的生长并不均匀, 因为介质的低剪切导致沿着铜绿假细菌较大的团群跟踪, 因此生物膜生长不是很光滑, 不均匀的样品也不可能是用荧光显微镜轻松分析 35,36 。
通过使用中等吞吐量的微流体生物反应器, 只需要毫升的介质, 反应板很小, 在高压灭菌37后容易一次性使用, 从而克服了一些常见的生物反应器限制。此外, 根据井数的不同, 许多复制只需在一个反应堆运行中就能进行, 这就提供了足够的数据, 可以进行有意义的统计分析。在图 1中, 显示了允许控制条件的微流体显微镜系统的不同组件, 包括温度和流速38、39、40。生物反应器与荧光显微镜结合, 可在低剪切条件下在应用的低剪切条件下显示 pa01 中 egfp 标记的荧光, 这将模拟在环境或生物医学领域遇到的更现实的场景。
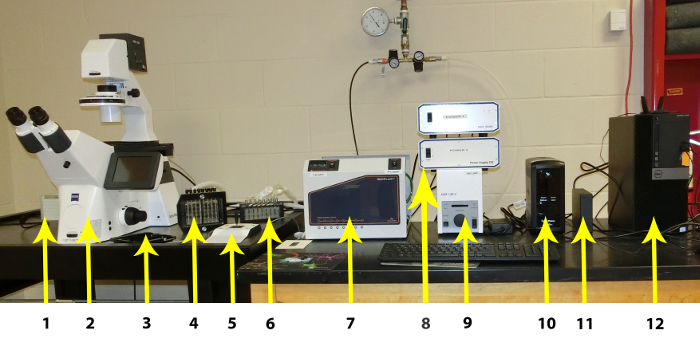
图 1: 微流体系统的各个组成部分.各个组件从左到右列出: 1. ccd 相机, 2. 带自动舞台的高分辨率倒置显微镜, 自动荧光模块和自动对焦模块, 3. 平板舞台, 4: 成像系统接口, 5: 手动显微镜级控制, 6:vapor 陷阱, 7: 成像系统控制器 (包括温度控制器), 8: 硬件控制器, 9: 荧光控制器, 10: 不间断电源, 11: 用于图像存储的外部硬盘驱动器, 12:pc 工作站。请点击这里查看此图的较大版本.
微流体板的节选如图 2所示。最常用的板块由48口井组成。一个实验需要一个入口和一个出口井, 总共2口井。这允许在各种实验条件下同时进行24个实验, 例如细菌菌株、抗菌处理和介质, 这些条件因渠道而异, 6个通道的每一列都有控制剪切流。实验温度也通过在整个板材中设定一个温度来控制。微流体通道显示, 每个通道都有一个蛇形区域, 以提供足够的背压和可控剪切。
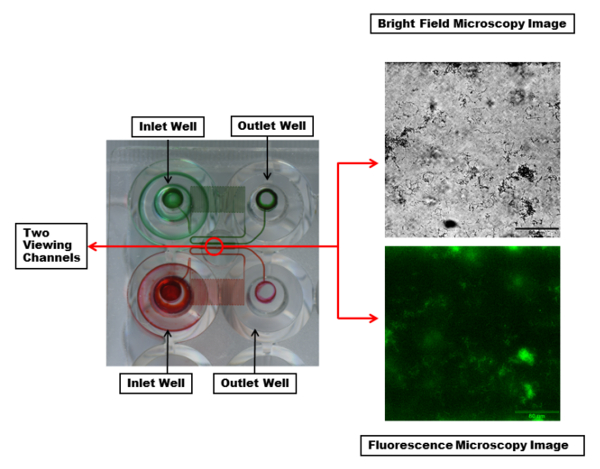
图 2:微流体通道和查看窗口的可视化.两个入口和出口井与连接它们的微流体通道突出了红色和绿色染料。染料使每个通道可见蛇形区域, 在流体流动过程中产生足够的背压和可控剪切。每个观察通道 (在红色圆圈内) 都可以用所需的波长进行成像。显示的是明亮的领域 (顶部) 和荧光 (底部) 显微镜图像的单个通道与 pa01-egfp 生物膜使用20x 的目标。刻度栏 = 80μm. 请点击这里查看此图的较大版本.
提供了一个一步一步的指南, 使使用与荧光显微镜耦合的微流体生物反应器的用户能够使用不同的剪切环境进行新的生物膜实验。这种方法将允许扩大涉及细菌以外的其他微生物的实验, 如真菌和藻类, 这些微生物具有医疗和环境应用 41,42,43。详细介绍了如何培养 pa01-egfp, 接种48孔板, 建立微流体器件和软件, 设置荧光显微镜, 并演示软件分析, 以获得生物膜的覆盖范围, 生长速度,形态特性, 如表面粗糙度。
研究方案
1. 媒体准备
- 准备最小介质 (mm) 与0.25% 葡萄糖。使 mm 的1升与0.25% 的葡萄糖, 加入200毫升的无菌 mL 盐溶液, 2 毫升的无菌 1 m mgso4, 100μl 的无菌 1 m ccl 2, 12.5 ml 的无菌 20% (w/v) 葡萄糖在水中 (dh 2o) 在最后体积为 1 l.
- 使用无菌技术将所需介质转移到无菌瓶中。根据所使用的通道数准备所需的金额。通常情况下, 每个通道的启动需要 200μl, 播种需要 300μl, 24小时实验需要1300μl。
- 将瓶子放在实验温度下的孵化器或水浴中。介质在使用前应处于实验温度, 以避免微通道中形成气泡。
2. pa01-egfp 的隔夜和实验培养的准备。
- 将15毫升的实验介质放入无菌侧臂瓶中, 并从带有 pa01-egfp 的琼脂板上接种一个或两个菌落。在37°c 和180-220 转/半表的孵化器振动台上, 将培养时间扩大12-16小时。
- 测量隔夜文化的 od600 。当 od600大于0.80 时, 使用新鲜的 mm 将隔夜培养稀释到0.8 的最终 od. 将实验培养物置于孵化器或37°c 的水浴中, 直到需要播种微流体通道。在播种前立即再次检查 od600 , 以确保它与目标 od600、0.8 在这种情况下没有明显变化。
3. 设备启动
- 根据用户指南设置系统工作站, 类似于图 1中的设置。为避免仪器连接到软件时出现错误, 请按以下顺序打开仪器:
电脑工作站
荧光模块。确保荧光快门处于打开状态 (通过打开快门按钮的蓝光)
硬件控制器
成像系统控制器 (见材料表)
ccd 摄像机
成像站 (显微镜)
注: 加热板的温度应调整到所需的实验温度。 - 启动控制应用程序, 并输入位于印版侧面标签上的车牌号。
注: 控制应用程序启动后, 将有两个单独的应用程序窗口, 一个用于控制显微镜/成像软件的软件, 另一个用于控制泵和井板接口的控制模块。如图 s1所示, 其中顶部打开了 "多维采集" 菜单, 并在底部为自动运行设置了控制模块。
4. 微流控板的启动和播种
注: 启动、播种、细菌附着和生长如图 3所示。
- 从包装中取出48口的微流体板, 确保不要接触板底的玻璃表面。用镜片组织、无皮棉或低皮棉擦拭将盘子底部的玻璃滑块清洁干净。
- 为了给微流体通道提供优质的输送器 200μl, 将 37°c mm 的移液器放入输出井中, 小心避免气泡。将板材放入板材舞台, 用乙醇擦拭界面, 使其干燥, 然后将其密封到板材舞台上。
- 在控制模块上的手动模式下, 在37°c 时将"流体" 设置为 lb, 在 5.00 dynecm2时将最大剪切设置为 lb。单击输出井以激活从输出到输入井的流量, 启动通道。5分钟启动后, 暂停流动, 为播种做准备。小心地从舞台上取出板, 从输出中取出移液器残留介质, 但不要从内圈中取出任何导致微流体通道的介质。
- 为了播种实验通道, 首先将 mm 的液器300μl 放入输入井中, 然后将细菌培养的300μl 移入输出井中。将板材放回板材舞台, 确保在将其放置在板材上之前擦拭界面。
- 在控制模块上, 将板舞台放置在显微镜舞台上后, 使用实时相机进料将焦点放在单个通道上。在现场直播的视觉监测中, 恢复流在 1.00-2.00 dynecm 2大约 2-4 s, 以允许细胞进入实验通道, 但不在蛇形通道。将板材放置在温度控制阶段 1小时, 以便进行细胞连接。在1小时的孵化过程中, 可以为自动运行设置控制模块和蒙太奇软件 (步骤 5)。
注: 播种所需的时间因介质和生物而异, 因此应由现场饲料密切监测, 直至优化并用作一般时间框架。整个板块的播种可能会有所不同, 这将需要更多的时间应用流到某些通道柱完成播种。 - 在附着期结束后, 轻轻地将板材从舞台上取出, 然后将细菌从输出中移出, 首先避免干扰通道。使用新的移液器尖端, 从输入井中取出介质。
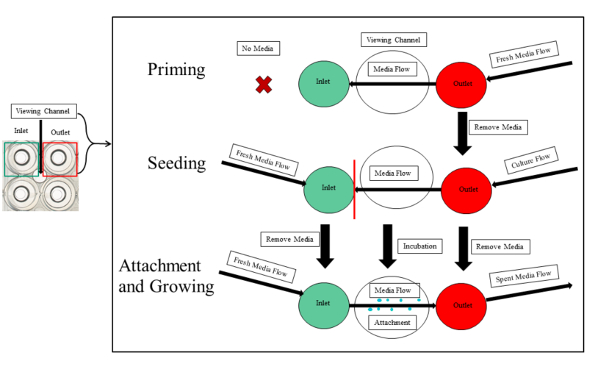
图 3: 在微流体通道中进行 pa01-egfp 的启动、播种和附着的实验综述.概述了启动、播种和附件。启动的第一步需要新的媒体引入到输出。播种分别要求输入井和输产量井中的培养基和细菌培养量相等。培养不应通过实验通道 (红线) 的观察段, 以避免堵塞蛇形通道。潜伏期结束后, 新鲜介质不断地从输入流到观察室, 并流入插座。这就引发了细菌生物膜的附着和生长。请点击这里查看此图的较大版本.
5. 设置软件
- 在软件中, 打开 "多维度采集" 来控制显微镜图像的采集。在 "主" 菜单下, 选择 "时间间隔"、"多个阶段位置" 和 "多个波长" (图 s1a)。
- 设置"保存设置", 创建一个简单的 "基本名称", 确保选中了"增量" 基本名称 (如果存在文件) 。单击 "选择目录"以选择将在其中保存所有文件的文件夹。在"说明" 中包括实验的基本详细信息。
- 在 "时间间隔" 选项卡下, 将实验时间的持续时间调整为24小时。若要控制获取图像的频率, 请设置"时间间隔" , 以便在整个实验过程中每5分钟获取一次图像。"时间点数" 会自动使用设置的参数进行调整。
- 在明亮的现场显微镜下使用实时相机进料设置舞台位置, 以便正确放置和对焦。从10倍目标开始, 将重点放在通道的中心, 位于刻在板材上的通道号的上方或下方。切换到20x 目标, 在通道内找到最佳的观看区域和焦平面。将位置添加到列表中, 或将预设通道位置替换为新设置。
- 在"波长" 菜单下, 将波长数设置为3。对于波长 1 (w1), 选择具有10毫秒曝光时间的 fitc 100% cam。对于波长 2 (w2), 选择亮度场 50% cam 50% vis, 最小曝光时间为3毫秒. 对于波长 3 (w3), 设置为"全闭合" 滤波器, 以便采集时间之间的最后通道上不保留光线。
- 设置控制模块时, 请检查以确保手动运行已停止。
- 在 "协议设置" 选项卡下的"编辑自动运行"菜单中, 为24小时的持续时间设置一个新协议, 并以所需的剪切速率向前移动。为了研究生物膜生长对剪切速率的影响, 剪切速率是不同的。单击 "添加"和"另存为协议"。
- 在 "序列设置" 选项卡下, 通过选择LB@37degrees作为所有通道的默认流体来创建新序列。在步骤迭代 1下, 对于通道 1-12, 选择具有所需剪切速率的协议并启用所有通道。对于通道 13-24, 选择具有所需的第二个剪切速率的协议, 并启用所有通道。选择"应用"和"另存为序列" 。
- 在 "自动运行"菜单中, 选择要用于自动运行的已保存序列。
6. 定时生物膜生长实验设置
- 将最大13300μl 的无菌 mm 输送到微流体板的输入井中。将板材放回板材舞台上, 用乙醇擦拭界面, 使乙醇完全干燥, 然后密封板材。
- 将板级放置在显微镜舞台上, 并确保正确设置协议和顺序。选择"开始"可快速启动自动运行, 然后单击 "获取"以启动显微镜图像收集。
- 调整图像获取之间所有舞台位置的焦点和位置。选择"暂停",然后使用明亮字段波长下的实时图像模式, 选择 "转到" 以查看每个舞台位置。通过选择"设置为当前" 设置新设置。单击 "在下一个计划的购置时间之前继续" .
7. 时间生物膜生长实验后图像序列的回顾与分析
- 若要查看24小时自动运行后的每个通道, 请在软件中打开"查看多维数据", 位于"分析工具" 下。点击选择基本文件选择 "目录"以导航到具有图像序列的文件夹, 以便进行感兴趣的实验。在出现的文件夹中的"数据集" 列表中, 选择感兴趣数据的基本名称。
- 选择感兴趣的数据后, 单击 "查看" 查看此数据。选择一个波长以查看"波长"窗口下的数据 (图 s2)。
- 通过选择舞台位置下的通道号并使用视频控件分析289个时间点的数据 (假设图像在24小时内每5分钟获得一次), 查看每个通道的图像序列。注意可用的增长数据。
注: 在每个通道中, 观察气泡和堵塞的发展, 这些气泡和木块会干扰通道内的生物膜生长, 从而影响数据。但是, 如果这些情况发生在以后的运行中, 则可能会发现这些情况之前的数据很有用。 - 在查看并选择要分析的所需数据后, 为每个通道创建图像堆栈。在 "文件"菜单下, 选择 "打开特殊状态"构建堆栈编号名称。在 "生成堆栈"窗口中, 选择"选择第一个图像" 按钮, 然后选择文件夹中堆栈的第一个图像;选择 "选择最后一个图像" 按钮, 然后选择与堆栈中最后一个图像相对应的文件。单击"确定" , 按时间顺序打开包含通道所有图像的堆栈。堆栈可以保存在"文件"菜单下, 另存为 tiff 文件。
注: 不要使用拇指图像创建堆栈。这些文件不是很有用, 可以删除, 以节省硬盘驱动器上的空间。此外, 图像文件保存为以下内容:
基本名称 _ wavelength _ sCHANNEL # _ timage #。
例如, 07062018_W1FITC 100_s12_t112 是通道 12, 时间点 112, 图像在第一个波长 fitc 100% 相机数据的基本名称为 "07062018"。 - 最后, 从"文件"下拉菜单中选择"另存为", 将序列另存为 tiff 堆栈。堆栈也可以叠加并保存为电影。步骤9对此进行了讨论。
- 在量化% 的表面覆盖范围之前, 请校准图像距离。在"分析工具"下, 单击 "校准距离".选择适当的校准测量, 然后单击 "应用于所有打开的图像"。
- 当堆栈位于序列的第一个图像上时, 单击"阈值" 按钮。使用光对象的 "自动阈值" 来确定荧光信号的阈值 (fitc 波长),使用 "暗对象的自动阈值" 来确定明亮字段中的阈值。将阈值调整为表示图像覆盖范围的覆盖范围。
- 对于荧光阈值, 使用带有非荧光单元的通道建立任何背景信号, 并设置最小阈值, 以排除在含有这些非荧光细胞的通道中检测到的信号, 从而确保信号测量不高估荧光面积。这可能与运行运行不同, 因此最好至少使用一个 (如果不是更多) 通道来进行非荧光控件。
- 对于明亮的字段阈值, 如果橙色阈值签名不涵盖所有单元格, 请通过滑动工具栏或使用阈值图像(在分析工具下找到) 调整最大阈值, 以便所有单元格都是覆盖, 但任何背景都被排除在外。
注: 虽然理想情况下, 这些阈值将用于堆栈中的所有图像和所有通道, 但为一个图像/通道选择的阈值可能不适用于其他图像或通道, 因此用户可能需要调整阈值范围定期。因此, 如果以后需要查看数据, 最好始终记录所使用的最大和最小阈值。
- 若要量化覆盖范围, 请在"分析工具"下, 单击 "显示区域统计信息"。确保选中了 "使用阈值" , 选择了 "整个图像", 并在"获取数据"下, 确保选择了所需的测量设置 (阈值面积、平均值、标准偏差、最小值、最大值和% 阈值)区)。单击 "打开日志" , 并确保在单击"确定"之前选择了 dde 文件。选择"microsoft excel" , 然后单击 "确定"。在将打开的电子表格中, 单击 "日志数据"以自动记录所分析图像的测量值。
- 保持此电子表格处于打开状态, 请使用相同的工作表收集单个堆栈的所有图像分析数据。在堆栈中, 转到第四个图像和阈值以获得最佳设置。单击 "显示区域统计信息" 屏幕中的 "日志数据" , 并将这些值记录到电子表格中。每15分钟重复分析一次图像。
8. 其他分析, 包括使用生物膜形态套件中的 python 脚本进行形态和表面覆盖测量
- 安装 python 3.6 的分布, 其中包括标准的科学模块, 通过使用标准的科学 python 分布, 如 anaconda, 可在 https://www.anaconda.com/download。
- 通过导航到 https://github.com/cdwentworth/Biofilm-Morphology-Suite.git, 从 github 中的 github 获取 biofilm 形态套件, 然后选择 "克隆" 或"下载" (绿色按钮), 然后选择 "下载 zip"。解压缩文件并将代码文件夹移动到工作目录。
- 通过将图像堆栈复制到 "覆盖范围"文件夹来测量覆盖率百分比。使用终端窗口导航到该文件夹, 然后使用命令执行脚本
python bf封面. py tiffstackname. tif
从命令行界面,其中 tiffstackname. tif 是包含图像的 tiff 堆栈的文件的名称。将创建一个包含每个时间点覆盖测量值的文本文件, 名称为 tiffstackname. txt, 并将创建一个图形文件, 其中包含覆盖图作为时间的函数. - 使用步骤8.3 中指定的相同过程来测量其他形态特征: 生物膜累积、粗糙度系数和纹理熵。使用 bfacc. py 脚本进行累积测量, 使用粗 coef. py 脚本进行粗糙度系数测量, tevstime. py 脚本用于纹理熵测量。
9. 其他软件应用程序-制作叠加和电影
- 如果使用多个波长, 则创建包括这些波长在内的叠加堆栈。打开所有感兴趣的堆栈, 并按照步骤7.6 中的步骤校准距离。
- 在"分析工具"下, 选择 "叠加图像"。将源设置为感兴趣的堆栈。在 fitc 堆栈中, 单击彩虹圆圈并选择要添加的绿色滤光片, 以突出显示此波长。
- 使用bal调整叠加, 以便 fitc 堆栈在图像中可见。进行所有调整后, 选择两个堆栈的"所有平面" , 然后单击 "应用"。以与步骤7.5 中描述的堆栈相同的方式保存叠加, 或保存步骤9.2 中描述的影片。
- 要将堆栈或叠加保存为时间流逝的影片, 请在"mm 标准" 和"堆栈" 下, 单击 "制作影片"。选择源作为所需的堆栈或覆盖。单击"保存"并将其另存为质量设置在70-80 之间的microsoft 视频 1 。
结果
图 4a显示了在0.2、0.5 和 1.0 dyne/cm 2 的剪切流中, 24小时运行时的阈值面积随时间的推移所占的百分比。所有三种剪切设置的生物膜覆盖范围或阈值表面积百分比 (c [%]) 都不同。生物膜覆盖最快, 在 1.0 dyne/cm2剪切, 其中阈值面积从2-5 增加到 100%, 在200分钟的生长和达到一个固定阶段后, 400分钟。在 0.5 dyne cm2 时, 生物膜覆盖延迟, 800分钟后开始增加, 400分钟达到100% 的覆盖率。在 0.2 dyne/cm 2 的最低剪切力清楚地显示了生物膜覆盖最慢的增加, 其中百分比覆盖率开始增加在 500分钟, 但从未达到65% 的阈值范围。这些结果表明, 剪切对生物膜表面覆盖有直接影响。较高的剪切力似乎是生物膜生长的一个更理想的条件, 可能是由于培养基为细菌提供了更多的营养物质, 使生物膜能够更快地增殖。
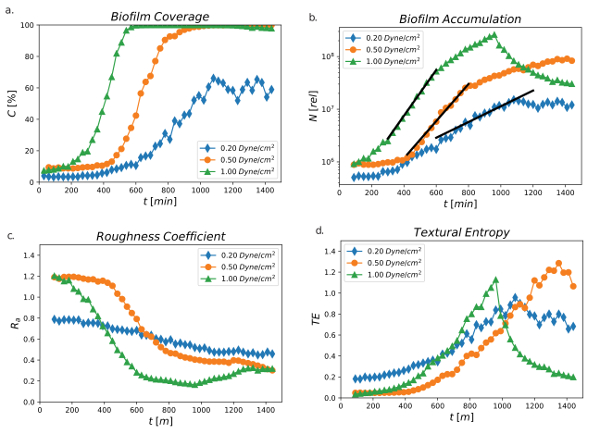
图 4:百分比阈值面积、生物膜积累总量、粗糙度系数和结构熵. a) 在一段时间内使用0.2、0.5 和1.0 动态2的百分比阈值面积 (c [%]), 在24小时内使用。每个剪切条件的数据都是从一个通道获得的。(b) 在24小时内使用0.2、0.5 和 1.0 dyne/cm 2 的时间函数, 生物膜总积累 (相对测量 )。黑线是最小的正方形, 适合指数模型。每个剪切条件的数据都是从一个通道获得的。(c) pa01-egfp 的粗糙度系数, 使用0.2、0.5 和 1.0 dyne/cm 2 的剪切应力值监测超过 24小时, 每个剪切条件从一个通道获得数据。d) 使用0.2、0.5 和 1.0 dynecm 2 的剪切应力值的 pa01-egfp纹理熵.每个剪切条件的数据来自一个通道 (valquier-fleynn, h., sutlief, a. l., wentworth, c. d., 2018)。请点击这里查看此图的较大版本.
图 4b与图 4a所示的结果一致。生物膜积累 (n[rel]) 是基于这样的假设测量的, 即图像中某一点的 gfp 信号与该位置的活细胞密度成正比。结果表明, 在24小时内, 使用0.2、0.5 和 1.0 dyne/cm 2 的总相对测量生物膜积累随着时间的增加而增加, 生长速率从最高剪切应力下降到最低剪切应力。有一个明确的时间段, 给指数增长, 从这个周期可以计算出定量增长率。
图 4c显示剪切流为0.2、0.5和 1.0 dynecm 2 时的粗糙度系数 (r a).表面粗糙度, 用粗糙度系数来量化, 测量薄膜厚度分布的方差。正式的定义是
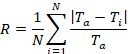
其中t i 是i-厚度测量, ta 是平均厚度, n是厚度测量的数量30。本调查中描述的程序测量与活细胞相关的厚度。单元的平面排列将产生零粗糙度系数, 而厚度与平均值的显著变化将产生大于1的粗糙度系数。与剪切对薄膜生长速度和阈值覆盖率的影响相似, 生物膜随着时间的推移表现出不同的地形。总体而言, 在所有剪切条件下, ra 会随着时间的推移而减小, 这表明所有表面变得更平滑。然而, 与 0.2 dynecm 的最低剪切相比, 0.5 和 1.0 dynecm 2 的较高剪切设置会导致表面随着时间的推移而平滑, 这表明更快的剪切流有助于使表面更平滑、更均匀, 最高的剪切力为1.0 dyne cm 2 到达 0.2 ra 以下.
表面的平滑度、规律性或粗糙度也可以用纹理熵 (te) 表示。te 是用于图像分析的一种属性, 用于测量二维图像中的随机性程度。它的计算基于由 haralick等人定义的灰度共现矩阵, 该矩阵查看一个位置的像素值是否与另一个位置44 上的像素值相关。高度的相关性会导致低熵。图 4d描述了在剪切流为0.2、0.5 和 1.0 dyne/cm2时的 te。所有剪切条件下的 te 随时间而增加, 但 1.0 dyne\ cm 的最大剪切应力比900分钟的较低剪切应力提前到最大 te (1.0)。最低剪切量 0.2 dynemcm2在1000分钟后, te (0.8) 最低达到最大值。然而, 0.5 dyne/cm2的中间剪切应力达到了其最大 te (1.2), 远远晚于高或低剪切应力条件。
粗糙度系数和 te 测量不同的特征。虽然平面薄膜具有较低的粗糙度系数和较低的熵, 但厚度差异较大的薄膜具有较高的粗糙度系数, 但如果这种变化是正弦而不是随机的, 则可能仍然是低熵。在这种情况下, ra随剪切应力和时间的增加而减小, 而 te 趋势与随着时间的推移应用于生物膜形成的剪切应力不能直接相关。
图 s1: 控制软件窗口的图像捕获.打开 "多维获取" 菜单的软件窗口 (顶部)。自动运行 (底部) 的控制模块集。请点击这里查看此图的较大版本.
图 s2用于查看数据的软件的图像捕获.使用"分析工具"菜单中的"查看多维数据" 工具选择感兴趣的数据集后的应用程序窗口。请点击这里查看此图的较大版本.
讨论
这里讨论的微流体系统和图像分析程序的重点是执行微流体生物膜实验, 以确定不需要通常从共聚焦中找到的完整三维信息的形态特性显微镜研究。其中包括微群体底层覆盖 (百分比覆盖率)、表面粗糙度测量的粗糙度系数和纹理熵。提出了一种估计总相对生物膜细胞积累的方法, 并从中计算出测井阶段的生长速率。
在此方法中, 应突出显示几个小但重要的步骤。在从实验到实验的同时, 也避免了从实验到实验的其他细菌的污染。启动和播种也非常重要, 因为启动可以让用户确定哪些通道允许媒体在没有任何干扰或堵塞的情况下通过通道。通道在启动后也不应受到干扰 (即它们应不断充满媒体), 以增加在没有气泡或堵塞的情况下成功实验的机会。播种步骤可以根据细菌的类型而改变, 因此应该针对细胞附着进行优化。例如, 如果细胞似乎没有附着, 在播种之前, 可能必须在微流体板上进行表面修改, 或者可能需要更长的孵育时间。同样重要的是, 要确保正确设置显微镜以获得聚焦图像, 并在整个实验过程中定期进行监测, 以确保获得高质量的图像。如果焦点关闭, 显微镜可以也应该随着实验的继续而调整。在图像采集期间, 应将最后一个波长设置为"全部关闭",以避免在图像采集之间的等待时间内, 筛选器和照明仅暴露给一个通道。此外, 确定% 表面覆盖率的图像分析是在内部设计的, 因为蒙太奇软件手册没有明确描述该过程。此外, 为了扩展图像分析并确定表面粗糙度等其他特性, 在内部开发了基于Python-based 的开源代码45 , 并在 github 存储库上共享。在本地硬盘驱动器上存储和管理的数据量也存在限制, 因此需要外部硬盘驱动器或在线数据共享, 如 cy诅咒46。
传统的生物反应器, 如 cdc 反应器和滴水流反应器34, 需要大量的介质, 提供较少的样品尺寸, 并要求大量的灭菌设备。相反, 这种高吞吐量平台的优点包括控制剪切力、流速的能力, 以及体外实验在体内条件中非常相似的假设。该系统的缺点包括多个附件和软件, 需要精心设置, 必须按照正确的事件顺序执行。此外, 为设备提供的手册并不能充分解释实验的每一步和软件命令, 因此, 在实验过程中会发生许多错误, 包括通道堵塞、缺乏生长或依恋,或缺乏高质量的显微镜图像或电影。仪器本身和消耗品, 如微流体板, 也相对昂贵, 每个板材的价格标签超过200美元, 无法再利用。因此, 虽然这项技术提供了强大的成果, 但使用该技术所需的技术专长相对较高, 需要该领域的专家反复培训。本报告试图通过为这些生物反应器的新用户研究生物膜特征提供指南来解决这一问题。
能够进行细胞分析的微流体系统在微生物学、免疫学、血液学、肿瘤学和干细胞研究等各种科学模式中受到了相当大的关注。更具体地说, 该技术已产生了许多出版物, 描述了与医疗应用高度相关的主题37,47, 包括微生物口服粘附 48, 确定的影响铜绿假单胞菌和金黄色葡萄球菌49,50的生物表面活性剂, 51大肠杆菌中宿主病原体相互作用,链球菌粘附 52, 及治疗囊性纤维化53.鉴于这种微流体系统用途广泛, 预计将在世界各地分布越来越多的系统。
应认真考虑一些具体的协议步骤。介质可以用 dh2o稀释到 50%, 以帮助防止气泡和堵塞, 但在这种情况下不是必需的。用于播种的 od600的具体值必须使用生长实验的试运行来确定, 以确定什么最适合所使用的特定条件集。在密封之前, 井内的气泡会导致微流体通道中的气泡, 应通过爆裂或用移液器尖端吸出来去除。重要的是要防止细菌进入小蛇形通道。在播种过程中, 由于输入和输出介质的数量相等, 因此由于液体体积的压力而产生的流量将受到控制, 因此流量仅由于来自系统的施加压力。校准距离应由公司代表在安装过程中设置。这些设置是针对每个摄像机的。
在为图像找到最具代表性的阈值时, 会出现几个挑战。如果背景区域的平均像素强度不一致, 则很难选择不在通道中心的舞台位置或板上的碎片。在mm 标准下, 单击 "进程", 然后选择 "背景" 和 "底纹校正" 工具以更正这些不一致。但是, 只有当用户在播种前拍摄了通道的图像, 并且可以用作参考图像时, 此工具通常才有帮助。或者, 如果背景/底纹参考图像不可用, 用户将需要使用其判断来设置一个阈值, 该阈值覆盖最多的单元格区域, 而不包括整个图像的背景。或者, 选择要测量的代表性区域, 以通过单击 "矩形区域"、 "椭圆区域" 或"跟踪区域" 来选择区域, 然后选择 "活动区域" 而不是"整个区域"" 显示区域统计信息" 窗口上的图像 (在 "分析工具"下)。如果使用代表性区域对明亮的场图像进行阈值, 则应使用同一区域测量相应的 fitc 图像。记录与该代表性区域关联的特定空间统计信息(左、上、宽、高度、面积、周长) 很有帮助, 这样就会找到相同的区域,在相应的 fitc 图像上进行测量。
为了防止硬盘驱动器上的数据积聚, 从而导致计算机变慢, 可以购买用于数据存储的外部硬盘驱动器。数据存储和促进数据共享的另一个选择是 cyverse 生物信息学平台。通过 http://www.cyverse.org/在 cy诅咒系统上创建一个帐户。登录后, 启动 "发现环境", 然后选择 "登录 cy诅咒"。选择 "数据" 并导航到您所在的文件夹。如果图像堆栈位于本地计算机上, 则选择 "上传", 然后选择 "从桌面进行简单上传"。找到图像堆栈文件并选择要上传。如果文件或文件夹具有 cy诅咒帐户并被授予权限, 则可以与协作者共享。向公众共享数据文件夹需要使用 cy诅咒批准的标准为每个文件添加元数据。这里将不讨论此过程, 因为这不属于本工作的范围。
披露声明
作者没有什么可透露的。
致谢
这项工作是通过国家普通医学研究所 (nigms) (5p20gm103427) 的资助而得以完成的, 该研究所是国家卫生研究院 (nih) 的一个组成部分。
材料
| Name | Company | Catalog Number | Comments |
| Ammonium Chloride, ACS | VWR | BDH9208-500G | Part of the minimal media composition |
| BioFlux 1000 48 Well Low Shear Plate | Fluxion Biosciences | 910-0047 | |
| BioFlux 1000Z Microfluidic Imaging System | Fluxion Biosciences | BF 1000Z | |
| Calcium Chloride Dihydride, ACD Grade | VWR | 97061-904 | Part of the minimal media composition |
| Dextrose, Anhydrous, ACS | VWR | BDH9230-500G | Part of the minimal media composition |
| Magnesium Sulfate ACS Grade | VWR | EM-MX0070-1 | Part of the minimal media composition |
| Potassium Phosphate Monobasic, ACS Grade | VWR | BDH9268-500G | Part of the minimal media composition |
| Pseudomonas Aeurginosa GFP | ATCC | 15692GFP | Pseudomonas aeurinosa bacterial strain PA01 with GFP modification used for this study. |
| Sodium Chloride, ACS | VWR | BDH9286-500G | Part of the minimal media composition |
| Sodium Phosphate, Monobasic, Anhydrous, Reagent Grade | VWR | 97061-942 | Part of the minimal media composition |
参考文献
- Donlan, R. M., et al. Model System for Growing and Quantifying Streptococcus pneumoniae Biofilms In Situ and in Real Time. Applied and Environmental Microbiology. 70 (8), 4980-4988 (2004).
- Lührig, K., et al. Bacterial Community Analysis of Drinking Water Biofilms in Southern Sweden. Microbes and Environments. 30 (1), 99-107 (2015).
- Singh, R., Paul, D., Jain, R. Biofilms: implications in bioremediation. Trends in Microbiology. 14 (9), 389-397 (2006).
- Veerachamy, S., et al. Bacterial adherence and biofilm formation on medical implants: A review. Proceedings of the Institution of Mechanical Engineers, Part H: Journal of Engineering in Medicine. 228 (10), 1083-1099 (2014).
- Lyczak, J. B., Cannon, C. L., Pier, G. B. Establishment of Pseudomonas aeruginosa infection: lessons from a versatile opportunist. Microbes and Infection. 2 (9), 1051-1060 (2000).
- Percival, S. L., Knapp, J. S., Edyvean, R., Wales, D. S. Biofilm Development On Stainless Steel In Mains Water. Water Research. 32 (1), 243-253 (1998).
- Percival, S. L., Knapp, J. S., Wales, D. S., Edyvean, R. G. J. The effect of turbulent flow and surface roughness on biofilm formation in drinking water. Journal of Industrial Microbiology and Biotechnology. 22 (3), 152-159 (1999).
- Ling, F., Whitaker, R., LeChevallier, M. W., Liu, W. -T. Drinking water microbiome assembly induced by water stagnation. The ISME Journal. , 1(2018).
- Moss, B. Water pollution by agriculture. Philosophical Transactions of the Royal Society B: Biological Sciences. 363 (1491), 659-666 (2008).
- Schultz, M. P., Bendick, J. A., Holm, E. R., Hertel, W. M. Economic impact of biofouling on a naval surface ship. Biofouling. 27 (1), 87-98 (2011).
- Tornero, V., Hanke, G. Chemical contaminants entering the marine environment from sea-based sources: A review with a focus on European seas. Marine Pollution Bulletin. 112 (1-2), 17-38 (2016).
- LaBauve, A. E., Wargo, M. J. Growth and Laboratory Maintenance of Pseudomonas aeruginosa. Current Protocols in Microbiology. 25 (1), 1-8 (2012).
- He, J., et al. The broad host range pathogen Pseudomonas aeruginosa strain PA14 carries two pathogenicity islands harboring plant and animal virulence genes. Proceedings of the National Academy of Sciences of the United States of America. 101 (8), 2530-2535 (2004).
- Shaner, N. C., Steinbach, P. A., Tsien, R. Y. A guide to choosing fluorescent proteins. Nature Methods. 2 (12), 905-909 (2005).
- Zhang, G., Gurtu, V., Kain, S. R. An Enhanced Green Fluorescent Protein Allows Sensitive Detection of Gene Transfer in Mammalian Cells. Biochemical and Biophysical Research Communications. 227 (3), 707-711 (1996).
- Patterson, G. H., Knobel, S. M., Sharif, W. D., Kain, S. R., Piston, D. W. Use of the green fluorescent protein and its mutants in quantitative fluorescence microscopy. Biophysical Journal. 73 (5), 2782-2790 (1997).
- Chalfie, M., Tu, Y., Euskirche, G., Ward, W. W., Prashert, D. C. Green fluorescent protein as a marker for gene expression. Science. 263 (5148), 802-805 (1994).
- Heim, R., Prasher, D., Tsien, R. Wavelength mutations and posttranslational autoxidation of green fluorescent protein. Proceedings of the National Academy of Sciences of the United States of America. 91, 12501-12504 (1994).
- Bakke, R., Kommedal, R., Kalvenes, S. Quantification of biofilm accumulation by an optical approach. Journal of Microbiological Methods. 44 (1), 13-26 (2001).
- Heydorn, A., et al. Statistical analysis of Pseudomonas aeruginosa biofilm development: impact of mutations in genes involved in twitching motility, cell-to-cell signaling, and stationary-phase sigma factor expression. Applied and Environmental Microbiology. 68 (4), 2008-2017 (2002).
- Wilson, E., et al. Using Fluorescence Intensity of Enhanced Green Fluorescent Protein to Quantify Pseudomonas aeruginosa. Chemosensors. 6 (21), (2018).
- Fang, H., Chen, Y., Huang, L., He, G. Analysis of biofilm bacterial communities under different shear stresses using size-fractionated sediment. Scientific Reports. 7 (1), 1299(2017).
- Saunders, K. A., Greenman, J. The formation of mixed culture biofilms of oral species along a gradient of shear stress. Journal of Applied Microbiology. 89 (4), 564-572 (2001).
- Valquier-Flynn, H., Wilson, C. L., Holmes, A. E., Wentworth, C. D. Growth Rate of Pseudomonas aeruginosa Biofilms on Slippery Butyl Methacrylate-Co-Ethylene Dimethacrylate (BMA-EDMA), Glass and Polycarbonate Surfaces. Journal of Biotechnology & Biomaterials. 7 (4), (2017).
- Trulear, M. G., Characklis, W. G. Dynamics of biofilm processes. Journal (Water Pollution Control Federation). 54 (9), 1288-1301 (1982).
- Machineni, L., Rajapantul, A., Nandamuri, V., Pawar, P. D. Influence of Nutrient Availability and Quorum Sensing on the Formation of Metabolically Inactive Microcolonies Within Structurally Heterogeneous Bacterial Biofilms: An Individual-Based 3D Cellular Automata Model. Bulletin of Mathematical Biology. 79 (3), 594-618 (2017).
- Jiao, Y., et al. Identification of Biofilm Matrix-Associated Proteins from an Acid Mine Drainage Microbial Community. Applied and Environmental Microbiology. 77 (15), 5230-5237 (2011).
- Flemming, H. C., Wingender, J. The Biofilm Matrix. Nature Reviews Microbiology. 8 (9), 632-633 (2010).
- Yang, X., Beyenal, H., Harkin, G., Lewandowski, Z. Quantifying biofilm structure using image analysis. Journal of Microbiological Methods. 39 (2), 109-119 (2000).
- Heydorn, A., et al. Quantification of biofilm structures by the novel computer program COMSTAT. Microbiology. 146 (10), 2395-2407 (2000).
- Azeredo, J., et al. Critical review on biofilm methods. Critical Reviews in Microbiology. 43 (3), 313-351 (2017).
- Goeres, D. M., et al. A method for growing a biofilm under low shear at the air liquid interface using the drip flow biofilm reactor. Nature Protocols. 4 (3), 783-788 (2009).
- Goeres, D. M., et al. Statistical assessment of a laboratory method for growing biofilms. Microbiology. 151, 757-762 (2005).
- Swartz, K., Stephenson, R., Hernandez, M., Jambang, N., Boles, B. R. The use of Drip Flow and Rotating Disk Reactors for Staphylococcus aureus biofilm analysis. Journal of Visual Experiments. (46), e2470(2010).
- Werner, E., et al. Stratified Growth in Pseudomonas aeruginosa Biofilms. Applied and Environmental Microbiology. 70 (10), 6188-6196 (2004).
- Wilson, C., et al. The Quantitative Assessment of Pseudomonas aeruginosa (PA)14 Biofilm Surface Coverage on Slippery Liquid Infused Polymer Surfaces (SLIPS). International Journal of Nanotechnology in Medicine & Engineering. 3 (3), 35-42 (2018).
- Samarian, D. S., Jakubovics, N. S., Luo, T. L., Rickard, A. H. Use of a High-throughput In Vitro Microfluidic System to Develop Oral Multi-species Biofilms. Journal of Visualized Experiments. (94), 52467(2014).
- Rusconi, R., Garren, M., Stocker, R. Microfluidics Expanding the Frontiers of Microbial Ecology. Annual Review of Biophysics. 43, 65-91 (2014).
- Kim, J., Park, H. -D., Chung, S. Microfluidic Approaches to Bacterial Biofilm Formation. Molecules. 17 (8), 9818-9834 (2012).
- Mosier, A. P., Cady, N. C. Analysis of bacterial surface interactions using microfluidic systems. Science Progress. 94 (4), 431-450 (2011).
- Nobile, C. J., Johnson, A. D. Candida albicans Biofilms and Human Disease. Annual Review of Microbiology. 69, 71-92 (2015).
- Chandra, J., Kuhn, D. M., Mukherjee, P. K., Hoyer, L. L., McCormick, T., Ghannoum, M. A. Biofilm Formation by the Fungal Pathogen Candida albicans: Development, Architecture, and Drug Resistance. Journal of Bacteriology. 183 (18), 5385-5394 (2001).
- Vasconcelos, M. A., et al. Effect of Algae and Plant Lectins on Planktonic Growth and Biofilm Formation in Clinically Relevant Bacteria and Yeasts. BioMed Research International. 2014, 9(2014).
- Haralick, R. M., Shanmugam, K., Dinstein, I. Textural Features for Image Classification. IEEE Transactions on Systems, Man, and Cybernetics. 3 (6), 610-621 (1973).
- Wentworth, C. D. Biofilm Morphology Suite. , Available from: https://github.com/cdwentworth/Biofilm-Morphology-Suite.git (2018).
- Cyverse. , Available from: https://user.cyverse.org/services/mine (2018).
- Benoit, M. R., Conant, C. G., Ionescu-Zanetti, C., Schwartz, M., Matin, A. New Device for High-Throughput Viability Screening of Flow Biofilms. Applied and Environmental Microbiology. 76 (13), 4136-4142 (2010).
- Ding, A. M., Palmer, R. J., Cisar, J. O., Kolenbrander, P. E. Shear-Enhanced Oral Microbial Adhesion. Applied and Environmental Microbiology. 76 (4), 1294-1297 (2010).
- Diaz De Rienzo, M. A., Stevenson, P. S., Marchant, R., Banat, I. M. Effect of biosurfactants on Pseudomonas aeruginosa and Staphylococcus aureus biofilms in a BioFlux channel. Applied Microbiology and Biotechnology. 100 (13), 5773-5779 (2016).
- Moormeier, D. E., Bayles, K. W. Staphylococcus aureus Biofilm: A Complex Developmental Organism. Molecular Microbiology. 104 (3), 365-376 (2017).
- Tremblay, Y. D. N., Vogeleer, P., Jacques, M., Harel, J. High-Throughput Microfluidic Method To Study Biofilm Formation and Host-Pathogen Interactions in Pathogenic Escherichia coli. Applied and Environmental Microbiology. 81 (8), 2827-2840 (2015).
- Nobbs, A. H., Lamont, R. J., Jenkinson, H. F. Streptococcus Adherence and Colonization. Microbiology and Molecular Biology Reviews. 73 (3), 407-450 (2009).
- Díez-Aguilar, M., Morosini, M. I., Köksal, E., Oliver, A., Ekkelenkamp, M., Cantón, R. Use of Calgary and Microfluidic BioFlux Systems To Test the Activity of Fosfomycin and Tobramycin Alone and in Combination against Cystic Fibrosis Pseudomonas aeruginosa Biofilms. Antimicrobial Agents and Chemotherapy. 62 (1), 01650-01717 (2018).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。