Method Article
Pseudomonas Aeruginosa mit einem automatisierten höheren Durchsatz mikrofluidischen System Zelle Analyse der Schubspannung weiterleben
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Hier beschreiben wir die Verwendung eines höheren Durchsatz mikrofluidischen Bioreaktors gepaart mit einem fluoreszierenden Mikroskop für die Analyse der Schubspannung Auswirkungen auf Pseudomonas Aeruginosa Biofilms grün fluoreszierende Proteine, einschließlich Instrument zum Ausdruck zu bringen eingerichtet, die Bestimmung der Biofilm Abdeckung, Wachstumsrate und morphologischen Eigenschaften.
Zusammenfassung
Ein höheren Durchsatz mikrofluidischen in-vitro- Bioreaktor gepaart mit Fluoreszenz-Mikroskopie wurde zur bakteriellen Biofilm Wachstum und Morphologie, einschließlich Pseudomonas Aeruginosa (p. Aeruginosa). Hier werden wir beschreiben, wie das System verwendet werden kann, zu studieren, die Wachstum-Kinetik und die morphologischen Eigenschaften wie die Oberflächenrauhigkeit und strukturelle Entropie von p. Aeruginosa Belastungen PA01, die ein verbesserte grünes fluoreszierendes Protein (PA01-EGFP ausdrückt ). Ein detailliertes Protokoll wird beschrieben, wie Sie wachsen und Samen PA01-EGFP Kulturen, wie das Mikroskop und Autorun, und die Durchführung der Bildanalyse, Wachstumsrate und morphologischen Eigenschaften, die mit einer Vielzahl von Scherkräften, die durch gesteuert werden die mikrofluidische Gerät. Dieser Artikel bietet eine detaillierte Beschreibung einer Technik, die Studie von PA01-EGFP Biofilmen zu verbessern, die schließlich gegen andere Stämme von Bakterien, Pilzen oder Algen Biofilme mit dem mikrofluidischen Plattform angewendet werden können.
Einleitung
Hier zeigen wir ein Verfahren zur Messung der Wirkung der Schubspannung auf die Bildung von Leuchtstofflampen Pseudomonas Aeruginosa (p. Aeruginosa) PA01 Biofilme mit einem automatisierten höheren Durchsatz mikrofluidischen System.
Biofilme sind Gemeinschaften von Mikroorganismen, wie Bakterien, organisiert von einer extrazellulären Polymeren Substanz, die an einem Träger befestigt sind, und sind in der Regel an der Grenzfläche zwischen einer Flüssigkeit und eine solide Oberfläche1gefunden. Dieser Biofilm Gemeinschaften kann vorteilhaft für die Umwelt, wie z. B. Verbesserung der Wasserqualität in Wasserleitungen und Bioremediation von widerspenstigen2,3Verbindungen. Biofilme kann jedoch auch sehr schädlich für die menschliche Gesundheit mit unerwünschten Folgen. Z. B. sind medizinische Geräte, wie z. B. Hüft- und Kniegelenke Implantate eine Art von Oberfläche wo Biofilm Ansammlung war eine Herausforderung und verursacht schwerwiegende medizinische Komplikationen4,5. Biofilme können auch geben Sie natürlichen Wasser-Systeme, wie Flüsse und Seen, und Wasserleitungen zu Bakterien Verunreinigungen im Trinkwasser, was zu Infektionen6,7,8zu infiltrieren. Biofilme Marineumwelt gegründet, Schiffen und anderen künstlichen Substraten haften und präsentieren ein großes Problem der wirtschaftlichen und ökologischen, wie erhöhter Reibung führt um zu Kraftstoff Verbrauch9,10zu erhöhen. Antimikrobielle Beschichtungen, wie z. B. Tributyltin, wurden entwickelt, um diese Probleme zu vermeiden, sondern sind giftig für Meereslebewesen11.
P. Aeruginosa ist ein gramnegatives Bakterium mit hohen blühende Fähigkeiten in einer Vielzahl von Umwelt-und nutrimental12. P. Aeruginosa ist eine häufige Ursache von Gemeinschaft und nosokomiale Infektionen und fand genau zu Verletzungen wie Verbrennungen und immungeschwächten Gastgeber, wie z. B. bei zystischer Fibrose (CF)5,12verbunden sind, 13, AIDS und Krebs Patienten5,13. Die Bildung von p. Aeruginosa Biofilms ist am schwersten zu CF, verbunden, wo chronische Lungenentzündungen die häufigste Todesursache für diese Krankheit5 sind.
Ein Referenz-Stamm von p. Aeruginosa, PA01, wird in diesem Bericht verwendet und ist genetisch verändert, um eine verbesserte grün fluoreszierendes Protein (PA-EGFP) auszudrücken. EGFP repräsentiert eine mutierte Form des GFP mit größeren Fluoreszenzeigenschaften ermöglicht eine in Situ Biofilm Analyse mit Fluoreszenz-Mikroskopie14,15,16. Diese Art der Fluoreszenzanalyse ist vorteilhaft für die Untersuchung von Biofilmen, weil GFP deutlich Zellwachstum und Funktion17nicht stört. Zum Beispiel wuchs Escherichia coli Zellen, die mit GFP markiert waren gut und kontinuierlich ohne toxischen Nebenwirkungen im Vergleich zu der Kontrolle Bakterien17erlitten. Andere Berichte belegen diesen Anspruch18,19,20. Darüber hinaus die Verwendung von fluoreszierenden Reporter wie EGFP ist schnell und einfach, aber nur lebende Zellen werden gemessen werden, da abgestorbene Zellen schnell aufhören zu21fluoreszieren.
Biofilme können unter verschiedenen Umweltbedingungen, einschließlich derjenigen mit unterschiedlichen Strömungsgeschwindigkeiten wachsen. Beispielsweise können Filme in Flüssen, wie z. B. in hohen Scherbeanspruchung wachsen wo Hochwasser Strömungsverhältnisse zu einer größeren mikrobiellen Diversität22 führen. Im Gegensatz dazu stehendes Wasser in Teichen oder orale Biofilme erleben Sie eine viel geringere Scherkräfte Kraft23. Neben Durchfluss, gibt es andere Faktoren, die Einfluss auf Biofilm Haftung, einschließlich Oberflächenrauhigkeit und Hydrophobie, Medienkomposition, und sogar die bakterielle Zelle Oberfläche1,4,7, 24. Bedingungen kann auch Unterschiede in der räumlichen Struktur oder Morphologie der Biofilm. Dazu gehören Umwelteinflüsse wie Scherbeanspruchung von sich bewegenden Flüssigkeit ausgeübten oder Farbverläufe in Nährstoffverfügbarkeit und biologische Faktoren wie die Art zu präsentieren, in das System, Beweglichkeit der Zellen und die spezifische Proteine in der extrazellulären Polymeren Substanz25,26,27. Unter bestimmten Bedingungen wird der Biofilm Rasen-wie sein (glatt und flach), während unter anderen Bedingungen der Biofilm wird rau, flauschige, oder auch Pilz-wie28. Während der qualitative Unterschied zwischen Biofilm Rasenflächen und Pilz Strukturen in mikroskopische Aufnahmen deutlich zu erkennen, erfordert Verständnis der Beziehung zwischen Filmstruktur und biologische Prozesse innerhalb des Films, systematische und quantitative Methoden zur Beschreibung der Morphologie. Morphologische Eigenschaften, die für die Studie von Forschern vorgeschlagen sind, Porosität, Fraktale Dimension, Diffusionsprozess Länge, Microcolony Bereich am Substrat, Microcolony Volumen, Rauheit Koeffizient und strukturelle Entropie29,30 .
Bioreaktoren werden in der Studie von Biofilmen zur realen Bedingungen31zu imitieren. Tropf-Fluss-Reaktoren (DFR) stellen eine Low-Shear Umwelt wo Nährstoffe in den Medien langsam über Zellen fließen, die an einer Oberfläche im Laufe der Zeit bilden einen Biofilm mit einer hohen Dichte32angeschlossen werden. CDC-Reaktoren sind Bioreaktoren, die sich ständig verändernden Umfeld eine hohe Scherspannung durch Kontrolle über einen Rührstab zu erstellen, die ständig in den Medien dreht Tank33gefüllt. Diese Arten von Bioreaktoren sind einfach einzurichten, aber sie sind im Umfang begrenzt, wegen des relativ geringen Stichprobenumfangs, der hohe Verbrauch von Medien, große Mengen an biogefährlichen Abfällen aus Medienströme Tropfen für Tropfen fließen Reaktoren 125 µL/Minute bis hin auf mehr als 1 mL/min für CDC-Reaktoren und die Notwendigkeit, Autoklaven große Mengen von Glaswaren und Abfall Medien34. Biofilme wachsen nicht gleichmäßig über die Oberfläche in einem Tropfen fließen Reaktor weil die geringe Scherkräfte Medien bewirkt, dass nachfolgende entlang der größeren Konglomeraten von p. Aeruginosa Bakterien daher, der Biofilm nicht sehr glatt wächst und die unebenen Proben nicht Fluoreszenz-Mikroskopie35,36 leicht analysiert werden.
Einige gemeinsame Bioreaktor Einschränkungen mithilfe eines Medium-Durchsatz mikrofluidischen Bioreaktors, wo nur Milliliter Medien erforderlich sind, und die Reaktion-Platten sind klein und leicht verfügbare nach Autoklavieren37. Darüber hinaus können je nach Anzahl der Bohrungen, viele Wiederholungen in einem Reaktor laufen, bietet die ausreichende Menge an Daten aussagekräftige statistische Analysen durchgeführt werden. In Abbildung 1sind die verschiedenen Komponenten des Systems der Mikrofluidik-Mikroskopie, die kontrollierten Bedingungen, einschließlich Temperatur ermöglichen und flow Rate38,39,40, dargestellt. Der Bioreaktor ist mit Fluoreszenz-Mikroskopie, die Fluoreszenz des EGFP Tags im PA01 unter visualisieren gekoppelt angewendet tief durch hohe Scherkräfte Bedingungen, die realistischere Szenarien zu imitieren, die in der Umgebung oder im biomedizinischen Bereich anzutreffen sind.
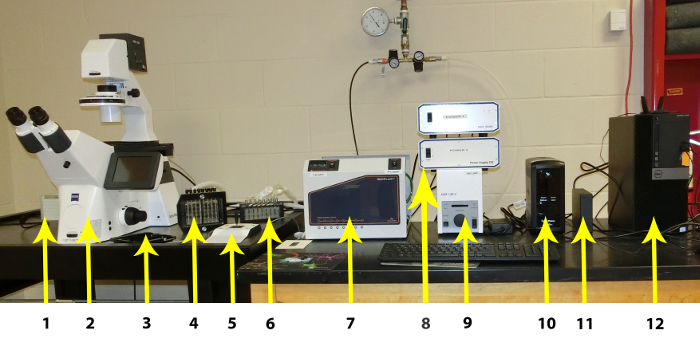
Abbildung 1 : Einzelkomponenten des Systems mikrofluidischen. Die einzelnen Komponenten sind von links nach rechts aufgeführt: 1. CCD-Kamera, 2. hohe Auflösung Inverted Mikroskop mit automatisierten Bühne, Fluoreszenz-Modul automatisiert und Autofokus-Modul, 3. Platte 4.Etappe: Imaging-System-Schnittstelle, 5: Handbuch Mikroskoptisch Regler 6: Dampf-Trap, 7: Imaging-System Controller (einschließlich Temperaturregler), 8: Hardware-Controller, 9: Fluoreszenz-Controller, 10: unterbrechungsfreie Stromversorgung, 11: externe Festplatte für Bildspeicher, 12: PC-Arbeitsplatz. Bitte klicken Sie hier für eine größere Version dieser Figur.
Ein Auszug aus der Mikrofluidik-Platte ist in Abbildung 2dargestellt. Die am häufigsten verwendeten Platten bestehen aus 48 Brunnen. Ein Experiment erfordert einen Einlass und einem Auslass Wells, insgesamt 2 Brunnen. Dies ermöglicht 24 gleichzeitige Experimente, die durchgeführt werden können mit verschiedenen experimentellen Bedingungen, wie z. B. Bakterienstämme, antimikrobielle Behandlung und Medien vielfältig von Kanal zu Kanal und Schubfluss für jede Spalte der sechs Kanäle gesteuert. Die experimentelle Temperaturregelung ist auch mit einer Temperatureinstellung in der Platte. Mikrofluidische Kanäle zeigen, dass jeder Kanal eine serpentine Region hat Bereitstellung ausreichender Gegendruck und kontrollierte Scherung.
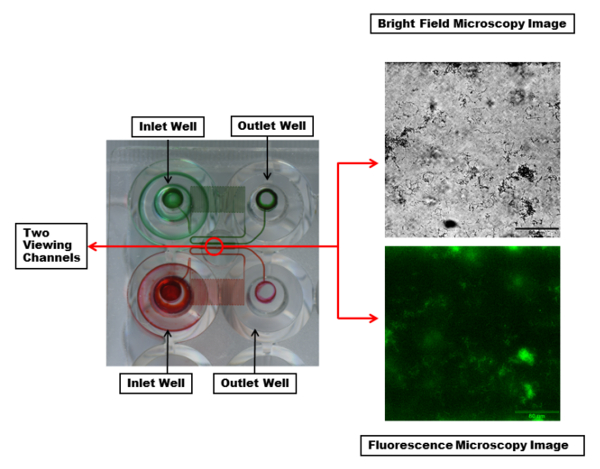
Abbildung 2: Visualisierung der mikrofluidische Kanäle und Sichtfenster. Zwei Einlass und Auslass Brunnen mit verbindenden mikrofluidische Kanäle sind mit roten und grünen Farbstoffen markiert. Der Farbstoff macht in jedem Kanal schafft genügend Gegendruck und kontrollierten Schub während Flüssigkeitsströmung eine serpentine Region sichtbar. Jedes Anzeigekanal (innerhalb des roten Kreises) kann mit gewünschten Wellenlängen abgebildet werden. Gezeigt werden Hellfeld (oben) und Leuchtstoffröhren (unten) Mikroskopie Bilder eines einzelnen Kanals mit einem PA01-EGFP Biofilm mit einem 20 X Objektiv. Maßstabsleiste = 80 µm. Bitte klicken Sie hier für eine größere Version dieser Figur.
Eine Schritt-für-Schritt-Anleitung wird bereitgestellt, damit die Benutzer von mikrofluidischen Bioreaktoren, die mit Fluoreszenz-Mikroskopie für neuartige Biofilm Experimente mit verschiedenen Scheren Umgebungen gekoppelt sind. Diese Methode ermöglicht für den Ausbau der Experimente mit anderen Mikroorganismen neben Bakterien, Pilzen und Algen, die Medizin- und Umwelttechnik Anwendungen41,42,43haben. Die detaillierte Vorgehensweise wird beschrieben, wie Kultur PA01-EGFP, impfen eine 48-Well-Platte und mikrofluidischen Gerät und Software einrichten, richten Sie die Fluoreszenz-Mikroskop und demonstrieren die Softwareanalyse, um zu erhalten die Biofilm-Abdeckung, Wachstumsrate, und morphologische Eigenschaften wie Rauheit der Oberfläche.
Protokoll
(1) Media-Vorbereitung
- Bereiten Sie minimal Medien (MM) mit 0,25 % Glukose. Um 1 L mm mit 0,25 % Glukose bilden, addieren Sie 200 mL einer sterilen M9 Salzlösung, 2 mL steril 1 M MgSO4, 100 µL steriler 1 M CaCl2und 12,5 mL steriler Glukose 20 % (w/V) zu Wasser (dH2O) zu einem Endvolumen von 1 L.
- Übertragen Sie benötigten Medien auf eine sterile Flasche mit steriler Techniken. Bereiten Sie die Füllmenge je nach der Anzahl der Kanäle verwendet wird. In der Regel erfordert jeder Kanal 200 µL für Grundierung, 300 µL für Aussaat und 1300 µL für das 24 h-Experiment.
- Stellen Sie die Flasche in einen Inkubator oder Wasserbad stellen auf die experimentelle Temperatur. Die Medien sollte die Experiment-Temperatur vor dem Gebrauch in den Mikrokanälen Blasenbildung zu vermeiden.
2. Vorbereitung einer Übernachtung und experimentelle Kultur von PA01-EGFP.
- Eine sterile sidearm Flasche 15 mL experimentelle Medien setzen und mit ein oder zwei Kolonien von einer Nährbodenplatte durchzogen von PA01-EGFP impfen. Die Kultur für 12-16 h auf einen Inkubator/Shaker-Tisch bei 37 ° C und 180-220 u/min zu erweitern.
- Messen Sie die OD600 der Übernacht-Kultur. Wenn die OD600 0,80 größer ist, verdünnen Sie die Übernachtung Kultur, die die endgültige OD von 0,8 mit frischen MM. Ort der experimentellen Kultur in einem Inkubator oder Wasserbad bei 37 ° C bis zur Aussaat mikrofluidische Kanäle benötigt. Überprüfen Sie die OD600 wieder unmittelbar vor der Aussaat um sicherzustellen, dass es nicht wesentlich vom Ziel OD600, 0,8 in diesem Fall verändert hat.
3. Geräte-Start
- Richten Sie die System Station entsprechend der Bedienungsanleitung, ähnlich wie in Abbildung 1das Setup. Um einen Fehler in der Verbindung des Instruments auf die Software zu vermeiden, schalten Sie das Gerät in der folgenden Reihenfolge:
PC-Arbeitsplatz
Fluoreszenz-Modul. Versichern Sie sich, der Fluoreszenz-Verschluss auf (Blaulicht durch Auslöser auf)
Hardware-Controller
Imaging-System Controller (siehe Tabelle der Materialien)
CCD-Kamera
Imaging Station (Mikroskop)
Hinweis: Die Temperatur der beheizten Platte sollte auf die gewünschte experimentelle Temperatur eingestellt werden. - Starten Sie die Anwendung, und geben Sie das Kennzeichen befindet sich auf dem Etikett auf der Seite der Platte.
Hinweis: Nachdem Kontrolle Anwendung gestartet werden zwei separate Anwendung Windows, eine für die Software, die die Mikroskop/Imaging Software steuert und eine für das Steuermodul, das die Pumpe und die Well-Platte-Schnittstelle steuert. Dies ist in Abbildung S1, gezeigt, wo das "Multi Dimensional Acquisition" Menü ist oben offen und das Steuermodul für ein Autorun festgelegt ist, auf der Unterseite.
4. grundieren und Aussaat der Mikrofluidik-Platte
Hinweis: Die Grundierung, Aussaat, bakterielle Anlage und das Wachstum sind in Abbildung 3dargestellt.
- Entfernen Sie die 48-Well mikrofluidischen aus der Verpackung und achten Sie auf die Glasoberfläche an der Unterseite der Platte nicht berühren. Reinigen Sie die Glas-Folie an der Unterseite der Platte gut mit ein Linsenpapier, einem fusselfreien Tuch oder ein fusselarme abwischen.
- Um die mikrofluidische Kanäle prime, 200 µL 37 ° C MM pipette in die Ausgabe nun, darauf achten, um Luftblasen zu vermeiden. Legen Sie die Platte in die Platte Stufe und wischen Sie die Schnittstelle mit Ethanol, trocknen, bevor Sie auf die Bühne der Platte abdichten, damit ab.
- Im Manuellen Modus auf dem Steuermodul festgesetzt Flüssigkeit als LB bei 37 ° C und Max Scherung 5,00 dyn/cm2. Klicken Sie auf die Ausgabe Brunnen nun, aktivieren Sie fließen vom Ausgang zum Eingang Grundieren der Kanäle. Unterbrechen Sie nach den 5 min Grundierung den Fluss für die Aussaat vorbereiten. Sorgfältig entfernen Sie die Platte von der Bühne und pipette verbleibende Medien aus der Ausgang aber nicht entfernen die Videos aus dem inneren Kreis, der die die mikrofluidische Kanäle führt.
- Um die experimentelle Kanäle seed, pipette zunächst 300 µL MM an den Eingang gut gefolgt von Pipettieren 300 µL der Bakterienkultur in die Ausgabe auch in die Ausgabe gut. Setzen Sie die Platte wieder in die Platte Bühne und achten Sie auf die Schnittstelle zu wischen, bevor sie auf dem Teller platziert.
- Konzentrieren Sie auf dem Steuermodul sich auf einen einzigen Kanal mit der live-Kamera nach dem Platzieren der Platte Bühne auf den Mikroskoptisch zu ernähren. Während visuelle Überwachung durch den live-Feed, fortgesetzt bei 1.00-2.00 dyn/cm2 für ca. 2-4 s damit Zellen den experimentellen Channel zu betreten, aber nicht in der serpentine Kanäle. Lassen Sie die Platte auf der Bühne kontrollierter Temperatur für 1 h bis zur Zelle Befestigung zu ermöglichen. Während der 1 h Inkubation kann die Steuermodul und Montage-Software für den Autorun (Schritt 5) eingerichtet werden.
Hinweis: Der Zeitaufwand für die Aussaat variiert mit Medien und Organismus, so dass es eng sollen, durch die Liveübertragung überwacht werden bis optimiert und als eine allgemeine zeitliche Vorgabe verwendet. Aussaat in die Platte kann unterschiedlich sein, die mehr Zeit angewandte Flow auf bestimmte Spalten von Kanälen für die komplette Aussaat erfordern würde. - Nach Ablauf der Anlage schonend entfernen Sie die Platte von der Bühne und pipette die Bakterien aus der Ausgabe zuerst zu vermeiden, den Kanal zu stören. Entfernen Sie mit einer neuen Pipettenspitze das Medium aus der Eingabe Brunnen.
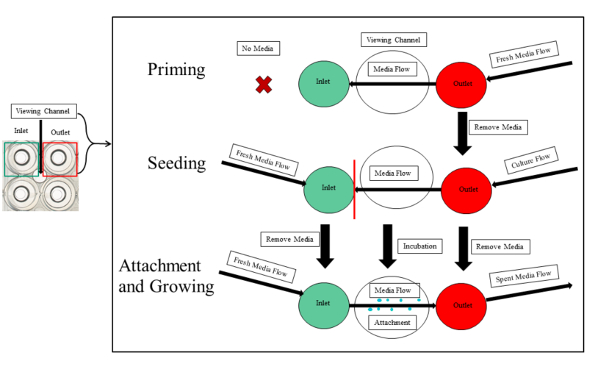
Abbildung 3 : Experimentelle Überblick Priming, Aussaat und Befestigung der PA01-EGFP in mikrofluidischen Kanälen. Die Grundierung, Aussaat und Anlage wird erläutert. Der erste Schritt der Grundierung erfordert neue Medien eingeführt, um die Ausgabe. Die Aussaat mit sich bringt gleich Mengen an Medien und bakterielle Kultur in der Eingabe und Ausgabe Brunnen, beziehungsweise. Die Kultur sollte nicht Segment Anzeige des experimentellen Kanals (rote Linie) zu vermeiden, verstopfen die serpentine Kanäle weiterzugeben. Nach Ablauf die Inkubationszeit fließt frische Medien kontinuierlich aus der Eingabe der Anzeige-Kammer und in die Steckdose. Dadurch wird die Anlage und das Wachstum des bakteriellen Biofilms. Bitte klicken Sie hier für eine größere Version dieser Figur.
5. Einstellung der Software
- Öffnen Sie in der Software "Mehrere dreidimensionale Erfassung", um die Bildaufnahme Mikroskop zu steuern. Klicken Sie im Menü "Main" "Zeitraffer", "Mehrere Phase-Positionen" und "Mehrere Wellenlängen" (Abbildung S1A).
- Einrichten der Einstellungen Speichern , erstellen Sie einen einfachen Basisnamen, um sicherzustellen, dass die Schrittweite Basis Name existiert Datei aktiviert ist. Klicken Sie auf Wählen Sie Verzeichnis um den Ordner auszuwählen, in dem alle Dateien gespeichert werden. Enthalten Sie wesentliche Details des Experiments in der Beschreibung.
- Passen Sie unter der Registerkarte " Zeitraffer " die Dauer der experimentellen Zeit bis 24 h. Das Zeitintervall festlegen, so dass Bilder alle 5 Minuten während des Experiments erworben werden, um Steuern, wie oft Bilder erworben werden. Die Anzahl der Zeitpunkte passt sich automatisch mit den eingestellten Parametern.
- Eingestellte Stufe Positionen mit der live-Kamera im Hellfeld-Mikroskop, so dass für die korrekte Platzierung und Ausrichtung zu ernähren. Beginnend mit der 10 X-Objektiv, Fokus auf der Mitte des Kanals, befindet sich oberhalb oder unterhalb der Kanalnummern eingraviert auf dem Teller. Wechseln Sie zu den 20 X Objektiv, Suche nach dem optimalen Sichtbereich und Brennebene innerhalb des Kanals. Fügen Sie die Position der Liste hinzu oder ersetzen Sie die preset-Kanal-Position mit den neuen Einstellungen.
- Legen Sie unter dem Menü " Wellenlängen " die Anzahl der Wellenlängen auf 3. Wellenlänge 1 (W1) wählen Sie in der FITC-100 %-CAM mit einer Belichtungszeit von 10 ms. Für Wellenlänge 2 (W2), wählen Sie das Hellfeld 50 % CAM 50 % VIS mit minimalen Belichtungszeit von 3 ms für Wellenlänge 3 (W3), als der Filter Alle geschlossen bleibt also kein Licht auf den letzten Kanal zwischen Erfassungszeiten.
- Bei der Einrichtung das Steuermodul überprüfen Sie, um sicherzustellen, dass die manuelle Ausführung gestoppt wurde.
- Legen Sie im Menü Bearbeiten Autorun unter der Registerkarte " Protokoll Setup " ein neues Protokoll für 24 h Dauer hinweg, fließt in Durchlassrichtung an die gewünschte Scherrate. Schergeschwindigkeit ist vielfältig, um die Wirkung der Biofilm Wachstum mit Scherrate zu studieren. Klicken Sie auf Hinzufügen "und" Speichern unter Protokoll.
- Erstellen Sie unter der Registerkarte " Sequenz Setup " eine neue Sequenz indem Sie LB@37degrees als die Standard-Flüssigkeit für alle Kanäle auswählen. Wählen Sie unter Schritt Iteration 1, für Kanäle 1-12 das Protokoll mit dem gewünschten Schergeschwindigkeit und aktivieren alle Kanäle. Wählen Sie für die Kanäle 13-24 das Protokoll mit dem zweiten gewünschten Schergeschwindigkeit und aktivieren alle Kanäle. Wählen Sie anwenden und Speichern unter der Sequenz.
- Wählen Sie im Autorun -Menü die gespeicherte Sequenz für den Autorun verwendet werden.
6. zeitgesteuerte Biofilm Wachstum Testeinrichtung
- Pipette bis zu 1.300 µL steriler MM an den Eingang auch der mikrofluidischen Platte. Die Platte wieder auf die Platte-Bühne und wischen Sie die Oberfläche mit Ethanol, Ethanol vollständig trocknen, bevor die Abdichtung der Plattenrandes ermöglicht.
- Platzieren die Platte auf den Mikroskoptisch zu inszenieren und sicher sein, dass die Protokoll(e) und Sequenzen korrekt eingerichtet sind. Wählen Sie starten , starten Sie den Autorun, schnell gefolgt von Acquire um die Mikroskop-Bildersammlung zu starten klicken.
- Stellen Sie den Fokus und die Platzierung aller Bühne Positionen zwischen Bild Rauhfutter. Wählen Sie Pause , und wählen Sie mithilfe des Livebild-Modus in der Hellfeld-Wellenlänge Gehe zu jeder Phase Positionen anzeigen. Legen Sie neue Einstellungen, indem Sie auswählen, auf Strom gesetzt. Klicken Sie auf fortsetzen , vor der nächsten geplanten Übernahme.
7. überprüfen und analysieren von Bildsequenzen nach dem zeitgesteuerten Biofilm-Wachstum-Experiment
- Überprüfen Sie jeden Kanal nach 24 h Autorun, in der Software öffnen Multidimensionale Daten überprüfen, befindet sich unter Analyse-Tools. Klicken Sie auf Wählen Sie Base-Datei | Wählen Sie Verzeichnis navigieren Sie zu dem Ordner mit Bildsequenzen für das Experiment von Interesse. Wählen Sie in der Liste der Datensätze in den Ordner, der angezeigt wird den Basisnamen der relevanten Daten.
- Nachdem die relevanten Daten ausgewählt haben, klicken Sie auf Ansicht , um diese Daten zu überprüfen. Wählen Sie eine Wellenlänge, die Daten unter Wellenlänge Fenster (Abbildung S2) zu überprüfen.
- Überprüfung der Bildsequenz für jeden Kanal, indem Sie auswählen die Kanalnummer unter Tischposition und die video-Steuerelemente zum Analysieren der Daten für die 289 mal Punkte (unter der Annahme, dass Bilder alle 5 Minuten über einen Zeitraum von 24 h erhalten werden). Nutzbare Wachstum Daten beachten.
Hinweis: In jedem Kanal für die Entwicklung von Luftblasen und Clogs, die stören, die Biofilm-Wachstum innerhalb des Kanals, die Auswirkungen auf die Daten zu sehen. Jedoch, wenn diese auftreten, später in die Laufdaten vor diesen Umständen nützlich finden. - Erstellen Sie nach Prüfung und Auswahl der gewünschten Daten für die Analyse Stapel von Bildern für jeden Kanal. Wählen Sie im Menü Datei geöffnet spezielle | Stapel zu bauen | Nummerierte Namen. Wählen Sie im Stapel bauen Fenster die Schaltfläche " Erste Bild auswählen " und wählen Sie dann das erste Bild für den Stack in den Dateiordner; Wählen Sie die Schaltfläche " Letzte Bild auswählen " und wählen Sie dann die Datei entsprechend bis zum letzten Bild im Stapel. Klicken Sie auf "OK" , um den Stapel mit den Bildern des Kanals in chronologischer Reihenfolge zu öffnen. Stapel können im Menü Datei Speichern unter als TIFF-Dateien gespeichert werden.
Hinweis: Erstellen Sie Stapel nicht mit dem Daumen Bilder. Diese Dateien sind nicht sehr nützlich und können gelöscht werden, um Platz auf der Festplatte zu speichern. Darüber hinaus werden Image-Dateien wie folgt gespeichert:
Basis NAME_WAVELENGTH_sCHANNEL #_tIMAGE #.
Zum Beispiel ist 07062018_W1FITC 100_s12_t112 für den Kanal 12, Zeitpunkt 112, Bild in den ersten Wellenlänge-FITC-100 % Kamera-Daten mit den Namen "07062018". - Schließlich wählen Sie Speichern unter aus dem Dropdown-Menü Datei Sequenz speichern als TIFF-Stack. Stapel können auch überlagert und als Film gespeichert werden. Dies ist in Schritt 9 gerichtet.
- Kalibrieren Sie vor der Quantifizierung der % Flächendeckung die Bild Strecken. Analyse-Toolsklicken Sie auf Kalibrieren Entfernungen. Wählen Sie die entsprechende Kalibrierung Messung und klicken Sie auf anwenden, um alle geöffneten Bilder.
- Während der Stapel auf das erste Bild der Sequenz ist, klicken Sie auf die Schaltfläche " Schwelle ". Verwenden Sie Automatische Schwellenwert für Licht Objekte Schwellenwert für Fluoreszenzsignal (FITC Wellenlänge) und Auto-Schwelle für dunkle Objekte Schwellenwert im Hellfeld. Stellen Sie Schwelle in der Berichterstattung, die die Abdeckung des Bildes darstellt.
- Fluoreszierende Schwellen nutzen Sie einen Kanal mit nicht-fluoreszierenden Zellen herstellen jeder Hintergrundsignal und Satz der minimalen Grenzwert auszuschließende Signal erkannt Zellen um die Signalmessung zu gewährleisten ist in Kanäle, enthält diese nicht fluoreszierenden überschätzen Sie nicht den Bereich der Fluoreszenz. Verschiedene von Ausführung zu Ausführung möglicherweise eine gute Idee, verwenden Sie mindestens eine, also, wenn nicht mehr, für nicht-fluoreszierende Steuerelemente Abflusskanäle.
- Für Hellfeld Schwellen wenn alle Zellen die orange Schwelle Signatur nicht erfasst sind, passen Sie den maximalen Grenzwert entweder über die gleitende Symbolleiste oder mit der Schwelle Bild (zu finden unter den Analyse-Tools), so dass alle Zellen sind an bedeckt, aber keinen Hintergrund ist ausgeschlossen.
Hinweis: Während diese Schwellenwerte im Idealfall für alle Bilder innerhalb eines Stapels und alle Kanäle verwendet werden würde, Schwellenwerte für ein Bild/Kanal ausgewählt möglicherweise nicht geeignet für andere Bilder oder Kanäle, daher, dass der Benutzer muss möglicherweise Grenzbereich anpassen in regelmäßigen Abständen. Aus diesem Grund ist es ratsam, immer die maximale aufzeichnen und Mindestschwelle Werte verwendet, sollten die Daten müssen später überprüft werden.
- Quantifizierung die Deckung unter Analyse-Tools, klicken Sie auf Statistiken zu einer Region zu zeigen. Darauf achten, dass die Verwendung Schwelle aktiviert ist, wird das Gesamtbild ausgewählt und unter Erhaltenen Daten, stellen Sie sicher die gewünschten Messungen/Einstellungen ausgewählt (Schwellenbereich, Mittelwert, Standardabweichung, min, Max und %-Schwelle Bereich). Klicken Sie auf Protokoll öffnen und stellen Sie sicher, dass DDE-Datei ausgewählt ist, bevor Sie auf "OK". Wählen Sie Microsoft Excel , und klicken Sie auf "OK". In der Tabelle, die öffnet, klicken Sie auf Log-Daten , um die Maße des Bildes analysiert automatisch aufzeichnen.
- Verwenden Sie diese Tabelle offengelassen, das gleiche Blatt um alle Bilddaten Analyse für einen einzigen Stapel sammeln. Gehen Sie im Stapel auf dem vierten Bild und Schwelle auf die optimalen Einstellungen. Klicken Sie auf Log-Daten auf dem Bildschirm Zeigen Statistiken zu einer Region und die Werte werden in die Tabelle protokolliert. Wiederholen Sie diesen Vorgang für die Analyse der Bilder alle 15 Minuten.
8. sonstige Analyse einschließlich Morphologie und Oberfläche Abdeckung Maßnahme verwenden Python Skripte aus der Biofilm-Morphologie-Suite
- Installieren Sie eine Verteilung der Python-3.6, die wissenschaftliche Standardmodule enthält mithilfe eine standard wissenschaftlichen Python-Distribution wie Anaconda, an https://www.anaconda.com/download erhältlich.
- Erhalten der Biofilm-Morphologie-Suite von GitHub in einem Browser durch navigieren zu https://github.com/cdwentworth/Biofilm-Morphology-Suite.git, dann wählen Sie Klon oder Download (grüne Taste) und Zip-Download. Entpacken Sie die Datei und verschieben Sie die Code-Ordner in ein Arbeitsverzeichnis.
- Messen Sie Prozent thresholded Abdeckung, indem Sie den Bildstapel in diesen "Abdeckung" Ordner kopieren. Verwenden Sie ein terminal-Fenster zu diesem Ordner navigieren und dann führen Sie das Skript mit dem Befehl
Python-bfCoverage.py- tiffStackName.tif
von der Kommandozeilen-Schnittstelle, wobei tiffStackName.tif der Name der Datei mit dem TIFF-Stapel von Bildern ist. Eine Textdatei mit der Abdeckung Messungen zu jedem Zeitpunkt erstellt werden mit dem Namen tiffStackName.txt und eine Grafikdatei mit einem Grundstück von Abdeckung als Funktion der Zeit mit dem Namen erstellt wird tiffStackName.png. - Verwenden Sie dasselbe Verfahren in Schritt 8.3 angegeben, um der morphologischen Merkmale zu messen: Biofilm Akkumulation, Rauheit Koeffizient und strukturelle Entropie. Nutzen Sie das bfAcc.py-Skript für die Messung der Akkumulation, das roughCoef.py-Skript für die Rauheitsmessung Koeffizienten und das TEvsTime.py-Skript für die strukturelle Entropie-Messung.
9. andere Software-Anwendungen - Overlays und Filme machen
- Wenn mehrere Wellenlängen verwendet werden, erstellen Sie Overlay Stapel mit diesen Wellenlängen. Öffnen Sie alle Stapel von Interesse und kalibrieren Sie die Entfernung zu, wie in Schritt 7.6 getan.
- Wählen Sie unter Analyse-Tools Overlay-Bilder. Legen Sie die Quellen als die Stapel von Interesse. FITC-Stack auf dem Regenbogen-Kreis klicken Sie und wählen Sie grüne Farbe Filter hinzugefügt werden, um diese Wellenlänge zu unterstreichen.
- Verwenden Sie bal die Überlagerung so anpassen, dass die FITC-Stack in den Bildern ersichtlich ist. Nachdem alle Einstellungen vorgenommen haben, wählen Sie Alle Ebenen für beide Stacks und klicken Sie auf anwenden. Speichern Sie die Overlays auf die gleiche Weise als Stapel im Schritt 7,5 beschrieben oder als Film im Schritt 9.2 beschrieben.
- Um Stacks oder Überlagerungen als einen Zeitraffer-Film unter MM Standard und Stack, zu speichern klicken Sie auf Film erstellen. Wählen Sie die Quelle als Stapel oder Overlay, das gewünscht wird. Klicken Sie auf Speichern und speichern Sie als Microsoft Video 1 mit einer Qualität zwischen 70-80.
Ergebnisse
Abbildung 4 eine zeigt die prozentuale thresholded Bereich im Laufe der Zeit aus einer 24 h laufen auf Scherung Ströme von 0,2, 0,5 und 1,0 dyn/cm2. Der Biofilm Abdeckung oder Prozent Schwellenfläche (C [%]) wurde für alle drei scher Einstellungen unterschiedlich. Die Biofilm-Berichterstattung war der schnellste auf 1,0 dyn/cm2 Scherung, wo erhöht die Schwellenbereich von 2-5 % bis 100 % nach 200 min des Wachstums und eine stationäre Phase nach 400 min erreicht. Bei 0,5 dyn/cm2die Biofilm-Berichterstattung wurde verzögert und fing an bei 400 min erreichen 100 % Abdeckung nach 800 min zu erhöhen. Die niedrigsten Scherkräfte bei 0,2 dyn/cm2 zeigte deutlich die langsamsten Anstieg der Biofilm Abdeckung, wo Prozent Abdeckung begann bei 500 min erhöhen aber nie außerhalb des Raumes 65 % Schwelle erreicht. Diese Ergebnisse zeigten Scherung im Biofilm Flächendeckung direkten Einfluss hatte. Die höhere Scherkräfte schien eine optimale Voraussetzung für Biofilm Wachstum, möglicherweise, da die Medien die Bakterien mit mehr Nährstoffen versehen, so dass der Biofilm schneller vermehren kann.
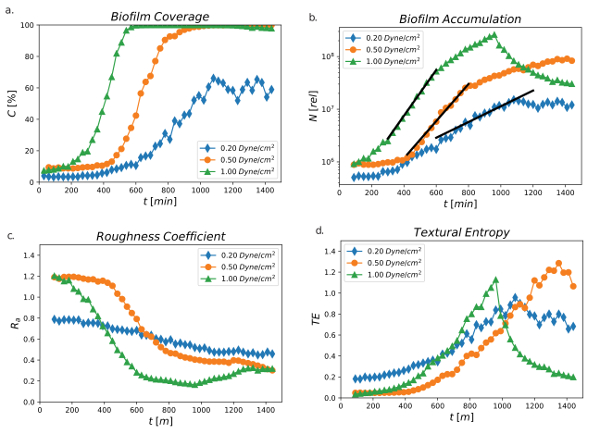
Abbildung 4: prozentuale Schwellenbereich, total Biofilm Akkumulation, Rauheit Koeffizient und strukturelle Entropie. a) Prozent Schwellenbereich (C [%]) im Laufe der Zeit mit 0,2, 0,5 und 1,0 dyn/cm2 über einen 24 h Zeitraum. Daten wurden von einem Kanal für jede Schere Bedingung ermittelt. (b) insgesamt Biofilm Akkumulation (relatives Maß) als Funktion der Zeit mit 0,2, 0,5 und 1,0 dyn/cm2 über einen 24 h Zeitraum. Die schwarzen Linien sind die kleinsten Quadrate zu einem exponentiellen Modell passt. Daten wurden von einem Kanal für jede Schere Bedingung ermittelt. (c) Rauheit Koeffizient von PA01-EGFP mit Schubspannung Werten von 0,2, 0,5 und 1,0 dyn/cm2 überwacht über 24 h. Daten wurde von einem Kanal für jede Schere Bedingung erhalten. (d) strukturelle Entropie von PA01-EGFP mit Schubspannung Werten von 0,2, 0,5 und 1,0 dyn/cm2. Daten wurden von einem Kanal für jede Schere Bedingung ermittelt (Valquier-Flynn, H., Sutlief, A.L., Wentworth, C.D., 2018). Bitte klicken Sie hier für eine größere Version dieser Figur.
Abbildung 4 b steht im Einklang mit den Erkenntnissen, die in Abbildung 4einengezeigt. Die Biofilm-Ansammlung (N[Rel]) gemessen wurde basiert auf der Annahme, dass das GFP-Signal an einem Punkt in einem Bild proportional zu der live Zelldichte an dieser Position ist. Es zeigt, dass insgesamt Relativmessung Biofilm Anhäufung als Funktion der Zeit mit 0,2, 0,5 und 1,0 dyn/cm2 über einen 24 h Zeitraum erhöht und die Wachstumsrate verringerte sich von höchsten Schubspannung auf niedrigsten Schubspannung. Es gibt eine klare geben exponentielles Wachstum, aus dem ein quantitatives Wachstum berechnet werden kann.
Abbildung 4 c zeigt die Rauheit Koeffizient (Rein) auf Scherung Ströme von 0,2, 0,5 und 1,0 dyn/cm2. Rauheit der Oberfläche, durch die Rauheit Koeffizient quantifiziert misst die Abweichung im Dickenprofil des Films. Die formale Definition ist
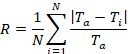
wo Tich der ich-th Dickenmessung, Tein ist die durchschnittliche Dicke, und N ist die Anzahl der Dicke Messung30. In dieser Untersuchung beschriebene Verfahren misst die Dicke mit lebenden Zellen verbunden. Eine flache Anordnung der Zellen erbringt eine Rauheit Koeffizient von Null, während erhebliche Abweichungen der Stärke aus dem Durchschnitt eine Rauheit Koeffizient größer als eins erbringt. Ähnlich wie bei der Scherung Einfluss auf das Wachstum und die prozentuale Schwellenwert Abdeckung des Films, die Biofilme ausgestellt unterschiedlichen Topographien im Laufe der Zeit. Insgesamt verringerte sich Reine im Laufe der Zeit für alle scher Bedingungen darauf hinweist, dass alle Oberflächen glatter geworden. Führten jedoch im Vergleich zu den niedrigsten Scherung von 0,2 dyn/cm, die höhere Scherkräfte Einstellungen von 0,5 und 1,0 dyn/cm2 eine glattere Oberfläche im Laufe der Zeit darauf hinweist, dass ein schneller Schubfluss an glatter und ebenmäßiger Oberfläche, und die höchsten Scherung der beigetragen 1,0 dyn/cm2 erreichen unter 0,2 Rein.
Die Glätte, Regelmäßigkeit oder Rauigkeit der Oberfläche kann auch in strukturellen Entropie (TE) ausgedrückt werden. TE ist eine Eigenschaft, die in Bild-Analyse verwendet, um den Grad an Zufälligkeit in ein zweidimensionales Bild messen. Die Berechnung basiert auf der grauen Level Co-Occurrence Matrix festgelegten Haralick Et Al., die schaut ob Pixelwerte an einem Standort mit Pixelwerte an einem anderen Standort44korreliert sind. Ein hohes Maß an Korrelation führt zu niedriger Entropie. Abbildung 4 d zeigt die TE bei Scherung Ströme von 0,2, 0,5 und 1,0 dyn/cm2. TE erhöht im Laufe der Zeit für alle scher Bedingungen aber die höchste Schubspannung mit 1,0 dyn/cm erreicht die maximale TE (1.0) früher als die niedrigeren Schubspannungen bei 900 min. Die niedrigsten Scherung von 0,2 dyn/cm2 hatte die niedrigsten TE (0,8) erreicht sein Maximum nach 1.000 min. Die mittlere Schubspannung von 0,5 dyn/cm2 erreichte jedoch viel später als die hohe oder niedrige Schubspannung Bedingungen seine maximale TE (1.2).
Die Rauheit Koeffizient und TE messen verschiedene Funktionen. Während eine Flachfolie eine geringe Rauheit Koeffizient und niedriger Entropie hätten, ein Film mit deutlichen Unterschieden in der Dicke hätte eine hohe Rauheit Koeffizient aber könnte noch niedriger Entropie sein, wenn die Variation sinusförmige als zufällige ist. In diesem Fall verringert Reine erhöhte Schubspannung und Zeit während der TE-Trends direkt zugeordnet werden können, um Stress angewendet zur Biofilmbildung im Laufe der Zeit zu scheren.
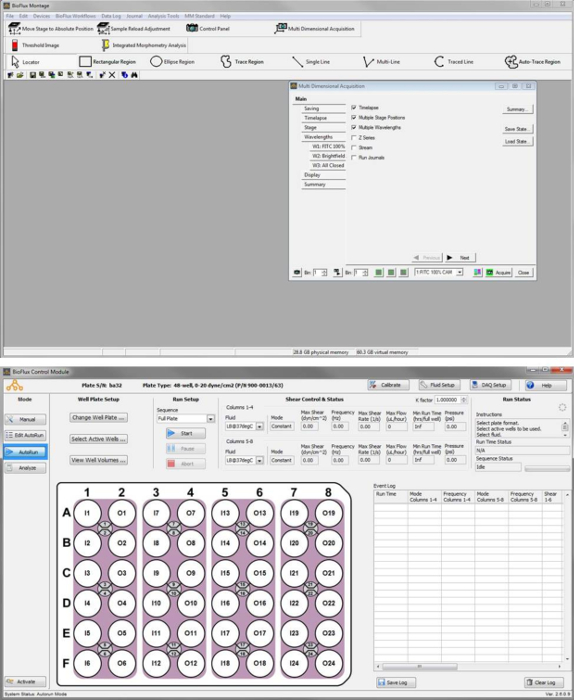
Abbildung S1 : Von der Software Windows Bilderfassung. Software mit Multi Dimensional Erwerb Menü Fenster (oben). Steuermodul für ein AutoRun (unten) festgelegt. Bitte klicken Sie hier für eine größere Version dieser Figur.
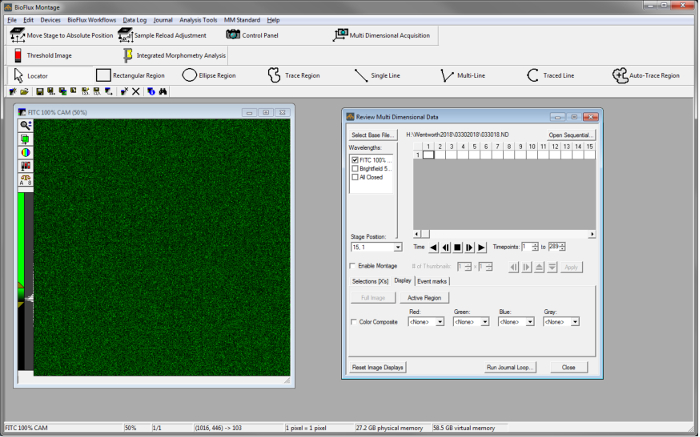
Abbildung S2 Bilderfassung der Software für die Überprüfung der Daten. Anwendungsfenster nach Auswahl des Datensatzes von Interesse mit dem Review Multi-dimensionalen Daten -Tool aus dem Analyse-Tools -Menü. Bitte klicken Sie hier für eine größere Version dieser Figur.
Diskussion
Das mikrofluidischen System und Bild Analyseverfahren diskutiert hier konzentriert sich auf die Ausführung von mikrofluidischen Biofilm Experimente zur morphologische Eigenschaften bestimmen, die nicht die volle dreidimensionale Informationen in der Regel aus konfokale benötigen Mikroskop-Studien. Dazu gehörten Microcolony Substrat Abdeckung (prozentuale Abdeckung), Oberflächenrauhigkeit, gemessen an der Rauheit Koeffizient und strukturelle Entropie. Eine Methode zur Schätzung der gesamten relativen Biofilm Zelle Ansammlung wird ebenfalls dargestellt von welchen Wachstumsraten im Protokoll Phase berechnet werden kann.
Es gibt mehrere kleine, aber wichtige Schritte, die in dieser Methode hervorgehoben werden sollte. Wischen die Oberfläche mit Alkohol vermeidet Kontamination von anderen Bakterien in going von Experiment zu Experiment, aber auch von Brunnen zu Brunnen in einem Experiment. Grundierung und Aussaat sind ebenfalls sehr wichtig, weil Priming ermöglicht es dem Benutzer zu bestimmen, welche Kanäle die Medien durch die Kanäle ungestört fließen zulassen oder Verstopfung. Kanäle sollte auch nicht gestört (d. h. sie sollten ständig voller Medien sein) nach dem Grundieren zur Erhöhung der Chancen auf ein erfolgreiches Experiment mit keine Luftblasen oder verstopfen. Die Aussaat Schritt variiert werden nach der Art der Bakterien und sollte also für Zellhaftung optimiert werden. Zum Beispiel wenn Zellen nicht zu legen scheinen, Oberflächenmodifikationen möglicherweise auf der Mikrofluidik-Platte vor der Aussaat auftreten oder längere Inkubationen Zeiten erforderlich sein. Es ist auch wichtig, um sicherzustellen, dass das Mikroskop korrekt eingerichtet ist, dass ein Bild im Fokus zu erhalten und sollten in regelmäßigen Abständen überwacht werden, während das Experiment zu versichern, dass die Bildqualität erzielt werden. Wenn der Fokus deaktiviert ist, wird das Mikroskop kann und sollte angepasst werden, wie das Experiment geht weiter. Während die Bild-Erfassungszeit sollte die letzte Wellenlänge Alle geschlossen festgelegt werden, zur Vermeidung die Exposition von Filter und Beleuchtung, nur einen Kanal während der Wartezeit, die zwischen den Bild-Akquisitionen auftritt. Auch war die Bildanalyse, die bestimmt, die % Flächendeckung im Haus konzipiert, da Montage-Software-Handbuch nicht explizit beschrieben. Darüber hinaus um zu erweitern auf die Bildanalyse und andere Merkmale wie Oberflächenrauhigkeit usw.zu bestimmen., open Source Python-basierten Code45 wurde im Haus und freigegebenen auf GitHub Repository entwickelt. Es gibt auch Einschränkungen auf wie viele Daten können gespeichert und verwaltet auf der lokalen Festplatte, so dass eine externe Festplatte oder Online-Datenaustausch wie CyVerse46erforderlich ist.
Herkömmlichen Bioreaktoren, wie dem CDC-Reaktor und die Tropfen fließen Reaktor34, erfordert viel Medien bieten weniger Stichprobengrößen und verlangen ein hohes Maß an Sterilisation der Geräte. Im Gegensatz dazu die Vorteile dieser höheren Durchsatz-Plattform gehört die Fähigkeit, Kontrolle Scherung, Durchflussmengen, und die Annahme, dass die in-vitro- Experimenten eng ähneln in Vivo -Bedingungen. Nachteile des Systems gehören mehrere Zubehör und Software, die erfordern eine sorgfältige einrichten, die in der richtigen Reihenfolge der Ereignisse durchgeführt werden muss. Darüber hinaus die Anleitung, die für die Ausrüstung bereitgestellt wird nicht vollständig zu erklären jeden Schritt der Experimente und die Softwarebefehle, und folglich viele Fehler auftreten, während der Experimente, einschließlich Verstopfung der Kanäle, Mangel an Wachstum oder Anlage, oder der Mangel an qualitativ hochwertige Mikroskopie Bilder oder Filme. Das Instrument selbst und Verbrauchsmaterialien, wie die mikrofluidischen Platten sind auch mit einem Preis von über $200 pro Platte relativ teuer und sind nicht wiederverwendbar. So, während die Technik starke Ergebnisse verleiht, technische Fachkenntnisse für seine Verwendung ist relativ hoch und erfordert wiederholte Ausbildung von Experten auf dem Gebiet. Dieser Bericht versucht, dieses Problem zu beheben, indem Sie eine Anleitung für neue Benutzer von diesen Bioreaktoren Biofilme Eigenschaften zu studieren.
Das mikrofluidischen System, das zelluläre Analyse durchführen kann, hat beträchtliche Aufmerksamkeit für verschiedene wissenschaftliche Modalitäten, wie z. B. in Mikrobiologie, Immunologie, Hämatologie, Onkologie und Stammzellenforschung gewonnen. Genauer gesagt, hat die Technologie führte in vielen Publikationen beschreiben Themen, die für medizinische Anwendungen37,47, einschließlich mikrobielle mündliche Adhäsion48, Bestimmung der Auswirkungen von großer Bedeutung sind Biosurfactants auf Pseudomonas Aeruginosa und Staphylococcus Aureus49,50, host-Pathogen Interaktionen in E. Coli51, Streptococcus Einhaltung52und Behandlung Mukoviszidose-53. Angesichts der Tatsache, dass dieses mikrofluidischen System sehr vielseitig ist, ist davon auszugehen, dass immer mehr Systeme in der ganzen Welt verteilt werden.
Einige spezifische Protokoll Schritte sollte sorgfältig abgewogen werden. Medien können verdünnt werden, um 50 % mit dH2O zur Vermeidung von Luftblasen und Verstopfungen aber war in diesem Fall nicht erforderlich. Der spezifische Wert der OD600 verwendet für die Aussaat muss ermittelt werden, mit Probeläufen eines Experiments Wachstum um zu sehen, was funktioniert am besten für die bestimmten Satz von Bedingungen verwendet. Luftblasen in den Brunnen vor dem Versiegeln zu Bläschen in mikrofluidischen Kanälen führen können und sollte entfernt werden, durch geknallt oder mit einer Pipettenspitze abgesaugt. Es ist wichtig, um Bakterien aus den kleinen Serpentinen Kanälen zu halten. Dadurch, dass gleiche Mengen an Medien in die ein- und Ausgabe bei der Aussaat, wird Fluss aufgrund des Drucks durch das Flüssigkeitsvolumen gesteuert werden, also nur durch den anliegenden Druck aus dem System läuft. Die Kalibrierung Entfernungen sollten durch die Vertreter des Unternehmens während der Installation eingerichtet werden. Diese Einstellungen sind spezifisch für jede Kamera.
Es gibt mehrere Herausforderungen, die auftreten, wenn die repräsentativsten Schwelle für ein Bild zu finden. Maximale Grenzwerte festlegen kann schwierig sein, wenn die durchschnittliche Pixel Intensität in den Regionen des Hintergrundes verursacht entweder wählen Sie eine Bühne-Position, die nicht in der Mitte des Kanals oder Trümmer auf der Platte ist nicht konsistent sind. Klicken Sie unter MM Standardauf Vorgang, und wählen Sie Hintergrund und Shading Korrektur -Tool zur Korrektur für diese Ungereimtheiten. Dieses Tool ist jedoch im Allgemeinen nur hilfreich, wenn der Nutzer Bilder der Kanäle vor der Aussaat getroffen hat, die sie als Referenzbilder verwenden können. Oder, wenn Hintergrundschattierung/Referenz Bilder sind nicht verfügbar, der Benutzer muss ihr Urteil zu verwenden, um ein Schwellenwert festgelegt, die die meisten Zelle Bereich abdeckt, ohne Hintergrund für das gesamte Bild. Alternativ wählen Sie repräsentative Bereiche zu messen, die Regionen der Inkonsistenz durch Klick Rechteckigen Bereich, Ellipse Regionoder Trace Region , wählen eine Region und Aktiven Region anstatt gesamte ausschließen Bild auf Fenster Zeigen Statistiken zu einer Region (unter Analyse-Tools). Wenn eine repräsentative Region genutzt wird für Schwellwerte das Hellfeld-Bild, die gleiche Region für Messung des entsprechenden Bildes FITC verwendet werden soll. Es ist hilfreich, erfassen die spezifische räumlichen Statistiken (Links, Top, Breite, Höhe, Fläche, Umfang) dieser repräsentativen Region zugeordnet, so dass die gleiche Region gefunden werden und gemessen auf das entsprechende Bild, FITC.
Um eine Ansammlung von Daten auf der Festplatte zu verhindern, durch die den Computer langsamer wird, kann eine externe Festplatte für die Datenspeicherung erworben werden. Eine weitere Option für die Datenspeicherung und erleichtern den Datenaustausch ist die CyVerse-Bioinformatik-Plattform. Erstellen Sie ein Konto auf dem CyVerse-System, indem Sie auf http://www.cyverse.org/. Einmal eingeloggt, starten Sie die Entdeckung-Umgebung zu und wählen Sie dann "Log in CyVerse". Wählen Sie "Daten" und navigieren Sie zu du hast den Ordner. Wenn der Bild-Stack auf dem lokalen Computer ist, wählen Sie "hochladen" und dann "Einfache Hochladen von Desktop". Finden Sie die Image-Stapel-Datei und wählen Sie für den Upload. Die Datei oder einen Ordner kann mit Mitarbeitern geteilt werden, wenn sie ein CyVerse-Konto und Berechtigung erteilt werden. Freigeben des Ordners Data für die breite Öffentlichkeit erfordert, dass die Metadaten hinzugefügt werden für jede Datei mit CyVerse Normen zugelassen. Dieses Verfahren wird hier nicht diskutiert werden, weil dies nicht im Rahmen dieser Arbeit ist.
Offenlegungen
Die Autoren haben nichts preisgeben.
Danksagungen
Diese Arbeit wurde durch Zuschüsse aus dem National Institute für allgemeine medizinische Wissenschaft (NIGMS) (5P20GM103427), eine Komponente des National Institute of Health (NIH) möglich
Materialien
| Name | Company | Catalog Number | Comments |
| Ammonium Chloride, ACS | VWR | BDH9208-500G | Part of the minimal media composition |
| BioFlux 1000 48 Well Low Shear Plate | Fluxion Biosciences | 910-0047 | |
| BioFlux 1000Z Microfluidic Imaging System | Fluxion Biosciences | BF 1000Z | |
| Calcium Chloride Dihydride, ACD Grade | VWR | 97061-904 | Part of the minimal media composition |
| Dextrose, Anhydrous, ACS | VWR | BDH9230-500G | Part of the minimal media composition |
| Magnesium Sulfate ACS Grade | VWR | EM-MX0070-1 | Part of the minimal media composition |
| Potassium Phosphate Monobasic, ACS Grade | VWR | BDH9268-500G | Part of the minimal media composition |
| Pseudomonas Aeurginosa GFP | ATCC | 15692GFP | Pseudomonas aeurinosa bacterial strain PA01 with GFP modification used for this study. |
| Sodium Chloride, ACS | VWR | BDH9286-500G | Part of the minimal media composition |
| Sodium Phosphate, Monobasic, Anhydrous, Reagent Grade | VWR | 97061-942 | Part of the minimal media composition |
Referenzen
- Donlan, R. M., et al. Model System for Growing and Quantifying Streptococcus pneumoniae Biofilms In Situ and in Real Time. Applied and Environmental Microbiology. 70 (8), 4980-4988 (2004).
- Lührig, K., et al. Bacterial Community Analysis of Drinking Water Biofilms in Southern Sweden. Microbes and Environments. 30 (1), 99-107 (2015).
- Singh, R., Paul, D., Jain, R. Biofilms: implications in bioremediation. Trends in Microbiology. 14 (9), 389-397 (2006).
- Veerachamy, S., et al. Bacterial adherence and biofilm formation on medical implants: A review. Proceedings of the Institution of Mechanical Engineers, Part H: Journal of Engineering in Medicine. 228 (10), 1083-1099 (2014).
- Lyczak, J. B., Cannon, C. L., Pier, G. B. Establishment of Pseudomonas aeruginosa infection: lessons from a versatile opportunist. Microbes and Infection. 2 (9), 1051-1060 (2000).
- Percival, S. L., Knapp, J. S., Edyvean, R., Wales, D. S. Biofilm Development On Stainless Steel In Mains Water. Water Research. 32 (1), 243-253 (1998).
- Percival, S. L., Knapp, J. S., Wales, D. S., Edyvean, R. G. J. The effect of turbulent flow and surface roughness on biofilm formation in drinking water. Journal of Industrial Microbiology and Biotechnology. 22 (3), 152-159 (1999).
- Ling, F., Whitaker, R., LeChevallier, M. W., Liu, W. -. T. Drinking water microbiome assembly induced by water stagnation. The ISME Journal. , 1 (2018).
- Moss, B. Water pollution by agriculture. Philosophical Transactions of the Royal Society B: Biological Sciences. 363 (1491), 659-666 (2008).
- Schultz, M. P., Bendick, J. A., Holm, E. R., Hertel, W. M. Economic impact of biofouling on a naval surface ship. Biofouling. 27 (1), 87-98 (2011).
- Tornero, V., Hanke, G. Chemical contaminants entering the marine environment from sea-based sources: A review with a focus on European seas. Marine Pollution Bulletin. 112 (1-2), 17-38 (2016).
- LaBauve, A. E., Wargo, M. J. Growth and Laboratory Maintenance of Pseudomonas aeruginosa. Current Protocols in Microbiology. 25 (1), 1-8 (2012).
- He, J., et al. The broad host range pathogen Pseudomonas aeruginosa strain PA14 carries two pathogenicity islands harboring plant and animal virulence genes. Proceedings of the National Academy of Sciences of the United States of America. 101 (8), 2530-2535 (2004).
- Shaner, N. C., Steinbach, P. A., Tsien, R. Y. A guide to choosing fluorescent proteins. Nature Methods. 2 (12), 905-909 (2005).
- Zhang, G., Gurtu, V., Kain, S. R. An Enhanced Green Fluorescent Protein Allows Sensitive Detection of Gene Transfer in Mammalian Cells. Biochemical and Biophysical Research Communications. 227 (3), 707-711 (1996).
- Patterson, G. H., Knobel, S. M., Sharif, W. D., Kain, S. R., Piston, D. W. Use of the green fluorescent protein and its mutants in quantitative fluorescence microscopy. Biophysical Journal. 73 (5), 2782-2790 (1997).
- Chalfie, M., Tu, Y., Euskirche, G., Ward, W. W., Prashert, D. C. Green fluorescent protein as a marker for gene expression. Science. 263 (5148), 802-805 (1994).
- Heim, R., Prasher, D., Tsien, R. Wavelength mutations and posttranslational autoxidation of green fluorescent protein. Proceedings of the National Academy of Sciences of the United States of America. 91, 12501-12504 (1994).
- Bakke, R., Kommedal, R., Kalvenes, S. Quantification of biofilm accumulation by an optical approach. Journal of Microbiological Methods. 44 (1), 13-26 (2001).
- Heydorn, A., et al. Statistical analysis of Pseudomonas aeruginosa biofilm development: impact of mutations in genes involved in twitching motility, cell-to-cell signaling, and stationary-phase sigma factor expression. Applied and Environmental Microbiology. 68 (4), 2008-2017 (2002).
- Wilson, E., et al. Using Fluorescence Intensity of Enhanced Green Fluorescent Protein to Quantify Pseudomonas aeruginosa. Chemosensors. 6 (21), (2018).
- Fang, H., Chen, Y., Huang, L., He, G. Analysis of biofilm bacterial communities under different shear stresses using size-fractionated sediment. Scientific Reports. 7 (1), 1299 (2017).
- Saunders, K. A., Greenman, J. The formation of mixed culture biofilms of oral species along a gradient of shear stress. Journal of Applied Microbiology. 89 (4), 564-572 (2001).
- Valquier-Flynn, H., Wilson, C. L., Holmes, A. E., Wentworth, C. D. Growth Rate of Pseudomonas aeruginosa Biofilms on Slippery Butyl Methacrylate-Co-Ethylene Dimethacrylate (BMA-EDMA), Glass and Polycarbonate Surfaces. Journal of Biotechnology & Biomaterials. 7 (4), (2017).
- Trulear, M. G., Characklis, W. G. Dynamics of biofilm processes. Journal (Water Pollution Control Federation). 54 (9), 1288-1301 (1982).
- Machineni, L., Rajapantul, A., Nandamuri, V., Pawar, P. D. Influence of Nutrient Availability and Quorum Sensing on the Formation of Metabolically Inactive Microcolonies Within Structurally Heterogeneous Bacterial Biofilms: An Individual-Based 3D Cellular Automata Model. Bulletin of Mathematical Biology. 79 (3), 594-618 (2017).
- Jiao, Y., et al. Identification of Biofilm Matrix-Associated Proteins from an Acid Mine Drainage Microbial Community. Applied and Environmental Microbiology. 77 (15), 5230-5237 (2011).
- Flemming, H. C., Wingender, J. The Biofilm Matrix. Nature Reviews Microbiology. 8 (9), 632-633 (2010).
- Yang, X., Beyenal, H., Harkin, G., Lewandowski, Z. Quantifying biofilm structure using image analysis. Journal of Microbiological Methods. 39 (2), 109-119 (2000).
- Heydorn, A., et al. Quantification of biofilm structures by the novel computer program COMSTAT. Microbiology. 146 (10), 2395-2407 (2000).
- Azeredo, J., et al. Critical review on biofilm methods. Critical Reviews in Microbiology. 43 (3), 313-351 (2017).
- Goeres, D. M., et al. A method for growing a biofilm under low shear at the air liquid interface using the drip flow biofilm reactor. Nature Protocols. 4 (3), 783-788 (2009).
- Goeres, D. M., et al. Statistical assessment of a laboratory method for growing biofilms. Microbiology. 151, 757-762 (2005).
- Swartz, K., Stephenson, R., Hernandez, M., Jambang, N., Boles, B. R. The use of Drip Flow and Rotating Disk Reactors for Staphylococcus aureus biofilm analysis. Journal of Visual Experiments. (46), e2470 (2010).
- Werner, E., et al. Stratified Growth in Pseudomonas aeruginosa Biofilms. Applied and Environmental Microbiology. 70 (10), 6188-6196 (2004).
- Wilson, C., et al. The Quantitative Assessment of Pseudomonas aeruginosa (PA)14 Biofilm Surface Coverage on Slippery Liquid Infused Polymer Surfaces (SLIPS). International Journal of Nanotechnology in Medicine & Engineering. 3 (3), 35-42 (2018).
- Samarian, D. S., Jakubovics, N. S., Luo, T. L., Rickard, A. H. Use of a High-throughput In Vitro Microfluidic System to Develop Oral Multi-species Biofilms. Journal of Visualized Experiments. (94), 52467 (2014).
- Rusconi, R., Garren, M., Stocker, R. Microfluidics Expanding the Frontiers of Microbial Ecology. Annual Review of Biophysics. 43, 65-91 (2014).
- Kim, J., Park, H. -. D., Chung, S. Microfluidic Approaches to Bacterial Biofilm Formation. Molecules. 17 (8), 9818-9834 (2012).
- Mosier, A. P., Cady, N. C. Analysis of bacterial surface interactions using microfluidic systems. Science Progress. 94 (4), 431-450 (2011).
- Nobile, C. J., Johnson, A. D. Candida albicans Biofilms and Human Disease. Annual Review of Microbiology. 69, 71-92 (2015).
- Chandra, J., Kuhn, D. M., Mukherjee, P. K., Hoyer, L. L., McCormick, T., Ghannoum, M. A. Biofilm Formation by the Fungal Pathogen Candida albicans: Development, Architecture, and Drug Resistance. Journal of Bacteriology. 183 (18), 5385-5394 (2001).
- Vasconcelos, M. A., et al. Effect of Algae and Plant Lectins on Planktonic Growth and Biofilm Formation in Clinically Relevant Bacteria and Yeasts. BioMed Research International. 2014, 9 (2014).
- Haralick, R. M., Shanmugam, K., Dinstein, I. Textural Features for Image Classification. IEEE Transactions on Systems, Man, and Cybernetics. 3 (6), 610-621 (1973).
- . Biofilm Morphology Suite Available from: https://github.com/cdwentworth/Biofilm-Morphology-Suite.git (2018)
- . Cyverse Available from: https://user.cyverse.org/services/mine (2018)
- Benoit, M. R., Conant, C. G., Ionescu-Zanetti, C., Schwartz, M., Matin, A. New Device for High-Throughput Viability Screening of Flow Biofilms. Applied and Environmental Microbiology. 76 (13), 4136-4142 (2010).
- Ding, A. M., Palmer, R. J., Cisar, J. O., Kolenbrander, P. E. Shear-Enhanced Oral Microbial Adhesion. Applied and Environmental Microbiology. 76 (4), 1294-1297 (2010).
- Diaz De Rienzo, M. A., Stevenson, P. S., Marchant, R., Banat, I. M. Effect of biosurfactants on Pseudomonas aeruginosa and Staphylococcus aureus biofilms in a BioFlux channel. Applied Microbiology and Biotechnology. 100 (13), 5773-5779 (2016).
- Moormeier, D. E., Bayles, K. W. Staphylococcus aureus Biofilm: A Complex Developmental Organism. Molecular Microbiology. 104 (3), 365-376 (2017).
- Tremblay, Y. D. N., Vogeleer, P., Jacques, M., Harel, J. High-Throughput Microfluidic Method To Study Biofilm Formation and Host-Pathogen Interactions in Pathogenic Escherichia coli. Applied and Environmental Microbiology. 81 (8), 2827-2840 (2015).
- Nobbs, A. H., Lamont, R. J., Jenkinson, H. F. Streptococcus Adherence and Colonization. Microbiology and Molecular Biology Reviews. 73 (3), 407-450 (2009).
- Díez-Aguilar, M., Morosini, M. I., Köksal, E., Oliver, A., Ekkelenkamp, M., Cantón, R. Use of Calgary and Microfluidic BioFlux Systems To Test the Activity of Fosfomycin and Tobramycin Alone and in Combination against Cystic Fibrosis Pseudomonas aeruginosa Biofilms. Antimicrobial Agents and Chemotherapy. 62 (1), 01650-01717 (2018).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten