Method Article
水生甲壳虫中的RNA干扰是特定发育时间点操纵基因表达的有力工具
摘要
RNA干扰是一种广泛适用、强大的技术,用于在特定发育阶段操纵基因表达。在这里,我们描述了在水生潜水甲虫热力学马莫拉图斯中实施这项技术的必要 步骤,从基因序列的获取到影响结构或行为的基因的敲击。
摘要
RNA干扰(RNAi)仍然是一种强大的技术,允许通过mRNA降解有针对性地减少基因表达。这种技术适用于各种各样的生物体,在物种丰富的秩序中效率很高。在这里,我们总结了在新型生物体中开发这种技术的必要步骤,并说明其在水生潜水甲虫热力学丸不同发育阶段的应用。目标基因序列可以通过与已知基因组学或新基因组学的近亲相比,以经济高效地获得转录组。候选基因克隆利用特定的克隆载体(pCR4-TOPO质粒),它允许合成双链RNA(dsRNA)的任何基因使用一个单一的共质基因。合成的dsRNA可以注射到胚胎中,用于早期发育过程或幼虫,用于后期发育过程。然后,我们说明RNAi如何可以注入水生幼虫使用固定在阿加罗斯。为了演示这项技术,我们提供了RNAi实验的几个示例,用预测的表型生成特定的击倒。具体来说,晒黑基因乳面2的RNAi在幼虫和成人中都会产生角质光,而眼睛色素沉着基因白的RNAi在眼管中产生增white光/缺乏色素沉着。此外,关键透镜蛋白的敲击导致幼虫的光学缺陷和猎杀猎物的能力下降。综合起来,这些结果体现了RNAi作为研究生物体形态模式和行为特征的工具,只有转录数据库。
引言
特定基因如何促进不同特征的进化是生物学中一个令人兴奋的话题。在过去的几十年里,在解剖一些模型生物体的发育过程的遗传基础方面取得了很大进展,如线虫卡诺哈布迪炎、果蝇果蝇果蝇黑色素和家鼠Musmusculus1。1最近,强大的基因编辑技术的发明,如聚类定期间穿短回语重复(CRISPR)/Cas92,提供了改变非模型生物体的遗传密码的能力(见示例3,4)。,4因此,对各种生物体的基因研究激增,而这些生物以前从未通过分子技术接触过。考虑到我们动物王国的巨大多样性,许多有趣的特征或特征差异,只代表特定物种,这一进展已经使进化-发育生物学("evo-devo")相关工作成为激动人心的时刻。然而,非模型生物体可用的基因组编辑技术在可应用的发育时间点方面相对有限,因此很难辨别与特定基因在任何特征中的作用相关的时间属性。此外,转基因技术往往仅限于对生存无关紧要的基因(即,其敲除不会导致致命性)。因此,虽然基因编辑技术已开始流行,但仍需要有效的技术,这些技术适用于特定发育时间点的各种不同生物体,并促进部分敲击(而不是完全丧失功能)。在这里,我们提请注意RNA干扰(RNAi),一种有点过时但强大的基因敲击技术5,作为基因编辑的协同方法特别有价值。具体来说,我们开发了一些程序,允许RNAi应用于水生潜水甲虫作为例子,说明该技术的实现,从获取必要的分子序列到成功将双链RNA(dsRNA)注射到卵子和幼虫中。
基于RNAi的基因敲击利用了生物体的先天防御机制,其中dsRNA分子促进入侵核酸序列的沉默,如病毒和转位子6。简言之,dsRNA被引入细胞,由D者酶切成20~25核苷酸片段。然后,这些片段激活RNA诱导沉默复合物(RISC)的形成,该复合物使用导链在特定位点与目标mRNA结合,从而抑制目标mRNA的形成。这个过程最终导致mRNA降解,从而干扰mRNA转化为各自的蛋白质6。因此,这里介绍的基于RNAi的基因敲击技术依赖于dsRNA的注射。对于动物模型,这项技术最初是在C.elegans7和D.黑色素8中开发的,但后来在非模型生物体9,10,中成为一种强大的功能遗传工具。RNAi由于其在一些昆虫中具有很高的传染性,甚至可以应用于害虫管理11。
作为研究工具,RNAi用于研究关键分子发育途径在非传统昆虫模型中的作用。例如,在面粉甲虫三叶虫Tribolium castaneum的RNAi在确定保存基因对甲虫中特定特性的贡献方面起了多大作用,例如为特殊形状的翅膀12、13、14,13,14和眼睛15、16,的发展。在T.castaneum中操作的技术已经很好地描述了17,并依赖于在粘性表面上固定相对干燥的鸡蛋和幼虫的能力。然而,这种固定对于水生生物的湿发育形式是不可能的,如太阳爆发潜水甲壳虫。和许多非传统模型生物体的情况一样,它缺乏一个带注释的基因组。为了操纵任何没有基因组的生物体的基因表达,一个合理且具有成本效益的第一步是生成转录组,并基于与相关但更成熟的模型生物体的序列相似性,确定感兴趣的表达基因的假设核苷酸序列,在这种情况下,主要是三叶草(Coleoptera)和果蝇基因。
在这里,为了演示RNAi如何可用于水生生物,我们首先讨论了RNA提取和转录组生成和组装的协议和软件,这些协议和软件允许识别特定的靶向基因序列。然后,我们总结了合成基因特异性 dsRNA 的必要步骤。随后,我们说明了如何在水生环境中注射卵子,并演示培养发育胚胎的孵化方案。此外,我们展示在注射过程中如何使用阿加罗斯凝胶完全固定幼虫,这种技术在各种手术中通常很有用,可以应用于各种节肢动物。为了演示RNAi如何应用于不同的发育阶段,我们包括一个我们沉默的胚胎中眼睛色素沉着基因白色的例子。此外,我们描述了一个的例子,其中晒黑基因乳酸laccase22(lac2)在第二幼虫星(影响第三幼虫星的幼虫)和第三幼虫星(影响成人)期间均被沉默。最后,我们证明注射浓度较低的dsRNA会导致部分击倒,这表明该技术也可以应用于已知功能丧失为致命基因的基因。
研究方案
1. RNA分离和 de novo 转录组组装
- 使用专为富含脂质的组织设计的RNA隔离试剂盒(见材料表),从晚期第三星潜水甲虫幼虫眼管和成年Table of Materials甲虫进行总RNA分离。从T.马莫拉图斯分离出总RNA,然后按照前面描述的方法18对它进行测序。
- 德诺沃转录本组装
注:要组装新转录组,可以使用各种生物信息学平台( 参见材料表)。这些平台是市售的,在某些情况下,作为基于 Unix 的命令行脚本存在。由于组装是 de novo,建议在两个平台上独立组装转录组,并比较结果以排除误报或误报。- 要开始组装转录组,请将原始读物上传到相应的生物信息学平台上。
注意:这些文件通常是压缩的,格式为 FASTQSANGER。广州。无需解压缩文件,因为下一步将接受此格式。 - 使用 Trimmomatic 命令行修剪原始读取以删除低于默认阈值的适配器序列或短读。解压缩修剪后的原始读取;文件现在将采用 FASTQ 格式。串联每个示例的 FASTQ 原始读取,以获取已准备好进行新程序集的文件。
- 使用任何命令行脚本组装串联的原始读取 de novo,该命令行脚本可以组装转录组 de novo。保存组装的转录组,该转录组现在将有一个包含其各自序列的连体列表,作为 FASTA 文件。为了扩大组装的覆盖范围,为同一物种组合和组装多个转录组。
注:这个过程增加了产生更准确的连体的机会,如果低拷贝数基因感兴趣,这一点尤其重要。 - 组装完成后,通过计算覆盖率分数来评估转录组是否完整(覆盖评估软件可免费获得, 请参阅材料表)。
注:覆盖分数为 +gt;75% 的转录组被认为是不错的。然而,这个分数的相关性取决于转录组生成的原因。例如,如果转录组用于识别低拷贝数基因,则分数 >85% 更好,因为它表示覆盖范围更大。 - 使用基本的局部对齐搜索工具(BLAST;参见材料表)对 de novo 组装的转 录组进行注释。
注:对于这个实验,转录组主要对照一个已知的 果蝇蛋白 数据库和已知的甲虫蛋白数据库进行注释。注释列表可以下载为电子表格文件,指示序列与其他生物体蛋白质相似的连体/连音可以下载为 FASTA 文件。 - 确定感兴趣的蛋白质的连数/数,并提取核苷酸序列。使用 BLAST 确定感兴趣的注附核苷酸序列中是否存在蛋白质特异性保守区域(如家用盒域)。检查具有相同注释的其他连音核苷酸序列重叠,以生成基因特异性转录的序列。
注:识别序列重叠的连体通常不是所有基因都不可能,尤其是低拷贝数基因。 - 如果需要该基因的全长序列,请从具有最高序列相似性的连体开始,作为使用 cDNA 端的 3° 和 5° 快速扩增等技术识别整个成熟转录笔核苷酸序列的起始点 (RACE19).
- 按照步骤 1.2.4=1.2.7 对从第二个平台获取的程序集进行注释。
注:理想情况下,结果应该与感兴趣的蛋白质相似;但是,不同平台的覆盖范围在一定程度上会有所不同。 - 使用组装的转录组合成物种特异性 dsRNA,如第 2 节所述。
- 要开始组装转录组,请将原始读物上传到相应的生物信息学平台上。
2. 克隆和基因特异性 dsRNA 合成
注:基因特异性克隆和dsRNA合成已详细描述T.卡斯塔纽姆2020,21,22。,21,22以下步骤是简要概述。
- 克隆、细菌转化和质粒测序
- 从生物体中分离总RNA(如步骤1.1所述),并使用逆转录试剂盒创建互补DNA(cDNA)( 参见材料表)。从对感兴趣的基因特定的连体中识别一个或两个 100–1000 bp 核苷酸序列。
- 设计跨越整个核苷酸的底端对,并通过聚合酶链反应(PCR)放大序列。在结束添加额外的 30 分钟步长,在放大的 DNA 产品中创建 3+ 腺苷悬垂。使用 PCR 纯化套件净化本产品( 参见材料表)。
- 按照供应商提供的克隆试剂盒协议,将纯化产品克隆成 pCR4-TOPO 质粒载体(参见材料表)。 Table of Materials使用热休克治疗,将克隆质粒转化为称职的细菌细胞(应变:大肠杆菌DH10B),按照供应商的称职细胞协议(见材料表),并筛选成功转化的细胞。
注:带菌落的板材可包裹在石蜡膜中,以保留水分,并在4°C下储存长达一个月。 - 在37°C和200 rpm的摇床培养箱中,使用10μL移液器尖端和含有安西林(50μg/mL)的利生根汤(LB)中培养转化细胞的菌落,持续24小时。
注:对于长期储存,培养细胞可用100%甘油稀释1:1,并在-80°C冷冻为甘油储存。 - 使用微型表分离试剂盒分离含有该培养物感兴趣的基因的质粒(参见材料表)。在评估屈服分光光度后,将分离的质粒(微活)储存在-20°C。具体地说,测量 260 nm、280 nm 和 230 nm 的样品吸光度。计算用于量化DNA的A 260/A280比率,以及用于量化 DNA 纯度的 A260 /A 230比率。260
注:260/280比率1.8被普遍接受为足够纯的DNA,低于1.5的比率表示存在残留萃取试剂,如苯酚。260/230 比率应大于或等于 260/280 比率。较低的比率表示残留试剂污染。产量通常为 100 至 500 ng/μL。 - 对分离的微片进行测序,确认基因特异性片段已成功整合,在质粒中5°T7和3°T3启动子区域之间;这可以使用通用T7和T3测序底像22完成。使用具有基因特异性序列的微头,用于 dsRNA 制备。
- 从生物体中分离总RNA(如步骤1.1所述),并使用逆转录试剂盒创建互补DNA(cDNA)( 参见材料表)。从对感兴趣的基因特定的连体中识别一个或两个 100–1000 bp 核苷酸序列。
- PCR扩增和体外 dsRNA 合成
- PCR 扩增和产品纯化
- 设计具有 T7 启动子序列的质粒特异性或基因特异性底转(详情见参考 22), 以放大插入基因的线性片段。通过设置至少 10 个 20 μL 的反应来优化产量。
注:所得产品侧翼有两个20bp T7聚合酶结合位。 - 使用 PCR 产品纯化套件(参见 材料表)按照供应商的基于列的洗脱协议进行净化 PCR 产品。要增加产量,请使用相同的洗脱剂重复最后的洗脱步骤达3次。以光度测量方式评估产量分光度。
注:产量通常为100至700纳克/μL。这种纯化产品可以储存在-20°C,直到进一步使用。
- 设计具有 T7 启动子序列的质粒特异性或基因特异性底转(详情见参考 22), 以放大插入基因的线性片段。通过设置至少 10 个 20 μL 的反应来优化产量。
- 体外 dsRNA 合成和纯化
- 使用基因特异性PCR纯化产品根据选择的dsRNA合成试剂盒的协议在体外合成dsRNA。如有必要,延长潜伏时间,增加 dsRNA 产量。
- 根据反应混合物的不透明度评估反应的进展(dsRNA 产品的量越高,浊度越高)。在孵育结束时,使用DNase治疗停止反应。为此,从2U/μL的库存中加入1 μL,加入反应混合物。将此处理延长至 1~2 小时,以确保模板 DNA 的最佳降解。
- 使用纯化套件或通过以下步骤净化 dsRNA。
- 用115μL无核酸水稀释所得产品,加入15μL的3M醋酸钠。将2体积的冰冷100%乙醇加入到这种反应混合物中,并在-20°C下将dsRNA沉淀过夜。
注:此时,样品可以安全地存储,而不会影响最终产量。 - 在 0 °C 下将沉淀物在 9000 x g 下离心 20 分钟, 然后丢弃上一代。用冰冷的70%乙醇清洗产品一次,在13000xg下离心机15 分钟。
- 取出上流液,风干颗粒5~15分钟。将颗粒重新在高达40μL的无核酸酶水中(此体积可根据分离的颗粒大小进行调整),并评估产量和纯度分光度,如2.1.4所述。
- 将纯化的 dsRNA 储存在 -20 °C 下,直到进一步使用。在所需的发育阶段使用纯化的 dsRNA 进行注射。
- 用115μL无核酸水稀释所得产品,加入15μL的3M醋酸钠。将2体积的冰冷100%乙醇加入到这种反应混合物中,并在-20°C下将dsRNA沉淀过夜。
- PCR 扩增和产品纯化
3. 收集和制备用于 dsRNA 注射的早期 T. 马莫拉图斯胚胎
- 准备一个糖板,用于 孵育T.玛莫拉图斯胚 胎。
- 首先,在100 mL的自溶蒸馏水(2%agarose)中溶解20毫克低熔化的阿加糖粉,然后在微波炉中煮沸这种混合物,直到获得明确的溶液。小心热溶液,因为气泡会导致其溢出。
- 将溶液放入小培养皿中。要在红玫瑰板表面制作凹槽(用于容纳胚胎),在液体加糖表面凝固之前,将三个 1~2 mm 宽的塑料管(如塑料转移移液器的尖端)放在其表面。
- 一旦气糖凝固,使用一对钝钳去除这些管子。使用 P1000 微管加入 500 μL 的自体蒸馏水,以覆盖气糖中的这些压痕。
- 使用精细的天然发刷,从充满自洗蒸馏水的玻璃腔盘中从甲虫筑巢点收集5~8小时老的 T.Marmoratus 胚胎。经常监测筑巢地点,以确保获得的卵子具有适当的年龄。
- 使用精细的解剖钳在立体显微镜下对胚胎进行解剖。为此,在显微镜下(在所需的放大倍率下)将胆小子可视化,以适当的角度定位光源,然后用锋利的钳子从两侧抓住胆小子,将其撕开,然后轻轻地将胚胎滑出。由于有两个图层,请确保同时删除这两个图层。
- 使用精细的天然发刷,小心地将胚胎从玻璃腔盘中转移到红糖盘中,并将它们排列在立体显微镜下的凹槽中。在这个过程中要小心,因为被肢解的胚胎非常脆弱。使用准备好的胚胎进行 dsRNA 注射。
注:稍厚的端是胚胎的一侧,头部将在其中发育。
4. 早期T.玛莫拉图斯胚胎 的dsRNA微 注射
- 要用基因特异性dsRNA注射早期胚胎,请使用微注射针拉拔器准备微注射针( 参见材料表)。在立体显微镜下可视化针尖时,使用一对细钳来打破针尖,从而形成锋利的边缘。理想情况下,对角线断开针尖,使一侧保持更锋利的边缘,从而允许它进入胚胎,同时造成最小的伤害。
- 在冰上解冻纯化库存 dsRNA 溶液,加入 10x 注射缓冲液 1 μL,用双蒸馏水将其顶上,使 10 μL 的注射溶液。对于注射,dsRNA浓度为1μg/μL通常是合适的,但可以根据结果动物的表型严重程度进行调整。
注:通过加入 10 μL 的 0.1 M 磷酸钠缓冲液(通过混合 8.5 mL 的 1 M Na2HPO4 和 1.5 mL 的 1,制备 1 mL 的 10x 注射缓冲液 22 M NaH2PO4 在 25 °C 时调整为 pH 7.6,100 μL 为 0.5 M KCl),100 μL 食品染料和 790 μL 双蒸馏水。22- 使用 P10 移液器,使用工作 dsRNA 溶液回填微注射针。溶液充满针头后,将其连接到使用压力喷射技术的微注射系统(参见材料表) 的 微针支架上。
- 打开微注射系统和加压空气供应。使用微注射系统的微价,将喷射压力调整为15 psi。使用时间调整旋钮调整喷射持续时间(设置为"秒")。
- 由于注射持续时间取决于所需的注射量,如有必要,在每次注射前调整微注射系统的此参数。对早期T.玛莫拉图斯胚胎使用1~2nL 的注射 体积。
注:注射量越高,往往干扰胚胎存活率。
- 由于注射持续时间取决于所需的注射量,如有必要,在每次注射前调整微注射系统的此参数。对早期T.玛莫拉图斯胚胎使用1~2nL 的注射 体积。
- 要测量微注射系统分配的注射液的体积,通过评估注射到空气中时在针头尖端形成的液泡的体积来校准注射针。对于 T. marmoratus 胚胎,使用 100~200 μm 的最佳气泡大小。如果注射持续时间为 ±3 s,注射针的质量良好,这一点可以在 15 psi 时实现。
- 将针头和含有胚胎的红糖板在立体显微镜下对齐,使针头以 45° 和 60° 的角度接近胚胎。
- 使用微操纵器,在通过立体显微镜监测进度的同时,缓慢移动微声。一旦针头接触胚胎表面,小心地向前移动针头,直到它刺穿胚胎表面。不要用针尖深入穿孔胚胎,因为这会影响胚胎的生存。为了减少变异性,在胚胎中间注射。
- 一旦针头进入胚胎内,小心地按下微注射器的注射按钮,将剂量的 dsRNA 送到胚胎。注射1~2nL的dSRNA后,慢慢从胚胎中收回针头,这样不会导致胚胎破裂。
- 以类似的方式注射其他胚胎。如果微注射针在手术过程中堵塞,则通过增加注射压力和持续时间来解除阻止。通过注射部位存在有色斑来直观地识别成功注射的胚胎(因为注射缓冲液含有食物色素)。
- 小心地将带注射胚胎的盘子放在湿度室中。
- 要建造这样的房间,请拿任何带盖子的塑料盒,用70%的乙醇消毒,让其干燥,并在底部将浸泡在自 <3> <3>化水中的组织放在底部,以提供潮湿的环境。将此湿度室放在温度适当(本例中为 25°C)且光循环为 14 小时、暗于 10 小时处的培养箱中。
- 允许注射胚胎发育成第一个星体幼虫,这需要4~5天。每天使用立体显微镜监测胚胎发育的进展情况,以识别图2中描述的形态可见的击倒 表型。
- 使用高分辨率摄像机拍摄数字图像,记录此进度。取出死胚胎,每天补充黄玫瑰板上的水,以避免干燥和微生物污染在孵化期间可能扩散到其他存活的胚胎。
- 对 dsRNA 注射进行适当的控制,包括缓冲注射、根据感兴趣的基因的感知链合成的 dsRNA 注射,以及针对目标生物体内不存在的序列合成的 dsRNA 注射。确认RNAi击倒使用额外的分子方法22。
5. 为 dsRNA 注射 准备 T. 马莫拉图斯幼虫
注:与早期胚胎 不同,T.玛 莫拉图斯幼虫相对坚固,可以注射更大的体积。例如,第二星可以注入高达2μL的dsRNA工作溶液,第三星可注射至少3μL,而不会对存活率产生明显的负面影响。注射dsRNA,通过将幼虫嵌入到阿加罗斯中,使幼虫固定不动是有帮助的。
- 在收集幼虫之前,熔化2%的气糖,并在60~70°C的水浴中保持液态。将融化的2%的阿加罗斯倒入小培养皿中,然后冷却直到凝固,准备固定盘。使用钝钳在阿加罗斯板中制作一个浅槽(大致相当于要嵌入的节肢动物的大小),用于注射的每个动物。
- 在适当的发育阶段(分析所需的阶段之前的一个阶段)收集幼年幼虫。为此,请小心地将水从含有幼虫的培养杯中排出,然后按照以下步骤操作。
- 在冰上麻醉(小心地从水杯中挑出幼虫,轻轻地放在冰上),直到幼虫没有明显的运动。使用钝的,软端钳小心地将幼虫从冰上提起,并放在固定阶段,其颈部和身体在凹槽中,但它的尾巴位于阿加罗斯表面上方,这样它便不会被覆盖,这样幼虫才能继续通过它的尾尖呼吸呼吸。
- 用一层厚厚的气糖覆盖幼虫,这种加糖仍然是液体,但热不危险。如果不小心被盖住,请使用钳子将尾巴和下方的两段从气管中释放。一旦阿加罗斯凝固,使用幼虫进行dsRNA注射。
6. T. 马莫拉图斯幼虫 的 dsRNA 微注射
- 使用所需量的基因特异性 dsRNA 工作溶液准备和回填微注射针(如步骤 4.1~4.2 中所述)。将针头连接到连接到手动控制的微注射注射器的针架上。在连接针头之前,将注射器柱塞一直拉出,以避免在中间过程中耗尽注射压力。
- 定位固定化幼虫,使注射部位位于第三和第四体板段之间的侧身位置,以相对平坦的角度(几乎与舞台平行)与微注射针的轨迹对齐。
注:将针头和幼虫插入此位置非常重要,因为它为针尖提供最小阻力路径,以最小的伤害打穿幼虫。 - 一旦针头和幼虫被定位,小心地将针尖移入幼虫,同时通过立体显微镜监测进展。
- 尖端穿孔组织后,缓慢而小心地向注射器施加压力。通过观察显微镜下针头中彩色注射液的运动来调节注射压力。
- 注射后小心地收回针头。使用软钳剥开,从而从红玫瑰中释放幼虫。将幼虫与水一起转移回容器(室温下),在25°u201228°C的培养箱或培养室中进一步发育,光循环为14小时,暗10小时。
- 采用适当的控制注射和击倒定量,如步骤 4.10 所述。
结果
使用上述协议,我们击倒了三个不同的基因,即白色,laccase2(lac2)和Lens3(表1),在太阳爆发潜水甲壳虫T.马莫拉图斯的各种不同发育阶段。lac2我们在胚胎形成的早期注射了dsRNA(图1A),在T.Marmoratus中进行了RNAi。由于一些胚胎不能存活下来的过程,并变成坏死(图1B),他们需要删除,以保持剩余的胚胎健康。这里的例子是对白色基因的 dsRNA注射。这种基因在果蝇中是众所周知的,是参与吸收和储存眼色素23前体的前体的三个ATP结合盒(ABC)运输者之一。因此,其功能丧失导致一种未发气的白眼表型。我们的结果表明,针对T.Marmoratus胚胎中正交white白基因的dsRNA注射会导致新兴幼虫眼睛色素沉着的丧失。在这种情况下,野生型幼虫的特点是眼睛色素严重,而白色RNAi的敲击会导致不同程度的减少,甚至完全消除眼睛色素。总的来说,我们观察到34%的存活胚胎(n = 35)眼睛着色至少有些减少。图 2A比较了眼睛颜色稍浅的控制个体和眼睛颜色稍浅的个体。图 2B显示了个体中更严重的击倒,其中群集的腹眼(眼睛 2+5)完全未感染,而后眼仍然表现出一些色素沉着。这些差异突出了敲击的效率如何因区域而异,这可能与密集组织中 dsRNA 渗透效果的差异有关。另一个个体显示所有眼睛基本上完全失去色素(图2C)。
为了研究RNAi在T.Marmoratus的幼虫阶段的效果如何,我们注射了dsRNA瞄准的tannic基因lac2进入第二个星幼虫前几天,他们是由于摩尔特到第三星(图3),并评估了对第三星幼虫的切分着色的影响。Lac2 是一种苯酚酶,氧化结合蛋白质,使蛋白质不溶,更硬,更暗。在面粉甲虫T.卡斯塔内姆的敲击已被证明导致较浅的彩色个体在低剂量,但被认为是致命的高剂量24。图4说明,这种治疗也导致较浅的太阳爆发潜水甲壳虫幼虫。具体来说,在这个实验中,75%的存活注射幼虫(n = 12)减少了色素沉着(相比之下,对照组为0%)。图 4A显示了一个相对轻度脱毛的个体,而图 4C显示了一个T. marmoratus幼虫的头部,其中角质层的黑暗着色几乎不存在。脱皮对于这些幼虫的典型中央黑暗图案尤为明显,而这种模式在控制注射个体中仍然清晰可见(图4B)。此外,观察到尾气管的闪电,如图4D所示。在lac2 dsRNA 被注射到第三个星幼虫中的情况下,获得较轻的成年个体(图 5)。请注意,击倒甲虫的翅膀有些变形,可能是由于其不寻常的柔软性。
除了改变形态特征外,还可以使用RNAi来靶向影响行为的基因。为了证明这一点,我们对一个关键透镜蛋白编码 基因 Lens318进行了RNAi,并注射到第二个星幼虫中,以影响第三星幼虫眼睛的光学特性。在这里观察到的对镜头的任何影响都可能是因为 T.Marmoratus幼虫 的眼睛在这个转变中经历了主要的眼睛生长,这也涉及到镜头25的重大光学变化。RNAi 在这个实验中的敲击效率很高。通过 qPCR 进行验证的成功率为 100%;在13个被测试个体中,有12人被击倒到控制个体表达水平的10%以下,其余个体的表达水平为控制水平的17%(未发表的观察)。在表型水平上,只有一些个体受到严重阻碍或无法捕捉猎物, 如图6所示,对于 从非常近距离反复接近猎物但始终超拍猎物的个人。
表1:白色、乳酸和 Lens3 蛋白的底漆序列和安普利素。请点击这里下载此表。
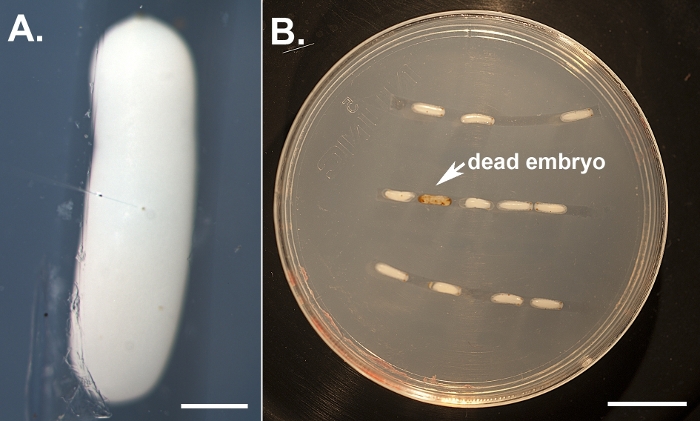
图1:胚胎注射图。(A) 在阿加罗斯板上排列的脱卵胚胎,使用填充 dsRNA 和含食物染料注射缓冲液的微注射针在中心附近注射。比例线表示 500 μm。(B) 有更严重击倒的个人.注射的胚胎保存在湿度室中,每天进行监测,以评分表型并清除死亡个体(变成棕色)。比例线代表 5 毫米 。请单击此处查看此图的较大版本。
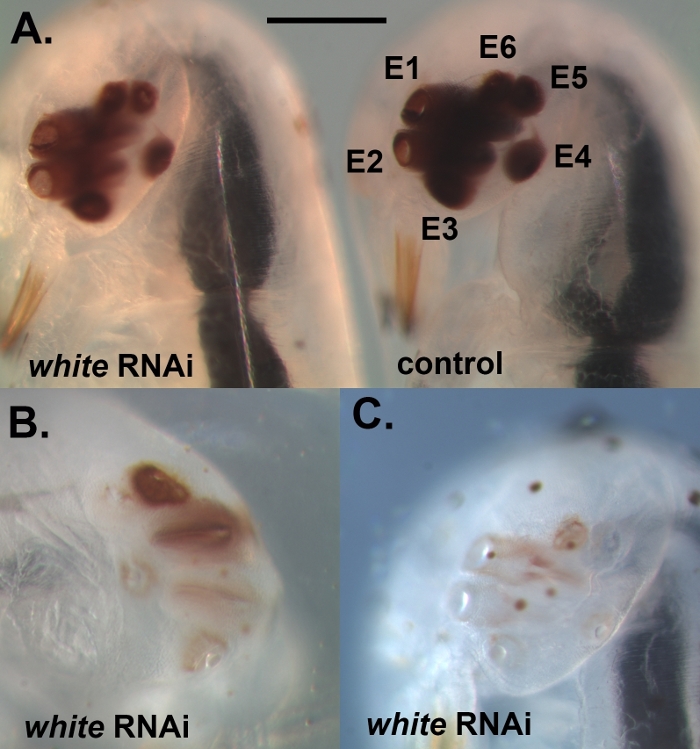
图2:注射dsRNA对白的完全发育的胚胎的例子。 (A) 眼睛色素沉着减少(左)和控制注射个体(右)的比较。E1+E6 指眼睛 1+6。(B) 具有更严重击倒表型的个人,说明有时,集群内一些眼睛受到击倒的影响比其他人更严重。(C) 眼睛色素沉着几乎完全丧失的个人。比例线表示 200 μm;面板 B 和 C 以相同的比例表示。请单击此处查看此图的较大版本。

图3:幼虫注射图。(A) 几个幼虫通过嵌入2%的气糖中,其尾尖远离任何气糖而固定。(B) 微选择含有放置的注射溶液,使其尖端可以穿透两段之间的细膜。(C) 注射后注射部位可见蓝色注射染料.比例线代表 1 厘米 。请单击此处查看此图的较大版本。

图4:RNAi的 laccase2 应用于第二星幼虫,导致第三星幼虫的角质层着色降低。(A) 与控制注入个体(顶部)相比 ,lac2 RNAi 个体(底部)的着色相对轻微。比例线表示 5 mm. (B) 控制个体的头部,显示头部中心的特征深色图案。(C) 相对严重的 lac2 击倒 , 导致中央头部着色几乎完全丧失。(D) 与控制注入个体(顶部) 相比,lac2 RNAi 个体(底部)的主要尾节的着色损失。B+u2012D 中的缩放条表示 1 mm。请单击此处查看此图的较大版本。

图5:RNAi 的 laccase2 应用于第三个星体幼虫,导致成人的角质层着色减少。与控件(右)相比,击倒个体(左)也具有非常柔软的电子力。比例线代表 5 毫米 。请单击此处查看此图的较大版本。
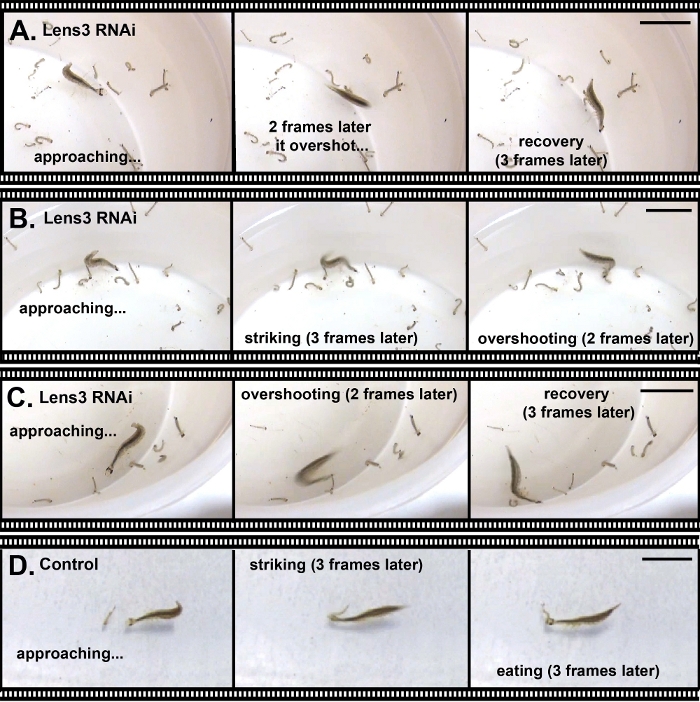
图6:敲击一种主要透镜蛋白,导致猎物捕获不足。(A +C)三个例子,其中太阳爆发潜水甲壳虫幼虫,其中光学缺陷诱导通过RNAi,无法捕捉猎物(蚊子幼虫)。代表性静止图像是从特征猎物捕获的录像中挑选的。(D) 控制幼虫捕捉猎物.所有比例杆代表 2 厘米。请单击此处查看此图的较大版本。
讨论
我们的目标是,这种方法汇编将使RNAi广泛可用,特别是因为这个工具仍然是一个强大的协同技术,CRISPR/Cas9为基础的基因编辑,其优点是,它可以应用于研究生物体的所需发育阶段。为了说明这种强度,我们把dsRNA注射到胚胎和不同的幼虫阶段。注射到卵子影响胚胎发育(图2),注射进入第二幼虫阶段对第三幼虫阶段有明显影响(图4和图6),注射到第三幼虫阶段显示对成人的影响(图5)。虽然确切的时间必须建立实验,一般来说,注射将在几天内生效。这个过程的成功可能受 dsRNA 序列的长度的影响。在这里,我们提出了使用200bp到800bp的例子。一般来说,100 和 600 bp 之间的序列是限制非目标效果的首选,但序列高达 1000 bp 产生良好的结果22。与RNAi有关的问题之一是通过这种技术可以实现的击倒持续时间。由于表型在每个阶段都是终端,因此我们无法根据我们提出的结果来评论这个问题。然而,以前已经注意到,RNAi效应通常寿命相对较长,浓度越高,导致更持久的击倒20。
这种技术的一个局限性是,它比对另一些生物效果更好,而且似乎没有直接的方法来预测它先验地工作得如何。然而,它已被发现对大量不同的生物体工作得很好。在节肢动物中,包括甲壳虫26只,甲壳类动物27只,以及各种昆虫,甲虫28的成功率特别高。另一个并发症是,尽管应用了相同数量的 dsRNA,但个体之间通常发生表型严重性差异。如图 2B所示,变异甚至可能发生在个人内部。在我们的RNAi研究中,针对参与 T.Marmoratus幼虫 眼发育的不同基因,我们经常发现,有些眼睛受到的影响比其他眼睛更严重。这种现象可能与眼睛群相对密集的组织有关,dsRNA可以更好地到达一些单位。
为了成功执行RNAi实验,对目标基因优化几个参数至关重要。例如,dsRNA的浓度和靶向基因的长度可以强烈地影响结果20。另一个关键参数是如何执行注射,因为这个过程可以极大地影响存活率。对于胚胎,我们通过瞄准胚胎中心取得了最佳效果。布局良好的板允许在一次会话中注射100个或更多胚胎。对于幼虫,在各段之间插入注射针至关重要。这些注射需要更多的dsRNA,根据幼虫的可用性,我们的注射集通常只包括几个动物的时间。对于所有的注射,这是至关重要的,以防止空气进入有机体。
在某些情况下,基因调控网络的反馈循环和基因冗余会影响RNAi表型的切兰,尽管不断的敲击。这似乎是我们的行为观察幼虫与非常成功的击倒著名的镜头蛋白,Lens318的情况。虽然我们通过 qPCR 验证了这些击倒的高效率,但相关表型中观察到了相当大的变化。此结果突出了正确量化 RNAi 击倒的必要性(有关选项的详细信息,请参见22)。如果对生成的表型没有明确的先验预期,那么控制RNAi的离目标效应的一个好方法就是将同一基因对靶对,并针对两个非重叠的dsRNA序列,并评估常见表型的结果。
与基因编辑技术相比,RNAi也是研究致命基因的有力工具,有两种方法可以做到这一点。例如,如果一个人对一个基因的功能贡献感兴趣,而在发育早期,已知功能丧失是致命的,那么只要让动物正常发育,然后在发育后期(即成人中),就可以通过RNAi击倒该基因,就可以对这种基因进行功能调查。或者,已知完全丧失功能为致命性的基因可以通过部分敲击来研究,这可以通过注射一系列 dsRNA 浓度实现。我们的一些结果显示了lac2的敲击,如果昆虫的角质层变得过于柔软,那么众所周知,这是致命的。即使是图5中描述的 lac2 RNAi甲虫也不太可能在实验室条件之外存活。另一个致命的基因是切割,它编码的转录因子是基础细胞命运规范在各种器官系统在节肢动物,并一直与胶质发展在果蝇视觉系统29。根据我们在T.Marmoratus胚胎中切割RNAi的经验,我们可以唤起胚胎中能够完成胚胎眼发育(未发表的观察)的翔实眼表型。 cut在这里, 更高的剂量似乎导致更高的致死率, 而低剂量导致可观察和翔实的表型.
我们的协议不仅列出了研究人员在T.Marmoratus上进行RNAi实验的必要步骤,而且一般也适用于其他生物体,特别是水生生物。在水生生物中,甲壳类动物中已经有几个例子,如水蚤Daphnia30和虾(最近的审查,见参考31)。 Daphnia水生昆虫有很多机会,据估计,它们占所有昆虫多样性的6%左右,可能超过20万种32种。此外,RNAi已经在水生环境33的水面上进行了。如果没有基因组存在,那么转录组就可以组装。只要这个过程揭示了几百个核苷酸的连体,就可以设计出针对特定基因的dsRNA。我们关于在阿加罗斯中固定昆虫的协议可能也会用于其他程序,特别是对于软体、可塑性生物和水生生物。综合起来,RNAi仍然是操纵不同生物体中基因表达的有力技术,即使没有其他分子和遗传工具可用。
披露声明
作者没有什么可透露的。
致谢
我们要感谢乔希·贝诺伊特博士在生物信息学方面的援助,海莉·托布勒帮助她提高了太阳爆发潜水甲壳虫和塔玛拉·佩斯的编辑援助。这项研究得到了国家科学基金会的资助,拨款为IOS-1456757和IOS-1856341至EKB,IOS1557936至YT。
材料
| Name | Company | Catalog Number | Comments |
| 1. RNA isolation and de novo transcriptome assembly | |||
| liquid nitrogen | University Stockroom | Typically locally available at research institutions | |
| pipette tips | Fisher Scientific | size dependent | Products from other vendors would work equally well |
| RNeasy lipid tissue mini kit (RNA isolation kit) | Qiagen | 74804 | Detailed specific protocol is provided with the kit |
| spectrophotometer (NnanoDrop) | Thermo Fisher | 9836674 | Other models would work equally well |
| 1.2. De novo transcriptome assembly. | |||
| BUSCO.v3 | https://busco.ezlab.org/ | Any freely available software for assessing trasncriptome completeness can be used. | |
| CLC Genomics workbench | Qiagen | 832021 | Other equivalent software packages are also available |
| Galaxy workbench | https://usegalaxy.org/ | An open source online transcriptome assembly & annotation pipeline | |
| NCBI BLASTx | https://blast.ncbi.nlm.nih.gov/Blast.cgi?LINK_LOC=blasthome&PAGE_TYPE=BlastSearch&PROGRAM=blastx | An open source online alignment and annotation pipeline | |
| 2. cDNA synthesis, Cloning & Miniprep isolation | |||
| ampicillin sodium salt | Gibco | 11593-027 | Products from other vendors would work equally well |
| glycerol | Fisher Scientific | G33-500 | Products from other vendors would work equally well |
| LB broth | Fisher Scientific | BP1426-500 | Products from other vendors would work equally well |
| Omniscript RT (reverse trasncription) kit | Qiagen | 205111 | Detailed specific protocol is provided with the kit |
| One Shot™ TOP10 Chemically Competent E. coli | Invitrogen | C404010 | Detailed specific protocol is provided with the kit |
| Petri dishes | Fisher Scientific | FB0875713 | Products from other vendors would work equally well |
| PureLink Quick Plasmid Miniprep Kit | ThermoFischer | 771471 | Detailed specific protocol is provided with the kit |
| QIAquick PCR purification kit | Qiagen | 28104 | Detailed specific protocol is provided with the kit |
| shaker-incubator | Labnet | 211DS | Other models would work equally well |
| spectrophotometer (NnanoDrop) | Thermo Fisher | 9836674 | Other models would work equally well |
| thermal cycler for PCR | BioRad | T100 | Other models would work equally well |
| TOPO TA Cloning kit | Invitrogen | 1845069 | Detailed specific protocol is provided with the kit |
| X-Gal Solution | Thermo Scientific | R0941 | Products from other vendors would work equally well |
| 2.2 PCR amplification & in vitro dsRNA synthesis | |||
| Centrifuge | Fisher Scientific | accuSpin Micro 17R | Other models would work equally well |
| ethanol | Fisher Scientific | A4094 | Products from other vendors would work equally well |
| FastTaq DNA Polymerase, dNTPack | Roche | 13873432 | This kit contains all the reagents necessary for a PCR |
| MEGAclear Transcriiption clean up kit | ThermoFischer | AM1908 | Detailed specific protocol is provided with the kit |
| MEGAscript T7 Transcription kit | ThermoFischer | AM1334 | Detailed specific protocol is provided with the kit |
| nuclease-free water | Fisher Scientific | AM9932 | Products from other vendors would work equally well |
| QIAquick PCR purification kit | Qiagen | 28104 | Detailed specific protocol is provided with the kit |
| sodium acetate | Fisher Scientific | BP333-500 | Make 3M working solution |
| Spectrophotometer (NnanoDrop) | Thermo Fisher | 9836674 | Other models would work equally well |
| 3. Collecting and preparing early stage T. marmoratus embryos for dsRNA injections | |||
| agarose | Fisher Scientific | 9012-36-6 | Products from other vendors would work equally well |
| distilled water | Fisher Scientific | 9180 | Products from other vendors would work equally well |
| forceps (Dumon #4 Biology) | Fine Science Tools | 11242-40 | Products from other vendors would work equally well |
| glass cavity dish (3 well-dish) | Fisher Scientific | 50-243-43 | Products from other vendors would work equally well |
| microwave | Welbilt | turn-table | Other models would work equally well |
| natural hair paintbrush | Amazon | Any fine brush will do | |
| P1000 micro-pipetter | Gilson | F123602 | Other models would work equally well |
| Petri dishes | Fisher Scientific | FB0875713 | Products from other vendors would work equally well |
| stereomicroscope | Microsocope Central | 10446293 | Any good stereomicroscope will work |
| transfer pipettes | Fisher Scientific | 21-200-109 | Products from other vendors would work equally well |
| 4. dsRNA micro-injections in early stage T. marmoratus embryos | |||
| digital camera | Edmund optics (Qimaging) | Retiga 2000R | Other models would work equally well |
| ethanol | Fisher Scientific | A4094 | Products from other vendors would work equally well |
| food dye | Kroger | Any available food dye should work fine | |
| humidity chamber | take any plastic box with a lid, sterilize it with 70% ethanol and let it dry | ||
| incubator | Labline | 203 | Other models would work equally well |
| injection buffer | Prepared for 1 mL following reference #22 : Mix 10 µL of 0.1 M sodium phosphate buffer, 100 µL of 0.5 M potassium chloride solution, 100 µL of food dye and 790 µL of double-distilled water. Store in 4 °C. | ||
| intracellular Microinjection Systems (Picospritzer) | Parker | 052-0500-900 | Currently the model III is available, but older models work also |
| micro needle holder | A-M Systems | 672441 | Other products would work equally well |
| microinjection needles (1.2 mm x 0.68 mm, 4 inches) | A-M Systems | 603000 | Other models would work equally well |
| micromanipulator | Drummond Scientific Company | 3-000-024-R | Any quality micromanipulator will work |
| monosodium phosphate | Fischer scientific | 7558-80-7 | To make 10 mL of 1 M working solution: Add 1.2 g of monosodium phosphate powder to 10 mL of Double distilled water and mix until clear solution is obtained |
| P-1000 Micropipette Puller | Sutter Instrument | P-1000 | Puller settings:Heat 575, Pull 60, Velocity 75 Delay 110, Pressure 700. |
| P10 micro-pipetter | Gilson | F144802 | Other models would work equally well |
| P1000 micro-pipetter | Gilson | F123602 | Other models would work equally well |
| potassium chloride | Fischer scientific | 7447-40-7 | To make 100 mL of 0.5 M potassium chloride solution: Add 3.73 g of potassium chloride crystals to 100 mL of double distilled water and mix until clear solution is obtained. |
| sodium phosphate buffer (0.1M) | To make 10 mL of 0.1 M of this buffer: Mix 8.5 mL of 1 M sodium phosphate dibasic solution with 1.5 mL of 1 M monosodium phosphate solution. Check the pH with a pH meter and adjust accordingly to pH 7.6 at room temprature. | ||
| sodium phosphate dibasic | Fischer scientific | 7558-79-4 | To make 10 mL of 1 M working solution: Add 1.42 g of sodium phosphate dibasic powder to 10 mL of Double distilled water and mix until clear solution is obtained |
| stereomicroscope | Microsocope Central | 10446293 | Any good stereomicroscope will work |
| 5. Preparing T. marmoratus larvae for dsRNA injections | |||
| agarose | Fisher Scientific | 9012-36-6 | Products from other vendors would work equally well |
| forceps (Dumon #4 Biology) | Fine Science Tools | 11242-40 | Products from other vendors would work equally well |
| Petri dishes | Fisher Scientific | FB0875713 | Products from other vendors would work equally well |
| 6. dsRNA micro-injections in T. marmoratus larvae | |||
| injection buffer (10x) | See section 4 | ||
| microinjection needles (1.2 mm x 0.68 mm, 4 inches) | A-M Systems | 603000 | Other models would work equally well |
| microinjection syringe | A-M Systems | 603000 | Other models would work equally well |
| micro needle holder | A-M Systems | 672441 | Other products would work equally well |
| P-1000 Micropipette Puller | Sutter Instrument | P-1000 | Puller settings:Heat 575, Pull 60, Velocity 75 Delay 110, Pressure 700. |
| stereomicroscope | Microsocope Central | 10446293 | Any good stereomicroscope will work |
参考文献
- Muller, B., Grossniklaus, U. Model organisms - A historical perspective. Journal of Proteomics. 73, 2054-2063 (2010).
- Gilbert, L. A., et al. CRISPR-Mediated Modular RNA-Guided Regulation of Transcription in Eukaryotes. Cell. 154, 442-451 (2013).
- Westerman, E. L., et al. Aristaless Controls Butterfly Wing Color Variation Used in Mimicry and Mate Choice. Current Biology. 28, 3469 (2018).
- Perry, M., et al. Molecular logic behind the three-way stochastic choices that expand butterfly colour vision. Nature. 535, 280 (2016).
- Boettcher, M., McManus, M. T. Choosing the Right Tool for the Job: RNAi, TALEN, or CRISPR. Molecular Cell. 58, 575-585 (2015).
- Meister, G., Tuschl, T. Mechanisms of gene silencing by double-stranded RNA. Nature. 431, 343-349 (2004).
- Fire, A., et al. Potent and specific genetic interference by double-stranded RNA in Caenorhabditis elegans. Nature. 391, 806-811 (1998).
- Kennerdell, J. R., Carthew, R. W. Use of dsRNA-mediated genetic interference to demonstrate that frizzled and frizzled 2 act in the wingless pathway. Cell. 95, 1017-1026 (1998).
- Mello, C. C., Conte, D. Revealing the world of RNA interference. Nature. 431, 338-342 (2004).
- Agrawal, N., et al. RNA interference: Biology, mechanism, and applications. Microbiol. Microbiology and Molecular Biology Reviews. 67, 657 (2003).
- Gu, L. Q., Knipple, D. C. Recent advances in RNA interference research in insects: Implications for future insect pest management strategies. Crop Protection. 45, 36-40 (2013).
- Ravisankar, P., Lai, Y. T., Sambrani, N., Tomoyasu, Y. Comparative developmental analysis of Drosophila and Tribolium reveals conserved and diverged roles of abrupt in insect wing evolution. Developmental Biology. 409, 518-529 (2016).
- Tomoyasu, Y., Wheeler, S. R., Denell, R. E. Ultrabithorax is required for membranous wing identity in the beetle Tribolium castaneum. Nature. 433, 643-647 (2005).
- Tomoyasu, Y., Arakane, Y., Kramer, K. J., Denell, R. E. Repeated Co-options of Exoskeleton Formation during Wing-to-Elytron Evolution in Beetles. Current Biology. 19, 2057-2065 (2009).
- Yang, X., et al. Probing the Drosophila retinal determination gene network in Tribolium (II): The Pax6 genes eyeless and twin of eyeless. Developmental Biology. 333, 215-227 (2009).
- ZarinKamar, N., et al. The Pax gene eyegone facilitates repression of eye development in Tribolium. Evodevo. 2, 15 (2011).
- Linz, D. M., Clark-Hachtel, C. M., Borras-Castells, F., Tomoyasu, Y. Larval RNA Interference in the Red Flour Beetle, Tribolium castaneum. Journal of Visualized Experiments. , e52059 (2014).
- Stahl, A., S, B. R., Cook, T. A., Buschbeck, E. K. A complex lens for a complex eye. Integrative and Comparative Biology. 57, 1071-1081 (2017).
- Yeku, O., Frohman, M. A. Rapid Amplification of cDNA Ends (RACE). RNA, Methods In Molecular Biology. , 107-122 (2011).
- Miller, S. C., Miyata, K., Brown, S. J., Tomoyasu, Y. Dissecting Systemic RNA Interference in the Red Flour Beetle Tribolium castaneum: Parameters Affecting the Efficiency of RNAi. Plos One. 7, (2012).
- Tomoyasu, Y., Denell, R. E. Larval RNAi in Tribolium (Coleoptera) for analyzing adult development. Development Genes and Evolution. 214, 575-578 (2004).
- Philip, B. N., Tomoyasu, Y., Orgogozo, V., Rockman, M. V. . Molecular Methods for Evolutionary Genetics. , 471-497 (2011).
- Mackenzie, S. M., Howells, A. J., Cox, G. B., Ewart, G. D. Sub-cellular localisation of the White/Scarlet ABC transporter to pigment granule membranes within the compound eye of Drosophila melanogaster. Genetica. 108, 239-252 (2000).
- Arakane, Y., Muthukrishnan, S., Beeman, R. W., Kanost, M. R., Kramer, K. J. Laccase 2 is the phenoloxidase gene required for beetle cuticle tanning. Proceedings of the National Academy of Sciences of the United States of America. 102, 11337-11342 (2005).
- Werner, S., Buschbeck, E. K. Rapid and step-wise eye growth in molting diving beetle larvae. Journal of Comparative Physiology. 201, 1091-1102 (2015).
- Prpic, N. M., Schoppmeier, M., Damen, W. G. Gene Silencing via Embryonic RNAi in Spider Embryos. Cold Spring Harbor protocols. 3, 1-4 (2008).
- Sagi, A., Manor, R., Ventura, T. Gene Silencing in Crustaceans: From Basic Research to Biotechnologies. Genes. 4, 620-645 (2013).
- Zhu, K. Y., Palli, S. R., Douglas, A. E. . Annual Review of Entomology. 65, 293-311 (2020).
- Bauke, A. C., Sasse, S., Matzat, T., Klaembt, C. A transcriptional network controlling glial development in the Drosophila visual system. Development. 142, 2184 (2015).
- Lin, C. Y., et al. RNAi analysis of doublesex1 expression in Daphnia pulex Leydig, 1860 (Cladocera). Crustaceana. 92, 137-154 (2019).
- Nguyen, D. V., Christiaens, O., Bossier, P., Smagghe, G. RNA interference in shrimp and potential applications in aquaculture. Reviews in Aquaculture. 10, 573-584 (2018).
- Dijkstra, K. D. B., Monaghan, M. T., Pauls, S. U., Berenbaum, M. R. . Annual Review of Entomology. 59, 143-163 (2014).
- Crumiere, A. J. J., Khila, A. Hox genes mediate the escalation of sexually antagonistic traits in water striders. Biology. Letters. 15, 5 (2019).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。