Method Article
특정 발달 시점에서 유전자 발현을 조작하기 위한 강력한 도구로 수생 딱정벌레의 RNA 간섭
요약
RNA 간섭은 특정 발달 단계에서 유전자 발현을 조작하기 위한 널리 적용가능하고 강력한 기술입니다. 여기서, 우리는 수중 다이빙 딱정벌레 Thermonectus marmoratus에서이 기술을 구현하기 위한 필요한 단계를 기술합니다, 구조 또는 행동에 영향을 미치는 유전자의 녹다운에 유전자 의 취득에서.
초록
RNA 간섭(RNAi)은 mRNA 분해를 통해 유전자 발현의 표적 감소를 허용하는 강력한 기술로 남아 있다. 이 기술은 다양한 유기체에 적용 가능하며 종이 풍부한 순서인 Coleoptera(딱정벌레)에서 매우 효율적입니다. 여기에서, 우리는 새로운 유기체에서 이 기술을 개발하기 위한 필요한 단계를 요약하고 수생 다이빙 딱정벌레 Thermonectus marmoratus의 다른 발달 단계에 그것의 응용프로그램을 설명합니다. 표적 유전자 서열은 알려진 유전체학 또는 드 노보와 가까운 상대에 대한 전사의 조립을 통해 비용 효율적으로 얻을 수 있다. 후보 유전자 복제는 단일 공통 프라이머를 사용하는 모든 유전자에 대해 이중 좌초 RNA(dsRNA)의 합성을 허용하는 특정 복제 벡터(pCR4-TOPO 플라스미드)를 활용한다. 합성된 dsRNA는 나중에 발달 프로세스를 위한 초기 발달 프로세스 또는 애벌레를 위한 태아 로 주입될 수 있습니다. 그런 다음 RNAi가 아가로즈의 고정화를 사용하여 수생 유충에 주입될 수 있는 방법을 설명합니다. 이 기술을 입증하기 위해 RNAi 실험의 몇 가지 예를 제공하여 예측된 표현형으로 특정 녹다운을 생성합니다. 구체적으로, 선탠 유전자 락케이스2에 대한 RNAi는 애벌레와 성인 모두에서 큐티클 번개로 이어지며, 눈 색소 침착 유전자를 위한 RNAi는 안구에서 색소 침착의 번개/부족을 일으킵니다. 또한, 키 렌즈 단백질의 넉다운은 광학 결핍과 먹이를 사냥하는 능력을 감소케하는 애벌레로 이어집니다. 결합, 이러한 결과는 단지 전사 데이터베이스와 유기체의 형태학적 패턴과 행동 특성을 조사하기위한 도구로 RNAi의 힘을 예시.
서문
특정 유전자가 다양한 특성의 진화에 기여하는 방법의 질문은 생물학에서 흥미로운 주제입니다. 지난 수십 년 동안, 선충 근 사엽다비티스 엘레간,과일 플라이 드로소필라 멜라노가스터,그리고 집 마우스 무근육1과같은 몇 가지 모델 유기체에서 발달 과정의 유전적 기초를 해부하는 것과 관련하여 많은 진전이 이루어졌습니다. 최근에는 군집된 짧은 palindromic 반복(CRISPR)/Cas92와 같은 강력한 유전자 편집 기술의 발명은 비모델 유기체의 유전 코드를 변경하는 기능을 제공하였다(예를 들어3,,4참조). 그 결과, 이전에 분자 기술을 통해 접근되지 않았던 다양한 유기체에 대한 유전 적 연구가 급증하고 있습니다. 특정 종에서만 표현되는 흥미로운 특성이나 특성 차이가 있는 우리 동물 왕국의 엄청난 다양성을 고려할 때, 이러한 진전은 진화 발달 생물학("evo-devo") 관련 작업을 위한 흥미로운 시기였습니다. 그러나, 비모델 유기체에 사용할 수 있는 게놈 편집 기술은 적용될 수 있는 발달 시간 점에 관하여 상대적으로 제한됩니다, 특정 유전자가 어떤 특성든지에서 재생하는 역할과 관련있는 시간적 특성을 분별하는 것을 어렵게 만듭니다. 또한, 형질 전환 기술은 종종 생존에 필수적이지 않은 유전자로 제한됩니다 (즉, 녹아웃이 치사증을 초래하지 않습니다). 따라서 유전자 편집 기술이 인기를 끌기 시작했지만, 특정 발달 시점의 다양한 유기체에 적용할 수 있는 효과적인 기술이 필요하며 부분적인 녹다운을 용이하게 할 필요가 있다(완전한 기능 상실보다는). 여기서는 유전자 편집에 대한 시너지 접근법으로서 특히 귀중한 다소 오래된 강력한 유전자 녹다운 기술5인 RNA 간섭(RNAi)에 주목합니다. 특히, 우리는 RNAi를 수중 다이빙 딱정벌레에 적용할 수 있도록 하는 절차를 개발하여 필요한 분자 서열의 획득에서부터 계란과 애벌레에 이중 좌초 RNA(dsRNA)를 성공적으로 주입하는 것까지 이 기술의 구현을 보여주는 예로서 개발했습니다.
RNAi 기반 유전자 녹다운은 dsRNA 분자가 바이러스 및 트랜스포슨6과같은 핵산 서열침을 침전시키는 것을 용이하게 하는 유기체의 타고난 방어 메커니즘을 활용한다. 간단히 말해서, dsRNA는 디커 효소에 의해 20-25 뉴클레오티드 조각으로 절단되는 세포로 채택됩니다. 이러한 조각들은 RNA 유도 침묵 복합체(RISC)의 형성을 활성화시켜, 이는 가이드 가닥을 사용하여 특정 부위에 결합하여 표적 mRNA를 억제한다. 이 과정은 궁극적으로 mRNA 분해로 이어지므로 mRNA를 각 단백질6으로변환하는 것을 방해한다. 여기에서 제출된 RNAi 기지를 둔 유전자 녹다운 기술은 그러므로 dsRNA의 주입에 의지합니다. 동물 모델의 경우, 이 기술은 원래 C. elegans7 및 D. 멜라노가스터8에서 개발되었지만 이후 비모델 유기체9,,10에서강력한 기능성 유전 도구로 부상했습니다. 일부 곤충의 매우 효과적인 특성으로 인해 RNAi는 해충 관리11에적용 될 수 있습니다.
연구 도구로서 RNAi는 비전통적인 곤충 모델에서 주요 분자 발달 경로가 어떻게 작동하는지 검사하는 데 사용되었습니다. 예를 들어, 밀가루 딱정벌레 트리볼륨 카스타뉴움의 RNAi는 특별히 모양의 날개,12, 13,14및 눈14 15,,16의발달을 예시하는 바와 같이, 그 딱정벌레의 특정 특성에 얼마나 깊이 보존된 유전자가 기여하는지 결정하는 데 중요한 역할을 해왔다., T. 카스타뉴움의 조작의 근간을 뒷받침하는 기술은17에 잘 설명되어 있으며 끈적끈적한 표면에 상대적으로 건조한 계란과 애벌레를 고정시키는 능력에 의존합니다. 그러나 이러한 고정은 Sunburst 다이빙 딱정벌레 열상모라투스와같은 수생 생물의 습한 발달 형태에 대해 가능하지 않다. 많은 비전통적인 모형 유기체에 대한 경우와 같이, 그것은 음부게놈이 부족합니다. 게놈이 없는 유기체에서 유전자 발현을 조작하기 위해, 합리적이고 비용 효율적인 첫 번째 단계는 전사를 생성하고 관련하지만 더 확립된 모델 유기체와의 서열 유사성에 Tribolium 기초하여 관심있는 발현 유전자의 퍼티블 뉴클레오티드 서열을 식별하는 것입니다. Drosophila
여기서, RNAi가 수생 유기체에서 어떻게 사용될 수 있는지 를 보여주기 위하여는, 우리는 먼저 특정 표적 유전자 서열의 확인을 허용하는 RNA 추출 및 전사 생성 및 조립을 위한 프로토콜 및 소프트웨어를 토론합니다. 그런 다음 유전자 특정 dsRNA를 합성하는 데 필요한 단계를 요약합니다. 그 후, 우리는 어떻게 계란수생 환경에서 주입될 수 있는지 설명하고 발전하는 배아를 배양하기 위한 잠복기 프로토콜을 보여줍니다. 또한, 사산과정에서 애벌레를 완전히 고정하는 데 아가로즈 젤을 어떻게 사용할 수 있는지 를 보여 주며, 다양한 시술 중에 일반적으로 유용하며 다양한 절지동물에 적용될 수 있는 기술이다. RNAi가 다른 발달 단계에 어떻게 적용될 수 있는지 보여주기 위하여는, 우리는 우리가 태아에 있는 눈 색소 침착 유전자 백색을 침묵하는 예가 포함되어 있습니다. 또한, 우리는 태닝 유전자 laccase2(lac2)가 두 번째 애벌레 인스타 (세 번째 애벌레 인스타의 애벌레에 영향을 미치기 위해) 및 세 번째 애벌레 인스타 (성인에 영향을 미치는)동안 침묵한 예를 설명합니다. 마지막으로, 우리는 dsRNA의 낮은 농도의 주입이 부분적인 녹다운으로 이끌어 낸다는 것을 보여줍니다, 이 기술은 또한 기능의 손실이 치명적인 것으로 알려져 있는 유전자에 적용될 수 있다는 것을 보여줍니다.
프로토콜
1. RNA 절연 및 드 노보 전사 조립
- 지질이 풍부한 조직을 위해 설계된 RNA 격리 키트(재료표참조)를 사용하여 후기 스테이지 3 인스타 다이빙 딱정벌레 애벌레 안구와 성인 딱정벌레로부터 총 RNA 절연을 수행합니다. T. marmoratus에서 총 RNA를 분리하고 이전에 설명된방법(18)에따라 서열한다.
- 드 노보 전사 어셈블리
참고: 전사 드 노보를 조립하려면 다양한 생물정보학 플랫폼을 사용할 수 있습니다(재료표참조). 이러한 플랫폼은 시판되고 있으며 경우에 따라 유닉스 기반 명령줄 스크립트로 존재합니다. 어셈블리는 드 노보이므로 두 플랫폼에서 전사를 독립적으로 조립하고 결과를 비교하여 거짓 긍정 또는 거짓 네거티브를 제외하는 것이 좋습니다.- 전사를 조립하기 시작하려면 각 생물 정보학 플랫폼에 원시 판독기를 업로드합니다.
참고: 이러한 파일은 일반적으로 압축되며 FASTQSANGER 형식으로 압축됩니다. Gz. 이 형식은 다음 단계에서 허용되기 때문에 파일 압축을 풀 필요가 없습니다. - Trimmomatic 명령줄을 사용하여 원시 읽기를 트리밍하여 기본 임계값 미만인 어댑터 시퀀스 또는 짧은 읽기를 제거합니다. 트리밍된 원시 읽기압축을 풀고 있습니다. 이제 파일이 FASTQ 형식으로 표시됩니다. 각 샘플에 대해 FASTQ 원시 판독을 연결하여 드 노보 어셈블리에 사용할 준비가 된 파일을 가져옵니다.
- 전사 드 노보를 조립할 수 있는 명령줄 스크립트를 사용하여 연결된 원시 읽기 드 노보를 조립합니다. 이제 FASTA 파일로 각 시퀀스와 연속의 목록이있을 것이다 조립 된 전사를 저장합니다. 어셈블리의 커버리지를 높이려면 동일한 종에 대해 여러 전사를 결합하고 조립합니다.
참고: 이 과정은 더 정확한 연속을 생성의 기회를 증가하고 낮은 복사 번호 유전자가 관심있는 경우에 특히 중요합니다. - 일단 조립되면 커버리지 점수를 계산하여 완전성을 위해 전사를 평가하십시오(커버리지 평가 소프트웨어는 자유롭게 사용할 수 있으며 재료 표참조).
참고: 커버리지 점수가 있는 전사체 >75%는 좋은 것으로 간주됩니다. 그러나,이 점수의 관련성은 전사 생성에 대한 이유에 따라 달라집니다. 예를 들어, 전사가 낮은 복사 번호 유전자를 식별하는 데 사용되는 경우, 점수 >85%는 더 큰 커버리지를 의미하기 때문에 더 좋습니다. - 기본 로컬 정렬 검색 도구를 사용하여 드 노보 조립 된 전사체에 음장 (BLAST; 재료의 표참조).
참고: 이 실험을 위해, 전사는 알려진 Drosophila 단백질의 데이터베이스 및 알려진 딱정벌레 단백질의 데이터베이스에 대하여 주로 부음되었습니다. 주석 목록은 다른 유기체의 단백질과 서열 유사성을 나타내는 스프레드시트 파일 및 연속/연속으로 다운로드할 수 있으며 FASTA 파일로 다운로드할 수 있다. - 관심 있는 단백질의 연속 수/숫자를 식별하고 뉴클레오티드 서열을 추출한다. BLAST를 사용하여 단백질 특이적 보존 영역(예: 홈로박스 도메인)이 인가된 뉴클레오티드 서열에 존재하는지 확인합니다. 뉴클레오티드 서열에 대해 동일한 성서가 겹치는 다른 연속체를 확인하여 유전자 특이적 전사체의 서열을 생성한다.
참고: 서열 중첩을 가진 연속을 확인하는 것은 일반적으로 낮은 복사 번호 유전자를 위해, 모든 유전자를 위해 가능하지 않습니다. - 유전자의 전체 길이 서열이 필요한 경우, cDNA 끝의 3′ 및 5′ 급속 증폭 (RACE19)와같은 기술을 사용하여 전체 성숙한 성적 체형의 뉴클레오티드 서열을 식별하기위한 개시 지점으로 가장 높은 서열 유사성을 가진 연속성으로 시작합니다.
- 1.2.4-1.2.7 단계에 따라 두 번째 플랫폼에서 얻은 어셈블리에 대해 노트윗합니다.
참고: 이상적으로, 결과는 관심있는 단백질에 대해 유사해야 합니다; 그러나 플랫폼 간에 는 범위가 어느 정도 다를 수 있습니다. - 조립된 전사체를 사용하여 섹션 2에 설명된 대로 종별 dsRNA를 합성합니다.
- 전사를 조립하기 시작하려면 각 생물 정보학 플랫폼에 원시 판독기를 업로드합니다.
2. 복제 및 유전자 특정 dsRNA 합성
참고: 유전자 특이적 복제 및 dsRNA 합성은 T. 카스타뉴움20,,21,,22에대해 자세히 설명되어 왔다. 다음 단계는 간략한 개요입니다.
- 복제, 세균 변환 및 플라스미드 시퀀싱
- 유기체로부터 총 RNA를 분리(1.1단계에서 설명한 대로) 역전사 키트를 사용하여 상호 보완적인 DNA(cDNA)를 생성한다(재료표참조). 관심 있는 유전자에 특이적인 연속으로부터 하나 또는 두 개의 100-1000 bp 뉴클레오티드 서열을 식별한다.
- 전체 규미에 걸친 설계 프라이머 쌍은 전체 규미성드에 걸쳐 중합효소 연쇄 반응(PCR)을 통해 서열을 증폭시한다. 추가 추가 30 증폭 된 DNA 제품에 adenine 오버행을 만들 끝에 분 단계. PCR 정제 키트를 사용하여 이 제품을 정화하십시오(재료 표참조).
- 복제 키트와 함께 제공된 공급업체의프로토콜에 따라 정제된 제품을 pCR4-TOPO 플라스미드 벡터(재료 표 참조)로 복제합니다. 열 충격 처리를 사용하여 복제된 플라스미드를 유능한 세균 세포(균주: E.coli DH10B)로 변환하여 유능한 세포(재료 표참조)에 대한 공급업체의 프로토콜에 따라 성공적으로 변형된 세포를 위한 화면을 변형시킵니다.
참고: 콜로니가 있는 플레이트는 파라핀 필름으로 싸서 수분을 유지하고 최대 1개월 동안 4°C에 보관할 수 있습니다. - 37°C의 셰이커 인큐베이터에서 암피실린(50 μg/mL)을 함유한 리소겐 국물(LB)의 10 μL 파이펫 팁 및 배양물을 사용하여 변형된 세포의 콜로니를 선택하고 24시간 동안 200rpm을 선택한다.
참고: 장기 보관을 위해 배양된 세포는 1:1로 희석되고 글리세롤 주식으로 -80°C에서 냉동될 수 있습니다. - 미니프레플 격리 키트를 사용하여 이 배양으로부터 관심 있는 유전자를 함유한 플라스미드를 분리한다(재료표참조). 수율을 분광성으로 평가한 후 -20°C에 격리된 플라스미드(minipreps)를 저장합니다. 구체적으로는 260nm, 280nm 및 230nm에서 시료 흡광도를 측정합니다. DNA 정량화에 대한 비율 A260/A280, DNA 순도를 정량화하기 위한 A260/A230을 계산합니다.
참고: 1.8의 260/280 비율은 일반적으로 충분히 순수한 DNA로 받아들여지고, 1.5보다 낮은 비율은 페놀과 같은 잔류 추출 시약의 존재를 나타낸다. 260/230 비율은 260/280 비율보다 크거나 같아야 합니다. 비율이 낮을수록 잔류 시약 오염이 있음을 나타냅니다. 수율은 일반적으로 100에서 500 ng/μL 범위입니다. - 유전자 특정 단편이 성공적으로 통합되었음을 확인하기 위해 격리 된 미니 프렙을 서열, 플라스미드에서 5′ T7과 3′ T3 프로모터 영역 사이에 측면되고; 이 작업은 범용 T7 및 T3 시퀀싱프라이머(22)를사용하여 수행할 수 있습니다. dsRNA 제제에 대한 유전자 특이적 관심 서열을 사용하여 미니프렙을 사용하십시오.
- 유기체로부터 총 RNA를 분리(1.1단계에서 설명한 대로) 역전사 키트를 사용하여 상호 보완적인 DNA(cDNA)를 생성한다(재료표참조). 관심 있는 유전자에 특이적인 연속으로부터 하나 또는 두 개의 100-1000 bp 뉴클레오티드 서열을 식별한다.
- PCR 증폭 및 시험관 내 dsRNA 합성
- PCR 증폭 및 제품 정화
- 삽입된 유전자의 선형 단편을 증폭시키기 위해 5' 측에 T7 프로모터22 서열을 갖는 플라스미드 특이적 또는 유전자 특이적 프라이머를 설계한다. 각각 20μL의 10회 이상의 반응을 설정하여 수율을 최적화합니다.
참고: 결과 제품은 20bp T7 폴리머라제 결합 부위에 의해 측면에 있습니다. - 공급 업체의 열 기반 용출 프로토콜에 따라 PCR 제품 정제 키트 (재료 표참조)를 사용하여 PCR 제품을 정화합니다. 수율을 높이려면 동일한 용출물로 최종 용출 단계를 최대 3배까지 반복합니다. 수율을 분광성으로 평가합니다.
참고: 수율은 일반적으로 100에서 700 ng/μL 범위입니다. 이 정제 된 제품은 추가 사용까지 -20 °C에 보관 할 수 있습니다.
- 삽입된 유전자의 선형 단편을 증폭시키기 위해 5' 측에 T7 프로모터22 서열을 갖는 플라스미드 특이적 또는 유전자 특이적 프라이머를 설계한다. 각각 20μL의 10회 이상의 반응을 설정하여 수율을 최적화합니다.
- 시험관 내 dsRNA 합성 및 정제
- 유전자 특이적 PCR 정제 제품을 사용하여 dsRNA 합성 키트의 프로토콜에 따라 시험관 내에서 dsRNA를 합성한다. 필요한 경우 dsRNA 생산 증가를 위해 인큐베이션 시간을 연장하십시오.
- 반응 혼합물의 불투명도에 기초하여 반응의 진행을 평가합니다(dsRNA 생성물의 양이 클수록 탁도가 높을수록). 인큐베이션의 끝에서, DNase 치료를 사용하여 반응을 중지합니다. 이렇게하려면 2 U / μL의 스톡에서 1 μL을 반응 혼합물에 추가합니다. 템플릿 DNA의 최적의 분해를 보장하기 위해이 치료를 1-2 h로 확장하십시오.
- 정제 키트 또는 다음 단계를 통해 dsRNA를 정화합니다.
- 생성 된 제품을 115 μL의 뉴클레아제없는 물로 희석하고 3 M 나트륨 아세테이트의 15 μL을 추가하십시오. 이 반응 혼합물에 얼음 차가운 100 % 에탄올 2 부피를 추가하고 -20 °C에서 하룻밤 dsRNA를 침전시한다.
참고: 이 시점에서 최종 수율에 영향을 주지 않고 샘플을 안전하게 저장할 수 있습니다. - 9000 x g에서 20 분 동안 0 °C에서 침전을 원심 분리하고 상체를 폐기하십시오. 13000 x g에서 15 분 동안 얼음 차가운 70 % 에탄올과 원심 분리기로 제품을 한 번 씻으십시오.
- 상피및 공기건조펠릿을 5-15분 동안 제거하였다.
- 정제 된 dsRNA를 -20 °C에서 추가 사용까지 저장합니다. 원하는 발달 단계에서 주입을 위해 정제 된 dsRNA를 사용합니다.
- 생성 된 제품을 115 μL의 뉴클레아제없는 물로 희석하고 3 M 나트륨 아세테이트의 15 μL을 추가하십시오. 이 반응 혼합물에 얼음 차가운 100 % 에탄올 2 부피를 추가하고 -20 °C에서 하룻밤 dsRNA를 침전시한다.
- PCR 증폭 및 제품 정화
3. dsRNA 주사를 위한 초기 단계 T. marmoratus 배아의 수집 및 준비
- T. marmoratus 배아의 잠복에 대 한 아가로즈 플레이트를 준비.
- 먼저, 20 mg의 저융 아가로즈 분말을 100mL의 오토클레이브 증류수(2% 아가로즈)에 녹인 다음, 투명한 용액이 얻어질 때까지 전자레인지에 이 혼합물을 끓입니다. 기포가 넘쳐 나면 뜨거운 용액을 주의하십시오.
- 이 솔루션을 작은 페트리 접시에 분배합니다. 아가로즈 플레이트 표면에 홈(배아를 보유하게 됩니다)을 만들기 위해, 액체 아가로즈 표면에 1-2mm 너비의 플라스틱 튜브(예: 플라스틱 이송 파이펫의 끝)를 배치한 후 고화시한다.
- 아가로즈가 고화되면 무딘 집게 한 쌍을 사용하여 이러한 튜브를 제거하십시오. 아가로즈의 이러한 들여쓰기를 커버하기 위해 P1000 마이크로 파이프를 사용하여 500 μL의 오토클레이브 증류수를 추가합니다.
- 미세한 천연 헤어 페인트 브러시를 사용하여, 자동 증류수로 채워진 유리 캐비티 접시에 딱정벌레 중첩 사이트에서 5-8 h 의 T. marmoratus 배아를 수집합니다. 중첩 부위를 자주 모니터링하여 얻은 계란이 적절한 나이인지 확인합니다.
- 미세 해부 집게를 사용하여 스테레오 현미경으로 배아를 부조합니다. 이렇게하려면, 현미경 (원하는 배율에서) 적당한 각도로 광원을 배치하여 현미경 (원하는 배율에서)에서 초리온을 시각화한 다음 날카로운 집게로 양면에서 초리온을 잡고 찢어서 배아를 부드럽게 밀어 냅니다. 두 개의 레이어가 있는 것처럼 두 레이어가 모두 제거되었는지 확인합니다.
- 미세한 천연 헤어 페인트 브러시를 사용하여 유리 캐비티 접시에서 아가로즈 플레이트로 배아를 조심스럽게 옮기고 스테레오 현미경으로 홈에 배치하십시오. 이 과정에서 비반향배아가 매우 깨지기 때문에 세심한 주의를 기울이십시오. dsRNA 주사를 위해 준비된 배아를 사용하십시오.
참고: 약간 두꺼운 끝은 머리가 발달할 배아의 측면입니다.
4. 초기 단계 T. marmoratus 배아에서 dsRNA 미세 주입
- 유전자 특정 dsRNA를 가진 초기 단계 배아를 주입하려면, 미세 주입 바늘 풀러를 사용하여 미세 주입 바늘을 준비 (재료의 표참조). 스테레오 현미경의 밑에 바늘 끝을 시각화하는 동안, 바늘 끝을 깰 미세 한 집게의 쌍을 사용하여 날카로운 가장자리를 만듭니다. 이상적으로, 바늘 끝을 대각선으로 깨고 날카로운 가장자리가 한쪽에 남아 있어 최소한의 부상을 입히면서 배아에 들어갈 수 있습니다.
- 얼음에 정제 된 재고 dsRNA 솔루션을 해동, 10 x 사출 버퍼의 1 μL을 추가하고 이중 증류수로 위로, 사출 솔루션의 10 μL을 만들기 위해. 주사의 경우, 1 μg/μL의 dsRNA 농도는 일반적으로 적합하지만 생성된 동물의 현상진 심각도에 따라 조정할 수 있다).
참고: 0.1M 나트륨 인산염 버퍼의 10 μL을 추가하여 10x 사출 버퍼22의 1mL을 준비합니다(M Na2HPO4의 8.5mL 및 1의 1.5mL를 혼합하여 제작 M NaH2PO4는 0.5M KCl의 100 μL, 100 μL 의 식품 염료 100 μL 및 이중 증류수 790 μL으로 25°C에서 pH 7.6으로 조정되었습니다.- P10 파이펫을 사용하여 작동 dsRNA 용액으로 미세 주입 바늘을 다시 채웁니다. 용액이 바늘의 끝을 채우면 압력 배출 기술을 활용하는 마이크로 주입 시스템에 연결된 마이크로 니들 홀더에 부착하십시오 (재료 표참조).
- 미세 주입 시스템과 가압 공기 공급을 켭니다. 마이크로 주입 시스템의 마이크로 밸브를 사용하여 사출 압력을 15 psi로 조정합니다. 시간 조정 노브를 사용하여 사출 기간을 조정합니다("초"로 설정).
- 주사 기간은 필요한 사출 볼륨에 따라 달라지므로 필요한 경우 각 주입 전에 미세 주입 시스템에 이 매개 변수를 조정합니다. 초기 단계 T. marmoratus 배아에 대 한 1-2 nL의 주입 볼륨을 사용 하 여.
참고: 주사량이 많을수록 배아 생존율을 방해하는 경향이 있습니다.
- 주사 기간은 필요한 사출 볼륨에 따라 달라지므로 필요한 경우 각 주입 전에 미세 주입 시스템에 이 매개 변수를 조정합니다. 초기 단계 T. marmoratus 배아에 대 한 1-2 nL의 주입 볼륨을 사용 하 여.
- 마이크로주입 시스템에 의해 분배되는 사출액의 부피를 측정하기 위해, 공기 중으로 주입할 때 바늘 끝에서 형성되는 유체 버블의 부피를 평가하여 주사 바늘을 보정한다. T. marmoratus 배아의 경우, 100-200 μm의 최적의 거품 크기를 사용합니다. 주사 바늘은 ~3s의 주사 지속 시간으로 15 psi에서 달성 될 수 있다면 좋은 품질입니다.
- 바늘이 45°와 60°사이의 각도로 배아에 접근하는 것을 예체 현미경으로 배아를 포함하는 바늘과 아가로즈 플레이트를 정렬합니다.
- 미세 조작기사용, 스테레오 현미경을 통해 진행 상황을 모니터링하는 동안 천천히 마이크로 니들 이동. 바늘끝이 배아의 표면에 닿으면 배아 표면을 관통할 때까지 바늘을 조심스럽게 앞으로 움직입니다. 이 배아의 생존에 영향을 미칠 수 있기 때문에 바늘 끝으로 깊은 배아를 천명하지 마십시오. 가변성을 줄이기 위해 배아 중간에 주사를 전달합니다.
- 바늘이 배아 내부에 들어가면 마이크로 인젝터의 주사 버튼을 조심스럽게 눌러 dsRNA의 용량을 배아에 전달하십시오. dsRNA의 1-2 nL을 주입 한 후, 천천히 배아에서 바늘을 철회, 그것은 파열 배아를 유발하지 않도록.
- 유사한 방식으로 다른 배아를 주입하십시오. 미세 주입 바늘이 시술 중에 차단되면 주입 압력과 지속 시간을 증가시켜 차단을 해제하십시오. 주사 부위에 착색 된 반점이 존재하여 성공적으로 주입 된 배아를 시각적으로 식별합니다 (사출 버퍼에는 음식 색이 포함되어 있기 때문에).
- 조심스럽게 습도 챔버에 주입 된 배아와 접시를 배치합니다.
- 이러한 챔버를 구성하려면 뚜껑이있는 플라스틱 상자를 가져 가서 70 % 에탄올로 살균하고 건조 할 수 있도록하고 바닥에 있는 오토클레이브 물에 담근 조직을 배치하여 습한 환경을 제공합니다. 이 습도 챔버를 적절한 온도(이 경우 25°C)와 14h 광 및 10h 어둡게 하는 인큐베이터에 놓습니다.
- 주입된 태아가 4-5일이 필요한 첫 번째 인스타 애벌레로 발전하도록 허용하십시오. 도 2에묘사된 바와 같이 형태학적으로 보이는 녹다운 표현형을 식별하기 위해 스테레오현미경을 사용하여 매일 배아 발달의 진행 상황을 모니터링한다.
- 고해상도 카메라를 사용하여 디지털 이미지를 촬영하여 이 진행 상황을 문서화합니다. 죽은 배아를 제거하고 잠복 기간 동안 다른 생존 배아에 미생물 오염의 가능한 확산을 피하기 위해 매일 아가로즈 플레이트에 물을 보충.
- dsRNA 주사에 대 한 적절 한 제어를 수행, 완충 주사를 포함 하 여, dsRNA의 주입 관심 의 유전자의 감각 가닥에 대 한 합성, DsRNA의 주사 대상 유기체 내에서 존재 하지 않는 서열에 대 한 합성. 추가 분자 방법을 사용하여 RNAi녹다운확인(22)
5. dsRNA 주사를 위한 T. marmoratus 애벌레의 준비
참고: 초기 단계 배아와는 달리, T. marmoratus 애벌레는 상대적으로 튼튼하고 더 큰 부피로 주입될 수 있습니다. 예를 들어, 제2 인스타는 생존율에 눈에 띄는 부정적인 영향 없이 최소 3μL를 가진 dsRNA 작동 용액의 최대 2 μL 및 세 번째 인별을 주입할 수 있다. dsRNA를 주입하기 위해 아가로즈에 포함시켜 애벌레를 고정시키는 것이 도움이됩니다.
- 애벌레를 수집하기 전에 2 % 아가로즈를 녹여 60-70 °C의 수조에 보관하여 액체 형태로 유지하십시오. 녹은 2% 아가로즈를 작은 페트리 접시에 붓고 굳어질 때까지 냉각하여 고정 접시를 준비합니다. 무딘 집게를 사용하여 주입 될 각 동물에 대해 아가로즈 플레이트 (대략 삽입 할 절지 동물의 크기)에 얕은 홈을 만듭니다.
- 적절한 발달 단계에서 어린 애벌레를 수집하십시오 (분석을 위해 원하는 단계 보다 한 단계 앞선 단계). 이렇게하려면 애벌레가 들어있는 배양 컵에서 물을 조심스럽게 배출하고 아래에 언급 된 단계를 따르십시오.
- 유충이 주목할만한 움직임을 보이지 않을 때까지 얼음에 마취 (조심스럽게 물 컵에서 애벌레를 선택하고 얼음에 부드럽게 배치). 무딘 부드러운 집게를 사용하여 얼음에서 유충을 조심스럽게 들어 올리고 목과 몸을 홈에 있는 고정 단계에 배치하지만, 아가로즈 표면 위에 있는 꼬리는 덮여 있지 않도록 하여 유충이 꼬리 첨탑을 통해 계속 호흡할 수 있게 합니다.
- 유충을 여전히 액체이지만 위험하게 뜨겁지 않은 아가로즈 두꺼운 층으로 덮습니다. 실수로 덮여 있는 경우, 집게를 사용하여 꼬리와 그 아래의 두 세그먼트를 아가로즈에서 풀어 주세요. 일단 아가로즈가 고화되면, dsRNA 주사에 대한 애벌레를 사용합니다.
6. T. marmoratus 애벌레에서 dsRNA 미세 주입
- 원하는 양의 유전자 특이적 dsRNA 작업 용액으로 미세 주입 바늘을 준비하고 백필하십시오(4.1-4.2 단계에서 설명된 대로). 수동으로 제어된 미세 주입 주사기에 연결된 바늘 홀더에 바늘을 부착합니다. 바늘을 부착하기 전에 주사기 플런저를 끝까지 당겨 주입 압력 중간 시술이 부족하지 않도록하십시오.
- 제3 체형과 제4체 체판 세그먼트 사이에 등등적으로 위치한 주입 부위가 미세 주입 바늘의 궤적(스테이지에 거의 평행)에 정렬되는 등 고정된 유충을 배치한다.
참고 : 바늘과 애벌레를 이 위치에 있는 앵글링하는 것은 바늘 팁에 대한 최소 저항의 경로를 제공하여 최소한의 손상으로 애벌레를 천포하기 때문에 중요합니다. - 바늘과 애벌레가 배치되면 스테레오 현미경을 통해 진행 상황을 모니터링하면서 미세 조작기를 사용하여 바늘 끝을 유충으로 조심스럽게 이동합니다.
- 팁이 조직을 천포화 한 후, 천천히 조심스럽게 주사기에 압력을 적용합니다. 현미경의 밑에 바늘에 있는 착색한 주입 액의 움직임을 관찰하여 사출 압력을 조정합니다.
- 조심스럽게 주입 후 바늘을 철회. 부드러운 집게를 사용하여 껍질을 벗기고 아가로즈에서 유충을 풀어냅니다. 유충을 물(실온)이 있는 용기로 다시 옮기고 25\u201228°C의 인큐베이터 또는 배양실에서 더 발전할 수 있으며 14h 빛과 10h 의 빛의 경사이클을 갖는다.
- 4.10 단계에서 설명한 바와 같이 적절한 제어 주사 및 넉다운 정량을 사용하십시오.
결과
상술한 프로토콜을 사용하여, 우리는 3개의 다른 유전자, 즉 백색, 락케이스2(lac2) 및 Lens3(표 1)를선버스트 다이빙 딱정벌레 T. 마마라투스의다양한 발달 단계에서 쓰러뜨렸다.lac2 우리는 배아 발생 중 매우 초기 단계에서 dsRNA를 주입하여 T. marmoratus에서 RNAi를 수행(도 1A). 일부 배아는 공정에서 살아남지 못하고 괴사(도1B)를돌리기 때문에 남은 배아를 건강하게 유지하기 위해 제거해야 합니다. 여기에서 예시된 것은 백색 유전자에 대하여 dsRNA의 주사입니다. 이 유전자는 Drosophila에서 안료(23)의전구체의 섭취 및 저장에 관여하는 3개의 ATP 결합 카세트(ABC) 수송기 중 하나로 잘 알려져 있다. 따라서 기능 상실로 인해 안색되지 않은 백색 눈 표현형이 발생합니다. 우리의 결과는 T. marmoratus 태아에 있는 정형 하 백색 유전자를 표적으로 하는 dsRNA의 주사가 새로 떠오르는 애벌레에 있는 눈 색소침착의 손실로 이끌어 낸다는 것을 보여줍니다. 이 경우 야생 형 애벌레는 심하게 색소된 눈을 특징으로하며, 흰색의 RNAi 녹다운은 다양한 수준의 감소와 눈 안료의 완전한 제거로 이어집니다. 전반적으로, 우리는 살아남은 태아의 34%에서 눈 착색에 있는 적어도 몇몇 감소를 관찰했습니다 (n = 35). 도 2A는 대조군 개인과 약간 밝은 눈 색깔을 가진 개인을 비교합니다. 도 2B는 개별에서 더 심한 녹다운을 보여 주며, 클러스터의 복부 눈(Eyes 2-5)이 완전히 색소화된 반면, 등쪽 눈은 여전히 약간의 색소 침착을 보여줍니다. 이러한 차이는 녹다운의 효율성이 지역적으로 변화할 수 있는 방법을 강조하며, 이는 조밀한 조직에서 dsRNA 침투의 효능의 차이와 관련이 있을 수 있습니다. 또 다른 개별은 모든 눈에서 본질적으로 완전한 안료 손실을 보여줍니다(그림 2C).
T. marmoratus의애벌레 단계에서 RNAi가 얼마나 잘 작동하는지 조사하기 위해, 우리는 탄닌 유전자 lac2를 제 3 의 별로 몰트로 인해Figure 3며칠 전에 두 번째 인스타 애벌레로 표적으로 하는 dsRNA를 주입하고 제 3 의 별 애벌레의 큐티큘러 착색에 미치는 영향을 평가했습니다. Lac2는 단백질을 산화적으로 유도하여 불용성, 단단하고 어둡게 만드는 페놀로시다제의 일종입니다. 밀가루 딱정벌레 T. 카스타뉴움에서 녹다운은 낮은 복용량에서 밝은 색깔의 개인으로 이끌어 내는 것으로 보였지만 고용량24에서치명적인 것으로 간주됩니다. 그림 4는 이 치료법이 밝은 색상의 선버스트 다이빙 딱정벌레 애벌레로 이어진다는 것을 보여줍니다. 구체적으로, 본 실험에서, 살아남은 주입된 애벌레의 75%(n=12)는 색소침착을 감소하였다(대조군내 0%에 비해). 도 4A는 비교적 온화한 탈색을 가진 개별을 나타내고, 도 4C는 표피의 어두운 착색이 거의 없는 T. marmoratus 애벌레의 머리를 보여줍니다. 탈색은 이러한 애벌레에 대한 전형적인 중앙 어두운 패터닝에 특히 분명했지만,이 패턴은 제어 주입 된 개인(도 4B)에서명확하게 볼 수 있었습니다. 또한, 도 4D에묘사된 바와 같이 꼬리 기관체의 번개가 관찰되었다. lac2 dsRNA가 제3의 인스타 애벌레로 주입된 경우, 가벼운 성인 개인이 얻어졌다(도5). 낙엽 딱정벌레의 날개는 특이한 부드러움으로 인해 다소 변형되어 있습니다.
형태학적 특성을 변경하는 것 외에도 RNAi를 사용하여 행동에 영향을 미치는 유전자를 대상으로 할 수 있습니다. 이를 입증하기 위해, 우리는 주요 렌즈 단백질 코딩 유전자에 대하여 RNAi를, Lens318,제 3 성급 애벌레 눈의 광학 적특성에 영향을 미치기 위하여 제2 별 애벌레로 주입했습니다. 여기서 관찰되는 렌즈에 미치는 영향은 T. marmoratus 애벌레의 눈이 이 전환에서 주요 눈 성장을 겪기 때문에 가능성이 높으며, 이는 또한렌즈(25)의주요 광학 변화를 수반한다. 이 실험에서 RNAi 넉다운은 매우 효율적이였습니다. qPCR을 통한 검증은 100% 성공률을 보였습니다. 13명의 시험된 개별 중 12명은 대조군 개인의 발현 수준의 10% 미만으로 쓰러졌으며, 나머지 개인은 대조군 수준의 17%(미공개 관찰)의 발현 수준을 갖는 것으로 나타났다. 현상 수준에서, 일부 개인은 심각하게 장애가 또는 먹이 캡처의 무능력했다, 반복적으로 매우 가까운 거리에서 먹이를 접근하지만 지속적으로 오버 샷 개인에 대한 그림 6에 설명된 바와 같이.
표 1: 화이트, 락2 및 Lens3 단백질을 위한 프라이머 서열 및 앰플리콘. 이 테이블을 다운로드하려면 여기를 클릭하십시오.
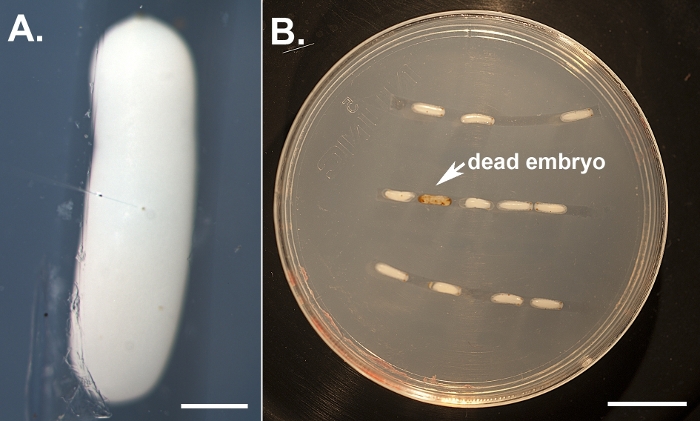
그림 1: 배아 주사의 그림. (A)dechorionated 배아는 아가로즈 플레이트에 줄지어 있고 dsRNA와 식품 염료 함유 주입 버퍼로 채워진 미세 주입 바늘을 사용하여 그들의 센터 근처에 주입. 스케일 바는 500 μm을 나타냅니다. (B)더 심한 넉다운을 가진 개인. 주입된 배아는 습도 챔버에 보관되고 표현형을 채점하고 죽은 개인을 제거하기 위해 매일 모니터링됩니다 (갈색으로 변함). 배율 막대는 5mm를 나타냅니다.
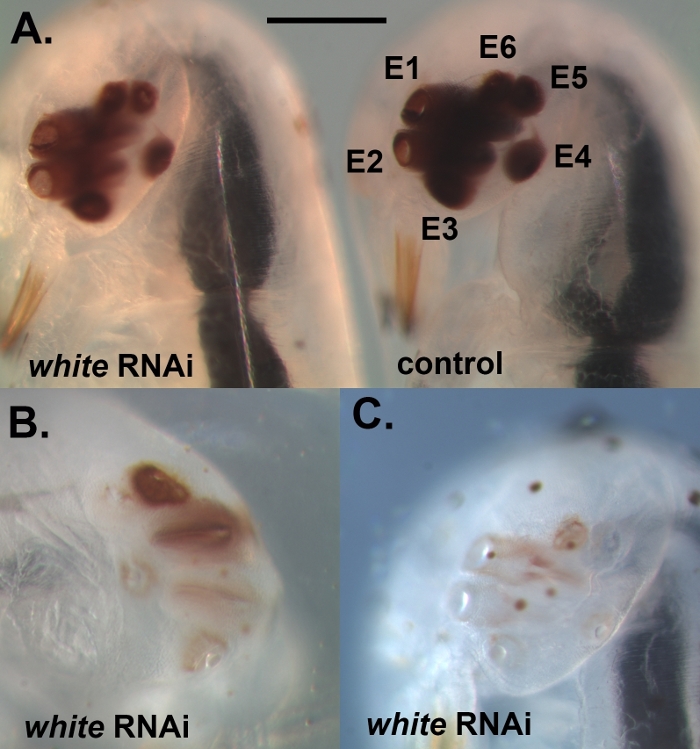
그림 2: 완전히 발달된 배아의 예로, 백색에 대하여 dsRNA로 주입되었다. (A)눈 색소 침착의 감소를 가진 개인의 비교(왼쪽) 및 제어 주입 된 개별 (오른쪽). E1-E6은 눈 1-6을 참조하십시오. (B)더 심한 녹다운 표현형을 가진 개인은 때때로 클러스터 내의 눈 중 일부가 다른 사람보다 녹다운에 의해 더 심각하게 영향을 받는다는 것을 보여줍니다. (C)눈 색소 침착의 거의 완전한 손실을 가진 개인. 스케일 바는 200 μm을 나타냅니다. 패널 B와 C는 동일한 축척으로 표시됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 3: 애벌레 주사의 그림. (A)꼬리 첨탑이 아가로즈를 제거한 2%의 아가로즈에 내장하여 고정된 여러 애벌레. (B)그 팁이 두 세그먼트 사이의 미세 멤브레인을 관통 할 수 있도록 배치 된 사출 용액을 포함하는 마이크로 전극. (C)주입 후 주사 부위에 보이는 청색 사출 염료. 배율 막대는 1cm를 나타냅니다.

도 4: 랑카2용 RNAi는 제3의 인스타 애벌레에서 큐티클 착색을 감소시켜 제2인스타 애벌레에 적용한다. (A)대조군 주입 개인(상단)과 비교했을 때 lac2 RNAi 개별(아래)에서 착색의 비교적 가벼운 손실. 스케일 바는 5mm를 나타낸다.(B)헤드의 중앙에 특징적인 어두운 색 패턴을 보여주는 대조군 개별의 헤드. (C)중앙 머리 착색의 거의 완전한 손실로 이어지는 lac2의 상대적으로 심각한 넉다운. (D)제어 주입 개인(상단)과 비교했을 때 lac2 RNAi 개별(아래)의 주요 꼬리 표절에서 착색의 손실. B\u2012D의 스케일 바는 1mm를 나타냅니다.

그림 5: 락카2용 RNAi는 제3의 인스타 애벌레에 적용되어 성인의 표피 색이 감소시됩니다. 녹다운 개인(왼쪽)은 컨트롤(오른쪽)과 비교할 때 매우 부드러운 엘리트라가 특징입니다. 배율 막대는 5mm를 나타냅니다.
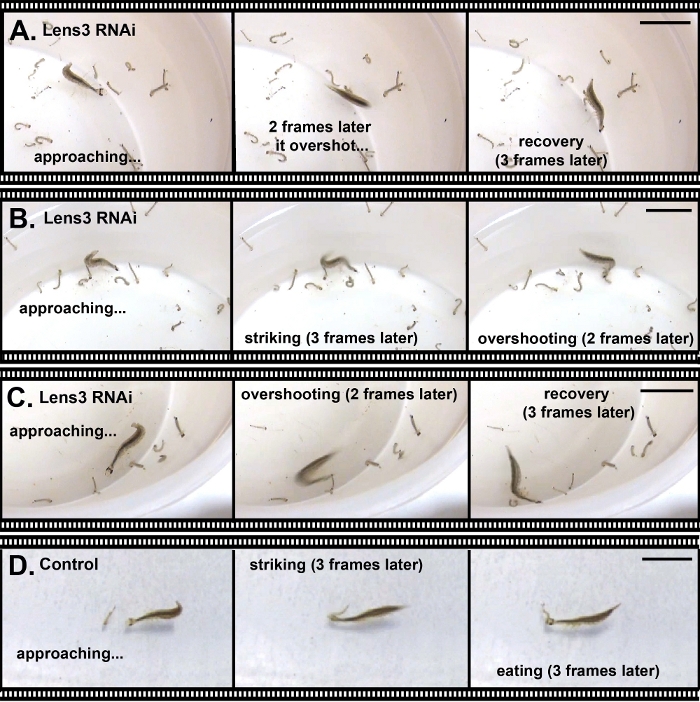
그림 6: 먹이 포획의 결핍으로 이어지는 주요 렌즈 단백질의 넉다운. (A-C). RNAi를 통해 광학 적자를 유도한 Sunburst 다이빙 딱정벌레 애벌레가 먹이(모기 애벌레)를 포획할 수 없었던 세 가지 예. 대표적인 스틸 이미지는 특징적인 먹이 캡처의 비디오 녹화에서 선택되었습니다. (D)먹이 잡기 애벌레 를 제어합니다. 모든 축척 막대는 2cm를 나타냅니다.
토론
우리의 목표는 이 방법의 편집이 RNAi를 널리 사용할 수 있게 할 것입니다, 특히이 도구는 CRISPR / Cas9 기반 유전자 편집에 강력한 시너지 기술을 유지로, 그것은 연구 된 유기체의 원하는 개발 단계에 적용 될 수 있다는 장점. 이 힘을 예시하기 위하여는, 우리는 dsRNA를 태아로 그리고 다른 애벌레 단계로 주입했습니다. 계란에 주사는 배아의 발달에 영향을 미쳤다(도 2),제 2 애벌레 단계로 주사는 세 번째 애벌레 단계에 명백한 효과를 했다(도 4 및 도 6),세 번째 애벌레 단계로 주사 성인에 미치는 영향을 보여 주었다(도 5). 정확한 타이밍실험적으로 설정 해야 하는 동안, 일반적으로, 주사 는 몇 일 이내에 적용. 이 프로세스의 성공은 dsRNA 서열의 길이에 의해 영향을 받을 수 있다. 여기에서 는 200 bp이상에서 800 bp 이상으로 조금 더 사용하는 예제를 제시했습니다. 일반적으로 100~600bp 사이의 시퀀스는 오프타겟 효과를 제한하는 것이 바람직하지만, 최대 1000bp의 시퀀스는 양호한 결과22를산출한다. RNAi에 관한 한 가지 질문은이 기술을 통해 달성 할 수있는 녹다운의 지속 기간입니다. 표현형은 각 단계에서 단자이기 때문에 제시된 결과에 따라 이 문제에 대해 언급할 수 없습니다. 그러나, 그것은 이전에 RNAi 효과 일반적으로 상대적으로 긴 수명이 다는 것을 지적 되었습니다., 그리고 높은 농도 더 오래 지속 녹다운 이어질20.
이 기술의 한 가지 제한은 다른 생물보다 일부 유기체에 더 잘 작동한다는 것입니다, 그것은 우선 순위가 얼마나 잘 작동하는지 예측하는 직접적인 방법이없는 것 같다. 그럼에도 불구 하 고, 그것은 다른 유기체의 큰 범위에 대 한 잘 작동 발견 되었습니다. 절지동물 내에서, 이것은거미류 26,갑각류27,및 딱정벌레28에서특히 높은 성공률을 가진 곤충의 다양한 포함합니다. 추가 합병증은 표현형 엄격에 있는 다름이 dsRNA의 동일 양의 적용에도 불구하고 개별 사이에서 수시로 생기는다는 것입니다. 도 2B에도시된 바와 같이, 개별 내에서도 변형이 발생할 수 있습니다. T. marmoratus 애벌레 눈 발달에 관련 되 었던 다른 유전자를 대상으로 하는 우리의 RNAi 연구 결과에서는, 우리는 몇몇 눈이 그 외 보다는 더 가혹하게 영향을 된다는 것을 자주 발견했습니다. 이 현상은 dsRNA가 단위의 일부에 더 잘 도달할 수 있는 눈 클러스터의 상대적으로 조밀한 조직과 관련될 수 있습니다.
RNAi 실험의 성공적인 실행을 위해, 몇몇 매개 변수는 표적 유전자를 위해 최적화되는 것이 중요합니다. 예를 들어, dsRNA의 농도와 표적 유전자의 길이는결과(20)에강하게 영향을 미칠 수 있다. 또 다른 중요한 매개 변수는 주사가 실행되는 방법입니다, 이 과정은 크게 생존율에 영향을 미칠 수 있기 때문에. 배아의 경우, 우리는 배아의 중심을 표적으로 삼아 최상의 결과를 달성했습니다. 잘 배치 된 플레이트는 단일 세션에서 100 개 이상의 배아를 주입 할 수 있습니다. 애벌레의 경우 세그먼트 사이에 주사 바늘을 삽입하는 것이 중요합니다. 이러한 주사는 더 많은 dsRNA를 필요로하고, 애벌레 가용성에 따라, 여기에 우리의 주사 세트는 일반적으로 한 번에 몇 동물로 구성. 모든 주사의 경우 공기가 유기체에 유입되는 것을 방지하는 것이 중요합니다.
경우에 따라, 유전자 규제 네트워크 및 유전 중복성의 피드백 루프는 일관된 녹다운에도 불구하고 RNAi 표현형의 침투에 영향을 미칠 수 있습니다. 이것은 눈에 띄는 렌즈 단백질의 매우 성공적인 녹다운을 가진 애벌레의 우리의 행동 관측에 대한 경우인 것 같다, Lens318. 우리는 qPCR을 통해 이러한 녹다운의 높은 효율을 확인했지만, 관련 표현형에서 상당한 변화가 관찰되었다. 이 결과는 RNAi 넉다운을 적절하게 정량화할 필요성을 강조합니다(옵션에 대한 자세한 내용은22참조). 결과 표현형에 관하여 명확한 우선 순위 기대가 없는 경우에, RNAi의 오프 표적 효력을 통제하는 좋은 쪽은 dsRNA의 2개의 비 겹치는 순서를 가진 동일 유전자를 표적으로 하고 일반적인 표현형에 대한 결과를 평가하는 것입니다.
유전자 편집 기술과는 달리 RNAi는 치명적인 유전자를 연구하기위한 강력한 도구이며 그렇게하는 방법에는 두 가지가 있습니다. 예를 들어, 개발 초기에 기능 상실이 치명적인 것으로 알려진 유전자의 기능적 기여에 관심이 있는 경우, 동물이 정상적으로 발달하도록 허용한 다음 나중에 개발중(즉, 성인)을 통해 유전자를 쓰러뜨리면 이러한 유전자의 기능적 조사를 달성할 수 있다. 대안적으로, 완전한 기능 상실이 치명적인 것으로 알려진 유전자는 부분적인 녹다운을 통해 조사될 수 있으며, 이는 다양한 dsRNA 농도를 주입하여 달성될 수 있다. 우리의 결과 중 일부는 곤충의 큐티클이 지나치게 부드러운24가되면치명적으로 알려진 lac2의녹다운을 보여줍니다. 도 5에 묘사 된 lac2 RNAi 딱정벌레조차도 외부 실험실 조건에서 살아남을 가능성이 낮습니다. 또 다른 치명적인 유전자는 절단되어,절지동물내의 다양한 장기 시스템에서 세포-운명 사양에 대한 근본적인 전사 인자에 대한 코드이며 드로소필라 시각시스템(29)의glia 발달과 관련이 있다. T. marmoratus 배아에서 절단 RNAi에 우리의 경험을 바탕으로, 우리는 그들의 배아 눈 발달을 완료할 수 있는 태아에 있는 유익한 눈 표현형을 불러 일으킬 수 있습니다 (공개되지 않은 관측). 여기, 더 높은 복용량 높은 치 사 율으로 이어질 표시, 낮은 복용량 관찰 및 유익한 표현형 귀 속 하는 동안.
우리의 프로토콜은 연구원이 T. marmoratus에RNAi 실험을 추구하는 데 필요한 단계를 나열할뿐만 아니라 다른 유기체, 특히 수생 생물에도 일반적으로 적용됩니다. 수생 유기체 중, 이미 물 벼룩 다프니아(30 및 새우와 같은 갑각류 안에 몇 가지 예가 있습니다)(최근 검토에 대 한 참조참조 31참조). 수생 곤충 중 충분한 기회가 있다, 그들은 모든 곤충 다양성의 약 6 %를 구성 하는 것으로 추정 되었습니다., 가능성이 이상 200,000 종32. 또한, RNAi는 이미 수중환경의표면에 서식하는 경향이 있는 물 스트라이더(33)에서 수행되었다. 게놈이 존재하지 않는 경우, 전사는 드 노보를 조립 할 수 있습니다. 이 과정이 수백 개의 뉴클레오티드의 연속을 밝히는 한 특정 유전자에 대한 dsRNA를 설계할 수 있습니다. 아가로즈에서 곤충을 고정하기위한 우리의 프로토콜은 특히 부드럽고 가단성 및 수생 생물에 대한 다른 절차에유용 할 것입니다. 종합하면 RNAi는 다른 분자 및 유전 적 도구가 제공되지 않더라도 다양한 유기체 그룹에서 유전자 발현을 조작하기위한 강력한 기술로 남아 있습니다.
공개
저자는 공개 할 것이 없습니다.
감사의 말
우리는 편집 지원을 위해 선버스트 다이빙 딱정벌레와 타마라 페이스를 키우는 데 도움을 준 생물정보학 인 헤일리 토블러 (Hailey Tobler)와의 도움을 준 조쉬 베노이트 박사에게 감사드립니다. 이 연구는 YT에 대한 IOS-1456757 및 IOS-1856341에 대한 국가 과학 재단에 의해 지원되었습니다.
자료
| Name | Company | Catalog Number | Comments |
| 1. RNA isolation and de novo transcriptome assembly | |||
| liquid nitrogen | University Stockroom | Typically locally available at research institutions | |
| pipette tips | Fisher Scientific | size dependent | Products from other vendors would work equally well |
| RNeasy lipid tissue mini kit (RNA isolation kit) | Qiagen | 74804 | Detailed specific protocol is provided with the kit |
| spectrophotometer (NnanoDrop) | Thermo Fisher | 9836674 | Other models would work equally well |
| 1.2. De novo transcriptome assembly. | |||
| BUSCO.v3 | https://busco.ezlab.org/ | Any freely available software for assessing trasncriptome completeness can be used. | |
| CLC Genomics workbench | Qiagen | 832021 | Other equivalent software packages are also available |
| Galaxy workbench | https://usegalaxy.org/ | An open source online transcriptome assembly & annotation pipeline | |
| NCBI BLASTx | https://blast.ncbi.nlm.nih.gov/Blast.cgi?LINK_LOC=blasthome&PAGE_TYPE=BlastSearch&PROGRAM=blastx | An open source online alignment and annotation pipeline | |
| 2. cDNA synthesis, Cloning & Miniprep isolation | |||
| ampicillin sodium salt | Gibco | 11593-027 | Products from other vendors would work equally well |
| glycerol | Fisher Scientific | G33-500 | Products from other vendors would work equally well |
| LB broth | Fisher Scientific | BP1426-500 | Products from other vendors would work equally well |
| Omniscript RT (reverse trasncription) kit | Qiagen | 205111 | Detailed specific protocol is provided with the kit |
| One Shot™ TOP10 Chemically Competent E. coli | Invitrogen | C404010 | Detailed specific protocol is provided with the kit |
| Petri dishes | Fisher Scientific | FB0875713 | Products from other vendors would work equally well |
| PureLink Quick Plasmid Miniprep Kit | ThermoFischer | 771471 | Detailed specific protocol is provided with the kit |
| QIAquick PCR purification kit | Qiagen | 28104 | Detailed specific protocol is provided with the kit |
| shaker-incubator | Labnet | 211DS | Other models would work equally well |
| spectrophotometer (NnanoDrop) | Thermo Fisher | 9836674 | Other models would work equally well |
| thermal cycler for PCR | BioRad | T100 | Other models would work equally well |
| TOPO TA Cloning kit | Invitrogen | 1845069 | Detailed specific protocol is provided with the kit |
| X-Gal Solution | Thermo Scientific | R0941 | Products from other vendors would work equally well |
| 2.2 PCR amplification & in vitro dsRNA synthesis | |||
| Centrifuge | Fisher Scientific | accuSpin Micro 17R | Other models would work equally well |
| ethanol | Fisher Scientific | A4094 | Products from other vendors would work equally well |
| FastTaq DNA Polymerase, dNTPack | Roche | 13873432 | This kit contains all the reagents necessary for a PCR |
| MEGAclear Transcriiption clean up kit | ThermoFischer | AM1908 | Detailed specific protocol is provided with the kit |
| MEGAscript T7 Transcription kit | ThermoFischer | AM1334 | Detailed specific protocol is provided with the kit |
| nuclease-free water | Fisher Scientific | AM9932 | Products from other vendors would work equally well |
| QIAquick PCR purification kit | Qiagen | 28104 | Detailed specific protocol is provided with the kit |
| sodium acetate | Fisher Scientific | BP333-500 | Make 3M working solution |
| Spectrophotometer (NnanoDrop) | Thermo Fisher | 9836674 | Other models would work equally well |
| 3. Collecting and preparing early stage T. marmoratus embryos for dsRNA injections | |||
| agarose | Fisher Scientific | 9012-36-6 | Products from other vendors would work equally well |
| distilled water | Fisher Scientific | 9180 | Products from other vendors would work equally well |
| forceps (Dumon #4 Biology) | Fine Science Tools | 11242-40 | Products from other vendors would work equally well |
| glass cavity dish (3 well-dish) | Fisher Scientific | 50-243-43 | Products from other vendors would work equally well |
| microwave | Welbilt | turn-table | Other models would work equally well |
| natural hair paintbrush | Amazon | Any fine brush will do | |
| P1000 micro-pipetter | Gilson | F123602 | Other models would work equally well |
| Petri dishes | Fisher Scientific | FB0875713 | Products from other vendors would work equally well |
| stereomicroscope | Microsocope Central | 10446293 | Any good stereomicroscope will work |
| transfer pipettes | Fisher Scientific | 21-200-109 | Products from other vendors would work equally well |
| 4. dsRNA micro-injections in early stage T. marmoratus embryos | |||
| digital camera | Edmund optics (Qimaging) | Retiga 2000R | Other models would work equally well |
| ethanol | Fisher Scientific | A4094 | Products from other vendors would work equally well |
| food dye | Kroger | Any available food dye should work fine | |
| humidity chamber | take any plastic box with a lid, sterilize it with 70% ethanol and let it dry | ||
| incubator | Labline | 203 | Other models would work equally well |
| injection buffer | Prepared for 1 mL following reference #22 : Mix 10 µL of 0.1 M sodium phosphate buffer, 100 µL of 0.5 M potassium chloride solution, 100 µL of food dye and 790 µL of double-distilled water. Store in 4 °C. | ||
| intracellular Microinjection Systems (Picospritzer) | Parker | 052-0500-900 | Currently the model III is available, but older models work also |
| micro needle holder | A-M Systems | 672441 | Other products would work equally well |
| microinjection needles (1.2 mm x 0.68 mm, 4 inches) | A-M Systems | 603000 | Other models would work equally well |
| micromanipulator | Drummond Scientific Company | 3-000-024-R | Any quality micromanipulator will work |
| monosodium phosphate | Fischer scientific | 7558-80-7 | To make 10 mL of 1 M working solution: Add 1.2 g of monosodium phosphate powder to 10 mL of Double distilled water and mix until clear solution is obtained |
| P-1000 Micropipette Puller | Sutter Instrument | P-1000 | Puller settings:Heat 575, Pull 60, Velocity 75 Delay 110, Pressure 700. |
| P10 micro-pipetter | Gilson | F144802 | Other models would work equally well |
| P1000 micro-pipetter | Gilson | F123602 | Other models would work equally well |
| potassium chloride | Fischer scientific | 7447-40-7 | To make 100 mL of 0.5 M potassium chloride solution: Add 3.73 g of potassium chloride crystals to 100 mL of double distilled water and mix until clear solution is obtained. |
| sodium phosphate buffer (0.1M) | To make 10 mL of 0.1 M of this buffer: Mix 8.5 mL of 1 M sodium phosphate dibasic solution with 1.5 mL of 1 M monosodium phosphate solution. Check the pH with a pH meter and adjust accordingly to pH 7.6 at room temprature. | ||
| sodium phosphate dibasic | Fischer scientific | 7558-79-4 | To make 10 mL of 1 M working solution: Add 1.42 g of sodium phosphate dibasic powder to 10 mL of Double distilled water and mix until clear solution is obtained |
| stereomicroscope | Microsocope Central | 10446293 | Any good stereomicroscope will work |
| 5. Preparing T. marmoratus larvae for dsRNA injections | |||
| agarose | Fisher Scientific | 9012-36-6 | Products from other vendors would work equally well |
| forceps (Dumon #4 Biology) | Fine Science Tools | 11242-40 | Products from other vendors would work equally well |
| Petri dishes | Fisher Scientific | FB0875713 | Products from other vendors would work equally well |
| 6. dsRNA micro-injections in T. marmoratus larvae | |||
| injection buffer (10x) | See section 4 | ||
| microinjection needles (1.2 mm x 0.68 mm, 4 inches) | A-M Systems | 603000 | Other models would work equally well |
| microinjection syringe | A-M Systems | 603000 | Other models would work equally well |
| micro needle holder | A-M Systems | 672441 | Other products would work equally well |
| P-1000 Micropipette Puller | Sutter Instrument | P-1000 | Puller settings:Heat 575, Pull 60, Velocity 75 Delay 110, Pressure 700. |
| stereomicroscope | Microsocope Central | 10446293 | Any good stereomicroscope will work |
참고문헌
- Muller, B., Grossniklaus, U. Model organisms - A historical perspective. Journal of Proteomics. 73, 2054-2063 (2010).
- Gilbert, L. A., et al. CRISPR-Mediated Modular RNA-Guided Regulation of Transcription in Eukaryotes. Cell. 154, 442-451 (2013).
- Westerman, E. L., et al. Aristaless Controls Butterfly Wing Color Variation Used in Mimicry and Mate Choice. Current Biology. 28, 3469(2018).
- Perry, M., et al. Molecular logic behind the three-way stochastic choices that expand butterfly colour vision. Nature. 535, 280(2016).
- Boettcher, M., McManus, M. T. Choosing the Right Tool for the Job: RNAi, TALEN, or CRISPR. Molecular Cell. 58, 575-585 (2015).
- Meister, G., Tuschl, T. Mechanisms of gene silencing by double-stranded RNA. Nature. 431, 343-349 (2004).
- Fire, A., et al. Potent and specific genetic interference by double-stranded RNA in Caenorhabditis elegans. Nature. 391, 806-811 (1998).
- Kennerdell, J. R., Carthew, R. W. Use of dsRNA-mediated genetic interference to demonstrate that frizzled and frizzled 2 act in the wingless pathway. Cell. 95, 1017-1026 (1998).
- Mello, C. C., Conte, D. Revealing the world of RNA interference. Nature. 431, 338-342 (2004).
- Agrawal, N., et al. RNA interference: Biology, mechanism, and applications. Microbiol. Microbiology and Molecular Biology Reviews. 67, 657(2003).
- Gu, L. Q., Knipple, D. C. Recent advances in RNA interference research in insects: Implications for future insect pest management strategies. Crop Protection. 45, 36-40 (2013).
- Ravisankar, P., Lai, Y. T., Sambrani, N., Tomoyasu, Y. Comparative developmental analysis of Drosophila and Tribolium reveals conserved and diverged roles of abrupt in insect wing evolution. Developmental Biology. 409, 518-529 (2016).
- Tomoyasu, Y., Wheeler, S. R., Denell, R. E. Ultrabithorax is required for membranous wing identity in the beetle Tribolium castaneum. Nature. 433, 643-647 (2005).
- Tomoyasu, Y., Arakane, Y., Kramer, K. J., Denell, R. E. Repeated Co-options of Exoskeleton Formation during Wing-to-Elytron Evolution in Beetles. Current Biology. 19, 2057-2065 (2009).
- Yang, X., et al. Probing the Drosophila retinal determination gene network in Tribolium (II): The Pax6 genes eyeless and twin of eyeless. Developmental Biology. 333, 215-227 (2009).
- ZarinKamar, N., et al. The Pax gene eyegone facilitates repression of eye development in Tribolium. Evodevo. 2, 15(2011).
- Linz, D. M., Clark-Hachtel, C. M., Borras-Castells, F., Tomoyasu, Y. Larval RNA Interference in the Red Flour Beetle, Tribolium castaneum. Journal of Visualized Experiments. , e52059(2014).
- Stahl, A., S, B. R., Cook, T. A., Buschbeck, E. K. A complex lens for a complex eye. Integrative and Comparative Biology. 57, 1071-1081 (2017).
- Yeku, O., Frohman, M. A. Rapid Amplification of cDNA Ends (RACE). RNA, Methods In Molecular Biology. , Springer. 107-122 (2011).
- Miller, S. C., Miyata, K., Brown, S. J., Tomoyasu, Y. Dissecting Systemic RNA Interference in the Red Flour Beetle Tribolium castaneum: Parameters Affecting the Efficiency of RNAi. Plos One. 7, (2012).
- Tomoyasu, Y., Denell, R. E. Larval RNAi in Tribolium (Coleoptera) for analyzing adult development. Development Genes and Evolution. 214, 575-578 (2004).
- Philip, B. N., Tomoyasu, Y. Molecular Methods for Evolutionary Genetics. Orgogozo, V., Rockman, M. V. , Humana Press. 471-497 (2011).
- Mackenzie, S. M., Howells, A. J., Cox, G. B., Ewart, G. D. Sub-cellular localisation of the White/Scarlet ABC transporter to pigment granule membranes within the compound eye of Drosophila melanogaster. Genetica. 108, 239-252 (2000).
- Arakane, Y., Muthukrishnan, S., Beeman, R. W., Kanost, M. R., Kramer, K. J. Laccase 2 is the phenoloxidase gene required for beetle cuticle tanning. Proceedings of the National Academy of Sciences of the United States of America. 102, 11337-11342 (2005).
- Werner, S., Buschbeck, E. K. Rapid and step-wise eye growth in molting diving beetle larvae. Journal of Comparative Physiology. 201, 1091-1102 (2015).
- Prpic, N. M., Schoppmeier, M., Damen, W. G. Gene Silencing via Embryonic RNAi in Spider Embryos. Cold Spring Harbor protocols. 3, 1-4 (2008).
- Sagi, A., Manor, R., Ventura, T. Gene Silencing in Crustaceans: From Basic Research to Biotechnologies. Genes. 4, 620-645 (2013).
- Zhu, K. Y., Palli, S. R. Annual Review of Entomology. Douglas, A. E. 65, Annual Reviews. 293-311 (2020).
- Bauke, A. C., Sasse, S., Matzat, T., Klaembt, C. A transcriptional network controlling glial development in the Drosophila visual system. Development. 142, 2184(2015).
- Lin, C. Y., et al. RNAi analysis of doublesex1 expression in Daphnia pulex Leydig, 1860 (Cladocera). Crustaceana. 92, 137-154 (2019).
- Nguyen, D. V., Christiaens, O., Bossier, P., Smagghe, G. RNA interference in shrimp and potential applications in aquaculture. Reviews in Aquaculture. 10, 573-584 (2018).
- Dijkstra, K. D. B., Monaghan, M. T., Pauls, S. U. Annual Review of Entomology. Berenbaum, M. R. 59, Annual Reviews. 143-163 (2014).
- Crumiere, A. J. J., Khila, A. Hox genes mediate the escalation of sexually antagonistic traits in water striders. Biology. Letters. 15, 5(2019).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유