需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
实现蛋白质快速光化学氧化的实时补偿,用于确定蛋白质地形变化
摘要
蛋白质的快速光化学氧化是蛋白质结构表征的新兴技术。不同的溶剂添加剂和配体具有不同的羟基基清除特性。为了比较不同条件下的蛋白质结构,需要实时补偿反应中产生的羟基基,使反应条件正常化。
摘要
蛋白质的快速光化学氧化(FPOP)是一种基于质谱的结构生物学技术,可探寻蛋白质的溶剂可访问表面积。该技术依赖于氨基酸侧链的反应,羟基基在溶液中自由扩散。FPOP通过激光光解过氧化氢产生这些基原,产生一阵羟基基,以微秒的速度耗尽。当这些羟基基与溶剂可访问的氨基酸侧链发生反应时,反应产品表现出质量变化,可以通过质谱法进行测量和量化。由于氨基酸的反应速率部分取决于该氨基酸的平均溶剂可访问表面,因此蛋白质给定区域的氧化量的测量变化可与不同构象之间该区域溶剂可访问性的变化直接相关(例如,配体结合与无配体、单体与聚合等)FPOP在生物学中已经应用了许多问题,包括蛋白质-蛋白质相互作用、蛋白质构象变化和蛋白质配体结合。由于羟基基的可用浓度因FPOP实验中的许多实验条件而异,因此监测蛋白质分析物所暴露的有效基量非常重要。通过采用内联剂量计测量来自FPOP反应的信号,实时调整激光荧光度,实现所需的氧化量,从而有效地实现了这种监测。通过这种补偿,可以在异质样品中使用相对较低的样本确定反映构象变化的蛋白质地形变化、配体结合表面和/或蛋白质-蛋白质相互作用界面。
引言
蛋白质的快速光化学氧化(FPOP)是一种新兴的技术,通过超快速共价修饰蛋白质的溶剂暴露表面积,然后通过LC-MS1检测,确定蛋白质地形变化。FPOP通过紫外线激光闪光光解过氧化氢,在原位产生高浓度的羟基基。这些羟基基是非常反应和短寿命,消耗约一微秒的时间尺度在FPOP条件2。这些羟基基通过水扩散,以动力学速率氧化溶液中的各种有机成分,通常从快速(+106 M-1 s-1)到扩散控制3。当羟基基遇到蛋白质表面时,该基会氧化蛋白质表面的氨基酸侧链,导致该氨基酸(最常见的是净添加一个氧原子)的质量转移4。任何氨基酸的氧化反应速率取决于两个因素:该氨基酸的固有反应性(取决于侧链和序列上下文)4、5,以及该侧链与扩散羟基基的可访问性,这与平均溶剂可访问表面积6、7,密切相关。除甘氨酸外,所有标准氨基酸在FPOP实验中被这些高活性羟基基所观察到,尽管产量差别很大;在实践中,Ser,Thr,Asn和Ala很少在大多数样品中被视为氧化,除了在高基剂量下,并经仔细和敏感的针对性ETD碎片8,9,识别。氧化后,对样品进行淬火,以去除过氧化氢和二次氧化剂(超氧化物、单氧、过氧化氢等)然后对淬火样品进行蛋白质化消化,产生氧化肽的混合物,其中结构信息作为化学"快照"冻结在各种肽的氧化产物的形态中(图1)。与质谱法 (LC-MS) 耦合的液相色谱用于测量给定蛋白糖解肽中氨基酸的氧化量,该肽的氧化和未氧化版本的相对强度。通过比较在不同构象条件下获得的相同蛋白质的氧化足迹(例如,配体结合与无配体),蛋白质给定区域的氧化量差异可与该区域6、7,的溶剂可访问表面积的差异直接相关。提供蛋白质地形信息的能力使FPOP成为蛋白质高阶结构测定的有吸引力的技术,包括在蛋白质治疗发现和开发10,11。10,

图1:FPOP概述。蛋白质的表面由高活性羟基基共价修饰。羟基基会以受侧链溶剂可访问性强烈影响的速率与蛋白质的氨基酸侧链发生反应。地形变化(例如,由于上文所示配体结合)将保护相互作用区域的氨基酸,防止与羟基基发生反应,从而降低LC-MS信号中改性肽的强度。 请单击此处查看此图的较大版本。
FPOP溶液中存在的不同成分(例如配体、辅料、缓冲液)对过氧化氢3激光光解产生的羟基基具有不同的清除活性。同样,过氧化物浓度、激光流量和缓冲成分的一个小变化可能会改变有效的基剂量,使FPOP数据的复制在样品之间和不同实验室之间具有挑战性。因此,重要的是能够比较羟基基剂量可用于与蛋白质在每个样品使用几个可用的羟基基剂量计之一12,13,14,15,16。,13,14,15,16羟基基剂量计通过与羟基基池中的分析剂(以及溶液中的所有清除剂)竞争来表现;通过测量剂量计的氧化量来测量羟基基的有效剂量。请注意,"有效的羟基基剂量"是羟基基的初始浓度和基的半寿命的函数。这两个参数部分相互依赖,使得理论动力学建模有些复杂(图2)。两个样本可能具有完全不同的初始激进半生命,同时仍然保持相同的有效基剂量,通过改变形成羟基基的初始浓度;它们仍然会产生相同的足迹17。腺素13和Tris12是方便的羟基基剂量计,因为它们的氧化水平可以通过紫外线光谱测量实时,使研究人员能够快速识别何时存在有效的羟基基剂量问题,并解决他们的问题。为了解决这个问题,一个直联剂量计位于流系统直接后,辐照站点,可以监测信号从腺素吸收变化实时是重要的。这有助于在缓冲液或任何其他辅料中进行FPOP实验,其羟基基清除能力水平差异很大17。这种基量补偿可以实时进行,通过调整有效基剂量,为同一顺从者产生统计上无法区分的结果。
在该协议中,我们有详细的程序,以使用腺宁作为内部光学基剂量计进行典型的FPOP实验,采用激进剂量补偿。此方法允许调查人员通过实时执行补偿来比较具有不同清理容量的 FPOP 条件下的足迹。
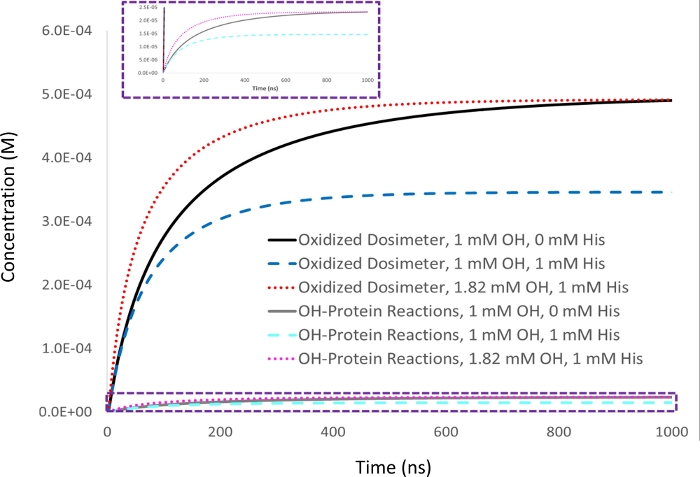
图2:基于剂量的补偿的动力学模拟。1 mM 腺苷剂量计响应以 5 μM 解酶分析素测量,初始羟基基浓度为 1 mM(▪OH t1/2=53 ns),并设定为目标剂量计响应(黑色)。在添加 1 mM 的清道夫辅料组蛋白时,剂量计响应(蓝色)会随着蛋白质氧化量的比例(青色)而降低。羟基基的半寿命也会降低(▪OH t1/2=39 ns)。当产生的羟基基量增加,以在样品中给予与1 mM组蛋白清除剂相当的氧化剂量,在没有清道夫(红色)的情况下,用1 mM羟基基达到,发生类似的蛋白质氧化量(品红色),而羟基半寿命进一步降低(▪OH t1/2=29 ns)。改编自夏普J.S.,Am Pharmaceut Rev 22, 50-55, 2019. 请单击此处查看此图的较大版本。
研究方案
1. 为 FPOP 准备光学台和毛细管
注意:KrF 兴奋激光器是极端对眼的危害,直接或反射光可造成永久性眼睛损伤。始终佩戴适当的眼罩,尽可能避免光束路径附近出现任何反射物体,并使用工程控制来防止未经授权访问有源激光并抑制任何杂散反射。
- 准备 FPOP 光学台。
- 打开激光进行预热。将激光设置为外部触发器、恒定能量、无气体更换。设置每个脉冲的激光能量(通常在 80-120 mJ/脉冲之间)。
- 直接在激光束的路径上设置带平面凸透镜(30 毫米直径 x 120 mm FL 未涂层)的光学台,并设置一个非反射后挡板来吸收光线,如图 3A 所示。

图3:FPOP实验的光学台。(A) 样本与H2 O2、腺素基剂量计、谷氨酰胺清除物混合并放入注射器中。2样品通过 KrF 兴奋紫外激光器的聚焦光束路径通过熔融石英毛细管。紫外线光解H2O2成羟基,氧化蛋白质和腺苷剂量计。在下一个激光脉冲之前,注射器流将发光样品从激光路径中推开,在照明区域之间具有未发光的排除体积。氧化后,样品立即通过内联紫外分光光度计,测量腺氨酸在265nm的紫外线吸收度。然后将样品沉积到淬火缓冲液中,以消除剩余的H2O2和二次氧化剂。(B) 在照射贴在毛细管后面的彩色便笺后,在248纳米时测量斑点尺寸。点宽用于计算采样流速,点中心毛细管的轮廓用于对齐光学台。请单击此处查看此图的较大版本。
- 切割适当长度的熔融石英毛细管(360 μm 外径和100μm内径),并使用套筒,使用低死体积连接器连接到气密注射器。
- 在样品激光照射后,内联剂量计在 265 nm 处读取吸收信号的地方用丁烷割炬轻轻燃烧毛细管的聚酰亚甲涂层。在无绒擦拭上轻轻使用甲醇擦拭毛细管上的碎屑。激光发生地点的聚酰亚胺涂层可以同样用丁烷割炬燃烧,也可以在低功耗下用兴奋激光燃烧。
注意:等待毛细管冷却,因为使用热毛细管上的甲醇是火灾危险。 - 将毛细管穿过激光的光束路径并放入内联剂量计中。
- 按下内联剂量计顶部的操纵杆以打开铰链。拆下磁座。将毛细管放在内联剂量计的加工槽中,使用磁座将毛细管保持到位。将剂量计铰链合上毛细管,按压它,直到操纵杆锁定到位。
- 使用剂量测量软件,单击"开始 闪光" 按钮开始发射兴奋激光。在激光控制软件本身上设置 50-100 mJ/脉冲之间的预设激光功率,并在剂量测量软件的"设置"选项卡中设置 10-20 Hz 之间的预设重复率。
- 使用安装在线性电动舞台上的平面凸透镜聚焦激光束。测量光管上粘胶上毛细管位置的激光点的宽度和高度,精确使用卡钳计算出事荧光(mJ/mm2),如图 3B所示。
- 在毛细管附近放置一个不透明的光圈,以确保毛细管的照明宽度一致,无论由于镜头的移动或激光18每个脉冲的能量的变化,光束大小如何变化。
- 使用激光发射,移动电动舞台通过其运动范围。确保光束保持居中于孔径,并在整个过程中可以观察到毛细管的轮廓。孔径的直径必须小于电动舞台范围内每个点的冲击聚焦光束的宽度。
- 以 20 μL/min 的热水穿过毛细管至少一分钟,清洗毛细管。
- 单击 剂量计软件上的"开始数据 + 自动零"按钮,将剂量计归水,并开始数据收集。
注:如果 FPOP 的缓冲系统在 265 nm 时具有显著的紫外线吸收度,则 FPOP 系统应对缓冲液进行零点,而不是对水进行归零。
- 单击 剂量计软件上的"开始数据 + 自动零"按钮,将剂量计归水,并开始数据收集。
- 在注射器泵上设置计算的流量。
- 蛋白质样本的流速取决于每枪辐照体积(VIrr)、秒激光拍摄次数(R)和所需的未辐照排除体积分数(FEx),以校正层流效应和样品扩散(建议为0.15-0.30)2、19、20。2,19,20使用以下方程,根据毛细血管的内径(毫米 (d)和冲击毛细管的激光点(即孔径宽度)(毫米的宽度)计算 VIrr(以μL 为单位)
VIrr = π (d/2)2w - 基于以下方程计算所需的流速(以 +L/分钟为单位)
流量 = 60R=VIrr (1 = FEx)=
- 蛋白质样本的流速取决于每枪辐照体积(VIrr)、秒激光拍摄次数(R)和所需的未辐照排除体积分数(FEx),以校正层流效应和样品扩散(建议为0.15-0.30)2、19、20。2,19,20使用以下方程,根据毛细血管的内径(毫米 (d)和冲击毛细管的激光点(即孔径宽度)(毫米的宽度)计算 VIrr(以μL 为单位)
2. 为FPOP制备蛋白质溶液
- 在两种或两种不同条件下准备蛋白质进行比较(例如,配体结合和无配体;聚合和单体;单独和与蛋白质结合伙伴;等等),以检测构象变化。
- 设置用于 FPOP 的总体积,以满足实验的需要。最小限值通常取决于辐照毛细管的体积以及强健检测和相对定量所需的材料,并且将主要取决于使用的 LC-MS/MS 系统和标签后样品处理方法。加入过氧化氢后,我们组常用的FPOP溶液的总量为20μL。蛋白质的最终浓度通常是1-10 μM,含有17 mM谷氨酰胺(以限制羟基基的使用寿命),1 mM腺苷(作为基质计)13、17,17和10m的磷酸盐缓冲液(羟基基的缓冲液)。样本通常使用多个复制进行准备,以便对结果进行统计建模。
- 对于大多数一般用途,在两种状态下制备三钙样品,以及至少一个样品作为无激光控制来测量背景氧化。准备此 FPOP 解决方案组合的 18 μL。
注:生物化学中常用的许多缓冲液和添加剂都是羟基基清除剂。可以使用这些添加剂和缓冲液;然而,由于羟基基清除缓冲液,氧化的减少可能发生。一般来说,将所有添加剂保持生物系统要求的最低水平,以最大限度地提高蛋白质氧化产量。应避免使用二甲基硫化物,因为二次基的倾向;二甲基酰胺一直是我们手中的有用替代品。当使用强羟基基清除液的缓冲器时,谷氨酰胺通常可以从FPOP溶液混合物中排除。
- 对于大多数一般用途,在两种状态下制备三钙样品,以及至少一个样品作为无激光控制来测量背景氧化。准备此 FPOP 解决方案组合的 18 μL。
- 在FPOP实验前立即准备1M过氧化氢。
注:供应商通常销售的30%过氧化氢包括稳定剂,可延长保质期。一旦稀释,过氧化氢应该迅速使用,肯定在同一天。过氧化氢还应定期测试FPOP使用羟基基剂量计进行分解。 - 制备含有25μL淬火溶液的微离心管,其内含5微克酸酯和0.5微克/μL的二恶英酶。如果将大于 20 μL 的样品体积用于 FPOP,请按比例增加淬火溶液体积。
3. 执行 FPOP 实验
- 在FPOP溶液混合物的18μL中加入2μL过氧化氢。将内装物轻轻与移液器混合,然后快速将溶液旋转到微离心管的底部。立即使用气密注射器收集并加载到注射器泵中。
- 单击剂量计软件上的"启动泵"按钮,以步骤 1.8.1(通常在 8-16 μL/min 之间)确定的Start Pump流量启动注射器泵上的流量。
- 使用内联剂量计监控实时腺素读数(参见 材料表),并收集废物中的样品。等待 Abs265 信号稳定。
- 单击剂量 计软件 中的"开始闪光"按钮,以预设的重复率和能量开始发射激光。
- 使用内联剂量计监控实时腺素读数(参见 材料表);Abs265 与激光关闭和激光打开的差异是 μAbs265 读数。
注:在存在过氧化氢的情况下发射激光时,高度不稳定的 Abs265 读数的出现是由于溶液中产生气泡。减少激光和/或过氧化氢浓度的荧光,以消除气泡。
4. 执行补偿
注:不同的配体、缓冲液等对羟基基体可能有不同的清除能力。重要的是要确保可比的有效羟基基剂量可用于与不同样品的蛋白质反应。这是通过确保样品之间的相等羟基基剂量计响应来完成的。使用腺苷剂量法,265 nm (+Abs265)的紫外线吸收率变化反映了有效的羟基基剂量;μAbs265越大,有效的羟基基剂量越高。
- 将获得的 +Abs265 读数与内联剂量计与先前实验或控制获得的所需 +Abs265 读数进行比较。μAbs265 读数低于所需读数,表示有效剂量的羟基基不足;a =Abs265 读数表示有效激进剂量过高。如果 +Abs265 读数处于所需水平,则在淬火缓冲液 17 中激光照射后立即收集样品。
- 补偿有效的激进剂量,使[Abs265]均等。这种补偿可以通过三种方式进行:改变过氧化氢浓度,通过改变每个脉冲的激光能量来增加激光荧光,或者通过改变对焦透镜的焦平面来增加激光荧光。
- 要在 +Abs265 读数中做出较大的更改 (>10 mAU),请使用或多或少的过氧化氢重新制作样品,然后根据第 3 节重新运行样品。
- 要实时更改 +Abs265 读数,请使用 50 mm 电动舞台调整对焦镜头的位置,调整射点光束的焦平面。使焦点平面更接近毛细管的位置将增加 +Abs265 读数 ;使焦平面远离毛细管的位置将减少 +Abs265 读数。
- 监测腺氨酸+Abs265, 以测量激光照射13后样品中存在的羟基基的有效量。使用内联紫外毛细管检测器进行实时监控,可实现 4.2.2 中所述的实时补偿;使用电动舞台调整镜头位置,直到 μAbs265 读数等于所需读数。使用紫外分光光度计的实验后吸收度测量也是准确的,但需要在每个有效激进剂量中使用新的样品。
5. 消化蛋白质样本
注:三辛是最常用的消化蛋白质样本的FPOP,是本协议中使用的蛋白酶。它是一种可靠的蛋白酶,在N-和C-术语中产生具有基本位点的肽,促进MS中的多增带电肽离子。此外,它在裂解后裂解裂解,精氨酸是两种氨基酸,对羟基基只具有中度反应;因此,由于分析剂氧化引起的消化模式变化是罕见的。其他蛋白酶已成功与FPOP21一起使用,但应注意确保消化模式在未氧化样品和氧化样品之间具有可比性。
- 测量淬火 FPOP 样品的最终体积。加入 500 mM Tris,pH 8.0 与 10 mM CaCl2 包含 50 mM 二核石醇 (DTT) 到蛋白溶液后淬火到 50 mM Tris 的最终浓度,1 mM CaCl2 和 5 mM DTT。
- 在95°C加热蛋白质样品15分钟。
- 立即将样品在冰上冷却 2 分钟。
- 将1:20的尝试性/蛋白质重量比添加到样品中。
- 在37°C下将蛋白质在37°C下与混合一起消化过夜。
- 通过添加 0.1% 的甲酸和/或加热样品到 95°C 10 分钟,停止消化反应。
- 将 2 mM DTT 添加到样品中,在 LC-MS/MS 之前在 60°C 下加热 15 分钟。
注:虽然其他组在FPOP实验中报告了硫醇的烷基化,但我们在手中注意到氧化蛋白烷基化的侧产物(可能是由于与作为小氧化产物形成的嗜核碳基的反应)。因此,我们尽可能避免硫醇的烷基化。
6. 执行液相色谱-串联质谱(LC-MS/MS)
- 准备移动阶段 A 由含有 0.1% 甲酸的水组成,移动阶段 B 由乙酰三叶酸和 0.1% 的甲酸组成。
- 首先将样品装到 C18 陷阀柱上(300 μm I.D. x 5 mm 100 μ 孔径,5 μm 颗粒大小)陷夹盒,用 2% 溶剂 B 在 5.0 μL/min 的流速下洗涤 3 分钟,以去除盐类和亲水小分子。
- 然后以 300 nL/min 的流速分离 C18 纳米柱上的肽(0.75 mm x 150 mm,2 μm 颗粒大小,100 μ孔径)。梯度包括从2%到35%的溶剂B在22分钟内线性增加,在5分钟内渐到95%溶剂B,并保持3分钟洗柱,然后在3分钟内返回2%B,并保持9分钟重新平衡柱。
注:此梯度足以满足大多数寻求肽水平定量的一蛋白和双蛋白FPOP混合物的LC-MS/MS。在极少数情况下,由于相似的保留时间和 m/z 值,多肽相互干扰,可能需要改变溶剂 B 的百分比以提高肽的分辨率。蛋白体尺度FPOP22或实验设计寻求分离肽氧化产物异构体1,1,23,24,25可能需要更长的LC梯度,超出了本报告的范围。23,24,25 - 使用导电纳米喷雾发射器将肽直接插入高分辨率质谱仪的纳米喷光源中。
- 以正离子模式获取数据。将喷雾电压设置为 2400 V,将离子输送管的温度设置为 300 °C。
- 以 m/z 250 到 2000 的标称分辨率获得从 m/z 250 到 2000 的完整 MS 扫描,然后是 8 个后续数据依赖线性离子陷阱 MS/MS 扫描,使用碰撞诱导的 35% 法化能量在 35% 的规范化能量下对前 8 个最丰富的肽离子进行分离,以识别肽。在 30 s 内将肽分割五次,然后转移到 60 s 的排除列表中。
7. 肽平均氧化的数据处理和计算
- 使用 MS/MS 蛋白质组学搜索引擎确定未氧化肽的蛋白质 、m/z 值和保留时间的顺序覆盖率。
- 将前体质量公差设置为 10 ppm,并使用标准的 trypsin 裂解特异性,为 trypsin 消化样品留出最多两个错过的裂解位点。
- 将肽质量碎片质量耐受性设置为 0.4 道尔顿。
- ,根据检测到的未修改肽的m/z比和主要氧化产物的已知质量变化,,计算每种肽4、26、27、28、29的各种,26理论氧化产物的m/z。,2927
- 使用软件识别这些 m/z 值的提取离子色谱图以查看质谱运行(图 4)。根据肽氧化产物的 m/z、其电荷状态以及洗脱时间与未修改肽的相似性确定肽氧化产物。在我们的手中,肽氧化产物在未修改肽使用上面的LC梯度后240秒至180秒之间。由于氧化通常会导致多种异构产物,因此在肽氧化产物的提取离子色谱中经常观察到多个部分解析的峰值,如图 4所示。肽氧化产物根据提取的离子色谱中的峰面积进行量化。
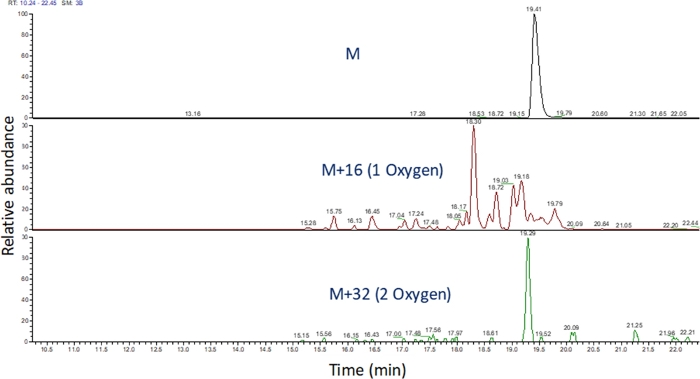
图4:FPOP后提取肽及其氧化产物的离子色谱。肽 氧化产物的m/z 基于未 氧化肽的m/z 和已知的氧化产物计算;确定这些肽产品的区域。然后,肽产物的面积用于计算每个肽的平均氧化事件。 请单击此处查看此图的较大版本。
- 使用以下方程计算肽的平均氧化。
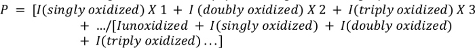
其中 P 表示每个肽分子的平均氧化事件数 ,I 表示未氧化肽(未氧化化)的峰值区域,以及具有 n 氧化事件的肽的峰值区域。请注意 ,I(单氧化) 不仅包括单个氧原子的添加,还包括研究者可能选择测量的其他不太常见的单氧化事件(例如,氧化脱氧、碳基形成等)。4,26,27,28,29。
结果
在磷酸盐缓冲液中对阿达利马布生物仿制药的重链肽足迹进行比较,在55°C加热1小时时,显示有趣的结果。学生的 t-检验用于识别这两个条件下有显著变化的肽(p ≤ 0.05)。肽20-38、99-125、215-222、223-252、260-278、376-413和414-420在蛋白质加热形成集料时对溶剂有显著的保护(图5)30。本实验确定了在加热和聚集时发生地形变化的肽区域。
讨论
基于质谱的结构技术,包括氢-铀交换、化学交汇、共价标记以及原生喷雾质谱和离子流动性,由于其灵活性、灵敏度和处理复杂混合物的能力,已迅速普及。FPOP 拥有几个优势,提高了其在质谱基结构技术领域的知名度。与大多数共价标记策略一样,它提供了与大多数贴标后工艺(如三辛消化、脱氧核二苯甲酸酯等)兼容的蛋白质地形稳定的化学快照,避免了阻碍氢-铀交换的回交换和扰动问题。...
披露声明
Joshua S. Sharp披露了GenNext技术公司的重大财务利益,该公司是一家寻求将蛋白质高阶结构分析(包括羟基基蛋白足迹)技术商业化的小型公司。
致谢
我们感谢国家普通医学科学研究所的研究资金,资助 R43GM125420-01 支持台式 FPOP 设备和 R01GM127267 的商业开发,用于开发高能 FPOP 的标准化和剂量测定协议。
材料
| Name | Company | Catalog Number | Comments |
| Adenine | Acros Organics | 147440250 | Soluble in water upto 3.5 mM |
| Aperture | Edmund Optics | 39-905 | 1000 μm Aperture Diameter, Gold-Plated Copper Aperture |
| Aperture holder | Edmund Optics | 53-287 | 25.8mm Outer Diameter, Precision Pinhole Mount |
| Catalse | Sigma Aldrich | C-40 | Catalase from bovine liver, lyophilized powder, ≥10,000 units/mg protein |
| COMPex Pro laser | Coherent | 1113836 | COMPexPRO 102, F-Vversion, KrF laser, No XeCl |
| Dithiotheitol (DTT) | Promega | V3151 | DTT, Molecular Grade (DL-Dithiothreitol) |
| Fraction collector | GenNext Technologies, Inc. | N/A | Automated fraction collector |
| Fused silica capillay | Molex | 1068150023 | Polymicro Flexible Fused Silica Capillary Tubing, Inner Diameter 100 µm, Outer Diameter 375 µm, TSP100375 |
| Glutamine | Acros Organics | 119951000 | L(+)-Glutamine, 99% |
| Holder for lens | Edmund Optics | 03-668 | 53 mm Outer Diameter, Three-Screw Adjustable Ring Mount |
| Hydrogen peroxide | Fisher Scientific | H325-100 | Hydrogen Peroxide, 30% (Certified ACS), Fisher Chemical |
| LC-MS/MS system | Thermo Scientific | IQLAAEGAAPFADBMBCX | Dionex Ultimate 3000 coupled to Orbitap Fusion Tribrid mass spectrometer |
| Mas spec grade Acetonitrile | Fisher Scientific | A955-1 | Acetonitrile, Optima LC/MS Grade, Fisher Chemical |
| Mass spec grade formic acid | Fisher Scientific | A117-50 | Formic Acid, 99.0+%, Optima™ LC/MS Grade, Fisher Chemical |
| Mass spec grade water | Fisher Scientific | W6-4 | Water, Optima LC/MS Grade, Fisher Chemical |
| MES buffer | Sigma Aldrich | M0164 | MES hemisodium salt |
| Methionine amide | Bachem | 4000594.0005 | H-met-NH2.HCl |
| Micro V clamp | Thor Labs | VK250 | Micro V-clamp with stainless steel blades |
| Motorized stage | Edmund Optics | 68-638 | 50mm Travel Motorized Stage System with Manual Control |
| Nano C18 colum | Thermo Scientific | 164534 | Acclaim PepMap 100 C18 HPLC Columns |
| Optical bench | Edmund Optics | 56-935 | 18" x 18" breadboard |
| Pioneer FPOP Module System | GenNext Technologies, Inc. | N/A | Inline FPOP Radical Dosimetry System |
| Post holder | Edmund Optics | 58-979 | 3" Length, ¼-20 Thread, Post Holder |
| Sodium phosphate dibasic | Fisher Scientific | BP331-500 | Sodium Phosphate Dibasic Heptahydrate (Colorless-to-White Crystals), Fisher BioReagents |
| Sodium phosphate monobasic | Fisher Scientific | BP330-500 | Sodium Phosphate Monobasic Monohydrate (Colorless-to-white Crystals), Fisher BioReagents |
| Syringe | Hamilton | 81065 | 100 µL, Model 1710 RN SYR, Small Removable NDL, 22s ga, 2 in, point style 3 |
| Syringe pump | KD Scientific | 788101 | Legato 101 syringe pump |
| Trap C18 column | Thermo Scientific | 160454 | Thermo Scientific Acclaim PepMap 100 C18 HPLC Columns |
| Tris | Sigma Aldrich | 252859 | Tris(hydroxymethyl)aminomethane |
| Trypsin | Promega | V5111 | Sequencing Grade Modified Trypsin |
| UV plano convex lens | Edmund Optics | 84-285 | 30 mm Dia. x 120 mm FL Uncoated, UV Plano-Convex Lens |
参考文献
- Kaur, P., Kiselar, J., Yang, S., Chance, M. R. Quantitative protein topography analysis and high-resolution structure prediction using hydroxyl radical labeling and tandem-ion mass spectrometry (MS). Molecular & Cellular Proteomics. 14 (4), 1159-1168 (2015).
- Hambly, D. M., Gross, M. L. Laser flash photolysis of hydrogen peroxide to oxidize protein solvent-accessible residues on the microsecond timescale. Journal of the American Society for Mass Spectrometry. 16 (12), 2057-2063 (2005).
- Buxton, G. V., Greenstock, C. L., Helman, W. P., Ross, A. B. Critical review of rate constants for reactions of hydrated electrons, hydrogen atoms and hydroxyl radicals (·OH/·O- in Aqueous Solution. Journal of Physical and Chemical Reference Data. 17 (2), 513 (1988).
- Xu, G., Chance, M. R. Radiolytic modification and reactivity of amino acid residues serving as structural probes for protein footprinting. Analytical Chemistry. 77 (14), 4549-4555 (2005).
- Sharp, J. S., Tomer, K. B. Effects of anion proximity in peptide primary sequence on the rate and mechanism of leucine oxidation. Analytical Chemistry. 78 (14), 4885-4893 (2006).
- Huang, W., Ravikumar, K. M., Chance, M. R., Yang, S. Quantitative mapping of protein structure by hydroxyl radical footprinting-mediated structural mass spectrometry: a protection factor analysis. Biophysical Journal. 108 (1), 107-115 (2015).
- Xie, B., Sood, A., Woods, R. J., Sharp, J. S. Quantitative protein topography measurements by high resolution hydroxyl radical protein footprinting enable accurate molecular model selection. Scientific Reports. 7 (1), 4552 (2017).
- Li, Z., et al. High structural resolution hydroxyl radical protein footprinting reveals an extended Robo1-heparin binding interface. Journal of Biological Chemistry. 290 (17), 10729-10740 (2015).
- Li, X., et al. Structural analysis of the glycosylated intact HIV-1 gp120-b12 antibody complex using hydroxyl radical protein footprinting. Biochemistry. 56 (7), 957-970 (2017).
- Li, K. S., Shi, L., Gross, M. L. Mass spectrometry-based fast photochemical oxidation of proteins (FPOP) for higher order structure characterization. Accounts of Chemical Research. 51 (3), 736-744 (2018).
- Li, J., Chen, G. The use of fast photochemical oxidation of proteins coupled with mass spectrometry in protein therapeutics discovery and development. Drug Discovery Today. 24 (3), 829-834 (2019).
- Roush, A. E., Riaz, M., Misra, S. K., Weinberger, S. R., Sharp, J. S. Intrinsic buffer hydroxyl radical dosimetry using Tris(hydroxymethyl)aminomethane. Journal of the American Society for Mass Spectrometry. 31 (2), 169-172 (2020).
- Xie, B., Sharp, J. S. Hydroxyl radical dosimetry for high flux hydroxyl radical protein footprinting applications using a simple optical detection method. Analytical Chemistry. 87 (21), 10719-10723 (2015).
- Niu, B., Zhang, H., Giblin, D., Rempel, D. L., Gross, M. L. Dosimetry determines the initial OH radical concentration in fast photochemical oxidation of proteins (FPOP). Journal of the American Society for Mass Spectrometry. 26 (5), 843-846 (2015).
- Niu, B., et al. Incorporation of a reporter peptide in FPOP compensates for adventitious scavengers and permits time-dependent measurements. Journal of the American Society for Mass Spectrometry. 28 (2), 389-392 (2017).
- Garcia, N. K., Sreedhara, A., Deperalta, G., Wecksler, A. T. Optimizing hydroxyl radical footprinting analysis of biotherapeutics using internal standard dosimetry. Journal of the American Society for Mass Spectrometry. 31 (7), 1563-1571 (2020).
- Sharp, J. S., Misra, S. K., Persoff, J. J., Egan, R. W., Weinberger, S. R. Real time normalization of fast photochemical oxidation of proteins experiments by inline adenine radical dosimetry. Analytical Chemistry. 90 (21), 12625-12630 (2018).
- Zhang, B., Cheng, M., Rempel, D., Gross, M. L. Implementing fast photochemical oxidation of proteins (FPOP) as a footprinting approach to solve diverse problems in structural biology. Methods. 144, 94-103 (2018).
- Konermann, L., Stocks, B. B., Czarny, T. Laminar flow effects during laser-induced oxidative labeling for protein structural studies by mass spectrometry. Analytical Chemistry. 82 (15), 6667-6674 (2010).
- Gau, B. C., Sharp, J. S., Rempel, D. L., Gross, M. L. Fast photochemical oxidation of protein footprints faster than protein unfolding. Analytical Chemistry. 81 (16), 6563-6571 (2009).
- Li, K. S., et al. Hydrogen-Deuterium exchange and hydroxyl radical footprinting for mapping hydrophobic interactions of human bromodomain with a small molecule Inhibitor. Journal of the American Society for Mass Spectrometry. 30 (12), 2795-2804 (2019).
- Espino, J. A., Jones, L. M. Illuminating biological interactions with in vivo protein footprinting. Analytical Chemistry. 91 (10), 6577-6584 (2019).
- Charvatova, O., et al. Quantifying protein interface footprinting by hydroxyl radical oxidation and molecular dynamics simulation: application to galectin-1. Journal of the American Society for Mass Spectrometry. 19 (11), 1692-1705 (2008).
- Gau, B., Garai, K., Frieden, C., Gross, M. L. Mass spectrometry-based protein footprinting characterizes the structures of oligomeric apolipoprotein E2, E3, and E4. Biochemistry. 50 (38), 8117-8126 (2011).
- Gau, B. C., Chen, J., Gross, M. L. Fast photochemical oxidation of proteins for comparing solvent-accessibility changes accompanying protein folding: Data processing and application to barstar. Biochimica et Biophysica Acta. 1834 (6), 1230-1238 (2013).
- Garrison, W. M. Reaction mechanisms in the radiolysis of peptides, polypeptides, and proteins. Chemical Reviews. 87 (2), 381-398 (1987).
- Xu, G., Chance, M. R. Radiolytic modification of sulfur-containing amino acid residues in model peptides: fundamental studies for protein footprinting. Analytical Chemistry. 77 (8), 2437-2449 (2005).
- Xu, G., Chance, M. R. Radiolytic modification of acidic amino acid residues in peptides: probes for examining protein-protein interactions. Analytical Chemistry. 76 (5), 1213-1221 (2004).
- Xu, G., Takamoto, K., Chance, M. R. Radiolytic modification of basic amino acid residues in peptides: probes for examining protein-protein interactions. Analytical Chemistry. 75 (24), 6995-7007 (2003).
- Misra, S. K., Orlando, R., Weinberger, S. R., Sharp, J. S. Compensated hydroxyl radical protein footprinting measures buffer and excipient effects on conformation and aggregation in an adalimumab biosimilar. AAPS Journal. 21 (5), 87 (2019).
- Simmons, D. A., Konermann, L. Characterization of transient protein folding intermediates during myoglobin reconstitution by time-resolved electrospray mass spectrometry with on-line isotopic pulse labeling. Biochemistry. 41 (6), 1906-1914 (2002).
- Vahidi, S., Konermann, L. Probing the time scale of FPOP (fast photochemical oxidation of proteins): radical reactions extend over tens of milliseconds. Journal of the American Society for Mass Spectrometry. 27 (7), 1156-1164 (2016).
- Chance, M. R. Unfolding of apomyoglobin examined by synchrotron footprinting. Biochemical and Biophysical Research Communications. 287 (3), 614-621 (2001).
- Xu, G., Chance, M. R. Hydroxyl radical-mediated modification of proteins as probes for structural proteomics. Chemical Reviews. 107 (8), 3514-3543 (2007).
- Zhang, Y., Rempel, D. L., Zhang, H., Gross, M. L. An improved fast photochemical oxidation of proteins (FPOP) platform for protein therapeutics. Journal of the American Society for Mass Spectrometry. 26 (3), 526-529 (2015).
- Cornwell, O., Radford, S. E., Ashcroft, A. E., Ault, J. R. Comparing hydrogen deuterium exchange and fast photochemical oxidation of proteins: a structural characterisation of wild-type and ΔN6 β(2)-microglobulin. Journal of the American Society for Mass Spectrometry. 29 (2), 2413-2426 (2018).
- Xie, B., Sharp, J. S. Relative Quantification of sites of peptide and protein modification using size exclusion chromatography coupled with electron transfer dissociation. Journal of the American Society for Mass Spectrometry. 27 (8), 1322-1327 (2016).
- Srikanth, R., Wilson, J., Vachet, R. W. Correct identification of oxidized histidine residues using electron-transfer dissociation. Journal of Mass Spectrometry. 44 (5), 755-762 (2009).
- Li, X., Li, Z., Xie, B., Sharp, J. S. Improved identification and relative quantification of sites of peptide and protein oxidation for hydroxyl radical footprinting. Journal of the American Society for Mass Spectrometry. 24 (11), 1767-1776 (2013).
- Li, X., Li, Z., Xie, B., Sharp, J. S. Supercharging by m-NBA Improves ETD-Based Quantification of Hydroxyl Radical Protein Footprinting. Journal of the American Society for Mass Spectrometry. 26 (8), 1424-1427 (2015).
- Khaje, N. A., Sharp, J. S. Rapid quantification of peptide oxidation isomers from complex mixtures. Analytical Chemistry. 92 (5), 3834-3843 (2020).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。