Method Article
RNA荧光 原位 杂交(FISH)可视化 秀丽隐杆线虫 肠道中的微生物定植和感染
摘要
肠道微生物,包括细胞外细菌和细胞内病原体,如奥赛病毒和微孢子虫(真菌),通常与野生 Caenorhabditis 线虫有关。本文介绍了一种方案,用于检测和量化定植和/或感染 秀丽隐杆线 虫的微生物,以及在实验室中控制感染后测量病原体负荷的方案。
摘要
野生Caenorhabditis线虫的肠道中栖息着多种微生物,包括肠道微生物组细菌和病原体,如微孢子虫和病毒。由于秀丽隐杆线虫和哺乳动物肠道细胞之间的相似性,以及秀丽隐杆线虫系统的力量,该宿主已成为研究体内宿主肠道-微生物相互作用的模型系统。虽然可以用明场显微镜观察这些相互作用的某些方面,但如果没有更精确的工具,很难准确分类微生物并表征定植或感染的程度。RNA荧光原位杂交(FISH)可用作识别和可视化野生线虫中微生物的工具,或在实验室中对感染微生物的线虫感染进行实验表征和定量。FISH探针标记高丰度的小亚基核糖体RNA,为细菌和微孢子虫细胞产生明亮的信号。设计用于靶向许多物种共有的核糖体RNA保守区域的探针可以检测广泛的微生物,而靶向核糖体RNA的不同区域可用于较窄的检测。同样,可以设计探针来标记病毒RNA。提出了用多聚甲醛(PFA)或丙酮固定对RNA FISH染色的方案。PFA 固定是与细菌、微孢子虫和病毒相关的线虫的理想选择,而丙酮固定对于小孢子虫孢子的可视化是必要的。首先将动物洗涤并固定在多聚甲醛或丙酮中。固定后,将FISH探针与样品一起孵育,以使探针与所需靶标杂交。再次清洗动物,然后在显微镜载玻片上或使用自动化方法进行检查。总体而言,该FISH协议能够检测,鉴定和定量居住在秀丽隐杆线虫肠道中的微生物,包括没有遗传工具可用的微生物。
引言
秀丽隐杆线虫已成为研究肠上皮细胞中先天免疫和宿主-微生物相互作用的强大模型系统1,2。由于具有透明的身体和只有20个肠道细胞,秀丽隐杆线虫代表了一种方便的系统,用于在完整生物体的背景下监测微生物肠道定植和感染的过程。线虫肠细胞与哺乳动物肠上皮细胞具有多种形态和功能相似性,使其成为可处理的体内模型,用于解剖控制微生物组定植和病原体感染的过程3,4,5,6。
野生秀丽隐杆线虫以各种定植和感染肠道的微生物为食,对这些线虫的采样导致发现了病毒、真核生物(真菌、卵菌)和与该宿主自然相关的细菌7,8,9,10。发现奥赛病毒感染肠道,是目前唯一已知的秀丽隐杆线虫9天然病毒。微孢子虫是真菌相关的专性细胞内病原体,是野生Caenorhabditis中最常见的感染,已发现几种感染秀丽隐杆线虫和相关线虫8,11的物种。许多细菌通常栖息在野生秀丽隐杆线虫的肠腔中,并且已经建立了几种物种作为秀丽隐杆线虫微生物组(CeMbio)的自然模型6,12,13,14。发现和表征自然定植和/或感染秀丽隐杆线虫的微生物对于理解控制这些宿主-微生物相互作用的遗传机制以及可视化仅在完整宿主动物环境中发生的新型微生物过程至关重要。
取样后,通过微分干涉对比(DIC)显微镜筛选野生线虫,以寻找表明感染或定植的表型。例如,肠细胞的刻板颗粒外观的变化可能与细胞内寄生虫感染的存在有关8。具体而言,肠道颗粒的丢失和胞质粘度的降低是病毒感染的迹象,而肠道颗粒重组为"凹槽"可能表明感染了杀线虫属中的小孢子虫8,9。由于野生秀丽隐杆线虫样品中存在多种微生物,因此很难通过DIC显微镜区分微生物。由于许多微生物的体积小,有关宿主内微生物空间分布的信息也可能难以检测15。此外,在体外培养任何感兴趣的特定微生物并不总是可能的,导致检测和/或定量困难。
RNA荧光原位杂交(FISH)提供了一种通过利用荧光探针与固定细胞中小核糖体亚基(SSU)的RNA结合来荧光标记微生物的方法。如果形态特征分析表明存在特定类别的微生物,则可以使用针对特定或广泛类别的此类微生物的FISH探针。例如,EUB338被认为是细菌SSU的通用探针,通常用于检测各种细菌16。此处描述的方案使用单链DNA探针,这些探针用荧光团末端标记,专门设计用于与目标微生物的靶SSU互补,尽管以前设计的探针可用16。靶向微生物SSU的主要优点是该RNA的丰度相对较大,其通常占细胞中所有RNA的80%-90%,导致染色具有非常高的信噪比17。探针也可以设计成靶向RNA以检测病毒,如奥赛病毒9,18,如果病毒正在积极复制,这些病毒通常以非常高的拷贝存在于受感染的细胞中。
根据已知探针的结果,可能需要获得进一步的序列信息,以设计更具体的探针以进行 原位物种确认。一种常见的方法是使用通用引物对抗SSU的保守区域(细菌为16S,真核生物为18S)以扩增(通过 PCR)更不同的区域8。利用这些序列信息,可以设计出具有更多物种特异性的探针。然后,这些FISH探针能够以独立于培养物的方式鉴定微生物8。此外,RNA FISH可以深入了解独特的形态定植和感染特征,包括细丝或组织定位模式19,20。可以同时使用不同颜色的FISH探针,这允许在视觉上区分野生线虫样品中的微生物,以及观察宿主内的微生物 - 微生物动力学15,20。此外,RNA FISH 染色可应用于宿主-病原体相互作用研究,其中已知物种的感染和定植可以很容易地手动或通过自动化方法量化,以提供有关病原体负荷的见解,例如,在比较对感染抵抗力增加或降低的秀 丽隐杆线虫 突变体时21。
研究方案
注意:线虫可以用多聚甲醛溶液(PFA)或丙酮固定。PFA允许比丙酮更好地可视化形态,并且可以保留被丙酮破坏的转基因绿色荧光蛋白(GFP)的信号。然而,丙酮固定对于透化微孢子虫孢子是必要的,以便能够标记这个生命阶段。此外,丙酮比PFA更方便,因为它的毒性更小,样品可以在-20°C冰箱中的丙酮中储存数天,而无需去除固定剂。以下是两种独立的方案,使用PFA溶液或丙酮作为固定剂。有关协议步骤的可视化,请参见 图 1。
1. 用PFA固定法染色鱼
- 制备具有相关微生物的线虫
- 在接种有适当食物来源的标准线虫生长培养基(NGM)平板上用所需的感兴趣微生物生长线虫。将线虫在20°C孵育,直到达到所需的生命阶段。
- 将 2 mL M9 最小盐培养基(42 mM Na 2 HPO 4、22 mM KH2PO 4、8.6 mM NaCl、19 mM NH4 Cl)+ 0.1% 吐温 20 加入含有感染或定植的 NGM 平板。
注意:添加洗涤剂是为了防止线虫粘附在移液器和微量离心管上,并帮助沉淀 L1-L2 阶段线虫。0.1% Triton-X 可用于代替吐温 20。 - 使用玻璃巴斯德移液管和灯泡从平板上移取线虫,并将它们转移到标记的 1.5 mL 微量离心管中。
注意:首选玻璃移液器,因为线虫会粘附在塑料移液器上,但添加洗涤剂(吐温 20 或 Triton-X)可以最大限度地减少此问题。 - 使用微量离心机,将含有定植或感染线虫的样品以 2,000 x g 旋转 L1 动物 60 秒,或 500 x g 旋转 L4 或成年动物 60 秒。所有后续离心步骤将以选定的速度进行。
- 使用移液管从微量离心管中取出上清液。通过小心地将上清液去除至沉淀上方 100 μL 来避免干扰线虫沉淀。这可以使用标记的 1.5 mL 微量离心管进行估算。
- 清洗线虫以消除外部污染
- 向微量离心管中加入 1 mL 1x PBS(137 mM NaCl、2.7 mM KCl、10 mM Na 2 HPO 4、1.8 mM KH2PO4)+ 0.1% 吐温 20 (PBS-T)。
- 在微量离心机中以适当的速度旋转样品(参见步骤1.1.4)。使用移液器除去除 100 μL 以外的所有上清液。这可以使用标记的 1.5 mL 微量离心管进行估算。
- 重复上述两个步骤2-3x。
注意:总共三次洗涤通常就足够了;但是,可以进行额外的清洗以去除任何多余的外部污染。
- 用PFA修复线虫
- 在通风橱中,将 33 μL 16% PFA 加入含有 100 μL 上清液的微量离心管中,高于从步骤 1.2.3 获得的线虫沉淀,最终浓度为 4% PFA。
注意:PFA是一种致癌物质。接触PFA可能导致皮肤过敏和刺激以及眼睛损伤。PFA释放有毒烟雾,可能导致呼吸道刺激或致敏。使用 PFA 时,请在通风橱中使用适当的个人防护设备工作,并在使用前参考相应的安全数据表。 - 将含有定植或感染感兴趣的微生物的线虫的样品在室温下孵育30-45分钟。孵育后,将样品储存在4°C的70%乙醇中,直到方案准备好继续。
注意:较短的孵育期更适合维持转基因菌株中的GFP信号,因为PFA会随着时间的推移降解GFP。更长的孵育时间使固定剂能够更好地渗透到样品中。最好根据样品的经验确定孵育时间。
- 在通风橱中,将 33 μL 16% PFA 加入含有 100 μL 上清液的微量离心管中,高于从步骤 1.2.3 获得的线虫沉淀,最终浓度为 4% PFA。
- 移除 PFA 溶液
- 在微量离心机中以适当的速度旋转样品(参见步骤1.1.4)。用移液管在不干扰沉淀的情况下尽可能多地去除上清液。
注意:上清液含有PFA,这是有毒的。丢弃上清液,至少前两次作为有毒废物在通风橱中洗涤。 - 向微量离心管中的样品中加入 0.5 mL PBS-T。
- 使用 PBS-T 遵循并重复步骤 1.4.1 和 1.4.2 2-3x。
注意:执行更多洗涤将有助于减少背景信号。建议总共至少清洗四次。 - 最后一次洗涤后,旋转样品并除去上清液,使沉淀不受干扰。
- 在微量离心机中以适当的速度旋转样品(参见步骤1.1.4)。用移液管在不干扰沉淀的情况下尽可能多地去除上清液。
- 准备杂交缓冲液(HB)并洗涤线虫
- 每个样品制备 1 mL HB(900 mM NaCl,20 mM Tris pH 7.5,0.01% SDS)。
注意:HB应在每次使用前新鲜制备,以避免沉淀。但是,可以提前制备一般缓冲液(900 mM NaCl,20 mM Tris pH 7.5)并在室温下储存,直到需要HB。使用前,每个样品制备1 mL的通用缓冲液,并加入SDS至终浓度为0.01%。 - 向含有线虫沉淀的微量离心管中加入 800 μL HB。在微量离心机中沉淀样品(参见步骤1.1.4)。在不干扰沉淀的情况下除去上清液。
- 每个样品制备 1 mL HB(900 mM NaCl,20 mM Tris pH 7.5,0.01% SDS)。
- 将 FISH 探针杂交到所需的靶序列
- 将每个制备的HB样品与所需的FISH探针混合100 μL,最终浓度为5-10 ng/μL探针。
注意:FISH 探针是 15-23 -mer 寡核苷酸,对目标微生物的 SSU 有反义,并用附着在 5' 或 3' 端的彩色荧光团标记(此处使用的探针参见 表 1 )。通常,原液 FISH 探针的储存量为 1 mg/mL。 - 向每个样品中加入 100 μL 含有 FISH 探针的 HB。通过轻轻轻弹或倒置试管进行混合。
注意:可以同时添加不同颜色的FISH探针,以可视化同一样品中的多个荧光信号(见 图1B)。 - 将样品在46-54°C的干浴中孵育过夜(6-24小时)或在46-54°C的热混合器中以1,200rpm孵育。
注意:通常在46-48°C下孵育用于杂交。但是,可能需要根据FISH探头的熔化温度调整该温度。一般来说,杂交温度比熔解温度低4°C。
- 将每个制备的HB样品与所需的FISH探针混合100 μL,最终浓度为5-10 ng/μL探针。
- 取下 FISH 探针并清洗线虫
- 每个样品制备 3 mL 洗涤缓冲液 (WB)(900 mM NaCl、20 mM Tris pH 7.5、5 mM EDTA、0.01% SDS)。
注意:每次使用前应新鲜准备WB,以避免沉淀。但是,可以提前制备一般缓冲液(900 mM NaCl,20 mM Tris pH 7.5)并在室温下储存,直到需要洗涤缓冲液。使用前,每个样品制备3 mL的通用缓冲液,并加入EDTA至终浓度为5 mM,SDS至终浓度为0.01%。 - 以适当的速度离心样品(见步骤1.1.4)。使用移液管取出HB,同时小心使线虫沉淀不受干扰。
- 向每个样品中加入 1 mL 制备的 WB。
- 以适当的速度离心样品(见步骤1.1.4)。使用移液器取出WB,同时小心不要打扰沉淀。
- 向每个样品中加入 1 mL 制备的 WB。
- 将样品在48-56°C的干浴中孵育1小时(或热混合器在48-56°C以1200rpm)。如果在干浴中孵育,每15-20分钟轻轻倒置试管。
注意:通常使用48°C作为标准洗涤温度;但是,如果背景信号较高,则可能需要调整此温度。洗涤温度通常比杂交温度高2°C。WB中的孵育时间可以缩短至30分钟以进一步降低背景,之后,应重复步骤1.7.4和1.7.5。随后在48°C下进行一次(对于细菌)或两个(对于微孢子虫孢子质)30分钟的孵育期。 - 以适当的速度离心样品(见步骤1.1.4)。使用移液器取出洗涤缓冲液,同时小心使沉淀不受干扰。
- 向每个样品中加入 100-500 μL PBS-T。此时,样品可以在4°C的PBS-T中储存长达一周,直到方案准备好继续。
- 每个样品制备 3 mL 洗涤缓冲液 (WB)(900 mM NaCl、20 mM Tris pH 7.5、5 mM EDTA、0.01% SDS)。
- 安装线虫
- 以适当的速度离心样品(见步骤1.1.4)。在不干扰线虫沉淀的情况下尽可能多地去除PBS-T。
- (可选)向样品中加入 20 μL 带有 DAPI(材料表)的抗淬灭封片剂。
- 用 200 μL 移液器吸头加载 20 μL 移液器,然后用剪刀剪掉移液器的尖端,以便移液较大的线虫。
- 使用切好的移液器吸头,将 5-10 μL 沉淀转移到显微镜载玻片上。用 22 x 22 盖玻片盖住。要储存载玻片,请用指甲油密封盖玻片的边缘,并将其保存在4°C的暗盒中,直到准备进一步使用。
2. 用丙酮固定法染色
- 按照步骤1.1中所述制备具有相关微生物的线虫。
- 清洗线虫以消除外部污染,如步骤1.2中所述。
- 用丙酮固定线虫
- 在不干扰沉淀的情况下去除上清液,并向样品中加入 1 mL 实验室级丙酮。
注意:丙酮是一种高度易燃的液体和蒸气。虽然它可以作为指甲油去除剂在柜台购买,但重要的是要记住丙酮会引起严重的眼睛刺激,并可能导致嗜睡或头晕。 - 将含有定植或感染感兴趣微生物的线虫的样品在室温下孵育15分钟。孵育后,样品可以在-20°C的丙酮中储存长达2周,直到方案准备好继续。
注意:如果需要保持荧光,请勿将此协议与表达GFP(或其等位基因突变形式)的转基因 秀丽隐杆线虫 菌株一起使用,因为丙酮会破坏该信号。
- 在不干扰沉淀的情况下去除上清液,并向样品中加入 1 mL 实验室级丙酮。
- 取出丙酮
- 在微量离心机中以适当的速度旋转样品(参见步骤1.1.4)。用移液管在不干扰沉淀的情况下除去上清液。
注意:上清液含有丙酮。丢弃上清液,至少前两次作为有毒废物在通风橱中洗涤。 - 向微量离心管中的样品中加入 0.5 mL PBS-T。
- 使用 PBS-T 遵循并重复步骤 2.4.1 和 2.4.2 2-4x。
注意:执行更多洗涤将有助于减少背景信号。建议总共进行四次洗涤。 - 最后一次洗涤后,旋转样品并除去上清液,确保沉淀不受干扰。
- 在微量离心机中以适当的速度旋转样品(参见步骤1.1.4)。用移液管在不干扰沉淀的情况下除去上清液。
- 制备杂交缓冲液(HB)并按照步骤1.5中所述洗涤线虫。
- 如步骤1.6中所述,将FISH探针杂交到所需的靶序列。
- 取下 FISH 探针并清洗线虫
- 每个样品制备 1.1 mL 洗涤缓冲液 (WB)(900 mM NaCl、20 mM Tris pH 7.5、5 mM EDTA、0.01% SDS)。
注意:每次使用前应新鲜准备WB,以避免沉淀。但是,可以提前制备一般缓冲液(900 mM NaCl,20 mM Tris pH 7.5)并在室温下储存,直到需要洗涤缓冲液。使用前,每个样品制备1.1 mL的通用缓冲液,并加入EDTA至终浓度为5 mM,SDS至终浓度为0.01%。 - 以适当的速度离心样品(见1.1.4)。使用移液管取出HB,同时小心使线虫沉淀不受干扰。
- 向每个样品中加入 100 μL 制备的 WB。
- 以适当的速度离心样品(见1.1.4)。使用移液器取出WB,同时小心不要打扰沉淀。
- 向每个样品中加入 1 mL 制备的 WB。
- 将样品在48-56°C的干浴中孵育1小时(或热混合器在48-56°C以1,200rpm)。如果在干浴中孵育,每15-20分钟轻轻倒置试管。
注意:通常使用48°C作为标准洗涤温度;但是,如果背景信号较高,则可能需要调整此温度。洗涤温度通常比杂交温度高2°C。 - 以适当的速度离心样品(见步骤1.1.4)。使用移液器取出洗涤缓冲液,同时小心使沉淀不受干扰。
- 向每个样品中加入 100-500 μL PBS-T。此时,样品可以在4°C的PBS-T中储存长达一周,直到方案准备好继续。
- 每个样品制备 1.1 mL 洗涤缓冲液 (WB)(900 mM NaCl、20 mM Tris pH 7.5、5 mM EDTA、0.01% SDS)。
- 按照步骤1.8中所述安装线虫。
结果
为了分析微生物组细菌,在野生分离的动物身上使用了针对细菌16S的特异性和通用的FISH探针。野生 热带Caenorhabditis 菌株(JU1848)是从法属圭亚那一条小河附近的Nouragues森林中从腐烂的棕榈树果实中取样的22。在微分干涉对比(DIC)显微镜下,发现这种线虫菌株被一种细菌定植,该细菌似乎定向地粘附在肠上皮上(图2A)。然后选择性地清洁JU1848以消除其他微生物污染物并富集所需的粘附细菌23。使用通用PCR方法,该细菌被鉴定为α变形杆菌类的新物种。然后将用Cal Fluor Red 610标记的FISH探针专门设计用于该细菌的16S rRNA序列,以允许对 C. tropicalis 内的定植进行荧光可视化(图2B)。能够结合多种细菌的通用16S rRNA FISH探针(EUB338)用6-羧基荧光素(FAM)标记,并被添加到该样品中。绿色和红色荧光信号完全重叠,表明大多数定植在肠道中的细菌是粘附的α变形杆菌细菌。这些动物在染色前固定在PFA中。
为了分析实验室中已知身份的细胞内病原体的实验感染,在具有野生型背景的秀丽隐杆线虫上使用Orsay病毒和微孢子虫特异性FISH探针。奥赛病毒是来自诺达病毒科的正链RNA病毒,也是秀丽隐杆线虫中发现的唯一天然病毒病原体。奥赛病毒的二分RNA基因组由RNA1和RNA2片段组成,并且已经开发出针对这两个片段的FISH探针(图3A,B)9,18。在肠道中,病毒RNA由RIG-I同源物DRH-1 24感知,这是激活称为细胞内病原体反应(IPR)的转录防御程序所必需的25,26,27。抗病毒IPR基因的转录至少部分由ZIP-1转录因子21控制。在这里,ZIP-1::GFP的表达位于细胞质中显示奥赛病毒FISH染色阳性的细胞的肠核中(图3A)21。显示用Orsay特异性FISH染色的多只动物以指示该信号的强度,以便于定量(图3B)。将图3A,B所示的动物固定在PFA中。
名为巴黎线虫的微孢子虫寄生虫,意思是来自巴黎的线虫杀手,是一种专性肠道细胞内病原体。已经使用了几种标记巴黎猪笼草18S rRNA的FISH探针,包括荧光标记的MicroA和MicroB探针。显示用MicroB FISH染色的多只动物指示该信号的强度,以便于定量(图3C)。此外,秀丽隐杆线虫被其他密切相关的微孢子虫感染。N2与巴黎猪笼草和相关奥苏贝利奈瑟菌的混合感染可以通过设计物种特异性FISH探针来区分,这些探针相互竞争以结合到18S rRNA上的不同区域(图3D)28。在本例中,巴黎猪笼草 FISH 探针与巴黎猪笼草 18S rRNA 具有完美的碱基配对,但与 N. ausubeli 18S rRNA 错配 7 bp。相反,对于N. ausubeli探测器也是如此。因此,每个物种特异性FISH探针在与同源物种18S的结合方面将优于非同源物种。此外,使用DAPI对细胞核进行染色可以在整个动物的背景下更好地定位感染,特别是对于具有大而易于识别的细胞核的肠道。图3C,D包含固定在PFA中的动物。后来感染巴黎猪笼草导致梅龙特发育成孢子。为了可视化巴黎猪笼草孢子,必须将动物固定在丙酮中,因为它比PFA更好地穿透孢子壁(图3E,F)8。所得的FISH染色显示了小而大的杆状结构,可能与巴黎猪笼草孢子相对应,后者用红色的巴黎猪笼草特异性探针染色。
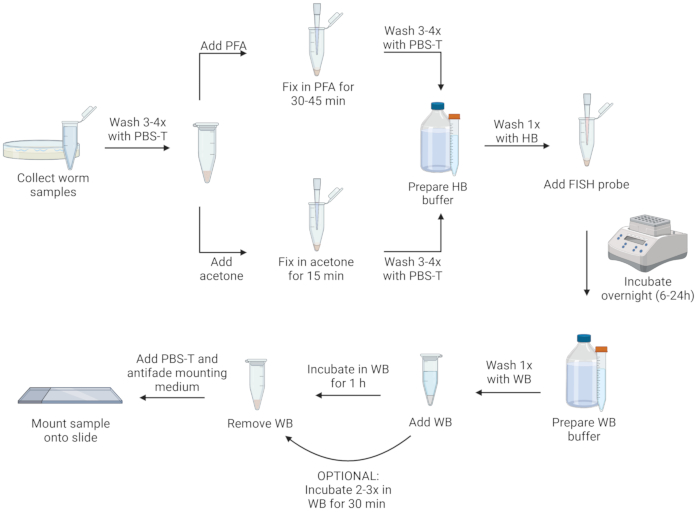
图 1:FISH 协议的可视化表示。 用 Biorender.com 创建。 请点击此处查看此图的大图。
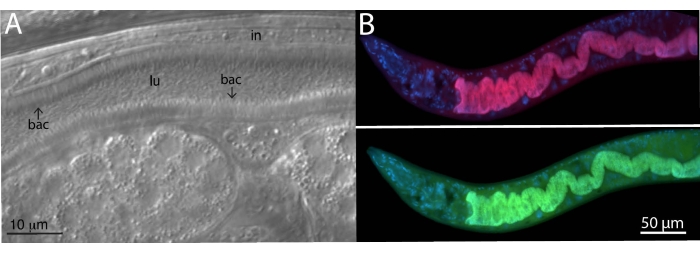
图 2:在肠道中用粘附细菌定植的野生 热带 C. tropicalis JU1848 菌株的 FISH 染色。 (A)Nomarski图像描绘了数千个细杆菌细菌(bac)定向结合到JU1848的肠道(in),在腔内(lu)内产生头发状表型。该图面板改编自摩根等人 (2021)23。(B)JU1848的FISH染色,固定在PFA中,使用红色标记探针(b002_16S_A-CF610)设计用于靶向粘附细菌的16S rRNA序列(顶部)和绿色标记的通用FISH探针(EUB338-FAM)旨在靶向16S细菌(底部)。宿主细胞核的DAPI染色以蓝色显示。有关探针序列,请参见 表1 。 请点击此处查看此图的大图。
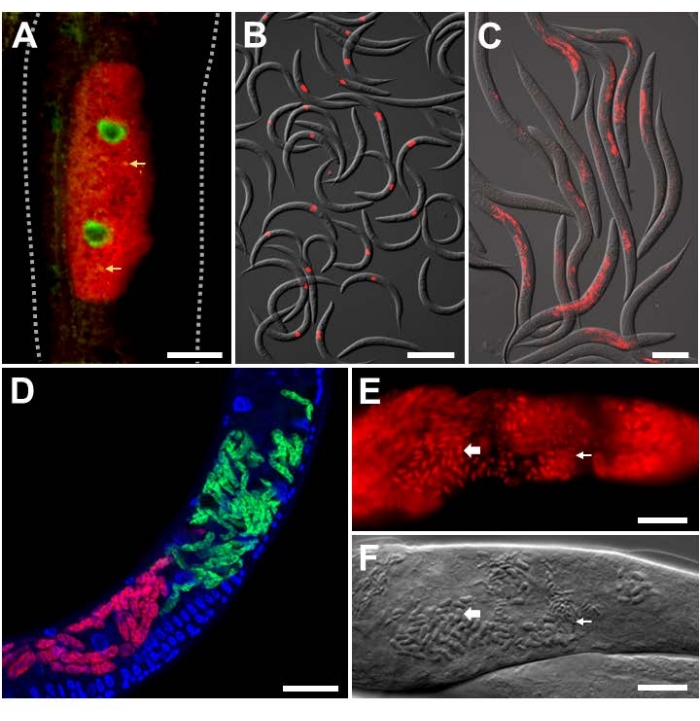
图3:感染细胞内病原体的秀丽隐杆线虫的FISH染色。 (A,B)表达ZIP-1::GFP并感染Orsay病毒的秀丽隐杆线虫的FISH染色,在染色前用PFA固定以保留GFP信号。奥赛 1 红探针和奥赛 2 红探针用于病原体染色。(A)合成图像由合并的红色和绿色荧光通道组成。核ZIP-1::GFP表达在奥赛病毒感染时诱导,并以绿色显示。肠道颗粒的自发荧光以黄色显示,并用黄色箭头表示。虚线勾勒出线虫体。比例尺 = 25 μm。 (B)合成图像由合并的红色荧光和DIC通道组成。比例尺 = 200 μm。 (C,D) 固定在 PFA 中感染微孢子虫的野生型秀丽隐杆线虫的 FISH 染色。(C)感染巴黎猪笼草并固定在PFA中的野生型秀丽隐杆线虫的FISH染色。微生物B-CF610探针用于病原体染色。合成图像由合并的红色荧光和DIC通道组成。比例尺 = 100 μm。 (D) 肠道中与巴黎猪笼草和奥苏贝利猪笼草共同感染的野生型秀丽隐杆线虫的 FISH 染色。使用一对特定的FISH探针对两种病原体进行共染色,这些探针竞争与18S rRNA的同一区域结合。巴黎猪笼草使用MicroF-CF610(红色)染色,Ausubeli猪笼草使用MicroSp1A-FAM(绿色)染色。宿主细胞核的DAPI染色呈蓝色。比例尺 = 25 μm。 (E) 用丙酮固定的野生型秀丽隐杆线虫染色 巴黎猪笼草孢子。MicroA-CF610(红色)用于染色(红色)。比例尺 = 15 μm。 (F) Nomarski 图像描绘了 (E) 中看到的巴黎猪笼草孢子。比例尺 = 15 μm。在(E)和(F)中,小和大的杆状结构分别用小箭头和大箭头标记,对应于巴黎猪笼草孢子。有关探针序列,请参见表1。(A)中显示的图像改编自Lažetić, V. et al. (2022)21。(B)和(C)中显示的图像改编自Reddy,K.C.等人(2019)26。(E)和(F)中显示的图像改编自Troemel,E.R.等人(2008)8。请点击此处查看此图的大图。
| 探测器名称 | 探针特异性 | 探针荧光基团 | 探针序列 |
| EUB338-FAM | 细菌 16S(通用) | 5' 6-荧光素 | GCTGCCTCCCGTAGGAGT |
| b002_16S_A-CF610 | α变形杆菌16S | 钙氟红 610 (CF610) | TGTACCGACCCTTAACGTTC |
| 奥赛1 红色 | 奥赛病毒RNA1 | 钙氟红 610 (CF610) | GACATATGTGATGCCGAGAC |
| 奥赛2 红色 | 奥赛病毒RNA2 | 钙氟红 610 (CF610) | GTAGTGTCATTGTAGGCAGC |
| 微A-CF610 | 巴黎线虫 18S | 钙氟红 610 (CF610) | CTCTGTCCATCCTCGGCAA |
| 微B-CF610 | 巴黎线虫 18S | 钙氟红 610 (CF610) | CTCTCGGCACTCCTTCCTG |
| 微F-CF610 | 巴黎线虫 18S | 钙氟红 610 (CF610) | AGACAAATCAGTCCACGAATT |
| MicroSp1A-FAM | 奥苏贝利线虫 18S | 5' 6-荧光素 | CAGGTCACCCCACGTGCT |
表1:FISH探针序列列表。 所有FISH探针均在商业上购买,荧光团连接到5'端(通过 定制寡核苷酸合成;参见 材料表),寡核苷酸通过反相HPLC纯化。
讨论
野生秀丽隐杆线虫与各种微生物天然相关。研究人员可以使用RNA FISH来检测和鉴定这些微生物,并深入了解它们在整个动物环境中的定位。通过该方法可以鉴定具有所需或有趣表型的微生物,然后分离以进行进一步表征和测序。来自野生秀丽隐杆线虫的众多细菌分离株的丰度也可以通过RNA FISH29进行定量。通过使用此处描述的协议,还可以观察宿主内的已知微生物并了解有关其相互作用的更多信息。重要的是,奥赛病毒和微孢子虫是专性寄生虫,不能独立于宿主进行培养,因此FISH是标准的可视化工具。定植或感染也可以通过RNA FISH进行量化,使用在接种有所需目标可培养细菌的平板上生长的线虫。除了对秀丽隐杆线虫肠道中的微生物进行染色外,该协议还可用于其他线虫菌株,如热带线虫或 Oscheius tipulae19,23。
FISH 方案的主要优点是它提供了一种简单、快速和稳健的方法来染色与秀丽隐杆线虫相关的微生物。从FISH染色产生的图像具有高信噪比,这是通过使用靶向样品中SSU丰富RNA的FISH探针来实现的。由于rRNA的含量通常为rDNA的30倍或更高,因此用靶向rRNA的探针染色的FISH的大部分信号是由于rRNA而不是rDNA30引起的。此外,RNA FISH可以在整个动物的背景下看到感染或定植。通过将宿主细胞核与DAPI共染色和/或使用荧光标记的秀丽隐杆线虫菌株来更好地突出样品中感染或定植的定位,可以促进这种可视化。例如,使用一组在不同组织中表达GFP的秀丽隐杆线虫菌株来确定小孢子虫特异性FISH来确定线虫的组织嗜性20。此外,该协议适用于允许研究人员确定适合其特定需求的理想条件(例如,调整固定期,增加杂交温度)的变化。
FISH 协议中的一个关键步骤是固定样品。添加固定剂后的孵育期是必要的,以便试剂有时间透化样品。对于含有转基因荧光蛋白的样品,较长的孵育时间并不理想,因为蛋白质会随着时间的推移被PFA降解。对于含有GFP的样品,必须确定最佳固定时间以允许透化,同时仍保持GFP信号。
FISH 可用于染色秀丽隐杆线虫中的细菌、病毒或微孢子虫。然而,用于FISH的最佳固定剂类型取决于样品和下游要求。该协议将PFA溶液作为染色细菌和病毒的主要固定剂。然而,PFA不足以观察微孢子虫孢子,因为它不能穿透孢子壁。对于孢子的可视化,应改用丙酮。虽然,PFA固定对于微孢子虫其他生命阶段的FISH标记是有效的,包括孢子质,Meronts和sporonts。丙酮固定和PFA固定之间的其他主要差异;丙酮更方便,因为添加后样品可以快速储存在冰箱中,无需清洗。然而,丙酮会迅速杀死转基因宿主中任何现有的GFP。如果保留宿主中的某些生理结构很重要,则PFA是首选的固定剂,因为丙酮固定动物似乎更降解,使得某些组织的鉴定更加困难。由于样品是固定的,因此该FISH协议不允许对体内宿主-微生物相互作用进行实时成像。然而,脉搏追逐感染时间过程,然后在不同时间点对样品进行FISH染色,可以让人们看到微生物感染的一些动态19,20,31。
整个方案中的另一个关键步骤是在杂交前后彻底清洗样品。在杂交之前,当将蠕虫收集到微量离心管中时,多余的细菌或来自NGM板的其他微生物可以随蠕虫样品一起携带。标配三次PBS-T清洗;然而,可能需要更多的洗涤才能充分消除外部微生物,特别是在使用严重污染、野生分离的 秀丽隐杆线虫时。在FISH之后查看安装的样品时,可能会有一些残留的FISH探针在样品的背景中产生大量信号。洗涤温度和洗涤次数对于去除多余的和非特异性结合的探头很重要。为了减少背景荧光,可以每30分钟用1mL WB进行两次或三次洗涤,而不是用1 mL WB洗涤一次一小时。不同的 FISH 探头可能需要不同的洗涤温度。通常,洗涤温度比杂交温度高2°C,但如果背景荧光过多(高噪声),则可以增加洗涤温度。
FISH 方案使用旨在靶向物种特异性微生物 RNA 的荧光探针,但 FISH 探针可以设计用于其他高拷贝转录本。其他FISH探针可能具有不同的熔化温度,因此孵育步骤可能需要在高于或低于描述的温度下进行。FISH 染色可以识别宿主内微生物定植或感染的空间分布,从而可以表征宿主-微生物和微生物-微生物相互作用。一个限制是只能同时使用几种常规荧光团,这减少了可以通过FISH 同时检测 的不同微生物的数量。这限制了其在 秀丽隐杆线虫中复杂微生物组研究的使用。然而,多色rRNA靶向FISH利用用非规范荧光团标记的探针,可以增加不同微生物组标记的数量15。另一个限制是很难区分具有高度相似的SSU序列的密切相关的物种,尤其是细菌。然而,微孢子虫物种之间的极端序列差异有助于通过该方案促进它们的分化(图3)32,33。
总体而言,该FISH协议描述了一种检测 秀丽隐杆线虫内微生物的技术。它允许研究人员使用透明且可遗传处理的模型系统来检测和量化完整动物背景下的定植和感染,以及识别宿主内独特的微生物行为或形态。该手稿的预印本版本在审查34 期间发布。
披露声明
作者没有利益冲突。
致谢
感谢Marie-Anne Félix博士为我们提供野生线虫菌株。这项工作得到了NSF在CAREER资助2143718和加州州立大学的COVER新研究者奖RJL,NIH在R01 AG052622和R01 GM114139下对ERT的支持,以及美国心脏协会奖学金到VL。
材料
| Name | Company | Catalog Number | Comments |
| 10% SDS | Invitrogen | AM9822 | |
| Acetone | Fisher Scientific | A-11-1 | |
| Antifade mounting serum with DAPI (Vectashield) | Vectalab | NC9524612 | |
| EDTA | Fisher Scientific | S311-500 | |
| FISH probes (see Table 1) | LGC Biosearch Technologies | FISH probes were commercially purchased via custom oligonucleotide synthesis | |
| KCl | Fisher Scientific | P217 | |
| KH2PO4 | Fisher Scientific | P-286 | |
| Na2HPO4 | Fisher Scientific | S375-500 | |
| NaCl | Fisher Scientific | S-671 | |
| NH4Cl | Fisher Scientific | A-661 | |
| Paraformaldehyde | Electron Microscopy Science | 50-980-487 | CAUTION: PFA is a carcinogen. Handle appropriately |
| Thermal mixer | Eppendorf | 5384000020 | |
| Tris base | Fisher Scientific | BP152 | |
| Triton X-100 | Fisher Scientific | BP-151 | |
| Tween-20 | Fisher Scientific | BP337-500 |
参考文献
- Pukkila-Worley, R., Ausubel, F. M. Immune defense mechanisms in the Caenorhabditis elegans intestinal epithelium. Current Opinion in Immunology. 24 (1), 3-9 (2012).
- Balla, K. M., Troemel, E. R. Caenorhabditis elegans as a model for intracellular pathogen infection. Cellular Microbiology. 15 (8), 1313-1322 (2013).
- Dimov, I., Maduro, M. F. The C. elegans intestine: organogenesis, digestion, and physiology. Cell and Tissue Research. 377 (3), 383-396 (2019).
- Bossinger, O., Fukushige, T., Claeys, M., Borgonie, G., McGhee, J. D. The apical disposition of the Caenorhabditis elegans intestinal terminal web is maintained by LET-413. Developmental Biology. 268 (2), 448-456 (2004).
- Szumowski, S. C., Botts, M. R., Popovich, J. J., Smelkinson, M. G., Troemel, E. R. The small GTPase RAB-11 directs polarized exocytosis of the intracellular pathogen N. parisii for fecal-oral transmission from C. elegans. Proceedings of the National Academy of Sciences. 111 (22), 8215-8220 (2014).
- Samuel, B. S., Rowedder, H., Braendle, C., Félix, M. A., Ruvkun, G. Caenorhabditis elegans responses to bacteria from its natural habitats. Proceedings of the National Academy of Sciences. 113 (27), 3941-3949 (2016).
- Zhang, F., et al. Caenorhabditis elegans as a model for microbiome research. Frontiers in Microbiology. 8, 485 (2017).
- Troemel, E. R., Félix, M. -. A., Whiteman, N. K., Barrière, A., Ausubel, F. M. Microsporidia are natural intracellular parasites of the nematode Caenorhabditis elegans. PLoS Biology. 6 (12), 2736-2752 (2008).
- Felix, M. A., et al. Natural and experimental infection of Caenorhabditis nematodes by novel viruses related to nodaviruses. PLoS Biology. 9 (1), 1000586 (2011).
- Osman, G. A., et al. Natural infection of C. elegans by an oomycete reveals a new pathogen-specific immune response. Current Biology. 28 (4), 640-648 (2018).
- Zhang, G. A large collection of novel nematode-infecting microsporidia and their diverse interactions with Caenorhabditis elegans and other related nematodes. PLOS Pathogens. 12 (12), 1006093 (2016).
- Clark, L. C., Hodgkin, J. Commensals, probiotics and pathogens in the Caenorhabditis elegans model. Cell Microbiology. 16 (1), 27-38 (2014).
- Dirksen, P., et al. CeMbio - The Caenorhabditis elegans microbiome resource. G3. Genes, Genomes, Genetics. 10 (9), 3025-3039 (2020).
- Berg, M., et al. Assembly of the Caenorhabditis elegans gut microbiota from diverse soil microbial environments. The ISME Journal. 10 (8), 1998-2009 (2016).
- Michael, L., Markus, S., Petra, P., Holger, D. A multicolor fluorescence in situ hybridization approach using an extended set of fluorophores to visualize microorganisms. Frontiers in Microbiology. 10, 1383 (2019).
- Fuchs, B. M., et al. Flow cytometric analysis of the in situ accessibility of Escherichia coli 16S rRNA for fluorescently labeled oligonucleotide probes. Applied and Environmental Microbiology. 64 (12), 4973-4982 (1998).
- O'Neil, D., Glowatz, H., Schlumpberger, M. Ribosomal RNA depletion for efficient use of RNA-seq capacity. Current Protocols in Molecular Biology. 103 (1), 4-19 (2013).
- Franz, C. J., et al. Santeuil and Le Blanc viruses primarily infect intestinal cells in Caenorhabditis nematodes. Virology. 448, 255-264 (2014).
- Tran, T. D., Ali, M. A., Lee, D., Luallen, R. J. Bacterial filamentation as a mechanism for cell-to-cell spread within an animal host. Nature Communications. 13 (1), 1-11 (2022).
- Luallen, R. J., et al. Discovery of a natural microsporidian pathogen with broad tissue tropism in Caenorhabditis elegans. PLOS Pathogens. 12 (6), 1005724 (2016).
- Lažetić, V., et al. et al The transcription factor ZIP-1 promotes resistance to intracellular infection in Caenorhabditis elegans. Nature Communications. 13 (1), 1-16 (2022).
- Félix, M. -. A., et al. Species richness, distribution and genetic diversity of Caenorhabditis nematodes in a remote tropical rainforest. BMC Evolutionary Biology. 13 (1), 10 (2013).
- Morgan, E., Longares, J. F., Félix, M. A., Luallen, R. J. Selective cleaning of wild Caenorhabditis nematodes to enrich for intestinal microbiome bacteria. Journal of Visualized Experiments. (174), e62937 (2021).
- Sowa, J. N., et al. The Caenorhabditis elegans RIGI homolog DRH-1 mediates the intracellular pathogen response upon viral infection. Journal of Virology. 94 (2), 01173 (2020).
- Bakowski, M. A., et al. Ubiquitin-mediated response to microsporidia and virus infection in C. elegans. PLOS Pathogens. 10 (6), 1004200 (2014).
- Reddy, K. C., et al. Antagonistic paralogs control a switch between growth and pathogen resistance in C. elegans. PLoS Pathogens. 15 (1), 1007528 (2019).
- Reddy, K. C., et al. An intracellular pathogen response pathway promotes proteostasis in C. elegans. Current Biology. 27 (22), 3544-3553 (2017).
- Balla, K. M., Lažetić, V., Troemel, E. R. Natural variation in the roles of C. elegans autophagy components during microsporidia infection. PloS One. 14 (4), 0216011 (2019).
- Dirksen, P., et al. The native microbiome of the nematode Caenorhabditis elegans: gateway to a new host-microbiome model. BMC Biology. 14 (1), 38 (2016).
- Fu, R., Gong, J. Single cell analysis linking ribosomal (r)DNA and rRNA copy numbers to cell size and growth rate provides insights into molecular protistan ecology. The Journal of Eukaryotic Microbiology. 64 (6), 885-896 (2017).
- Willis, A. R., et al. A parental transcriptional response to microsporidia infection induces inherited immunity in offspring. Science Advances. 7 (19), (2021).
- Cuomo, C. A., et al. Microsporidian genome analysis reveals evolutionary strategies for obligate intracellular growth. Genome Research. 22 (12), 2478-2488 (2012).
- Reinke, A. W., Balla, K. M., Bennett, E. J., Troemel, E. R. Identification of microsporidia host-exposed proteins reveals a repertoire of rapidly evolving proteins. Nature Communications. 8 (1), 14023 (2017).
- Rivera, D. E., Lažetić, V., Troemel, E. R., Luallen, R. J. RNA fluorescence in situ hybridization (FISH) to visualize microbial colonization and infection in the Caenorhabditis elegans intestines. bioRxiv. , (2022).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。