Method Article
体外研究 用于靶向表观遗传沉默的工程化转录抑制因子的选择
摘要
在这里,我们提出了一种用于 体外 选择工程转录抑制因子 (ETR) 的方案,该方案具有高、长期、稳定、靶向沉默效率和低全基因组脱靶活性。该工作流程允许将初始的、复杂的候选 ETR 库减少到一个简短的列表,适合在治疗相关环境中进行进一步评估。
摘要
基因灭活有助于研究基因功能,是治疗多种疾病的有前途的策略。在传统技术中,RNA干扰存在部分靶标消除和终身治疗的需要。相比之下,人工核酸酶可以通过诱导DNA双链断裂(DSB)来实现稳定的基因失活,但最近的研究质疑这种方法的安全性。 通过 工程转录阻遏因子 (ETR) 进行靶向表观遗传编辑可能是一种解决方案,因为单次施用特定的 ETR 组合可以导致持久的沉默,而不会诱导 DNA 断裂。
ETR 是含有可编程 DNA 结合域 (DBD) 和来自天然转录阻遏蛋白的效应子的蛋白质。具体而言,配备人 ZNF10 的 KRAB 结构域、人 DNMT3A 和人 DNMT3L 的催化结构域的三种 ETR 的组合被证明可以在 ETR 靶基因上诱导可遗传的抑制性表观遗传状态。该平台的肇事逃逸性质,对靶标的DNA序列没有影响,以及通过按需DNA去甲基化恢复到抑制状态的可能性,使表观遗传沉默成为改变游戏规则的工具。一个关键步骤是确定ETR在靶基因上的正确位置,以最大限度地提高靶向和最小化脱靶沉默。在最终的 离体 或 体内 临床前环境中执行此步骤可能很麻烦。
本文以 CRISPR/催化死亡的 Cas9 系统作为 ETR 的范式 DBD,描述了一种方案,包括体 外 筛选向导 RNA (gRNA) 与三重 ETR 组合偶联,以实现有效的靶向沉默,然后评估热门命中的全基因组特异性特征。这允许将候选 gRNA 的初始库减少到一小段有前途的 gRNA,其复杂性适合在感兴趣的治疗相关环境中进行最终评估。
引言
传统上,基因失活在研究细胞和动物模型中的基因功能方面发挥着关键作用。此外,在过去的二十年中,随着基因疗法的兴起,它已被提议作为一种潜在的改变游戏规则的方法,用于治疗由功能获得性突变1、传染病2 或一种基因沉默可以补偿另一个基因的遗传缺陷3 的病理学引起的疾病。最后,提出了细胞适应性和功能控制关键调节因子的遗传失活,以提高细胞产品在癌症免疫治疗4 和再生医学5 中的效率。
在实现基因失活的不同技术中,最有前途的技术之一是靶向表观遗传沉默6,7。该技术的核心是所谓的工程转录抑制因子(ETR),即由可编程DNA结合结构域(DBD)和具有表观遗传抑制功能的效应结构域(ED)组成的嵌合蛋白。锌指蛋白 (ZFP)8、转录激活因子样效应子 (TALEs)9 或基于 CRISPR/dCas910 的 DBD 可以设计为选择性地将 ED 拴系在待沉默的靶基因的启动子/增强子序列上。一旦到达那里,ETR 的 ED 通过施加异染色质诱导的抑制性表观遗传标记来执行其沉默活性,例如组蛋白修饰(H3K9 11,12 或 H3K27 13 甲基化、H3 或 H4 脱乙酰化14)和 CpG DNA 甲基化 15,根据所使用的抑制结构域。
特别是,受植入前胚胎16 中发生的内源性逆转录病毒永久转录抑制的分子过程的启发,已经产生了三种 ETR 的组合来利用以下 ED:i) 人 ZNF10 的 Krüppel 相关盒 (KRAB) 结构域;ii) 人从头 DNA 甲基转移酶 3A (DNMT3A) 的催化结构域;iii) 全长人类 DNA 甲基转移酶 3 样 (DNMT3L)。KRAB 是高等脊椎动物中多个 ZFP 共享的保守抑制结构域17,18,其沉默活性主要基于其募集 KAP119-a 支架蛋白的能力,然后与其他几种异染色质诱导剂相互作用 20-包括核小体重塑和脱乙酰化 (NuRD) 复合物 21、H3K9 组蛋白甲基转移酶 SETDB1 22 和 H3K9 甲基化读取器 HP1 23,24 等。
DNMT3A 在 CpG 序列25 处主动转移 DNA 上的甲基。DNMT3A 的催化活性通过其与 DNMT3L 的物理结合而增强,DNMT3L 是 DNMT3A 的胚胎和生殖细胞限制性旁系同源物,缺乏负责甲基转移的催化结构域26,27。嵌入哺乳动物基因启动子/增强子元件的富含 CpG 的区域(称为 CpG 岛 (CGI))的 DNA 甲基化通常与转录沉默有关28。重要的是,一旦沉积,CpG 甲基化可以通过基于 UHRF1-DNMT1 的分子复合物在整个有丝分裂过程中稳定遗传29。
ETRs在靶细胞中的稳定过表达可能是有问题的,这可能是因为随着时间的推移,脱靶活性和内源性相互作用物从其生理靶位点被抑制的风险增加。然而,单 ETR 部分的瞬时表达可能无法以高效率诱导长期沉默30,从而阻碍了它们的治疗应用。因此,该领域的一项开创性突破是有证据表明,三种基于 KRAB、DNMT3A 和 DNMT3L 的 ETR 的组合可以协同作用,即使只是瞬时共递送,也会对靶基因 H3K9 和 CpG 甲基化的启动子序列施加影响。然后,这些细胞在整个有丝分裂过程中被细胞读取和繁殖,导致多种人和小鼠细胞系以及 离体 培养的原代细胞中的可遗传沉默30。
值得注意的是,ETR 施加的表观遗传沉默可以通过靶向(例如,在沉默位点上募集基于 CRISPR/dCas9 的 TET1 DNA 去甲基化酶)或药理学(施用 DNA 甲基转移酶抑制剂 5-Aza)DNA 去甲基化30 来按需恢复,这是 ETR 相关不良事件的潜在解毒剂。还描述了携带三种基于 KRAB、DNMT3A 和 DNMT3L 的 ED 的多合一 ETR,显示出细胞系31,32 对绝大多数蛋白质编码基因的显着沉默效率。此外,几项采用 ETR 的研究报告了高安全性,在从头 CpG 甲基化或染色质可及性改变方面没有重大脱靶活性30,31,32。然而,建议在临床应用之前对配备新设计的 DBD 的 ETR 的特异性特征进行专门分析。
从临床角度来看,靶向表观遗传沉默可能为基于 RNA 干扰 (RNAi) 的敲低33 和基于人工核酸酶的基因破坏8 提供关键优势。与RNAi相比,靶向表观遗传沉默可以诱导每个细胞的靶标完全消失,并且不需要定期治疗来确保长期沉默;与基因破坏相比,它使 DNA 序列保持不变,避免了 DNA 双链断裂 (DSB) 的产生。然后,DSB 可以诱导细胞凋亡和细胞周期停滞,可能导致针对具有功能性 p53 通路的细胞的选择 34,35,尤其是在多重基因编辑环境中,染色体重排35。此外,通过传递非同源末端连接介导的 DNA DSB 修复36 的不可逆镶嵌结果,基因破坏无法避免将靶标的框内修复为功能编码序列作为最终结果之一,并且与表观遗传沉默相比,不能按需擦除。
最后,表观遗传沉默有可能将可靶向遗传元件的范围扩大到完全或至少部分对 RNAi 和基因破坏难治的类别,例如非转录调控元件和非编码 RNA30,32。任何靶向表观遗传沉默应用的第一个关键步骤是设计一组涵盖靶基因不同调控序列的 ETR,并确定性能最佳的 ETR。考虑到不断开发的可编程 DNA 结合技术可以靶向的基因组部分越来越多,要测试的 ETR 数量可能至关重要37。直接在细胞类型上进行ETR的筛选,以治疗性地沉默靶基因将代表最相关的选择。然而,高通量筛选在原代细胞中可能技术上很麻烦,因为它们在培养中的存活率有限,而且其工程能力通常不理想。大屏幕在体内可能更加不可行。
一种更实用的替代方案包括首先在易于工程化的细胞系中对大量 ETR 进行初步筛选,然后只验证治疗相关细胞类型中最有希望的 ETR。一个平行的问题是选择适当的读数来测量ETR的沉默效率。通过RT-qPCR、蛋白质印迹或ELISA直接评估靶基因的转录本或蛋白水平可能既昂贵又耗时,并且可能缺乏足够的灵敏度,从而限制了它们在高通量规模上的应用。在靶基因调控序列的转录控制下,产生 临时 工程化报告细胞系,允许利用基于流式细胞术的方法在单细胞水平和高通量速度读取表观遗传沉默。
遵循这些一般考虑,本文描述了一种方案,该方案包括用于靶向沉默效率的 体外 阵列 ETR 筛选,然后评估顶级命中的全基因组脱靶活性。该工作流程允许将候选 ETR 的初始库减少到一个有前途的短列表,其复杂性适合于在感兴趣的治疗相关细胞类型中进行最终评估。
在可用于生成 ETR 的不同可编程 DBD 中,该协议将专注于基于 CRISPR/dCas9 的技术,因为易于在高通量规模上设计跨越靶基因启动子的 gRNA。然而,可以采用下面描述的相同概念工作流程来评估配备其他DBD的ETR的效率和特异性。
研究方案
1. 设计基于荧光的报告细胞系,通过流式细胞术监测靶基因的转录活性
- 鉴定表达要沉默的靶基因的细胞系。浏览人类蛋白质图谱38 中要沉默的靶基因,并浏览其"细胞系"部分,以识别代表目标体细胞组织的细胞系(例如,如果最终靶标是肝细胞,则为肝细胞系)。或者,查询公开可用的 RNA 测序 (RNA-Seq) 数据库(例如 NCBI GEO)。
- 在候选细胞中,优先考虑具有高效瞬时基因递送方案(用于ETR递送)的细胞系。
注:在不同的模式中,核转染是最佳选择之一,因为它确保了高转染效率。在这里,人红血病 K-562 细胞被选中产生报告 β-2-微球蛋白 (B2M) 基因(以下简称 B2MTdTomato K-562 细胞)转录活性的细胞系。 - 进一步优先考虑候选基因,以避免靶基因对细胞活力至关重要的细胞系,因为这会损害细胞的维持,并在培养中稳定沉默靶基因。如果之前没有报道过,为了了解靶基因在所选细胞类型中的重要性,通过用 Cas9 核酸酶和靶向基因的第一个编码外显子之一的 gRNA 转染细胞来产生遗传破坏对照。
注意:根据定义,遗传破坏是一个稳定的事件;随着时间的推移,对被破坏细胞的反选择表明靶基因对所选细胞的生理学至关重要。- 确定在所选细胞系中优先使用的靶剪接亚型( B2M 基因的亚型NM_004048.4在本方案中靶向)。
- 鉴定一种能够在靶亚型的第一个编码外显子中有效且特异性切割的 gRNA(例如,Chopchop (http://chopchop.cbu.uib.no/)39,这是一种有效且用户友好的在线 gRNA 选择工具)。
- 对于 K-562 细胞,每 5 个× 105 个细胞转染 1 μg spCas9 编码质粒(hCas9;参见材料表)和 250 ng gRNA 编码质粒(phU6-gRNA;参见材料表)通过核转染(根据制造商的说明)。
- 培养细胞(K-562细胞在37°C下,在补充有10%胎牛血清[FBS],L-谷氨酰胺和青霉素/链霉素[100U / mL]的RPMI-1640中,并监测基因破坏水平随时间的变化,利用突变检测试剂盒(遵循制造商的说明)。
- 克隆供体模板,用于在目标基因的转录控制下进行荧光报告基因的同源重组介导的整合(图1)。
- 鉴定靶基因的区域以整合荧光团表达盒。
注意:避免靶向转录相关元件,例如富集 H3K27 乙酰化的 CpG 岛和区域(活性启动子和增强子的标志物)。改变这些调控元件(ETR靶向以指示表观遗传沉默可能很重要)使报告细胞系对靶基因的生理调控的预测性降低。- 如果靶基因不编码分泌蛋白,则通过2A自裂解肽将报告基因融合到靶基因的最后一个密码子,以维持靶基因的功能。
- 如果靶基因确实编码分泌蛋白,为避免潜在分泌报告基因,请将报告基因置于靶基因的内含子区域。通过剪接受体位点 (SA) 将报告基因强行整合到剪接转录本中,随后通过内部核糖体进入序列 (IRES) 进行翻译会损害靶基因的功能。
- 使用 Chopchop 选择靶区中的 gRNA 切割(这里,选择 gRNA 靶向 B2M 基因第一个内含子的序列 5′-AGGCTACTAGCCCCATCAAGAGG-3′)。
- 为 gRNA 切割位点设计供体模板,包括:i) 左同源臂(n 个碱基对 [bp] 与 gRNA 切割位点上游区域匹配);ii) 无启动子的转基因表达盒(在此所示的情况下,SA-3X 终止密码子-IRES-tdTomato-BGH poly(A) 有利于与 B2M 基因的第一个内含子剪接);iii) 右同源臂(n bp 与 gRNA 切割位点下游区域匹配)。
注意:有效诱导同源重组所需的同源臂的长度可能因不同细胞类型而异(100-500 bp 是 K-562 细胞的适当范围)。
- 鉴定靶基因的区域以整合荧光团表达盒。
- 将 CRISPR/Cas9 核酸酶系统和供体模板递送至靶细胞系内。对于 K-562 细胞,通过核转染每 5 个× 10 5个细胞转染 1 μg spCas9 编码质粒(hCas9;参见材料表)、250 ng g(p250-5)编码质粒(根据制造商的说明)。
- 培养细胞至少14天(对于K-562细胞),并使用流式细胞仪监测荧光报告基因的表达水平随时间变化(激活藻红蛋白(PE)通道以测量tdTomato报告基因的荧光强度,并按照制造商的说明进行流式细胞术)。
注:供体模板(特别是基于质粒的供体模板)可以包含隐秘的启动子序列,导致荧光报告基因从非整合供体拷贝中表达。培养细胞允许通过细胞分裂稀释这些非整合拷贝,并最终仅从整合在靶基因组中的供体拷贝中维持报告基因的表达。 - 通过单细胞水平的荧光激活细胞分选 (FACS) 克隆报告阳性细胞。对于该协议,激活PE通道以测量tdTomato报告基因的荧光强度,并按照制造商的说明对96孔板的每个孔分选单个tdTomato阳性K-562细胞。
- 在培养物中细胞扩增后(K-562 细胞通常为 20-30 天),通过 PCR 筛选报告阳性克隆,以选择一个在靶基因座内具有报告盒双等位基因整合的克隆。
注意:这最大限度地提高了报告基因表达,并促进了ETR处理后通过流式细胞术进一步分离报告基因表达细胞和报告基因沉默细胞之间的分辨率。- 使用 DNA 提取试剂盒(按照制造商的说明)从每个报告×阳性克隆的 1 个10 个 细胞中提取基因组 DNA。
- 按照PCR扩增试剂盒的说明,用正向(5'-GTATTTGCTGGTTATGTTAG-3')和反向(5'-AATGGTTGAGTTGGAC-3')引物扩增 B2M 靶区。这对引物的退火温度为47.7°C,使用基于Taq的DNA聚合酶,引物浓度为0.5μM。
- 使用1%琼脂糖凝胶电泳(按照制造商的说明)分析PCR产物。筛选显示与 tdTomato 整合在 B2M 靶位点 (3,413 bp) 相关的条带,而没有与野生型靶位点相关的条带 (1,027 bp)。
2. 设计用于基于 CRISPR/dCas 9 的靶基因表观遗传沉默的 gRNA
- 在UCSC基因组浏览器40中浏览靶基因,并提取可能调节其转录活性的区域的核苷酸序列,例如CpG岛和富集H3K27乙酰化的位点(活性启动子和增强子的标志物)。
注:根据最近的一项研究,最佳靶向区域是以靶基因32 的转录起始位点为中心的 1 千碱基 (kb) 窗口。 - 将所选序列粘贴到 Chopchop 在线工具中,然后选择 抑制 作为要检索的 gRNA 的目的。等待 Chopchop 提供映射在目标基因序列上的 gRNA 列表,并根据考虑脱靶匹配数量和预测靶向效率的分数列出(图 2)。
- 每个靶序列至少选择 10 个 gRNA。如果可能,尝试选择跨越整个区域的gRNA进行检测,与整个基因组中的其他基因内序列没有完全匹配。
3. 基于CRISPR/dCas 9的ETR在报告细胞系中的阵列瞬时递送
- 在先前报道的靶细胞系转基因递送系统中,选择仅允许瞬时转基因表达的系统,最大限度地提高递送效率,并最大限度地减少细胞操作相关毒性。对于 K-562 细胞,强烈建议同时进行质粒和 mRNA 核转染,质粒生产在技术上是一种更简单、更便宜的替代方案。
- 在所选的转基因递送系统中克隆第 2 节中选择的 gRNA 和基于 CRISPR/dCas9 的 ETR。有关在质粒DNA中克隆ETR的方案,请参阅以下步骤。
注:单独编码dCas9:KRAB、dCas9:DNMT3A和dCas9:DNMT3L30的质粒在Addgene上不可用。通过将质粒 pAC154-dual-dCas9VP160-sgExpression41 中的 VP160 反式激活剂替换为 KRAB、DNMT3A 或 DNMT3L 结构域编码序列30 来克隆它们。一种用于多合一 ETR 的质粒编码,称为 CRISPRoff-v2.132(参见材料表)。- 在化学感受态 大肠杆菌 细胞中转化编码ETR的质粒(遵循制造商的说明)。通过限制性内切酶消化和Sanger测序筛选菌落是否存在携带ETR的质粒,最后选择一种阳性菌落用于质粒DNA Midiprep生产(遵循制造商的说明)。
- 克隆phU6-gRNA骨架内的gRNA(图3)。
- 使用分子生物学设计软件,在原间隔区(所选 gRNA 的第一个可变 20 个核苷酸 [nt])上游附加一个 5'-CACCG-3' 序列,以生成 25 nt 长的 计算机寡核苷酸,称为 SGfw。
- 类似地,在所选 gRNA 的原间隔区反向补体的上游附加一个 5'-AAAC-3' 序列,在其下游附加一个 5'-C-3' 序列,以产生一个 25 nt 长的寡核苷酸,称为 SGrv。
- 订购 SGfw 和 SGrv 序列作为无盐单链 DNA 寡核苷酸,以 100 μM 重悬于水中。
- 将 1 μL 每种寡核苷酸加入 2 μL 退火缓冲液(10 mM Tris [pH 7.5-8.0]、50 mM NaCl、1 mM EDTA)和 16 μL 水中。
- 通过将溶液置于编程为在95°C下开始10分钟的热循环仪中进行寡核苷酸退火。然后,在45分钟内逐渐冷却至25°C。
- 用 99 μL 无核酸酶水稀释 1 μL 退火寡核苷酸,然后用先前用 BsaI 限制性内切酶消化的 50 ng phU6-gRNA 质粒连接 1 μL 该稀释液(按照 BsaI 和连接酶试剂盒供应商的说明进行消化和连接程序)。
- 用 2 μL 连接产物转化 20 μL 化学感受态 大肠杆 菌细胞(按照制造商的转化程序说明进行转化)。
- 选择多个菌落用于质粒DNA Miniprep生产(遵循供应商的说明),并使用以下与U6启动子5'-GAGGGCCTATTTCCCATGATT-3'匹配的引物通过Sanger测序控制原间隔区的成功克隆。
- 选择一种阳性菌落用于质粒DNA Midiprep生产(按照制造商的说明)。
- 在阵列报告细胞系中递送选定的 gRNA 和基于 CRISPR/dCas9 的 ETR(每种条件一个特定的 gRNA)(图 3)。
注:作为一个代表性案例,对编码 dCas9:KRAB、dCas9:DNMT3A、dCas9:DNMT3L 和靶向 B2M CpG 岛的 gRNA 的质粒进行核转染的工作流程如下TdTomato K-562 细胞所示。- 准备含有500ngdCas9:KRAB-,dCas9:DNMT3A-和dCas9:DNMT3L编码质粒的单独试管,但要测试的gRNA不同(每管125ngg编码质粒)。将不含 gRNA 和 ETR 的核转染条件作为模拟处理的样品。对每个样品进行至少三次技术重复的筛选。
- 每管沉淀 5 个× 10个 5 个B2M TdTomato K-562 细胞,并用质粒混合物对它们进行核化(按照制造商的说明)。
- 将细胞重悬于先前加热的 RPMI-1640 哺乳动物细胞培养基的 200 μL 中,并将它们放回培养箱中。
4. 分析靶基因随时间变化的转录活性
- 使用流式细胞术测量ETR递送后不同时间点沉默细胞的百分比(图4)。使用不携带荧光团编码序列的野生型 (WT) 细胞来设置报告阴性细胞的阈值。使用模拟处理的样品为报告阳性细胞设置门。
注意:如步骤1.3所示,在实验中包括遗传破坏对照。这对于监测CRISPR递送效率和被剥夺靶基因的细胞的适应性都非常有用;随着时间的推移,转录沉默和遗传破坏的丧失可归因于靶基因在所选细胞类型中的重要性。包括短期(第 3 天、第 7 天、第 10 天)和长期(第 21 天、第 35 天)时间点,以指示沉默的急性和长期效率。 - 确定长期沉默效率排名前三的 gRNA。使用FACS选择在这些样本中稳定维持的报告阴性亚群。此外,对大量模拟处理的样品进行流式细胞仪,使其处于与测试样品相同的处理状态,以便在随后的分析中进行适当的比较。
5. 通过 RNA-seq 和甲基化 DNA 免疫沉淀 (MeDIP)-seq 评估 ETR 治疗的特异性
- 使用 RNA-seq 评估 ETR 递送时任何最终的全基因组转录失调。
- 对于用三种性能最好的 gRNA 处理的样品的报告沉默亚群和模拟处理的细胞,使用市售试剂盒提取 RNA。使用市售试剂盒评估RNA的质量和浓度。
- 使用市售试剂盒进行RNA片段化、逆转录和文库制备,以制备RNA-seq文库(遵循制造商的说明)。
- 使用与下一代测序和数字电泳兼容的质控仪器进行文库定量和质量控制。
- 下一代测序仪上的序列库遵循制造商的说明,采用 100 bp 的双端方案,目标是平均 45 M reads/样本。
- 将读取标签与适当的参考基因组对齐并量化转录本表达。对 tdTomato 序列进行比对并单独量化。
注意:这里使用带有默认参数的 STAR aligner (v 2.3.0)43,与 Rsubread 包44 耦合。 - 根据已发布的最佳实践对RNA-seq数据进行分析45。
注:此处使用 R/Bioconductor 封装 edgeR46,在至少三个样品中应用至少百万分之一计数 (cpm) 的过滤器,以丢弃低表达基因。或者,可以使用 edgeR 中的 filterByExpr 函数。 - 使用实施的负二项式广义对数线性模型评估差异基因表达 edgeR(函数 glmFit)47。将调整后的 p 值(Benjamini-Hochberg [BH]校正)的阈值设置为0.01,以保留差异调控的基因。
- 通过 MeDIP-seq 评估 ETR 的任何最终脱靶 CpG 甲基化活性。
- 对于用前三个 gRNA 处理的样品的报告沉默亚群和模拟处理的细胞,使用市售试剂盒(按照制造商的说明)提取基因组 DNA。
- 使用超声仪和以下参数超声处理 500 ng 基因组 DNA: 占空比:20 %;点:175;每次连发周期数:200;时间:40秒。
- 使用市售的MeDIP-seq试剂盒制备测序文库(遵循制造商的说明)。
- 在接头连接步骤之后,通过荧光测定法定量文库,并使用市售文库定量试剂盒(按照制造商的说明)通过qPCR检查连接效率。
- 通过混合随机选择的库来获得库库,以减少技术偏差。对每个库使用相等的库量 (ng) 来平衡池。向每个池中加入试剂盒中提供的甲基化和非甲基化加标对照 DNA。出于对照目的,保留 10% 体积的文库,标记为"输入"且未免疫沉淀。使用 MeDIP-seq 试剂盒中提供的针对 5-甲基胞嘧啶的单克隆抗体对剩余的 90% 文库进行免疫沉淀。
- 按照制造商的说明,使用用于纯化 5-甲基胞嘧啶免疫沉淀产物的试剂盒纯化富集的文库和输入文库。
- 通过使用试剂盒随附的引物对对照的内部加标进行定量实时荧光定量PCR来评估富集效率。对于每种免疫沉淀 (IP),使用 MeDIP 的循环阈值 (Ct) 值和从 qPCR 反应获得的输入组分(参见公式 1 和 2)计算甲基化和非甲基化 DNA 回收率的富集特异性:
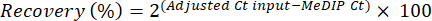 (1)
(1)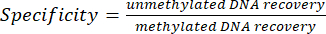 (2)
(2)
注:如果特异性值为 ≥0.95,则认为 IP 文库成功。 - 按照制造商的说明,使用 MeDIP-seq 文库制备试剂盒扩增文库,并对产物进行定量和文库大小分布分析。
- 在下一代测序仪上进行文库测序。使用双端测序,读取长度为 100 bp,目标是平均 30 M reads/样本。
- 使用 bwa(v 0.7.5 或更高)48 将测序读长标签与适当的参考基因组(例如 hg38)对齐,然后使用 MACS(v 2.0.10 或更高)49 鉴定峰,从而鉴定宽峰 (-slocal = 0,-llocal = 500000)。
- 使用 BEDTools 的多交集工具50 从不同样本创建一组通用区域,启用聚类选项。
- 使用 BEDTools 的 multicov 计算最终区域列表中的每个样本覆盖率,丢弃重复的读长。
- 使用 edgeR 执行矩阵计数分析。在至少三个样品中应用至少百万分之一计数 (cpm) 的过滤器,以丢弃低富集区域。
注意:或者,可以使用 edgeR 中的 filterByExpr 函数。 - 通过采用 edgeR(函数 glmFit)中实现的广义对数线性模型并使用条件分位数归一化51 进行归一化来校正区域 GC 含量,从而识别差异甲基化。通过对 BH 调整的 p 值应用阈值 0.01 来选择差异甲基化区域。按如下方式对重复序列进行分析。
注意:有关选项和参数的完整列表,请参阅 edgeR 用户指南(https://bioconductor.org/packages/release/bioc/html/edgeR.html)。- 过滤 MeDIP-seq 结果以获得标称 p 值 <0.01,并创建两组区域:选择第一组中具有 logFC >1 的区域,以及第二组中具有 logFC <-1 的区域。
- 将所选基因组的 RepeatMasker 注释检索为床文件,并计算每组中的元素数。将计数转换为每个数据集的区域数的比率。
- 提取与每类重复重叠的甲基化组的比率,并进行卡方检验以检测任何显着富集。
- 评估 ETR 和模拟处理样品之间的差异转录或差异甲基化区域是否映射到 计算机预测的脱靶 gRNA 结合。
- 使用 CRISPR 设计套件52 作为脱靶 gRNA 结合预测工具。
- 对于每个假定的脱靶区域,查看最近的转录起始位点 (TSS) 和最接近的甲基化区域。将与 FDR <0.01 调控且与 TSS 距离小于 10 Kb 的基因相关的区域,或与 FDR <0.01 调控且距离小于 1 Kb 的甲基化区域相关的区域视为真正的脱靶效应。
- 与模拟处理的样品相比,确定用三种性能最好的 gRNA 处理的样品中转录改变或过度甲基化区域的数量和特征(图 5),以确定 gRNA 中特异性最高的区域。按以下顺序对脱靶位点对靶细胞生理学的潜在影响进行排序(从影响最大到影响较小):
i) 基因内调控区生理表达基因
ii) 基因内、外显子区域生理表达基因
iii) 基因内含子区生理表达基因
iv) 基因内调控区未表达基因
v) 基因内、外显子区不表达基因
vi) 基因内含子区域未表达基因
vii) 基因间区
结果
在与CRISPR/Cas9系统偶联的靶位点中同源重组介导的荧光报告基因整合的供体模板(例如,在K-562细胞的情况下通过质粒核转染)递送供体模板后,报告基因阳性细胞出现在处理的样品中(图1,底部)。如果没有发生这种情况,请重新检查供体模板和 CRISPR/Cas9 试剂的设计和克隆的准确性。如果确认,请尝试优化试剂剂量和递送方案本身。
获得报告细胞系后,选择靶基因的启动子/增强子序列并设计一组匹配的 gRNA。在计算机预测工具(如 Chopchop)中使用,既可以识别 gRNA 序列,又可以根据预测的效率和特异性对它们进行排名(图 2)。如果未检索到gRNA,则控制靶序列中典型Cas9原间隔区相邻基序(PAM)(5'-NGG-3')的存在。如果不存在 PAM 序列,请考虑转向基于替代 PAM 非依赖性 Cas9 变体的 ETR(尚未发布带有这些变体的 ETR)或切换到替代 DNA 结合域平台,例如 ZFP53 或 TALEs30。但是,如果仅检索到预测功效/特异性较低的 gRNA,请考虑 1) 测试这些低质量的 gRNA 或 2) 扩展靶 DNA 序列,以寻找更好的 gRNA。在瞬时递送三重 ETR 组合(或 CRISPRoff;v2.1)与 gRNA 一起后,对荧光报告基因的表达进行纵向流式细胞术分析,通常在急性分析中观察到报告基因抑制的峰值,然后由于 ETR 编码质粒的有丝分裂稀释随着时间的推移至少部分重吸收(图 4C).如果 ETRs/gRNA 组合有效地将 CpG 甲基化沉积在靶基因座上,则在相当一部分处理的细胞中将发生报告基因的永久抑制(图 4C)。不同的gRNA可以显示出不同的长期沉默效率(图4B,C)。
对于全基因组特异性评估,使用 MeDIP-seq 鉴定靶标长期沉默的细胞和未处理细胞之间的差异甲基化区域。理想情况下,高度特异性的 gRNA 只会在靶位点诱导从 头 CpG 甲基化峰(图 5)。如果没有,可以考虑表征在靶向效率列表中排名较低的 gRNA 的脱靶活性。
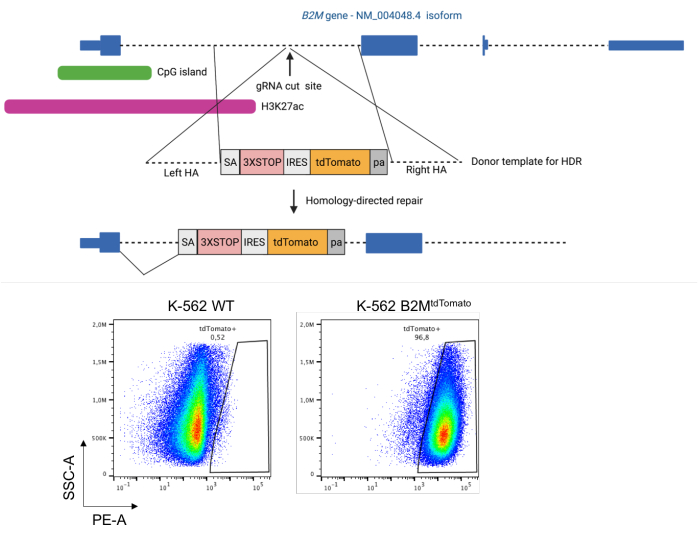
图 1:通过同源定向修复将 tdTomato 报告基因整合到人类 B2M 基因的调控元件下。 上图:通过CRISPR/Cas9诱导的同源定向修复将tdTomato荧光报告基因整合到人类 B2M 基因的第一个内含子中的策略示意图。下图:K-562 细胞在 B2M 基因第一个内含子中 tdTomato 报告基因整合前和整合后的代表性点图。缩写:HA = 同源臂;HDR = 同源定向修复;IRES = 内部核糖体进入位点;pa = BGH poly(A);SA = 剪接受体位点;WT = 野生型;3XSTOP = 三个串联终止密码子。 请点击这里查看此图的较大版本.
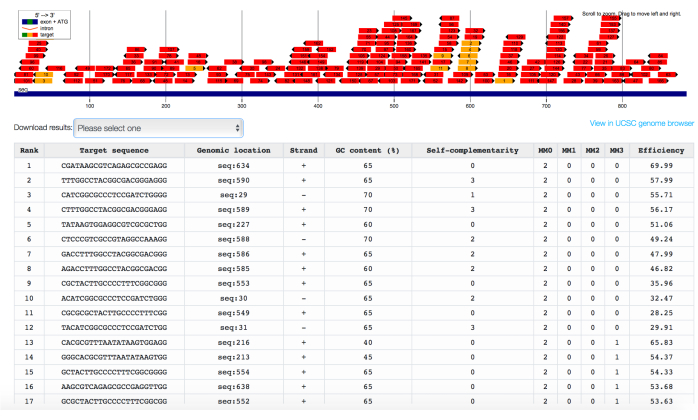
图 2:靶向 B2M 基因 CpG 岛的 gRNA 的计算机鉴定,根据预测效率和特异性进行排名。 Chopchop 的输出界面显示了靶向嵌入 B2M 基因启动子序列中的 CpG 岛的 gRNA,根据错配为 0 (MM0)、1 (MM1)、2 (MM2) 或 3 (MM3) 错配的脱靶序列数量和预测的靶向效率进行排名。请点击这里查看此图的较大版本.
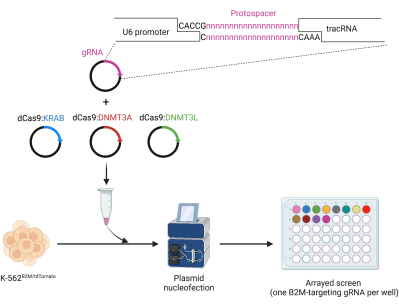
图 3:用于 dCas9 ETR 介导的表观遗传沉默的 gRNA 的克隆和阵列核转染。 上图:寡核苷酸介导的表达人U6-gRNA质粒中的原间隔区克隆。下图:靶向 B2M 的 gRNA 阵列筛选,用于在 K-562B2M/tdTomato 细胞中通过质粒核转染进行基于 CRISPR/dCas9 的表观遗传沉默。 请点击这里查看此图的较大版本.
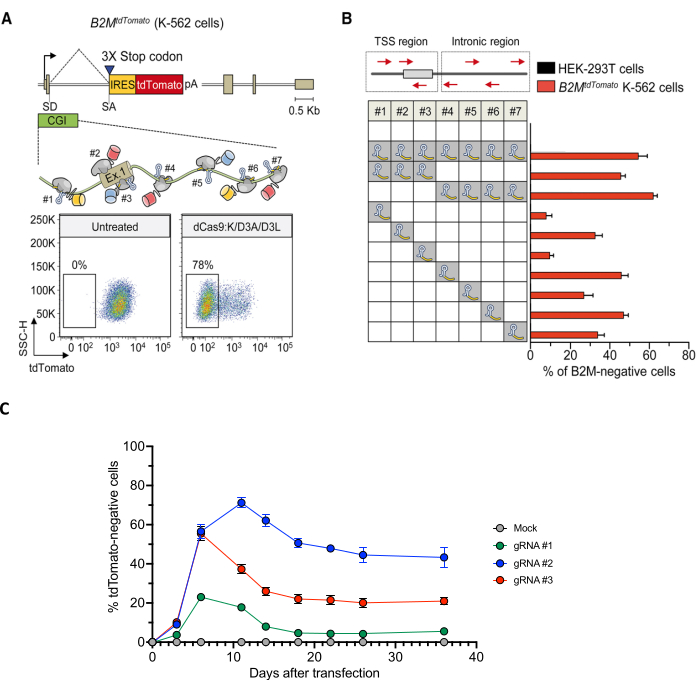
图 4:筛选有效诱导 B2M 基因长期沉默的 gRNA。 (A) 上图:放大区域描绘的 B2M tdTomato 基因示意图,基于 dCas9 的 ETR 与 gRNA 复合物结合的相对顺序和方向。下图:在ETR沉默之前(左)或之后(右)B2MtdTomato K-562细胞的代表性点图。在转染后 30 天使用编码 gRNA 的质粒和三重 dCas9:KRAB+ dCas9:D3A + dCas9:D3L 组合进行分析。(B) 转染后第 30 天,K-562 B2MtdTomato 细胞中靶向 B2M 的 CpG 岛的指示 gRNA(在池中或作为单个 gRNA)的沉默活性(顶部示意图中的红色箭头表示 gRNA 的方向)。数据显示tdTomato阴性细胞的百分比(SEM±平均值;n = 每种处理条件下的四次独立转染)。(C) 使用表达三重 ETR 组合和指示的 B2M CpG 岛靶向 gRNA 或模拟(无 gRNA 和 ETR 转染)的质粒转染 B2M tdTomato K-562 细胞的时程分析。数据显示tdTomato阴性细胞的百分比(SEM±平均值;n = 每种处理条件下的三次独立转染)。未发布的数据。图A和B改编自Amabile等人30。缩写:CGI = CpG island;IRES = 内部核糖体进入位点;pa = BGH poly(A);SA = 剪接受体位点;SD = 剪接供体位点;TSS = 转录起始位点;UT = 未转染;3XSTOP = 3 个串联终止密码子。请点击这里查看此图的较大版本.
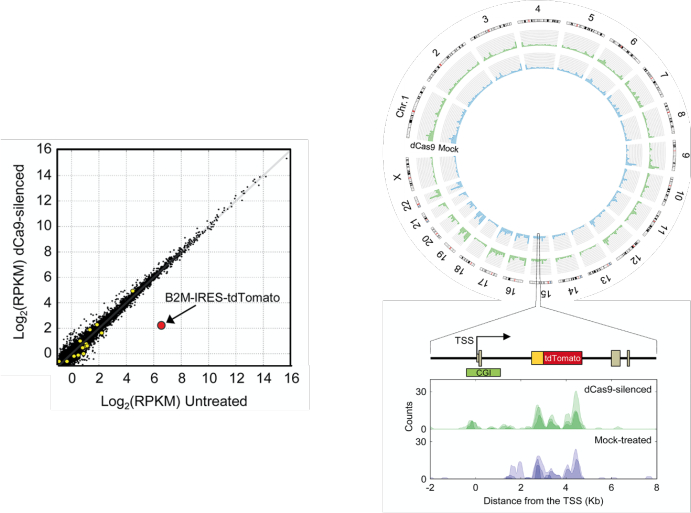
图 5:通过 RNA-seq 和 MeDIP-seq 对 ETR 特异性进行全基因组分析。 左图:模拟处理的 B2MtdTomato K-562 细胞和用三重 dCas9:KRAB、dCas9:DNMT3A、dCas9:DNMT3L ETR 组合和靶向 B2M 基因的 CpG 岛的 gRNA 处理的细胞的表达水平比较。值以映射读取的每千碱基每千碱基 (RPKM) 读取数的 log2 表示。黑点表示在所有条件下以可比水平表达的基因;黄色圆圈代表在FDR<0.01下受到差异调控的基因;红色圆圈代表 B2M-IRES-tdTomato 成绩单。右上图:circos 图显示了模拟处理的 B2MtdTomato K-562 细胞(蓝色)或用三重 dCas9:KRAB、dCas9:DNMT3A、dCas9:DNMT3L ETR 组合和靶向 B2M 基因的 CpG 岛 (CGI) 的 gRNA 处理的细胞(绿色)的全基因组 MeDIP-seq 图谱。右下:显示了指定样品中 B2MtdTomato 位点的甲基化状态。每个堆积中代表三个重复;使用高斯窗口对齐读数的堆积进行平滑处理。该图改编自 Amabile et al.30。缩写:TSS = 转录起始位点。请点击这里查看此图的较大版本.
讨论
靶向表观遗传沉默可能代表了一种很有前途的解决方案,可以治疗可以从永久性基因失活中受益的疾病,包括由功能获得性突变引起的疾病 1、传染病2 以及一种基因沉默可以补偿另一个基因的遗传缺陷3 或释放过继细胞疗法的全部潜力4,5.通过在染色质水平上起作用并由细胞 7,30,32 自动繁殖,表观遗传沉默可以避免靶基因 DNA 序列的毒性改变(例如,染色体重排)和靶基因的部分、瞬时沉默,这分别是基于人工核酸酶的基因破坏8、34、35 和基于 RNAi 的敲低 33 的限制。
任何表观遗传沉默方案中的关键初步步骤之一是确定靶基因上的适当位置,以引导 ETR,这些 ETR 将沉积关闭靶标转录活性所需的抑制性表观遗传标记。不同的转录起始位点近端和远端调控元件可能同时支持给定人类基因的转录输出54。此外,由于越来越多的可编程 DNA 结合技术,现在可以识别每个特定调控元件的不同靶位点6。因此,在开始对最终治疗环境中的最佳候选药物进行繁琐且耗时的评估工作之前,可以使用方案(例如本文在工程细胞系中描述的方案)来指定适合ETR介导的表观遗传沉默的单个靶位点和/或基因组区域。该协议的一些关键方面将在下面进一步描述。
预测最终治疗靶点的报告细胞系工程
尽管细胞工程方案不断优化,需要在靶基因中插入荧光报告基因编码的盒并递送ETR,但不能想当然地认为它们可能已经可用于最接近最终治疗靶点的细胞系。在这种情况下,可以应用不同的缓解策略:a)利用供应商提供的优化试剂盒来优化目标细胞系的内部转染方案;b)切换到仍在表达该靶基因但属于最终治疗靶标以外的组织的其他细胞系 - 工程方案已被广泛优化。如果这些选项不可用,可以考虑切换到代表最终靶标的原代细胞类型或类器官。作为基于ETR的表观遗传沉默的临床前研究和治疗应用的一般考虑因素,应摒弃靶基因对靶细胞类型至关重要的情况。随着时间的流逝,ETRs强加的必需基因的完全、长期沉默将导致靶细胞的反选择(以及潜在的治疗毒性)。在这些情况下,首选提供部分靶标消除的替代技术,例如 RNAi33。
评估ETR的靶向沉默活性
基于 KRAB、DNMT3A 和 DNMT3L 效应结构域组合的 ETR 已被证明对绝大多数蛋白质编码基因有效,其广泛的 1 kb碱基长许可靶向窗口以转录起始位点32 为中心。为了了解表现良好的表观遗传沉默实验的外观,这里提供了在K-562细胞中沉默 B2M 基因的技术细节和结果。这可以被认为是一个重要的阳性对照,不仅被研究K-562细胞的研究人员包括在内,而且被那些首次接近基于ETR的技术的研究人员所包括。如方案所述,推荐使用基于人工核酸酶(例如CRISPR / Cas9)的基因破坏作为对目标细胞类型中基因递送效率和被剥夺靶基因的细胞表型的额外控制。在与基于 CRISPR/dCas9 的 ETR 偶联的 gRNA 进行初始筛选后,如果测试的 gRNA 都不能永久沉默靶基因,则应按以下顺序考虑:1) 增加递送的 gRNA 和 ETR 的数量;2)测试顶级gRNA的池,寻找它们之间的协同效应。如果仍未实现长期沉默,则应考虑:3)测试额外的gRNA,这些gRNA可能靶向与指导表观遗传沉默更相关的位点;4)切换到基于ZFP8或TALE9的DBD平台,其与靶染色质的结合能力可能更高;5)从瞬时切换到稳定,例如,整合基于病毒载体的ETR表达(采用CRISPR/dCas9技术时,gRNA或dCas9融合构建体或两者兼而有之)。由于我们小组开发了三个单独的 ETR30 的共同交付,并且拥有丰富的经验,因此此处显示的协议和结果都基于这种方法。然而,类似的概念工作流程可能适用于基于CRISPR的多合一系统32。
评估ETR的脱靶活性
多项研究表明,基于 KRAB、DNMT3A 和 DNMT3L 效应结构域组合的 ETR 特异性初步体外适应症30,31,32。然而,如果在测试的 gRNA 中,它们都没有在转录调控和/或从头 DNA 甲基化方面显示出令人满意的特异性特征,则可以采用两种非相互排斥的策略:a) 通过降低 ETR 的剂量或测试替代递送系统来减少 ETR 在细胞内的停留时间(从而减少它们潜在的脱靶活性)。例如,与质粒相比,mRNA 和蛋白质递送都有望减少细胞暴露于 ETR 的时间,从而减少脱靶活性的可能性55;b) 切换到更新的 Cas9 变体,优化以减少平台56 的脱靶结合,或替代基于 ZFP8 或 TALE9 的 DNA 结合技术。重要的是要考虑到,与模拟处理的样本相比,基因沉默的靶向和脱靶活性不仅受到DBD与其靶序列结合的影响,还受到表观遗传效应结构域的潜在能力的影响,这些能力被其天然的内源性辅因子募集到其他位点。因此,减少 ETR 在靶细胞中的停留时间不仅可以降低 DBD 与脱靶位点结合的可能性,还可以降低 ETR 与内源性辅因子相互作用的可能性,在特异性方面具有潜在的优势,在靶向活性方面具有劣势。最后,与模拟处理的样本相比,在沉默细胞中测量到的一些转录和不太可能的 CpG 甲基化改变可以简单地通过剥夺靶基因来得出。这些不被视为消音技术的偏离目标。为了识别它们,还应在实验面板8、9、10 中包括人工核酸酶的基因破坏。由于靶基因功能丧失而导致的生物学改变将在表观遗传沉默和这种替代技术之间共享。
披露声明
AL 是 Chroma Medicine, Inc. 的联合创始人、配额持有者和顾问。
致谢
作者要感谢 Angelo Amabile、Paola Capasso、Ilaria Caserta、Tania Baccega、Alice Reschigna、Valeria Mollica 和 Deborah Cipria 多年来在开发表观遗传沉默技术方面的合作努力;Dejan Lazarevic 和 Francesca Giannese 对协议中描述的 RNA-seq 和 MeDIP-seq 分析进行了严格审查。这项工作得到了Telethon基金会(TIGET资助号F1)和欧盟地平线2020计划(UPGRADE)对A.L.的资助。插图是用 BioRender.com 创作的。
作者贡献
A.M、M.A.C.、F.G. 和 A.C. 为协议的设计和撰写手稿做出了贡献;S.V.、I.M. 和 D.C. 设计了协议的生物信息学部分并修订了手稿;A.M.和A.L.设计了协议,构思并撰写了手稿,并听取了所有作者的意见。
材料
| Name | Company | Catalog Number | Comments |
| 4200 TapeStation System | Agilent | G2991BA | DNA quantification |
| 4D-Nucleofector X Unit | Lonza Bioscience | AAF-1003X | Nucleofection |
| B2M silencing gRNA #1 | Lombardo's lab | GCAATCAGGACAAGGCCCGC | Gene silencing |
| B2M silencing gRNA #2 | Lombardo's lab | GGGGTAGGAGAGACTCACGC | Gene silencing |
| B2M silencing gRNA #3 | Lombardo's lab | GAGTCCAGGGCTGGATCTCG | Gene silencing |
| BD FACSAria Fusion Flow Cytometer | BD Biosciences | https://www.bdbiosciences.com/en-us/products/instruments/flow-cytometers/research-cell-sorters/bd-facsaria-fusion | Fluorescence Activated Cell Sorting |
| bedtools | Bedtools | http://bedtools.readthedocs.io/en/latest/ | Processing of genomic intervals |
| bwa | Ih3 | https://github.com/lh3/bwa | Alignment of MeDIP-seq reads |
| Chopchop | Valen's lab | http://chopchop.cbu.uib.no/ | gRNA selection software |
| Corning RPMI 1640 Medium (Mod.) 1x with L-Glutamine | Corning | 10-040-CV | Cell culture |
| cqn | Bioconductor | http://bioconductor.org/packages/release/bioc/html/cqn.html | Region-wise normalization by GC-content |
| CRISPR design suite | Zhang's lab | https://zlab.bio/resources-2 | off-target gRNA binding prediction |
| CRISPRoff-v2.1 plasmid | Addgene | 167981 | Gene silencing |
| CytoFLEX S V4-B4-R3-I2 Flow Cytometer | Beckman Coulter | C01161 | Flow cytometry |
| Donor template sequence for tdTomato integration in the first intron of the B2M gene | Lombardo's lab | ctcctcctctgacctgtgtgtgggttttgtttttgtttt | Genetic engineering |
| E220 Focused-ultrasonicator | Covaris | 500239 | DNA sonication |
| edgeR | Bioconductor | https://bioconductor.org/packages/release/bioc/html/edgeR.html | Differential abundance testing |
| EnGen Mutation Detection Kit | NEB | E3321 | Gene disruption quantification |
| Fetal Bovine Serum | Sigma-Aldrich | F2442 | Cell culture |
| Flowjo | BD Biosciences | https://www.flowjo.com/solutions/flowjo | Flow cytometry data analysis software |
| Gel Loading Dye, Purple (6x) | NEB | B7024S | DNA gel loading |
| Go Taq G2 Hot Start DNA Polymerase | Promega | M7401 | PCR amplification |
| gRNA sequence for dTomato integration in the first intron of the B2M gene | Lombardo's lab | AGGCTACTAGCCCCATCAAG | Genetic engineering |
| hCas9 plasmid | Addgene | 41815 | Genetic engineering |
| High Sensitivity D1000 Reagents | Agilent | 5067-5585 | DNA quantification |
| High Sensitivity D1000 ScreenTape | Agilent | 5067-5584 | DNA quantification |
| High Sensitivity RNA ScreenTape | Agilent | 5067-5579 | RNA quantification |
| High Sensitivity RNA ScreenTape Ladder | Agilent | 5067-5581 | RNA quantification |
| High Sensitivity RNA ScreenTape Sample Buffer | Agilent | 5067-5580 | RNA quantification |
| IPure kit | Diagenode | C03010011 | Purification of the 5-methylcytosine immunoprecipitation product |
| K-562 cells | ATCC | CCL-243 | Cell engineering |
| KAPA Library Quantification Kit | Roche | KK4824 | MeDIP-Seq libraries preparation |
| MACS2 | Taoliu | https://github.com/macs3-project/MACS | Identification of methyl-enriched regions |
| MagMeDIP kit | Diagenode | C02010020 | 5-methylcytosine immunoprecipitation |
| NextFlex Methylseq kit 1 | Bioo Scientific | 5118-01 | MeDIP-Seq libraries preparation |
| NextSeq 500 / NovaSeq 6000 | Illumina | SY-415-1002 / 20012850 | Next Generation Sequencing |
| NucleoBond Xtra Midi kit for transfection-grade plasmid DNA | Macherey-nagel | 740410.50 | Midiprep plasmid preparation |
| One Shot TOP10 Chemically Competent cells | ThermoFisher | C404010 | Plasmid transformation |
| pAC154-dual-dCas9VP160-sgExpression plasmid | Addgene | 48240 | Gene activation |
| pcDNA.CMV.dCas9:KRAB plasmid | Lombardo's lab | available on request (lombardo.angelo@hsr.it) | Gene silencing |
| pcDNA.CMV.dCas9:KRAB plasmid | Lombardo's lab | available on request (lombardo.angelo@hsr.it) | Gene silencing |
| pcDNA.CMV.dCas9:KRAB plasmid | Lombardo's lab | available on request (lombardo.angelo@hsr.it) | Gene silencing |
| Penicillin/Streptomycin | Sigma-Aldrich | P0781 | Cell culture |
| phU6.sgRNA plasmid | Addgene | 53188 | Genetic engineering |
| PowerPac Basic Power Supply | Biorad | 1645050 | Agarose gel electrophoresis |
| Primer forward sequence to check tdTomato integration in the first intron of the B2M gene | Lombardo's lab | GTATTTGCTGGTTATGTTAG | Genetic engineering |
| Primer reverse sequence to check tdTomato integration in the first intron of the B2M gene | Lombardo's lab | AATGGTTGAGTTGGAC | Genetic engineering |
| QIAamp DNA Mini Kit | Qiagen | 51304 | DNA extraction |
| Qubit | Thermo Fisher | Q33238 | DNA quantification |
| Qubit dsDNA HS (high sensitivity) Assay Kit | Thermo Fisher | Q32851 | DNA quantification |
| Qubit RNA HS (high sensitivity) Assay Kit | Thermo Fisher | Q32852 | RNA quantification |
| Restriction enzymes | NEB | DNA digestion | |
| RNeasy Mini kit | Qiagen | 74106 | RNA extraction |
| Rsubread | Bioconductor | https://bioconductor.org/packages/release/bioc/html/Rsubread.html | Quantification of gene expression |
| SF Cell Line 4D-Nucleofector X Kit S (32 RCT) | Lonza Bioscience | V4XC-2032 | Nucleofection |
| SnapGene | Dotmatics | https://www.snapgene.com/ | Molecular biology design software |
| STAR | Alexander Dobin | https://github.com/alexdobin/STAR | Alignment of RNA-seq reads |
| T100 Thermal Cycler | Biorad | 1861096 | PCR amplification |
| T4 DNA Ligase | Promega | M1801 | DNA ligation |
| TAE Buffer | Fisher scientific | BP1332500 | Agarose gel electrophoresis |
| TruSeq Stranded Total RNA kit | Illumina | 20020597 | RNA-Seq library preparation |
| UltraPure Agarose | ThermoFisher | 16500500 | Agarose gel |
参考文献
- Cummings, C. J., Zoghbi, H. Y. Fourteen and counting: unraveling trinucleotide repeat diseases. Human Molecular Genetics. 9 (6), 909-916 (2000).
- Tebas, P., et al. Gene editing of CCR5 in autologous CD4 T cells of persons infected with HIV. The New England Journal of Medicine. 370 (10), 901-910 (2014).
- Frangoul, H., et al. CRISPR-Cas9 gene editing for sickle cell disease and β-thalassemia. The New England Journal of Medicine. 384 (3), 252-260 (2021).
- Murty, T., Gene Mackall, C. L. Gene editing to enhance the efficacy of cancer cell therapies. Molecular Therapy. 29 (11), 3153-3162 (2021).
- Lanza, R., Russell, D. W., Nagy, A. Engineering universal cells that evade immune detection. Nature Reviews Immunology. 19 (12), 723-733 (2019).
- Matharu, N., Ahituv, N. Modulating gene regulation to treat genetic disorders. Nature Reviews Drug Discovery. 19 (11), 757-775 (2020).
- Sgro, A., Blancafort, P. Epigenome engineering: New technologies for precision medicine. Nucleic Acids Research. 48 (22), 12453-12482 (2020).
- Urnov, F. D., Rebar, E. J., Holmes, M. C., Zhang, H. S., Gregory, P. D. Genome editing with engineered zinc finger nucleases. Nature Reviews Genetics. 11 (9), 636-646 (2010).
- Joung, J. K., Sander, J. D. TALENs: A widely applicable technology for targeted genome editing. Nature Reviews Molecular Cell Biology. 14 (1), 49-55 (2013).
- Adli, M. The CRISPR tool kit for genome editing and beyond. Nature Communications. 9 (1), 1911(2018).
- Gilbert, L. A., et al. XCRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes. Cell. 154 (2), 442(2013).
- Snowden, A. W., Gregory, P. D., Case, C. C., Pabo, C. O. Gene-specific targeting of H3K9 methylation is sufficient for initiating repression in vivo. Current Biology. 12 (24), 2159-2166 (2002).
- Chen, X., et al. Construction and validation of the CRISPR/dCas9-EZH2 system for targeted H3K27Me3 modification. Biochemical and Biophysical Research Communications. 511 (2), 246-252 (2019).
- Kwon, D. Y., Zhao, Y. T., Lamonica, J. M., Zhou, Z. Locus-specific histone deacetylation using a synthetic CRISPR-Cas9-based HDAC. Nature Communications. 8, 15215(2017).
- Stepper, P., et al. Efficient targeted DNA methylation with chimeric dCas9-Dnmt3a-Dnmt3L methyltransferase. Nucleic Acids Research. 45 (4), 1703-1713 (2017).
- Ecco, G., Imbeault, M., Trono, D. KRAB zinc finger proteins. Development. 144 (15), 2719-2729 (2017).
- Witzgall, R., O'leary, E., Leaf, A., Onaldi, D., Bonventre, J. The Kruppel-associated box-A (KRAB-A) domain of zinc finger proteins mediates transcriptional repression. Proceedings of the National Academy of Sciences. 91 (10), 4514-4518 (1994).
- Mannini, R., et al. Structure/function of KRAB repression domains: Structural properties of KRAB modules inferred from hydrodynamic, circular dichroism, and FTIR spectroscopic analyses. Proteins: Structure, Function and Genetics. 62 (3), 604-616 (2006).
- Friedman, J. R., et al. KAP-1, a novel corepressor for the highly conserved KRAB repression domain. Genes and Development. 10 (16), 2067-2078 (1996).
- Iyengar, S., Farnham, P. J. KAP1 protein: An enigmatic master regulator of the genome. The Journal of Biological Chemistry. 286 (30), 26267-26276 (2011).
- Schultz, D. C., Friedman, J. R., Rauscher, F. J. Targeting histone deacetylase complexes via KRAB-zinc finger proteins: The PHD and bromodomains of KAP-1 form a cooperative unit that recruits a novel isoform of the Mi-2α subunit of NuRD. Genes and Development. 15 (4), 428-443 (2001).
- Schultz, D. C., Ayyanathan, K., Negorev, D., Maul, G. G., Rauscher, F. J. SETDB1: A novel KAP-1-associated histone H3, lysine 9-specific methyltransferase that contributes to HP1-mediated silencing of euchromatic genes by KRAB zinc-finger proteins. Genes and Development. 16 (8), 919-932 (2002).
- Nielsen, A. L., et al. Interaction with members of the heterochromatin protein 1 (HP1) family and histone deacetylation are differentially involved in transcriptional silencing by members of the TIF1 family. The EMBO Journal. 18 (22), 6385-6395 (1999).
- Sripathy, S. P., Stevens, J., Schultz, D. C. The KAP1 corepressor functions to coordinate the assembly of de novo HP1-demarcated microenvironments of heterochromatin required for KRAB zinc finger protein-mediated transcriptional repression. Molecular and Cellular Biology. 26 (22), 8623-8638 (2006).
- Jurkowska, R. Z., Jurkowski, T. P., Jeltsch, A. Structure and function of mammalian DNA methyltransferases. ChemBioChem. 12 (2), 206-222 (2011).
- Jia, D., Jurkowska, R. Z., Zhang, X., Jeltsch, A., Cheng, X. Structure of Dnmt3a bound to Dnmt3L suggests a model for de novo DNA methylation. Nature. 449 (7159), 248-251 (2007).
- Tajima, S., Suetake, I., Takeshita, K., Nakagawa, A., Kimura, H. Domain structure of the Dnmt1, Dnmt3a, and Dnmt3b DNA methyltransferases. Advances in Experimental Medicine and Biology. 945, 63-86 (2016).
- Greenberg, M. V. C., Bourc'his, D. The diverse roles of DNA methylation in mammalian development and disease. Nature Reviews Molecular Cell Biology. 20 (10), 590-607 (2019).
- Ishiyama, S., et al. Structure of the Dnmt1 reader module complexed with a unique two-mono-ubiquitin mark on histone H3 reveals the basis for DNA methylation maintenance. Molecular Cell. 68 (2), 350.e7-360.e7 (2017).
- Amabile, A., et al. Inheritable silencing of endogenous genes by hit-and-run targeted epigenetic editing. Cell. 167 (1), 219.e14-232.e14 (2016).
- Mlambo, T., et al. Designer epigenome modifiers enable robust and sustained gene silencing in clinically relevant human cells. Nucleic Acids Research. 46 (9), 4456-4468 (2018).
- Nuñez, J. K., et al. Genome-wide programmable transcriptional memory by CRISPR-based epigenome editing. Cell. 184 (9), 2503.e17-2519.e17 (2021).
- Davidson, B. L., McCray, P. B. Current prospects for RNA interference-based therapies. Nature Reviews Genetics. 12 (5), 329-340 (2011).
- Haapaniemi, E., Botla, S., Persson, J., Schmierer, B., Taipale, J. CRISPR-Cas9 genome editing induces a p53-mediated DNA damage response. Nature Medicine. 24 (7), 927-930 (2018).
- Kosicki, M., Tomberg, K., Bradley, A. Repair of double-strand breaks induced by CRISPR-Cas9 leads to large deletions and complex rearrangements. Nature Biotechnology. 36 (8), 765-771 (2018).
- Ciccia, A., Elledge, S. J. The DNA damage response: making it safe to play with knives. Molecular Cell. 40 (2), 179-204 (2010).
- Hu, J. H., et al. Evolved Cas9 variants with broad PAM compatibility and high DNA specificity. Nature. 556 (7699), 57-63 (2018).
- Uhlén, M., et al. Proteomics. Tissue-based map of the human proteome. Science. 347 (6220), (2015).
- Labun, K., et al. CHOPCHOP v3: Expanding the CRISPR web toolbox beyond genome editing. Nucleic Acids Research. 47 (W1), W171-W174 (2019).
- Kent, W. J., et al. The Human Genome Browser at UCSC. Genome Research. 12 (6), 996-1006 (2002).
- Cheng, A. W., et al. Multiplexed activation of endogenous genes by CRISPR-on, an RNA-guided transcriptional activator system. Cell Research. 23 (10), 1163-1171 (2013).
- Kabadi, A. M., Ousterout, D. G., Hilton, I. B., Gersbach, C. A. Multiplex CRISPR/Cas9-based genome engineering from a single lentiviral vector. Nucleic Acids Research. 42 (19), e147(2014).
- Dobin, A., et al. STAR: ultrafast universal RNA-seq aligner. Bioinformatics. 29 (1), 15-21 (2013).
- Liao, Y., Smyth, G. K., Shi, W. The Subread aligner: fast, accurate and scalable read mapping by seed-and-vote. Nucleic Acids Research. 41 (10), e108-e108 (2013).
- Conesa, A., et al. A survey of best practices for RNA-seq data analysis. GenomeBiology. 17, 13(2016).
- Robinson, M. D., Oshlack, A. A scaling normalization method for differential expression analysis of RNA-seq data. Genome Biology. 11 (3), R25(2010).
- Robinson, M. D., McCarthy, D. J., Smyth, G. K. edgeR: a Bioconductor package for differential expression analysis of digital gene expression data. Bioinformatics. 26 (1), 139-140 (2010).
- Li, H., Durbin, R. Fast and accurate long-read alignment with Burrows-Wheeler transform. Bioinformatics. 26 (5), 589-595 (2010).
- Zhang, Y., et al. Model-based analysis of ChIP-Seq (MACS). Genome Biology. 9 (9), R137(2008).
- Quinlan, A. R. BEDTools: The Swiss-Army tool for genome feature analysis. Current Protocols in Bioinformatics. 47, (2014).
- Hansen, K. D., Irizarry, R. A., Wu, Z. Removing technical variability in RNA-seq data using conditional quantile normalization. Biostatistics. 13 (2), 204-216 (2012).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nature Biotechnology. 31 (9), 827-832 (2013).
- Zeitler, B., et al. Allele-selective transcriptional repression of mutant HTT for the treatment of Huntington's disease. Nature Medicine. 25 (7), 1131-1142 (2019).
- Andersson, R., Sandelin, A. Determinants of enhancer and promoter activities of regulatory elements. Nature Reviews Genetics. 21 (2), 71-87 (2020).
- Liang, X., et al. Rapid and highly efficient mammalian cell engineering via Cas9 protein transfection. Journal of Biotechnology. 208, 44-53 (2015).
- Han, H. A., Pang, J. K. S., Soh, B. -S. Mitigating off-target effects in CRISPR/Cas9-mediated in vivo gene editing. Journal of Molecular Medicine. 98 (5), 615-632 (2020).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。