Method Article
In vitro Sélection de répresseurs transcriptionnels modifiés pour le silençage épigénétique ciblé
Dans cet article
Résumé
Nous présentons ici un protocole pour la sélection in vitro de répresseurs transcriptionnels modifiés (ETR) avec une efficacité de silençage élevée, à long terme, stable et sur cible et une faible activité hors cible à l’échelle du génome. Ce flux de travail permet de réduire un répertoire initial et complexe de RTE candidats à une liste restreinte, adaptée à une évaluation plus approfondie dans des contextes thérapeutiquement pertinents.
Résumé
L’inactivation génique joue un rôle déterminant dans l’étude de la fonction des gènes et représente une stratégie prometteuse pour le traitement d’un large éventail de maladies. Parmi les technologies traditionnelles, l’interférence de l’ARN souffre de l’abrogation partielle de la cible et de la nécessité de traitements à vie. En revanche, les nucléases artificielles peuvent imposer une inactivation génique stable par induction d’une cassure double brin de l’ADN (DSB), mais des études récentes remettent en question l’innocuité de cette approche. L’édition épigénétique ciblée via des répresseurs transcriptionnels modifiés (ETR) peut représenter une solution, car une seule administration de combinaisons spécifiques d’ETR peut conduire à un silence durable sans induire de cassures de l’ADN.
Les ETR sont des protéines contenant un domaine de liaison à l’ADN programmable (DBD) et des effecteurs de répresseurs transcriptionnels naturels. Plus précisément, il a été démontré qu’une combinaison de trois ETR équipés du domaine KRAB du ZNF10 humain, du domaine catalytique du DNMT3A humain et du DNMT3L humain, induit des états épigénétiques répressifs héréditaires sur le gène cible de l’ETR. La nature aléatoire de cette plate-forme, l’absence d’impact sur la séquence d’ADN de la cible et la possibilité de revenir à l’état répressif par déméthylation de l’ADN à la demande, font du silençage épigénétique un outil qui change la donne. Une étape cruciale est l’identification de la position appropriée des ETR sur le gène cible afin de maximiser la ciblage et de minimiser le silençage hors cible. La réalisation de cette étape dans le cadre préclinique final ex vivo ou in vivo peut s’avérer fastidieuse.
En prenant le système CRISPR/Cas9 en état de mort catalytique comme DBD paradigmatique pour les ETR, cet article décrit un protocole consistant en un criblage in vitro d’ARN guides (ARNg) couplé à la combinaison triple-ETR pour un silençage efficace sur la cible, suivi d’une évaluation du profil de spécificité à l’échelle du génome des meilleurs résultats. Cela permet de réduire le répertoire initial des ARNg candidats à une courte liste d’ARNg prometteurs, dont la complexité est adaptée à leur évaluation finale dans le contexte thérapeutiquement pertinent d’intérêt.
Introduction
L’inactivation génique a traditionnellement joué un rôle clé dans l’étude de la fonction des gènes dans les modèles cellulaires et animaux. De plus, au cours des deux dernières décennies, avec l’essor de la thérapie génique, elle a été proposée comme une approche potentiellement révolutionnaire pour traiter les maladies causées par des mutations de gain de fonction1, les maladies infectieuses2 ou les pathologies dans lesquelles le silence d’un gène peut compenser un défaut héréditaire dans un autre3. Enfin, l’inactivation génétique des principaux régulateurs de la fitness cellulaire et du contrôle fonctionnel a été proposée pour améliorer l’efficacité des produits cellulaires pour l’immunothérapie du cancer4 et la médecine régénérative5.
Parmi les différentes technologies permettant d’accomplir l’inactivation des gènes, l’une des plus prometteuses est le silençage épigénétique ciblé 6,7. Au cœur de cette technologie se trouvent les répresseurs transcriptionnels modifiés (ETR), des protéines chimériques constituées d’un domaine de liaison à l’ADN programmable (DBD) et d’un domaine effecteur (ED) ayant une fonction répressive épigénétique. Les protéines à doigt de zinc (ZFP)8, les effecteurs de type activateur de transcription (TALE)9 ou les DBD à base de CRISPR/dCas910 peuvent être conçus pour attacher sélectivement la dysfonction érectile à la séquence promoteur/amplificateur du gène cible à réduire au silence. Une fois sur place, l’ED de l’ETR exerce son activité de silençage en imposant des marques épigénétiques répressives induisant l’hétérochromatine telles que les modifications des histones (méthylation H3K9 11,12 ou H3K27 13, désacétylation H3 ou H4 14) et la méthylation de l’ADN CpG 15, selon le domaine répressif utilisé.
En particulier, en s’inspirant des processus moléculaires de répression transcriptionnelle permanente des rétrovirus endogènes survenant dans l’embryonpréimplantatoire 16, une combinaison de trois ETR a été générée pour exploiter les ED suivantes : i) le domaine de la boîte associée à Krüppel (KRAB) du ZNF10 humain ; ii) le domaine catalytique de l’ADN méthyltransférase 3A humaine de novo (DNMT3A) ; et iii) l’ADN méthyltransférase 3-like humain pleine longueur (DNMT3L). KRAB est un domaine répressif conservé partagé par plusieurs ZFPs chez les vertébrés supérieurs17,18, dont l’activité de silençage repose principalement sur sa capacité à recruter KAP1 19-une protéine d’échafaudage qui interagit ensuite avec plusieurs autres inducteurs d’hétérochromatine20-comprenant le complexe de remodelage et de désacétylation des nucléosomes (NuRD) 21, l’histone méthyltransférase H3K9 SETDB122, et le lecteur de méthylation H3K9 HP1 23, 24, entre autres.
DNMT3A transfère activement les groupes méthyle sur l’ADN au niveau des séquences CpG25. L’activité catalytique de DNMT3A est renforcée par son association physique avec DNMT3L, un paralogue de DNMT3A restreint aux cellules embryonnaires et germinales dépourvu du domaine catalytique responsable du transfert du groupe méthyle26,27. La méthylation de l’ADN dans les régions riches en CpG - appelées îlots CpG (CGI) - intégrée dans les éléments promoteurs/amplificateurs des gènes des mammifères est généralement associée au silençage de la transcription28. Il est important de noter qu’une fois déposée, la méthylation de CpG peut être héritée de manière stable tout au long de la mitose par un complexe moléculaire à base d’UHRF1-DNMT129.
La surexpression stable des ETR dans la cellule cible peut être problématique, probablement en raison des risques croissants d’activité hors cible et d’étouffement des interacteurs endogènes de leurs sites cibles physiologiques au fil du temps. Cependant, l’expression transitoire de fractions ETR uniques peut ne pas induire de silençage à long terme avec une efficacité élevée30, ce qui entrave leur application thérapeutique. Par conséquent, une percée majeure dans le domaine a été la preuve que la combinaison des trois ETR basés sur KRAB-, DNMT3A et DNMT3L peut créer une synergie et, même lorsqu’elle n’est co-délivrée que transitoirement, s’imposer sur la séquence promotrice du gène cible H3K9 et la méthylation de CpG. Ceux-ci sont ensuite lus et propagés par la cellule tout au long de la mitose, conduisant à un silençage héréditaire dans de multiples lignées cellulaires humaines et murines, ainsi que dans des cellules primaires cultivées ex vivo 30.
Il convient de noter que le silençage épigénétique imposé par les ETR peut être inversé à la demande par une déméthylation de l’ADN30 ciblée (par exemple, le recrutement de l’ADN déméthylase TET1 à base de CRISPR/dCas9 sur le locus silencieux) ou pharmacologique (administration de l’inhibiteur de l’ADN méthyltransférase 5-Aza), un antidote potentiel en cas d’événements indésirables liés à l’ETR. Des ETR tout-en-un portant les trois EDs à base de KRAB, DNMT3A et DNMT3L ont également été décrits, montrant des efficacités significatives de silençage dans les lignées cellulaires31,32 contre la grande majorité des gènes codant pour des protéines. De plus, plusieurs études utilisant les ETR ont fait état d’un profil d’innocuité élevé, sans activité hors cible majeure en termes de méthylation de novo de CpG ou d’altération de l’accessibilité de la chromatine30,31,32. Cependant, une analyse spécifique du profil de spécificité des ETR équipés d’un DBD nouvellement conçu est recommandée avant les applications cliniques.
D’un point de vue clinique, le silençage épigénétique ciblé peut offrir des avantages essentiels à la fois à l’inhibition basée sur l’interférence de l’ARN (ARNi)33 et à la perturbation génique artificielle basée sur la nucléase8. Contrairement à l’ARNi, le silençage épigénétique ciblé peut induire l’abrogation complète de sa cible par cellule et ne nécessite pas de traitement périodique pour assurer un silence à long terme ; contrairement à la perturbation génétique, elle laisse la séquence d’ADN inaltérée, évitant ainsi la génération de cassures double brin de l’ADN (DSB). Les DSB peuvent alors induire l’apoptose et l’arrêt du cycle cellulaire, conduisant potentiellement à une sélection contre les cellules ayant une voie fonctionnelle p5334,35 et, en particulier dans les contextes d’édition de gènes multiplex, à des réarrangements chromosomiques 35. De plus, en relayant le résultat irréversible de la mosaïque irréversible de la réparation de l’ADN DSB médiée par la jonction d’extrémitésnon homologues 36, la perturbation génique ne peut pas éviter la réparation dans le cadre de la cible en séquences codantes fonctionnelles comme l’un des résultats finaux et, contrairement au silençage épigénétique, ne peut pas être effacée à la demande.
Enfin, le silençage épigénétique a le potentiel d’élargir la gamme d’éléments génétiques ciblables à des classes entièrement ou au moins partiellement réfractaires à l’ARNi et à la perturbation des gènes, telles que les éléments régulateurs non transcrits et les ARN non codants30,32. La première étape critique de toute application de silençage épigénétique ciblé consiste à concevoir un panel d’ETR couvrant les différentes séquences régulatrices du gène cible et à identifier les plus performantes. Le nombre d’ETR à tester peut être crucial, compte tenu de la part croissante du génome qui peut être ciblée par les technologies de liaison à l’ADN programmables en cours de développement37. Effectuer le criblage des ETR directement sur le type de cellule dans lequel réduire au silence thérapeutiquement le gène cible représenterait l’option la plus pertinente. Cependant, les cribles à haut débit peuvent être techniquement encombrants dans les cellules primaires en raison de leur survie limitée en culture et de leur capacité d’ingénierie souvent sous-optimale. Les écrans à grande échelle peuvent être encore plus irréalisables in vivo.
Une alternative plus pratique consiste à effectuer un criblage initial d’un large panel d’ETR dans des lignées cellulaires facilement modifiables dans un premier temps, puis à ne valider que les plus prometteuses dans le type de cellule thérapeutiquement pertinent. Une question parallèle est le choix d’une lecture appropriée pour mesurer l’efficacité de l’insonorisation des ETR. L’évaluation directe des niveaux de transcription ou de protéines du gène cible par RT-qPCR, Western Blot ou ELISA peut être coûteuse et prendre beaucoup de temps et peut manquer d’une sensibilité suffisante, limitant ainsi leur application à des échelles à haut débit. La génération de lignées cellulaires rapporteures modifiées ad hoc dans lesquelles un fluorophore est placé sous le contrôle transcriptionnel des séquences régulatrices du gène cible permet d’exploiter l’approche basée sur la cytométrie en flux pour lire le silençage épigénétique au niveau de la cellule unique et à un rythme à haut débit.
À la suite de ces considérations générales, cet article décrit un protocole consistant en un criblage in vitro d’ETR en réseau pour l’efficacité du silençage sur cible, suivi d’une évaluation de l’activité hors cible à l’échelle du génome des meilleurs résultats. Ce flux de travail permet de réduire le répertoire initial des ETR candidats à une courte liste de candidats prometteurs, dont la complexité est adaptée à leur évaluation finale dans le type de cellule d’intérêt thérapeutiquement pertinent.
Parmi les différents DBD programmables qui peuvent être exploités pour générer des ETR, ce protocole se concentrera sur la technologie basée sur CRISPR/dCas9, en raison de la facilité de conception d’ARNg couvrant le promoteur du gène cible à une échelle à haut débit. Cependant, le même flux de travail conceptuel décrit ci-dessous peut être adopté pour évaluer l’efficacité et la spécificité des RTE équipés d’autres DBD.
Protocole
1. Ingénierie d’une lignée cellulaire rapporteure basée sur la fluorescence pour surveiller l’activité transcriptionnelle du gène cible par cytométrie en flux
- Identifier les lignées cellulaires exprimant le gène cible à réduire au silence. Parcourez le gène cible à réduire au silence dans l’Atlas des protéines humaines38 et naviguez dans sa section « Lignée cellulaire » pour identifier les lignées représentatives du tissu somatique d’intérêt (par exemple, une lignée cellulaire hépatique si les cibles finales sont des hépatocytes hépatiques). Vous pouvez également interroger une base de données de séquençage de l’ARN (RNA-Seq) accessible au public (p. ex., NCBI GEO).
- Parmi les candidats, donner la priorité aux lignées cellulaires pour lesquelles des protocoles efficaces d’administration de gènes transitoires - essentiels à l’administration de l’ETR - sont disponibles.
REMARQUE : Parmi les différentes modalités, la nucléofection représente l’une des meilleures options, car elle assure une efficacité de transfection élevée. Ici, des cellules d’érythroleukémie humaine K-562 ont été choisies pour générer une lignée cellulaire rapportant l’activité transcriptionnelle du gène bêta-2-microglobuline (B2M) (ci-après dénommé les cellules B2MTdTomato K-562). - Donnez davantage la priorité aux candidats pour éviter les lignées cellulaires dans lesquelles le gène cible est essentiel à la viabilité cellulaire, car cela nuit au maintien des cellules avec un silençage stable du gène cible en culture. S’il n’a pas déjà été signalé, pour avoir une idée de l’essentialité du gène cible dans le type de cellule choisi, générer un contrôle de perturbation génétique en transfectant les cellules avec la nucléase Cas9 et un ARNg ciblant l’un des premiers exons codants du gène.
REMARQUE : La perturbation génétique est un événement stable par définition ; La contre-sélection des cellules perturbées au fil du temps indique que le gène cible est essentiel à la physiologie de la cellule sélectionnée.- Identifier l’isoforme d’épissage cible préférentiellement utilisée dans la lignée cellulaire sélectionnée (l’isoforme NM_004048.4 du gène B2M est ciblée dans ce protocole).
- Identifier un ARNg qui peut se couper efficacement et spécifiquement dans le premier exon codant de l’isoforme cible (p. ex., Chopchop (http://chopchop.cbu.uib.no/)39, qui est un outil de sélection d’ARNg en ligne valide et convivial).
- Pour les cellules K-562, transfectez 1 μg de plasmide codant pourCas9 sp (hCas9 ; voir le tableau des matériaux) et 250 ng de plasmide codant pour l’ARNg (phU6-gRNA ; voir le tableau des matériaux) par 5 × 105 cellules par nucléofection (selon les instructions du fabricant).
- Cultivez les cellules (cellules K-562 à 37 °C sous 5 % de CO2 dans le RPMI-1640 complété par 10 % de sérum de veau fœtal [FBS], de L-glutamine et de pénicilline/streptomycine [100 U/mL]) et surveillez les niveaux de perturbation génique au fil du temps en exploitant un kit de détection de mutation (suivez les instructions du fabricant).
- Cloner un modèle de donneur pour l’intégration médiée par la recombinaison homologue d’un rapporteur fluorescent sous le contrôle transcriptionnel du gène cible d’intérêt (Figure 1).
- Identifier la région du gène cible pour intégrer la cassette d’expression du fluorophore.
NOTE : Évitez de cibler les éléments transcriptionnellement pertinents, tels que les îlots CpG et les régions enrichies pour l’acétylation de H3K27 (marqueur des promoteurs actifs et des activateurs). L’altération de ces éléments régulateurs (potentiellement importants pour être ciblés par les ETR afin d’instruire le silençage épigénétique) rend la lignée cellulaire rapporteure moins prédictive de la régulation physiologique du gène cible.- Si le gène cible ne code pas pour une protéine sécrétée, fusionner le rapporteur avec le dernier codon du gène cible par l’intermédiaire d’un peptide d’autoclivage 2A pour maintenir la fonctionnalité du gène cible.
- Si le gène cible code pour une protéine sécrétée, pour éviter la sécrétion potentielle du rapporteur, placez le rapporteur dans une région intronique du gène cible. L’intégration forcée du rapporteur dans le transcrit épissé par un site accepteur d’épissage (SA) et la traduction subséquente par une séquence d’entrée interne du ribosome (IRES) altèrent la fonctionnalité du gène cible.
- Utilisez Chopchop pour sélectionner la coupure de l’ARNg dans la région cible (ici, un ARNg est sélectionné pour cibler la séquence 5′-AGGCTACTAGCCCCATCAAGAGG-3′ du premier intron du gène B2M ).
- Concevoir un modèle donneur pour le site de coupure de l’ARNg composé de : i) un bras d’homologie gauche (n paires de bases [pb] correspondant à la région juste en amont du site de coupure de l’ARNg) ; ii) une cassette d’expression transgénique sans promoteur (dans le cas présenté ici, un poly(A) SA-3X Stop Codon-IRES-tdTomato-BGH favorisant l’épissage avec le premier intron du gène B2M ) ; et iii) un bras d’homologie droite (n pb correspondant à la région en aval du site de coupure de l’ARNg).
REMARQUE : La longueur des bras d’homologie nécessaires pour induire efficacement une recombinaison homologue peut varier selon les différents types de cellules (100-500 pb est une plage appropriée pour les cellules K-562).
- Identifier la région du gène cible pour intégrer la cassette d’expression du fluorophore.
- Délivrer le système de nucléase CRISPR/Cas9 et le modèle donneur à l’intérieur de la lignée cellulaire cible. Pour les cellules K-562, transfectez 1 μg de plasmide codantpour Cas9 sp (hCas9 ; voir le tableau des matériaux), 250 ng de plasmide codant pour l’ARNg (phU6-gRNA ; voir le tableau des matériaux) et 1 μg de plasmide codant pour la matrice du donneur par 5 × 105 cellules par nucléofection (selon les instructions du fabricant).
- Cultivez les cellules pendant au moins 14 jours (pour les cellules K-562) et surveillez les niveaux d’expression du rapporteur fluorescent au fil du temps à l’aide d’un cytomètre en flux (activez le canal phycoérythrine (PE) pour mesurer l’intensité de fluorescence du rapporteur tdTomato et suivez les instructions du fabricant pour effectuer une cytométrie en flux).
NOTE : Le modèle du donneur, en particulier s’il est à base de plasmide, peut contenir des séquences de promoteurs cryptiques, conduisant à l’expression du rapporteur fluorescent à partir de copies de donneurs non intégrées. La culture des cellules permet de diluer ces copies non intégrées par division cellulaire et finalement de maintenir l’expression du rapporteur uniquement à partir des copies donneuses intégrées dans le génome cible. - Cloner des cellules rapporteures positives par tri cellulaire activé par fluorescence (FACS) au niveau d’une seule cellule. Pour ce protocole, activez le canal PE pour mesurer l’intensité de fluorescence du rapporteur tdTomato et suivez les instructions du fabricant pour trier les cellules K-562 tdTomato-positives par puits d’une plaque à 96 puits.
- Lors de l’expansion cellulaire en culture (généralement 20 à 30 jours pour les cellules K-562), cribler les clones rapporteurs positifs par PCR pour en sélectionner un porteur portant une intégration bi-allélique de la cassette rapporteur à l’intérieur du locus cible.
REMARQUE : Cela maximise l’expression du rapporteur et facilite la résolution entre les cellules exprimant le rapporteur et les cellules silencieuses du rapporteur par cytométrie en flux lors du traitement ETR.- Extraire l’ADN génomique de 1 × 105 cellules par clone rapporteur-positif à l’aide d’un kit d’extraction d’ADN (en suivant les instructions du fabricant).
- Amplifiez la région cible B2M avec des amorces directes (5'-GTATTTGCTGGTTATGTTAG-3') et inverses (5'-AATGGTTGAGTTGGAC-3') en suivant les instructions du kit d’amplification PCR. La température de recuit de cette paire d’amorces est de 47,7 °C avec des ADN polymérases à base de Taq et des concentrations d’amorces de 0,5 μM.
- Analyser le produit PCR à l’aide d’une électrophorèse sur gel d’agarose à 1 % (en suivant les instructions du fabricant). Criblage des clones montrant la bande liée à l’intégration de la tdTomato dans le locus cible B2M (3 413 pb), sans la bande liée au locus cible de type sauvage (1 027 pb).
2. Conception d’ARNg pour le silençage épigénétique du gène cible basé sur CRISPR/dCas 9
- Parcourez le gène cible dans le navigateur génomique40 de l’UCSC et extrayez la séquence nucléotidique des régions régulant potentiellement son activité transcriptionnelle, telles que les îlots CpG et les sites enrichis pour l’acétylation de H3K27 (marqueur des promoteurs et enhancers actifs).
NOTE : Selon une étude récente, la meilleure région de ciblage est une fenêtre de 1 kilobase (kb) centrée sur le site de départ de la transcription du gènecible 32. - Collez les séquences sélectionnées dans l’outil en ligne Chopchop et sélectionnez la répression comme objectif des ARNg à récupérer. Attendez que Chopchop fournisse une liste d’ARNg cartographiés sur la séquence génétique d’intérêt et répertoriés en fonction d’un score tenant compte à la fois du nombre de correspondances hors cible et de l’efficacité prédite sur la cible (Figure 2).
- Sélectionnez au moins 10 ARNg par séquence cible. Si possible, essayez de sélectionner des ARNg couvrant toute la région à interroger, sans correspondance complète avec d’autres séquences intragéniques dans tout le génome.
3. Distribution transitoire en réseau d’ETR basés sur CRISPR/dCas 9 dans la lignée cellulaire rapporteure
- Parmi les systèmes d’administration de transgènes précédemment signalés pour la lignée cellulaire cible, choisissez ceux qui ne permettent que l’expression transitoire du transgène, maximisant l’efficacité de l’administration et minimisant la toxicité liée à la manipulation cellulaire. Dans le cas des cellules K-562, la nucléofection plasmidique et l’ARNm sont fortement recommandées, la production de plasmides représentant une alternative techniquement plus facile et moins chère.
- Cloner à la fois les ARNg sélectionnés dans la section 2 et les ETR basés sur CRISPR/dCas9 dans le système d’administration de transgènes de choix. Reportez-vous aux étapes suivantes pour connaître le protocole de clonage des ETR dans l’ADN plasmidique.
REMARQUE : Les plasmides codant séparément pour dCas9 :KRAB, dCas9 :DNMT3A et dCas9 :DNMT3L30 ne sont pas disponibles sur Addgene. Ils ont été clonés en remplaçant le trans-activateur VP160 du plasmide pAC154-dual-dCas9VP160-sgExpression41 (voir le tableau des matériaux) par la séquence de codage de domaine30 KRAB, DNMT3A ou DNMT3L. Un plasmide codant pour un ETR tout-en-un, appelé CRISPRoff-v2.132, est disponible (voir Tableau des matériaux).- Transformer les plasmides codant pour les ETR dans les cellules E. coli chimiquement compétentes (en suivant les instructions du fabricant). Criblez les colonies pour la présence du plasmide contenant l’ETR par digestion enzymatique de restriction et séquençage de Sanger, et enfin choisissez l’une des colonies positives pour la production d’ADN plasmidique Midiprep (en suivant les instructions du fabricant).
- Clonez les ARNg à l’intérieur du squelette de l’ARNg phU6 (Figure 3).
- À l’aide d’un logiciel de conception de biologie moléculaire, ajouter une séquence 5'-CACCG-3' en amont du protoespaceur (la première variable de 20 nucléotides [nt] de l’ARNg sélectionné) pour générer un oligo in silico de 25 nt de long, appelé SGfw.
- De même, ajouter une séquence 5'-AAAC-3' en amont du complément inverse du protoespaceur de l’ARNg sélectionné et une séquence 5'-C-3' en aval de celui-ci pour générer un oligo de 25 nt de long appelé SGrv.
- Commander les séquences SGfw et SGrv sous forme d’oligos d’ADN monocaténaire sans sel, remis en suspension dans l’eau à 100 μM.
- Ajouter 1 μL de chaque oligo à 2 μL de tampon de recuit (10 mM de Tris [pH 7,5-8,0], 50 mM de NaCl, 1 mM d’EDTA) et 16 μL d’eau.
- Effectuer un oligo-recuit en plaçant la solution dans un thermocycleur programmé pour démarrer à 95 °C pendant 10 min. Ensuite, laisser refroidir progressivement jusqu’à 25 °C pendant 45 min.
- Diluer 1 μL des oligos recuits avec 99 μL d’eau sans nucléase, puis ligaturer 1 μL de cette dilution avec 50 ng de plasmide d’ARNg phU6-gRNA préalablement digéré avec l’enzyme de restriction BsaI (suivre les instructions des fournisseurs de kits BsaI et ligase pour la procédure de digestion et de ligase).
- Transformer 20 μL de cellules E. coli chimiquement compétentes avec 2 μL de produit de ligature (suivre les instructions du fabricant pour la procédure de transformation).
- Choisissez plusieurs colonies pour la production d’ADN plasmidique Miniprep (suivez les instructions du fournisseur) et contrôlez le clonage réussi du protospacer par séquençage Sanger avec l’amorce suivante correspondant au promoteur U6 5'-GAGGGCCTATTTCCCATGATT-3'.
- Choisissez l’une des colonies positives pour la production d’ADN plasmidique Midiprep (en suivant les instructions du fabricant).
- Délivrer les ARNg sélectionnés et les ETR basés sur CRISPR/dCas9 dans la lignée cellulaire rapporteure en réseau (un ARNg spécifique par condition) (Figure 3).
REMARQUE : À titre de cas représentatif, le flux de travail pour la nucléofection des plasmides codant pour dCas9 :KRAB, dCas9 :DNMT3A, dCas9 :DNMT3L et les ARNg ciblant l’îlot B2M CpG dans les cellules B2MTdTomato K-562 est illustré dans les étapes suivantes.- Préparez des tubes séparés contenant 500 ng de chacun des plasmides codant pour dCas9 :KRAB-, dCas9 :DNMT3A- et dCas9 :DNMT3L, mais différents pour l’ARNg à tester (125 ng d’un plasmide codant pour l’ARNg par tube). Inclure une condition de nucléofection sans ARNg et ETR en tant qu’échantillon traité par simulation. Effectuez le criblage avec au moins trois répétitions techniques par échantillon.
- Pelleter 5 × 105 cellules B2MTdTomato K-562 par tube et les nucléo-utiliser avec le mélange plasmidique (en suivant les instructions du fabricant).
- Remettre les cellules en suspension dans 200 μL de milieux de culture de cellules de mammifères RPMI-1640 préalablement réchauffés et les remettre dans l’incubateur.
4. Analyser l’activité transcriptionnelle du gène cible au fil du temps
- Utiliser la cytométrie en flux pour mesurer le pourcentage de cellules réduites au silence à différents moments après l’administration des ETR (Figure 4). Utilisez des cellules de type sauvage (WT) - ne portant pas la séquence codant pour les fluorophores - pour définir le seuil de cellules rapporteures négatives. Utilisez l’échantillon traité par simulation pour définir la porte pour les cellules rapporteures positives.
REMARQUE : Comme suggéré à l’étape 1.3, incluez un contrôle de perturbation génétique dans l’expérience. Cela peut être utile à la fois pour surveiller l’efficacité de l’administration de CRISPR et la valeur adaptative des cellules privées du gène cible ; La perte à la fois du silençage transcriptionnel et de la perturbation génétique au fil du temps peut être attribuée à l’essentialité du gène cible dans le type de cellule choisi. Incluez à la fois des points temporels à court terme (jour 3, jour 7, jour 10) et à long terme (jour 21, jour 35) pour obtenir une indication de l’efficacité à court et à long terme du silence. - Identifiez les trois principaux ARNg en termes d’efficacité de silençage à long terme. Utilisez FACS pour sélectionner la sous-population déclarante négative maintenue de manière stable dans ces échantillons. De plus, effectuer des FACS sur les échantillons en vrac traités par simulation afin de les soumettre au même traitement que les échantillons d’essai afin de permettre une comparaison appropriée dans les analyses ultérieures.
5. Évaluation de la spécificité du traitement ETR par RNA-seq et immunoprécipitation d’ADN méthylé (MeDIP)-seq
- Utilisez le séquençage de l’ARN pour évaluer toute éventuelle dérégulation transcriptionnelle à l’échelle du génome lors de l’administration de l’ETR.
- Pour la sous-population des échantillons traités avec les trois ARNg les plus performants et pour les cellules traitées par simulation, extraire l’ARN à l’aide de kits disponibles dans le commerce. Évaluez la qualité et la concentration de l’ARN à l’aide de trousses disponibles dans le commerce.
- Effectuer la fragmentation de l’ARN, la rétrotranscription et la préparation des banques à l’aide de kits disponibles dans le commerce pour la préparation des banques de séquençage de l’ARN (en suivant les instructions du fabricant).
- Effectuez la quantification de la bibliothèque et le contrôle de la qualité à l’aide d’instruments de contrôle de la qualité compatibles avec le séquençage de nouvelle génération et l’électrophorèse numérique.
- Bibliothèques de séquences sur un séquenceur de nouvelle génération suivant les instructions du fabricant, avec un protocole apparié de 100 bp et visant une moyenne de 45 millions de lectures/échantillon.
- Alignez les balises de lecture sur le génome de référence approprié et quantifiez l’expression des transcrits. Effectuez l’alignement sur la séquence tdTomato et quantifiez-la séparément.
NOTE : Ici, l’aligneur STAR (v 2.3.0)43, avec les paramètres par défaut, couplé au paquet Rsubread44 est utilisé. - Effectuer l’analyse des données de séquençage de l’ARN selon les meilleures pratiques publiées45.
REMARQUE : Le boîtier R/Bioconductor edgeR46 est utilisé ici, en appliquant un filtre d’au moins un comptage par million (cpm) dans au moins trois échantillons pour éliminer les gènes faiblement exprimés. Vous pouvez également utiliser la fonction filterByExpr dans edgeR. - Évaluer l’expression différentielle des gènes à l’aide d’un modèle log-linéaire binomial généralisé négatif implémenté edgeR (function glmFit)47. Fixer un seuil de 0,01 sur les valeurs p ajustées (correction de Benjamini-Hochberg [BH]) pour conserver les gènes régulés différentiellement.
- Évaluer toute éventuelle activité de méthylation CpG hors cible des ETR par MeDIP-seq.
- Pour la sous-population des échantillons traités avec les trois premiers ARNg et pour les cellules traitées simulées, extraire l’ADN génomique à l’aide de trousses disponibles dans le commerce (en suivant les instructions du fabricant).
- Sonicer 500 ng d’ADN génomique à l’aide d’un ultrasonicateur et des paramètres suivants : Droit : 20 % ; PIP : 175 ; Cycles par rafale : 200 ; Durée : 40 s.
- Préparez des bibliothèques de séquençage avec les kits disponibles dans le commerce pour MeDIP-seq (en suivant les instructions du fabricant).
- Après l’étape de ligature par adaptateur, quantifier les bibliothèques par dosage fluorométrique et vérifier l’efficacité de la ligature par qPCR à l’aide de kits de quantification de bibliothèque disponibles dans le commerce (en suivant les instructions du fabricant).
- Obtenez des pools de bibliothèques en mélangeant des bibliothèques sélectionnées au hasard pour réduire les biais techniques. Utilisez une quantité de bibliothèque égale (ng) pour chaque bibliothèque afin d’équilibrer les pools. À chaque pool, ajoutez l’ADN de contrôle de pointe méthylé et non méthylé fourni dans la trousse. À des fins de contrôle, conservez un volume de 10 % de la bibliothèque, étiqueté comme « entrée » et non immunoprécipité. Effectuer l’immunoprécipitation sur les 90 % restants de la bibliothèque à l’aide de l’anticorps monoclonal dirigé contre la 5-méthylcytosine fourni dans le kit MeDIP-seq.
- Purifier les bibliothèques enrichies et d’entrée à l’aide des kits de purification du produit d’immunoprécipitation de la 5-méthylcytosine, en suivant les instructions des fabricants.
- Évaluez l’efficacité de l’enrichissement en effectuant une PCR quantitative en temps réel sur le pic interne des contrôles à l’aide d’amorces fournies avec le kit. Pour chaque immunoprécipitation (IP), calculer la spécificité d’enrichissement à partir de la récupération de l’ADN méthylé et non méthylé, en utilisant les valeurs de seuil de cycle (Ct) de MeDIP et les fractions d’entrée obtenues à partir de la réaction qPCR (voir équations 1 et 2) :
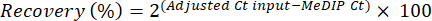 (1)
(1)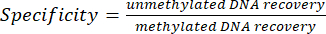 (2)
(2)
REMARQUE : Considérez que les bibliothèques IP réussissent si les valeurs de spécificité sont de ≥0,95. - Amplifier les bibliothèques à l’aide des kits de préparation de bibliothèques MeDIP-seq, en suivant les instructions des fabricants, et effectuer la quantification et l’analyse de la distribution de la taille des bibliothèques du produit.
- Effectuez le séquençage de la bibliothèque sur les séquenceurs de nouvelle génération. Utilisez le séquençage par paires, avec une longueur de lecture de 100 pb, visant une moyenne de 30 millions de lectures/échantillon.
- Aligner les balises de lecture de séquençage sur le génome de référence approprié (p. ex., hg38) à l’aide de bwa (v 0.7.5 ou supérieur)48, puis identifier les pics à l’aide de MACS (v 2.0.10 ou supérieur)49, ce qui permet d’identifier les pics larges (-slocal = 0,-llocal = 500000).
- Créez un ensemble commun de régions à partir de différents échantillons à l’aide de l’outil multiintersection50 de BEDTools, en activant l’option de clustering.
- Calculez la couverture par échantillon sur la liste finale des régions à l’aide du multicov de BEDTools, en éliminant les lectures dupliquées.
- Effectuez une analyse du nombre de matrices à l’aide d’edgeR. Appliquez un filtre d’au moins un comptage par million (cpm) dans au moins trois échantillons pour éliminer les régions faiblement enrichies.
REMARQUE : Vous pouvez également utiliser la fonction filterByExpr dans edgeR. - Identifiez la méthylation différentielle en adoptant le modèle log-linéaire généralisé implémenté dans edgeR (fonction glmFit) et en normalisant à l’aide de la normalisation conditionnelle des quantiles51 pour corriger le contenu en GC par région. Sélectionnez les régions méthylées différentiellement en appliquant un seuil de 0,01 sur les valeurs p ajustées en BH. Effectuez l’analyse des séquences répétées comme suit.
REMARQUE : Reportez-vous au guide de l’utilisateur edgeR pour obtenir la liste complète des options et des paramètres (https://bioconductor.org/packages/release/bioc/html/edgeR.html).- Filtrez les résultats MeDIP-seq pour une valeur nominale de p <0,01 et créez deux ensembles de régions : sélectionnez les régions ayant logFC >1 dans le premier ensemble et les régions ayant logFC <-1 dans le deuxième ensemble.
- Récupérez l’annotation RepeatMasker pour le génome choisi sous la forme d’un fichier de lit et comptez le nombre d’éléments dans chaque ensemble. Convertissez le nombre sous la forme d’un ratio sur le nombre de régions pour chaque jeu de données.
- Extraire le rapport de méthylome qui chevauche chaque classe de répétitions et effectuer un test du Khi carré pour détecter tout enrichissement significatif.
- Évaluer si les régions transcrites différentiellement ou différentiellement méthylées entre les échantillons traités ETR et simulés correspondent à la liaison de l’ARNg hors cible prédite in silico.
- Utilisez CRISPR design suite52 comme outil de prédiction de la liaison de l’ARNg hors cible.
- Pour chaque région hors cible putative, examinez le site de départ de transcription (TSS) le plus proche et la région méthylée la plus proche. Considérons comme véritable effet hors cible une région associée soit à un gène régulé par FDR <0,01 et une distance au MES inférieure à 10 Kb, soit une région méthylée régulée par FDR <0,01 et une distance inférieure à 1 Kb.
- Identifier le nombre et les caractéristiques des régions transcriptionnellement altérées ou surméthylées dans les échantillons traités avec chacun des trois ARNg les plus performants par rapport aux échantillons traités simulés (Figure 5) afin d’identifier le plus spécifique parmi les ARNg. Classez l’impact potentiel d’un site hors cible sur la physiologie des cellules cibles dans l’ordre suivant (du plus impactant au moins impactant) :
i) Gène intragénique, régulateur, physiologiquement exprimé
ii) Gène intragénique, région exonique - physiologiquement exprimé
iii) Gène intragénique, région intronique, physiologiquement exprimé
iv) Gène intragénique, région régulatrice non exprimée
v) Gène intragénique, région exonique non exprimée
vi) Gène intragénique, région intronique non exprimé
vii) Région intergénique
Résultats
Lors de la livraison d’un modèle de donneur pour l’intégration médiée par la recombinaison homologue du rapporteur fluorescent dans le locus cible couplé au système CRISPR/Cas9 (par exemple, par nucléofection plasmidique dans le cas des cellules K-562), des cellules rapporteur-positives apparaissent dans l’échantillon traité (Figure 1, en bas). Si ce n’est pas le cas, vérifiez à nouveau l’exactitude de la conception et du clonage du modèle donneur et des réactifs CRISPR/Cas9. Si cela se confirme, essayez d’optimiser les doses de réactifs et le protocole d’administration lui-même.
Une fois la lignée cellulaire rapporteure obtenue, sélectionnez les séquences promoteur/amplificateur du gène cible et concevez un panel d’ARNg correspondants. Utiliser des outils de prédiction in silico tels que Chopchop pour identifier les séquences d’ARNg et les classer en termes d’efficacité et de spécificité prédites (Figure 2). Si aucun ARNg n’est récupéré, contrôlez la présence du motif adjacent au protoespaceur Cas9 canonique (PAM) (5'-NGG-3') dans la séquence cible. S’il n’y a pas de séquences PAM, envisagez soit de passer à des ETR basés sur d’autres variantes Cas9 indépendantes de PAM (aucun ETR n’a encore été publié avec ces variantes), soit de passer à d’autres plates-formes de domaine de liaison à l’ADN, telles que ZFPs53 ou TALEs30. Cependant, si seuls les ARNg ayant une faible efficacité/spécificité prédite sont récupérés, envisagez soit 1) de tester ces ARNg de faible qualité, soit 2) d’élargir la séquence d’ADN cible, à la recherche de meilleurs ARNg. Lors de l’administration transitoire de la combinaison triple ETR (ou CRISPRoff ; v2.1) avec les ARNg, effectuer des analyses de cytométrie en flux longitudinal pour l’expression du rapporteur fluorescent, en observant souvent un pic de répression du rapporteur lors des analyses aiguës, qui est ensuite au moins partiellement réabsorbé en raison de la dilution mitotique des plasmides codant pour ETR au fil du temps (Figure 4C). Si la combinaison ETRs/ARNg dépose efficacement la méthylation de CpG sur le locus cible, une répression permanente du rapporteur se produira dans une fraction importante des cellules traitées (Figure 4C). Différents ARNg peuvent montrer une efficacité de silençage variable à long terme (Figure 4B,C).
Pour les évaluations de la spécificité à l’échelle du génome, utilisez MeDIP-seq pour identifier les régions méthylées différentiellement entre les cellules dans lesquelles la cible a été réduite au silence à long terme et les cellules non traitées. Idéalement, un ARNg hautement spécifique n’induira qu’un pic de méthylation de novo de CpG sur le site cible (Figure 5). Si ce n’est pas le cas, on peut envisager de caractériser l’activité hors cible des ARNg classés dans une position inférieure dans la liste d’efficacité sur cible.
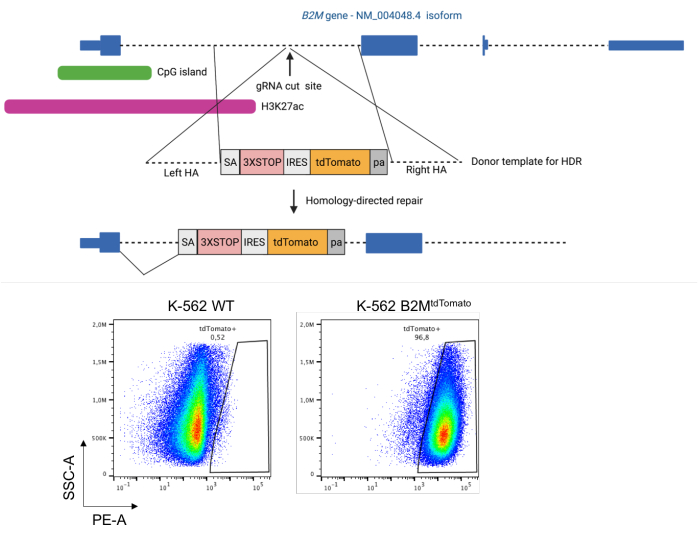
Figure 1 : Intégration d’un rapporteur tdTomato sous les éléments régulateurs du gène B2M humain par réparation dirigée par homologie. En haut : Schémas de la stratégie d’intégration d’un rapporteur fluorescent tdTomato dans le premier intron du gène B2M humain par réparation dirigée par homologie induite par CRISPR/Cas9. En bas : diagrammes de points représentatifs des cellules K-562 avant et après l’intégration du rapporteur tdTomato dans le premier intron du gène B2M. Abréviations : HA = bras d’homologie ; HDR = réparation dirigée par homologie ; IRES = site d’entrée interne du ribosome ; pa = BGH poly(A) ; SA = site accepteur d’épissure ; WT = type sauvage ; 3XSTOP = trois codons stop en tandem. Veuillez cliquer ici pour voir une version agrandie de cette figure.
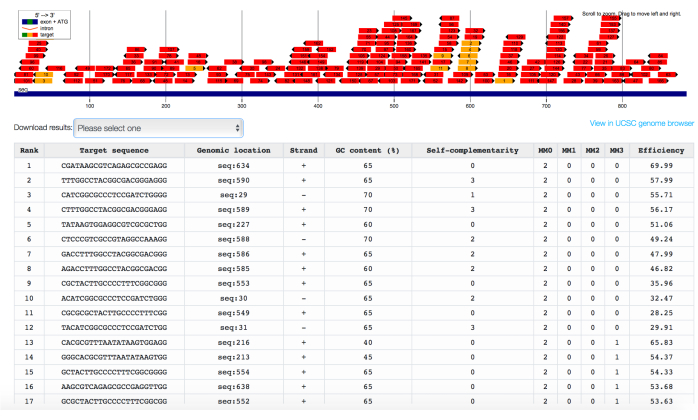
Figure 2 : Identification in silico d’ARNg ciblant l’îlot CpG du gène B2M, classés en fonction de leur efficacité et de leur spécificité prédites. L’interface de sortie de Chopchop montrant les ARNg ciblant l’îlot CpG intégré dans la séquence promotrice du gène B2M, classée à la fois pour le nombre de séquences hors cible avec 0 (MM0), 1 (MM1), 2 (MM2) ou 3 (MM3) et l’efficacité prédite sur la cible. Veuillez cliquer ici pour voir une version agrandie de cette figure.
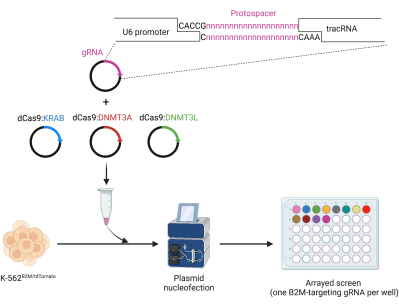
Figure 3 : Clonage et nucléofection matricielle d’ARNg pour le silençage épigénétique médié par dCas9 ETR. En haut : clonage de protospacer à médiation oligoscopique dans un plasmide humain exprimant l’ARNg U6. En bas : criblage en réseau d’ARNg ciblant B2M pour le silençage épigénétique basé sur CRISPR/dCas9 par nucléofection plasmidique dans les cellules K-562B2M/tdTomi. Veuillez cliquer ici pour voir une version agrandie de cette figure.
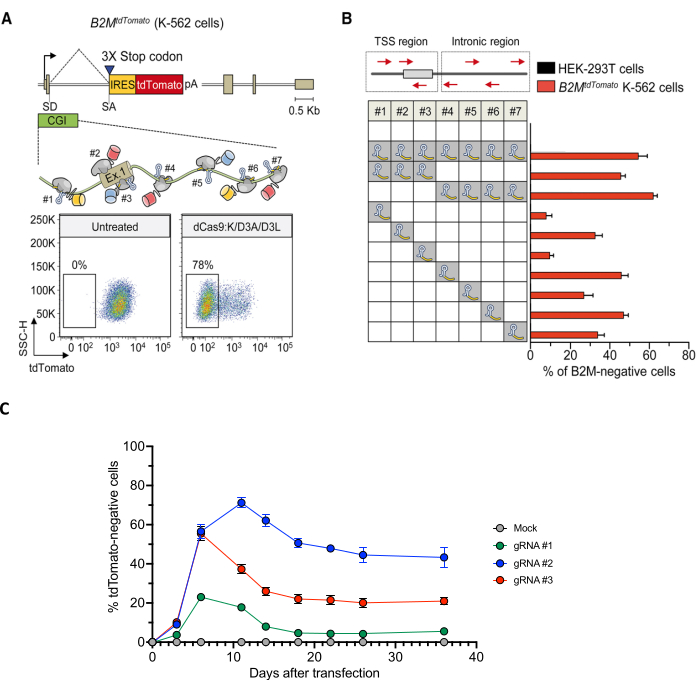
Figure 4 : Criblage des ARNg induisant efficacement le silençage à long terme du gène B2M. (A) En haut : schémas du gèneB2M tdTomato représenté dans la zone agrandie, l’ordre relatif et l’orientation de la liaison des ETR à base de dCas9 complexés avec des ARNg. En bas : diagrammes de points représentatifs des cellulesB2M tdTomato K-562 avant (à gauche) ou après (à droite) l’inactivation de l’ETR. Analyses à 30 jours après la transfection avec des plasmides codant pour les ARNg et la triple combinaison dCas9 :KRAB+ dCas9 :D3A + dCas9 :D3L. (B) Activité de silençage des ARNg indiqués (soit dans des pools, soit sous forme d’ARNg individuels) ciblant l’îlot CpG de B2M (les flèches rouges dans le schéma supérieur indiquent l’orientation des ARNg) dans les cellules K-562B2M tdTomato au jour 30 après la transfection. Les données montrent le pourcentage de cellules tdTomates négatives (moyenne ± MEB ; n = quatre transfections indépendantes pour chaque condition de traitement). (C) Analyse temporelle des cellules B2MtdTomato K-562 lors de la transfection avec des plasmides exprimant la combinaison triple ETR et les ARNg B2M CpG ciblant l’îlot ou simulé (transfection sans ARNg et ETR). Les données indiquent le pourcentage de cellules tdTomates négatives (moyenne ± MEB ; n = trois transfections indépendantes pour chaque condition de traitement). Données non publiées. Panneaux A et B adaptés d’Amabile et al.30. Abréviations : CGI = îlot CpG ; IRES = site d’entrée interne du ribosome ; pa = BGH poly(A) ; SA = site accepteur d’épissure ; SD = site donneur d’épissure ; TSS = site de départ de la transcription ; UT = non transfecté ; 3XSTOP = 3 codons stop en tandem. Veuillez cliquer ici pour voir une version agrandie de cette figure.
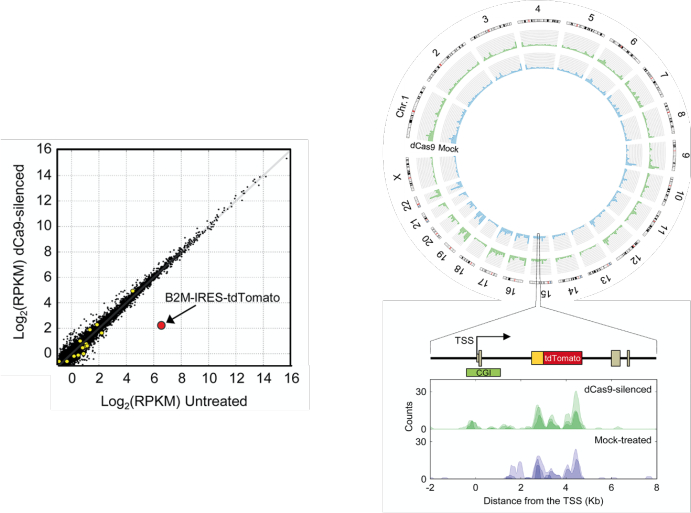
Figure 5 : Analyse à l’échelle du génome de la spécificité de l’ETR par RNA-seq et par MeDIP-seq. À gauche : comparaison des niveaux d’expression dans les cellulesB2M tdTomato K-562 traitées en simulacre et les cellules traitées avec la combinaison triple dCas9 :KRAB, dCas9 :DNMT3A, dCas9 :DNMT3L ETR et avec un ARNg ciblant l’îlot CpG du gène B2M. Les valeurs sont exprimées en log2 de lectures par kilobase par million (RPKM) de lectures mappées. Les points noirs représentent les gènes exprimés à des niveaux comparables dans toutes les conditions ; les cercles jaunes représentent les gènes régulés de manière différentielle dans le cadre d’un RAD <0,01 ; Le cercle rouge représente la transcription B2M-IRES-tdTomato. En haut à droite : tracé circos montrant les profils MeDIP-seq du génome entier de cellulesB2M tdTomato K-562 traitées simulées (en bleu) ou de cellules traitées avec la combinaison triple dCas9 :KRAB, dCas9 :DNMT3A, dCas9 :DNMT3L ETR et avec un ARNg ciblant l’îlot CpG (CGI) du gène B2M (vert). En bas à droite : l’état de méthylation du locus B2MtdTomato dans les échantillons indiqués est indiqué. Trois répétitions sont représentées dans chaque carambolage ; L’empilement des lectures alignées a été lissé à l’aide d’une fenêtre gaussienne. Cette figure a été adaptée d’Amabile et al.30. Abréviation : TSS = site de début de la transcription. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Discussion
Le silençage épigénétique ciblé peut représenter une solution prometteuse pour traiter les troubles qui peuvent bénéficier d’une inactivation permanente des gènes, y compris les maladies causées par des mutations de gain de fonction1, les maladies infectieuses2 et les pathologies dans lesquelles le silençage d’un gène peut soit compenser une anomalie héréditairedans un autre 3, soit libérer tout le potentiel des thérapies cellulaires adoptives4, 5. Le En agissant au niveau de la chromatine et en étant auto-propagé par la cellule 7,30,32, le silençage épigénétique peut éviter les altérations toxiques (par exemple, les réarrangements chromosomiques) de la séquence d’ADN du gène cible et le silençage partiel et transitoire de la cible, qui sont des limitations de la perturbation génique artificielle basée sur la nucléase8,34,35 et de l’inhibition basée sur l’ARNi 33, respectivement.
L’une des étapes préliminaires clés de tout protocole de silençage épigénétique consiste à identifier la position appropriée sur le gène cible pour diriger les ETR qui déposeront les marques épigénétiques répressives nécessaires pour désactiver l’activité transcriptionnelle de la cible. Différents éléments régulateurs de départ transcriptionnels, proximaux et distaux peuvent concourir à soutenir la production transcriptionnelle d’un gène humain donné54. De plus, il est désormais possible d’identifier différents sites cibles par élément de régulation spécifique grâce au nombre croissant de technologies programmables de liaison à l’ADN6. Par conséquent, des protocoles, tels que celui décrit ici dans les lignées cellulaires modifiées, peuvent être utilisés pour désigner des sites cibles individuels et/ou des régions génomiques susceptibles d’être réduites au silence épigénétique médié par l’ETR, avant de se lancer dans des efforts d’évaluation fastidieux et chronophages des meilleurs candidats dans le cadre thérapeutique final. Certains aspects critiques du protocole sont décrits plus en détail ci-dessous.
Ingénierie d’une lignée cellulaire rapporteure prédictive de la cible thérapeutique finale
Malgré l’optimisation constante des protocoles d’ingénierie cellulaire - nécessaires ici pour insérer la cassette codant pour le rapporteur fluorescent dans le gène cible et pour délivrer les ETR - on ne peut pas tenir pour acquis qu’ils pourraient déjà être disponibles pour la lignée cellulaire qui ressemble le plus à la cible thérapeutique finale. Dans ce cas, différentes stratégies d’atténuation peuvent être appliquées : a) tirer parti des kits d’optimisation fournis par les fournisseurs pour optimiser en interne le protocole de transfection de la lignée cellulaire cible ; b) le passage à d’autres lignées cellulaires exprimant toujours ce gène cible, mais appartenant à des tissus autres que la cible thérapeutique finale, pour lesquels les protocoles d’ingénierie ont été largement optimisés. Dans le cas où ces options ne sont pas disponibles, on peut envisager de passer à des types de cellules primaires ou à des organoïdes représentant la cible finale. D’une manière générale, tant pour les études précliniques que pour les applications thérapeutiques du silençage épigénétique basé sur l’ETR, les scénarios dans lesquels le gène cible est essentiel pour le type de cellule cible doivent être écartés. L’inhibition complète et à long terme d’un gène essentiel imposé par les ETR conduira à une contre-sélection des cellules cibles au fil du temps (et, potentiellement, à la toxicité du traitement). Dans ces cas, il est préférable d’utiliser d’autres technologies permettant l’abrogation partielle de la cible, comme l’ARNi33.
Évaluation de l’activité de réduction au silence ciblée des ETR
Les ETR basés sur la combinaison des domaines effecteurs KRAB, DNMT3A et DNMT3L se sont avérés efficaces contre la grande majorité des gènes codant pour des protéines, avec une large fenêtre de ciblage permissive d’environ 1 kilobase centrée sur le site de départ de la transcription32. Pour avoir une idée de ce à quoi ressemble une expérience de silençage épigénétique bien réalisée, les détails techniques et les résultats de l’inhibition du gène B2M dans les cellules K-562 sont fournis ici. Cela peut être considéré comme un contrôle positif important à inclure non seulement par les chercheurs travaillant avec des cellules K-562, mais aussi par ceux qui s’approchent de la technologie basée sur l’ETR pour la première fois. Comme indiqué dans le protocole, la perturbation génique basée sur la nucléase artificielle (par exemple, CRISPR/Cas9) est recommandée comme contrôle supplémentaire de l’efficacité de l’administration du gène dans le type cellulaire d’intérêt et du phénotype des cellules privées du gène cible. Après le criblage initial des ARNg à coupler avec les ETR basés sur CRISPR/dCas9, si aucun des ARNg testés n’est capable de faire taire définitivement le gène cible, il faut envisager, dans l’ordre suivant : 1) d’augmenter la quantité d’ARNg et d’ETR délivrés ; 2) tester des pools des meilleurs ARNg à la recherche d’effets synergiques entre eux. Si l’inhibition à long terme n’est toujours pas atteinte, il faut envisager : 3) de tester des ARNg supplémentaires, qui peuvent cibler des sites plus pertinents pour instruire le silençage épigénétique ; 4) le passage à des plates-formes DBD basées sur ZFP8 ou TALE9, qui peuvent avoir une capacité de liaison améliorée à la chromatine cible ; 5) le passage d’un transitoire à un stable, par exemple en intégrant l’expression vectorielle virale des ETR (soit les constructions de fusion de l’ARNg, soit les deux lors de l’adoption de la technologie CRISPR/dCas9). Étant donné que notre groupe a mis au point et possède une solide expérience de la co-livraison des trois ETR distincts30, le protocole et les résultats présentés ici sont basés sur cette approche. Cependant, un flux de travail conceptuel similaire peut probablement être appliqué à un système tout-en-un basé sur CRISPR32.
Évaluation de l’activité hors cible des ETR
De nombreuses études ont montré des indications préliminaires in vitro de la spécificité des ETR basées sur la combinaison des domaines effecteurs KRAB, DNMT3A et DNMT3L30,31,32. Cependant, si parmi les ARNg testés, aucun d’entre eux ne présente un profil de spécificité satisfaisant en termes de régulation transcriptionnelle et/ou de méthylation de novo de l’ADN, on peut poursuivre deux stratégies non mutuellement exclusives : a) réduire le temps de séjour des ETR à l’intérieur de la cellule (et par conséquent leur activité potentielle hors cible) soit en diminuant les doses d’ETR, soit en testant des systèmes d’administration alternatifs. Par exemple, par rapport aux plasmides, on s’attend à ce que l’ARNm et l’administration de protéines réduisent le temps d’exposition des cellules aux ETR et, par conséquent, la probabilité d’une activité hors cible55 ; b) le passage à des variantes Cas9 plus récentes, optimisées pour réduire la liaison hors cible de la plate-forme56, ou à d’autres technologies de liaison à l’ADN basées sur ZFP8 ou TALE9. Il est important de considérer que, par rapport aux échantillons traités simulés, l’activité sur cible et hors cible du silençage génique est affectée non seulement par la liaison de la DBD à leur séquence cible, mais aussi par la capacité potentielle des domaines effecteurs épigénétiques à être recrutés sur d’autres loci par leurs cofacteurs endogènes naturels. Par conséquent, la réduction du temps de séjour des ETR dans la cellule cible peut diminuer non seulement la probabilité de liaison du DBD aux sites hors cible, mais aussi la probabilité que les ETR interagissent avec des cofacteurs endogènes, avec des avantages potentiels en termes de spécificité et des inconvénients en termes d’activité sur cible. Enfin, par rapport aux échantillons traités de manière simulée, certaines des altérations transcriptionnelles et moins probables de la méthylation de CpG mesurées dans les cellules réduites au silence peuvent simplement être dérivées par la privation du gène cible. Ceux-ci ne sont pas considérés comme des cibles hors cible de la technologie de réduction au silence. Pour les identifier, il faut également inclure la perturbation génique par nucléase artificielle dans le panel expérimental 8,9,10. Les altérations biologiques dues à la perte fonctionnelle du gène cible seront partagées entre le silençage épigénétique et cette technologie alternative.
Déclarations de divulgation
AL est cofondateur, détenteur de quotas et consultant de Chroma Medicine, Inc.
Remerciements
Les auteurs tiennent à remercier Angelo Amabile, Paola Capasso, Ilaria Caserta, Tania Baccega, Alice Reschigna, Valeria Mollica et Deborah Cipria pour l’effort de collaboration visant à développer la technologie de silençage épigénétique au fil des ans ; Dejan Lazarevic et Francesca Giannese pour l’examen critique des analyses RNA-seq et MeDIP-seq décrites dans le protocole. Ces travaux ont été soutenus par des subventions à A.L. de la Fondation Téléthon (subvention TIGET n° F1) et du programme européen Horizon 2020 (UPGRADE). Les illustrations ont été créées avec BioRender.com.
CONTRIBUTION DES AUTEURS
A.M, M.A.C., F.G. et A.C. ont contribué à la conception du protocole et à la rédaction du manuscrit ; S.V., I.M. et D.C. ont conçu les sections bioinformatiques du protocole et révisé le manuscrit ; A.M. et A.L. ont conçu le protocole, conçu et rédigé le manuscrit avec la participation de tous les auteurs.
matériels
| Name | Company | Catalog Number | Comments |
| 4200 TapeStation System | Agilent | G2991BA | DNA quantification |
| 4D-Nucleofector X Unit | Lonza Bioscience | AAF-1003X | Nucleofection |
| B2M silencing gRNA #1 | Lombardo's lab | GCAATCAGGACAAGGCCCGC | Gene silencing |
| B2M silencing gRNA #2 | Lombardo's lab | GGGGTAGGAGAGACTCACGC | Gene silencing |
| B2M silencing gRNA #3 | Lombardo's lab | GAGTCCAGGGCTGGATCTCG | Gene silencing |
| BD FACSAria Fusion Flow Cytometer | BD Biosciences | https://www.bdbiosciences.com/en-us/products/instruments/flow-cytometers/research-cell-sorters/bd-facsaria-fusion | Fluorescence Activated Cell Sorting |
| bedtools | Bedtools | http://bedtools.readthedocs.io/en/latest/ | Processing of genomic intervals |
| bwa | Ih3 | https://github.com/lh3/bwa | Alignment of MeDIP-seq reads |
| Chopchop | Valen's lab | http://chopchop.cbu.uib.no/ | gRNA selection software |
| Corning RPMI 1640 Medium (Mod.) 1x with L-Glutamine | Corning | 10-040-CV | Cell culture |
| cqn | Bioconductor | http://bioconductor.org/packages/release/bioc/html/cqn.html | Region-wise normalization by GC-content |
| CRISPR design suite | Zhang's lab | https://zlab.bio/resources-2 | off-target gRNA binding prediction |
| CRISPRoff-v2.1 plasmid | Addgene | 167981 | Gene silencing |
| CytoFLEX S V4-B4-R3-I2 Flow Cytometer | Beckman Coulter | C01161 | Flow cytometry |
| Donor template sequence for tdTomato integration in the first intron of the B2M gene | Lombardo's lab | ctcctcctctgacctgtgtgtgggttttgtttttgtttt | Genetic engineering |
| E220 Focused-ultrasonicator | Covaris | 500239 | DNA sonication |
| edgeR | Bioconductor | https://bioconductor.org/packages/release/bioc/html/edgeR.html | Differential abundance testing |
| EnGen Mutation Detection Kit | NEB | E3321 | Gene disruption quantification |
| Fetal Bovine Serum | Sigma-Aldrich | F2442 | Cell culture |
| Flowjo | BD Biosciences | https://www.flowjo.com/solutions/flowjo | Flow cytometry data analysis software |
| Gel Loading Dye, Purple (6x) | NEB | B7024S | DNA gel loading |
| Go Taq G2 Hot Start DNA Polymerase | Promega | M7401 | PCR amplification |
| gRNA sequence for dTomato integration in the first intron of the B2M gene | Lombardo's lab | AGGCTACTAGCCCCATCAAG | Genetic engineering |
| hCas9 plasmid | Addgene | 41815 | Genetic engineering |
| High Sensitivity D1000 Reagents | Agilent | 5067-5585 | DNA quantification |
| High Sensitivity D1000 ScreenTape | Agilent | 5067-5584 | DNA quantification |
| High Sensitivity RNA ScreenTape | Agilent | 5067-5579 | RNA quantification |
| High Sensitivity RNA ScreenTape Ladder | Agilent | 5067-5581 | RNA quantification |
| High Sensitivity RNA ScreenTape Sample Buffer | Agilent | 5067-5580 | RNA quantification |
| IPure kit | Diagenode | C03010011 | Purification of the 5-methylcytosine immunoprecipitation product |
| K-562 cells | ATCC | CCL-243 | Cell engineering |
| KAPA Library Quantification Kit | Roche | KK4824 | MeDIP-Seq libraries preparation |
| MACS2 | Taoliu | https://github.com/macs3-project/MACS | Identification of methyl-enriched regions |
| MagMeDIP kit | Diagenode | C02010020 | 5-methylcytosine immunoprecipitation |
| NextFlex Methylseq kit 1 | Bioo Scientific | 5118-01 | MeDIP-Seq libraries preparation |
| NextSeq 500 / NovaSeq 6000 | Illumina | SY-415-1002 / 20012850 | Next Generation Sequencing |
| NucleoBond Xtra Midi kit for transfection-grade plasmid DNA | Macherey-nagel | 740410.50 | Midiprep plasmid preparation |
| One Shot TOP10 Chemically Competent cells | ThermoFisher | C404010 | Plasmid transformation |
| pAC154-dual-dCas9VP160-sgExpression plasmid | Addgene | 48240 | Gene activation |
| pcDNA.CMV.dCas9:KRAB plasmid | Lombardo's lab | available on request (lombardo.angelo@hsr.it) | Gene silencing |
| pcDNA.CMV.dCas9:KRAB plasmid | Lombardo's lab | available on request (lombardo.angelo@hsr.it) | Gene silencing |
| pcDNA.CMV.dCas9:KRAB plasmid | Lombardo's lab | available on request (lombardo.angelo@hsr.it) | Gene silencing |
| Penicillin/Streptomycin | Sigma-Aldrich | P0781 | Cell culture |
| phU6.sgRNA plasmid | Addgene | 53188 | Genetic engineering |
| PowerPac Basic Power Supply | Biorad | 1645050 | Agarose gel electrophoresis |
| Primer forward sequence to check tdTomato integration in the first intron of the B2M gene | Lombardo's lab | GTATTTGCTGGTTATGTTAG | Genetic engineering |
| Primer reverse sequence to check tdTomato integration in the first intron of the B2M gene | Lombardo's lab | AATGGTTGAGTTGGAC | Genetic engineering |
| QIAamp DNA Mini Kit | Qiagen | 51304 | DNA extraction |
| Qubit | Thermo Fisher | Q33238 | DNA quantification |
| Qubit dsDNA HS (high sensitivity) Assay Kit | Thermo Fisher | Q32851 | DNA quantification |
| Qubit RNA HS (high sensitivity) Assay Kit | Thermo Fisher | Q32852 | RNA quantification |
| Restriction enzymes | NEB | DNA digestion | |
| RNeasy Mini kit | Qiagen | 74106 | RNA extraction |
| Rsubread | Bioconductor | https://bioconductor.org/packages/release/bioc/html/Rsubread.html | Quantification of gene expression |
| SF Cell Line 4D-Nucleofector X Kit S (32 RCT) | Lonza Bioscience | V4XC-2032 | Nucleofection |
| SnapGene | Dotmatics | https://www.snapgene.com/ | Molecular biology design software |
| STAR | Alexander Dobin | https://github.com/alexdobin/STAR | Alignment of RNA-seq reads |
| T100 Thermal Cycler | Biorad | 1861096 | PCR amplification |
| T4 DNA Ligase | Promega | M1801 | DNA ligation |
| TAE Buffer | Fisher scientific | BP1332500 | Agarose gel electrophoresis |
| TruSeq Stranded Total RNA kit | Illumina | 20020597 | RNA-Seq library preparation |
| UltraPure Agarose | ThermoFisher | 16500500 | Agarose gel |
Références
- Cummings, C. J., Zoghbi, H. Y. Fourteen and counting: unraveling trinucleotide repeat diseases. Human Molecular Genetics. 9 (6), 909-916 (2000).
- Tebas, P., et al. Gene editing of CCR5 in autologous CD4 T cells of persons infected with HIV. The New England Journal of Medicine. 370 (10), 901-910 (2014).
- Frangoul, H., et al. CRISPR-Cas9 gene editing for sickle cell disease and β-thalassemia. The New England Journal of Medicine. 384 (3), 252-260 (2021).
- Murty, T., Gene Mackall, C. L. Gene editing to enhance the efficacy of cancer cell therapies. Molecular Therapy. 29 (11), 3153-3162 (2021).
- Lanza, R., Russell, D. W., Nagy, A. Engineering universal cells that evade immune detection. Nature Reviews Immunology. 19 (12), 723-733 (2019).
- Matharu, N., Ahituv, N. Modulating gene regulation to treat genetic disorders. Nature Reviews Drug Discovery. 19 (11), 757-775 (2020).
- Sgro, A., Blancafort, P. Epigenome engineering: New technologies for precision medicine. Nucleic Acids Research. 48 (22), 12453-12482 (2020).
- Urnov, F. D., Rebar, E. J., Holmes, M. C., Zhang, H. S., Gregory, P. D. Genome editing with engineered zinc finger nucleases. Nature Reviews Genetics. 11 (9), 636-646 (2010).
- Joung, J. K., Sander, J. D. TALENs: A widely applicable technology for targeted genome editing. Nature Reviews Molecular Cell Biology. 14 (1), 49-55 (2013).
- Adli, M. The CRISPR tool kit for genome editing and beyond. Nature Communications. 9 (1), 1911 (2018).
- Gilbert, L. A., et al. XCRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes. Cell. 154 (2), 442 (2013).
- Snowden, A. W., Gregory, P. D., Case, C. C., Pabo, C. O. Gene-specific targeting of H3K9 methylation is sufficient for initiating repression in vivo. Current Biology. 12 (24), 2159-2166 (2002).
- Chen, X., et al. Construction and validation of the CRISPR/dCas9-EZH2 system for targeted H3K27Me3 modification. Biochemical and Biophysical Research Communications. 511 (2), 246-252 (2019).
- Kwon, D. Y., Zhao, Y. T., Lamonica, J. M., Zhou, Z. Locus-specific histone deacetylation using a synthetic CRISPR-Cas9-based HDAC. Nature Communications. 8, 15215 (2017).
- Stepper, P., et al. Efficient targeted DNA methylation with chimeric dCas9-Dnmt3a-Dnmt3L methyltransferase. Nucleic Acids Research. 45 (4), 1703-1713 (2017).
- Ecco, G., Imbeault, M., Trono, D. KRAB zinc finger proteins. Development. 144 (15), 2719-2729 (2017).
- Witzgall, R., O'leary, E., Leaf, A., Onaldi, D., Bonventre, J. The Kruppel-associated box-A (KRAB-A) domain of zinc finger proteins mediates transcriptional repression. Proceedings of the National Academy of Sciences. 91 (10), 4514-4518 (1994).
- Mannini, R., et al. Structure/function of KRAB repression domains: Structural properties of KRAB modules inferred from hydrodynamic, circular dichroism, and FTIR spectroscopic analyses. Proteins: Structure, Function and Genetics. 62 (3), 604-616 (2006).
- Friedman, J. R., et al. KAP-1, a novel corepressor for the highly conserved KRAB repression domain. Genes and Development. 10 (16), 2067-2078 (1996).
- Iyengar, S., Farnham, P. J. KAP1 protein: An enigmatic master regulator of the genome. The Journal of Biological Chemistry. 286 (30), 26267-26276 (2011).
- Schultz, D. C., Friedman, J. R., Rauscher, F. J. Targeting histone deacetylase complexes via KRAB-zinc finger proteins: The PHD and bromodomains of KAP-1 form a cooperative unit that recruits a novel isoform of the Mi-2α subunit of NuRD. Genes and Development. 15 (4), 428-443 (2001).
- Schultz, D. C., Ayyanathan, K., Negorev, D., Maul, G. G., Rauscher, F. J. SETDB1: A novel KAP-1-associated histone H3, lysine 9-specific methyltransferase that contributes to HP1-mediated silencing of euchromatic genes by KRAB zinc-finger proteins. Genes and Development. 16 (8), 919-932 (2002).
- Nielsen, A. L., et al. Interaction with members of the heterochromatin protein 1 (HP1) family and histone deacetylation are differentially involved in transcriptional silencing by members of the TIF1 family. The EMBO Journal. 18 (22), 6385-6395 (1999).
- Sripathy, S. P., Stevens, J., Schultz, D. C. The KAP1 corepressor functions to coordinate the assembly of de novo HP1-demarcated microenvironments of heterochromatin required for KRAB zinc finger protein-mediated transcriptional repression. Molecular and Cellular Biology. 26 (22), 8623-8638 (2006).
- Jurkowska, R. Z., Jurkowski, T. P., Jeltsch, A. Structure and function of mammalian DNA methyltransferases. ChemBioChem. 12 (2), 206-222 (2011).
- Jia, D., Jurkowska, R. Z., Zhang, X., Jeltsch, A., Cheng, X. Structure of Dnmt3a bound to Dnmt3L suggests a model for de novo DNA methylation. Nature. 449 (7159), 248-251 (2007).
- Tajima, S., Suetake, I., Takeshita, K., Nakagawa, A., Kimura, H. Domain structure of the Dnmt1, Dnmt3a, and Dnmt3b DNA methyltransferases. Advances in Experimental Medicine and Biology. 945, 63-86 (2016).
- Greenberg, M. V. C., Bourc'his, D. The diverse roles of DNA methylation in mammalian development and disease. Nature Reviews Molecular Cell Biology. 20 (10), 590-607 (2019).
- Ishiyama, S., et al. Structure of the Dnmt1 reader module complexed with a unique two-mono-ubiquitin mark on histone H3 reveals the basis for DNA methylation maintenance. Molecular Cell. 68 (2), 350.e7-360.e7 (2017).
- Amabile, A., et al. Inheritable silencing of endogenous genes by hit-and-run targeted epigenetic editing. Cell. 167 (1), 219.e14-232.e14 (2016).
- Mlambo, T., et al. Designer epigenome modifiers enable robust and sustained gene silencing in clinically relevant human cells. Nucleic Acids Research. 46 (9), 4456-4468 (2018).
- Nuñez, J. K., et al. Genome-wide programmable transcriptional memory by CRISPR-based epigenome editing. Cell. 184 (9), 2503.e17-2519.e17 (2021).
- Davidson, B. L., McCray, P. B. Current prospects for RNA interference-based therapies. Nature Reviews Genetics. 12 (5), 329-340 (2011).
- Haapaniemi, E., Botla, S., Persson, J., Schmierer, B., Taipale, J. CRISPR-Cas9 genome editing induces a p53-mediated DNA damage response. Nature Medicine. 24 (7), 927-930 (2018).
- Kosicki, M., Tomberg, K., Bradley, A. Repair of double-strand breaks induced by CRISPR-Cas9 leads to large deletions and complex rearrangements. Nature Biotechnology. 36 (8), 765-771 (2018).
- Ciccia, A., Elledge, S. J. The DNA damage response: making it safe to play with knives. Molecular Cell. 40 (2), 179-204 (2010).
- Hu, J. H., et al. Evolved Cas9 variants with broad PAM compatibility and high DNA specificity. Nature. 556 (7699), 57-63 (2018).
- Uhlén, M., et al. Proteomics. Tissue-based map of the human proteome. Science. 347 (6220), (2015).
- Labun, K., et al. CHOPCHOP v3: Expanding the CRISPR web toolbox beyond genome editing. Nucleic Acids Research. 47 (W1), W171-W174 (2019).
- Kent, W. J., et al. The Human Genome Browser at UCSC. Genome Research. 12 (6), 996-1006 (2002).
- Cheng, A. W., et al. Multiplexed activation of endogenous genes by CRISPR-on, an RNA-guided transcriptional activator system. Cell Research. 23 (10), 1163-1171 (2013).
- Kabadi, A. M., Ousterout, D. G., Hilton, I. B., Gersbach, C. A. Multiplex CRISPR/Cas9-based genome engineering from a single lentiviral vector. Nucleic Acids Research. 42 (19), e147 (2014).
- Dobin, A., et al. STAR: ultrafast universal RNA-seq aligner. Bioinformatics. 29 (1), 15-21 (2013).
- Liao, Y., Smyth, G. K., Shi, W. The Subread aligner: fast, accurate and scalable read mapping by seed-and-vote. Nucleic Acids Research. 41 (10), e108-e108 (2013).
- Conesa, A., et al. A survey of best practices for RNA-seq data analysis. GenomeBiology. 17, 13 (2016).
- Robinson, M. D., Oshlack, A. A scaling normalization method for differential expression analysis of RNA-seq data. Genome Biology. 11 (3), R25 (2010).
- Robinson, M. D., McCarthy, D. J., Smyth, G. K. edgeR: a Bioconductor package for differential expression analysis of digital gene expression data. Bioinformatics. 26 (1), 139-140 (2010).
- Li, H., Durbin, R. Fast and accurate long-read alignment with Burrows-Wheeler transform. Bioinformatics. 26 (5), 589-595 (2010).
- Zhang, Y., et al. Model-based analysis of ChIP-Seq (MACS). Genome Biology. 9 (9), R137 (2008).
- Quinlan, A. R. BEDTools: The Swiss-Army tool for genome feature analysis. Current Protocols in Bioinformatics. 47, (2014).
- Hansen, K. D., Irizarry, R. A., Wu, Z. Removing technical variability in RNA-seq data using conditional quantile normalization. Biostatistics. 13 (2), 204-216 (2012).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nature Biotechnology. 31 (9), 827-832 (2013).
- Zeitler, B., et al. Allele-selective transcriptional repression of mutant HTT for the treatment of Huntington's disease. Nature Medicine. 25 (7), 1131-1142 (2019).
- Andersson, R., Sandelin, A. Determinants of enhancer and promoter activities of regulatory elements. Nature Reviews Genetics. 21 (2), 71-87 (2020).
- Liang, X., et al. Rapid and highly efficient mammalian cell engineering via Cas9 protein transfection. Journal of Biotechnology. 208, 44-53 (2015).
- Han, H. A., Pang, J. K. S., Soh, B. -. S. Mitigating off-target effects in CRISPR/Cas9-mediated in vivo gene editing. Journal of Molecular Medicine. 98 (5), 615-632 (2020).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon