Method Article
イン・ビトロ 標的エピジェネティックサイレンシングのための遺伝子操作転写リプレッサーの選択
要約
ここでは、高い、長期にわたる、安定したオンターゲットサイレンシング効率と、低いゲノムワイドなオフターゲット活性を持つ、遺伝子操作された転写抑制因子(ETR)の in vitro 選択のためのプロトコルを提示します。このワークフローにより、候補となるETRの初期の複雑なレパートリーを、治療上重要な設定でのさらなる評価に適した短いリストに減らすことができます。
要約
遺伝子の不活性化は、遺伝子機能の研究に役立ち、幅広い疾患の治療のための有望な戦略を表しています。従来の技術では、RNA干渉は、部分的な標的の除去と生涯にわたる治療の必要性に悩まされています。一方、人工ヌクレアーゼはDNA二本鎖切断(DSB)を誘導することで安定した遺伝子不活性化が可能だが、最近の研究ではこのアプローチの安全性が疑問視されている。遺伝子操作された転写リプレッサー(ETR) による 標的エピジェネティック編集は、特定のETRの組み合わせを1回投与するだけで、DNA切断を誘発することなく耐久性のあるサイレンシングが得られるため、解決策となる可能性があります。
ETRは、プログラム可能なDNA結合ドメイン(DBD)と天然の転写抑制因子由来のエフェクターを含むタンパク質です。具体的には、ヒトDNMT3AとヒトDNMT3Lの触媒ドメインであるヒトZNF10のKRABドメインを備えた3つのETRの組み合わせが、ETR標的遺伝子に遺伝性の抑制的なエピジェネティック状態を誘導することが示されました。このプラットフォームのヒットアンドランの性質、ターゲットのDNA配列への影響の欠如、およびオンデマンドでのDNA脱メチル化によって抑圧状態に戻る可能性により、エピジェネティックサイレンシングはゲームチェンジャーツールになります。重要なステップは、オンターゲットを最大化し、オフターゲットサイレンシングを最小限に抑えるために、ターゲット遺伝子上の適切なETRの位置を特定することです。このステップを最終的な ex vivoまたは in vivo の前臨床設定で実行することは、面倒な場合があります。
本稿では、CRISPR/触媒死Cas9システムをETRのパラダイムDBDとしてとらえ、効率的なオンターゲットサイレンシングのためのトリプルETRの組み合わせと組み合わせたガイドRNA(gRNA)の in vitro スクリーニングと、それに続くトップヒットのゲノムワイド特異性プロファイルの評価からなるプロトコルについて説明しています。これにより、候補gRNAの初期レパートリーを有望なgRNAの短いリストに減らすことができ、その複雑さは、治療的に関連性のある関心のある設定での最終評価に適しています。
概要
遺伝子の不活性化は、従来、細胞モデルと動物モデルの両方で遺伝子機能を研究する上で重要な役割を果たしてきました。さらに、過去20年間で、遺伝子治療の台頭により、機能獲得型変異1、感染症2、またはある遺伝子のサイレンシングが別の遺伝子の遺伝性欠陥を補う可能性のある病状3によって引き起こされる疾患を治療するための潜在的に革新的なアプローチとして提案されてきました。最後に、がん免疫療法4および再生医療5のための細胞産物の効率を高めるために、細胞の適応度と機能制御の主要な調節因子の遺伝子不活性化が提案されている。
遺伝子不活性化を達成するためのさまざまな技術の中で、最も有望なものの1つは、標的エピジェネティックサイレンシング6,7です。この技術の中核をなすのは、プログラム可能なDNA結合ドメイン(DBD)とエピジェネティックな抑制機能を持つエフェクタードメイン(ED)からなるキメラタンパク質、いわゆる遺伝子組換え転写抑制因子(ETR)です。ジンクフィンガータンパク質(ZFP)8、転写活性化因子様エフェクター(TALE)9、またはCRISPR/dCas910ベースのDBDは、EDをサイレンシングする標的遺伝子のプロモーター/エンハンサー配列に選択的につなぎ合わせるように設計することができます。一旦そこに着くと、ETRのEDは、使用される抑制ドメインに応じて、ヒストン修飾(H3K9 11,12またはH3K27 13メチル化、H3またはH4脱アセチル化14)およびCpG DNAメチル化15などのヘテロクロマチン誘導抑制エピジェネティックマークを課すことによってサイレンシング活性を実行します。
特に、着床前胚16で発生する内因性レトロウイルスの永久転写抑制の分子プロセスに触発されて、以下のEDを利用するために3つのETRの組み合わせが生成されました:i)ヒトZNF10のKrüppel関連ボックス(KRAB)ドメイン;ii)ヒトde novo DNAメチルトランスフェラーゼ3A(DNMT3A)の触媒ドメイン;iii)完全長ヒトDNAメチルトランスフェラーゼ3様(DNMT3L)。KRABは、高等脊椎動物におけるいくつかのZFPによって共有される保存された抑制ドメインであり17,18、そのサイレンシング活性は、主に、その後、ヌクレオソームリモデリングおよび脱アセチル化(NuRD)複合体21、H3K9ヒストンメチルトランスフェラーゼSETDB1 22、およびH3K9メチル化リーダーHP123を含むいくつかの他のヘテロクロマチン誘導剤20と相互作用する足場タンパク質をリクルートする能力に基づいている。とりわけ24。
DNMT3Aは、CpG配列25でDNA上のメチル基を積極的に転写します。DNMT3Aの触媒活性は、メチル基の転移に関与する触媒ドメインを欠くDNMT3Aの胚細胞および生殖細胞制限パラログであるDNMT3Lとの物理的会合によって増強される26,27。哺乳類遺伝子のプロモーター/エンハンサー要素に埋め込まれたCpGが豊富な領域(CpGアイランド(CGI)と呼ばれる)でのDNAメチル化は、通常、転写サイレンシングと関連している28。重要なことに、一旦沈着すると、CpGメチル化はUHRF1-DNMT1ベースの分子複合体によって有糸分裂全体を通して安定して受け継がれ得る29。
標的細胞におけるETRの安定した過剰発現は、時間の経過とともに生理学的標的部位からの内因性相互作用因子のオフターゲット活性およびスケルチのリスクが高まるため、問題となる可能性があります。しかしながら、単一ETR部分の一過性発現は、高効率で長期サイレンシングを誘導できず30、それらの治療応用を妨げる可能性がある。したがって、この分野における独創的なブレークスルーは、3つのKRAB、DNMT3A、およびDNMT3LベースのETRの組み合わせが相乗効果を発揮し、一過性にしか同時送達されない場合でも、標的遺伝子H3K9のプロモーター配列とCpGメチル化を課すことができるという証拠でした。これらはその後、有糸分裂を通して細胞によって読み取られて増殖し、複数のヒトおよびマウス細胞株、ならびに ex vivo 培養された初代細胞における遺伝性サイレンシングをもたらす30。
注目すべきは、ETRによって課されるエピジェネティックなサイレンシングは、標的(例えば、サイレンシングされた遺伝子座へのCRISPR/dCas9ベースのTET1 DNAデメチラーゼの動員)または薬理学的(DNAメチルトランスフェラーゼ阻害剤5-Azaの投与)DNA脱メチル化30、ETR関連の有害事象の場合の潜在的な解毒剤によって、要求に応じて元に戻すことができることである。3つのKRAB-、DNMT3A-、およびDNMT3LベースのEDを有するオールインワンETRも記載されており、タンパク質をコードする遺伝子の大部分に対して細胞株31,32で有意なサイレンシング効率が示されました。さらに、ETRを用いたいくつかの研究では、de novoCpGメチル化またはクロマチンアクセシビリティの変化に関して大きなオフターゲット活性はなく、高い安全性プロファイルが報告されています30,31,32。ただし、臨床応用の前に、新しく設計されたDBDを装備したETRの特異性プロファイルの専用分析が推奨されます。
臨床的観点からは、標的エピジェネティックサイレンシングは、RNA干渉(RNAi)ベースのノックダウン33と人工ヌクレアーゼベースの遺伝子破壊8の両方に決定的な利点をもたらす可能性があります。RNAiとは対照的に、標的エピジェネティックサイレンシングは、細胞ごとの標的の完全な除去を誘導する可能性があり、長期的なサイレンシングを確実にするための定期的な処理を必要としません。遺伝子破壊とは対照的に、DNA配列は変化せず、DNA二本鎖切断(DSB)の発生を回避します。DSBは、その後、アポトーシスと細胞周期停止を誘導し、機能的なp53経路34,35、特にマルチプレックス遺伝子編集設定では、染色体再構成35を持つ細胞に対する選択につながる可能性があります。さらに、非相同末端結合媒介性DNA DSB修復の不可逆的なモザイク結果を中継することによって36、遺伝子破壊は、最終結果の1つとして、標的の機能的コード配列へのインフレーム修復を避けることができず、エピジェネティックサイレンシングとは対照的に、要求に応じて消去することができない。
最後に、エピジェネティックサイレンシングは、非転写調節要素やノンコーディングRNAなど、RNAiおよび遺伝子破壊に対して完全または少なくとも部分的に抵抗性のクラスに、標的化可能な遺伝的要素の範囲を広げる可能性を秘めています30,32。ターゲットエピジェネティックサイレンシングアプリケーションの最初の重要なステップは、標的遺伝子のさまざまな調節配列をカバーするETRのパネルを設計し、最もパフォーマンスの高いものを特定することです。常に開発中のプログラム可能なDNA結合技術によって標的とすることができるゲノムの部分が増加していることを考えると、テストされるETRの数は非常に重要です37。標的遺伝子を治療的にサイレンシングする細胞タイプに対してETRのスクリーニングを直接行うことが、最も適切な選択肢となります。しかし、ハイスループットスクリーニングは、培養中での生存期間が限られており、エンジニアリング能力が最適でないことが多いため、初代細胞では技術的に扱いにくい場合があります。大規模なスクリーニングは、in vivoではさらに実現不可能になる可能性があります。
より実用的な代替案は、最初に簡単に操作可能な細胞株でETRの大規模なパネルの初期スクリーニングを実施し、次に治療上重要な細胞タイプで最も有望なもののみを検証することです。これと並行して、ETRの消音効率を測定するための適切な読み出しを選択することも課題です。RT-qPCR、ウェスタンブロット、またはELISAによる標的遺伝子の転写産物またはタンパク質レベルの直接評価は、コストと時間がかかり、十分な感度を欠く可能性があるため、ハイスループットスケールでのアプリケーションが制限される可能性があります。蛍光色素を標的遺伝子の調節配列の転写制御下に置いた アドホック 改変レポーター細胞株の作製により、フローサイトメトリーベースのアプローチを利用して、単一細胞レベルでハイスループットペースでエピジェネティックサイレンシングを読み取ることができます。
これらの一般的な考慮事項に従って、この論文では、オンターゲットサイレンシング効率のためのETRの in vitro アレイスクリーニングと、それに続くトップヒットのゲノムワイドなオフターゲット活性の評価で構成されるプロトコルについて説明します。このワークフローにより、候補となるETRの初期レパートリーを、治療上重要な目的の細胞タイプでの最終評価に適した複雑さを持つ有望なETRの短いリストに減らすことができます。
ETRの生成に利用できるさまざまなプログラム可能なDBDの中で、このプロトコルでは、標的遺伝子プロモーターにまたがるgRNAをハイスループットスケールで設計することが容易であるため、CRISPR/dCas9ベースの技術に焦点を当てます。ただし、以下で説明するのと同じ概念ワークフローを採用して、他のDBDを備えたETRの効率と特異性を評価することができます。
プロトコル
1. フローサイトメトリーによる標的遺伝子の転写活性をモニターするための蛍光ベースのレポーター細胞株の操作
- サイレンシングする標的遺伝子を発現する細胞株を同定します。Human Protein Atlas38でサイレンシングする標的遺伝子を閲覧し、その「細胞株」セクションをナビゲートして、目的の体細胞組織を代表する細胞株(例えば、最終標的が肝肝細胞の場合は肝細胞株)を同定します。または、一般に公開されているRNAシーケンシング(RNA-Seq)データベース(NCBI GEOなど)を調べます。
- 候補の中から、効率的な一過性遺伝子送達プロトコル(ETR送達に有用)が利用可能な細胞株を優先します。
注:さまざまなモダリティの中で、ヌクレオフェクションは高いトランスフェクション効率を保証するため、最良の選択肢の1つです。ここでは、β-2-ミクログロブリン(B2M)遺伝子の転写活性を報告する細胞株(以下、B2M TdTomato K-562細胞と呼ぶ)を生成するために、ヒト赤白血病K-562細胞が選択されている。 - さらに、標的遺伝子が細胞の生存に不可欠な細胞株は、培養中の標的遺伝子の安定したサイレンシングによる細胞の維持を損なうため、候補を優先します。以前に報告されていない場合は、選択した細胞タイプにおける標的遺伝子の必須性を理解するために、Cas9ヌクレアーゼと遺伝子の最初のコードエクソンの1つを標的とするgRNAを細胞にトランスフェクションすることにより、遺伝的破壊制御を生成します。
注:遺伝的破壊は、定義上、安定したイベントです。時間の経過に伴う破壊された細胞の逆選択は、標的遺伝子が選択された細胞の生理機能に不可欠であることを示しています。- 選択した細胞株で優先的に使用されるターゲットスプライシングアイソフォームを特定します(このプロトコルでは、 B2M 遺伝子のアイソフォームNM_004048.4がターゲットとされています)。
- 標的アイソフォームの最初のコードエクソンを効果的かつ特異的に切断できるgRNAを同定します(例えば、有効でユーザーフレンドリーなオンラインgRNA選択ツールであるChopchop(http://chopchop.cbu.uib.no/)39)。
- K-562細胞の場合、5細胞あたり1μgのspCas9コードプラスミド(hCas9;材料表を参照)および250 ngのgRNAコードプラスミド(phU6-gRNA;材料表を参照)をヌクレオフェクション×トランスフェクションします(メーカーの指示による)。
- 細胞を培養し(10%ウシ胎児血清[FBS]、L-グルタミン、ペニシリン/ストレプトマイシン[100 U/mL]を添加したRPMI-1640中の5%CO2下で37°CのK-562細胞)、変異検出キットを利用して経時的な遺伝子破壊のレベルをモニターします(メーカーの指示に従ってください)。
- 目的遺伝子の転写制御下で蛍光レポーターを相同組換え媒介で組み込むためのドナーテンプレートをクローニングします(図1)。
- 標的遺伝子の領域を同定し、蛍光色素発現カセットを組み込みます。
注:H3K27アセチル化(活性プロモーターおよびエンハンサーのマーカー)に富むCpGアイランドや領域など、転写に関連する要素を標的にすることは避けてください。これらの調節要素(エピジェネティックなサイレンシングを指示するためにETRが標的とすることが重要である可能性がある)を変化させると、レポーター細胞株は標的遺伝子の生理学的調節を予測しにくくなります。- 標的遺伝子が分泌タンパク質をコードしていない場合は、2A自己切断ペプチドを介してレポーターを標的遺伝子の最後のコドンに融合させ、標的遺伝子の機能を維持します。
- 標的遺伝子が分泌タンパク質をコードしている場合、レポーターの分泌の可能性を避けるために、レポーターを標的遺伝子のイントロニック領域に配置します。スプライスアクセプター部位(SA)を介したスプライシングされた転写産物へのレポーターの強制的な組み込みと、その後の内部リボソームエントリー配列(IRES)を介した翻訳は、標的遺伝子の機能を損なう。
- Chopchopを使用して、標的領域におけるgRNA切断を選択する(ここでは、 B2M 遺伝子の最初のイントロンの配列5'-AGGCTACTAGCCCCATCAAGAGG-3'を標的とするgRNAを選択する)。
- 以下からなるgRNA切断部位のドナーテンプレートを設計する:i)左相同性アーム(gRNA切断部位のすぐ上流の領域に一致するn塩基対[bp]);ii)プロモーターフリー導入遺伝子発現カセット(ここに示したケースでは、 B2M 遺伝子の第1イントロンとのスプライシングを支持するSA-3Xストップコドン-IRES-tdTomato-BGHポリ(A));iii)右相同性アーム(nbpはgRNA切断部位の下流の領域と一致する)。
注:相同組換えを効果的に誘導するために必要な相同アームの長さは、細胞タイプによって異なる場合があります(K-562細胞では100〜500 bpが適切な範囲です)。
- 標的遺伝子の領域を同定し、蛍光色素発現カセットを組み込みます。
- CRISPR/Cas9ヌクレアーゼシステムとドナーテンプレートを標的細胞株内に導入します。K-562細胞の場合、1 μgの spCas9コードプラスミド(hCas9;材料 表を参照)、250 ngのgRNAコードプラスミド(phU6-gRNA; 材料表を参照)、および1 μgのドナーテンプレートコード化プラスミドを5細胞×105 細胞あたり1 μgをヌクレオフェクションでトランスフェクションします(メーカーの説明書による)。
- 細胞を少なくとも14日間培養し(K-562細胞の場合)、フローサイトメーターを使用して蛍光レポーターの発現レベルを経時的にモニターします(フィコエリトリン(PE)チャネルを活性化してtdTomatoレポーターの蛍光強度を測定し、メーカーの指示に従ってフローサイトメトリーを実施します)。
注:ドナーテンプレートは、特にプラスミドベースの場合、不可解なプロモーター配列を含む可能性があり、非統合ドナーコピーからの蛍光レポーターの発現につながります。細胞を培養することで、細胞分裂によってこれらの非統合コピーを希釈し、最終的に標的ゲノムに組み込まれたドナーコピーからのみレポーターの発現を維持することができます。 - 単一細胞レベルでの蛍光活性化セルソーティング(FACS)によるレポーター陽性細胞のクローン化。このプロトコルでは、PEチャンネルを活性化してtdTomatoレポーターの蛍光強度を測定し、メーカーの指示に従って、96ウェルプレートのウェルごとに単一のtdTomato陽性K-562細胞を選別します。
- 培養中の細胞増殖時(K-562細胞の場合は通常20〜30日)、PCRでレポーター陽性クローンをスクリーニングし、標的遺伝子座内のレポーターカセットの両対立遺伝子統合を有するクローンを選択します。
注:これにより、レポーターの発現が最大化され、ETR処理時のフローサイトメトリーにより、レポーター発現細胞とレポーターサイレンシング細胞の間のさらなる分離が容易になります。- DNA抽出キットを使用して、レポーター陽性クローンあたり1〜10×5 個の細胞からゲノムDNAを抽出します(製造元の指示に従ってください)。
- PCR増幅キットの指示に従って、フォワード(5'-GTATTTGCTGGTTATTTAG-3')およびリバース(5'-AATGGTTGAGTTGGAC-3')プライマーで B2M 標的領域を増幅します。このプライマーペアのアニーリング温度は、TaqベースのDNAポリメラーゼと0.5 μMのプライマー濃度で47.7°Cです。
- 1%アガロースゲル電気泳動を用いてPCR産物を分析します(製造元の指示に従ってください)。野生型標的遺伝子座(1,027 bp)に関連するバンドを含まない、 B2M 標的遺伝子座(3,413 bp)へのtdTomatoの組み込みに関連するバンドを示すクローンのスクリーニング。
2. CRISPR/dCas 9に基づく標的遺伝子のエピジェネティックサイレンシングのためのgRNAの設計
- UCSCゲノムブラウザ40で標的遺伝子を閲覧し、CpGアイランドおよびH3K27アセチル化(活性プロモーターおよびエンハンサーのマーカー)のために濃縮された部位など、その転写活性を調節する可能性のある領域のヌクレオチド配列を抽出する。
注:最近の研究によると、最良の標的領域は、標的遺伝子32の転写開始部位を中心とする1キロベース(kb)のウィンドウである。 - 選択した配列をChopchopオンラインツールに貼り付け、取得するgRNAの目的として 抑制 を選択します。Chopchopが目的の遺伝子配列にマッピングされ、オフターゲットマッチの数と予測されるオンターゲット効率の両方を考慮したスコアに従ってリストされたgRNAのリストを提供するのを待ちます(図2)。
- ターゲット配列ごとに少なくとも 10 個の gRNA を選択します。可能であれば、ゲノム全体の他の遺伝子内配列と完全に一致しない状態で、調査する領域全体にまたがるgRNAを選択するようにしてください。
3. レポーター細胞株におけるCRISPR/dCas 9ベースのETRのアレイ型一過性送達
- 標的細胞株についてこれまでに報告された導入遺伝子送達システムの中から、一過性の導入遺伝子発現のみを可能にし、導入効率を最大化し、細胞操作に関連する毒性を最小限に抑えるものを選択してください。K-562細胞の場合、プラスミドとmRNAの両方の核形成を強く推奨し、プラスミド産生は技術的に簡単で安価な代替手段となります。
- セクション 2 で選択した gRNA と CRISPR/dCas9 ベースの ETR の両方を、選択した導入遺伝子送達システムでクローニングします。プラスミドDNA中のETRをクローニングするためのプロトコルについては、以下のステップを参照してください。
注:dCas9:KRAB、dCas9:DNMT3A、およびdCas9:DNMT3L30を個別にコードするプラスミドは、Addgeneでは入手できません。これらは、プラスミドpAC154-dual-dCas9VP160-sgExpression41( 資料表参照)のVP160トランスアクチベーターを、KRAB、DNMT3A、またはDNMT3Lドメインコード配列30のいずれかに置き換えてクローニングしました。CRISPRoff-v2.132と呼ばれるオールインワンETR用のプラスミドエンコーディングが利用可能です( 材料表を参照)。- 化学的に有能な 大腸菌 細胞のETRをコードする形質転換プラスミド(メーカーの指示に従う)。制限酵素消化およびサンガーシーケンシングにより、ETR含有プラスミドの存在についてコロニーをスクリーニングし、最後にプラスミドDNAミディプレップ産生用の陽性コロニーの1つを選択します(メーカーの指示に従ってください)。
- phU6-gRNAバックボーン内のgRNAをクローニングします(図3)。
- 分子生物学デザインソフトウェアを使用して、プロトスペーサーの上流に5'-CACCG-3'配列(選択したgRNAの最初の可変20ヌクレオチド[nt])を付加し、SGfwと呼ばれる25nt長のin silicoオリゴを生成します。
- 同様に、選択したgRNAのプロトスペーサーの逆補体の上流に5'-AAAC-3'配列を付加し、その下流に5'-C-3'配列を付加して、 SGrvと呼ばれる25 nt長のオリゴを生成します。
- SGfw 配列と SGrv 配列の両方を、100 μM の水に再懸濁した無塩一本鎖 DNA オリゴとして注文します。
- 各オリゴ1 μLを2 μLのアニーリングバッファー(10 mM Tris [pH 7.5-8.0]、50 mM NaCl、1 mM EDTA)および16 μLの水に加えます。
- 95°Cで10分間開始するようにプログラムされたサーモサイクラーに溶液を入れて、オリゴアニーリングを行います。その後、45分かけて25°Cまで徐々に冷却します。
- アニーリングしたオリゴ 1 μL を 99 μL のヌクレアーゼフリー水で希釈し、次にこの希釈液 1 μL を 、以前に BsaI 制限酵素で消化した 50 ng の phU6-gRNA プラスミドでライゲーションします(消化およびライゲーション手順については、 BsaI およびリガーゼキットベンダーの指示に従ってください)。
- 20 μLの化学的にコンピテントな 大腸菌 細胞を2 μLのライゲーション産物で形質転換します(形質転換手順については、メーカーの指示に従ってください)。
- プラスミドDNAミニプレップ産生のために複数のコロニーを選び(ベンダーの指示に従ってください)、U6プロモーター5'-GAGGGCCTATTTCCCATGATT-3'に一致する以下のプライマーを用いてサンガーシーケンシングにより、プロトスペーサーのクローニングを成功させます。
- プラスミドDNAミディプレップ産生用のポジティブコロニーの1つを選択します(メーカーの指示に従ってください)。
- 選択したgRNAおよびCRISPR/dCas9ベースのETRをレポーター細胞株にアレイで導入します(条件ごとに1つの特異的gRNA)(図3)。
注:代表的なケースとして、B2MTdTomato K-562細胞のB2M CpGアイランドを標的とするdCas9:KRAB、dCas9:DNMT3A、dCas9:DNMT3L、およびgRNAをコードするプラスミドのヌクレオフェクションのワークフローを以下のステップに示します。- dCas9:KRAB-、dCas9:DNMT3A-、および dCas9:DNMT3L をコードするプラスミドをそれぞれ 500 ng ずつ入れた別々のチューブを調製しますが、試験する gRNA によって異なります(チューブあたり 125 ng の gRNA コードプラスミド)。gRNAおよびETRフリーの核酸発現条件を模擬処理サンプルとして含めます。サンプルごとに少なくとも 3 つのテクニカル反復でスクリーニングを実行します。
- チューブあたり5×10個のB2M TdTomatoK-562 細胞5個をペレット化し、プラスミドミックスで核形成します(メーカーの指示に従ってください)。
- 温めた哺乳類細胞培養培地200 μLに細胞を再懸濁し、インキュベーターに戻します。
4. 標的遺伝子の転写活性の経時解析
- フローサイトメトリーを使用して、ETR送達後のさまざまな時点でサイレンシングされた細胞の割合を測定します(図4)。蛍光色素をコードする配列を持たない野生型(WT)細胞を使用して、レポーター陰性細胞の閾値を設定します。モック処理したサンプルを使用して、レポーター陽性細胞のゲートを設定します。
注:ステップ1.3で提案されているように、実験に遺伝的破壊コントロールを含めます。これは、CRISPR送達効率と標的遺伝子を奪われた細胞の適応度の両方を監視するのに役立ちます。転写サイレンシングと遺伝的破壊の両方が時間の経過とともに失われるのは、選択した細胞タイプにおける標的遺伝子の必須性に起因する可能性があります。短期(3日目、7日目、10日目)と長期(21日目、35日目)の両方の時点を含めて、サイレンシングの急性および長期の両方の効率を示します。 - 長期的なサイレンシング効率の観点から上位 3 つの gRNA を特定します。FACSを使用して、これらのサンプルで安定的に維持されるレポーター陰性のサブポピュレーションを選択します。また、バルク模擬処理されたサンプルのFACSを実行して、テストサンプルと同じ処理下に保持し、その後の分析で適切な比較を可能にします。
5. RNA-seqおよびメチル化DNA免疫沈降(MeDIP)-seqによるETR処理の特異性評価
- RNA-seqを使用して、ETR導入時の最終的なゲノムワイドな転写脱制御を評価します。
- 最も性能の高い 3 つの gRNA で処理したサンプルのレポーターサイレンシングサブポピュレーションとモック処理細胞の両方について、市販のキットを使用して RNA を抽出します。市販のキットを使用してRNAの品質と濃度を評価します。
- RNA-seqライブラリを調製するための市販のキットを使用して、RNAフラグメンテーション、レトロトランスクリプション、およびライブラリ調製を行います(メーカーの指示に従ってください)。
- 次世代シーケンシングおよびデジタル電気泳動に対応した品質管理装置を使用して、ライブラリの定量と品質管理を行います。
- 次世代シーケンサーのシーケンスライブラリは、メーカーの指示に従い、100 bpのペアエンドプロトコルを使用し、平均45 Mリード/サンプルを目標としています。
- リードタグを適切なリファレンスゲノムにアライメントし、転写産物の発現を定量します。tdTomato配列のアライメントを行い、個別に定量します。
注:ここでは、STARアライナー(v 2.3.0)43は、デフォルトパラメータで、Rsubreadパッケージ44に結合されて使用されます。 - RNA-seqデータの解析は、公表されているベストプラクティス45に従って行う。
注:ここでは、R/BioconductorパッケージedgeR46を使用して、少なくとも3つのサンプルで100万あたり少なくとも1カウント(cpm)のフィルターを適用して、低発現遺伝子を破棄します。または、edgeR の filterByExpr 関数を使用することもできます。 - edgeR (関数 glmFit)47 を実装した負の二項一般化対数線形モデルを使用して、遺伝子発現の差を評価します。調整済み p 値(Benjamini-Hochberg[BH]補正)に閾値を0.01に設定して、差異的に制御された遺伝子を保持します。
- MeDIP-seq による ETR の最終的なオフターゲット CpG メチル化活性を評価します。
- 上位 3 つの gRNA で処理したサンプルのレポーターサイレンシングサブポピュレーションとモック処理した細胞の両方について、市販のキットを使用してゲノム DNA を抽出します(メーカーの指示に従ってください)。
- 超音波装置と以下のパラメータを使用して500ngのゲノムDNAを超音波処理する: デューティ:20%;ピップ:175;バーストあたりのサイクル数:200;時間:40秒。
- MeDIP-seq用の市販キットでシーケンシングライブラリを調製します(メーカーの指示に従ってください)。
- アダプターライゲーションステップの後、蛍光アッセイでライブラリを定量し、市販のライブラリ定量キットを使用してqPCRでライゲーション効率を確認します(メーカーの指示に従ってください)。
- ランダムに選択されたライブラリを混在させることでライブラリプールを取得し、技術的な偏りを減らします。各ライブラリーに等しいライブラリー量 (ng) を使用して、プールのバランスを取ります。各プールに、キットに同梱されているメチル化および非メチル化スパイクインコントロール DNA を添加します。コントロールの目的で、ライブラリの10%の容量を保持し、「インプット」とラベル付けし、免疫沈降させないでください。MeDIP-seqキットに同梱されている5-メチルシトシンに対するモノクローナル抗体を用いて、ライブラリーの残りの90%に対して免疫沈降を行います。
- 5-メチルシトシン免疫沈降産物の精製キットを使用して、濃縮ライブラリとインプットライブラリをメーカーの指示に従って精製します。
- キットに付属のプライマーを使用して、コントロールの内部スパイクに対して定量的リアルタイムPCRを実施することにより、濃縮効率を評価します。各免疫沈降(IP)について、MeDIPのサイクル閾値(Ct)値とqPCR反応から得られたインプットフラクションを使用して、メチル化および非メチル化DNAの回収率から濃縮特異性を計算します(式1および式2を参照)。
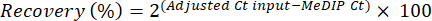 (1)
(1)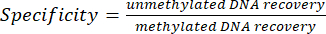 (2)
(2)
メモ : 特異度の値が ≥0.95 の場合、IP ライブラリは正常であると考えてください。 - MeDIP-seqライブラリ調製キットを用いて、メーカーの指示に従いライブラリを増幅し、製品の定量およびライブラリサイズ分布解析を行います。
- 次世代シーケンサーでライブラリシーケンシングを実行します。リード長 100 bp のペアエンドシーケンシングを使用し、平均 30 M リード/サンプルを目指します。
- bwa(v 0.7.5以上)48を用いてシーケンシングリードタグを適切な参照ゲノム(例えば、hg38)にアライメントし、次にMACS(v 2.0.10以上)49を用いてピークを同定し、ブロードピーク(-slocal = 0,-llocal = 500000)の同定を可能にする。
- BEDToolsの多重交差ツール50を使用して、異なるサンプルから領域の共通セットを作成し、クラスタリングオプションを有効にします。
- BEDTools の multicov を使用して最終領域リストのサンプルごとのカバレッジを計算し、重複した読み取りを破棄します。
- edgeR を使用して行列数の解析を実行します。少なくとも 3 つのサンプルで 1 00 万あたりのカウント (cpm) のフィルターを適用して、エンリッチの低い領域を破棄します。
注: または、edgeR の filterByExpr 関数を使用することもできます。 - edgeR (関数 glmFit) に実装された一般化対数線形モデルを採用し、条件付き分位点正規化51 を使用して正規化して領域ごとの GC 含有量を補正することにより、メチル化の差を特定します。BH調整 p 値に0.01の閾値を適用することにより、メチル化差領域を選択します。繰り返しシーケンスの解析は、次のように実行します。
注: オプションとパラメーターの完全なリストについては、edgeR ユーザー ガイドを参照してください (https://bioconductor.org/packages/release/bioc/html/edgeR.html)。- MeDIP-seq の結果を公称 p 値 <0.01 でフィルター処理し、2 つの領域セットを作成します: 最初のセットに logFC >1 を持つ領域を選択し、2 番目のセットに logFC <-1 を持つ領域を選択します。
- 選択したゲノムのRepeatMaskerアノテーションをベッドファイルとして取得し、各セット内の要素数をカウントします。各データセットのリージョン数に対する比率としてカウントを変換します。
- リピートの各クラスが重複するメチロームの比率を抽出し、カイ二乗検定を実行して有意な濃縮を検出します。
- ETR処理サンプルとモック処理サンプル間の転写またはメチル化の差異領域が、 in silicoで予測されたオフターゲットgRNA結合にマッピングされるかどうかを評価します。
- CRISPR design suite52をオフターゲットgRNA結合予測ツールとして使用します。
- すべての推定オフターゲット領域について、最も近い転写開始部位(TSS)と最も近いメチル化領域を調べます。FDR <0.01 で制御され、TSS までの距離が 10 Kb 未満の遺伝子に関連する領域、または FDR <0.01 で制御され、距離が 1 Kb 未満のメチル化領域のいずれかに関連する領域を真のオフターゲット効果として考えます。
- モック処理したサンプルと比較して、最も性能の高い 3 つの gRNA で処理したサンプルのそれぞれで転写変化または過剰メチル化された領域の数と特徴を同定し(図 5)、gRNA の中で最も特異的なものを特定します。標的細胞の生理機能に対するオフターゲット部位の潜在的な影響を、次の順序でランク付けします(最も影響の大きいものから影響の少ないものへ)。
i) 遺伝子内調節領域生理学的発現遺伝子
ii) 遺伝子内エクソン領域生理学的発現遺伝子
iii) 遺伝子内イントロニック領域生理学的発現遺伝子
iv)遺伝子内調節領域-発現していない遺伝子
v)遺伝子内、エクソン領域-発現していない遺伝子
vi)遺伝子内、イントロニック領域-発現していない遺伝子
vii)遺伝子間領域
結果
CRISPR/Cas9システムと結合した標的遺伝子座への蛍光レポーターの相同組換え媒介統合のためのドナーテンプレートを送達すると(例えば、K-562細胞の場合はプラスミドヌクレオフェクションによって)、レポーター陽性細胞が処理されたサンプルに現れます(図1、下)。これが起こらない場合は、ドナーテンプレートとCRISPR/Cas9試薬の両方のデザインとクローニングの精度を再確認してください。確認された場合は、試薬の用量と送達プロトコル自体を最適化してみてください。
レポーター細胞株が得られたら、標的遺伝子のプロモーター/エンハンサー配列を選択し、一致するgRNAのパネルを設計します。Chopchopなどの in silico 予測ツールを使用して、gRNA配列を同定し、予測効率と特異性の観点からランク付けします(図2)。gRNAが回収されない場合は、標的配列中の標準的なCas9プロトスペーサー隣接モチーフ(PAM)(5'-NGG-3')の存在を制御します。PAM 配列が存在しない場合は、PAM 非依存の代替 Cas9 変異体に基づく ETR に移行するか(これらの変異体ではまだ ETR は公開されていません)、ZFP53 や TALEs30 などの代替 DNA 結合ドメインプラットフォームへの切り替えを検討してください。ただし、有効性/特異性が予測される低いgRNAのみが回収される場合は、1)これらの低品質のgRNAをテストするか、2)より良いgRNAを求めて標的DNA配列を拡張することを検討してください。トリプルETRの組み合わせ(またはCRISPRoff;v2.1)とgRNAの一過性送達後、蛍光レポーターの発現について縦断的フローサイトメトリー解析を行い、多くの場合、急性解析でレポーター抑制のピークを観察し、その後、ETRをコードするプラスミドの有糸分裂希釈により少なくとも部分的に再吸収されます(図4C)).ETR/gRNAの組み合わせが標的遺伝子座にCpGメチル化を効果的に沈着させる場合、レポーターの永久的な抑制は、処理された細胞のかなりの部分で起こります(図4C)。gRNAが異なれば、長期的なサイレンシング効率も異なります(図4B、C)。
ゲノムワイドな特異性評価には、MeDIP-seqを使用して、ターゲットが長期間サイレンシングされた細胞と未処理の細胞の間のメチル化領域を同定します。理想的には、特異性の高いgRNAは、標的部位でのみ de novoCpG メチル化のピークを誘導します(図5)。そうでない場合は、オンターゲット効率リストの下位にランクされているgRNAのオフターゲット活性を特徴付けることを検討できます。
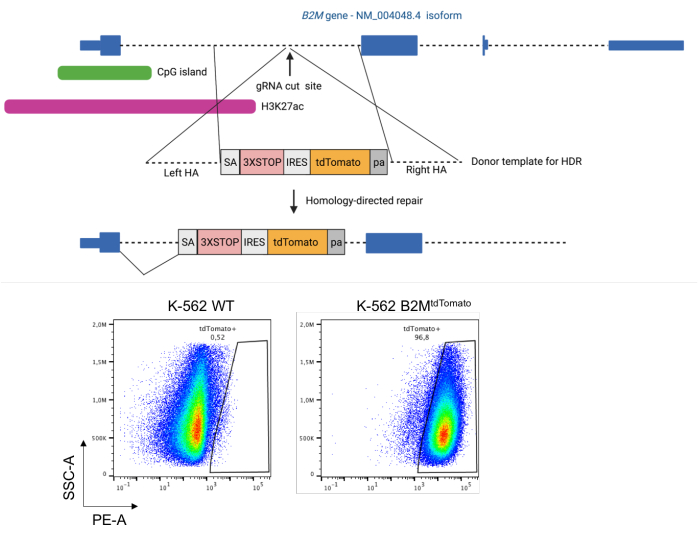
図1:相同性指向性修復によるヒト B2M 遺伝子の調節要素下でのtdTomatoレポーターの統合。 上:CRISPR/Cas9誘導相同性指向性修復により、ヒト B2M 遺伝子の最初のイントロンにtdTomato蛍光レポーターを組み込む戦略の概略図。下: B2M 遺伝子の最初のイントロンにおけるtdTomatoレポーターのインテグレーション前後のK-562細胞の代表的なドットプロット。略語:HA =相同性アーム;HDR = 相同性指向性修復;IRES = 内部リボソーム侵入部位;pa = BGHポリ(A);SA = スプライス アクセプター サイト;WT = 野生型;3XSTOP = 3つのタンデム終止コドン。 この図の拡大版をご覧になるには、ここをクリックしてください。
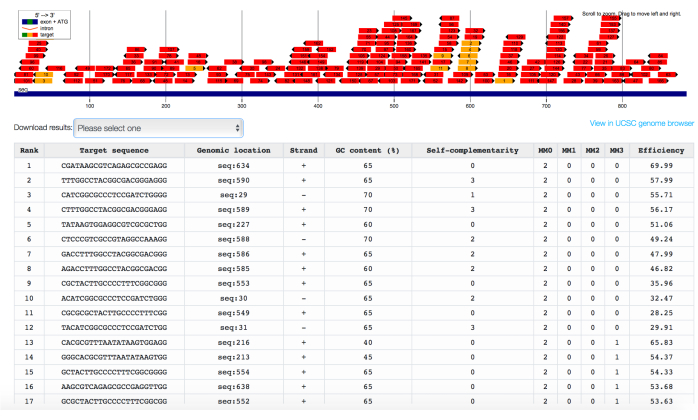
図2:B2M遺伝子のCpGアイランドを標的とするgRNAのin silico同定、予測される効率と特異性でランク付け。B2M遺伝子のプロモーター配列に埋め込まれたCpGアイランドを標的とするgRNAを示すChopchopの出力インターフェースは、0(MM0)、1(MM1)、2(MM2)、または3(MM3)の不一致を持つオフターゲット配列の数と予測されるオンターゲット効率の両方でランク付けされています。この図の拡大版をご覧になるには、ここをクリックしてください。
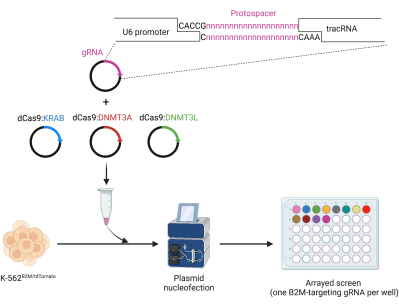
図3:dCas9 ETRを介したエピジェネティックサイレンシングのためのgRNAのクローニングとアレイヌクレオフェクション。 上:プラスミドを発現するヒトU6-gRNAにおけるオリゴ媒介プロトスペーサークローニング。下:K-562B2M/tdTomato細胞におけるプラスミドヌクレオフェクションによるCRISPR/dCas9ベースのエピジェネティックサイレンシングのためのB2M標的gRNAのアレイスクリーン。この図の拡大版をご覧になるには、ここをクリックしてください。
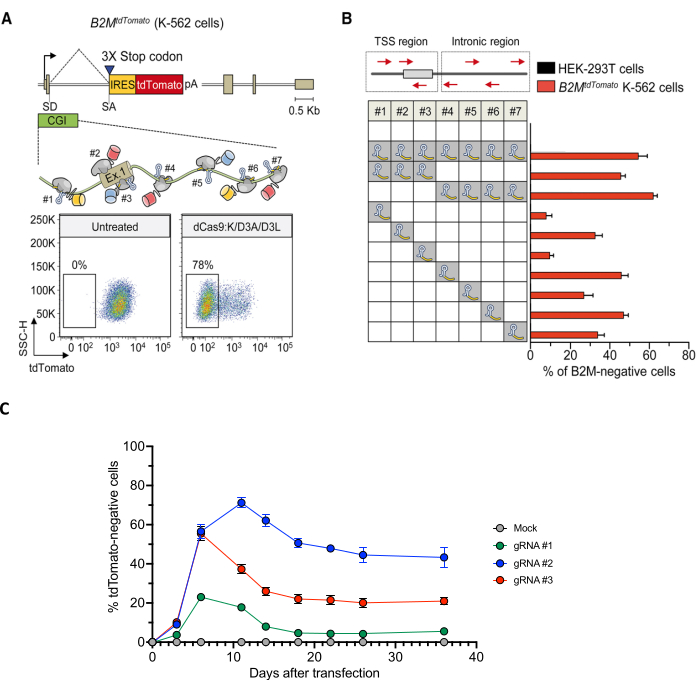
図4:B2M遺伝子の長期サイレンシングを効果的に誘導するgRNAのスクリーニング。 (A)上:拡大領域に描かれたB2MtdTomato遺伝子の模式図、gRNAと複合体を形成したdCas9ベースのETRの結合の相対的な順序と配向。下:ETRサイレンシング前(左)または後(右)のB2MtdTomato K-562細胞の代表的なドットプロット。トランスフェクション後30日で、gRNAとトリプルdCas9:KRAB+ dCas9:D3A + dCas9:D3Lの組み合わせをコードするプラスミドを用いて分析しました。(B)トランスフェクション後30日目のK-562 B2MtdTomato細胞におけるB2MのCpGアイランド(上図の赤い矢印はgRNAの配向を示す)を標的とする示されたgRNA(プール内または個々のgRNAとして)のサイレンシング活性。データは、tdTomato陰性細胞の割合を示しています(SEM±平均;n = 各処理条件で4つの独立したトランスフェクション)。(C)トリプルETRの組み合わせを発現するプラスミドと、示されたB2M CpGアイランドターゲティングgRNAまたはモックを用いたトランスフェクション時のB2MtdTomato K-562細胞の経時的解析(gRNAおよびETRフリートランスフェクション)。データは、tdTomato陰性細胞の割合を示しています(SEM±平均;n = 各処理条件で3つの独立したトランスフェクション)。未公開データ。パネルAとBは、Amabile et al.30から引用した。略語:CGI = CpG島;IRES = 内部リボソーム侵入部位;pa = BGHポリ(A);SA = スプライス アクセプター サイト;SD = スプライス ドナー サイト。TSS = 転写開始部位;UT = トランスフェクトなし;3XSTOP = 3つのタンデム終止コドン。この図の拡大版をご覧になるには、ここをクリックしてください。
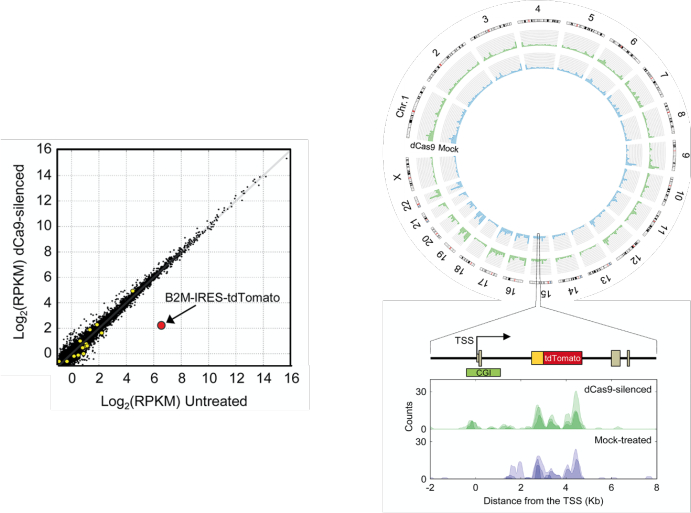
図5:RNA-seqおよびMeDIP-seqによるETR特異性のゲノムワイド解析。 左:模擬処理したB2MtdTomato K-562細胞と、トリプルdCas9:KRAB、dCas9:DNMT3A、dCas9:DNMT3L ETRの組み合わせとB2M遺伝子のCpGアイランドを標的とするgRNAで処理した細胞の発現量の比較。値は、マッピングされた読み取りの RPKM (キロベース/百万キロベースあたりの読み取り数) の log2 で表されます。黒い点は、すべての条件で同等のレベルで発現している遺伝子を表します。黄色の円は、FDR <0.01;赤丸はB2M-IRES-tdTomato転写産物。右上:模擬処理したB2MtdTomato K-562細胞(青)またはトリプルdCas9:KRAB、dCas9:DNMT3A、dCas9:DNMT3L ETRの組み合わせとB2M遺伝子のCpGアイランド(CGI)を標的とするgRNAで処理した細胞(緑)の全ゲノムMeDIP-seqプロファイルを示すサーカスプロット。右下:示されたサンプル中のB2MtdTomato遺伝子座のメチル化状態を示す。各パイルアップには 3 つの反復が表されます。アライメントされた読み取りのパイルアップは、ガウスウィンドウを使用して平滑化されました。この図は、Amabile et al.30 から引用したものです。略称:TSS = transcription start site。この図の拡大版をご覧になるには、ここをクリックしてください。
ディスカッション
標的エピジェネティックサイレンシングは、機能獲得型変異によって引き起こされる疾患1、感染症2、およびある遺伝子のサイレンシングが別の遺伝子の遺伝性欠陥を補う3か、養子細胞療法の可能性を最大限に引き出す4病状など、永久的な遺伝子不活性化の恩恵を受けることができる疾患を治療するための有望な解決策となる可能性があります。5.クロマチンレベルで作用し、細胞7,30,32によって自動増殖することにより、エピジェネティックサイレンシングは、標的遺伝子のDNA配列の毒性変化(例えば、染色体再構成)および標的の部分的で一過性のサイレンシングを回避することができ、これらはそれぞれ人工ヌクレアーゼベースの遺伝子破壊8,34,35およびRNAiベースのノックダウン33の限界である。
エピジェネティックサイレンシングプロトコルにおける重要な予備ステップの1つは、ターゲットの転写活性をオフにするために必要な抑制的なエピジェネティックマークを沈着させるETRを指示するために、ターゲット遺伝子上の適切な位置を特定することです。異なる転写開始部位-近位および遠位の調節要素は、所与のヒト遺伝子の転写出力を支持するために一致し得る54。さらに、プログラム可能なDNA結合技術の数が増えているおかげで、特定の調節要素ごとに異なる標的部位を同定できるようになりました6。したがって、遺伝子組換え細胞株でここに記載されているようなプロトコルを使用して、最終的な治療環境で最良の候補の煩雑で時間のかかる評価作業に着手する前に、ETRを介したエピジェネティックサイレンシングに適した個々の標的部位および/またはゲノム領域を指名することができます。プロトコルのいくつかの重要な側面について、以下で詳しく説明します。
最終治療標的を予測するレポーター細胞株のエンジニアリング
蛍光レポーターのコードするカセットを標的遺伝子に挿入し、ETRを送達するために、細胞工学プロトコルの絶え間ない最適化が行われているにもかかわらず、最終的な治療標的に最もよく似た細胞株でETRがすでに利用可能であることは当然のこととは考えられません。この場合、異なる緩和戦略を適用することができます:a)ベンダーが提供する最適化キットを利用して、標的細胞株のトランスフェクションプロトコルを社内で最適化する。b)工学プロトコルが広範囲に最適化されている最終治療標的以外の組織に属する、まだその標的遺伝子を発現しているが、他の細胞株に切り替える。これらの選択肢が利用できない場合は、最終標的となる初代細胞タイプまたはオルガノイドへの切り替えを検討することができます。ETRベースのエピジェネティックサイレンシングの前臨床試験と治療応用の両方における一般的な考慮事項として、標的遺伝子が標的細胞タイプに必須であるシナリオは破棄する必要があります。ETRによって課せられた必須遺伝子の完全かつ長期的なサイレンシングは、時間の経過とともに標的細胞の逆選択につながります(そして、潜在的には治療の毒性)。このような場合には、RNAi33などの部分的な標的除去を提供する代替技術が好まれます。
ETRのオンターゲットサイレンシング活性の評価
KRAB、DNMT3A、およびDNMT3Lエフェクタードメインの組み合わせに基づくETRは、転写開始部位を中心とする約1キロ塩基の広いロングパーミッシブターゲティングウィンドウにより、タンパク質をコードする遺伝子の大部分に対して有効であることが証明されています32。エピジェネティックなサイレンシング実験がどのようなものかを理解するために、K-562細胞における B2M 遺伝子のサイレンシングの技術的な詳細と結果をここに掲載します。これは、K-562細胞を扱う研究者だけでなく、ETRベースの技術に初めてアプローチする研究者にも含まれる重要なポジティブコントロールと見なすことができます。プロトコルに記載されているように、人工ヌクレアーゼ(CRISPR/Cas9など)ベースの遺伝子破壊は、目的の細胞タイプにおける遺伝子送達の効率と、標的遺伝子を奪われた細胞の表現型の両方の追加の制御として推奨されます。CRISPR/dCas9ベースのETRと結合するgRNAの最初のスクリーニング後、テストされたgRNAのいずれも標的遺伝子を永久にサイレンシングできない場合は、次の順序で検討する必要があります:1)送達されるgRNAとETRの量を増やす。2)上位gRNAのプールをテストし、それらの間の相乗効果を探します。それでも長期的なサイレンシングが達成されない場合は、次のことを検討する必要があります:3)エピジェネティックサイレンシングを指示するためにより関連性の高い部位を標的とする可能性のある追加のgRNAのテスト。4)ZFP8またはTALE9ベースのDBDプラットフォームに切り替えると、標的クロマチンへの結合能力が向上する可能性があります。5)一過性から安定への切り替え-例えば、ETR(CRISPR/dCas9技術を採用する場合は、gRNAまたはdCas9融合コンストラクトのいずれか、またはその両方)のウイルスベクターベースの発現を統合する。私たちのグループは、3つの別々のETR30の共同デリバリーを開発し、豊富な経験を持っているため、ここに示したプロトコルと結果は、このアプローチに基づいています。しかしながら、同様の概念的ワークフローは、オールインワンCRISPRベースのシステム32にも適用され得る可能性が高い。
ETRのオフターゲット活性の評価
複数の研究により、KRAB、DNMT3A、およびDNMT3Lエフェクタードメインの組み合わせに基づくETRの特異性の予備的なin vitro適応症が示されています30,31,32。しかし、試験したgRNAの中で、転写調節および/またはde novoDNAメチル化に関して満足のいく特異性プロファイルを示さない場合は、2つの非相互に排他的な戦略を追求することができます:a)ETRの用量を減らすか、代替送達システムをテストすることにより、細胞内のETRの滞留時間(およびその結果、潜在的なオフターゲット活性)を短縮します。例えば、プラスミドと比較して、mRNAとタンパク質の送達はどちらも、ETRへの細胞曝露時間を短縮し、その結果、オフターゲット活性の可能性を低減することが期待されます55。b)プラットホーム56のオフターゲット結合を減少させるように最適化された、より最近のCas9変異体への切り替え、または代替的なZFP8−またはTALE9ベースのDNA結合技術への切り替え。模擬処理されたサンプルと比較して、遺伝子サイレンシングのオンターゲットおよびオフターゲット活性は、DBDの標的配列への結合だけでなく、エピジェネティックエフェクタードメインが天然の内因性補因子によって他の遺伝子座にリクルートされる潜在的な能力によっても影響を受けることを考慮することが重要です。したがって、標的細胞におけるETRの滞留時間を短縮すると、DBDがオフターゲット部位に結合する可能性が低下するだけでなく、ETRが内因性補因子と相互作用する可能性も低下し、特異性の点でメリットがあり、オンターゲット活性の点でデメリットが生じる可能性があります。最後に、模擬処理されたサンプルと比較して、サイレンサー細胞で測定された転写および可能性の低いCpGメチル化変化の一部は、標的遺伝子の欠乏によって単純に導き出すことができます。これらは、サイレンシング技術のオフターゲットとは見なされません。それらを同定するには、人工ヌクレアーゼによる遺伝子破壊も実験パネルに含める必要があります8,9,10。標的遺伝子の機能喪失による生物学的変化は、エピジェネティックサイレンシングとこの代替技術の間で共有されます。
開示事項
ALは、Chroma Medicine, Inc.の共同設立者、クォータ保有者、コンサルタントです。
謝辞
著者らは、長年にわたるエピジェネティックサイレンシング技術の開発に協力してくれたAngelo Amabile氏、Paola Capasso氏、Ilaria Caserta氏、Tania Baccega氏、Alice Reschigna氏、Valeria Mollica氏、Deborah Cipria氏に感謝します。Dejan Lazarevic と Francesca Giannese は、プロトコルに記載されている RNA-seq および MeDIP-seq 解析の批判的レビューについて述べています。この研究は、Telethon Foundation(TIGET助成金番号F1)およびEU Horizon 2020プログラム(UPGRADE)からのA.L.への助成金によって支援されました。イラストは BioRender.com で作成されています。
著者の貢献
A.M.、M.A.C.、F.G.、およびA.C.は、プロトコルの設計と原稿の作成に貢献しました。S.V.、I.M.、およびD.C.は、プロトコルのバイオインフォマティクスセクションを設計し、原稿を改訂しました。A.M.とA.L.は、すべての著者の意見を取り入れてプロトコルを設計し、原稿を考案し、執筆しました。
資料
| Name | Company | Catalog Number | Comments |
| 4200 TapeStation System | Agilent | G2991BA | DNA quantification |
| 4D-Nucleofector X Unit | Lonza Bioscience | AAF-1003X | Nucleofection |
| B2M silencing gRNA #1 | Lombardo's lab | GCAATCAGGACAAGGCCCGC | Gene silencing |
| B2M silencing gRNA #2 | Lombardo's lab | GGGGTAGGAGAGACTCACGC | Gene silencing |
| B2M silencing gRNA #3 | Lombardo's lab | GAGTCCAGGGCTGGATCTCG | Gene silencing |
| BD FACSAria Fusion Flow Cytometer | BD Biosciences | https://www.bdbiosciences.com/en-us/products/instruments/flow-cytometers/research-cell-sorters/bd-facsaria-fusion | Fluorescence Activated Cell Sorting |
| bedtools | Bedtools | http://bedtools.readthedocs.io/en/latest/ | Processing of genomic intervals |
| bwa | Ih3 | https://github.com/lh3/bwa | Alignment of MeDIP-seq reads |
| Chopchop | Valen's lab | http://chopchop.cbu.uib.no/ | gRNA selection software |
| Corning RPMI 1640 Medium (Mod.) 1x with L-Glutamine | Corning | 10-040-CV | Cell culture |
| cqn | Bioconductor | http://bioconductor.org/packages/release/bioc/html/cqn.html | Region-wise normalization by GC-content |
| CRISPR design suite | Zhang's lab | https://zlab.bio/resources-2 | off-target gRNA binding prediction |
| CRISPRoff-v2.1 plasmid | Addgene | 167981 | Gene silencing |
| CytoFLEX S V4-B4-R3-I2 Flow Cytometer | Beckman Coulter | C01161 | Flow cytometry |
| Donor template sequence for tdTomato integration in the first intron of the B2M gene | Lombardo's lab | ctcctcctctgacctgtgtgtgggttttgtttttgtttt | Genetic engineering |
| E220 Focused-ultrasonicator | Covaris | 500239 | DNA sonication |
| edgeR | Bioconductor | https://bioconductor.org/packages/release/bioc/html/edgeR.html | Differential abundance testing |
| EnGen Mutation Detection Kit | NEB | E3321 | Gene disruption quantification |
| Fetal Bovine Serum | Sigma-Aldrich | F2442 | Cell culture |
| Flowjo | BD Biosciences | https://www.flowjo.com/solutions/flowjo | Flow cytometry data analysis software |
| Gel Loading Dye, Purple (6x) | NEB | B7024S | DNA gel loading |
| Go Taq G2 Hot Start DNA Polymerase | Promega | M7401 | PCR amplification |
| gRNA sequence for dTomato integration in the first intron of the B2M gene | Lombardo's lab | AGGCTACTAGCCCCATCAAG | Genetic engineering |
| hCas9 plasmid | Addgene | 41815 | Genetic engineering |
| High Sensitivity D1000 Reagents | Agilent | 5067-5585 | DNA quantification |
| High Sensitivity D1000 ScreenTape | Agilent | 5067-5584 | DNA quantification |
| High Sensitivity RNA ScreenTape | Agilent | 5067-5579 | RNA quantification |
| High Sensitivity RNA ScreenTape Ladder | Agilent | 5067-5581 | RNA quantification |
| High Sensitivity RNA ScreenTape Sample Buffer | Agilent | 5067-5580 | RNA quantification |
| IPure kit | Diagenode | C03010011 | Purification of the 5-methylcytosine immunoprecipitation product |
| K-562 cells | ATCC | CCL-243 | Cell engineering |
| KAPA Library Quantification Kit | Roche | KK4824 | MeDIP-Seq libraries preparation |
| MACS2 | Taoliu | https://github.com/macs3-project/MACS | Identification of methyl-enriched regions |
| MagMeDIP kit | Diagenode | C02010020 | 5-methylcytosine immunoprecipitation |
| NextFlex Methylseq kit 1 | Bioo Scientific | 5118-01 | MeDIP-Seq libraries preparation |
| NextSeq 500 / NovaSeq 6000 | Illumina | SY-415-1002 / 20012850 | Next Generation Sequencing |
| NucleoBond Xtra Midi kit for transfection-grade plasmid DNA | Macherey-nagel | 740410.50 | Midiprep plasmid preparation |
| One Shot TOP10 Chemically Competent cells | ThermoFisher | C404010 | Plasmid transformation |
| pAC154-dual-dCas9VP160-sgExpression plasmid | Addgene | 48240 | Gene activation |
| pcDNA.CMV.dCas9:KRAB plasmid | Lombardo's lab | available on request (lombardo.angelo@hsr.it) | Gene silencing |
| pcDNA.CMV.dCas9:KRAB plasmid | Lombardo's lab | available on request (lombardo.angelo@hsr.it) | Gene silencing |
| pcDNA.CMV.dCas9:KRAB plasmid | Lombardo's lab | available on request (lombardo.angelo@hsr.it) | Gene silencing |
| Penicillin/Streptomycin | Sigma-Aldrich | P0781 | Cell culture |
| phU6.sgRNA plasmid | Addgene | 53188 | Genetic engineering |
| PowerPac Basic Power Supply | Biorad | 1645050 | Agarose gel electrophoresis |
| Primer forward sequence to check tdTomato integration in the first intron of the B2M gene | Lombardo's lab | GTATTTGCTGGTTATGTTAG | Genetic engineering |
| Primer reverse sequence to check tdTomato integration in the first intron of the B2M gene | Lombardo's lab | AATGGTTGAGTTGGAC | Genetic engineering |
| QIAamp DNA Mini Kit | Qiagen | 51304 | DNA extraction |
| Qubit | Thermo Fisher | Q33238 | DNA quantification |
| Qubit dsDNA HS (high sensitivity) Assay Kit | Thermo Fisher | Q32851 | DNA quantification |
| Qubit RNA HS (high sensitivity) Assay Kit | Thermo Fisher | Q32852 | RNA quantification |
| Restriction enzymes | NEB | DNA digestion | |
| RNeasy Mini kit | Qiagen | 74106 | RNA extraction |
| Rsubread | Bioconductor | https://bioconductor.org/packages/release/bioc/html/Rsubread.html | Quantification of gene expression |
| SF Cell Line 4D-Nucleofector X Kit S (32 RCT) | Lonza Bioscience | V4XC-2032 | Nucleofection |
| SnapGene | Dotmatics | https://www.snapgene.com/ | Molecular biology design software |
| STAR | Alexander Dobin | https://github.com/alexdobin/STAR | Alignment of RNA-seq reads |
| T100 Thermal Cycler | Biorad | 1861096 | PCR amplification |
| T4 DNA Ligase | Promega | M1801 | DNA ligation |
| TAE Buffer | Fisher scientific | BP1332500 | Agarose gel electrophoresis |
| TruSeq Stranded Total RNA kit | Illumina | 20020597 | RNA-Seq library preparation |
| UltraPure Agarose | ThermoFisher | 16500500 | Agarose gel |
参考文献
- Cummings, C. J., Zoghbi, H. Y. Fourteen and counting: unraveling trinucleotide repeat diseases. Human Molecular Genetics. 9 (6), 909-916 (2000).
- Tebas, P., et al. Gene editing of CCR5 in autologous CD4 T cells of persons infected with HIV. The New England Journal of Medicine. 370 (10), 901-910 (2014).
- Frangoul, H., et al. CRISPR-Cas9 gene editing for sickle cell disease and β-thalassemia. The New England Journal of Medicine. 384 (3), 252-260 (2021).
- Murty, T., Gene Mackall, C. L. Gene editing to enhance the efficacy of cancer cell therapies. Molecular Therapy. 29 (11), 3153-3162 (2021).
- Lanza, R., Russell, D. W., Nagy, A. Engineering universal cells that evade immune detection. Nature Reviews Immunology. 19 (12), 723-733 (2019).
- Matharu, N., Ahituv, N. Modulating gene regulation to treat genetic disorders. Nature Reviews Drug Discovery. 19 (11), 757-775 (2020).
- Sgro, A., Blancafort, P. Epigenome engineering: New technologies for precision medicine. Nucleic Acids Research. 48 (22), 12453-12482 (2020).
- Urnov, F. D., Rebar, E. J., Holmes, M. C., Zhang, H. S., Gregory, P. D. Genome editing with engineered zinc finger nucleases. Nature Reviews Genetics. 11 (9), 636-646 (2010).
- Joung, J. K., Sander, J. D. TALENs: A widely applicable technology for targeted genome editing. Nature Reviews Molecular Cell Biology. 14 (1), 49-55 (2013).
- Adli, M. The CRISPR tool kit for genome editing and beyond. Nature Communications. 9 (1), 1911 (2018).
- Gilbert, L. A., et al. XCRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes. Cell. 154 (2), 442 (2013).
- Snowden, A. W., Gregory, P. D., Case, C. C., Pabo, C. O. Gene-specific targeting of H3K9 methylation is sufficient for initiating repression in vivo. Current Biology. 12 (24), 2159-2166 (2002).
- Chen, X., et al. Construction and validation of the CRISPR/dCas9-EZH2 system for targeted H3K27Me3 modification. Biochemical and Biophysical Research Communications. 511 (2), 246-252 (2019).
- Kwon, D. Y., Zhao, Y. T., Lamonica, J. M., Zhou, Z. Locus-specific histone deacetylation using a synthetic CRISPR-Cas9-based HDAC. Nature Communications. 8, 15215 (2017).
- Stepper, P., et al. Efficient targeted DNA methylation with chimeric dCas9-Dnmt3a-Dnmt3L methyltransferase. Nucleic Acids Research. 45 (4), 1703-1713 (2017).
- Ecco, G., Imbeault, M., Trono, D. KRAB zinc finger proteins. Development. 144 (15), 2719-2729 (2017).
- Witzgall, R., O'leary, E., Leaf, A., Onaldi, D., Bonventre, J. The Kruppel-associated box-A (KRAB-A) domain of zinc finger proteins mediates transcriptional repression. Proceedings of the National Academy of Sciences. 91 (10), 4514-4518 (1994).
- Mannini, R., et al. Structure/function of KRAB repression domains: Structural properties of KRAB modules inferred from hydrodynamic, circular dichroism, and FTIR spectroscopic analyses. Proteins: Structure, Function and Genetics. 62 (3), 604-616 (2006).
- Friedman, J. R., et al. KAP-1, a novel corepressor for the highly conserved KRAB repression domain. Genes and Development. 10 (16), 2067-2078 (1996).
- Iyengar, S., Farnham, P. J. KAP1 protein: An enigmatic master regulator of the genome. The Journal of Biological Chemistry. 286 (30), 26267-26276 (2011).
- Schultz, D. C., Friedman, J. R., Rauscher, F. J. Targeting histone deacetylase complexes via KRAB-zinc finger proteins: The PHD and bromodomains of KAP-1 form a cooperative unit that recruits a novel isoform of the Mi-2α subunit of NuRD. Genes and Development. 15 (4), 428-443 (2001).
- Schultz, D. C., Ayyanathan, K., Negorev, D., Maul, G. G., Rauscher, F. J. SETDB1: A novel KAP-1-associated histone H3, lysine 9-specific methyltransferase that contributes to HP1-mediated silencing of euchromatic genes by KRAB zinc-finger proteins. Genes and Development. 16 (8), 919-932 (2002).
- Nielsen, A. L., et al. Interaction with members of the heterochromatin protein 1 (HP1) family and histone deacetylation are differentially involved in transcriptional silencing by members of the TIF1 family. The EMBO Journal. 18 (22), 6385-6395 (1999).
- Sripathy, S. P., Stevens, J., Schultz, D. C. The KAP1 corepressor functions to coordinate the assembly of de novo HP1-demarcated microenvironments of heterochromatin required for KRAB zinc finger protein-mediated transcriptional repression. Molecular and Cellular Biology. 26 (22), 8623-8638 (2006).
- Jurkowska, R. Z., Jurkowski, T. P., Jeltsch, A. Structure and function of mammalian DNA methyltransferases. ChemBioChem. 12 (2), 206-222 (2011).
- Jia, D., Jurkowska, R. Z., Zhang, X., Jeltsch, A., Cheng, X. Structure of Dnmt3a bound to Dnmt3L suggests a model for de novo DNA methylation. Nature. 449 (7159), 248-251 (2007).
- Tajima, S., Suetake, I., Takeshita, K., Nakagawa, A., Kimura, H. Domain structure of the Dnmt1, Dnmt3a, and Dnmt3b DNA methyltransferases. Advances in Experimental Medicine and Biology. 945, 63-86 (2016).
- Greenberg, M. V. C., Bourc'his, D. The diverse roles of DNA methylation in mammalian development and disease. Nature Reviews Molecular Cell Biology. 20 (10), 590-607 (2019).
- Ishiyama, S., et al. Structure of the Dnmt1 reader module complexed with a unique two-mono-ubiquitin mark on histone H3 reveals the basis for DNA methylation maintenance. Molecular Cell. 68 (2), 350.e7-360.e7 (2017).
- Amabile, A., et al. Inheritable silencing of endogenous genes by hit-and-run targeted epigenetic editing. Cell. 167 (1), 219.e14-232.e14 (2016).
- Mlambo, T., et al. Designer epigenome modifiers enable robust and sustained gene silencing in clinically relevant human cells. Nucleic Acids Research. 46 (9), 4456-4468 (2018).
- Nuñez, J. K., et al. Genome-wide programmable transcriptional memory by CRISPR-based epigenome editing. Cell. 184 (9), 2503.e17-2519.e17 (2021).
- Davidson, B. L., McCray, P. B. Current prospects for RNA interference-based therapies. Nature Reviews Genetics. 12 (5), 329-340 (2011).
- Haapaniemi, E., Botla, S., Persson, J., Schmierer, B., Taipale, J. CRISPR-Cas9 genome editing induces a p53-mediated DNA damage response. Nature Medicine. 24 (7), 927-930 (2018).
- Kosicki, M., Tomberg, K., Bradley, A. Repair of double-strand breaks induced by CRISPR-Cas9 leads to large deletions and complex rearrangements. Nature Biotechnology. 36 (8), 765-771 (2018).
- Ciccia, A., Elledge, S. J. The DNA damage response: making it safe to play with knives. Molecular Cell. 40 (2), 179-204 (2010).
- Hu, J. H., et al. Evolved Cas9 variants with broad PAM compatibility and high DNA specificity. Nature. 556 (7699), 57-63 (2018).
- Uhlén, M., et al. Proteomics. Tissue-based map of the human proteome. Science. 347 (6220), (2015).
- Labun, K., et al. CHOPCHOP v3: Expanding the CRISPR web toolbox beyond genome editing. Nucleic Acids Research. 47 (W1), W171-W174 (2019).
- Kent, W. J., et al. The Human Genome Browser at UCSC. Genome Research. 12 (6), 996-1006 (2002).
- Cheng, A. W., et al. Multiplexed activation of endogenous genes by CRISPR-on, an RNA-guided transcriptional activator system. Cell Research. 23 (10), 1163-1171 (2013).
- Kabadi, A. M., Ousterout, D. G., Hilton, I. B., Gersbach, C. A. Multiplex CRISPR/Cas9-based genome engineering from a single lentiviral vector. Nucleic Acids Research. 42 (19), e147 (2014).
- Dobin, A., et al. STAR: ultrafast universal RNA-seq aligner. Bioinformatics. 29 (1), 15-21 (2013).
- Liao, Y., Smyth, G. K., Shi, W. The Subread aligner: fast, accurate and scalable read mapping by seed-and-vote. Nucleic Acids Research. 41 (10), e108-e108 (2013).
- Conesa, A., et al. A survey of best practices for RNA-seq data analysis. GenomeBiology. 17, 13 (2016).
- Robinson, M. D., Oshlack, A. A scaling normalization method for differential expression analysis of RNA-seq data. Genome Biology. 11 (3), R25 (2010).
- Robinson, M. D., McCarthy, D. J., Smyth, G. K. edgeR: a Bioconductor package for differential expression analysis of digital gene expression data. Bioinformatics. 26 (1), 139-140 (2010).
- Li, H., Durbin, R. Fast and accurate long-read alignment with Burrows-Wheeler transform. Bioinformatics. 26 (5), 589-595 (2010).
- Zhang, Y., et al. Model-based analysis of ChIP-Seq (MACS). Genome Biology. 9 (9), R137 (2008).
- Quinlan, A. R. BEDTools: The Swiss-Army tool for genome feature analysis. Current Protocols in Bioinformatics. 47, (2014).
- Hansen, K. D., Irizarry, R. A., Wu, Z. Removing technical variability in RNA-seq data using conditional quantile normalization. Biostatistics. 13 (2), 204-216 (2012).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nature Biotechnology. 31 (9), 827-832 (2013).
- Zeitler, B., et al. Allele-selective transcriptional repression of mutant HTT for the treatment of Huntington's disease. Nature Medicine. 25 (7), 1131-1142 (2019).
- Andersson, R., Sandelin, A. Determinants of enhancer and promoter activities of regulatory elements. Nature Reviews Genetics. 21 (2), 71-87 (2020).
- Liang, X., et al. Rapid and highly efficient mammalian cell engineering via Cas9 protein transfection. Journal of Biotechnology. 208, 44-53 (2015).
- Han, H. A., Pang, J. K. S., Soh, B. -. S. Mitigating off-target effects in CRISPR/Cas9-mediated in vivo gene editing. Journal of Molecular Medicine. 98 (5), 615-632 (2020).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved