Method Article
使用 ARL13B-CENTRIN-2 小鼠模型对肌肉骨骼组织中原代纤毛进行体积成像和分析
摘要
该方法描述了在 ARL13B-CENTRIN-2 转基因小鼠模型中利用内源信号对初级纤毛进行 原位成像,从组织收集到纤毛组织分析,在大面积矿化组织中进行三维成像。该方案也适用于其他类型的组织。
摘要
初级纤毛是由大多数细胞类型组装的非运动、孤立的细胞器。它对于骨骼的发育和体内平衡以及协调细胞对来自周围环境的生化和机械线索的反应至关重要。由于其微米级尺寸,初级纤毛很难以高通量方式在组织中进行 原 位成像表征,且无光学偏差。了解其 在体内 3D 环境中的组织将是理解纤毛在生理学和疾病中的作用的关键。ARL13B-CENTRIN-2 小鼠系在睫状轴丝蛋白 ARL13B 上呈上呈 mCherry 标签,在中心粒蛋白 CENTRIN-2 上呈绿色荧光蛋白 (GFP) 标签,位于纤毛基部。该小鼠系无需染色即可对初级纤毛进行成像,从而减少了图像采集的步骤数并解决了信噪比问题。本出版物描述了一种分步方案,用于在保留荧光信号的同时对肌肉骨骼组织中的初级纤毛进行成像。它还描述了一个普遍适用的图像分析管道,能够对纤毛特征(如长度和 3D 方向)进行无偏量化。
引言
初级纤毛是许多组织和器官(包括骨骼)发育和稳态中必不可少的细胞器。在出生后生长过程中,它调节软骨内骨化过程中的软骨矿化1。在人类中,影响纤毛相关蛋白的基因突变会导致一系列疾病和发育障碍,称为纤毛病。了解组织内初级纤毛的组织对于了解它们如何在器官水平调节细胞行为非常重要。初级纤毛的发生率、长度和方向已被证明在组织内部和组织之间有所不同,这表明组织组织对其功能的作用 2,3,4。生长板是长骨末端的软骨层,从早期到青春期,大多数产后骨骼生长发生在此,是一种高度组织化的组织,软骨细胞细胞呈柱状排列。在生长板中,软骨细胞经历分化阶段,从静止到增殖再到肥大。
这种细胞组织在一些纤毛病中丢失,这表明初级纤毛可能在协调这种细胞组织和/或至少与细胞极化有关。此外,初级纤毛方向似乎在其机械感觉功能中发挥作用,在关节软骨的承重区域和非承重区域之间观察到差异4。我们自己最近的研究表明,关节软骨和生长板的不同区域存在不同的作用 5,6。以前对生长板中的初级纤毛进行成像的研究仅限于 2D,这丢失了与组织中纤毛组织相关的信息,或者仅限于 3D 中的少量细胞,这降低了分析的规模和功效 7,8。
对初级纤毛进行成像的挑战包括其微米级大小和免疫组化抗体的非特异性。使用天然荧光信号可确保强大的定位和特异性。然而,天然荧光信号在矿化组织中可能难以成像,因为将组织包埋在石蜡中的漫长脱钙过程会导致信号丢失。在天然状态下冷冻的样品冷冻切片通过加快包埋和切片过程来防止这种损失。
为了对原代纤毛进行原位成像,Bangs 等人生成了双转基因小鼠系,该细胞系表达与 mCherry 融合的 ADP-核糖基化因子样蛋白 (ARL13B) 和与 GFP9 融合的 CENTRIN-2。ARL13B 是一种定位于初级纤毛膜的小 GTP 酶,而 CENTRIN-2 是一种定位于纤毛基部两个中心体的中心粒蛋白10,11。因此,该小鼠系无需免疫组织化学即可对荧光标记的初级纤毛进行成像。
此处描述的方案利用该转基因系用共聚焦显微镜在小鼠出生后生长板中对初级纤毛进行 3D 成像,只需要额外的 4',6-二脒基-2-苯基吲哚 (DAPI) 染色。数百个细胞中的数百个初级纤毛可以在组织环境中进行集体成像和分析。随后,图像分析管道测量生长板不同区域的初级纤毛入射、长度和方向。该管道还能够量化中心粒的这些特征。图 1 显示了协议中的步骤概述。
研究方案
畜牧业和实验是根据牛津大学道德框架和英国内政部授予的项目许可进行的。该方案在 2 至 10 周龄的小鼠身上进行,但将在此年龄以上的小鼠软骨中起作用。
1. 组织采集
- 通过在装有小鼠的盒子中填充二氧化碳(体积高达 80%)并保持其浓度 5 分钟来对小鼠实施安乐死。进行颈椎脱位术。
- 解剖出感兴趣的组织。
注意:以下是解剖后腿膝盖的方案,包括胫骨和股骨生长板以及关节软骨。- 切开脚部的皮肤,切开到大腿顶部。用镊子从切口线上去除皮肤。
- 修剪腿部的肌肉和脂肪组织。
- 要将腿从身体上分离,请干净利落地切开臀部顶部的股骨。切开胫骨底部的脚。
- 去除腿部残留的过多肌肉和脂肪组织。
2. 组织固定和包埋
注:在整个实验步骤中,请注意将组织置于黑暗中,以避免荧光信号损失。
- 在室温下将组织固定在 10% 福尔马林中 4 小时。
- 将组织在 4 °C 下转移到 2.5% 福尔马林中过夜。
- 将组织转移到蒸馏水 (dH2O) 中的 30% 蔗糖中,并在室温下轻轻旋转过夜或在 4 °C 下旋转 3 天。 将试管填充到顶部,以确保在旋转过程中组织完全浸入。孵育后,组织将沉到试管底部。
- 将少量干冰放入单独的盒子中,并将冷冻模具放在顶部。描画一条超级冷冻包埋介质 (SCEM) 线,并将肢体沿其放置,膝盖骨朝下,将胫骨和股骨分开,使腿尽可能平坦。用镊子固定到位,直到 SCEM 开始凝固并且肢体在不按住时不再移动。
- 用 SCEM 完全覆盖,将模具填充到顶部。将样品放入干冰盒中,保持样品平整以避免组织移动,直到 SCEM 完全凝固。
- 转移至 -20 °C。
3. 切片
- 从模具中取出块并修剪成适合低温恒温器卡盘的尺寸。通过在卡盘上应用 SCEM 并将块放在顶部,将块连接到卡盘上。
- 在 -20 °C 下在低温恒温器中凝固至少 5 分钟。
- 将卡盘连接到样品架上,并将刀片连接到刀架上。
- 修剪块,直到到达胫骨生长板。理想情况下,胫骨朝上的截面。
- 将冷冻胶片胶带剪成适合样品的尺寸,并为其周围留出足够的空间。将冷冻膜安装到切割面上,确保冷冻膜与安装工具紧密粘合。切割 40-60 μm 厚的切片以获得两细胞厚的切片。用镊子收集胶带并放在载玻片上,样品面朝上,胫骨生长板与载玻片的短载玻片对齐。
- 将样品载玻片储存在 -20 °C。
4. 幻灯片准备和安装
- 在室温下水平解冻载玻片。
- 用一滴 PBS 洗涤载玻片 3 x 5 分钟以去除 SCEM。
- 用 10 μM DAPI 染色 1 分钟。
- 用一滴 PBS 洗涤载玻片 2 x 5 分钟。
- 安装,盖上盖玻片,在室温下放置过夜。用盖玻片密封剂密封盖玻片的边缘。
- 第二天的图片。
5. 共聚焦显微镜成像
注意:在所使用的共聚焦显微镜上,40x/1.4 物镜的分辨率为 240 nm。
- 设置显微镜。
- 打开显微镜并打开软件。点击 System 按钮。
- 在 Acquisition 选项卡下,单击 Smart Setup 以设置渠道。选择并添加 GFP、mCherry 和 DAPI 通道。在 Airyscan 三角形中选择 Airyscan 选项和 SR8Y 功能(在分辨率和速度多路复用 8Y 之间)。点击 Best Signal |正常 (补充图 S1A)。
- 在 Locate 选项卡的 Microscope Control 下,选择 油浸的 40x/1.4 物镜。
- 找到感兴趣的区域。
- 将载玻片放入载玻片架中,然后向上移动物镜,直到油接触到盖玻片。
- 将生长板或其他感兴趣的区域放置在光路下方。
- 在 定位 选项卡中,单击 荧光,选择 DAPI,然后使用目镜,用纵杆移动载玻片支架,将生长板或感兴趣区域定位在视野下。
- 在 Acquisition 选项卡中,首先仅选择 DAPI 通道 。
- 单击 Continuous (连续 ) 在屏幕上查看该通道的图像,并确保生长板在视图中。
- 通过单击 Tiles 选项卡中的 Show viewer 打开 Tile 查看器。添加所需的图块数量(通常在 x 方向上 1 个图块,在 y 方向上 3-5 个图块),然后单击预览和开始(补充图 S1B)。
- 检查瓦片是否覆盖了生长板的三个区域 - 休息、增殖和肥大。根据需要调整 平铺位置 和 数量 。
- 设置成像参数。
- 单击 Acquisition 选项卡中的 Continuous。在 Channels 选项卡中,调整 Gain (Master) 和 Laser power 以在屏幕上清晰地查看原子核。允许 Airyscan 检测器对齐。通过单击窗口底部的平铺玫瑰花结符号打开 Airyscan Detector Adjustment 窗口;对齐后,所有图块都将变为绿色(补充图 S1C)。
- 分别选择 GFP 和 mCherry 通道并重复这些步骤 - 设置 增益(主)、激光功率并对齐 Airyscan 检测器。
- 选择所有三个通道,然后单击 Continuous。允许 Airyscan 检测器对齐。
- 在 Acquisition Mode 选项卡中,定义 Image Size、Pixel、Size、Frame Size、Speed 和 Averaging 值。有关所用通道的 Acquisition Mode 设置、Gain (Master) 和 Laser power 的值,请参见补充图 S1D,E。
- 获取图像。
- 在 Acquisition 选项卡中,单击 Z-Stack 复选框。
- 仅选择 DAPI 通道后,单击 Continuous 在屏幕上查看图像。将物镜向下移动到最近的要成像的平面。在 Z-Stack 选项卡中,单击 Set First plane,将焦点移开到要查看的最后一个平面,然后单击 Set Last plane。图像 30-40 μm z 堆栈。
- 将 间隔 设置为 0.15 μm。
- 移动到 z 堆栈的中心。在瓷砖选项卡的瓷砖区域下,点击验证并点击设置Z并移至下一步。
- 在 Focus Strategy 选项卡下,检查焦点策略是否设置为 Use Z Values/Focus Surface 在 Tiles Setup 中定义。
- 单击 Start Experiment 开始图像采集。
- 图像处理
- 在 Processing 选项卡,选择 Single 或 Batch 模式。选择 Airyscan processing。勾选 3D processing 并选择 Standard Autofilter。选择获取的原始图像并开始处理(补充图 S2A)。
- Airyscan 处理完成后,在处理方法中选择 拼合 。选择 New Output、Fuse Tiles、Correct Shading 和 All by reference,并将参考作为 DAPI。选择 Airyscan 处理后的图像并开始处理(补充图 S2B)。
- 保存 Raw 和双重处理的图像。后者会自动保存在 Batch 模式下。
6. 图像分析
注意:此处使用的分析管道是根据本研究的目的定制的。管道作的详细信息如 补充图 S3 所示。
- 将 czi 图像文件导入软件。打开分析面板;在下拉列表中选择 New Pipeline...
- 要创建细胞检测管道,请将 Cellpose Python Segmenter 导入管道12。将 Model_Name 设置为 cyto2,将 Input_channel 设置为 2,将 Second_channel 设置为 3,将 Diameter_in_ μm 设置为 10,将 Flow_threshold 设置为 0.4,将 Cellprob_threshold 设置为 0。通过单击 + Add Operation,在管道之前添加 Import Document Objects(重命名为 Import Manual Labels),并在管道之后添加 Object Feature Filter(重命名为 Size Filter)运算符。有关管道设置的值,请参阅补充图 S3A。
- 将查看器移动到第一个感兴趣的平面
- 单击 Draw Objects Tool 图标
- 单击 Polygon mode 图标
- 绘制要分析的区域
- 将查看器移动到最后一个感兴趣的平面
- 绘制要分析的区域
- 单击绿色对号 应用更改
- 单击 Show Objects Table(显示对象表 )图标
- 右键单击创建的对象,然后选择 Rename Object (重命名对象 ) 以将对象重命名为适当的名称。右键单击创建的对象,然后单击 Add Tag。添加名为 Analysis Region 的标签。
- 通过单击 Analysis (分析) 面板顶部的向前蓝色箭头来运行此管道。
- 要创建初级纤毛和中心粒检测管道,请打开分析面板,在下拉列表中选择 New Pipeline...。将以下运算符添加到管道中: Import Document Objects (重命名为 Import Manual Labels)、两个 Intensity Threshold Segmenter 运算符(重命名为 Cilia Threshold 和 Centriole Threshold)、 Splitting (重命名为 Centriole Splitting)、 Object Feature Filter (重命名为 Centriole Splitting 大小过滤器)和 Compartment (重命名为 Compartment - Cell)。有关管道设置的值,请参见 补充图 S3B 。
- 通过单击 Analysis (分析) 面板顶部的向前蓝色箭头来运行此管道。
- 打开 Objects (对象) 窗口。单击 Im/Export...,选择 Excel Export...。选择要保存的以下功能: # 子项, 名称, 父项名称, 3D 定向边界 短边, 中边, 长边, 角度 XY, 角度 XZ, 角度 YZ, 边界框 X1, X2, Y1, Y2, Z1, Z2, 大小 X, 大小 Y, 大小 Z, 几何中心 X, Y, Z, 平面第一, 平面 最后, 计数, 球度 (网格), 体积 (网格), 表面积 (网格)。
结果
使用描述的方案对 6 周龄小鼠的生长板进行成像,并在组织中映射初级纤毛组织。捕获生长板的所有三个区域(静止、增殖和肥大)的图像,并通过分析管道运行(图 2A)。可以通过 DAPI 染色来识别单个细胞,它们的睫状轴丝为红色,其中心粒为绿色。使用这种方法的分辨率足够高,可以区分单个中心粒(图 2B)。在这种分辨率下,纤毛可以在其 3D 环境中可视化,指向不同的方向(视频 1)。
也可以捕获具有较高放大倍率物镜的较低分辨率图像,以可视化较大组织区域的初级纤毛;但是,这些不适用于 3D 纤毛取向分析。例如,关节软骨中的初级纤毛用 20 倍物镜成像,表明该方法也适用于其他组织(图 2C)。在此分辨率下,可以识别红色的睫状轴丝,但中心粒更难区分。软骨下骨往往呈现大量 GFP 背景信号,使得该组织中中心粒的识别变得困难。
该管道可以检测单个细胞、初级纤毛和中心粒,它适用于高度组织化的年轻生长板和柱不太明确的老年生长板,如 10 周龄小鼠(图 3)。细胞和纤毛以 3D 形式检测,可以在 z 堆栈中查看。
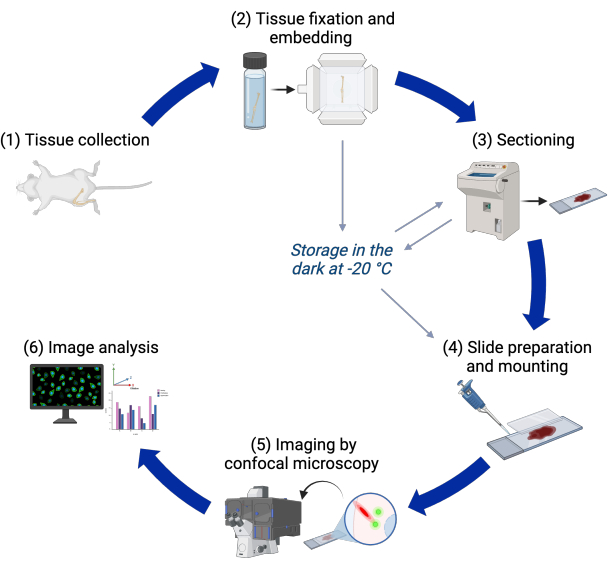
图 1:实验工作流程。 方案中从组织收集到图像分析的不同步骤的流程图。 请单击此处查看此图的较大版本。
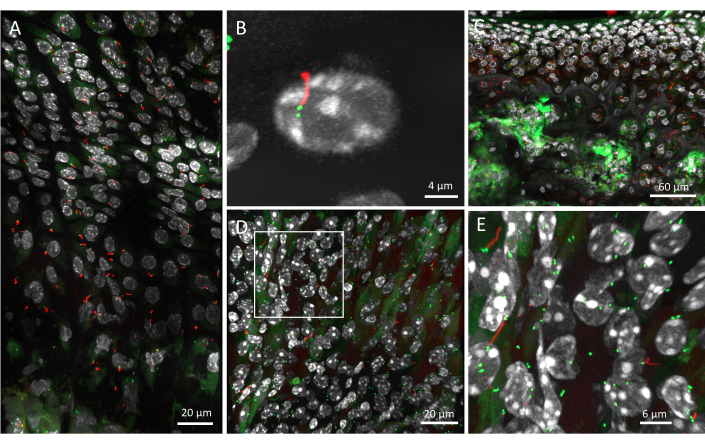
图 2:ARL13B-CENTRIN-2 小鼠小鼠组织中的初级纤毛。 (A) 一个 6 周龄的生长板,DAPI为白色,CENTRIN-2 为绿色,ARL13B 为红色,使用 40 倍物镜成像。(B) 放大单个初级纤毛及其两个中心粒。(C) 用 20 倍物镜成像的 4 周龄小鼠的关节软骨和软骨下骨。(D) 使用与肌肉骨骼组织相同的管道成像的 6 周龄小鼠的肾脏。(E) D 中白框的放大倍率,显示肾脏中单个初级纤毛及其中心粒。比例尺 = 20 μm (A,D)、4 μm (B)、60 μm (C)、6 μm (E)。 请单击此处查看此图的较大版本。
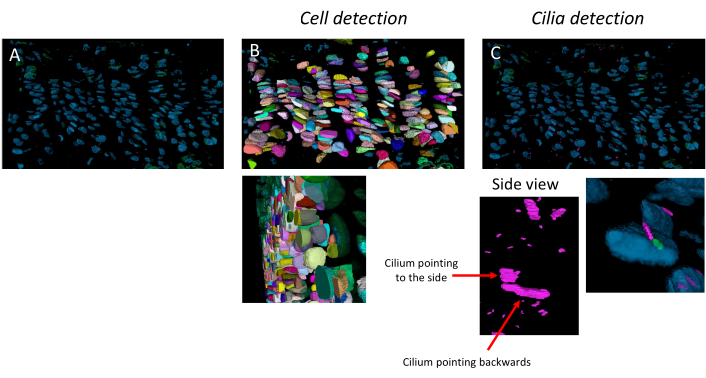
图 3:细胞和纤毛检测管道。 图像分析管道在 10 周龄生长板上的示例应用。(A) 在软件中打开的映像。(B) 细胞检测流程的结果。(C) 纤毛和中心粒检测管道的结果。纤毛为粉红色,中心粒为绿色。侧视图表明,分辨率足够高,可以区分 3D 中朝向不同方向的纤毛。 请单击此处查看此图的较大版本。
视频 1:ARL13B-CENTRIN-2 小鼠 6 周龄生长板的三维重建。 在 Imaris 软件中进行三维重建,并录制为动画视频。 请点击此处下载此视频。
补充图 S1:显微镜系统上的图像采集设置。 (A) 智能设置 窗口,用于添加频道并选择 Airyscan 模式。(B) Tile Viewer 选择并预览要成像的图块。(C) Airyscan 检测器调整 窗口,一旦所有图块都变为绿色,即确认 Airyscan 检测器的对齐。(D) 三个通道的激光设置。(E) 采集模式 和 Z 堆栈设置。 请点击此处下载此文件。
补充图 S2:显微镜系统上的图像处理设置。 (A) Airyscan 处理设置。(B) 拼接设置。 请点击此处下载此文件。
补充图 S3:自定义图像分析管道。 (A) 使用 GFP 和 DAPI 通道的细胞检测管道。(B) 分别使用 mCherry 和 GFP 通道的初级纤毛和中心粒检测管道。 请点击此处下载此文件。
讨论
在该协议中,必须在收集后立即进行组织处理和包埋步骤。然而,组织块和载玻片可以在 -20 °C 下保存至少 3 个月。 可以修改该协议中的几个步骤以适应不同的应用,特别是切片厚度、放大倍率和 z 堆栈参数。这将影响成像的深度和获得的分辨率。数值孔径和分辨率模式的变化可用于获得更高的分辨率,并且 z 堆栈间隔大小减小。但是,这会增加成像时间或减小可成像的视场大小。所描述的成像参数是初级纤毛方向的 3D 分析的最低要求。
封片剂的选择对于获得高分辨率非常重要。封片剂的折射率必须与浸没介质的折射率相似,以最大限度地减少 z 堆栈中的图像退化。此处使用的封片剂和浸油的折射率分别为 1.52 和 1.518。这里使用的共聚焦显微镜上的 Airyscan 功能可提供超分辨率图像。可以使用其他超分辨率显微镜;然而,我们发现仅共聚焦模式不足以获得成像区域大小所需的分辨率。多重 SR-8Y Airyscan 模式的最大分辨率为 x/y 为 120/160 nm,z 轴为 450 nm,最大帧数为 47.5。我们发现了多重 SR-4Y Airyscan 模式,它在 x/y 上的最大分辨率为 140 nm,在 z 轴上的最大分辨率为 450 nm,但成像速度只有一半,最大帧数为 25,在 3D 中对初级纤毛成像没有显着差异。同样,在这款显微镜上提供最高分辨率的 Airyscan 超分辨率 (SR) 模式(x/y 轴为 120 nm,z 轴为 350 nm),成像时间会更长,因为其最大帧数为 4.713。
背景 GFP 信号可以在其他组织(如肌肉)中观察到。为了最大限度地减少这种情况,可以缩小激光器的范围以减少与其他波长的重叠。该系以前用于对胚胎中的原代纤毛、原代细胞培养物、肌纤维和视网膜色素上皮细胞进行成像 9,14,15。我们已经成功地对肾脏、软骨和骨组织中的初级纤毛进行了成像。对于不同的组织,可能需要对成像设置进行一些优化;但是,该冷冻切片方案应确保信号的保留。
双细胞厚切片 (40-60 μm) 足以在生长板中获得 3D 方向信息,因为这允许对整个细胞及其原代纤毛进行成像。对于细胞大于软骨细胞的其他组织,例如脂肪细胞,可能需要更厚的切片来收集细胞群数据。根据所用显微镜的深度分辨率,可能无法对较厚的切片进行深入成像。由于需要时间,组织透明化技术可能会导致内源性 mCherry 和 GFP 信号丢失。对此进行了探索,因为清除方法用于肢体中的其他成像,但在这种情况下发现没有必要。我们在内源性 CD31-RFP 方面的经验表明,清除技术需要仔细、针对样品进行定制和优化。
该 ARL13B-CENTRIN-2 系可以与其他小鼠系杂交,以进一步研究组织中初级纤毛的组织。例如,我们将其与聚集蛋白聚糖-Cre IFT88fl/fl 杂交,以有条件地敲除表达聚集蛋白聚糖的细胞(出生后软骨细胞)中的 IFT88,这是一种初级纤毛蛋白,其缺失会阻止纤毛发生5,6。然后,这些 ARL13B 和 CENTRIN-2 荧光信号可用于测量组织纤毛结构水平的遗传扰动效率以及纤毛组织的任何相关变化。正在进行的工作目前正在做这件事。我们的第一个目标是用这种更稳健的方法量化 IFT88 缺失对纤毛患病率的影响。以前,我们通过使用免疫荧光来衡量这一点,并估计这减少了 20%6。该方法还可以与免疫组织化学相结合,以将初级纤毛与其他蛋白质(例如细胞膜蛋白)结合成像。
ARL13B 上的转基因可能对 ARL13B 表达和初级纤毛长度有影响。以前的研究在小鼠大脑的某些区域测量了与非转基因 ARL13B 相比,ARL13B-CENTRIN-2 系测量了不同的初级纤毛长度,在其他区域没有差异16。另一项研究观察到 GFP 标记的 ARL13B 小鼠品系与未标记的小鼠胚胎成纤维细胞中的 ARL13B 相比,初级纤毛长度存在差异10。正在进行的工作是通过比较使用 ARL13B-CENTRIN-2 小鼠系与针对非转基因 ARL13B 的抗体染色的结果来验证生长板和其他组织中初级纤毛长度的任何变化。
该协议中的图像分析管道旨在随后测量初级纤毛的纤毛百分比、长度和 3D 方向,以及生长板中的中心粒数量和位置。图像分析软件允许通过更改所涉及的步骤来制作自定义管道。此管道的输出是一个电子表格,其中包含检测到的细胞、纤毛和中心粒的所选特征的测量值。然后可以根据研究的目的在 R 或其他软件中进行后分析。与成像设置一样,可以根据研究的目的更改管道步骤和特定设置以处理不同的图像。总体而言,该方法允许对初级纤毛进行比以前在生长板中发表的高通量分析。可以研究的细胞和相关纤毛的数量允许进行更大、更可靠的测量。
披露声明
作者没有需要声明的利益冲突。
致谢
我们感谢肯尼迪风湿病研究所的所有 BSU 工作人员,特别是 Albertino Bonifacio 的畜牧业。我们感谢牛津-蔡司生物医学成像卓越中心在使用显微镜方面的帮助,特别是 Jacky (Ka Long) Ko 博士在开发图像分析管道方面的帮助。这项工作得到了肯尼迪风湿病研究学生奖学金 (Johnson) 和生物技术与生物科学研究委员会 (BBSRC, BB/X007049/1) 的支持。
材料
| Name | Company | Catalog Number | Comments |
| 22 x 50 mm cover slips | fisherscientific | 12373128 | |
| arivis Vision4D software | Zeiss | N/A | Image analysis software |
| Confocal laser scanning microscope | Zeiss | Zeiss 980 Airyscan 2 | |
| CoverGrip Coverslip Sealant | Biotium | 23005 | |
| Cryofilm type 3C(16UF) (sheet type) 2.0 cm width | Section-lab | C-FUF303 | To be purchased from Section-lab directly |
| Cryostat | Leica | CM1900 UV | |
| DAPI | invitrogen | D1306 | |
| Disposable Base Molds | epredia | 58952 | |
| Dulbecco's Phosphate Buffered Saline | gibco | 14190 | |
| Formalin | Sigma | 20260630 | |
| Imaris software | Oxford Instruments | N/A | Image analysis software for 3D reconstruction |
| Immersion Oil 518F | Zeiss | ISO 8036 | |
| Microtome blades | Feather | 02.075.00.006 | |
| SlowFade Glass Antifade Mountant | invitrogen | S36917 | |
| Sucrose | Sigma | S9378 | |
| Super Cryoembedding Medium (SCEM) | Section-lab | C-EM001 | To be purchased from Section-lab directly |
| Superfrost Plus Microscope slides | avantor | 631-0108 | |
| Zen software | Zeiss | N/A | Freeware for Confocal laser scanning microscopy |
参考文献
- Wheatley, D. N., Wang, A. M., Strugnell, G. E. Expression of primary cilia in mammalian cells. Cell Biol Int. 20 (1), 73-81 (1996).
- Donnelly, E., Ascenzi, M. G., Farnum, C. Primary cilia are highly oriented with respect to collagen direction and long axis of extensor tendon. J Orthop Res. 28 (1), 77-82 (2010).
- McGlashan, S. R., et al. Mechanical loading modulates chondrocyte primary cilia incidence and length. Cell Biol Int. 34 (5), 441-446 (2010).
- Farnum, C. E., Wilsman, N. J. Orientation of primary cilia of articular chondrocytes in three-dimensional space. Anat Rec. 294 (3), 533-549 (2011).
- Coveney, C. R., et al. The ciliary protein IFT88 controls post-natal cartilage thickness and influences development of osteoarthritis. Arth Rheumatol. 74 (1), 49-59 (2021).
- Coveney, C. R., et al. Ciliary IFT88 protects coordinated adolescent growth plate ossification from disruptive physiological mechanical forces. J Bone Miner Res. 37 (6), 1081-1086 (2022).
- Andrea, C. E. D., et al. Primary cilia organization reflects polarity in the growth plate and implies loss of polarity and mosaicism in osteochondroma. Lab Invest. 90 (7), 1091-1101 (2010).
- Ascenzi, M. -. G., et al. Effect of localization, length and orientation of chondrocytic primary cilium on murine growth plate organization. J. Theor Biol. 285 (1), 147-155 (2011).
- Bangs, F. K., Schrode, N., Hadjantonakis, A. K., Anderson, K. V. Lineage specificity of primary cilia in the mouse embryo. Nat. Cell Biol. 17 (2), 113-122 (2015).
- Larkins, C. E., Aviles, G. D. G., East, M. P., Kahn, R. A., Caspary, T. Arl13b regulates ciliogenesis and the dynamic localization of Shh signaling proteins. Mol Biol Cell. 22 (23), 4694-4703 (2011).
- Higginbotham, H., Bielas, S., Tanaka, T., Gleeson, J. G. Transgenic mouse line with green-fluorescent protein-labeled Centrin 2 allows visualization of the centrosome in living cells. Transgenic Res. 13 (2), 155-164 (2004).
- arivis. . Application Note #47 - Applying Cellpose models in arivis Vision4D. , 1-20 (2021).
- Huff, J., Bergter, A., Luebbers, B. Application Note: Multiplex mode for the LSM 9 series with Airyscan 2: fast and gentle confocal super-resolution in large volumes. Nat Methods. , (2019).
- Ning, K., et al. Cilia-associated wound repair mediated by IFT88 in retinal pigment epithelium. Sci Rep. 13 (1), 8205-8219 (2023).
- Palla, A. R., et al. Primary cilia on muscle stem cells are critical to maintain regenerative capacity and are lost during aging. Nat Commun. 13 (1), 1439-1451 (2022).
- Brewer, K. K., et al. Postnatal dynamic ciliary ARL13B and ADCY3 localization in the mouse brain. Cells. 13 (3), 259-277 (2024).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。