Method Article
ARL13B-CENTRIN-2マウスモデルを用いた筋骨格組織における原発性繊毛の体積イメージングと解析
要約
この方法は、ARL13B-CENTRIN-2トランスジェニックマウスモデルにおける内因性シグナルの利用により、組織収集から繊毛組織の解析まで、石灰化組織の広い領域にわたって一次繊毛 をin situでイメージングする方法を示しています。このプロトコルは、他の種類の組織にも適用できます。
要約
一次繊毛は、ほとんどの細胞タイプによって組み立てられた非運動性の孤立性オルガネラです。これは、骨格の発達と恒常性維持、および周囲の環境からの生化学的および機械的手がかりに対する細胞応答の調整に不可欠です。そのマイクロメートルスケールのサイズにより、一次繊毛は、ハイスループットな方法で、光学バイアスなしで組織内で特性評価するために in situ でイメージングすることが困難です。 in vivo の3D環境でのその組織を理解することは、生理学と疾患における繊毛の役割を理解するための鍵となります。ARL13B-CENTRIN-2マウス系統は、繊毛軸索タンパク質ARL13BにmCherryタグを、繊毛の基部に局在する中心小体タンパク質CENTRIN-2に緑色蛍光タンパク質(GFP)タグを示します。このマウス系統により、染色を必要とせずに一次繊毛のイメージングが可能となり、画像収集のステップ数を減らし、S/N比の問題を回避します。この出版物は、蛍光シグナルを維持しながら筋骨格組織の一次繊毛を画像化するための段階的なプロトコルについて説明しています。また、普遍的に適用可能な画像解析パイプラインについても説明されており、長さや3D方向などの繊毛の特徴を偏りなく定量化できます。
概要
一次繊毛は、骨格を含む多くの組織や器官の発達と恒常性維持に不可欠な細胞小器官です。出生後の成長期には、軟骨内骨化中の軟骨の石灰化を調節します1。ヒトでは、繊毛関連タンパク質に影響を与える遺伝子変異が、繊毛症として知られるさまざまな疾患や発達障害を引き起こします。組織内の一次繊毛の組織化を理解することは、一次繊毛が臓器レベルで細胞の挙動をどのように制御するかを理解する上で重要です。一次繊毛の発生率、長さ、および向きは、組織内および組織間で異なることが示されており、組織組織がその機能に果たす役割を示唆しています2,3,4。成長板は、出生後の骨の成長のほとんどが幼少期から青年期にかけて起こる長骨の端にある軟骨の層であり、軟骨細胞が列状に配置された高度に組織化された組織です。成長プレートでは、軟骨細胞は休息から増殖、肥大まで、分化の段階を経ます。
この細胞組織は、一部の繊毛障害では失われており、これは、一次繊毛がこの細胞組織を調整する役割を果たしている可能性があり、および/または少なくとも細胞分極に関連している可能性があることを示唆しています。さらに、一次繊毛の配向は、それらの機械感覚機能に役割を果たしているようであり、関節軟骨の耐荷重領域と非耐荷重領域の間に違いが観察されます4。私たち自身の最近の研究では、関節軟骨と成長板の異なる領域内で異なる役割が示唆されています5,6。成長プレートで一次繊毛を画像化した以前の研究では、2Dに限定されており、組織内の繊毛組織に関連する情報が失われているか、3Dの少数の細胞に限定されているため、分析の規模と検出力が低下しています7,8。
一次繊毛のイメージングにおける課題の中には、そのマイクロメートルスケールのサイズと免疫組織化学抗体の非特異性があります。ネイティブの蛍光シグナルを使用することで、強力な局在性と特異性が保証されます。しかし、天然の蛍光シグナルは、パラフィンに組織を埋め込むための長い脱灰プロセスによりシグナルが失われる可能性があるため、石灰化組織でのイメージングが困難な場合があります。ネイティブ状態で凍結したサンプルを凍結切片化すると、包埋および切片化プロセスが高速化され、この損失が防止されます。
一次繊毛in situを画像化するために、Bangsらによって、mCherryに融合したADP-リボシル化因子様タンパク質(ARL13B)とGFP9に融合したCENTRIN-2を発現するダブルトランスジェニックマウス系統を作製しました。ARL13Bは一次繊毛膜に局在する小さなGTPアーゼであり、CENTRIN-2は繊毛10,11の基部にある2つの中心体に局在する中心小体タンパク質である。したがって、このマウス系統では、蛍光標識された一次繊毛を免疫組織化学を必要とせずにイメージングすることができます。
ここで説明するプロトコルは、このトランスジェニックラインを利用して、共焦点顕微鏡法でマウスの出生後成長プレートで一次繊毛を3Dで画像化します。必要なのは、追加の4',6-ジアミジノ-2-フェニルインドール(DAPI)染色のみです。数百の細胞に含まれる数百の一次繊毛を、組織状況でまとめてイメージングおよび解析できます。その後、画像解析パイプラインにより、成長プレートのさまざまな領域における一次繊毛の発生率、長さ、および配向が測定されます。パイプラインは、中心小体についてこれらの特徴を定量化することもできます。プロトコルの手順の概要を図 1 に示します。
プロトコル
畜産と実験は、オックスフォード大学の倫理的枠組みに従って、英国内務省から付与されたプロジェクトライセンスの下で実施されました。このプロトコルは、2週齢から10週齢までのマウスで実施されましたが、この年齢を超えてマウスの軟骨で機能します。
1. ティッシュコレクション
- マウスの入った箱に二酸化炭素を体積の80%まで満たし、その濃度を5分間維持することにより、マウスを安楽死させます。頸部脱臼を行います。
- 目的の組織を解剖します。
注:以下は、脛骨と大腿骨の成長板、関節軟骨を含む後肢の膝を解剖するためのプロトコルです。- 足元の皮膚を切開し、太ももの上まで切り込みます。鉗子で切開線から皮膚を取り除きます。
- 脚の筋肉と脂肪組織をトリミングします。
- 脚を体から取り外すには、股関節の上部にある大腿骨をきれいに切り取ります。脛骨の底で足を切ります。
- 脚に残っている余分な筋肉や脂肪組織をすべて取り除きます。
2. 組織の固定と埋め込み
注:プロトコール全体を通して、蛍光シグナルの損失を避けるために、組織を暗所に保つように注意してください。
- 組織を10%ホルマリンに室温で4時間固定します。
- 組織を2.5%ホルマリンに4°Cで一晩移します。
- 組織を蒸留水(dH2O)中の30%スクロースに移し、室温で一晩または4°Cで3日間静かに回転させます。 回転中に組織が完全に浸るように、チューブを上部まで満たします。このインキュベーション後、組織はチューブの底に沈みます。
- 少量のドライアイスを別の箱に入れ、クライオモールドを上に置きます。スーパークライオエンベデッドメディウム(SCEM)のラインをトレースし、それに沿って手足を置き、膝蓋骨を下に向けて脛骨と大腿骨を離して脚をできるだけ平らにします。SCIMが固まり始め、押さえていないときに手足が動かなくなるまで、鉗子で所定の位置に保持します。
- SCIMで完全に覆い、型を上部まで満たします。SCEMが完全に固まるまで、組織の動きを避けるためにサンプルを平らに保ちながら、サンプルをドライアイスボックスに入れます。
- -20°Cに移します。
3. セクショニング
- ブロックを型から取り外し、クライオスタットのチャックに収まるサイズにトリミングします。チャックにSCIMを塗布し、ブロックを上に置いて、ブロックをチャックに取り付けます。
- クライオスタットで-20°Cで少なくとも5分間固化させます。
- チャックをサンプルホルダーに、ブレードをブレードホルダーに取り付けます。
- 脛骨成長板に到達するまでブロックをトリミングします。理想的には、脛骨が上を向いているセクション。
- クライオフィルムテープは、サンプルの周囲に十分なスペースを確保してサンプルにフィットするサイズにカットします。クライオフィルムを切断面に取り付け、フィッティングツールでクライオフィルムがしっかりと接着されるようにします。厚さ40〜60μmの切片を切断して、2セルの厚さ切片を取得します。鉗子を使用してテープを収集し、スライド上、サンプル側を上にして、スライドの短いスライドに合わせて脛骨成長プレートを置きます。
- サンプルスライドは-20°Cで保存してください。
4. スライドの準備と取り付け
- スライドを室温で水平に解凍します。
- スライドをPBSの滴で3 x 5分間洗浄して、SCEMを取り外します。
- 10 μM DAPIで1分間染色します。
- スライドをPBSを一滴で2 x 5分間洗浄します。
- 取り付け、カバースリップで覆い、室温で一晩放置します。カバースリップの端をカバースリップシーラントでシールします。
- 翌日の画像。
5. 共焦点顕微鏡によるイメージング
注:使用した共焦点顕微鏡の40x/1.4対物レンズの解像度は240nmです。
- 顕微鏡をセットアップします。
- 顕微鏡の電源を入れ、ソフトウェアを開きます。 [システム ]ボタンをクリックします。
- 「Acquisition」タブで、「Smart Setup」をクリックしてチャネルを設定します。GFP、mCherry、および DAPI チャネルを選択して追加します。AiryscanオプションとAiryscanトライアングル(解像度と速度多重8Yの間)のSR8Y機能を選択します。[Best Signal]をクリックします。OK(補足図S1A)。
- 「Locate」タブの「Microscope Control」で、油浸の40x/1.4対物レンズを選択します。
- 対象地域を特定します。
- スライドをスライドホルダーに置き、オイルがカバーガラスに触れるまで対物レンズを持ち上げます。
- 成長プレートまたはその他の関心領域を光路の下に配置します。
- [ Locate ]タブで、[ Fluorescence]をクリックし、[ DAPI]を選択し、接眼レンズを使用して、ジョイスティックでスライドホルダーを動かして、成長プレートまたは関心領域を視野の下に配置します。
- [Acquisition]タブで、DAPIチャネルのみを選択して開始します。
- [Continuous]をクリックして、そのチャネルの画像を画面に表示し、成長プレートが表示されていることを確認します。
- [Tiles] タブの [Show viewer] をクリックして、Tile Viewer を開きます。必要なタイルの数(通常はx方向に1タイル、y方向に3〜5タイル)を追加し、[プレビューと開始]をクリックします(補足図S1B)。
- タイルが成長プレートの3つのゾーン(休止、増殖、肥大)をカバーしていることを確認します。必要に応じて 、タイルの位置 と 数 を調整します。
- イメージングパラメータを設定します。
- [Acquisition] タブで [Continuous] をクリックします。[チャンネル]タブで、[ゲイン(マスター)]と[レーザー出力]を調整して、核が画面上にはっきりと見えるようにします。Airyscan Detectorの位置を合わせます。Airyscan Detector Adjustmentウィンドウを開くには、ウィンドウの下部にあるタイル状のロゼット記号をクリックします。整列すると、すべてのタイルが緑色に変わります(補足図S1C)。
- GFPチャンネルとmCherryチャンネルを個別に選択し、ゲイン(マスター)、レーザー出力を設定し、Airyscan検出器の位置を合わせる手順を繰り返します。
- 3つのチャンネルをすべて選択し、[Continuous]をクリックします。Airyscan Detectorの位置を合わせます。
- [取り込みモード] タブで、[画像サイズ]、[ピクセル]、[サイズ]、[フレーム サイズ]、[速度]、および [平均化] の値を定義します。アクイジションモード設定、ゲイン(マスター)、および使用されるチャンネルのレーザーパワーの値については、補足図S1D、Eを参照してください。
- 画像を取得します。
- [Acquisition] タブで、[Z-Stack] チェックボックスをクリックします。
- DAPIチャンネルのみを選択した状態で、[ Continuous ]をクリックして画面に画像を表示します。対物レンズを、撮像する最も近い平面に移動します。 Zスタック タブで、 最初の平面を設定 をクリックし、表示する最後の平面に焦点を合わせて、 最後の平面を設定 をクリックします。画像30-40μmのzスタック。
- 間隔を0.15μmに設定します。
- Z スタックの中央に移動します。 「Tiles 」タブの 「Tile Regions」で、「 Verify 」をクリックし、「 Set Z & move to」をクリックします。
- [フォーカス戦略] タブで、フォーカス戦略が [タイル設定] で定義された [Z 値の使用/フォーカス サーフェス] に設定されていることを確認します。
- 「実験を開始」をクリックして、画像取得を開始します。
- 画像処理
- 「処理」タブで、「 単一 」モードまたは 「バッチ 」モードを選択します。 Airyscan処理を選択します。 「3D処理 」にチェックを入れ、「 標準オートフィルター」を選択します。取得したRAW画像を選択し、処理を開始します(補足図S2A)。
- Airyscan処理が完了したら、処理方法で スティッチング を選択します。 [New Output]、[Fuse Tiles]、[Correct Shading]、および [All by reference] を選択し、参照を DAPI として使用します。Airyscanで処理された画像を選択し、処理を開始します(補足図S2B)。
- 未加工の画像と二重処理された画像を保存します。後者は バッチ モードで自動的に保存されます。
6. 画像解析
注: ここで使用した解析パイプラインは、この調査の目的に合わせてカスタマイズされています。パイプライン操作の詳細は、 補足図 S3 に示されています。
- cziイメージファイルをソフトウェアにインポートします。解析パネルを開きます。ドロップダウンで [新しいパイプライン] を選択します。
- 細胞検出パイプラインを作成するには、Cellpose Python Segmenter をパイプライン12 にインポートします。Model_Nameをcyto2に、Input_channelを2に、Second_channelを3に、Diameter_in_μmを10に、Flow_thresholdを0.4に、Cellprob_thresholdを0に設定します。パイプラインの前に Import Document Objects (名前が Import Manual Labels に変更) を追加し、パイプラインの後に Object Feature Filter (名前が Size Filter に変更) 演算子を追加するには、 [+ 操作の追加] をクリックします。パイプライン設定の値については、補足図 S3A を参照してください。
- ビューアを最初の関心のある平面に移動します
- オブジェクト描画ツールアイコンをクリックします
- ポリゴンモードアイコンをクリックします
- 解析する描画領域
- ビューアを最後の関心のある平面に移動します
- 解析する描画領域
- 緑色のチェックマークをクリックします 変更 を適用
- 「オブジェクトテーブルを表示」アイコンをクリックします
- 作成したオブジェクトを右クリックし、[ オブジェクト名の変更 ] を選択して、オブジェクトの名前を適切な名前に変更します。作成したオブジェクトを右クリックし、[ タグの追加]をクリックします。Analysis Region というタグを追加します。
- このパイプラインを実行するには、 [分析] パネルの上部にある前方の青い矢印をクリックします。
- 一次繊毛および中心小体検出パイプラインを作成するには、解析パネルを開き、ドロップダウンで [New Pipeline...]を選択します。パイプラインに次の演算子を追加します: Import Document Objects (Import Manual Labels に名前が変更されました)、2 つの Intensity Threshold Segmenter オペレータ ( Cilia Threshold と Centriole Threshold に名前が変更されました)、 Splitting ( Centriole Splitting に名前が変更されました)、 Object Feature Filter ( Centriole Splitting size filter に名前が変更されました)、 および Compartment ( Compartment - Cell に名前が変更されました)。パイプライン設定の値については、 補足図 S3B を参照してください。
- このパイプラインを実行するには、 [分析] パネルの上部にある前方の青い矢印をクリックします。
- [オブジェクト] ウィンドウを開きます。[Im/Export...] をクリックし、[Excel Export...] を選択します。保存する機能として、# Children, Name, Parent Names, 3D oriented bounds Short side, Middle side, Long side, Angle XY, Angle XZ, Angle YZ, Bounding Box X1, X2, Y1, Y2, Z1, Z2, SizeX, SizeY, SizeZ, Center of Geometry X, Y, Z, PLANE FIRST, LAST, COUNT, Sphericity (Mesh), Volume (Mesh), Surface Area (Mesh)を選択します。
結果
6週齢マウスの成長プレートを、記載されたプロトコルおよび組織にマッピングされた一次繊毛組織を用いて画像化した。成長プレートの3つの領域(静止状態、増殖性、肥大性)すべての画像がキャプチャされ、分析パイプラインを通じて実行されました(図2A)。個々の細胞はDAPI染色で同定でき、毛様体軸索は赤、中心小体は緑です。この方法を使用した分解能は、個々の中心小体を識別するのに十分なほど高いです(図2B)。この解像度では、繊毛を3D環境で視覚化し、さまざまな方向を向くことができます(ビデオ1)。
また、低解像度の画像や高倍率の対物レンズを撮影することで、組織のより広い領域にわたる一次繊毛を可視化することもできます。ただし、これらは3Dでの繊毛配向の解析には適していません。例えば、関節軟骨の一次繊毛を20倍対物レンズで画像化し、この方法が他の組織にも有効であることを示しました(図2C)。この解像度では、赤の毛様体軸索を識別できますが、中心小体は識別が難しくなります。軟骨下骨はGFPバックグラウンドシグナルを多く提示する傾向があり、この組織における中心小体の同定を困難にしています。
このパイプラインは、個々の細胞、初代繊毛、中心小体を検出でき、高度に組織化された若い成長プレートや、10週齢のマウスのようにカラムがあまり明確に定義されていない古い成長プレートで機能します(図3)。細胞と繊毛は3Dで検出され、zスタック全体で表示できます。
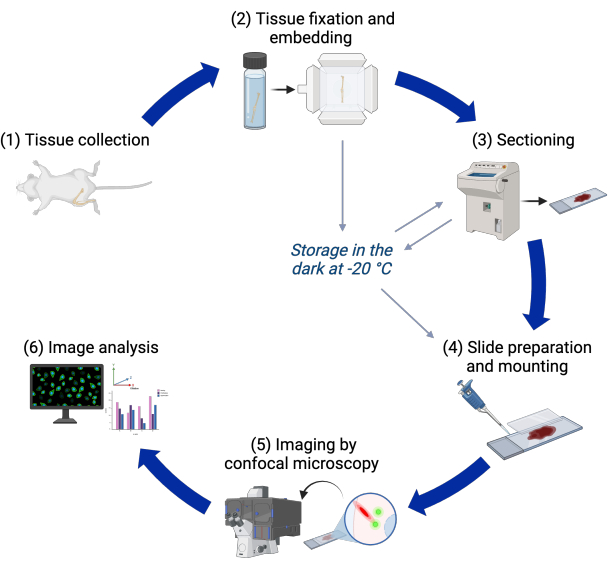
図1:実験ワークフロー。 組織収集から画像解析までのプロトコールのさまざまなステップのフローチャート。 この図の拡大版を表示するには、ここをクリックしてください。
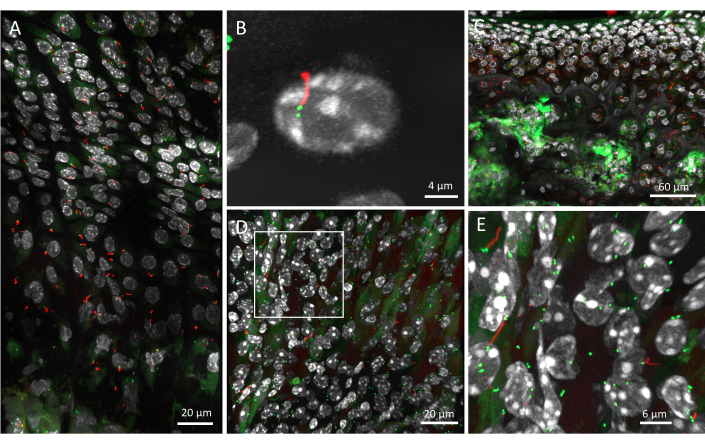
図2:ARL13B-CENTRIN-2マウスのマウス組織における一次繊毛。 (A)6週齢の成長プレートで、DAPIが白、CENTRIN-2が緑、ARL13Bが赤で、40倍対物レンズで画像化。(B)個々の一次繊毛とその2つの中心小体を拡大します。(C)20倍対物レンズで画像化された4週齢マウスの関節軟骨と軟骨下骨。(D)筋骨格組織と同じパイプラインを使用して画像化された6週齢マウスの腎臓。(E)腎臓内の個々の一次繊毛とその中心小体を示す D の白いボックスの拡大。スケールバー = 20 μm (A,D)、4 μm (B)、60 μm (C)、6 μm (E)。 この図の拡大版を表示するには、ここをクリックしてください。
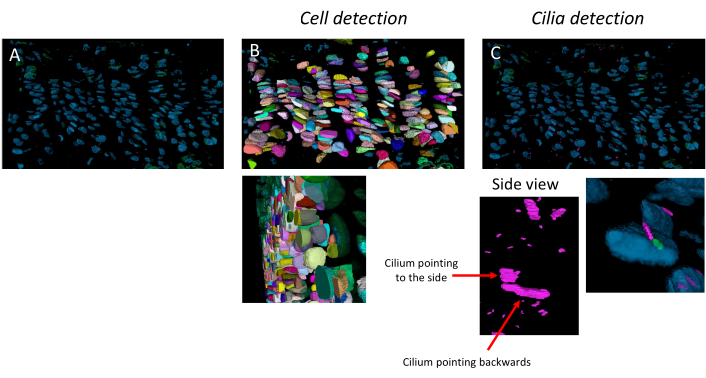
図3:細胞および繊毛検出パイプライン。 10 週齢の成長プレート上の画像解析パイプラインの適用例。(A)ソフトウェアで開いた画像。(B)細胞検出パイプラインの結果。(C)繊毛および中心小体検出パイプラインの結果。繊毛はピンク、中心小体は緑です。側面図は、解像度が3Dで異なる方向に向けられた繊毛を識別するのに十分な高いことを例示しています。 この図の拡大版を表示するには、ここをクリックしてください。
ビデオ1:ARL13B-CENTRIN-2マウスからの6週齢の成長板の3次元再構成。 立体再構成はImarisソフトウェアで行い、アニメーションビデオとして記録しました。 このビデオをダウンロードするには、ここをクリックしてください。
補足図S1:顕微鏡システムでの画像取得設定。 (A)チャネルを追加し、Airyscanモードを選択するためのスマートセットアップウィンドウ。(B)タイルビューアは、画像にタイルを選択してプレビューします。(C)Airyscan検出器の調整ウィンドウ、すべてのタイルが緑色に変わると、Airyscan検出器の位置合わせが確認されます。(D)3つのチャンネルのレーザー設定。(E)取得モードとZスタック設定。このファイルをダウンロードするには、ここをクリックしてください。
補足図S2:顕微鏡システムの画像処理設定。 (A)Airyscan処理設定。(B)ステッチ設定。 このファイルをダウンロードするには、ここをクリックしてください。
補足図 S3: カスタム画像分析パイプライン。 (A)GFPおよびDAPIチャネルを使用した細胞検出パイプライン。(B)mCherryチャネルとGFPチャネルをそれぞれ使用した一次繊毛および中心小体検出パイプライン。 このファイルをダウンロードするには、ここをクリックしてください。
ディスカッション
このプロトコールでは、採取後すぐに組織処理と包埋のステップを進めることが不可欠です。ただし、組織のブロックとスライドは、-20°Cで少なくとも3か月間保持できます。 このプロトコルのいくつかのステップは、さまざまなアプリケーション、特にセクションの厚さ、倍率、およびzスタックパラメータに合わせて変更できます。これは、イメージングの深さと得られる解像度に影響を与えます。開口数と解像度モードの変更を使用して、さらに高い解像度を得ることができ、zスタックの間隔サイズが小さくなりました。ただし、これにより、イメージング時間が長くなったり、イメージングできる視野のサイズが小さくなったりします。記載されているイメージングパラメータは、一次繊毛配向の3D解析で特定された最小要件です。
高分解能を得るためには、封入剤の選択が重要です。封入剤の屈折率は、zスタックでの画像の劣化を最小限に抑えるために、液浸剤の屈折率と同程度でなければなりません。ここで使用する封入剤の屈折率は1.52、浸漬油の屈折率は1.518です。ここで使用されている共焦点顕微鏡のAiryscan機能は、超解像画像を提供します。他の超解像顕微鏡を使用することもできます。しかし、共焦点モードだけでは、撮像された領域のサイズに必要な解像度を得るのに十分ではないことがわかりました。マルチプレックスSR-8Y Airyscanモードの最大解像度は、x / yで120/160 nm、z で450 nmで、最大フレーム数/秒は47.5です。Multiplex SR-4Y Airyscanモードは、最大解像度がx / yで140 nm、zで450 nmですが、最大フレーム数が25の半分の速度で画像は、3Dで一次繊毛をイメージングするのに大きな違いを示さないことがわかりました。同様に、この顕微鏡で最も高い解像度を提供するAiryscan超解像(SR)モード(x/yで120 nm、zで350 nm)は、最大フレーム数/秒が4.713であるため、イメージングに時間がかかります。
バックグラウンドGFPシグナルは、筋肉などの他の組織で観察できます。これを最小限に抑えるために、レーザーの範囲を狭めて、他の波長とのオーバーラップを減らすことができます。この系統は、以前に胚、初代細胞培養、筋線維、および網膜色素上皮9,14,15の初代繊毛を画像化するために使用されてきました。私たちは、腎臓、軟骨、骨組織における一次繊毛のイメージングに成功しています。イメージング設定の最適化は、さまざまな組織に対して必要になる可能性があります。ただし、この凍結切片プロトコルは、シグナルの保存を確実にする必要があります。
2細胞の厚い切片(40-60μm)切片は、全細胞およびその一次繊毛のイメージングを可能にするため、成長プレートで3D配向情報を取得するのに十分です。脂肪細胞など、細胞が軟骨細胞よりも大きい他の組織では、細胞集団データを収集するためにより厚い切片が必要になる場合があります。使用する顕微鏡の深さ分解能によっては、厚い部分の奥深くまでイメージングできない場合があります。組織透明化技術は、必要な時間のために内因性mCherryおよびGFPシグナルの喪失につながる可能性があります。これは、四肢の他のイメージングにクリアリング方法が使用されていたため、調査されましたが、この文脈では必要ではないことがわかりました。内因性CD31-RFPに関する私たちの経験は、クリアリング技術には慎重でオーダーメイドのサンプルの最適化が必要であることを示唆していました。
このARL13B-CENTRIN-2系統は、他のマウス系統と交配して、組織中の一次繊毛の組織化をさらに研究することができます。例えば、アグリカン-Cre IFT88fl/flと交配して、欠失が繊毛を妨げる一次繊毛タンパク質であるIFT88をアグリカン発現細胞(出生後軟骨細胞)で条件付きでノックアウトしました5,6。これらのARL13BおよびCENTRIN-2蛍光シグナルは、その後、組織内の毛様体構造のレベルでの遺伝的摂動の効率と、それに関連する毛様体組織の変化を測定するために使用できます。現在進行中の作業はまさにこれを行っています。私たちの最初の目標は、このより堅牢な方法論を使用して、IFT88 欠失が繊毛の有病率に及ぼす影響を定量化することです。以前は、免疫蛍光を使用してこれを測定し、これを 20% の減少と推定していました6。この方法は、免疫組織化学と組み合わせて、他のタンパク質、例えば細胞膜タンパク質と組み合わせて一次繊毛を画像化することもできます。
ARL13Bに導入された遺伝子は、ARL13Bの発現と一次繊毛の長さに影響を与える可能性があります。以前の研究では、マウス脳の特定の領域において、非導入遺伝子ARL13Bと比較して、ARL13B-CENTRIN-2系統を用いた異なる一次繊毛の長さが測定されており、他の領域では差はなかった16。別の研究では、マウス胚性線維芽細胞において、GFPタグ付きARL13Bマウス系統とタグなしARL13Bと比較して、一次繊毛の長さの違いが観察された10。現在進行中の研究では、ARL13B-CENTRIN-2マウス株と非トランスジェニックARL13Bに対する抗体染色の結果を比較することにより、成長プレートや他の組織における一次繊毛の長さの変化を検証しています。
このプロトコルの画像解析パイプラインは、その後、一次繊毛の繊毛率、長さ、3D配向、および中心小体の数と成長プレート内の位置を測定するように設計されています。画像解析ソフトウェアでは、関連する手順を変更することでカスタムパイプラインを作成できます。このパイプラインの出力は、検出された細胞、繊毛、および中心小体について選択された特徴の測定値を含むスプレッドシートです。その後、研究の目的に応じて、Rまたは他のソフトウェアで事後分析を実行できます。イメージング設定と同様に、パイプラインのステップと特定の設定は、研究の目的に応じて、さまざまな画像で動作するように変更できます。全体として、この分析法は、以前にGrowth Plateで発表された一次繊毛のハイスループット解析を可能にします。研究可能な細胞と関連する繊毛の数により、より大規模で信頼性の高い測定が可能になります。
開示事項
著者は、宣言する利益相反を持っていません。
謝辞
ケネディリウマチ研究所のBSUスタッフ全員、特にアルベルティーノ・ボニファシオの畜産に感謝します。顕微鏡の使用に協力してくださったOxford-ZEISS Centre of Excellence of Biomedical Imaging、特に画像解析パイプラインの開発にご協力いただいたJacky (Ka Long) Ko博士に感謝いたします。この研究は、ケネディ・トラスト・オブ・リウマチ学研究学生(ジョンソン)およびバイオテクノロジー・生物科学研究評議会(BBSRC、BB/X007049/1)の支援を受けた。
資料
| Name | Company | Catalog Number | Comments |
| 22 x 50 mm cover slips | fisherscientific | 12373128 | |
| arivis Vision4D software | Zeiss | N/A | Image analysis software |
| Confocal laser scanning microscope | Zeiss | Zeiss 980 Airyscan 2 | |
| CoverGrip Coverslip Sealant | Biotium | 23005 | |
| Cryofilm type 3C(16UF) (sheet type) 2.0 cm width | Section-lab | C-FUF303 | To be purchased from Section-lab directly |
| Cryostat | Leica | CM1900 UV | |
| DAPI | invitrogen | D1306 | |
| Disposable Base Molds | epredia | 58952 | |
| Dulbecco's Phosphate Buffered Saline | gibco | 14190 | |
| Formalin | Sigma | 20260630 | |
| Imaris software | Oxford Instruments | N/A | Image analysis software for 3D reconstruction |
| Immersion Oil 518F | Zeiss | ISO 8036 | |
| Microtome blades | Feather | 02.075.00.006 | |
| SlowFade Glass Antifade Mountant | invitrogen | S36917 | |
| Sucrose | Sigma | S9378 | |
| Super Cryoembedding Medium (SCEM) | Section-lab | C-EM001 | To be purchased from Section-lab directly |
| Superfrost Plus Microscope slides | avantor | 631-0108 | |
| Zen software | Zeiss | N/A | Freeware for Confocal laser scanning microscopy |
参考文献
- Wheatley, D. N., Wang, A. M., Strugnell, G. E. Expression of primary cilia in mammalian cells. Cell Biol Int. 20 (1), 73-81 (1996).
- Donnelly, E., Ascenzi, M. G., Farnum, C. Primary cilia are highly oriented with respect to collagen direction and long axis of extensor tendon. J Orthop Res. 28 (1), 77-82 (2010).
- McGlashan, S. R., et al. Mechanical loading modulates chondrocyte primary cilia incidence and length. Cell Biol Int. 34 (5), 441-446 (2010).
- Farnum, C. E., Wilsman, N. J. Orientation of primary cilia of articular chondrocytes in three-dimensional space. Anat Rec. 294 (3), 533-549 (2011).
- Coveney, C. R., et al. The ciliary protein IFT88 controls post-natal cartilage thickness and influences development of osteoarthritis. Arth Rheumatol. 74 (1), 49-59 (2021).
- Coveney, C. R., et al. Ciliary IFT88 protects coordinated adolescent growth plate ossification from disruptive physiological mechanical forces. J Bone Miner Res. 37 (6), 1081-1086 (2022).
- Andrea, C. E. D., et al. Primary cilia organization reflects polarity in the growth plate and implies loss of polarity and mosaicism in osteochondroma. Lab Invest. 90 (7), 1091-1101 (2010).
- Ascenzi, M. -. G., et al. Effect of localization, length and orientation of chondrocytic primary cilium on murine growth plate organization. J. Theor Biol. 285 (1), 147-155 (2011).
- Bangs, F. K., Schrode, N., Hadjantonakis, A. K., Anderson, K. V. Lineage specificity of primary cilia in the mouse embryo. Nat. Cell Biol. 17 (2), 113-122 (2015).
- Larkins, C. E., Aviles, G. D. G., East, M. P., Kahn, R. A., Caspary, T. Arl13b regulates ciliogenesis and the dynamic localization of Shh signaling proteins. Mol Biol Cell. 22 (23), 4694-4703 (2011).
- Higginbotham, H., Bielas, S., Tanaka, T., Gleeson, J. G. Transgenic mouse line with green-fluorescent protein-labeled Centrin 2 allows visualization of the centrosome in living cells. Transgenic Res. 13 (2), 155-164 (2004).
- arivis. . Application Note #47 - Applying Cellpose models in arivis Vision4D. , 1-20 (2021).
- Huff, J., Bergter, A., Luebbers, B. Application Note: Multiplex mode for the LSM 9 series with Airyscan 2: fast and gentle confocal super-resolution in large volumes. Nat Methods. , (2019).
- Ning, K., et al. Cilia-associated wound repair mediated by IFT88 in retinal pigment epithelium. Sci Rep. 13 (1), 8205-8219 (2023).
- Palla, A. R., et al. Primary cilia on muscle stem cells are critical to maintain regenerative capacity and are lost during aging. Nat Commun. 13 (1), 1439-1451 (2022).
- Brewer, K. K., et al. Postnatal dynamic ciliary ARL13B and ADCY3 localization in the mouse brain. Cells. 13 (3), 259-277 (2024).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved