Method Article
ARL13B-CENTRIN-2 마우스 모델을 사용한 근골격계 조직의 원발성 섬모의 체적 이미징 및 분석
요약
이 방법은 조직 수집에서 섬모 조직 분석에 이르기까지 광물화된 조직의 넓은 영역에 걸쳐 원발성 섬모를 3차원으로 제 자리에서 이미지화하기 위해 ARL13B-CENTRIN-2 형질전환 마우스 모델에서 내인성 신호를 활용하는 방법을 설명합니다. 이 프로토콜은 다른 유형의 조직에도 적용될 수 있습니다.
초록
1차 섬모는 대부분의 세포 유형에 의해 조립된 비운동성, 단독 소기관입니다. 이는 골격의 발달 및 항상성(homeostasis)에 필수적이며, 주변 환경의 생화학적, 기계적 신호에 대한 세포 반응을 조정하는 데 필수적입니다. 마이크로미터 크기의 크기로 인해 1차 섬모는 조직에서 특성화하기 위해 현장에서 이미지화하는 것이 어려우며, 높은 처리량 방식으로 광학 편향 없이 수행할 수 있습니다. 생체 내 3D 환경에서 조직을 이해하는 것은 생리학 및 질병에서 섬모 역할을 이해하는 데 핵심이 될 것입니다. ARL13B-CENTRIN-2 마우스 라인은 섬모 축삭 단백질 ARL13B에 mCherry 태그를, 섬모체 기저부에 국한된 중심소체 단백질 CENTRIN-2에 녹색 형광 단백질(GFP) 태그를 제공합니다. 이 마우스 라인을 사용하면 염색할 필요 없이 1차 섬모를 이미징할 수 있으며, 이는 이미지 수집의 단계 수를 줄이고 신호 대 잡음비와 관련된 문제를 우회합니다. 이 간행물은 형광 신호를 보존하면서 근골격계 조직의 원발성 섬모를 이미지화하는 단계별 프로토콜을 설명합니다. 또한 보편적으로 적용 가능한 이미지 분석 파이프라인에 대해 설명하여 길이 및 3D 방향과 같은 섬모 특징을 편향 없이 정량화할 수 있습니다.
서문
일차 섬모는 골격을 포함한 많은 조직과 기관의 발달과 항상성에 필수적인 세포 기관입니다. 출생 후 성장 중에는 연골 내 골화 (endochondral ossification) 동안 연골 광물화를 조절합니다1. 인간의 경우 섬모 관련 단백질에 영향을 미치는 유전적 돌연변이는 섬모병증으로 알려진 다양한 질병과 발달 장애를 유발합니다. 조직 내부의 원발성 섬모의 조직에 대한 이해는 기관 수준에서 세포 행동을 조절하는 방법을 이해하는 데 중요합니다. 원발성 섬모의 발생률, 길이 및 방향은 조직 내부 및 조직 간에 차이가 있는 것으로 나타났으며, 이는 조직 조직이 조직 기능에 미치는 역할을 시사합니다 2,3,4. 성장판(growth plate)은 긴 뼈 끝에 있는 연골층으로, 출생 후 대부분의 뼈 성장이 유아기부터 청소년기까지 이루어지는 곳으로, 연골 세포 세포가 기둥으로 배열되어 있는 고도로 조직화된 조직입니다. 성장판에서 연골세포는 휴지기에서 증식기, 비대기에 이르기까지 분화 단계를 거칩니다.
이 세포 조직은 일부 섬모병증에서 손실되는데, 이는 원발성 섬모가 이 세포 조직을 조정하는 역할을 하거나 최소한 세포 분극과 관련이 있을 수 있음을 시사합니다. 또한, 일차적 섬모 방향은 관절 연골4에서 하중을 견디는 영역과 하중을 견디지 않는 영역 간에 차이가 관찰되는 가운데 기계감각 기능에 중요한 역할을 하는 것으로 보인다. 본 연구의 최근 연구는 관절 연골과 성장판의 서로 다른 부위 내에서 차이가 있음을 시사한다 5,6. 성장판에서 원발성 섬모를 이미지화한 이전 연구는 2D로 제한되어 조직의 섬모 조직과 관련된 정보가 손실되거나 3D에서 소수의 세포로 제한되어 분석의 규모와 검정력이 감소합니다 7,8.
1차 섬모를 이미징하는 데 있어 어려운 점 중에는 마이크로미터 크기의 크기와 면역조직화학 항체의 비특이성이 있습니다. 기본 형광 신호를 사용하면 강력한 국소화와 특이성을 보장할 수 있습니다. 그러나 천연 형광 신호는 파라핀에 조직을 삽입하는 긴 석회질 제거 과정으로 인해 신호가 손실될 수 있기 때문에 광물화된 조직에서 이미지화하기 어려울 수 있습니다. native 상태에서 동결된 시료 동결 절편은 embedding 및 sectioning 프로세스의 속도를 높여 이러한 손실을 방지합니다.
원발성 섬모를 제자리에서 이미지화하기 위해 Bangs 등에 의해 이중 형질전환 마우스 라인이 생성되었으며, 이는 mCherry에 융합된 ADP-리보실화 인자 유사 단백질(ARL13B)과 GFP9에 융합된 CENTRIN-2를 발현합니다. ARL13B는 일차 섬모막에 국한된 작은 GTPase이고, CENTRIN-2는 섬모막10,11의 기저부에 있는 두 개의 중심체에 국한된 중심소체 단백질입니다. 따라서 이 마우스 라인을 사용하면 면역조직화학(immunohistochemistry) 없이 형광 표지된 1차 섬모를 이미지화할 수 있습니다.
여기에 설명된 프로토콜은 이 형질전환 라인을 사용하여 컨포칼 현미경으로 쥐 출생 후 성장 플레이트에서 원발성 섬모를 3D로 이미지화하며, 추가적인 4',6-디아미디노-2-페닐린돌(DAPI) 염색만 필요합니다. 수백 개의 세포에 있는 수백 개의 원발성 섬모를 조직 맥락에서 집합적으로 이미지화하고 분석할 수 있습니다. 이후 이미지 분석 파이프라인은 성장판의 여러 영역에서 원발성 섬모 발생률, 길이 및 방향을 측정합니다. 파이프라인은 또한 중심소체에 대해 이러한 기능을 정량화할 수 있습니다. 프로토콜의 단계에 대한 개요는 그림 1에 나와 있습니다.
프로토콜
축산 및 실험은 옥스퍼드 대학의 윤리적 프레임워크에 따라 영국 내무부에서 부여한 프로젝트 라이선스에 따라 수행되었습니다. 이 프로토콜은 2주에서 10주 사이의 마우스에서 수행되었지만 이 연령을 초과하는 마우스의 연골에서 작동합니다.
1. 조직 채취
- 생쥐가 들어있는 상자에 이산화탄소를 최대 80%까지 채우고 농도를 5분 동안 유지하여 생쥐를 안락사시킵니다. 경추 탈구를 수행합니다.
- 관심 있는 조직을 해부합니다.
참고: 다음은 경골 및 대퇴 성장판과 관절 연골을 포함한 뒷다리 무릎을 해부하기 위한 프로토콜입니다.- 발밑 피부를 절개하여 허벅지 위쪽까지 자릅니다. 집게로 절개 라인의 피부를 제거합니다.
- 다리에서 근육과 지방 조직을 잘라냅니다.
- 다리를 몸에서 분리하려면 엉덩이 상단의 대퇴골을 깨끗하게 자릅니다. 경골 아래쪽에서 발을 자릅니다.
- 다리에 남아 있는 과도한 근육과 지방 조직을 제거하십시오.
2. 조직 정착과 끼워넣기
참고: 프로토콜 전반에 걸쳐 형광 신호 손실을 방지하기 위해 조직을 어두운 곳에 보관하도록 주의하십시오.
- 조직을 10% 포르말린으로 실온에서 4시간 동안 고정합니다.
- 조직을 4°C에서 하룻밤 동안 2.5% 포르말린으로 옮깁니다.
- 조직을 증류수 (dH2O)에 30 % 자당으로 옮기고 실온에서 하룻밤 또는 4 °C에서 3 일 동안 부드럽게 회전시킵니다. 회전하는 동안 조직이 완전히 잠길 수 있도록 튜브를 위쪽까지 채웁니다. 이 배양 후 조직은 튜브 바닥으로 가라앉습니다.
- 별도의 상자에 소량의 드라이아이스를 넣고 그 위에 크라이오몰드를 놓습니다. SCEM(Super Cryoembedding Medium)의 선을 따라 슬개골이 아래를 향하도록 하고 다리가 가능한 한 평평하도록 경골과 대퇴골을 벌리도록 팔다리를 배치합니다. SCEM이 굳어지기 시작하고 누르고 있지 않으면 팔다리가 더 이상 움직이지 않을 때까지 집게로 제자리에 고정합니다.
- SCEM으로 완전히 덮고 금형을 맨 위까지 채웁니다. SCEM이 완전히 응고될 때까지 조직의 움직임을 방지하기 위해 샘플을 평평하게 유지하면서 샘플을 드라이아이스 상자에 넣습니다.
- -20 °C로 옮깁니다.
3. 단면화
- 금형에서 블록을 제거하고 저온 유지 장치의 척에 맞는 크기로 자릅니다. 척에 SCEM을 적용하고 블록을 맨 위에 놓아 블록을 척에 부착합니다.
- 저온 유지 장치에서 -20 °C에서 최소 5분 동안 응고되도록 합니다.
- 척을 샘플 홀더에 부착하고 블레이드를 블레이드 홀더에 부착합니다.
- 경골 성장판에 도달할 때까지 블록을 자릅니다. 이상적으로는 경골이 위쪽을 가리키는 부분이어야 합니다.
- 극저온 필름 테이프를 주변에 충분한 공간이 있는 샘플에 맞는 크기로 자릅니다. 극저온 필름을 절단면에 장착하고 피팅 도구로 극저온 필름이 단단히 접착되도록 합니다. 40-60μm 두께의 절편을 절단하여 2셀 두께의 절편을 얻습니다. 집게를 사용하여 테이프를 모아 슬라이드, 샘플 면이 위로 향하게 하고 슬라이드의 짧은 슬라이드와 일직선으로 경골 성장판에 놓습니다.
- 샘플 슬라이드를 -20°C에서 보관하십시오.
4. 슬라이드 준비 및 장착
- 슬라이드를 실온에서 수평으로 해동합니다.
- PBS 한 방울로 슬라이드를 3 x 5분 동안 세척하여 SCEM을 제거합니다.
- 10μM DAPI로 1분 동안 염색합니다.
- PBS 한 방울로 슬라이드를 2 x 5분 동안 씻습니다.
- 장착하고 커버 슬립으로 덮고 실온에서 밤새 그대로 두십시오. 커버슬립 밀봉제로 커버슬립의 가장자리를 밀봉합니다.
- 다음 날 이미지.
5. 컨포칼 현미경에 의한 이미징
참고: 사용된 컨포칼 현미경의 40x/1.4 대물렌즈의 해상도는 240nm입니다.
- 현미경을 설정합니다.
- 현미경을 켜고 소프트웨어를 엽니다. 시스템 버튼을 클릭합니다.
- Acquisition 탭에서 Smart Setup을 클릭하여 채널을 설정합니다. GFP, mCherry 및 DAPI 채널을 선택하고 추가합니다. Airyscan 삼각형(해상도와 속도 다중화 8Y 사이)에서 Airyscan 옵션과 SR8Y 기능을 선택합니다. Best Signal(최고의 신호)을 클릭합니다. OK(보충 그림 S1A).
- Locate 탭의 Microscope Control에서 오일 이멀젼이 있는 40x/1.4 대물렌즈를 선택합니다.
- 관심 영역을 찾습니다.
- 슬라이드를 슬라이드 홀더에 놓고 오일이 커버슬립에 닿을 때까지 대물렌즈를 위로 올립니다.
- 성장판 또는 기타 관심 영역을 광 경로 아래에 배치합니다.
- 위치 탭에서 형광을 클릭하고 DAPI를 선택한 다음 접안렌즈를 사용하여 조이스틱으로 슬라이드 홀더를 이동하여 성장 플레이트 또는 관심 영역을 시야 아래에 배치합니다.
- 획득 탭에서 DAPI 채널만 선택하여 시작합니다.
- 연속을 클릭하여 화면에서 해당 채널의 이미지를 보고 성장판이 보이는지 확인합니다.
- Tiles 탭에서 Show viewer를 클릭하여 Tile viewer를 엽니다. 필요한 타일 수(일반적으로 x 방향으로 1개, y 방향으로 3-5개)를 추가하고 미리보기 및 시작을 클릭합니다(보충 그림 S1B).
- 타일이 성장판의 세 영역(휴지기, 증식 및 비대)을 덮고 있는지 확인하십시오. 필요에 따라 타일 위치와 번호를 조정합니다.
- 이미징 매개변수를 설정합니다.
- Acquisition 탭에서 Continuous를 클릭합니다. 채널 탭에서 게인(마스터) 및 레이저 출력을 조정하여 화면에서 핵을 명확하게 봅니다. Airyscan 감지기를 정렬합니다. 창 하단에 있는 바둑판식 장미 기호를 클릭하여 Airyscan Detector Adjustment 창을 엽니다. 정렬되면 모든 타일이 녹색으로 바뀝니다(보충 그림 S1C).
- GFP 및 mCherry 채널을 개별적으로 선택하고 게인(마스터), 레이저 파워를 설정하고 Airyscan 감지기를 정렬하는 이 단계를 반복합니다.
- 세 채널을 모두 선택하고 연속을 클릭합니다. Airyscan 감지기를 정렬합니다.
- Acquisition Mode 탭에서 Image Size, Pixel, Size, Frame Size, Speed, Averaging 값을 정의합니다. 사용된 채널에 대한 수집 모드 설정, 게인(마스터) 및 레이저 출력 값은 보충 그림 S1D,E를 참조하십시오.
- 이미지를 획득합니다.
- Acquisition 탭에서 Z-Stack 체크박스를 클릭합니다.
- DAPI 채널만 선택한 상태에서 연속 을 클릭하여 화면에서 이미지를 봅니다. 이미징할 가장 가까운 평면으로 대물렌즈를 아래로 이동합니다. Z-Stack 탭에서 Set First plane(첫 번째 평면 설정 )을 클릭하고, 보려는 마지막 평면에 초점을 맞춘 다음 Set Last plane(마지막 평면 설정)을 클릭합니다. 이미지: 30-40μm z-stacks.
- 간격을 0.15μm로 설정합니다.
- z 스택의 중심으로 이동합니다. Tiles(타일 ) 탭의 Tile Regions(타일 영역)에서 Verify(검증 )를 클릭하고 Set Z(Z를 설정하고 다음으로 이동)를 클릭합니다.
- Focus Strategy 탭에서 Focus Strategy가 Tiles Setup에 정의된 Use Z Values/Focus Surface로 설정되어 있는지 확인합니다.
- Start Experiment(실험 시작)를 클릭하여 이미지 획득을 시작합니다.
- 이미지 처리
- Processing(처리) 탭에서 Single(단일 ) 또는 Batch mode( 배치 모드)를 선택합니다. Airyscan 처리를 선택합니다. 3D 처리를 선택하고 표준 자동 필터를 선택합니다. 획득한 원시 이미지를 선택하고 처리를 시작합니다(보충 그림 S2A).
- Airyscan 처리가 완료되면 처리 방법에서 스티칭 을 선택합니다. New Output(새 출력), Fuse Tiles(퓨즈 타일), Correct Shading(올바른 음영) 및 All by reference(모두 참조)를 선택하고 참조는 DAPI로 선택합니다. Airyscan 처리된 이미지를 선택하고 처리를 시작합니다(보충 그림 S2B).
- 원시 이미지와 이중 처리된 이미지를 저장합니다. 후자는 배치 모드에서 자동으로 저장됩니다.
6. 이미지 분석
참고: 여기에 사용된 분석 파이프라인은 이 연구의 목적에 맞게 사용자 지정되었습니다. 파이프라인 작업에 대한 자세한 내용은 보충 그림 S3에 나와 있습니다.
- czi 이미지 파일을 소프트웨어로 가져옵니다. 분석 패널을 엽니다. 드롭다운에서 새 파이프라인...
- 세포 검출 파이프라인을 생성하려면, Cellpose Python Segmenter를 파이프라인(12)으로 가져오십시오. Model_Name를 cyto2로, Input_channel를 2로, Second_channel를 3으로, Diameter_in_μm를 10으로, Flow_threshold를 0.4로, Cellprob_threshold를 0으로 설정합니다. + Add Operation을 클릭하여 파이프라인 앞에 Import Document Objects(Import Manual Labels로 이름이 변경됨)를 추가하고 파이프라인 뒤에 Object Feature Filter(Size Filter로 이름이 변경됨) 연산자를 추가합니다. 파이프라인 설정 값은 보충 그림 S3A를 참조하십시오.
- 뷰어를 첫 번째 관심 평면으로 이동
- Draw Objects Tool 아이콘을 클릭합니다.
- 폴리곤 모드 아이콘을 클릭합니다.
- 분석할 그리기 영역
- 뷰어를 관심 있는 마지막 평면으로 이동
- 분석할 그리기 영역
- 녹색 체크 표시를 클릭하십시오 Apply Changes
- Show Objects Table(개체 테이블 표시) 아이콘을 클릭합니다
- 생성된 개체를 마우스 오른쪽 버튼으로 클릭하고 개체 이름 바꾸기를 선택하여 개체의 이름을 적절한 이름으로 바꿉니다. 생성된 개체를 마우스 오른쪽 버튼으로 클릭하고 Add Tag를 클릭합니다. 분석 영역이라는 태그를 추가합니다.
- 분석 패널 맨 위에 있는 파란색 화살표를 클릭하여 이 파이프라인을 실행합니다.
- 기본 섬모 및 중심소체 검출 파이프라인을 만들려면 분석 패널을 열고 드롭다운에서 New Pipeline...을 선택합니다. 파이프라인에 다음 연산자를 추가합니다: Import Document Objects ( Import Manual Labels로 이름이 변경됨), 두 개의 Intensity Threshold Segmenter 연산자( Cilia Threshold 및 Centriole Threshold로 이름이 변경됨), Splitting ( Centriole Splitting으로 이름이 변경됨), Object Feature Filter ( Centriole Splitting size filter로 이름이 변경됨) 및 Compartment ( Compartment - Cell로 이름이 변경됨). 파이프라인 설정의 값은 보충 그림 S3B 를 참조하십시오.
- 분석 패널 맨 위에 있는 파란색 화살표를 클릭하여 이 파이프라인을 실행합니다.
- 개체 창을 엽니다. Im/Export...를 클릭하고 Excel Export...를 선택합니다. 저장할 다음 기능을 선택합니다: # 자식, 이름, 부모 이름, 3D 지향 경계 짧은 쪽, 중간, 긴 쪽, 각도 XY, 각도 XZ, 각도 YZ, 경계 상자 X1, X2, Y1, Y2, Z1, Z2, 크기X, 크기Y, Z, 형상 중심 X, Y, Z, 평면 첫 번째, 마지막, 개수, 구형도(메쉬), 볼륨(메쉬), 표면적(메쉬).
결과
6주 된 마우스의 성장판은 기술된 프로토콜과 조직에 매핑된 1차 섬모 조직을 사용하여 이미지화되었습니다. 성장판의 세 영역(휴지기, 증식성 및 비대성)의 이미지를 모두 캡처하여 분석 파이프라인을 통해 실행했습니다(그림 2A). 개별 세포는 DAPI 염색으로 식별할 수 있으며, 섬모 축삭은 빨간색, 중심소체는 녹색입니다. 이 방법을 사용하는 분해능은 개별 중심소체를 구별할 수 있을 만큼 충분히 높습니다(그림 2B). 이 해상도에서는 섬모를 3D 환경에서 시각화하여 다른 방향을 가리킬 수 있습니다(비디오 1).
더 높은 배율 목표를 가진 저해상도 이미지를 캡처하여 더 큰 조직 영역에 걸쳐 1차 섬모를 시각화할 수도 있습니다. 그러나 이는 3D에서 섬모 방향을 해석하는 데 적합하지 않습니다. 예를 들어, 관절 연골의 일차 섬모는 20x 대물렌즈로 이미지화되었으며, 이는 이 방법이 다른 조직에서도 작동한다는 것을 보여주었습니다(그림 2C). 이 해상도에서는 빨간색의 섬모 축삭을 식별할 수 있지만 중심소체는 구별하기가 더 어렵습니다. 연골하골은 많은 GFP 배경 신호를 나타내는 경향이 있어 이 조직에서 중심소체를 식별하기 어렵습니다.
이 파이프라인은 개별 세포, 일차 섬모 및 중심소체를 검출할 수 있으며, 10주 된 마우스와 같이 컬럼이 잘 정의되지 않은 고도로 조직화된 젊은 성장판과 오래된 성장판에 적합합니다(그림 3). 세포와 섬모는 z-stack에서 볼 수 있는 것처럼 3D로 감지됩니다.
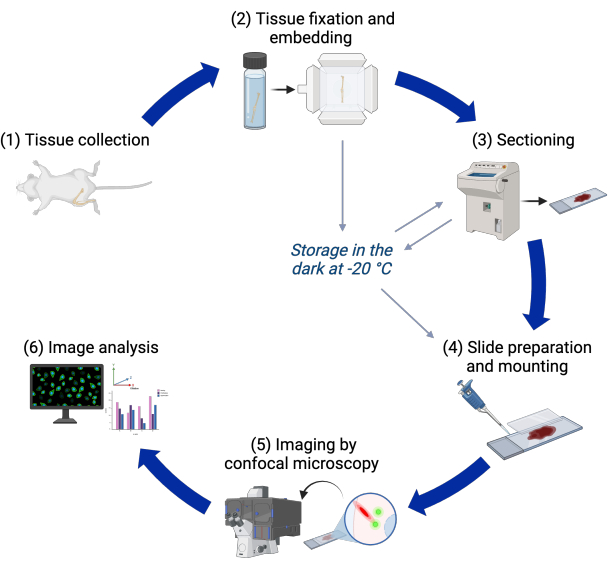
그림 1: 실험적 워크플로우. 조직 채취에서 이미지 분석에 이르기까지 프로토콜의 여러 단계에 대한 순서도. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
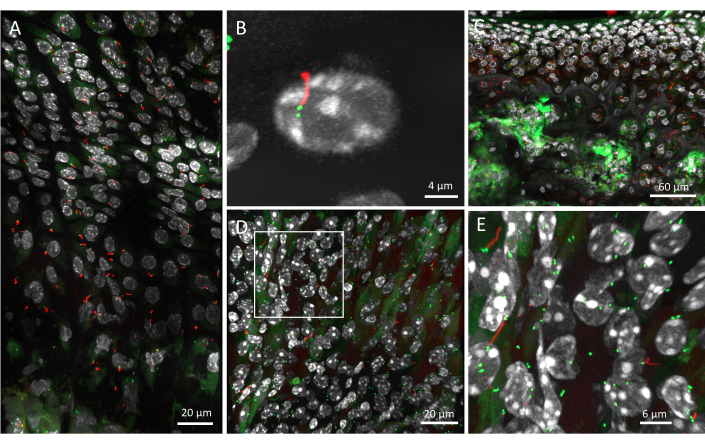
그림 2: ARL13B-CENTRIN-2 마우스의 쥐 조직에 있는 원발성 섬모. (A) 40x 대물렌즈로 이미지화한 DAPI(흰색), CENTRIN-2(녹색), ARL13B(빨간색)의 6주 된 성장판. (B) 개별 1차 섬모와 두 개의 중심소체를 확대합니다. (C) 20x 대물렌즈로 촬영한 4주 된 쥐의 관절 연골 및 연골하골. (D) 근골격계 조직과 동일한 파이프라인을 사용하여 이미지화한 6주 된 마우스의 신장. (E) 신장의 개별 원발성 섬모와 중심소체를 보여주는 D 의 흰색 상자 확대. 눈금자 = 20μm(A,D), 4μm(B), 60μm(C), 6μm(E). 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
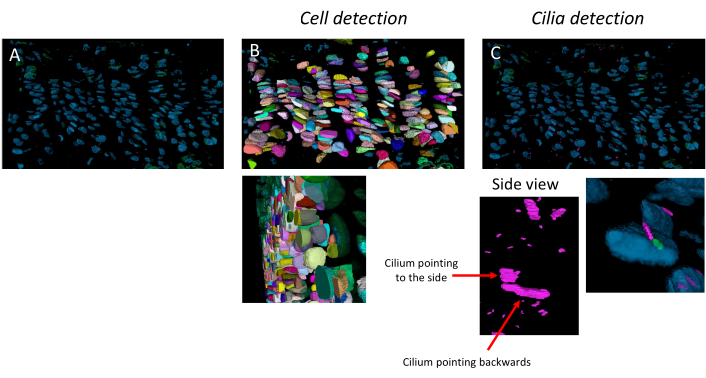
그림 3: 세포 및 섬모 검출 파이프라인. 10주 된 성장판에 이미지 분석 파이프라인을 적용하는 예. (A) 소프트웨어에서 열린 이미지. (B) 세포 검출 파이프라인의 결과. (C) 섬모 및 중심소체 검출 파이프라인의 결과. 섬모는 분홍색이고 중심소체는 녹색입니다. 측면도는 해상도가 3D에서 다른 방향으로 향하는 섬모를 구별할 수 있을 만큼 충분히 높다는 것을 보여줍니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
비디오 1: ARL13B-CENTRIN-2 마우스에서 6주 된 성장판의 3차원 재구성. 3차원 재구성은 Imaris 소프트웨어에서 수행되었으며 애니메이션 비디오로 녹화되었습니다. 이 비디오를 다운로드하려면 여기를 클릭하십시오.
보충 그림 S1: 현미경 시스템의 이미지 획득 설정. (A) 스마트 설정 창을 사용하여 채널을 추가하고 Airyscan 모드를 선택합니다. (B) 타일 뷰어를 사용하여 이미지화할 타일을 선택하고 미리 볼 수 있습니다. (C) Airyscan 감지기 조정 창, 모든 타일이 녹색으로 바뀌면 Airyscan 감지기의 정렬이 확인됩니다. (D) 3개 채널에 대한 레이저 설정. (E) 획득 모드 및 Z-Stack 설정. 이 파일을 다운로드하려면 여기를 클릭하십시오.
보충 그림 S2: 현미경 시스템의 이미지 처리 설정. (A) Airyscan 처리 설정. (B) 스티칭 설정. 이 파일을 다운로드하려면 여기를 클릭하십시오.
보충 그림 S3: 사용자 지정 이미지 분석 파이프라인. (A) GFP 및 DAPI 채널을 사용한 세포 검출 파이프라인. (B) mCherry 및 GFP 채널을 각각 사용하는 1차 섬모 및 중심소체 검출 파이프라인. 이 파일을 다운로드하려면 여기를 클릭하십시오.
토론
이 프로토콜에서는 채취 직후 조직 처리 및 포매 단계를 진행하는 것이 필수적입니다. 그러나 조직의 블록과 슬라이드는 -20 °C에서 최소 3 개월 동안 보관할 수 있습니다. 이 프로토콜의 여러 단계는 다양한 애플리케이션, 특히 단면 두께, 배율 및 z-stack 매개변수에 맞게 수정할 수 있습니다. 이것은 이미징의 깊이와 얻은 해상도에 영향을 미칩니다. 개구수 및 해상도 모드를 변경하면 훨씬 더 높은 해상도를 얻을 수 있으며 z-스택 간격 크기를 줄일 수 있습니다. 그러나 이렇게 하면 이미징 시간이 늘어나거나 이미징할 수 있는 시야의 크기가 줄어듭니다. 설명된 이미징 매개변수는 기본 섬모 방향의 3D 분석을 위해 식별된 최소 요구 사항입니다.
장착 매체의 선택은 고해상도를 얻는 데 중요합니다. 장착 매체의 굴절률은 z-스택에서 이미지 저하를 최소화하기 위해 침지 매체의 굴절률과 유사해야 합니다. 여기에 사용된 마운트런트와 이멀젼 오일의 굴절률은 각각 1.52와 1.518의 굴절률을 가지고 있습니다. 여기에 사용된 컨포칼 현미경의 Airyscan 기능은 초고해상도 이미지를 제공합니다. 다른 초해상도 현미경을 사용할 수 있습니다. 그러나 컨포칼 모드만으로는 이미징된 영역의 크기에 필요한 해상도를 얻기에 충분하지 않다는 것을 발견했습니다. 멀티플렉스 SR-8Y Airyscan 모드의 최대 해상도는 x/y에서 120/160nm 및 z에서 450nm이며 최대 프레임 수는 47.5입니다. 멀티플렉스 SR-4Y Airyscan 모드는 x/y에서 140nm, z에서 450nm의 최대 해상도를 제공하지만 최대 25프레임/초로 절반의 속도로 이미지를 제공하지만, 3D에서 1차 섬모를 이미징하는 데 큰 차이를 나타내지 않는 것으로 나타났습니다. 마찬가지로, 이 현미경에서 가장 높은 해상도(x/y에서 120nm, z에서 350nm)를 제공하는 Airyscan 초해상도(SR) 모드는 최대 프레임/초가 4.713이기 때문에 이미징하는 데 시간이 더 오래 걸립니다.
배경 GFP 신호는 근육과 같은 다른 조직에서 관찰될 수 있습니다. 이를 최소화하기 위해 레이저의 범위를 좁혀 다른 파장과의 중복을 줄일 수 있습니다. 이 라인은 이전에 배아, 1차 세포 배양, 근육 섬유 및 망막 색소 상피 9,14,15의 원발성 섬모를 이미지화하는 데 사용되었습니다. 우리는 신장, 연골 및 뼈 조직의 원발성 섬모를 성공적으로 이미지화했습니다. 이미징 설정의 일부 최적화는 다른 조직에 필요할 수 있습니다. 그러나 이 극저온 절편 프로토콜은 신호의 보존을 보장해야 합니다.
2세포 두께의 절편(40-60μm) 절편은 전체 세포와 1차 섬모를 이미징할 수 있기 때문에 성장판에서 3D 배향 정보를 얻기에 충분합니다. 지방세포와 같이 세포가 연골세포보다 큰 다른 조직의 경우, 세포 집단 데이터를 수집하기 위해 더 두꺼운 절편이 필요할 수 있습니다. 사용된 현미경의 깊이 해상도에 따라 더 두꺼운 부분까지 이미지화하지 못할 수 있습니다. 조직 제거 기술은 필요한 시간으로 인해 내인성 mCherry 및 GFP 신호의 손실로 이어질 수 있습니다. 사지의 다른 이미징을 위해 클리어링 방법이 사용되었기 때문에 이를 조사했지만 이러한 맥락에서는 필요하지 않은 것으로 밝혀졌습니다. 내인성 CD31-RFP에 대한 당사의 경험은 투명화 기법에 신중한 맞춤형 최적화가 필요하다고 제안했습니다.
이 ARL13B-CENTRIN-2 라인은 다른 마우스 라인과 교차하여 조직의 원발성 섬모 조직을 추가로 연구할 수 있습니다. 예를 들어, 우리는 aggrecan-Cre IFT88fl/fl과 교차하여 응그레칸 발현 세포(출생 후 연골 세포)에서 섬모를 방해하는 일차 섬모 단백질인 IFT88을 조건부로 녹아웃했습니다5,6. 이러한 ARL13B 및 CENTRIN-2 형광 신호는 조직의 섬모 구조 수준에서 유전적 섭동의 효율성과 섬모 조직의 관련 변화를 측정하는 데 사용할 수 있습니다. 현재 진행 중인 작업이 정확히 이 작업을 수행하고 있습니다. 우리의 첫 번째 목표는 이 보다 강력한 방법론을 사용하여 섬모 유병률에 대한 IFT88 결실의 영향을 정량화하는 것입니다. 이전에는 면역형광을 사용하여 이를 측정했으며 이를 20% 감소로 추정했습니다6. 이 방법은 또한 면역조직화학과 결합하여 다른 단백질, 예를 들어 세포막 단백질과 함께 원발성 섬모를 이미지화할 수 있습니다.
ARL13B의 전이유전자는 ARL13B 발현 및 원발성 섬모 길이에 영향을 미칠 수 있습니다. 이전 연구에서는 쥐 뇌의 특정 영역에서 ARL13B-CENTRIN-2 계통과 비이식유전자 ARL13B의 원발성 섬모 길이가 서로 다른 것으로 측정되었으며, 다른 영역에서는 차이가 없었다16. 또 다른 연구에서는 마우스 배아 섬유아세포에서 태그가 지정되지 않은 ARL13B와 비교하여 GFP 태그가 지정된 ARL13B 마우스 라인에서 원발성 섬모 길이의 차이를 관찰했습니다10. 진행 중인 연구는 ARL13B-CENTRIN-2 마우스 라인과 비형질전환 ARL13B에 대한 항체 염색을 사용하여 결과를 비교함으로써 성장판 및 기타 조직의 원발성 섬모 길이의 변화를 확인하는 것입니다.
이 프로토콜의 이미지 분석 파이프라인은 일차 섬모의 섬모 비율, 길이 및 3D 방향뿐만 아니라 성장판의 중심소체 수 및 위치를 이후에 측정하도록 설계되었습니다. 이미지 분석 소프트웨어를 사용하면 관련 단계를 변경하여 맞춤형 파이프라인을 만들 수 있습니다. 이 파이프라인의 출력은 감지된 세포, 섬모 및 중심소체에 대해 선택한 기능의 측정값이 포함된 스프레드시트입니다. 그런 다음 연구 목적에 따라 R 또는 다른 소프트웨어에서 사후 분석을 수행할 수 있습니다. 이미징 설정과 마찬가지로 파이프라인 단계 및 특정 설정을 변경하여 다른 이미지에서 그리고 연구의 목적에 따라 작업할 수 있습니다. 전반적으로 이 방법은 성장판에 이전에 발표된 것보다 원발성 섬모에 대한 고처리량 분석을 가능하게 합니다. 연구할 수 있는 세포 및 관련 섬모의 수는 더 크고 신뢰할 수 있는 측정을 가능하게 합니다.
공개
저자는 선언할 이해 상충이 없습니다.
감사의 말
케네디 류마티스 연구소(Kennedy Institute of Rheumatology)의 모든 BSU 직원, 특히 축산에 대해 알베르티노 보니파시오(Albertino Bonifacio)에게 감사드립니다. 현미경 사용에 도움을 주신 Oxford-ZEISS Centre of Excellence of Biomedical Imaging, 특히 이미지 분석 파이프라인 개발에 도움을 주신 Jacky (Ka Long) Ko 박사님께 감사드립니다. 이 연구는 Kennedy Trust of Rheumatology Research studentship(Johnson)과 Biotechnology and Biological Sciences Research Council(BBSRC, BB/X007049/1)의 지원을 받았습니다.
자료
| Name | Company | Catalog Number | Comments |
| 22 x 50 mm cover slips | fisherscientific | 12373128 | |
| arivis Vision4D software | Zeiss | N/A | Image analysis software |
| Confocal laser scanning microscope | Zeiss | Zeiss 980 Airyscan 2 | |
| CoverGrip Coverslip Sealant | Biotium | 23005 | |
| Cryofilm type 3C(16UF) (sheet type) 2.0 cm width | Section-lab | C-FUF303 | To be purchased from Section-lab directly |
| Cryostat | Leica | CM1900 UV | |
| DAPI | invitrogen | D1306 | |
| Disposable Base Molds | epredia | 58952 | |
| Dulbecco's Phosphate Buffered Saline | gibco | 14190 | |
| Formalin | Sigma | 20260630 | |
| Imaris software | Oxford Instruments | N/A | Image analysis software for 3D reconstruction |
| Immersion Oil 518F | Zeiss | ISO 8036 | |
| Microtome blades | Feather | 02.075.00.006 | |
| SlowFade Glass Antifade Mountant | invitrogen | S36917 | |
| Sucrose | Sigma | S9378 | |
| Super Cryoembedding Medium (SCEM) | Section-lab | C-EM001 | To be purchased from Section-lab directly |
| Superfrost Plus Microscope slides | avantor | 631-0108 | |
| Zen software | Zeiss | N/A | Freeware for Confocal laser scanning microscopy |
참고문헌
- Wheatley, D. N., Wang, A. M., Strugnell, G. E. Expression of primary cilia in mammalian cells. Cell Biol Int. 20 (1), 73-81 (1996).
- Donnelly, E., Ascenzi, M. G., Farnum, C. Primary cilia are highly oriented with respect to collagen direction and long axis of extensor tendon. J Orthop Res. 28 (1), 77-82 (2010).
- McGlashan, S. R., et al. Mechanical loading modulates chondrocyte primary cilia incidence and length. Cell Biol Int. 34 (5), 441-446 (2010).
- Farnum, C. E., Wilsman, N. J. Orientation of primary cilia of articular chondrocytes in three-dimensional space. Anat Rec. 294 (3), 533-549 (2011).
- Coveney, C. R., et al. The ciliary protein IFT88 controls post-natal cartilage thickness and influences development of osteoarthritis. Arth Rheumatol. 74 (1), 49-59 (2021).
- Coveney, C. R., et al. Ciliary IFT88 protects coordinated adolescent growth plate ossification from disruptive physiological mechanical forces. J Bone Miner Res. 37 (6), 1081-1086 (2022).
- Andrea, C. E. D., et al. Primary cilia organization reflects polarity in the growth plate and implies loss of polarity and mosaicism in osteochondroma. Lab Invest. 90 (7), 1091-1101 (2010).
- Ascenzi, M. -. G., et al. Effect of localization, length and orientation of chondrocytic primary cilium on murine growth plate organization. J. Theor Biol. 285 (1), 147-155 (2011).
- Bangs, F. K., Schrode, N., Hadjantonakis, A. K., Anderson, K. V. Lineage specificity of primary cilia in the mouse embryo. Nat. Cell Biol. 17 (2), 113-122 (2015).
- Larkins, C. E., Aviles, G. D. G., East, M. P., Kahn, R. A., Caspary, T. Arl13b regulates ciliogenesis and the dynamic localization of Shh signaling proteins. Mol Biol Cell. 22 (23), 4694-4703 (2011).
- Higginbotham, H., Bielas, S., Tanaka, T., Gleeson, J. G. Transgenic mouse line with green-fluorescent protein-labeled Centrin 2 allows visualization of the centrosome in living cells. Transgenic Res. 13 (2), 155-164 (2004).
- arivis. . Application Note #47 - Applying Cellpose models in arivis Vision4D. , 1-20 (2021).
- Huff, J., Bergter, A., Luebbers, B. Application Note: Multiplex mode for the LSM 9 series with Airyscan 2: fast and gentle confocal super-resolution in large volumes. Nat Methods. , (2019).
- Ning, K., et al. Cilia-associated wound repair mediated by IFT88 in retinal pigment epithelium. Sci Rep. 13 (1), 8205-8219 (2023).
- Palla, A. R., et al. Primary cilia on muscle stem cells are critical to maintain regenerative capacity and are lost during aging. Nat Commun. 13 (1), 1439-1451 (2022).
- Brewer, K. K., et al. Postnatal dynamic ciliary ARL13B and ADCY3 localization in the mouse brain. Cells. 13 (3), 259-277 (2024).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유