Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Fluorescence Activated Cell Sorting von Pflanzenprotoplasten
In diesem Artikel
Zusammenfassung
Ein Verfahren zur Isolierung von spezifischen Zelltypen aus pflanzlichem Material nachgewiesen ist. Diese Technik bedient sich transgene Marker exprimieren fluoreszierenden Proteinen in bestimmten Zelltypen, Zell-Dissoziation und Fluorescence Activated Cell Sorting. Zusätzlich wird ein Wachstum Setup hier erleichtert Behandlung von etablierten Arabidopsis thaliana Setzlinge vor cell sorting.
Zusammenfassung
Hochauflösende, Zelltyp-spezifische Analyse der Genexpression stark erhöht das Verständnis von Entwicklungs-Regelung und Antworten auf Reize aus der Umwelt in allen vielzelligen Organismus. In-situ-Hybridisierung und Reporter-Gen-Visualisierung kann in begrenztem Umfang zu diesem Zweck verwendet werden, aber für hochauflösende quantitative RT-PCR oder High-Throughput-Transkriptom-weite Analyse der Isolierung von RNA aus bestimmten Zelltypen ist Voraussetzung. Cellular Dissoziation von Gewebe exprimiert ein fluoreszierendes Protein-Marker in einem bestimmten Zelltyp und anschließende Fluorescence Activated Cell Sorting (FACS) macht es möglich, ausreichende Mengen an Material für die RNA-Extraktion, cDNA-Synthese /-Amplifikation und Microarray-Analyse zu sammeln.
Ein umfangreiches Set von Zelltyp-spezifischen fluoreszierenden Reporter-Linien ist für die Pflanze verfügbar Forschungsgemeinschaft. In diesem Fall sind zwei Markierungslinien der Arabidopsis thaliana Wurzel verwendet: P SCR:: GFP (Endodermis und ruhenden Mitte) und P WOX5:: GFP (Ruhe-Center). Große Zahlen (in Tausend) der Sämlinge sind hydroponically oder auf Agarplatten gewachsen und geerntet, um genügend root Material für weitere Analysen zu erhalten. Cellular Dissoziation von Pflanzenmaterial wird durch enzymatische Verdauung der Zellwand erreicht. Dieses Verfahren nutzt hohe Osmolarität induzierte Plasmolyse und kommerziell erhältliche Cellulasen, um Pektinasen und Hemicellulasen Protoplasten in Lösung freigeben.
FACS von GFP-positiven Zellen nutzt die Visualisierung der grünen gegen die roten Emissionsspektren von Protoplasten von einem 488 nm Laser angeregt. GFP-positive Protoplasten können durch ihre erhöhte Verhältnis von Grün auf Rot-Emission unterschieden werden. Protoplasten werden in der Regel direkt in RNA-Extraktionspuffer sortiert und für die weitere Verarbeitung zu einem späteren Zeitpunkt.
Diese Technik wird enthüllt werden einfach und praktikabel ist. Darüber hinaus wird gezeigt, dass sie ohne Schwierigkeiten eine ausreichende Anzahl von Zellen für die Transkriptom-Analyse zu isolieren können verwendet werden, auch bei sehr knappen Zelltypen (zB Ruhe-Zentrum-Zellen). Schließlich ist ein Wachstumsmarkt Setup für Arabidopsis-Keimlinge zeigten, dass ermöglicht eine unkomplizierte Behandlung der Pflanzen vor Zellsortierung (zB für die Zelltyp-spezifische Analyse der biotischen und abiotischen Stress-Reaktionen). Mögliche zusätzliche Einsatzmöglichkeiten für FACS von pflanzlichen Protoplasten werden diskutiert.
Protokoll
1) Herstellung des pflanzlichen Materials
- Protoplasten können aus vielen verschiedenen Pflanzenarten und Geweben, sofern die richtige Mischung aus Zellwand verdauenden Enzyme 1 wird verwendet, abgeleitet werden. Bevor eine groß angelegte Experiment durchgeführt wird, ist eine kleine Verdauung des Materials empfohlen, um die Protoplastierung Effizienz des Gewebes, Enzyme, etc. zu bewerten und die Prozent-positiven Zellen für Zellsortierung zu schätzen. Hier Protoplasten aus den Wurzeln von Arabidopsis thaliana Keimlinge, die Zelltyp-spezifisch ausdrücken grün fluoreszierende Protein (GFP) eingesetzt werden abgeleitet. Die Endodermis und ruhenden Zentrum von P SCR markiert:: GFP und den ruhenden Mittelpunkt von P WOX5:: GFP 2,3 (Abbildung 1).
- Sämlinge sind hydroponically in phytatrays gewachsen (Sigma; Abbildung 2a) auf eine Nylon-Filter (250 &mgr; m mesh; NITEX), die die Wurzeln wachsen durch in das Wachstumsmedium (0,22% w / v Murashige und Skoog Basal Medium [Sigma], 1% lässt w / v Saccharose, 0,05% w / v MES [2 - (N-Morpholino) ethansulfonsäure], pH 5,7 mit KOH). Alternativ können die Pflanzen auf einer Nylon-Filter (100 um Maschenweite) in vertikal angeordneten 1% Agar-Platten (Abb. 2b) angebaut werden.
- Die Verwendung der oben genannten Filter hilft nicht nur bei der Ernte der Wurzeln, es erleichtert auch eine zusätzliche Behandlung der Sämlinge, wenn gewünscht. Die Filter ermöglichen die Sämlinge en masse, neue phytatrays oder Agar-Platten mit einem Katalysator von Interesse ergänzt übertragen werden. Zum Beispiel die phytatray eingerichtet hat Zelltyp-spezifische Analyse der transkriptionellen Antwort auf Stickstoff-Behandlung in Arabidopsis-Keimlinge 4 verwendet.
- Stellen Sie sicher, dass Ihre mikroskopisch Fluoreszenzmarker korrekt ausgedrückt ist (insbesondere bei Verwendung der Behandlungsmöglichkeit, da die Zelltyp-spezifische Marker sich durch die Behandlung beeinflusst sein könnten). In diesem Fall sind Sämlinge unter einem Fluoreszenzmikroskop (; Abbildung 1 Nikon) inspiziert. Beachten Sie, dass Zelltyp-spezifischen fluoreszierenden Marker Linien zunächst sollte unter einem konfokalen Mikroskop genau, welche Zelltypen sind markiert ermitteln und festzustellen, Variabilität im Ausdruck gekennzeichnet werden.
2) Herstellung der Protoplastierung Lösung
- Dissolve 1,25% w / v Cellulase (Yakult), 0,3% w / v Macerozyme (Yakult), 0,4 M D-Mannitol, 20 mM MES, 20 mM KCl (aus einer 1 M Stammlösung) in VE-Wasser und den pH-Wert auf 5,7 mit 1 M Tris / HCl pH 7,5. Diese Lösung wird leicht getrübt werden.
- Erhitzen Sie die Lösung auf 55 ° C für 10 Minuten (die Lösung wird wiederum klar) und abkühlen lassen auf Raumtemperatur, bevor die Zugabe von 0,1% w / v BSA (Rinderserumalbumin), 10 mM CaCl 2, und 5 mM β-Mercaptoethanol .
3) Ernte und Protoplastierung des Pflanzenmaterials
- Die Wurzeln sind durch Kratzen sie aus dem Nylonnetz mit einem Skalpell geerntet und hinterlegt in einen Kolben mit dem Protoplastierung Lösung. Im Allgemeinen ist 10 ml Protoplastierung Lösung pro 1.500 Keimling Wurzeln verwendet.
- Schütteln Sie die Kolben vorsichtig (75 rpm) bei Raumtemperatur für eine Stunde. Eine längere Inkubationszeit kann sich die Protoplasten Ertrag, sondern auch um die Wirkung von Protoplastierung sich auf die Genexpression hinzuzufügen.
- Filtern Sie die Protoplasten-Lösung mit einem 40 um Zellsieb (BD Falcon) und teilen sich die Lösung über konische 15 ml Röhrchen (BD Falcon).
- Drehen Sie das Röhrchen in einem Ausschwingrotor Zentrifuge für 10 Minuten bei 500 G. Beachten Sie, dass dieser Zentrifugation Geschwindigkeit hängt von der Art des Protoplasten verwendet, ihre Zerbrechlichkeit und die Höhe der Zelltrümmer während enzymatische Behandlung produziert wird.
- Entfernen Sie die meisten der Überstand resuspendieren die Protoplasten in der verbleibenden Lösung und untersuchen diese mikroskopisch (Abbildung 3).
- Nutzen Sie die Hämazytometer, um die Anzahl und Dichte der Protoplasten zu schätzen. Die Zelldichte bestimmt Probeninjektion Geschwindigkeit, die Ereignisse pro Sekunde und damit die gesamte Zeit benötigt, um die Zellen bei der FACS-Sortierung (siehe auch Abschnitt 4.3).
- 3,7) Entweder gehen Sie direkt zu FACS oder waschen und resuspendieren die Protoplasten in einer Inkubationslösung, wie W5 (154 mM NaCl, 125 mM CaCl2, 5 mM KCl, 5 mM MES, pH-Wert einstellen auf 5,7 mit KOH) oder Protoplastierung Lösung ohne zugesetzten Enzyme. Besonders, wenn man transkriptionelle Veränderungen ist es von Bedeutung, um die Exposition der Proben, die Bedingungen, die die Genexpression beeinflussen können, wie das Ändern der Puffer der Protoplasten in. Es wird daher empfohlen, den Protoplasten in Protoplastierung Lösung zu halten und weiter gehalten werden minimiert zu FACS so schnell wie möglich.
4) Fluorescence Activated Cell Sorting von Protoplasten
- Schalten Sie und bereiten die Zell-Sorter. Hier wird ein FACSAria (BD) verwendet.
- Einrichten einer Strömung mit einem 100 & mu, m Düse und einem 20 psi Mantel Druck.
- Die Zelldichte und Probenaufgabe Geschwindigkeit an die jeweilige Experiment, ob eine bestmögliche Ausbeute oder schnellste erreichbare Geschwindigkeit gewünscht wird, basiert eingestellt werden. Wir haben erfolgreich mit Dichten bis zu 10.000.000 Zellen / ml sortiert.
- Verwenden Sie die Probe Agitation Option auf der FACS zur Sedimentation der Protoplasten zu verhindern. Wenn das Verstopfen der FACS ist ein Problem, gibt es drei mögliche Schritte zur Fehlerbehebung: 1. Führen Sie eine Probe-line Rückspülung. 2. Verdünnen Sie Ihre Protoplastensuspension, um die Dichte zu reduzieren. 3. Clean up the Protoplasten Lösung durch Wiederholung der Filtration (3,3) nach Zentrifugation und Resuspension.
- Bereiten Sie das Gerät auf forward scatter (FSC), Side Scatter (SSC) und Emission bei 530/30 nm für GFP und 610/20 nm für Rot Spektrum Autofluoreszenz (RSA) nach Anregung durch einen 488 nm Laser zu messen. Dies sind im Wesentlichen die einzigen Parameter verwendet, um GFP-positive Protoplasten isolieren. Hier wurden die Spannung Einstellungen wie folgt: FSC - 60V, SSC 250V, 350V und GFP RSA 335V. Beachten Sie, dass die optimale Spannung Einstellungen werden bei jedem anders FACS und sogar müssen während der gesamten Lebensdauer des Zellsortierers eingestellt werden.
- Starten Sie durch die Einrichtung einer Dotplot für Vorwärtsstreuung gegenüber side scatter. Übernehmen Sie die Spannung so, dass die gemessenen Ereignisse in die Handlung zentriert sind.
- Anschließend erstellen Sie eine Dot-Plot von grünen und roten Fluoreszenz-Signale. Übernehmen Sie die Spannung so, dass die gemessenen Ereignisse eine zentrierte diagonal Bevölkerung in die Handlung ergeben, wenn man eine Wildtyp-(non-GFP) Protoplastensuspension. Ein Protoplastensuspension aus einer GFP-Marker Linie abgeleitet wird einen klaren Bevölkerung des grün fluoreszierenden Ereignisse nie in Wildtyp-Proben zu sehen.
- Set Entschädigung Einschränkungen für spektrale Überlappung zwischen GFP und RSA einstellen. Angemessene Entschädigung Einschränkung Einstellungen werden für eine bessere Trennung der GFP-positive Protoplasten aus dem nicht-GFP Protoplasten und Schutt zu ermöglichen. Die Einschränkungen werden dabei wie folgt: RSA, minus 17,91% GFP.
- Richten Sie ein Tor zur GFP-positive Ereignisse, eine negative Kontrolle von Nicht-GFP Protoplasten verwendet werden, um bei der Festlegung der Grenzen Tor (Abbildung 4) Hilfe soll zu identifizieren.
- Implementieren Sie eine Vorwärtsstreuung Cutoff, um kleine Fremdkörper aus der Analyse zu verlassen. Visualisieren Sie die GFP-positive Ereignisse in der FSC vs SSC Plot um die Platzierung der Cutoff. Hier wurde der Cutoff bei 5.000 gesetzt. Beachten Sie, dass die FACS wird Schmutz als eine Art Veranstaltungen und eine Probe mit einem hohen Grad von Schmutz rechnen können eine unterschiedliche Prozent GFP positive Ereignisse als erwartet. Dies ist nicht unbedingt ein Problem. Allerdings, je mehr Schmutz in die Probe, desto länger die Art stattfinden wird.
- Je nach Experiment und die Fülle der Zelltyp zu analysieren, stellen Sie die FACS Präzisions-Modus entweder für eine optimale Ausbeute oder die optimale Reinheit der sortierten Zellen.
- Für die RNA-Extraktion, bereiten Sammelröhrchen (1,5 ml Mikrozentrifugenröhrchen) mit der entsprechenden Menge an RNA-Extraktionspuffer. Mit diesem Setup werden 20.000 Sortierung Ereignisse ergeben ein Gesamtvolumen von rund 100 ul, die in auf 350 ul Extraktionspuffer (RNeasy Micro Kit, QIAGEN) sortiert wurden. Mix Proben nach Abschluss der Art, wie Zellsuspension Pool an der Spitze.
- Shop-Proben oder gehen Sie direkt zur Extraktion RNA. Erfolgreiche Microarray-Analysen wurden mit der RNA aus so wenig wie 500 Sortieren Ereignisse extrahiert wurden vorgeformt. Hier haben wir eine RNeasy Micro Extraction Kit (QIAGEN), der WT-Ovation Pico RNA Amplification System und FL-Ovation cDNA Biotin-Modul V2 (NuGEN).
Repräsentative Ergebnisse
Ein phytatray von etwa 1.500 Ein-Wochen alten P SCR:: GFP Keimlinge ergab rund 60.000 Protoplasten (wie von Hämazytometer gemessen). 2,6% von 65.000 FACS-verarbeiteten Ereignisse wurden als GFP-positiv definiert und sortiert wurden (Abbildung 4b).
Acht Platten von etwa 1.500 Vier-Tage-alten P WOX5:: GFP Sämlinge jeder (12.000 insgesamt) ergab ca. 30.000.000 Protoplasten (wie von Hämazytometer gemessen). 0,063% von 16.000.000 FACS-verarbeiteten Ereignisse wurden als GFP-positive definiert und sortiert werden (Abbildung 4c).
10.000 sortiert Ereignisse sind in der Regel für die RNA-Extraktion verwendet und kann von 20 bis 140 ng Gesamt-RNA (Abbildung 5) ergeben.
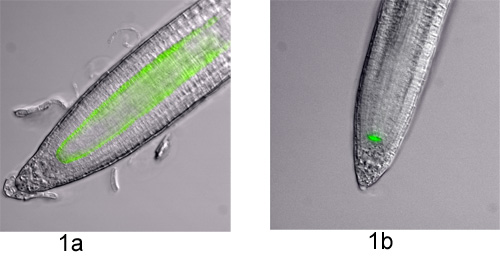
Abbildung 1. Zelltyp-spezifischen GFP Markierungslinien in der Arabidopsis root. Fluoreszenzmikroskopie Bilder wurden mit Differential-Interferenz-Kontrast (DIC) und GFP-Filter auf eine Eclipse 90i-Mikroskop (Nikon), die auf Metamorph-Software (Molecular Devices) übernommen. Die DIC-und GFP-Bilder wurden zur Visualisierung überlagert. Die beiden Markierungslinien in dieser visuellen Experiment verwendet werden gezeigt;a) P SCR:: GFP und b) P WOX5:: GFP.

Abbildung 2. Das Pflanzenwachstum Bedingungen. Sämlinge wurden in ein Umwelt-Controller hydroponically in phytatrays (a) oder vertikal positioniert Agarplatten (b) gewachsen.
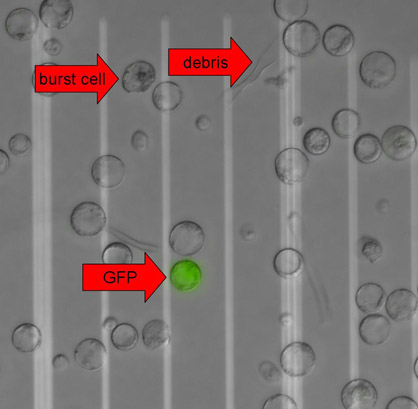
Abbildung 3. Pflanzenprotoplasten, die GFP. Fluoreszenzmikroskopie Bilder wurden mit Differential-Interferenz-Kontrast (DIC) und GFP-Filter auf eine Eclipse 90i-Mikroskop (Nikon), die auf Metamorph-Software (Molecular Devices) übernommen. Die DIC-und GFP-Bilder wurden zur Visualisierung überlagert. Die Pfeile zeigen eine Burst-Zelle, Zelltrümmer und einem GFP-positive Protoplasten. Der Abstand zwischen zwei weißen Linien ist 50 pm.
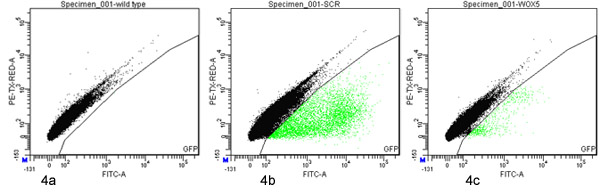
Abbildung 4. Fluorescence Activated Cell Sorting von GFP-positive Protoplasten Protoplasten aus dem Wildtyp (a) P SCR abgeleitet.:: GFP (b) oder P WOX5:: GFP (c) Markierungslinien wurden analysiert und mit einem FACSAria (BD) sortiert mit Toren definiert auf einem Dotplot von grün (530/30 nm; x-Achse) gegen Rot (610/20 nm; y-Achse) Fluoreszenz. 100.000 Veranstaltungen sind in jeder Parzelle vorgestellt. Die Ereignisse, die in den GFP Weiche sind grün markiert.
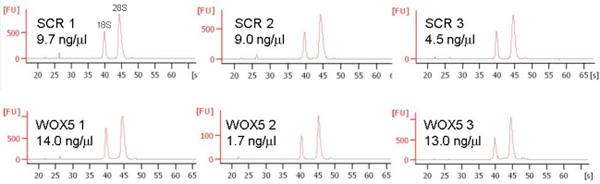
Abbildung 5. Vertreter RNA Extraktion von 10.000 sortierten Zellen. Die Zellen wurden direkt in RNA-Extraktionspuffer (QIAGEN) sortiert wurde die RNA gereinigt und geprüft für Konzentration, Reinheit und Integrität auf einem 2100 Bioanalyzer (Agilent). Drei Wiederholungen für beide Marker-Linien angezeigt werden.
Diskussion
Protoplasten können im Prinzip aus einer Vielzahl von pflanzlichen Geweben abgeleitet werden, die Optimierung der günstigen Bedingungen ermöglicht eine wesentlich bessere RNA Qualität und Quantität. Sowohl die Protoplastierung Lösung und die Wahl Inkubationspuffer beeinflussen diesen Aspekt.
Viele verschiedene fluoreszierende Proteine verwendet werden können, je nach den Fähigkeiten der FACS verwendet werden, zB GFP, RFP, YFP, CFP oder ihren vielen Varianten und Deriva...
Danksagungen
Diese Arbeit wurde von der National Science Foundation (Grant No. DBI 0519984) und die National Institutes of Health (Grant No. 5R01GM078279) unterstützt ..
Materialien
| Name | Company | Catalog Number | Comments |
| 250 μm nylon mesh | Sefar Filtration | NITEX 03-250/50 | |
| 100 μm nylon mesh | Sefar Filtration | NITEX 03-100/47 | |
| Square petri dishes | Fisher Scientific | 08-757-10k | |
| Phytatrays | Sigma-Aldrich | P1552 | |
| Murashige and Skoog Basal Medium (MS) | Sigma-Aldrich | M5519 | |
| sucrose | Fisher Scientific | S5-3 | |
| MES | Sigma-Aldrich | M2933 | |
| KOH | Sigma-Aldrich | P1767 | 10 M stock |
| Eclipse 90i microscope | Nikon Instruments | ||
| Cellulase R-10 | Yakult Pharmaceutical | ||
| Macerozyme R-10 | Yakult Pharmaceutical | ||
| D-mannitol | Sigma-Aldrich | M9546 | |
| KCl | Sigma-Aldrich | P8041 | 1 M stock |
| BSA | Sigma-Aldrich | A3912 | |
| β-mercapt–thanol | Calbiochem | 444203 | |
| CaCl2 | Sigma-Aldrich | C2536 | 1 M stock |
| orbital shaker | Labline Instruments | ||
| 40 μm cell strainer | BD Biosciences | 352340 | |
| conical 15 ml tubes | BD Biosciences | 352196 | |
| table centrifuge | Sorvall, Thermo Scientific | Legend RT | |
| NaCl | Sigma-Aldrich | S3014 | |
| FACSAria | BD Biosciences | ||
| 1.5 ml microfuge tubes | VWR international | 20170-38 | |
| RNeasy micro kit | Qiagen | 74004 | |
| WT-Ovation Pico RNA Amplification System | NuGEN | 3300_12 | |
| FL-Ovation cDNA Biotin Module V2 | NuGEN | 4200_12 |
Referenzen
- Sheen, J. Signal transduction in maize and Arabidopsis mesophyll protoplasts. Plant Physiol. 127, 1466-1475 (2001).
- Wysocka-Diller, J. W., Helariutta, Y., Fukaki, H., Malamy, J. E., Benfey, P. N. Molecular analysis of SCARECROW function reveals a radial patterning mechanism common to root and shoot. Development. 127, 595-603 (2000).
- Blilou, I., Xu, J., Wildwater, M., Willemsen, V., Paponov, I., Friml, J., Heidstra, R., Aida, M., Palme, K., Scheres, B. The PIN auxin efflux facilitator network controls growth and patterning in Arabidopsis roots. Nature. 433, 39-44 (2005).
- Gifford, M. L., Dean, A., Gutierrez, R. A., Coruzzi, G. M., Birnbaum, K. D. Cell-specific nitrogen responses mediate developmental plasticity. Proc Natl Acad Sci U S A. 105, 803-808 (2008).
- Bargmann, B. O. R., Birnbaum, K. D. Positive fluorescent selection permits precise, rapid, and in-depth overexpression analysis in plant protoplasts. Plant Physiol. 149, 1231-1239 (2009).
- Petersson, S. V., Johansson, A. I., Kowalczyk, M., Makoveychuk, A., Wang, J. Y., Moritz, T., Grebe, M., Benfey, P. N., Sandberg, G., Ljung, K. An Auxin Gradient and Maximum in the Arabidopsis Root Apex Shown by High-Resolution Cell-Specific Analysis of IAA Distribution and Synthesis. Plant Cell. 21, 1659-1668 (2009).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten