É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Separação de células de fluorescência Activated de Protoplastos Usina
Neste Artigo
Resumo
Um método para isolar tipos específicos de células a partir de material vegetal é demonstrada. Esta técnica emprega linhas marcador transgênicos expressando proteínas fluorescentes em tipos de células particular, celular dissociação e classificação de fluorescência celular ativado. Além disso, uma instalação de crescimento é estabelecido aqui que facilita o tratamento de Arabidopsis thaliana Mudas antes da separação de células.
Resumo
De alta resolução, tipo celular específico de análise da expressão do gene aumenta muito a compreensão da regulação do desenvolvimento e as respostas aos estímulos do ambiente em qualquer organismo multicelular. A hibridização in situ e visualização gene repórter pode até certo ponto ser utilizados para este fim, mas em alta resolução quantitativa RT-PCR ou high-throughput análise de transcriptoma-wide o isolamento de RNA a partir de tipos de células em particular é necessária. Dissociação do tecido celular expressando um marcador da proteína fluorescente em um tipo específico de célula e separação de células subseqüentes Fluorescence Activated (FACS) faz com que seja possível recolher uma quantidade suficiente de material para extração de RNA, análise, síntese / amplificação e microarray de cDNA.
Um extenso conjunto de células de tipo específico linhas repórter fluorescente está disponível para a comunidade de pesquisa da planta. Neste caso, duas linhas marcador da raiz de Arabidopsis thaliana são usados: P SCR:: GFP (endoderme e centro quiescente) e P WOX5:: GFP (centro quiescente). Um grande número (milhares) de mudas são cultivadas em sistema hidropônico ou em placas de ágar e colhida para obter material suficiente raiz para análise posterior. Dissociação celular de material vegetal é obtido por digestão enzimática da parede celular. Este procedimento faz uso de plasmólise osmolaridade induzida por alta e celulases comercialmente disponíveis, pectinases e hemicelulases para liberar protoplastos em solução.
FACS da GFP células positivas faz uso da visualização do verde contra o espectro de emissão vermelha de protoplastos animado por um laser nm 488. GFP-positivos protoplastos podem ser distinguidos pelo seu aumento da proporção de verde para emissão vermelha. Protoplastos são normalmente classificadas diretamente em tampão de extração de RNA e armazenados para posterior processamento em um momento posterior.
Esta técnica é revelado para ser simples e viável. Além disso, é mostrado que ele pode ser usado sem dificuldades para isolar um número suficiente de células para análise de transcriptoma, mesmo para tipos de células muito escassos (por exemplo, células centro quiescente). Por último, uma instalação de crescimento para mudas de Arabidopsis é demonstrado que permite tratamento sem complicações da plantas, antes da separação de células (por exemplo, para o tipo específico de célula de análise de respostas de estresse bióticos ou abióticos). Suplementares potenciais usos para FACS de protoplastos de plantas são discutidos.
Protocolo
1) Preparação do material vegetal
- Protoplastos pode ser derivado de muitas espécies de plantas diferentes e tecidos, desde que a mistura certa de parede celular enzimas digestivas é utilizado 1. Antes de um experimento em larga escala é realizada, a digestão de pequena escala do material é aconselhável, a fim de avaliar a eficiência protoplasting do tecido, enzimas, etc, e para estimar as células por cento positivo para separação de células. Aqui, protoplastos derivados das raízes de mudas de Arabidopsis thaliana que tipo de célula, especificamente expressam a proteína verde fluorescente (GFP) são usados. A endoderme e do centro de repouso são marcadas por P SCR:: GFP e do centro quiescente por P WOX5:: GFP 2,3 (Figura 1).
- Mudas são cultivadas em sistema hidropônico em phytatrays (Sigma; Figura 2a) em um filtro de nylon (malha 250 mm; NITEX) que permite que as raízes crescem através no meio de crescimento (0,22% w / v Murashige e Skoog Médio Basal [Sigma], 1% w / v de sacarose, 0,05% w / v MES [2 - (N-morfolino) ácido ethanesulfonic], pH 5,7 com KOH). Alternativamente, as plantas podem ser cultivadas em cima de um filtro de nylon (100 mesh mm) posicionados verticalmente em placas de ágar 1% (Figura 2b).
- O uso dos filtros acima referidos, não só ajuda na colheita das raízes, ele também facilita o tratamento adicional fácil das mudas, se desejar. Os filtros permitem que as mudas a serem transferidos em massa para phytatrays novo ou placas de ágar suplementado com um catalisador de interesse. Por exemplo, o conjunto phytatray até tem sido utilizado para análise de células de tipo específico da resposta transcricional a tratamento de nitrogênio em mudas de Arabidopsis 4.
- Microscopicamente garantir que o seu marcador fluorescente é corretamente expressa (especialmente quando se utiliza a opção de tratamento, como a célula tipo específico marcadores si pode ser influenciada pelo tratamento). Neste caso, as mudas são inspecionadas no microscópio de fluorescência (Nikon; Figura 1). Note-se que células de tipo específico linhas marcador fluorescente deve ser caracterizada inicialmente sob um microscópio confocal para determinar exatamente que tipos de células são marcadas e determinar a variabilidade de expressão.
2) Preparação da solução protoplasting
- Dissolver 1,25% w / v celulase (Yakult), 0,3% w / v Macerozyme (Yakult), 0,4 M D-manitol, 20 mM MES e 20 mM KCl (a partir de um estoque de M 1) em água desmineralizada e ajustar o pH para 5,7 com 1 M Tris / HCl pH 7.5. Esta solução será ligeiramente turvo.
- Aquecer a solução a 55 ° C por 10 minutos (a solução ficará claro) e deixe esfriar até a temperatura ambiente antes da adição de 0,1% w / v BSA (albumina bovina), 10 mM CaCl 2 e 5 mM β-mercaptoetanol .
3) A colheita e protoplasting do material vegetal
- As raízes são colhidas por raspagem-los fora da tela de náilon com um bisturi e depositado em um frasco contendo a solução protoplasting. Geralmente, 10 ml de solução protoplasting é utilizado por 1.500 raízes das mudas.
- Agitar o frasco suavemente (75 rpm) em temperatura ambiente por uma hora. Um maior tempo de incubação pode aumentar o rendimento de protoplastos, mas também irá adicionar ao efeito da protoplasting se na expressão do gene.
- Filtrar a solução com um filtro de protoplastos de células 40 mM (BD Falcon) e dividir a solução sobre tubos de 15 ml cônico (BD Falcon).
- Girar os tubos em uma centrífuga basculante para 10 minutos a 500 G. Observe que esta velocidade de centrifugação depende do tipo de protoplastos utilizados, a sua fragilidade e da quantidade de restos celulares produzidos durante o tratamento enzimático.
- Remover a maior parte do sobrenadante, ressuspender o protoplastos na solução restante e inspecioná-los ao microscópio (Figura 3).
- Fazer uso de um hemocitômetro para estimar o número e densidade de protoplastos. A densidade celular vai determinar a velocidade de injeção da amostra, os eventos por segunda taxa e, portanto, o tempo total necessário para classificar as células na FACS (ver também secção 4.3).
- 3.7) Ou vá diretamente para FACS ou lavar e ressuspender o protoplastos em uma solução de incubação, como a W5 (154 mM NaCl, 125 mM CaCl2, KCl 5 mM, 5 mM MES, pH ajustado para 5,7 com KOH) ou solução protoplasting sem a adicionado enzimas. Especialmente quando olhar para as mudanças de transcrição, é de importância para minimizar a exposição das amostras às condições que podem influenciar a expressão gênica, como alterar o buffer os protoplastos são mantidos dentro Portanto, é recomendável manter o protoplastos em solução e proceder protoplasting FACS para o mais rapidamente possível.
4) separação de células de fluorescência Activated de protoplastos
- Ligar e preparar o classificador de celular. Aqui, um FACSAria (BD) é usado.
- Criar um fluxo de fluxo com 100 m &u; m bico e uma pressão de 20 psi bainha.
- A densidade de células ea velocidade de injeção de amostra pode ser ajustado para o experimento em particular com base em se um rendimento melhor ou maior velocidade alcançável é desejado. Temos classificadas com sucesso com densidades de até 10 milhões de células / ml.
- Use a opção de agitação da amostra na FACS para evitar a sedimentação dos protoplastos. Se o entupimento da FACS é um problema, existem três etapas para solucionar problemas possíveis: 1. Executar uma amostra backflush-line. 2. Diluir sua suspensão de protoplastos para reduzir a densidade. 3. Limpar a solução de protoplastos, repetindo a etapa de filtração (3.3) após a centrifugação e ressuspensão.
- Prepare o aparelho para medir a dispersão para a frente (FSC), dispersão lateral (SSC) e emissão a 530 / 30 nm para a GFP e 610/20 nm do espectro vermelho autofluorescência (RSA), após a excitação por um laser nm 488. Estes são em essência os únicos parâmetros utilizados para isolar a GFP positivas protoplastos. Aqui, as configurações de tensão foram os seguintes: FSC - 60V, SSC 250V, 350V e GFP RSA 335V. Observe que as configurações de tensão ideal será diferente para cada FACS e vai mesmo ter de ser ajustada ao longo de todo o ordenador de celular.
- Começar pela criação de um dotplot para dispersão frontal versus dispersão lateral. Aplicar as configurações de tensão de forma que os eventos medidos estão centrados na trama.
- Em seguida, crie um gráfico de pontos de verde contra vermelho sinais de fluorescência. Aplicar as configurações de tensão de forma que os eventos medidos produzir uma população centrada diagonal na trama quando se olha para uma suspensão de protoplastos do tipo selvagem (não-GFP). A suspensão de protoplastos derivados de uma linha de marcador GFP irá produzir uma população claras de verde fluorescente eventos nunca vistos em amostras do tipo selvagem.
- Definir restrições de compensação para ajustar sobreposição espectral entre a GFP e RSA. Configurações restrição adequada compensação permitirá uma melhor separação dos protoplastos GFP-positivo do protoplastos não-GFP e detritos. As restrições usadas aqui são as seguintes: RSA, menos GFP 17,91%.
- Configurar uma porta para identificar GFP-positivos eventos, um controle negativo de não-GFP protoplastos deve ser utilizada para auxiliar na definição dos limites do portão (Figura 4).
- Implementar um corte de dispersão para a frente a fim de deixar pequenos detritos para fora da análise. Visualize os eventos GFP-positivo na trama FSC versus SSC para ajudar a determinar o posicionamento da corte. Aqui, o corte foi fixada em 5000. Note que o FACS contará detritos como ordenar os eventos e uma amostra com níveis elevados de detritos pode ter um por cento eventos diferentes GFP positivos que o esperado. Isso não é necessariamente um problema. No entanto, mais detritos na amostra, o mais longo do tipo vai demorar.
- Dependendo da experiência e da abundância do tipo de célula a ser analisada, defina o modo de precisão FACS tanto para o rendimento ideal ou ótima pureza das células classificadas.
- Para a extração de RNA, preparar tubos de coleta (tubos de 1,5 ml de microcentrífuga) com a quantidade adequada de RNA tampão de extração. Com esta configuração, 20.000 eventos de classificação vai render um volume total de aproximadamente 100 l, que foram classificados em até 350 mL de tampão de extração (RNeasy micro kit, QIAGEN). Mix amostras após a conclusão do tipo de suspensão de células pode piscina no topo.
- Armazenar amostras ou ir diretamente para extração de RNA. Análises de microarranjos de sucesso foram pré-formados com o RNA extraído de tão pouco como 500 eventos classificados. Aqui, nós usamos um kit de extração RNeasy micro (QIAGEN), o WT-Ovation Pico sistema de amplificação de RNA e FL-Ovation cDNA Biotina Módulo V2 (NuGEN).
Resultados representante
Um phytatray de aproximadamente 1.500 de uma semana de idade SCR P:: mudas GFP rendeu cerca de 60.000 protoplastos (medida por hemocitômetro). 2,6% dos 65 mil eventos FACS-processados foram definidos como sendo GFP-positivos e foram classificadas (Figura 4b).
Oito placas de aproximadamente 1.500 de quatro dias de idade WOX5 P:: mudas GFP cada (12.000 total) rendeu cerca de 30 milhões de protoplastos (medida por hemocitômetro). 0,063% dos 16 milhões eventos FACS-processados foram definidos como sendo GFP-positivos e foram classificadas (Figura 4c).
10.000 eventos classificados são normalmente utilizados para extração de RNA e pode render 20-140 ng total de RNA (Figura 5).
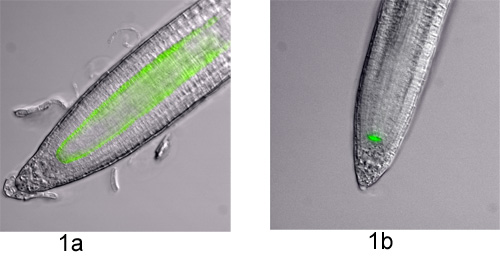
Figura 1. Tipo específico de células GFP linhas marcador na raiz Arabidopsis. Microscopia de fluorescência imagens foram tiradas com contraste de interferência diferencial (DIC) e um filtro de GFP em um microscópio Eclipse 90i (Nikon) rodando em software Metamorph (dispositivos Molecular). As imagens DIC e GFP foram sobrepostos para fins de visualização. As duas linhas de marcador usado neste experimento visuais são mostradas;a) P SCR:: GFP e b) P WOX5:: GFP.

Figura 2. Mudas de plantas condições de crescimento. Foram cultivadas em um controlador ambientais hydroponically em phytatrays (a) ou em placas de ágar posicionados verticalmente (b).
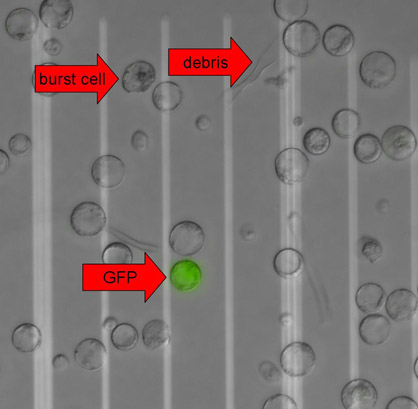
Figura 3. Protoplastos de plantas expressando GFP. Microscopia de fluorescência imagens foram tiradas com contraste de interferência diferencial (DIC) e um filtro de GFP em um microscópio Eclipse 90i (Nikon) rodando em software Metamorph (dispositivos Molecular). As imagens DIC e GFP foram sobrepostos para fins de visualização. Setas indicam uma célula estourar, restos celulares e um protoplastos GFP-positivo. A distância entre duas linhas brancas é 50 mm.
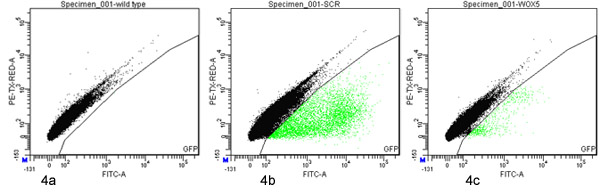
Figura 4. Separação de células ativadas por fluorescência da GFP positivas protoplastos Protoplastos derivados de tipo selvagem (a) P SCR:.: GFP (b) ou P WOX5:: GFP (c) linhas marcador foram analisados e classificados com uma FACSAria (BD) usando portas definidas em um dotplot de verde (530/30 nm; eixo x) versus vermelho (610/20 nm; eixo y) de fluorescência. 100 mil eventos são apresentados em cada parcela. Os eventos abrangidos pelo portão de separação GFP são destaque verde.
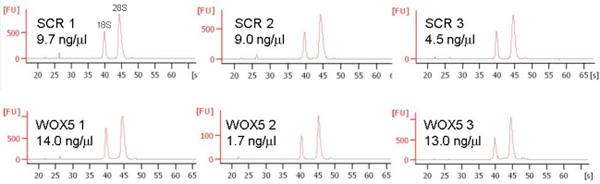
Figura 5. Representante RNA extrações de 10.000 células classificadas. As células foram classificadas diretamente para extração de RNA buffer (QIAGEN), o RNA foi purificado e sua pureza, concentração e integridade em um Bioanalyzer 2100 (Agilent). Três repetições para ambas as linhas marcador são mostrados.
Discussão
Protoplastos pode, em princípio, ser derivado de uma variedade de tecidos vegetais, otimizando as condições favoráveis irá aumentar RNA qualidade e quantidade. Tanto a solução protoplasting eo buffer de incubação eletiva utilizado irá influenciar nesse aspecto.
Muitos diferentes proteínas fluorescentes pode ser usada, dependendo das capacidades do FACS usado, por exemplo, GFP, RFP, YFP, PCP ou suas variantes muitos e derivados. A expressão dos marcadores pode ser c...
Agradecimentos
Este trabalho foi financiado pela National Science Foundation (Grant no. DBI 0.519.984) e os Institutos Nacionais de Saúde (Grant no. 5R01GM078279) ..
Materiais
| Name | Company | Catalog Number | Comments |
| 250 μm nylon mesh | Sefar Filtration | NITEX 03-250/50 | |
| 100 μm nylon mesh | Sefar Filtration | NITEX 03-100/47 | |
| Square petri dishes | Fisher Scientific | 08-757-10k | |
| Phytatrays | Sigma-Aldrich | P1552 | |
| Murashige and Skoog Basal Medium (MS) | Sigma-Aldrich | M5519 | |
| sucrose | Fisher Scientific | S5-3 | |
| MES | Sigma-Aldrich | M2933 | |
| KOH | Sigma-Aldrich | P1767 | 10 M stock |
| Eclipse 90i microscope | Nikon Instruments | ||
| Cellulase R-10 | Yakult Pharmaceutical | ||
| Macerozyme R-10 | Yakult Pharmaceutical | ||
| D-mannitol | Sigma-Aldrich | M9546 | |
| KCl | Sigma-Aldrich | P8041 | 1 M stock |
| BSA | Sigma-Aldrich | A3912 | |
| β-mercapt–thanol | Calbiochem | 444203 | |
| CaCl2 | Sigma-Aldrich | C2536 | 1 M stock |
| orbital shaker | Labline Instruments | ||
| 40 μm cell strainer | BD Biosciences | 352340 | |
| conical 15 ml tubes | BD Biosciences | 352196 | |
| table centrifuge | Sorvall, Thermo Scientific | Legend RT | |
| NaCl | Sigma-Aldrich | S3014 | |
| FACSAria | BD Biosciences | ||
| 1.5 ml microfuge tubes | VWR international | 20170-38 | |
| RNeasy micro kit | Qiagen | 74004 | |
| WT-Ovation Pico RNA Amplification System | NuGEN | 3300_12 | |
| FL-Ovation cDNA Biotin Module V2 | NuGEN | 4200_12 |
Referências
- Sheen, J. Signal transduction in maize and Arabidopsis mesophyll protoplasts. Plant Physiol. 127, 1466-1475 (2001).
- Wysocka-Diller, J. W., Helariutta, Y., Fukaki, H., Malamy, J. E., Benfey, P. N. Molecular analysis of SCARECROW function reveals a radial patterning mechanism common to root and shoot. Development. 127, 595-603 (2000).
- Blilou, I., Xu, J., Wildwater, M., Willemsen, V., Paponov, I., Friml, J., Heidstra, R., Aida, M., Palme, K., Scheres, B. The PIN auxin efflux facilitator network controls growth and patterning in Arabidopsis roots. Nature. 433, 39-44 (2005).
- Gifford, M. L., Dean, A., Gutierrez, R. A., Coruzzi, G. M., Birnbaum, K. D. Cell-specific nitrogen responses mediate developmental plasticity. Proc Natl Acad Sci U S A. 105, 803-808 (2008).
- Bargmann, B. O. R., Birnbaum, K. D. Positive fluorescent selection permits precise, rapid, and in-depth overexpression analysis in plant protoplasts. Plant Physiol. 149, 1231-1239 (2009).
- Petersson, S. V., Johansson, A. I., Kowalczyk, M., Makoveychuk, A., Wang, J. Y., Moritz, T., Grebe, M., Benfey, P. N., Sandberg, G., Ljung, K. An Auxin Gradient and Maximum in the Arabidopsis Root Apex Shown by High-Resolution Cell-Specific Analysis of IAA Distribution and Synthesis. Plant Cell. 21, 1659-1668 (2009).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados