Method Article
Avidität-basierte extrazelluläre Wechselwirkung Screening (AVEXIS) für die Erkennung von Scalable Low-Affinität extrazellulären Rezeptor-Ligand-Wechselwirkungen
In diesem Artikel
Zusammenfassung
AVEXIS ist ein Hochdurchsatz-Protein-Interaktion Assay entwickelt, um systematisch Bildschirm für neue extrazelluläre Rezeptor-Ligand-Paare in der Zellerkennung Prozessen beteiligt sind. Es ist speziell entwickelt, um transienten Protein-Interaktionen, die schwer zu identifizieren, die mit anderen hohen Durchsatz Ansätze sind zu erkennen.
Zusammenfassung
Extrazelluläre Protein-Protein-Wechselwirkungen zwischen sezernierten oder Membran-gebundene Proteine sind entscheidend für die Einleitung sowohl Kommunikation und die Kohäsion in vielzelligen Organismen. Vorhergesagten Proteine zu bilden extrazelluläre Interaktionen um rund ein Viertel der menschlichen Gene 1 kodiert sind, aber trotz ihrer Bedeutung und Fülle, die Mehrzahl dieser Proteine haben keine Bindungspartner dokumentiert. In erster Linie ist dies auf Grund ihrer biochemischen Widerspenstigkeit: Membran-Proteine eingebettet sind schwer in nativer Konformation solubilisieren und enthalten strukturell wichtige posttranslationale Modifikationen. Auch sind die Interaktion Affinitäten zwischen Rezeptor-Proteine oft durch äußerst geringe Stärke der Wechselwirkung (Halbwertszeiten <1 Sekunde) entgegensteht, ihr Nachweis mit vielen häufig verwendeten Hochdurchsatzmethoden 2 gekennzeichnet.
Hier beschreiben wir einen Test, AVEXIS (Gier-basierte extrazellulärenlar Interaction Screen), die diese technischen Herausforderungen ermöglicht die Detektion von sehr schwachen Protein-Wechselwirkungen (t 1/2 ≤ 0,1 sec) mit einer niedrigen Rate an falsch positiven 3 umgeht. Der Assay wird in der Regel in einem Format mit hohem Durchsatz durchgeführt, um die systematische Screening von vielen Tausenden von Wechselwirkungen in einem geeigneten Mikrotiterplatten-Format (1) zu ermöglichen. Es stützt sich auf die Produktion von löslichem rekombinanten Protein-Bibliotheken, die die Ektodomäne Fragmente von Zelloberflächenrezeptoren oder sekretierte Proteine in dem für Wechselwirkungen zu screenen, um enthalten, daher ist dieser Ansatz für Typ I, Typ II, GPI-gebundene Rezeptoren auf der Zelloberfläche und sezernierte Proteine, nicht aber für Multipass-Membranproteine wie Ionenkanäle oder transporter.
Die rekombinanten Protein-Bibliotheken werden unter Verwendung eines bequemen und High-Level-Säuger-Expressionssystem 4, an, dass wichtige posttranslationale Modifikationen wie Glycosylierung gewährleistention und Disulfidbindungen hinzugefügt werden. Ein biotinylierter Köder, an einer mit Streptavidin beschichtete feste Phase für das Screening geeignet erfasst werden kann, und einer enzymmarkierten pentamerisierten (β-Lactamase) Beute: exprimierten rekombinanten Proteine in das Medium und produziert in zwei Formen sezerniert. Die Bait und Prey-Proteine sind miteinander in einer binären Weise direkten Wechselwirkungen zwischen ihnen, ähnlich einem herkömmlichen ELISA (Abb. 1) zu erfassen dargestellt. Die pentamerisation der Proteine in der Beute durch eine Peptidsequenz aus dem Knorpel oligomere Matrix-Protein (COMP) erreicht und erhöht die lokale Konzentration der Ektodomänen damit eine signifikante Avidität Gewinne zu ermöglichen, auch sehr kurzlebige Wechselwirkungen zu detektieren. Durch die Normierung der Aktivitäten sowohl der Köder und Beute zu vorgegebenen Niveaus vor dem Screening, haben wir gezeigt, dass Wechselwirkungen mit monomeren Halbwertszeiten von 0,1 s mit niedrige False-Positive Raten 3 erkannt werden können.
Protokoll
1. Kompilieren einer Bibliothek von Bait und Prey-Plasmid-Expressionsvektoren
- Stellen Sie eine Liste von Zelloberflächen-Rezeptor und sezernierte Proteine von Interesse für die Wechselwirkungen abgeschirmt werden und rufen ihre Sequenzen aus einem geeigneten Protein-Sequenz-Datenbank. Die meisten Proteine mit einer N-terminalen sekretorisches Signalpeptid geeignet sind (diese sind vom Typ I, GPI-linked und sekretierte Proteine). Typischerweise würde ein Protein-Familie (zB Immunoglobulin-Superfamilie oder alle Rezeptoren beschränkt auf einen spezifischen Zelltyp wie die Erythrozyten) ausgewählt werden.
- Bestimmen des Ausmaßes der extrazellulären Regionen durch Identifizieren der Stelle des Signalpeptids und Transmembran-Region unter Verwendung von Protein-Funktion Vorhersage-Software. Wir verwenden in der Regel SignalP v3.0 5 und 6 TMHMM v2.0.
- Design-Primer-Sets, die den gesamten Ektodomäne Fragment für jeden Rezeptor verstärken. Wir haben eine Reihe von Bait und Prey-Vektoren, die für diesen Zweck geeignet sind, die eine sindvailable aus Addgene ( http://www.addgene.org/~~V ). Entwerfen der Sense-Primer um das Start-Methionin eine NotI Restriktionsenzym und eine optimale Kozak-Sequenz umfassen, das um 25 Basenpaare genaue Übereinstimmung Gen-spezifischen Sequenz (2A) gefolgt.
- Entwerfen des Antisense-Primers an Typ I-Rezeptor-Proteine unmittelbar vor der vorhergesagten Transmembrandomäne, um so die gesamte Ektodomäne als lösliches rekombinantes Protein zu exprimieren abzuschneiden. Einschließen einer AscI Restriktionsstelle in diesem Primer, die dann in demselben Raster mit einer C-terminalen Ratte Cd4d3 4 Protein-Tag 7 (2A) sollte übersetzen.
- Verstärken der Ektodomäne Fragmente aus allen Genen unter Verwendung dieser Primer und zu klonen in den Köder und Beute-Vektor. Wir finden es manchmal hilfreich, zuerst zu klonen sie in Shuttle-Vektoren vor Subklonierung in einen geeigneten Säugerexpressionsvektor wir ein Derivat des Vektors pTT3 3,4 zu verwenden. Sowohl die Köder und Beute Vektoren Contain C-terminalen Protein-Tags wie folgt (Abb. 2B).
- Köder: Ein Etikett mit Domänen 3 und 4 der Ratten-CD4 (Cd4d3 +4)-Protein durch eine Peptidsequenz, die ein Substrat für die E. coli BirA Enzym gefolgt und kann daher zu einem bestimmten Lysinrest monobiotinylierte werden (2B) . Die Cd4d3 4-Tag wird vom OX68 monoklonalen Antikörper erkannt wird und verwendet wird, um die Expression der Köderproteine durch ELISA quantifiziert. Die Köder-Proteine sind daher monomeren und monobiotinylierte. Zusätzliche Köderprotein Vektoren, wo die biotinylatable Peptid, das durch eine 6-His-Tag zur Aufreinigung ersetzt ist, sind verfügbar.
- Beute: Die Beute auch eine Ratte Cd4d3 4-Tag durch eine Peptidsequenz aus der Ratte Knorpel oligomere Matrix-Protein (COMP) und einer C-terminalen β-Lactamase-Enzym an. Die COMP-Peptid sorgt die Beute Formen Pentamere einmal zum Ausdruck gebracht (Abb. 2B).
2. Prey-und Bait-Protein-Expression
Wir verwendenein bequemer Expression auf hohem Niveau unter Verwendung Suspensionskultur der HEK293-Zelllinie 4 an unsere Protein-Bibliotheken zu erzeugen, sondern auch jedes Säuger-Expressionssystem geeignet sein. Eine handelsübliche Alternative ist die Freestyle-System. Die Zellen werden routinemäßig auf einem Schüttler (125 rpm) bei 37 ° C, 5% CO 2 bei 70% relativer Luftfeuchtigkeit. Sowohl positive als auch negative Kontrolle Proteine müssen ebenfalls angegeben werden. Die Ratte Cd4d3 4-Tag-nur-Fragment ist sowohl als Köder und Beute und eignen sich negative Kontrollen. Ebenso sind Köder und Beute-Vektoren, die eine positive Kontrolle kodieren verfügbar (Addgene); wir verwenden in der Regel die Ratte CD200-Interaktion CD200R 8.
- Saatgut den HEK293 Zellen am Tag vor der Transfektion in einer Dichte von 2,5 x 10 5 Zellen ml -1. Wir routinemäßig Kultur der Zellen in Volumina von 50 ml in Freestyle293 Medien nach den Empfehlungen des Herstellers. Um eine effiziente Biotinylierung zu gewährleisten, zu ergänzen ter Zellkulturmedium verwendet, um Köderproteine mit D-Biotin zu einer Endkonzentration von 100 pM zu erzeugen. Medium verwendet, um die Beute-Proteine zu exprimieren muss nicht mit zusätzlichen D-Biotin ergänzt werden.
- Am folgenden Tag Transfektion von Zellen mit einer geeigneten Transfektionen Reagenz (zB 293fectin). Verwenden 25 ug des Köders oder Prey-Plasmids Konstrukte, um eine 50 ml-Kultur zu transfizieren. An die biotinylierten Köder zu erzeugen, Co-Transfektion der Plasmid, welches das Köder mit einem Plasmid, das eine sekretierte Form des E. coli-Protein-Biotin-Ligase kodiert Konstrukt ( BirA), die vom Addgene ist. Co-Transfektion von Plasmiden Köder bei einem Verhältnis von 10:1 (2,5 ug) des Plasmid, welches das sezernierte Protein BirA.
- Ernte Kulturen fünf Tage nach der Transfektion durch erste Pelletierung der Zellen durch Zentrifugation bei 3000 × g für 20 Minuten durch Filtration des Überstands durch einen 0,22 uM Filter.
[Tipp: Bait und Prey-Proteins sollte unverdünnt gelagert bei 4 ° C und als eine allgemeine Richtlinie sollten innerhalb eines Jahres verwendet werden können. Fügen Sie Bakteriostatika wie 50 pg / ml Polymyxin B Sulfat oder 10 mM NaN 3, eine bakterielle Kontamination der Überstände zu verhindern.]
3. Die Normalisierung des Bait und Prey Proben
Eines der wichtigsten Merkmale des AVEXIS Test ist, dass, weil die rekombinanten Proteine sezerniert werden sie verdünnt werden kann oder konzentriert (normalisiert), um innerhalb von festgelegten Schwellenwert Aktivitäten, bevor sie in den Test eingesetzt. Wir haben gefunden, dass die Ebenen, auf denen die rekombinanten Proteine exprimiert werden eine breite Palette erstrecken - bis 4 Größenordnungen - und so die Normalisierung Schritt reduziert signifikant die Anzahl von Fehlalarmen in den Bildschirmen.
3,1 Bait Normalisierung
Hinweis: Freies D-Biotin muss von den Medien (typischerweise durch Dialyse) entfernt werden, da es mit dem biotinylierten bai konkurriertt-Protein für die Bindung an Bindungsstellen Streptavidin.
- Übertragen Köder Überstände in Dialyseschlauch, Labels, sicher befestigen und Dialyse umfassend gegen einen geeigneten Puffer wie PBS zu freiem Biotin zu entfernen.
[Tipp: Wir haben festgestellt, dass eine ausreichende Dialyse wird normalerweise für 6 x 50 ml Köder Überstände gegen ein Volumen von 4,5 l PBS mit 4 Veränderungen über einen Zeitraum von 24 Stunden erreicht. Unzureichende Dialyse kann durch eine Hemmung der Protein-Bindung bei niedrigen Verdünnungen bei der Köder Schritt der Normalisierung beobachtet werden (Abb. 3A).
- Führen eines ELISA, um die relativen Ebenen, auf denen die Proteine exprimiert werden Köder zu bestimmen. Herstellung einer Verdünnungsreihe der biotinylierten Köderproteine und fangen sie an einer Streptavidin-beschichteten Mikrotiterplatte. Verwenden Sie die Ratte Cd4d3 4 tag zur Feststellung und Quantifizierung der aufgenommenen Köder-Proteine mit einem monoklonalen Antikörper (OX68). Konzentrierter oder verdünnter Köderproteine dem Mindestbetrag erforderlich ist, um alle zu sättigen Biotin Bindungsstellen in die Vertiefung einer Streptavidin-beschichteten Platte.
- Block die Vertiefungen einer mit Streptavidin beschichteten Platte mit Phosphat-gepufferter saline/0.1% Tween-20 (PBST) / 2% Rinderserumalbumin (BSA) für 15 Minuten.
- In einer separaten Platte, um eine Verdünnungsreihe (vier 1.03 Verdünnungen sind normalerweise ausreichend) der Köderproteine in PBST / 2% BSA und Übertragung in die Vertiefungen einer mit Streptavidin beschichteten Platte. Inkubation für 1 Stunde bei Raumtemperatur.
- Waschen in PBST 3x. Geben Sie 100 ul 2 ug / ml Maus-anti-Ratte IgG Cd4d3 4 (OX68) Inkubation für 1 Stunde bei Raumtemperatur.
- Waschen in PBST 3x. Fügen Sie 100 ul Anti-Maus-alkalische Phosphatase bei 1:5.000-Verdünnung in PBST / 2% BSA und Inkubation für 1 Stunde bei Raumtemperatur.
- Waschen in PBST 3x und Erfüllung eines letzten Waschen in PBS, um das restliche Reinigungsmittel zu entfernen. Geben Sie 100 ul 1 mg / ml Substrat in Diethanolamin-Puffer (10% Diethanolamin (v / v), 0,5 mM MgCl 2, 10 mM NaN 3, pH9.8, gespeichert vor Licht geschützt bei 4 104° C).
- Nach einer Stunde messen Absorption bei 405 nm. Repräsentative Köder Normalisierung Ergebnisse sind in 3A gezeigt.
- Köder-Proteine, die auf einem Niveau, das ausreicht, um alle Biotin-Bindungsstellen eines Streptavidin-beschichteten Platte sättigen sind ausgedrückt werden, wenn unverdünnt verwendet, sollte konzentriert mit Vivaspin Konzentratoren (Vivascience, 10 kDa MWCO) werden. Als Richtwert schätzen wir, dass ~ 5 ug eines typischen Köderprotein (~ 80 kDa) ausreichen, um eine 96-Well-Platte zu sättigen ist.
- Köder-Proteine in großen Mengen exprimiert in PBST / 2% BSA auf eine Konzentration ausreicht, um die Streptavidin-beschichtete Platte zu sättigen verdünnt werden. Die Aktivität der Köder sollte nach Verdünnung oder Konzentration nachgeprüft werden, um sicherzustellen, dass sie all die Biotin-Bindungsstellen in der Streptavidin-beschichteten Platte zu sättigen.
3,2 Prey Normalisierung
- Quantifizierung der relativen Ebenen, auf denen die Beute-Proteine exprimiert werden mit dem β-LactamaseEnzymaktivität.
- Stellen Sie zunächst eine Verdünnungsreihe der Überstand, der die Beute Proteine in PBST / 2% BSA-Puffer. Dann fügen Sie 20 ul der Verdünnung auf 60 ul einer 242 uM (0,125 mg / mL) Nitrocefin Lösung (Calbiochem).
- Sofortige Umsetzen der Platte zu einem Mikrotiterplatten, die eine Absorptionsmessung bei 485 nm pro Minute für 20 Minuten bei Raumtemperatur erfolgt. Repräsentative Beute Normalisierung Ergebnisse sind in 3B gezeigt ist.
- Konzentrat Proben unter Verwendung Zentrifugalkonzentratoren oder verdünnter sie in PBST / 2% BSA, einen Schwellenwert-Aktivität von ~ 2 nmol min -1 Umsatz von Nitrocefin erreichen. Praktisch wird dies erreicht, wenn alle Nitrocefin innerhalb von ~ 7 Minuten hydrolysiert wird.
4. AVEXIS Screening Procedure
- Waschen Sie eine Streptavidin-beschichtete Platte in PBST.
- Geben Sie 100 ul PBST / 2% BSA in jede Vertiefung und inkubieren Sie für 30 Minuten, um alle störenden Protein-Bindungsstellen innerhalb jeder gut zu blockieren.
[Tipp: auf jeden Waschpufferrückstände nach Waschschritte entfernen, wird die Platte geklopft - kräftig - auf Küchenpapier]
- Blocking-Lösung mit einer Smart-Flick von der Platte über einem Waschbecken und 100 ul normalisierte biotinylierten monomeren Köder in die entsprechenden Vertiefungen pipettieren und für 1 Stunde bei Raumtemperatur.
- Entfernen Sie die Köder Proben aus der Platte durch Umdrehen der Platte und elegant flicking über ein Waschbecken, und 3 x waschen in PBST.
- Geben Sie 100 ul der normierten pentamerized Beute in jede Vertiefung konstruieren und inkubieren Sie bei Raumtemperatur für 1 Stunde, waschen, wie in Schritt 4.4 und führen Sie eine letzte Waschung mit PBS nur.
- In 60 ul 242 uM Nitrocefin-Lösung (5 mg auflösen Nitrocefin in 500 l DMSO, gut durchmischen und dann weiter verdünnen Nitrocefin in 40 ml PBS und durch ein 0,22 um Filter zu passieren, bevor Sie zu den Platten) und bei Raumtemperatur inkubieren . Positive Interaktionen beobachtet werden kannmit dem Auge, da die gelbe Nitrocefin wird zu einer roten Farbe hydrolysiert. Quantifizierung durch Messung der Absorption bei 485 nm mit einem Platten-Lesegerät. Eine typische Platte und Quantifizierung ist in Abb. 4.
[Tipp: Positive Interaktionen sind in der Regel innerhalb von 2 Stunden bei Raumtemperatur beobachtet, obwohl Platten sollte bei 4 ° C für 16 Stunden angeordnet werden, um sicherzustellen, dass alle potenziellen Wechselwirkungen erkannt werden. Wir haben auch festgestellt, dass gefällte Nitrocefin und Unvollkommenheiten auf dem Kunststoff der Mikrotiterplatte wie Kratzer / Fingerabdrücke können gelegentlich zu artifizielle Fehlalarme bei 485 nm führen trotz keiner offensichtlichen Nitrocefin Hydrolyse. Es ist daher ratsam, Fotos von den Platten zu ergreifen, um visuell aufzuzeichnen Nitrocefin Umsatzes in die Vertiefungen geben.]
5. Repräsentative Ergebnisse
Ein Beispiel eines repräsentativen AVEXIS Abschirmblech ist in 4 gezeigt. Man sollte Hydrolyse (gelb bis rot) des Nitrocefin Substrat in das beobachtenpositiven Kontrollvertiefungen (sowohl der Antikörper-vermittelten Kontrolle Beutefang und auch positive Kontrolle Interaktion) innerhalb von etwa zehn Minuten. Einige Wechselwirkungen innerhalb des Bildschirms werden darüber hinaus in dieser Zeitskala beobachtet, aber andere können länger dauern (einige Stunden) zu erscheinen. Typischerweise ist eine Hit-Rate von rund 0,4 bis 0,6% (eine positive Wechselwirkung für jeweils 150-250 Interaktionen durchleuchtenden) typisch, abhängig von den Protein-Bibliotheken werden gezeigt.

Abbildung 1. Identifizierung von Rezeptor-Ligand-Interaktionen vermitteln zelluläre Prozesse mit Hilfe AVEXIS Anerkennung. Das Schema zeigt zwei interagierenden Zellen (A und B) Austausch von Informationen durch Wechselwirkungen zwischen Membran-Embedded-Rezeptor-Proteine hergestellt. AVEXIS ist eine skalierbare Protein-Interaktions-Test speziell für die neuen Rezeptor-Ligand-Paaren, die durch sehr schwache Wechselwirkung Stärken auszeichnen zu identifizieren. Rekombinante soUnlöslich Protein-Bibliotheken, die die gesamte Ektodomäne des Zelloberflächenrezeptor Repertoire der beiden Zellen werden entweder als pentamere β-Lactamase-markierte Beute kompiliert (Zelle A) oder Biotin-markierten monomeren Köder (für die Zelle B). Die pentamerisation der Beute erhöht die lokale Konzentration der Ektodomänen zu einem Gesamtbild Avidität Verstärkung zu bewirken, so dass sogar sehr kurzlebige Wechselwirkungen detektiert werden kann. Der Köder Bibliothek wird auf Streptavidin-beschichteten Platten angeordnet sind und systematisch sondiert auf Wechselwirkungen mit den Beuteproteine. Nach einer kurzen Waschung, werden alle erfassten Beute unter Verwendung der β-Lactamase-Aktivität mit der Beute zugeordnet.
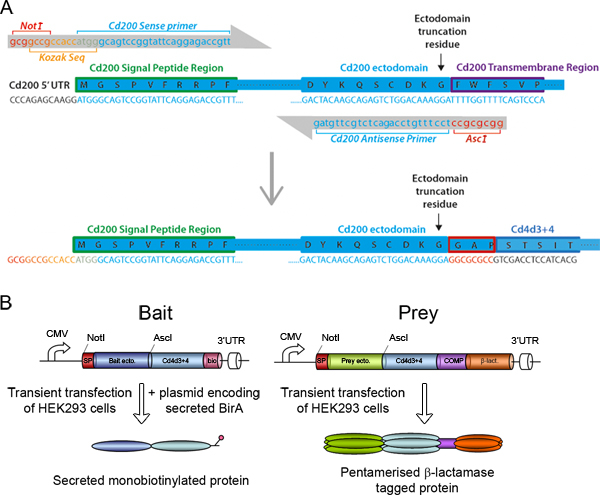
Abbildung 2. Bait-und Prey-Konstruktes Design. (A) Die Ratte CD200-Rezeptor-Protein wird als ein Beispiel dafür, wie die gesamte Ektodomänen von Zelloberflächen-Rezeptorproteinen bestimmt und Primer werden entworfen, um sowohl Köder zu bereitgestelltund Beute Expressionskonstrukte. Die cDNA und translatierten Proteinsequenz aus der N-terminalen Signalpeptid der Membran-überspannenden Transmembranregion von Ratten-CD200 gezeigt. Ein Sense-Primer wurde entwickelt, um eine 5'-NotI-Restriktionsenzym-Stelle und eine optimale Kozak-Sequenz von 25 Basen der Sequenz genaue Übereinstimmung der CD200-cDNA-Sequenz umfassen, gefolgt. Die Antisense-Primer enthält eine AscI Ort und 25 Basen genaue Übereinstimmung Sequenz unmittelbar stromaufwärts des ausgewählten Ektodomäne Abschneiden Rückstand. Um die Ektodomäne in Rahmen mit der Ratte Cd4d3 4 Tag halten, sollte die AscI Standort wie Gly-Ala-Pro übersetzen. (B) schematische Darstellungen der Köder und Beute Expressionskonstrukte. Jeder Rezeptor enthält Ektodomänen durch NotI und AscI Standorten und einem 4-Tag Cd4d3 flankiert. Die Köder enthalten eine C-terminale Peptid-Sequenz, die spezifisch durch Co-Transfektion kann mit einem Plasmid die Expression eines sezernierten BirA Enzym monobiotinylierte. Die Beute von der Sequenz folgt pentamerisationaus dem Knorpel oligomere Matrix-Protein (COMP) und der β-Lactamase-Enzym.

Abbildung 3. Die Normalisierung der Köder und Beute-Proteine. (A) Repräsentative Beispiele für Köder Normalisierung. Eine Verdünnungsreihe von vier verschiedenen dialysiert Köder Überstände wurden durch ELISA unter Verwendung eines Anti-CD4-Tag monoklonaler Antikörper ist. Die vier Köder werden auf verschiedenen Ebenen 1> 2> 3> 4 ausgedrückt. Köder sollte auf einen Schwellenwert, der die Biotin-Bindungsstellen in jeder Vertiefung sättigt normalisiert werden. Absolut ausgedrückt-Köder können verdünnt werden, um Protein zu erhalten (zB Köder 1 könnte ~ 1:100 verdünnt werden), und schlecht exprimiert Köder eingeengt. Verminderte Werte bei niedrigen Verdünnungen werden manchmal in einigen Köder (zB Köder 2), die auf das Vorhandensein von nicht-konjugiertem D-Biotin durch unvollständige Dialyse beobachtet. (B) Die Ebenen, auf denen Proteine Beute sind exgedrückt wird quantifiziert mit der Hydrolysegeschwindigkeit von einem β-Lactamase-Substrat, Nitrocefin. In dem gezeigten Beispiel sind die drei Beute auf verschiedenen Ebenen ausgedrückt: Beute 1> 2> 3. Preys sollte auf eine Aktivität von ca. 2 nmol min -1, die Hydrolyse von 14,5 nmol Nitrocefin in ~ 7 Minuten zu vollenden (zB Beute 1) entspricht normalisiert werden. Die andere Beutetiere in diesem Beispiel sollten vor der Verwendung in Screening konzentriert werden.

Abbildung 4. Vertreter AVEXIS Screening-Ergebnisse. (A) Ein Foto von einem Vertreter AVEXIS Abschirmblech. Ein Beute-Protein wird gegen 41 verschiedene Köder sondiert und eine positive Wechselwirkung ist in gut E2 erkannt. Kontrollen in den Screening-Platten verwendet werden, umfassen: ein biotinylierter Anti-CD4-Antikörper, alle Prey-Protein zu erfassen zugegeben, um das Bohrloch (G6), eine Steuereinheit Wechselwirkung: Ratten CD200 (Köder)-CD200R (Beute) mit dem CD200R der Schwelle Verdünnung Beute verwendet (H3), 1.10 (H4) und 1:100 (H5). Negative Kontrollen waren: a Cd4d3 4 Köder sondiert mit der Prey-Protein das gesiebt (H1) und der CD200R Beute (H2). (B) Quantifizierung der Extinktionswerte des gleichen Bildschirms in dreifacher Ausführung durchgeführt. Die Datenpunkte sind der Durchschnittswert ± SD.
Diskussion
Bei lebenden Zellen in vivo beobachtet werden, zeigen ihre Plasmamembranen hochdynamische Verhalten: Ein-und Ausfahren Membranverfahren, wie sie und wenden sich mit ihren Nachbarn 2. Die physikalische Wechselwirkungen zwischen der Membran eingelagerten Rezeptorproteine, die viele dieser Kontakte zu vermitteln haben sich als äußerst schwach, so dass diese sehr beweglichen Verhalten zu ermöglichen. Es wurde geschätzt, dass monomere Stärke der Wechselwirkung so schwach wie 50 uM könnte ausreichend sein, um spontane Interaktionen bei physiologischen Rezeptor Dichten 9, die Erkennung neuartiger Interaktionen extrem herausfordernd 2,10 macht zu fahren. Die AVEXIS hier beschriebene Methode stellt eine empfindliche Methode zum Nachweis von transienten extrazellulären Protein-Protein-Interaktionen, die in eine skalierbare Art und Weise mit einer niedrigen Rate an falsch positiven umgesetzt werden können. Während andere skalierbare Verfahren wurden entwickelt, um auf extrazelluläre Wechselwirkungen 11-15,Ein Vorteil der AVEXIS ist, dass die experimentellen Parameter wie die Köder und Beute Aktivitäten wurden quantifiziert, um selbst die schwächste von Interaktionen (t 1/2 ≤ 0,1 s) unter Beibehaltung eines niedrige False-Positive-Rate 3. Darüber hinaus haben sich die schlanke und robuste Verfahren zur Probenaufbereitung, diesen Test aktivieren, damit Sie auf einem viel größeren Maßstab als andere Assays durchgeführt werden, und wir haben vor kurzem eine systematische Interaktion Bildschirm im Gesamtwert von über 16.500 potentielle Wechselwirkungen ~ 16 beschrieben.
Der Assay kann auf jedem Protein, für das es möglich ist, ein lösliches rekombinantes Protein-Fragment exprimieren durchgeführt werden. Umfasst werden also Proteine, die eine N-terminale Signalpeptid wie Typ I, GPI-linked und sekretierte Proteine enthalten. Wir haben kürzlich eine Reihe von Vektoren, die uns nun ermöglichen, Typ-II-Proteine als Köder 17 Express konzipiert. Der Test ist im Allgemeinen nicht geeignet für Multipass Membranproteine wie Ionen-Transporters oder Pumpen, da sie wahrscheinlich nicht vollständig außerhalb des Kontextes eines Plasmamembran gefaltet werden. Wir führen dies auf eine Vielzahl von unterschiedlichen Systemen angewandt und haben erfolgreich exprimiert extrazelluläre Proteine aus Zebrafisch 3,16,18, von Mensch, Maus und S. falciparum für die Interaktion Bildschirme.
Im Hinblick auf die erforderlichen Ressourcen zur Einrichtung eines AVEXIS Bildschirm haben wir festgestellt, dass ein erheblicher Engpass die Auswahl, den Bau und die vollständige Sequenzierung der Expressionsplasmide ist. Dieser Aspekt des Verfahrens kann bis zu einem Jahr auf ~ 100 Bait und Prey-Konstrukte machen, jedoch mit der sinkenden Kosten für Gensynthese haben wir jetzt im Handel begünstigen Auslagerung dieser Aspekt des Projekts. Die Säuger-Expression System zusammen mit einer großen Schütteln Gewebekultur-Inkubator (wir verwenden eine INFORS HT Multitron Cell) ermöglicht einem einzelnen Forscher zu äußern und zu normalisieren bis ~ 100 Köder und Beute-Proteine im Monat. Sobald die Proteine produziert werden und normiert, screening für Wechselwirkungen ist schnell mit bis zu zwölf 96-well Platten in bequemer pro Tag verarbeitet. Die einzige maßgeschneiderte Ausrüstung, die wir entwickelt haben, um die Herstellung einer solchen großen Anzahl von Proteinen erleichtern, ist eine Druckluft-Kolbenmotor, die verwendet werden, um bis zu fünf übergeben Überstände durch einen 0,22 &mgr; m Filter parallel ist.
Die Hauptklasse der Interaktionen, die vielleicht entgehen würden, wenn die Wechselwirkungen aus der Literatur (falsch negativ) erwartet im Vergleich sind homophile Interaktionen, obwohl einige dieser Interaktionen 3 nachgewiesen werden kann. In den Fällen, in denen zu erwartenden Wechselwirkungen nicht erkannt werden, glauben wir, dass dies auf die Bildung von homophile Interaktionen von den Beute-Proteine (a Beute "Maskierung"-Effekt), die dann verhindert die weitere Interaktionen mit Proteinen immobilisiert Köder ist. Die Frequenz, bei der Interaktionen erfasst hängt von dem Gehalt der Protein-Bibliothek gescreent werden. Als Leitfaden für den Leser, ein Bildschirm von> 30, 000 Wechselwirkungen innerhalb der Zebrafisch-Immunglobulin-Superfamilie resultierte in 188 Positives - eine Frequenz von 0,6% 3,16,18. Eine sehr schnelle Überprüfung vorgenommen, um die positive gespielt werden kann, ist zu überprüfen, um festzustellen, ob die Interaktion in beiden Köder-Beute-Orientierungen erfasst werden kann, dh, kann der gleiche Effekt unabhängig davon, ob die bindenden Proteine vorhanden sind detektiert werden entweder als ein Köder oder eine Beute. Wenn wir diese Hin und Herbewegung Verhalten beobachtet, hat die Bindung erfolgt wurde gezeigt, dass durch den Nachweis spezifischer direkten gesättigte Bindung der gereinigten Proteine mit Oberflächenplasmonresonanz. Es sollte betont werden, dass, weil wir Bindungsavidität erhöhen künstlich die pentamerising Beuteproteine, wir sind nicht der Ansicht des Tests für den Vergleich quantitativ Interaktion Stärken werden. Um biophysikalischen Bindungsparameter der Interaktionen verwenden wir monomeren Proteinen und Oberflächen-Plasmon-Resonanz erhalten. Schließlich, wenn eine Wechselwirkung identifiziert worden ist, die hallogh Durchsatz Art des Assays ist es relativ einfach, den Test zum Screening auf Reagenzien wie Antikörper oder kleine Moleküle, die die Interaktion blockiert anzupassen.
Offenlegungen
Die Autoren haben nichts zu offenbaren.
Danksagungen
Diese Arbeit wurde vom Wellcome Trust Grant Number [077108] unterstützt, um GJW ausgezeichnet.
Materialien
| Name | Company | Catalog Number | Comments |
| Name des Reagenz | Firma | Cat. Anzahl | Kommentare |
| Freestyle Medien | Invitrogen | 12338018 | |
| Nitrocefin | Calbiochem | 484400 | |
| SA-beschichteten Mikrotiterplatten | Nunc | 734-1284 | |
| OX68 Antikörper | AbDSerotec | MCA1022R | |
| Dialyseschlauch | Durchstechen | PN68100 | |
| Polymyxin B | Sigma | P4932 | |
| D-Biotin | Sigma | B4639-5G | |
| Substrat-104 | Sigma | 1040-506 | |
| Vivaspin-20 Zentrifugalkonzentratoren | Sartorius | VS2002 | |
| 0,22 um Filter | Nalgene | 190-2520 |
Referenzen
- Fagerberg, L., Jonasson, K., von Heijne, G. Prediction of the human membrane proteome. Proteomics. 10, 1141-1141 (2010).
- Wright, G. J. Signal initiation in biological systems: the properties and detection of transient extracellular protein interactions. Molecular bioSystems. 5, 1405-1405 (2009).
- Bushell, K. M., Sollner, C., Schuster-Boeckler, B. Large-scale screening for novel low-affinity extracellular protein interactions. Genome research. 18, 622-622 (2008).
- Durocher, Y., Perret, S., Kamen, A. High-level and high-throughput recombinant protein production by transient transfection of suspension-growing human 293-EBNA1 cells. Nucleic acids research. 30, e9-e9 (2002).
- Bendtsen, J. D., Nielsen, H., von Heijne, G. Improved Prediction of Signal Peptides: SignalP 3.0. Journal of molecular biology. 340, 783-783 (2004).
- Krogh, A., Larsson, B., von Heijne, G. Predicting transmembrane protein topology with a hidden markov model: application to complete genomes. Journal of molecular biology. 305, 567-567 (2001).
- Brown, M. H., Barclay, A. N. Expression of immunoglobulin and scavenger receptor superfamily domains as chimeric proteins with domains 3 and 4 of CD4 for ligand analysis. Protein engineering. 7, 515-515 (1994).
- Wright, G. J., Puklavec, M. J., Willis, A. C. Lymphoid/neuronal cell surface OX2 glycoprotein recognizes a novel receptor on macrophages implicated in the control of their function. Immunity. 13, 233-23 (2000).
- Dustin, M. L., Golan, D. E., Zhu, D. M. Low affinity interaction of human or rat T cell adhesion molecule CD2 with its ligand aligns adhering membranes to achieve high physiological affinity. The Journal of biological chemistry. 272, (1997).
- Wright, G. J., Martin, S., Bushell, K. M. High-throughput identification of transient extracellular protein interactions. Biochemical Society transactions. 38, (2010).
- de Wildt, R. M., Tomlinson, I. M., Ong, J. L. Isolation of receptor-ligand pairs by capture of long-lived multivalent interaction complexes. Proceedings of the National Academy of Sciences of the United States of America. 99, (2002).
- Jiang, L., Barclay, A. N. Identification of leucocyte surface protein interactions by high-throughput screening with multivalent reagents. Immunology. 129, 55-55 (2010).
- Urech, D. M., Lichtlen, P., Barberis, A. Cell growth selection system to detect extracellular and transmembrane protein interactions. Biochimica et biophysica acta. 1622, 117-117 (2003).
- Voulgaraki, D., Mitnacht-Kraus, R., Letarte, M. Multivalent recombinant proteins for probing functions of leucocyte surface proteins such as the CD200 receptor. Immunology. 115, 337-337 (2005).
- Wojtowicz, W. M., Wu, W., Andre, I. A vast repertoire of Dscam binding specificities arises from modular interactions of variable Ig domains. Cell. 130, 1134-1134 (2007).
- Martin, S., Sollner, C., Charoensawan, V. Construction of a Large Extracellular Protein Interaction Network and Its Resolution by Spatiotemporal Expression Profiling. Mol. Cell. Proteomics. 9, 2654-2654 (2010).
- Crosnier, C., Bustamante, L. Y., Bartholdson, S. J., Bei, A. K., Theron, M., Uchikawa, M., Mboup, S., Ndir, O., Kwiatkowski, D. P., Duraisingh, M. T., Rayner, J. C., Wright, G. J. Basigin is a receptor essential for erythrocyte invasion by Plasmodium falciparum. Nature. 480, 534-537 (2011).
- Sollner, C., Wright, G. J. A cell surface interaction network of neural leucine-rich repeat receptors. Genome biology. 10, R99-R99 (2009).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten