Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Analyse von Proteindynamik mit Wasserstoffwechsel Massenspektrometrie
In diesem Artikel
Zusammenfassung
Protein-Konformation und Dynamik sind der Schlüssel zum Verständnis der Beziehung zwischen Struktur und Funktion. Wasserstoffaustausch gekoppelt mit hochauflösender Massenspektrometrie ist eine vielseitige Methode, um die Konformationsänderungen Dynamik von Proteinen als auch studieren Charakterisierung von Protein-Ligand und Protein-Protein-Wechselwirkungen, einschließlich Kontaktschnittstellen und allosterische Effekte.
Zusammenfassung
Alle zellulären Prozesse abhängig von der Funktionalität der Proteine. Obwohl die Funktionalität eines gegebenen Proteins ist die direkte Folge der einzigartigen Aminosäuresequenz wird nur durch die Faltung der Polypeptidkette in einer einzigen definierten dreidimensionalen Anordnung oder häufiger in einem Ensemble von Konformationen ineinander Umwandeln realisiert. Die Untersuchung der Verbindung zwischen Protein-Konformation und seine Funktion ist für ein komplettes Verständnis davon, wie Proteine in der Lage sind, ihre große Vielfalt von Aufgaben zu erfüllen daher unerlässlich. Eine Möglichkeit, ein Protein Konformationsänderungen erfährt, während voran durch seine Funktionszyklus studieren Wasserstoff-1 H / 2 H-Austausch in Kombination mit hochauflösender Massenspektrometrie (HX-MS). HX-MS ist eine vielseitige und robuste Methode, die eine neue Dimension der Strukturinformationen, zB durch Kristallographie gewonnen erstellt. Es wird verwendet, um Protein zu studieren und Ausklappen Bindung von kleinen molkül Liganden, Protein-Protein-Wechselwirkungen, Konformationsänderungen verbunden Enzymkatalyse und Allosterie. Zusätzlich wird HX-MS häufig verwendet, wenn die Menge an Protein ist sehr begrenzt oder Kristallisation des Proteins nicht möglich ist. Hier stellen wir ein allgemeines Protokoll für die Untersuchung der Proteindynamik mit HX-MS und zu beschreiben als ein Beispiel, wie man die Interaktionsschnittstelle von zwei Proteinen in einem Komplex zu offenbaren.
Einleitung
Die Anzahl von Kristallstrukturen von Proteinen und Proteinkomplexen schnell erhöht in den letzten Jahren. Sie präsentieren unschätzbare Schnappschüsse von der strukturellen Organisation dieser Proteine und eine Grundlage für die Struktur-Funktions-Analyse. Jedoch die Dynamik von Proteinen und Konformationsänderungen, die für ihre Funktionen sind, werden selten durch Röntgenkristallographie ergab. Cryo-Elektronenmikroskopie, auf der anderen Seite, ist in der Lage, Protein-und Protein-Komplexe in verschiedenen Konformationen zu erfassen, aber im Allgemeinen können Konformationsänderungen nicht lösen auf Sekundärstrukturebene ein. Konformationsdynamik von Proteinen in Lösung in atomarer Informationen sind nur für NMR gelöst werden, aber dieses Verfahren ist immer noch relativ kleine Proteine von Größen (in der Regel ≤ 30 kDa) beschränkt und erfordert hohe Konzentrationen von Proteinen (≥ 100 &mgr; M), die Experimente mit behindert Oligomerisierung oder Aggregation neigen Proteine 2. Eine Methode, dieder Lage ist, zwischen hochauflösende Röntgen-Kristallographie und Kryo-Elektronenmikroskopie und die Brücke nicht durch Größe oder Protein-Konzentration begrenzt ist Amid-Wasserstoff-1 H / 2 H-Austausch (HX) in Kombination mit der Massenspektrometrie (MS). In den letzten Jahren hat dieses Verfahren einen wertvollen analytisches Werkzeug für die Analyse von Proteindynamik, Proteinfaltung, der Proteinstabilität und Konformationsänderungen 3-5 entwickelt. Die molekulare Grundlage dieses Verfahrens ist die Labilität Rückgrat Amidwasserstoffe in Proteinen, die durch Deuteriumatome austauscht, wenn das Protein in D 2 O-Lösung gegeben. Die anschließende Erhöhung der Proteinmasse im Laufe der Zeit wird mit hochauflösenden MS gemessen.
Kurz unstrukturierten Peptiden HX hängt nur von der Temperatur, Katalysatorkonzentration (OH -, H 3 O +, dh pH-Wert, siehe Abbildung 3) und Aminosäure-Seitenketten der benachbarten Resten durch induktive, Katzekatalytische und sterische Effekte. Diese Auswirkungen auf die intrinsische chemischen Austauschrate k ch wurden elegant von Bai et al. 6 quantifiziert und ein Programm zur Verfügung (mit freundlicher Genehmigung Z. Zhang), die k ch für jede Aminosäure in einem Polypeptid abhängig von pH-Wert und Temperatur berechnet. Bei neutralem pH-Wert und Umgebungstemperaturen ch k in der Größenordnung von 10 1 bis 10 3 sec -1. Ionen in das Innere einer eng gefalteten Protein - in gefalteter Proteine HX 2-9 Größenordnungen langsamer hauptsächlich auf Wasserstoffbrückenbindung in der Sekundärstruktur und aufgrund eingeschränkter Zugangs hydratisierter OH geringem Ausmaß sein. HX in nativen Proteinen impliziert daher teilweise oder globale Entfaltung, chemischen Austausch und die Rückfaltung in den nativen Zustand nach Gleichung (1) und die beobachteten Wechselkurse k obs abhängig von der Öffnungsrate k op, die Schlussrate k cl und der intrinsischen chemischen Austausch rate ch k gemäß Gleichung (2).
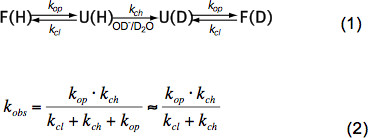
Unter nativen Zustand Bedingungen k op ist viel kleiner als k ch und kann im Nenner vernachlässigt werden. Es gibt zwei extreme Wechsel Regime genannt EX1 und EX2. Wenn der k Cl ist viel kleiner als k CH (EX1) die beobachtete Geschwindigkeit praktisch gleich der Öffnungsrate und HX unmittelbare Beobachtung der Entfaltung eines Strukturelements. Ein solcher Austausch Regime, in dem alle Amidprotonen Austausch auf einmal beim Öffnen des Strukturelements ist leicht beobachtbaren MS durch eine bimodale Verteilung der Isotopenpeaks 7. Wenn k Cl ist viel größer als k CH (EX2) die beobachtete Rate ist proportional zu k ch wobei die Proportionalitätskonstante gleich dem Faltungs-Entfaltungs-Gleichgewichte ist die Konstante K u = K op K / cl. Unter diesen Bedingungen sind viele Öffnungs-und Schließereignisse notwendig, bevor alle Amidprotonen gegen Deuteronen, was zu einem allmählichen Anstieg der Durchschnittsmasse, während die Isotopenverteilung etwa gleich bleibt. Die EX2 Regime ermöglicht die Bestimmung der freien Energie der Entfaltung &Dgr; G u und damit der Stabilität eines Strukturelements. Unter nativen Zustand Zustand der EX2-Regime ist am häufigsten. Erhöhung des pH-Wertes und Zugabe von chaotropen Mitteln können die Austauschmechanismus zu EX1 verschieben. Daher kann HX-MS verwendet werden, um thermodynamische erkunden sowie kinetischen Parameter der Proteinfaltung und Konformationsänderungen werden.
Wie oben erwähnt ist HX eigen pH-und temperaturabhängig und der Austausch Halbwertszeit von einer völlig löse ausgesetzt Protonen des Rückgrats Amidgruppe zwischen 5-400 ms bei einem physiologischen pH-Wert (pH 7,6) und 30 ° C, aber mindestens 10 min auf> 15 Stunden mit einem Mittelwert von> 2 h bei pH 2,9 und 0 °C (mit Ausnahme des Protons des ersten Gerüst-Amidbindung eines Polypeptids, das mit einer Halbwertszeit von ca. 1-2 min austauscht). Unter solchen langsamen Austausch Bedingungen ist es möglich, die Probe zu verdauen Proteasen (z. B. Pepsin), die aktiv sind, unter diesen Bedingungen mit verlieren sich alle in den eingeDeuteRonen enthaltenen Informationen. Seit der Einführung des Magen-Darm-Verdauung unter langsamem Austausch Bedingungen kann nicht nur die Gesamt HX Kinetik der vollständigen Proteine analysiert werden, aber HX kann auf bestimmte Regionen 8,9 lokalisiert werden. Ortsauflösung wird noch auf die Größe der Fragmente Magen erzeugt, die im allgemeinen zwischen 10-30 Reste beschränkt. Jedoch könnte überlappende Fragmente aufgrund der unspezifischen Art der Spaltung durch Pepsin erzeugt eine Erhöhung der räumlichen Auflösung führen. Darüber hinaus wurden mehrere andere Proteasen gefunden unter Abschrecken Bedingungen aktiv zu sein, aber viel weniger effizient als Pepsin 10. Weitere zunehse der räumlichen Auflösung können durch Fragmentierung von Peptiden in der Gasphase durch Methoden, die Deuterierung Muster wie Elektroneneinfang-Dissoziation (ECD), Elektronentransfer-Dissoziation (ETD) und Infrarot-Multiphoton-Dissoziation (IRMPD) 11-13 erhaltenen erreichen. Diese Methoden verhindern den Verlust an räumlicher Auflösung durch intramolekulare Protonenwanderung ("Scrambling"), die durch stoßinduzierte Dissoziation (CID) zu beobachten ist die am häufigsten verwendeten Fragmentierungstechnik. Allerdings erfordern diese Verfahren die Optimierung für jedes einzelne Peptid und ist damit immer noch ziemlich schwierig.
HX-MS wurde verwendet, um Protein-Liganden und Protein-Protein-Wechselwirkungen, einschließlich virale Kapsid-Zusammensetzung 14-17 analysieren. Protein-Entfaltung und Rückfaltung sowie temperaturinduzierte Konformationsänderung untersucht 7,18,19. Phosphorylierung und einzigen Aminosäure mutationsbedingte Konformationsänderungen 16,20 und nucleotide-induzierten Veränderungen wurden analysiert 21,22. Daher scheint diese Methode ideal geeignet zur Montage und Dynamik von molekularen Maschinen zu analysieren. Ein attraktiver Kandidat, dessen Mechanismus ist von großem allgemeinem Interesse ist das Hsp90-Chaperon-Komplex.
Protokoll
1. Herstellung von Puffern und Protein-Proben
- Bereiten H 2 O-Puffer. Verwendung Hsp90 Standardpuffer (40 mM HEPES / KOH, pH 7,5, 50 mM KCl, 5 mM MgCl 2, 10% Glycerin) und H 2 O-Puffer.
Hinweis: Wenn die Probe vor der Analyse dialysiert, verwenden Sie den Dialysepuffer als H 2 O-Puffer. Es ist wesentlich, daß die D 2 O-Puffer unterscheidet sich von der H 2 O-Puffer nur in der Wasserstoffisotop. Flüchtige Puffer wie NH 4 CO 3 oder NH 4-Acetat-oder Pufferkomponenten sind nicht geeignet! - Bereiten D 2 O-Puffer durch Gefriertrocknung von Hsp90 Standardpuffer mit Hilfe eines Vakuum-Konzentrator. Nach vollständiger Verdampfung von H 2 O hinzufügen reines D 2 O an das Rohr, um die anfängliche Volumen (zB 1 ml mit 15% Glycerin Puffer erfordern die Zugabe von 850 ul D 2 O) zu erreichen. Wiederholen Sie die vollständige Verdampfung des Puffer abgeblasen, und der Puffer / Salz-comKomponenten in D 2 O 2x.
- Vorbereitung Quench-Puffer (0,4 M KH 2 PO 4 / H 3 PO 4 pH 2,2).
Hinweis: Um die Effizienz des Magen-Darm-Digest von sehr stabilen Proteinen, 4 M Guanidin-Hydrochlorid und 0,5 M Tris (2-carboxyethyl) phosphin (TCEP-HCl) zugegeben werden, zu verbessern. - Bereiten Sie 100% Kontrolle Probe (6 M Guanidin-Hydrochlorid, D 2 O). Hinzufügen Guanidinhydrochlorid zu einem Hsp90-Aliquot auf eine Endkonzentration von 6 M zu erreichen vollständig verdampfen H 2 O aus der Probe und fügen D 2 O, um das Rohr um die anfängliche Volumen (z. B. 100 &mgr; l Probe mit 10% Glycerin erreichen die Zugabe 90 &mgr; l D 2 O). Wiederholen Sie die vollständige Verdampfung des Puffer abgeblasen, und der Puffer / Salzkomponenten in D 2 O.
Hinweis: 20-100 pmol der Probe für jede Injektion erforderlich. Bereiten Sie genügend Probe, um eine 100%-Kontrolle für jeden Tag des HX-MS-Experimente zu haben. - Bereiten 50 pmol Hsp90 in 5 ul Hsp90 Standardpuffer.
Hinweis: Ein Betrag in Höhe von 20-100 pmol Probe wird für jeden Punkt der Rohdaten erforderlich. Das Volumen der Probe in der Reaktion ist 1-5 &mgr; ideal. Stellen Sie die Konzentration, um diese Anforderungen zu passen. Jeder Puffer kann, solange es keine Reinigungsmittel oder flüchtige Bestandteile enthalten, verwendet werden.
2. Vorbereitung von immobilisiertem Pepsin auf Aldehyd-Aktivperlen
- Man löst 80 mg frischem Pepsin in 2 ml 50 mM Natriumcitrat (pH 5).
- Lösen Sie 20 mg Natriumcyanoborhydrid, mit Vorsicht hand (sehr giftig!) In 1 ml 2 M Na 2 SO 4 und in den Pepsin-Lösung.
- Inkubieren Gemisch 10 min bei Raumtemperatur unter leichtem Rühren (z. B. Kopfschüttler).
- Hinzufügen von 600 mg Kügelchen mit immobilisierten Aldehydgruppen zu der Mischung und Inkubation für 5-10 min bei Raumtemperatur.
- Hinzufügen von 2,2 ml 2 M Na 2 SO4 (pH 5) in 100 &mgr; l Aliquots alle 3 min über eine Stunde langsam Salz aus dem Pepsin. Vorsichtig mischen die Probe zwischen den Zugaben in einem Überkopfschüttler bei Raumtemperatur.
- Die Pepsin Perlen Inkubation bei 4 ° C für 14-16 h / über Nacht in einem Überkopfschüttler.
- Die Reaktion wird durch Zugabe von 1 ml 1 M Ethanolamin und Inkubation bei Raumtemperatur für 2 Stunden.
- Spin down die Perlen in einem 50 ml Falcon-Röhrchen bei 500 Upm, den Überstand verwerfen und resuspendieren die Perlen in 0,1% Ameisensäure. Wiederholen Sie diesen Schritt 2x. Nach dem letzten Zentrifugationsschritt, Überstand verwerfen und schätzen Volumen von Perlen. In eine gleichwertige Menge von 0,1% Ameisensäure und speichern bei 4 ° C
3. Herstellung von Säulen für Amid Wasserstoff-Austausch
- Verwenden Vorsäule mit einem Innendurchmesser von 1 mm zum Einfangen und Spalten mit 2 mm für Pepsin Spalten.
- Lösen Sie eine Seite der Schutzsäule und den Filter zu entfernen. Schrauben Sie die Verpackung funnel auf das offene Ende der Säule. Verwenden eine 1/16 Zoll-Adapter und Schlauch eine leere Spritze (5 ml) zu dem unteren Auslass der Säule zu befestigen. Stellen Sie sicher, dass es gasdicht auf den Guard-Säule befestigen.
- Geben Sie einige Tropfen von Gülle Perlenmaterial auf der Oberseite des Trichters. Ziehen Sie den Kolben der Spritze, um den Schlamm durch den Trichter in die Guard-Säule saugen. Bewerben mehr Gülle Perlenmaterial auf den Trichter und das Verfahren fortsetzen, bis die Schutzsäule ist komplett mit Perlenmaterial gefüllt. Nehmen Sie den Trichter und legen Filter und Filterring auf das offene Ende. Schrauben Sie die Spalte Kappe auf den Guard-Säule und entfernen Sie die Spritze von der anderen Seite. Schließen Sie beide Enden der Schutzsäulen mit Steckern, um das Trocknen von Säulenmaterial zu vermeiden.
4. Einrichten des Systems für die Wasserstoffwechsel-Massenspektrometrie (HX-MS)
- Man verbindet die Falle Spalte mit dem HPLC-System (Fig. 1). Äquilibrieren der Säule durch Einstellen der Strömungsrate desPumpe A auf 0,4 ml / min mit 0,1% iger Ameisensäure als Lösungsmittel. Sie das Pepsin Spalte noch die analytische Säule noch nicht an.
- Kalibrieren des Massenspektrometers und verbinden den Ausgang der HPLC mit der Quelle des Massenspektrometers.
5. Bestimmung der Dynamic Range von Exchange
- Bereiten hochreines Lösungsmittel A (0,1% Ameisensäure in Wasser) und hochreines Lösungsmittel B (0,1% Ameisensäure in Acetonitril); fertig gemischte Lösungsmittel sind im Handel erhältlich. Spülen Sie die HPLC-Pumpen. Richten Sie die Chromatographie und Massenspektrometrie-Methoden in der Steuersoftware, indem Sie ein Programm mit einem Stufengradienten nach kurzer Entsalzungsschritt. Für voller Länge verwenden Hsp90 eine 1-2 min Entsalzungsschritt, gefolgt von Einschalten der 6-Port-Ventil von entsalzen / LOAD zur Elution und einen Schritt Gradienten von 90% Lösungsmittel A/10% Lösungsmittel B zu 5% Lösungsmittel A/95% Lösungsmittel B. Vor der Injektion gesetzt das Einspritzventil zu laden und den 6-Wege-Ventil zum / Ladeposition zu entsalzen.
Anmerkung: Verwenden Sie kein Pepsin oder analytische Säule während dieses Experiments. - Bereiten 100-200 pmol von Hsp90 in 1-10 ul H 2 O-Puffer und Inkubation für 10 min bei 30 ° C Hinzufügen Temperatur eingestellt D 2 O-Puffer, um das Probenvolumen bis zu 100 &mgr; l zu bringen und Inkubieren für eine genau definierte Zeitspanne (z. B. 10 Sekunden, 100 Sekunden, 1000 Sekunden). 100 l Puffer Quench zugeben und durch Auf-und Abpipettieren. Spritzen die 200 ul Probe in die Einspritzöffnung des Einspritzventils mit einer Hamilton-Spritze. Starten Sie das Programm-Chromatographie und schalten Sie das Einspritzventil in INJECT-Position. Nach 2 min schalten Sie die 6-Port-Ventil von entsalzen / LADEN zu eluieren. Wiederholen Sie dies für mindestens drei Zeitpunkten.
- Wiederholen Sie Schritt 5.2 sondern fügen 2-3x Überschuss STI1 zum Hsp90-Probe vor der Inkubation für 10 min bei 30 ° C.
- Bestimmen Sie die in voller Länge Proteinmassen durch Entfaltung von Spektren in der Massenspektrometrie-Software. Berechnen Sie die Anzahl der Incorporated Deuteronen durch Vergleichen des Molekulargewichts von Hsp90 in voller Länge mit der beobachteten Masse nach jedem Durchlauf (z. B. 10 Sekunden, 100 Sekunden, 1000 Sekunden).
- Zeichnen Sie die eingeDeuteRonen für Hsp90 in Abwesenheit und Gegenwart von STI1 (y-Achse) gegen die Zeit (x-Achse). Bestimmen den Zeitpunkt, in dem dynamischen Bereich, wo die Differenz zwischen den beiden Kurven ist maximal. Verwenden Sie diesen Wert für die Inkubationszeit in D 2 O bei der Identifizierung Hsp90-STI1 Schnittstelle und Dynamik auf Peptidebene.
6. Bestimmung der Magen Peptiden mithilfe der MS / MS-Spektren
- Verbinden des Pepsin-Säule und analytische Säule des Systems.
- Stellen Sie die Parameter für die Chromatographie und Massenspektrometrie in der Steuerungssoftware, indem Sie Verlaufstyp und Massenspektrometrie-Methode. Wählen Sie ein langes Gefälle (zB mehr als 90 min), um eine gute chromatographische Auflösung zu gewährleisten. Aktivieren MS / MS-Spektren zu dem Massenspektrometer.
Hinweis: Gute Auflösungtion auf der HPLC und hohe Massengenauigkeit haben die höchste Priorität in diesem Schritt. - Bereiten 100-200 pmol von Hsp90 in 100 ul H 2 O-Puffer. 100 l Puffer Quench zugeben und durch Auf-und Abpipettieren. Spritzen die 200 ul Probe in die Einspritzöffnung des Einspritzventils mit einer Hamilton-Spritze. Starten Sie das Programm-Chromatographie und schalten Sie das Einspritzventil in INJECT-Position. Nach 2 min schalten Sie die 6-Port-Ventil von entsalzen / LADEN Position eluieren.
Hinweis: Wenn mehr als ein Protein in HX-MS (dh Protein-Protein-Interaktionsschnittstellen) analysiert werden die Peptide für jedes Protein haben individuell bestimmt werden. - Identifizieren Magen Peptiden von Hsp90 durch Durchsuchen einer Datenbank (dh Maskottchen) für die resultierenden Peptide.
Hinweis: Es ist möglich, eine benutzerdefinierte Datenbank verwenden, da es das Ziel ist, so viele Magen-Darm-Peptide wie möglich zu bestimmen, und die Analyse wird mit gereinigtem Protein getan. Probenreinheit wird have berücksichtigt werden. - Wiederholen diesen Schritt ohne MS / MS und der Gradient für die eigentliche HX Experiment verwendet wird. Für Hsp90 und STI1 verwenden eine 10 min-Gradienten von 90% Lösungsmittel A/10% Lösungsmittel B auf 45% Lösungsmittel A/55% Lösungsmittel B
Hinweis: Farbverläufe sind in der Regel zwischen 5-15 min und stark abhängig von der Komplexität und der Probe HX Systemspezifikationen. - Bestimmen Sie die Retentionszeiten der Magen-Darm-Peptide in 6,4 im Gradienten in 6,5 verwendet, identifiziert und erstellt eine Liste mit 1.) Peptid-Sequenz 2). Peptid Ladezustand und 3). Retentionszeit. Dies wird verwendet, um jedes Peptid nach HX-Experimente zu identifizieren.
Hinweis: Beachten Sie, dass Peptide mit kleinen m / z Unterschiede, gleiche Ladezustand und identische Retentionszeit kann eine Quelle der Zweideutigkeit.
7. Identifizierung von Protein-Protein-Interaktionsschnitt
- Stellen Sie den Gradienten und Massenspektrometrie-Methode in der Steuer SoftwarE. Verwenden Sie eine 10 min linearen Gradienten von 90% Lösungsmittel A/10% Lösungsmittel B auf 45% Lösungsmittel A/55% Lösungsmittel B Legen Sie eine Massenspektrometrie-Methode, die für die Erkennung von Massen zwischen 300-1.500 m / z optimiert ist, obwohl die meisten Die Peptide werden unter 1000 m / z sein. Stellen Sie das Einspritzventil in LOAD-Position, der 6-Wege-Ventil in LADE / Entsalzung Position. Die Durchflussrate der Ladepumpe auf 0,4 ml / min.
Hinweis: Länge und Art des Gradienten sind Beispielabhängig und können optimiert werden. Längere Steigungen verbessern die Auflösung der Chromatographie, sondern verringern die Aufnahme von Deuteronen in Proteine durch Back-Austausch. Die gewählten Methoden müssen die gleichen sein für alle Experimente verglichen werden. - Für die nicht ausgetauschten Referenz vorbereiten 20-100 pmol von Hsp90 in 100 ul H 2 O-Puffer. 100 l eiskaltem Quenchpuffer, Pipette und zweimal nach unten und injizieren Probe in das Einspritzventil der HPLC. Sofort die Chromatographie-Programm und starten swjucken das Einspritzventil in INJECT-Position. Nach 2 min schalten Sie die 6-Port-Ventil von entsalzen / LADEN Position eluieren. Dies für Hsp90 und STI1 einzeln und für die Mischung von Proteinen.
- Vorbereitung 20-100 pmol von Hsp90 in einem Volumen von 1-5 ul. In Temperatur angepasst D 2 O-Puffer, das Probenvolumen bis zu 100 ul zu bringen und genau Inkubation für einen definierten Zeitraum (zB 30 sec; für Konformationsdynamik siehe Hinweis). 100 l eiskaltem Quenchpuffer, Pipette auf und ab und zweimal schnell spritzen die 200 ul in die Einspritzventil der HPLC. Sofort die Chromatographie-Programm zu starten und schalten Sie das Einspritzventil in INJECT-Position. Nach 2 min schalten Sie die 6-Port-Ventil von entsalzen / LADEN Position eluieren. Tun Sie dies für jedes Protein einzeln, um die Deuteronen Einbau in jedes Peptid in Abwesenheit des interagierenden Proteins zu bestimmen.
Hinweis: Bei der Untersuchung der Dynamik der KonMational Änderungen, wiederholen Sie das Experiment mit unterschiedlichen Inkubationszeiten von Hsp90 in D 2 O-Puffer. Versuchen Sie, eine große Zeitskala durch die Wahl Inkubationszeiten logarithmisch (zB 10 Sek., 30 Sek., 100 Sek., 300 Sek., 1.000 sec, usw.). D 2 O-Puffer und Probenpuffer exakt identisch mit Ausnahme des Wasserstoffisotops sein. - Bereiten Sie eine gleiche Menge von 100% Kontrollprobe (20-100 pmol) und fügen D 2 O-Puffer, das Probenvolumen bis zu 100 ul zu bringen. 100 l eiskaltem Quenchpuffer, Pipette auf und ab und zweimal schnell spritzen die 200 ul in die Einspritzventil der HPLC. Sofort die Chromatographie-Programm zu starten und schalten Sie das Einspritzventil in INJECT-Position. Nach 2 min schalten Sie die 6-Port-Ventil von entsalzen / LADEN Position eluieren.
- Um die Interaktionsfläche bestimmen mischen Hsp90 mit einer mindestens 2-fachen Überschuß an STI1 das Gleichgewicht auf dem gebundenen Zustand zu wechseln (siehe Hinweis) und Inkubationauf die gewünschte Temperatur, bis die Komplexbildung im Gleichgewicht. Hinzufügen Temperatur eingestellt D 2 O-Puffer, um das Probenvolumen bis zu 100 &mgr; l zu bringen und Inkubieren für eine genau definierte Zeitspanne (z. B. 30 Sekunden). 100 l eiskaltem Quenchpuffer, Pipette auf und ab und zweimal schnell spritzen in die Einspritzventil der HPLC. Nach 2 min schalten Sie die 6-Port-Ventil von entsalzen / LADEN Position eluieren. Wiederholen Sie diesen Versuch mit 20-100 pmol STI1 und überschüssige von Hsp90.
Hinweis: Die absoluten Konzentrationen hängen von der Dissoziation Gleichgewichtskonstante der Wechselwirkung. Idealerweise nach der Verdünnung in D 2 O die Konzentration des Proteins im Überschuß zugegeben mindestens 10x K D (entsprechend> 85% der unteren konzentrierten Protein gebunden) sein. - Analysieren Sie die erfassten Daten mit einer geeigneten Software und verwenden Sie die Retentionszeiten in Schritt 6.5 festgelegt, jede Peptid in der Analyse zu finden. Berechnungente der Schwerpunkt des Isotopenverteilung für die nicht ausgetauschten Protein (Schritt 7.2) und für die Experimente HX (Schritt 7.3).
Hinweis: Obwohl die Isotopenmuster der Peptide ausgetauscht sehen anders aus als ihre nicht ausgetauschten Gegenstücke, sowohl das Wissen über Ladezustand des Peptids und die hohe Massengenauigkeit des Massenspektrometers ermöglicht die einfache Bestimmung der Schwerpunkte. - Vergleichen Einbau von Deuteronen an Zielprotein allein und mit mehr als Bindungspartner. Dies kann automatisch mit kommerzieller Software oder manuell mit einem Tabellenkalkulationsprogramm durchgeführt werden. Die 100% Kontrolle Probenwerte bezeichnen den maximalen Wechsel für jedes Peptid und kann verwendet werden, um die Menge von D 2 O Etikett verloren während des Experiments durch Rückaustausch bestimmen.
Ergebnisse
Hsp90 ist ein Chaperon in Hefe und Mitglied der Hsp90-Chaperon-Familie. Indem wir durch eine komplexe ATPase-Zyklus hilft es spät Klapptritt vieler Protein Kunden. Effiziente Faltung erfordert Transfer von Clients von Hsp70 und Wechselwirkung des Co-Chaperon Sti1/Hop. STI1 direkt bindet an Hsp90 und erleichtert die Client-Bindung durch die Hemmung von Hsp90-ATPase-Aktivität. Interaktion von Hsp90 mit STI1 wurde vor kurzem mit HX-MS-23 untersucht. Hier stellen wir repräsentative Ergebnisse der zugrunde lieg...
Diskussion
Bindung eines Interaktionspartners zu einem Protein führt unweigerlich zu Veränderungen der Lösungsmittelzugänglichkeit an der Bindungsstelle. Darüber hinaus haben viele Proteine werden dynamisch Konformationsänderungen bei der Bindung, die anderen Regionen beeinflussen als die tatsächliche Bindung Schnittstelle. HX-MS ist eine robuste Methode, um diese Änderungen zu überwachen und ist sogar in der Lage, enthüllt Konformationsänderungen in Proteinen auf Zeitskalen, dass andere Verfahren nicht abdecken k...
Offenlegungen
Wir haben nichts zu offenbaren.
Danksagungen
Wir danken M. Boysen für Kommentare zum Manuskript. Dieses Projekt wurde von der Deutschen Forschungsgemeinschaft (: Cellnetworks EXC 81/1 SFB638 und MA 1278/4-1 MPM und Exzellenzcluster) finanziert. MPM ist Investigator des Exzellenzclusters: Cellnetworks.
Materialien
| Name | Company | Catalog Number | Comments |
| maXis QTOF | Bruker | ||
| nanoAcquity UPLC | Waters Corp. | ||
| Shimadzu 10AD-VP | Shimadzu | ||
| 6-port Valve EPC6W with microelectric actuator | Valco | #EPC6W | |
| Injection valve (manual) | Rheodyne | #7725 | |
| Poros AL20 media | Applied Biosystems | #1-6029-06 | |
| Poros R2 | Applied Biosystems | #1-1118-02 | |
| Pepsin | Sigma | #P6887 | use fresh pepsin |
| Microbore (1 mm) | IDEX | #C-128 | |
| Microbore (2 mm) | IDEX | #C-130B | |
| Acquity UPLC BEH C8 Column | Waters Corp. | #186002876 | |
| Thermomixer | Eppendorf | #5355000.011 | |
| Tubing (various diameters) | IDEX | ||
| Fittings | IDEX | #PK-110 with PK-100 |
Referenzen
- Saibil, H. R. Conformational changes studied by cryo-electron microscopy. Nat. Struct. Biol. 7 (9), 711-714 (2000).
- Mittermaier, A., Kay, L. E. New tools provide new insights in NMR studies of protein dynamics. Science. 312 (5771), 224-228 (2006).
- Hoofnagle, A. N., Resing, K. A., Ahn, N. G. Protein analysis by hydrogen exchange mass spectrometry. Annu. Rev. Biophys. Biomol. Struct. 32, 1-25 (2003).
- Wales, T. E., Engen, J. R. Hydrogen exchange mass spectrometry for the analysis of protein dynamics. Mass. Spectrom. Rev. 25 (1), 158-170 (2006).
- Konermann, L., Pan, J., Liu, Y. -. H. Hydrogen exchange mass spectrometry for studying protein structure and dynamics. Chem Soc. Rev. 40 (3), 1224-1210 (2011).
- Bai, Y., Milne, J. S., Mayne, L., Englander, S. W. Primary structure effects on peptide group hydrogen exchange. Proteins. 17 (1), 75-86 (1993).
- Rist, W., Jørgensen, T. J. D., Roepstorff, P., Bukau, B., Mayer, M. P. Mapping temperature-induced conformational changes in the Escherichia coli heat shock transcription factor sigma 32 by amide hydrogen exchange. The Journal of biological chemistry. 278 (51), 51415-51421 (1074).
- Englander, J. J., Rogero, J. R., Englander, S. W. Protein hydrogen exchange studied by the fragment separation method. Anal Biochem. 147 (1), 234-244 (1985).
- Zhang, Z., Smith, D. L. Determination of amide hydrogen exchange by mass spectrometry: a new tool for protein structure elucidation. Protein Sci. 2 (4), 522-531 (1993).
- Cravello, L., Lascoux, D., Forest, E. Use of different proteases working in acidic conditions to improve sequence coverage and resolution in hydrogen/deuterium exchange of large proteins. Rapid Commun. Mass Spectrom. : RCM. 17 (21), 2387-2393 (2003).
- Rand, K. D., Zehl, M., Jensen, O. N., Jørgensen, T. J. D. Protein hydrogen exchange measured at single-residue resolution by electron transfer dissociation mass spectrometry. Anal chem. 81 (14), 5577-5584 (2009).
- Pan, J., Han, J., Borchers, C. H., Konermann, L. Electron capture dissociation of electrosprayed protein ions for spatially resolved hydrogen exchange measurements. J. Am. Chem. Soc. 130 (35), 11574-11575 (2008).
- Yamada, N., Suzuki, E. -. I., Hirayama, K. Identification of the interface of a large protein-protein complex using H/D exchange and Fourier transform ion cyclotron resonance mass spectrometry. Rapid Commun. Mass Spectrom. 16 (4), 293-299 (2002).
- Lee, T., Hoofnagle, A. N., et al. Docking motif interactions in MAP kinases revealed by hydrogen exchange mass spectrometry. Mol. Cell. 14 (1), 43-55 (2004).
- Hasan, A., Smith, D. L., Smith, J. B. Alpha-crystallin regions affected by adenosine 5'-triphosphate identified by hydrogen-deuterium exchange. Biochem. 41 (52), 15876-15882 (2002).
- Lanman, J., Lam, T. T., Emmett, M. R., Marshall, A. G., Sakalian, M., Prevelige, P. E. Key interactions in HIV-1 maturation identified by hydrogen-deuterium exchange. Nat. Struct. Mol. Biol. 11 (7), 676-677 (2004).
- Wang, L., Lane, L. C., Smith, D. L. Detecting structural changes in viral capsids by hydrogen exchange and mass spectrometry. Protein Sci. 10 (6), 1234-1243 (2001).
- Pan, H., Raza, A. S., Smith, D. L. Equilibrium and kinetic folding of rabbit muscle triosephosphate isomerase by hydrogen exchange mass spectrometry. J. Mol. Biol. 336 (5), 1251-1263 (2004).
- Mazon, H., Marcillat, O., Forest, E., Smith, D. L., Vial, C. Conformational dynamics of the GdmHCl-induced molten globule state of creatine kinase monitored by hydrogen exchange and mass spectrometry. Biochem. 43 (17), 5045-5054 (2004).
- Lanman, J., Lam, T. T., et al. Identification of novel interactions in HIV-1 capsid protein assembly by high-resolution mass spectrometry. J. Mol. Biol. 325 (4), 759-772 (2003).
- Rist, W., Graf, C., Bukau, B., Mayer, M. P. Amide hydrogen exchange reveals conformational changes in hsp70 chaperones important for allosteric regulation. J. Biol. chem. 281 (24), 16493-16501 (2006).
- Graf, C., Stankiewicz, M., Kramer, G., Mayer, M. P. Spatially and kinetically resolved changes in the conformational dynamics of the Hsp90 chaperone machine. EMBO J. 28 (5), 602-613 (2009).
- Lee, C. -. T., Graf, C., Mayer, F. J., Richter, S. M., Mayer, M. P. Dynamics of the regulation of Hsp90 by the co-chaperone Sti1. EMBO J. 31 (6), 1518-1528 (2012).
- Lou, X., Kirchner, M., et al. Deuteration distribution estimation with improved sequence coverage for HX/MS experiments. Bioinformatics. 26 (12), 1535-1541 (2010).
- Kreshuk, A., Stankiewicz, M., Lou, X., Kirchner, M., Hamprecht, F. A., Mayer, M. P. Automated detection and analysis of bimodal isotope peak distributions in H/D exchange mass spectrometry using HeXicon. Intl. J. mass spectrom. 302 (1-3), 125-131 (2011).
- Walters, B. T., Ricciuti, A., Mayne, L., Englander, S. W. Minimizing back exchange in the hydrogen exchange-mass spectrometry experiment. J. Am. Soc. Mass Spectrom. 23 (12), 2132-2139 (2012).
- Weis, D. D., Wales, T. E., Engen, J. R., Hotchko, M., Ten Eyck, L. F. Identification and characterization of EX1 kinetics in H/D exchange mass spectrometry by peak width analysis. J. Am. Soc. Mass Spectrom. 17 (11), 1498-1509 (2006).
- Fang, J., Rand, K. D., Beuning, P. J., Engen, J. R. False EX1 signatures caused by sample carryover during HX MS analyses. Intl. J. Mass Spectrom. 302 (1-3), 19-25 (2011).
- Deng, Y., Smith, D. L. Identification of unfolding domains in large proteins by their unfolding rates. Biochem. 37 (18), 6256-6262 (1998).
- Wales, T. E., Engen, J. R. Partial unfolding of diverse SH3 domains on a wide timescale. J. Mol. Biol. 357 (5), 1592-1604 (2006).
- Pan, Y., Piyadasa, H., O'Neil, J. D., Konermann, L. Conformational dynamics of a membrane transport protein probed by h/d exchange and covalent labeling: the glycerol facilitator. J. Mol. Biol. 416 (3), 400-413 (2012).
- Hebling, C. M., Morgan, C. R., Stafford, D. W., Jorgenson, J. W., Rand, K. D., Engen, J. R. Conformational analysis of membrane proteins in phospholipid bilayer nanodiscs by hydrogen exchange mass spectrometry. Anal chem. 82 (13), 5415-5419 (2010).
- Abzalimov, R. R., Kaplan, D. A., Easterling, M. L., Kaltashov, I. A. Protein conformations can be probed in top-down HDX MS experiments utilizing electron transfer dissociation of protein ions without hydrogen scrambling. J. Am. Soc. Mass Spectrom. 20 (8), 1514-1517 (2009).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten