JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
수소 환율 질량 분석을 사용하여 단백질의 역학 분석
요약
단백질의 입체 구조와 역학은 단백질 구조 및 기능 사이의 관계를 이해하기위한 열쇠이다. 고해상도 질량 분광법과 결합 수소 교환은 단백질의 형태 적 역학 연구뿐만 아니라 접촉 인터페이스 및 알로 스테 릭 효과를 포함하여 단백질 - 리간드와 단백질 - 단백질 상호 작용을 특성화하기 위해 다양한 방법이다.
초록
모든 세포 과정은 단백질의 기능에 따라 달라집니다. 주어진 단백질의 기능은 독특한 아미노산 서열의 직접적인 결과이지만, 그것은 오직 하나의 정의 된 입체 배열 또는 더 일반적 interconverting 배좌의 앙상블로로 폴리펩티드 사슬의 접힘에 의해 실현된다. 단백질의 입체 구조와 기능 사이의 연결을 조사하는 것은 단백질이 작업에 자신의 큰 다양성을 충족 할 수있는 방법을 완전히 이해 필수적이다. 기능적 사이클을 통해 진행하면서 겪는 단백질 형태 적 변화를 연구하는 하나의 가능성은 수소 한 고분해능 질량 분석법 (HX-MS)와 조합 H / H 2 - 교환이다. HX-MS는 예를 들어, 결정학에 의해 얻어진 구조 정보에 새로운 차원을 추가하는 다양하고 강력한 방법입니다. 그것은 작은 몰의 결합, 폴딩 및 전개 단백질을 연구하는 데 사용됩니다ecule 리간드, 단백질 - 단백질 상호 작용, 촉매 반응을 효소 연결된 구조적 변경 및 allostery. 단백질의 양이 매우 제한적이거나 단백질의 결정화가 가능하지 않을 경우 또, HX-MS가 종종 사용된다. 여기에서 우리는 HX-MS와 단백질 역학을 공부하는 일반적인 프로토콜을 제공하는 방법과 복잡한에 두 단백질의 상호 작용 인터페이스를 공개하는 예를 들어 설명합니다.
서문
단백질 및 단백질 복합체의 결정 구조의 수는 최근에 급격히 증가 하였다. 그들은 이러한 단백질의 구조적 조직의 귀중한 스냅 선물 구조 - 기능 분석을위한 기초를 제공한다. 그러나, 단백질의 기능에 필수적인 구조적 변화의 역학은 거의 X-선 결정학에 의해 공개되지 않습니다. 극저온 electronmicroscopy, 반면에, 상이한 입체 형태로 단백질 및 단백질 복합체를 캡처 할 수 있지만, 일반적으로 이차 구조 레벨 1에 형태 적 변화를 해결할 수 없다. 원자 세부 사항 용액에 단백질의 구조적 역학은 NMR에 의해 해결 될 수 있지만,이 방법은 여전히 상대적으로 작은 크기 (일반적으로 ≤ 30 kDa의)의 단백질로 제한하고 실험을 방해하는 단백질의 높은 농도 (≥ 100 μM)를 필요로한다 올리고머 또는 집계 발생하기 쉬운 단백질 2. 한 가지 방법이고해상도의 X-선 결정학 및 크라이 electronmicroscopy 어느 사이 해소 할 수는 단백질의 크기 나 농도에 의해 제한되지 않는다 아미드 수소 한 질량 분석계 (MS)와 결합 H / 2 H 교환기 (HX)입니다. 최근 몇 년 동안이 방법은 단백질 역학, 단백질 접힘, 단백질의 안정성과 구조적 변화 3-5의 분석을위한 유용한 분석 도구를 개발했습니다. 이 방법의 분자 기초는 단백질 D 2 O 용액에 배치 될 때, 중수소 원자와 교환 할 것이다 단백질 백본 아미드 수소의 불안정한 성질이다. 시간에 따른 단백질 질량의 후속 증가는 고해상도 MS로 측정된다.
짧은 비정형 펩티드 만 온도, 촉매 농도에 따라 HX (OH -도 3 참조, H 3 O + 즉 산도) 및 유도, 고양이에 의한 인접 잔기의 아미노산 측쇄alytic 및 입체 효과. 고유 화학 환율 K의 채널에 이러한 효과는 우아 바이 등. (6)에 의해 정량화하고 프로그램은 pH와 온도에 따라 폴리펩티드 내의 각 아미노산 K의 채널을 계산 (예의 Z. 장), 사용할 수 있습니다. 중성 pH 및 주위 온도에서 K의 채널 10 1 -10 3 초 -1의 순서입니다. 단단히 접힌 단백질의 내부에 이온 - 접힌 단백질 HX는 주로 이차 구조의 수소 결합과 OH 수화의 접근 제한으로 인해 작은 정도 느리다 2-9 명령 할 수있다. 천연 단백질 HX 따라서 일부 또는 글로벌 전개, 화학 교환 및 식에 따라 기본 상태로 폴딩 연루 (1)과 K OBS 여는 속도 K의 영업 이익, 마감 환율 케이의 CL과 고유 화학 교환에 따라 관찰 환율 라TE K CH 식에 따라 (2).
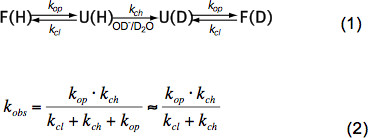
원시 상태의 조건에서 K의 영업 이익은 K의 채널보다 훨씬 작고 분모에서 무시 될 수있다. EX1과 EX2라는 두 개의 극단적 인 환율 제도가 있습니다. K 개의 CL은 K 개의 CH (EX1)보다 훨씬 작은 경우 관찰 속도는 개구율 실질적 같고 HX는 구조 부재의 전개를 즉시 관찰 할 수있다. 구조 요소의 열 때 한 번에 모든 아미드 양성자 교환, 동위 원소 피크 (7)의 바이 모달 분포에 의해 MS에서 쉽게 관찰 할 수 이러한 교환 정권. K의 CL은 K의 채널보다 훨씬 큰 경우 비례 상수는 접이식 전개 equilibriums에 해당된다 (EX2)는 관찰 된 비율은 K의 채널에 비례 상수 K U = K 영업 이익 / K CL. 동위 원소의 분포가 거의 동일하게 유지하면서 이러한 조건에서, 많은 개폐 이벤트는 평균 질량의 점진적인 증가로 이어지는, deuterons에 대한 모든 아미드 양성자 교환하기 전에 필요합니다. EX2 정권은 ΔG U 전개의 자유 에너지의 결정과 구조적 요소 때문에 안정성을 할 수 있습니다. 원시 상태의 조건에서 EX2 정권은 가장 일반적입니다. pH와 카오 트로픽 요원의 추가의 증가는 EX1에 교환 메커니즘을 전환 할 수 있습니다. 따라서, HX-MS는 열역학적을 탐구뿐만 아니라 단백질 접힘 및 구조적 변화의 운동 매개 변수를 사용할 수 있습니다.
HX 위에서 언급 한 바와 같이하는 것은 본질적으로 pH와 온도에 의존하고 백본 아미드 기의 완전히 용매 노출 된 양성자 교환 반감기>에 생리적 pH에서 5-400 밀리 초 (산도 7.6), 30 ° C,하지만 10 분 사이 pH가 2.9에서> 2 시간의 평균과 0 °와 15 시간(캘리포니아의 반감기. 1 ~ 2 분으로 교환하는 폴리 펩타이드의 첫 번째 백본 아미드 결합의 양자 제외) C. 느린 교환 조건 하에서 아웃이 혼입 deuterons에 포함 된 모든 정보를 잃고,이 조건 하에서 활성 단백질 분해 효소 (예 : 펩신)를 사용하여 샘플을 소화 할 수있다. 느린 교환 조건에서 소화 소화의 도입 이후, 전체 길이 단백질의 전체 HX 반응 속도뿐만 아니라 분석 할 수 있지만, HX는 특정 지역 8,9에 지역화 할 수 있습니다. 공간 해상도는 현재 10 ~ 30 잔기 사이에 일반에 발생하는 소화 파편의 크기로 제한됩니다. 그러나, 펩신으로 인해 분열의 비특이적 인 특성으로 생성 된 중복 조각은 공간 해상도의 증가로 이어질 수 있습니다. 또, 여러 다른 프로테아제는 급냉 조건 하에서 그러나 훨씬 덜 효율적인 열 펩신보다 활성 인 것으로 밝혀졌다. 또한 increa공간 해상도의 SE는 전자 캡쳐 해리 (ECD), 전자 전달 해리 (ETD) 및 적외선 다 광자 해리 (IRMPD) 11-13로서 중수소 패턴을 보존 방법에 의해 기체 상에있는 펩티드의 분열에 의해 도달 될 수있다. 이러한 기술에 의한 충돌 - 유도 해리 (CID)에 의해 관찰되는 분자 내 수소 이온 마이그레이션 ( "스크램블링"), 가장 일반적으로 사용되는 기술로 조각화 공간 해상도의 손실을 방지한다. 그러나 이러한 방법은 모든 개인 펩타이드에 대한 최적화를 필요로하고, 따라서 여전히 매우 도전이다.
HX-MS는 바이러스 캡시드 어셈블리 14-17 포함한 단백질 - 리간드와 단백질 - 단백질 상호 작용을 분석하는데 사용되어왔다. 단백질 전개 및 온도에 의한 구조적 변화가 7,18,19을 조사 하였다뿐만 아니라 접힘. 인산화 및 단일 아미노산 변이 관련 구조적는 16, 20 및 nucleot 변경IDE에 의한 변경 (21, 22)를 분석 하였다. 따라서,이 방법은 조립 및 분자 기계의 역학을 분석 할 이상적으로 적합한 것 같습니다. 그 메커니즘 위대한 장군 관심의 한 매력적인 후보,의 Hsp90 보호자 복잡합니다.
프로토콜
1. 버퍼의 준비 및 단백질 샘플
- H 2 O에게 버퍼를 준비합니다. 사용 Hsp90의 표준 버퍼 (40 mM의 HEPES / KOH, 산도 7.5, 50 밀리미터의 KCl, 5 mM의 MgCl2를, 10 % 글리세롤) H 2와 O 버퍼.
주 : 샘플을 분석하기 전에 투석 경우, H 2 O 버퍼로 투석 버퍼를 사용합니다. 그것은 D 2 O 버퍼는 수소의 동위 원소의 H 2 O 버퍼와 다른 것이 필수적이다. NH 4와 같은 휘발성 버퍼는 CO 3 또는 NH 4-아세테이트 또는 버퍼 구성 요소는 적합하지 않습니다! - 진공 농축기를 이용하여 Hsp90의 표준 버퍼의 동결 건조에 의해 D 2 O 버퍼를 준비합니다. H 2 O의 완료를 증발시킨 후 초기 전체 볼륨 (예 : 1 850 μL의 D 2 O를 추가해야 15 % 글리세롤 버퍼 ㎖)에 도달하기 위해 관에 순수 D 2 O를 추가합니다. 버퍼의 완전한 증발을 반복하고 버퍼 / 소금 컴 재용D 2 O 2 배에 ponents.
- (0.4 M KH 2 PO 4 / H 3 PO 4의 pH 2.2) 급냉 버퍼를 준비합니다.
참고 : 4 M 구아니딘 히드로 클로라이드, 0.5 M 트리스 (2 - 카르복시 에틸) 포스 핀 (TCEP - 염산)을 첨가 할 수있는 매우 안정한 단백질의 소화성 다이제스트의 효율을 개선한다. - 100 % 제어 샘플 (6 M 구아니딘 염산염, D 2 O)을 준비합니다. 완전 샘플로부터 H 2 O를 증발 및 10 % 글리세롤로 초기 전량 (예 100 μL 샘플에 도달하는 튜브에 D 2 O를 첨가에게 요구하는 추가 6 M.의 최종 농도에 도달하기의 Hsp90 분취 액에 염산 구아니딘을 추가 90 ㎕의 D 2 O의). 버퍼의 완전한 증발을 반복 D 2 O.에서 버퍼 / 염 성분을 재용
참고 : 샘플의 20 ~ 100 pmol의 각 주입이 필요합니다. HX-MS 실험의 매일을위한 100 % 제어 할 충분한 샘플을 준비합니다. - 5 ㎕의 Hsp90의 표준 버퍼에 50 pmol의 Hsp90의를 준비합니다.
참고 : 샘플의 20 ~ 100 pmol의의 양이 원시 데이터의 각 지점이 필요합니다. 반응 된 샘플의 체적은 이상적 1-5 μL이다. 이러한 요구 사항에 맞게 농도를 조절합니다. 상관 버퍼로는, 세제 또는 휘발성 성분을 포함하지 않는 한 사용될 수있다.
2. 알데히드에 고정 펩신의 준비 구슬을 활성화
- 2 ML 50 밀리미터 구연산 나트륨 (산도 5)에 80 mg의 신선한 펩신을 녹입니다.
- , 시아 노 수소화 붕소 나트륨 20mg을 녹여 1 ㎖ 2 M 나 2 SO 4의 치료 (매우 독성!)으로 처리하고 펩신 솔루션을 추가 할 수 있습니다.
- 부드럽게 (예 : 오버 헤드 통)를 교반하면서 실온에서 10 분 동안 혼합물을 품어.
- 혼합물에 고정 알데히드 그룹과 600 밀리그램의 구슬을 추가하고 실온에서 5 ~ 10 분 동안 품어.
- 2 M 나 2의 2.2 ML을 추가 SO100 ㎕의 분취 량 4 (산도 5) 원 시간에 걸쳐 매 3 분 천천히 펩신을 소금. 부드럽게 실온에서 오버 헤드 흔드는에 추가 사이의 샘플을 섞는다.
- 오버 헤드 통에 14 ~ 16 시간 / 밤새 4 ° C에서 펩신의 구슬을 품어.
- 실온에서 2 시간 동안 1 ㎖의 1 M 에탄올 아민 및 배양을 가하여 반응을 급냉.
- , 500 rpm에서 50 ML 팔콘 튜브에 비즈를 스핀 다운 뜨는을 취소하고 0.1 % 포름산에 구슬을 재현 탁. 이 단계의 2 배를 반복합니다. 마지막으로 원심 분리 단계 후, 상층 액과 비즈의 예상 볼륨을 폐기합니다. 0.1 % 포름산의 동일한 볼륨을 추가하고 4 ℃에서 저장
3. 아미드 수소 교환의 열 준비
- 열을 트래핑 1 ㎜의 내경과 펩신 열의 2mm 가드 컬럼을 사용합니다.
- 보호 컬럼의 한 쪽을 풀고 필터를 제거합니다. 단단히 FUNN 포장 나사열 열린 끝에 엘. 칼럼의 바닥 콘센트에 빈 주사기 (5 ㎖)를 연결하는 1 / 16 인치 어댑터와 튜브를 사용합니다. 보호 컬럼에 기밀을 해결해야합니다.
- 깔때기 위에 슬러리 비드 재료의 몇 방울을 적용합니다. 가드 컬럼에 깔때기를 통해 슬러리를 빨아 주사기의 플런저를 잡아 당겨. 깔때기에 더 슬러리 비드 재질을 적용하고, 보호 컬럼이 완전히 비드 재료로 채워질 때까지 절차를 계속합니다. 깔때기를 제거하고 오픈 엔드에 필터 및 필터 링을 배치합니다. 단단히 가드 칼럼에 열 스크류 캡과 다른 측면에서 주사기를 제거합니다. 열 재료의 건조를 방지하기 위해 플러그가 보호 컬럼의 양쪽 끝을 닫습니다.
4. 수소 환율 질량 분석 (HX-MS)을위한 시스템 설정
- HPLC 시스템과 트랩 열 (그림 1)에 연결합니다. 의 유량을 설정하여 컬럼을 평형화용매 0.1 % 포름산 0.4 ㎖ / 분으로 펌프. 아직 펩신 칼럼이나 분석 컬럼을 연결하지 마십시오.
- 질량 분석기를 보정 및 질량 분석기의 소스에 HPLC의 출구를 연결한다.
5. 환율의 동적 범위의 결정
- 초순수 용매 A (물 중 0.1 % 포름산) 및 초순수 용매 B (아세토 니트릴 중 0.1 % 포름산)을 제조; 준비 혼합 용매는 시판되고있다. HPLC 펌프를 제거. 짧은 탈염 단계 이후 단계의 그라데이션 프로그램을 선택하여 크로마토 그래피 및 제어 소프트웨어의 질량 분석 방법을 설정합니다. 전체 길이의 Hsp90은 5 % 용매 A/95 %의 용매에게 90 % 용매 A/10 %의 용매 B에서 용출 탈염 / LOAD에서 6 포트 밸브와 단계 구배를 전환 한 다음 1 ~ 2 분의 탈염 공정을 사용 B. 주입하기 전에 / 로딩 위치를 탈염 부하에 주입 밸브와 6 포트 밸브를 설정합니다.
주의이 실험 중 펩신이나 분석 컬럼을 사용하지 마십시오. - 1-10 μL의 H 2 O 버퍼의 Hsp90의 100 ~ 200 pmol의를 준비하고 30 ℃에서 10 분 동안 배양 100 μL까지 샘플의 볼륨을 일정하게하고 지정된 기간 (예 : 10 초, 100 초, 1000 초)에 대해 정확히 부화 온도 조절 D 2 O 버퍼를 추가합니다. 100 μL의 급냉 버퍼를 추가하고 아래로 피펫 팅에 의해 혼합. 해밀턴 주사기 분사 밸브의 분사 포트에 200 μL 샘플을 주입한다. 크로마토 그래피 프로그램을 시작하고 주입 위치로 주입 밸브를 전환합니다. 2 분 용출 탈염 / LOADING에서 6 포트 밸브를 전환 한 후에. 적어도 세 시간 점에 대해이 단계를 반복합니다.
- 5.2 단계를 반복하지만, 이전에 30 ℃에서 10 분 동안 배양에의 Hsp90 샘플 Sti1의 2 - 3 배를 초과하는 추가
- 질량 분석 소프트웨어 스펙트럼의 컨볼 루션하여 전체 길이의 단백질 질량을 결정합니다. incorporat의 수를 계산각 실행 (예를 들어, 10 초, 100 초, 1000 초) 후 관찰 된 질량 전체 길이의 Hsp90의 분자량을 비교하여 deuterons 에드.
- 부재와 시간 (x 축) 대 Sti1 (y 축)의 존재의 Hsp90에 대한 통합 deuterons 플롯. 두 곡선 사이의 차이가 최대 인 동적 범위의 시간 점을 확인합니다. 의 Hsp90-Sti1 인터페이스 및 펩티드 수준에서 역학을 식별 할 때 D 2 O의 배양 시간에 대해이 값을 사용합니다.
6. MS / MS 스펙트럼을 사용하여 소화 펩티드의 결정
- 시스템에 펩신 칼럼과 분석 컬럼을 연결합니다.
- 구배 분류 및 질량 분석 방법을 선택하여 제어 소프트웨어에서 크로마토 그래피 질량 분석을위한 파라미터를 설정. 좋은 크로마토 그래피 해상도를 보장하기 위해 긴 그라데이션 (예 : 90 분)을 선택합니다. 질량 분석기에 MS / MS 스펙트럼을 사용합니다.
참고 : 좋은 분해능을HPLC 높은 질량 정확성에 기이 단계에서 가장 높은 우선 순위를 갖습니다. - 100 μL의 H 2 O 버퍼의 Hsp90의 100 ~ 200 pmol의를 준비합니다. 100 μL의 급냉 버퍼를 추가하고 아래로 피펫 팅에 의해 혼합. 해밀턴 주사기 분사 밸브의 분사 포트에 200 μL 샘플을 주입한다. 크로마토 그래피 프로그램을 시작하고 주입 위치로 주입 밸브를 전환합니다. 2 분 위치를 용출 탈염 / LOADING에서 6 포트 밸브를 전환 한 후에.
참고 : 하나 이상의 단백질 HX-MS (즉, 단백질 - 단백질 상호 작용 인터페이스)에 분석되어야하는 경우, 각 단백질 펩티드는 개별적으로 결정되어야한다. - 결과 펩티드에 대한 데이터베이스 (즉, 마스코트)를 검색하여의 Hsp90의 소화성 펩티드를 확인합니다.
참고 : 목표는 가능한 한 많은 화성 펩타이드를 판별하고 분석은 정제 된 단백질로 수행되는 것처럼 그것은 지정 데이터베이스를 사용하는 것이 가능하다. 샘플 순도 것입니다 H고려되어야 번가. - MS / MS없이 실제 HX 실험에 사용되는 그라데이션이 단계를 반복합니다. 의 Hsp90와 Sti1 45 % 용매 A/55 %의 용매 B에 90 % 용매 A/10 %의 용매 B에서 10 분 구배를 사용하십시오
참고 : 그라디언트 ~ 15 분 사이에 높은 샘플의 복잡성과 HX 시스템 사양에 따라 일반적이다. - 6.5에 사용되는 그라데이션 6.4에서 확인 된 소화성 펩티드의 머무름 시간을 결정하고 1을 포함하는 목록을 만들 수 있습니다.) 펩타이드 서열 2.) 펩타이드의 충전 상태 및 3.) 유지 시간. 이 HX 실험 후 각각의 펩타이드를 식별하는 데 사용됩니다.
참고 : 작은 M / Z의 차이, 동일한 충전 상태와 동일한 머무름 시간이 펩티드는 모호성의 원천이 될 수 있음을 유의하십시오.
7. 단백질 - 단백질 상호 작용 인터페이스의 식별
- 제어 softwar를의 그라데이션과 질량 분석 방법을 설정전자. , 300-1,500 m / Z 사이에 대중의 검출에 최적화 된 질량 분석 방법을로드 45 % 용매 A/55 %의 용매 B에 90 % 용매 A/10 %의 용매 B에서 10 분 선형 그라데이션을 사용하지만 대부분의 펩티드 1,000 M / Z 아래에있을 것입니다. LOAD 위치, 로딩 / 탈염 위치에있는 6 포트 밸브로 주입 밸브를 설정합니다. 0.4 ㎖ / 분으로 적하 펌프의 유량을 설정한다.
참고 : 길이 및 그라데이션의 유형은 샘플 의존하고 최적화해야 할 수 있습니다. 긴 그라데이션 크로마토 그래피의 해상도를 개선하지만 백 교환에 의한 단백질로 deuterons의 결합을 감소시킨다. 선택 방법은 모든 실험이 비교 될 때까지 동일 할 필요가있다. - unexchanged 기준 100 μL의 H 2 O 버퍼의 Hsp90의 20 ~ 100 pmol의 준비를합니다. 두 번 아래로 100 ㎕의 얼음처럼 차가운 급냉 버퍼, 피펫 업을 추가하고 HPLC의 분사 밸브에 샘플을 주입. 즉시 크로마토 그래피 프로그램과 SW 시작분사 위치로 분사 밸브 가려움. 2 분 위치를 용출 탈염 / LOADING에서 6 포트 밸브를 전환 한 후에. 의 Hsp90와 Sti1 개별적으로 단백질의 혼합물에 대해이 작업을 수행합니다.
- 1 ~ 5 μL의 볼륨의 Hsp90의 20 ~ 100 pmol의를 준비합니다. (; 구조적 역학 주를 참조하십시오 예를 들면 30 초) 100 μL까지 샘플 볼륨을 가져오고 정의 된 시간에 정확히 부화 온도 조절 D 2 O 버퍼를 추가합니다. 배 아래 얼음처럼 차가운 급냉 버퍼, 피펫 위로 100 μl를 추가하고 신속하게 HPLC의 분사 밸브에 200 μl를 주입. 즉시 크로마토 그래피 프로그램을 시작하고 주입 위치로 주입 밸브를 전환합니다. 2 분 위치를 용출 탈염 / LOADING에서 6 포트 밸브를 전환 한 후에. 상호 작용하는 단백질의 부재에서 펩타이드로 DEUTERON의 설립을 결정하기 위해, 개별적으로 각각의 단백질에 대해이 작업을 수행합니다.
참고 : confor의 역학을 공부하는 경우변경 사항을 mational D 2 O 버퍼의 Hsp90의 다른 배양 시간과 실험을 반복한다. (예를 들어, 10 초, 30 초, 100 초, 300 초, 1000 초 등) 대수적으로 배양 시간을 선택하여 다양한 척도를 커버하려고합니다. D 2 O 버퍼와 샘플 버퍼는 수소의 동위 원소를 제외하고 정확히 동일해야합니다. - 100 % 제어 샘플 (20-100 pmol의)의 동일한 금액을 준비하고 100 μL까지 샘플 볼륨을 가지고 D 2 O 버퍼를 추가합니다. 배 아래 얼음처럼 차가운 급냉 버퍼, 피펫 위로 100 μl를 추가하고 신속하게 HPLC의 분사 밸브에 200 μl를 주입. 즉시 크로마토 그래피 프로그램을 시작하고 주입 위치로 주입 밸브를 전환합니다. 2 분 위치를 용출 탈염 / LOADING에서 6 포트 밸브를 전환 한 후에.
- 상호 작용의 표면을 확인하려면 (주 참조) 바인딩 된 상태로 평형을 이동하고 부화 Sti1의 최소 2 배 과량의 Hsp90 혼합원하는 온도에서 복잡한 형성 될 때까지 평형에 있습니다. 100 μL까지 샘플 볼륨을 가지고와 시간 (예 : 30 초)의 정의 된 기간에 대해 정확히 부화 온도 조절 D 2 O 버퍼를 추가합니다. 두 번 빠르게 HPLC의 분사 밸브에 주입 아래 얼음처럼 차가운 급냉 버퍼, 피펫 위로 100 μl를 추가합니다. 2 분 위치를 용출 탈염 / LOADING에서 6 포트 밸브를 전환 한 후에. 20 ~ 100 Sti1의 pmol의와의 Hsp90의 과잉이 실험을 반복합니다.
주 : 절대 농도는 상호 작용의 해리 평형 상수에 달려있다. 이상적으로, D 2 O로 희석 한 후 단백질의 농도 (하한 농축 단백질의> 85 %에 대응하는) 적어도 10X K D이어야 과량 첨가. - 적합한 소프트웨어로 수집 된 데이터를 분석하고 분석에 각 펩티드를 찾는 단계 6.5에서 결정된 유지 시간을 사용한다. 계산 등테 unexchanged 단백질 (단계 7.2) 및 HX 실험 (단계 7.3)의 동위 원소 분포의 중심.
참고 : 교환 펩티드의 동위 원소 패턴이 펩티드의 충전 상태에 대한 지식과 무게 중심을 쉽게 결정을 허용 질량 분석기의 높은 질량 정확성을 모두 자신의 unexchanged 대응이 다르게 보일 수 있지만. - 표적 단백질의 deuterons 단독으로 파트너를 바인딩 초과와의 결합을 비교합니다. 이것은 스프레드 시트 프로그램을 수동으로 상용 소프트웨어 또는 자동으로 수행 할 수 있습니다. 100 % 컨트롤 샘플 값은 각 펩티드에 대한 최대한의 교환을 나타낸다 및 O 라벨 인해 뒤 교환 실험 도중 분실 D (2)의 양을 결정하는데 사용될 수있다.
결과
HSP90은 효모의 Hsp90 보호자 가족의 구성원의 분자 보호자입니다. 복잡한 ATPase의주기를 통과하여 많은 단백질 고객의 늦은 접는 단계를 지원합니다. 효율적인 폴딩은 Hsp70의에서 클라이언트의 전송 및 공동 보호자 Sti1/Hop의 상호 작용을 필요로한다. Sti1 직접의 Hsp90에 바인딩과의 Hsp90의 ATPase의 활성을 억제하여 바인딩 클라이언트를 용이하게한다. Sti1와의 Hsp90의 상호 작용은 최근 HX-MS (23)를...
토론
단백질 상호 작용 파트너의 결합은 필연적으로 결합 부위에 용매 접근성의 변화가 발생합니다. 또한, 많은 단백질은 실제 바인딩 인터페이스가 아닌 다른 지역에 영향을 바인딩에 따라 동적 구조적인 변화를 겪는다. HX-MS는 이러한 변경 사항을 모니터링 할 수있는 강력한 방법이며 다른 방법으로 커버 할 수없는 척도에 단백질의 구조적 변화를 드러내는도 할 수있다.
성공적...
공개
우리는 공개 아무것도 없어.
감사의 말
우리는 원고에 대한 의견에 대한 M. Boysen 감사합니다. 이 프로젝트는 (: CellNetworks EXC 1분의 81 우수 SFB638 및 MA MPM에 1278/4-1 및 클러스터) 도이치 Forschungsgemeinschaft에 의해 투자되었다. MPM은 우수 클러스터의 수사관이다 : CellNetworks.
자료
| Name | Company | Catalog Number | Comments |
| maXis QTOF | Bruker | ||
| nanoAcquity UPLC | Waters Corp. | ||
| Shimadzu 10AD-VP | Shimadzu | ||
| 6-port Valve EPC6W with microelectric actuator | Valco | #EPC6W | |
| Injection valve (manual) | Rheodyne | #7725 | |
| Poros AL20 media | Applied Biosystems | #1-6029-06 | |
| Poros R2 | Applied Biosystems | #1-1118-02 | |
| Pepsin | Sigma | #P6887 | use fresh pepsin |
| Microbore (1 mm) | IDEX | #C-128 | |
| Microbore (2 mm) | IDEX | #C-130B | |
| Acquity UPLC BEH C8 Column | Waters Corp. | #186002876 | |
| Thermomixer | Eppendorf | #5355000.011 | |
| Tubing (various diameters) | IDEX | ||
| Fittings | IDEX | #PK-110 with PK-100 |
참고문헌
- Saibil, H. R. Conformational changes studied by cryo-electron microscopy. Nat. Struct. Biol. 7 (9), 711-714 (2000).
- Mittermaier, A., Kay, L. E. New tools provide new insights in NMR studies of protein dynamics. Science. 312 (5771), 224-228 (2006).
- Hoofnagle, A. N., Resing, K. A., Ahn, N. G. Protein analysis by hydrogen exchange mass spectrometry. Annu. Rev. Biophys. Biomol. Struct. 32, 1-25 (2003).
- Wales, T. E., Engen, J. R. Hydrogen exchange mass spectrometry for the analysis of protein dynamics. Mass. Spectrom. Rev. 25 (1), 158-170 (2006).
- Konermann, L., Pan, J., Liu, Y. -. H. Hydrogen exchange mass spectrometry for studying protein structure and dynamics. Chem Soc. Rev. 40 (3), 1224-1210 (2011).
- Bai, Y., Milne, J. S., Mayne, L., Englander, S. W. Primary structure effects on peptide group hydrogen exchange. Proteins. 17 (1), 75-86 (1993).
- Rist, W., Jørgensen, T. J. D., Roepstorff, P., Bukau, B., Mayer, M. P. Mapping temperature-induced conformational changes in the Escherichia coli heat shock transcription factor sigma 32 by amide hydrogen exchange. The Journal of biological chemistry. 278 (51), 51415-51421 (1074).
- Englander, J. J., Rogero, J. R., Englander, S. W. Protein hydrogen exchange studied by the fragment separation method. Anal Biochem. 147 (1), 234-244 (1985).
- Zhang, Z., Smith, D. L. Determination of amide hydrogen exchange by mass spectrometry: a new tool for protein structure elucidation. Protein Sci. 2 (4), 522-531 (1993).
- Cravello, L., Lascoux, D., Forest, E. Use of different proteases working in acidic conditions to improve sequence coverage and resolution in hydrogen/deuterium exchange of large proteins. Rapid Commun. Mass Spectrom. : RCM. 17 (21), 2387-2393 (2003).
- Rand, K. D., Zehl, M., Jensen, O. N., Jørgensen, T. J. D. Protein hydrogen exchange measured at single-residue resolution by electron transfer dissociation mass spectrometry. Anal chem. 81 (14), 5577-5584 (2009).
- Pan, J., Han, J., Borchers, C. H., Konermann, L. Electron capture dissociation of electrosprayed protein ions for spatially resolved hydrogen exchange measurements. J. Am. Chem. Soc. 130 (35), 11574-11575 (2008).
- Yamada, N., Suzuki, E. -. I., Hirayama, K. Identification of the interface of a large protein-protein complex using H/D exchange and Fourier transform ion cyclotron resonance mass spectrometry. Rapid Commun. Mass Spectrom. 16 (4), 293-299 (2002).
- Lee, T., Hoofnagle, A. N., et al. Docking motif interactions in MAP kinases revealed by hydrogen exchange mass spectrometry. Mol. Cell. 14 (1), 43-55 (2004).
- Hasan, A., Smith, D. L., Smith, J. B. Alpha-crystallin regions affected by adenosine 5'-triphosphate identified by hydrogen-deuterium exchange. Biochem. 41 (52), 15876-15882 (2002).
- Lanman, J., Lam, T. T., Emmett, M. R., Marshall, A. G., Sakalian, M., Prevelige, P. E. Key interactions in HIV-1 maturation identified by hydrogen-deuterium exchange. Nat. Struct. Mol. Biol. 11 (7), 676-677 (2004).
- Wang, L., Lane, L. C., Smith, D. L. Detecting structural changes in viral capsids by hydrogen exchange and mass spectrometry. Protein Sci. 10 (6), 1234-1243 (2001).
- Pan, H., Raza, A. S., Smith, D. L. Equilibrium and kinetic folding of rabbit muscle triosephosphate isomerase by hydrogen exchange mass spectrometry. J. Mol. Biol. 336 (5), 1251-1263 (2004).
- Mazon, H., Marcillat, O., Forest, E., Smith, D. L., Vial, C. Conformational dynamics of the GdmHCl-induced molten globule state of creatine kinase monitored by hydrogen exchange and mass spectrometry. Biochem. 43 (17), 5045-5054 (2004).
- Lanman, J., Lam, T. T., et al. Identification of novel interactions in HIV-1 capsid protein assembly by high-resolution mass spectrometry. J. Mol. Biol. 325 (4), 759-772 (2003).
- Rist, W., Graf, C., Bukau, B., Mayer, M. P. Amide hydrogen exchange reveals conformational changes in hsp70 chaperones important for allosteric regulation. J. Biol. chem. 281 (24), 16493-16501 (2006).
- Graf, C., Stankiewicz, M., Kramer, G., Mayer, M. P. Spatially and kinetically resolved changes in the conformational dynamics of the Hsp90 chaperone machine. EMBO J. 28 (5), 602-613 (2009).
- Lee, C. -. T., Graf, C., Mayer, F. J., Richter, S. M., Mayer, M. P. Dynamics of the regulation of Hsp90 by the co-chaperone Sti1. EMBO J. 31 (6), 1518-1528 (2012).
- Lou, X., Kirchner, M., et al. Deuteration distribution estimation with improved sequence coverage for HX/MS experiments. Bioinformatics. 26 (12), 1535-1541 (2010).
- Kreshuk, A., Stankiewicz, M., Lou, X., Kirchner, M., Hamprecht, F. A., Mayer, M. P. Automated detection and analysis of bimodal isotope peak distributions in H/D exchange mass spectrometry using HeXicon. Intl. J. mass spectrom. 302 (1-3), 125-131 (2011).
- Walters, B. T., Ricciuti, A., Mayne, L., Englander, S. W. Minimizing back exchange in the hydrogen exchange-mass spectrometry experiment. J. Am. Soc. Mass Spectrom. 23 (12), 2132-2139 (2012).
- Weis, D. D., Wales, T. E., Engen, J. R., Hotchko, M., Ten Eyck, L. F. Identification and characterization of EX1 kinetics in H/D exchange mass spectrometry by peak width analysis. J. Am. Soc. Mass Spectrom. 17 (11), 1498-1509 (2006).
- Fang, J., Rand, K. D., Beuning, P. J., Engen, J. R. False EX1 signatures caused by sample carryover during HX MS analyses. Intl. J. Mass Spectrom. 302 (1-3), 19-25 (2011).
- Deng, Y., Smith, D. L. Identification of unfolding domains in large proteins by their unfolding rates. Biochem. 37 (18), 6256-6262 (1998).
- Wales, T. E., Engen, J. R. Partial unfolding of diverse SH3 domains on a wide timescale. J. Mol. Biol. 357 (5), 1592-1604 (2006).
- Pan, Y., Piyadasa, H., O'Neil, J. D., Konermann, L. Conformational dynamics of a membrane transport protein probed by h/d exchange and covalent labeling: the glycerol facilitator. J. Mol. Biol. 416 (3), 400-413 (2012).
- Hebling, C. M., Morgan, C. R., Stafford, D. W., Jorgenson, J. W., Rand, K. D., Engen, J. R. Conformational analysis of membrane proteins in phospholipid bilayer nanodiscs by hydrogen exchange mass spectrometry. Anal chem. 82 (13), 5415-5419 (2010).
- Abzalimov, R. R., Kaplan, D. A., Easterling, M. L., Kaltashov, I. A. Protein conformations can be probed in top-down HDX MS experiments utilizing electron transfer dissociation of protein ions without hydrogen scrambling. J. Am. Soc. Mass Spectrom. 20 (8), 1514-1517 (2009).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유