Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Concentric Gel-System, um die biophysikalische Rolle der Matrix Mikromilieu auf 3D-Zellmigration Studieren
In diesem Artikel
Zusammenfassung
Die mechanischen Eigenschaften und die Mikrostruktur der extrazellulären Matrix stark beeinflussen 3D Migration von Zellen. In vitro-Verfahren, um die Raumzeit-Zellmigrationsverhalten in biophysikalisch variable Umgebungen sowohl Bevölkerung und einzelnen Zellebenen zu studieren, wird beschrieben.
Zusammenfassung
Die Fähigkeit von Zellen zur Migration entscheidend ist in einer Vielzahl von Zellfunktionen während des gesamten Lebens aus embryonalen Entwicklung und bei der Wundheilung zur Tumor- und Krebsmetastasierung. Trotz intensiver Forschung, die grundlegenden biochemischen und biophysikalischen Prinzipien der Zellwanderung immer noch nicht vollständig verstanden, insbesondere in ihre physiologisch relevanten dreidimensionalen (3D) Mikroumgebungen. Hier beschreiben wir ein in vitro Test zur quantitativen Untersuchung der 3D-Zellmigration Verhalten ermöglichen. Das Verfahren nutzt mechanosensing Fähigkeit der Zelle, und die Neigung, in vorher unbesetzten extrazellulären Matrix (ECM) zu migrieren. Wir nutzen die Invasion von hochinvasiven Brustkrebs-Zellen, MDA-MB-231, in Kollagengele als Modellsystem. Die Verbreitung der Zellpopulation und die Migrationsdynamik einzelner Zellen über Wochen Kultur kann mit Abbildung lebender Zellen überwacht und analysiert, um raumzeitlich aufgelösten Daten zu extrahieren. Darueber HinausDas Verfahren ist für verschiedene extrazelluläre Matrizen leicht an und bietet so eine einfache, aber leistungsstarke Möglichkeit, die Rolle der biophysikalischen Faktoren in der Mikroumgebung auf die Zellmigration zu untersuchen.
Einleitung
Migration von Zellen spielt eine wichtige Rolle bei verschiedenen physiologischen Reaktionen, wie der Embryonalentwicklung, Hämostase und Immunantwort als auch in pathologischen Prozessen, wie Gefäßerkrankungen, Entzündungen und Krebs 1. Sezieren der biochemischen und biophysikalischen Faktoren Zellmigration ist es daher von grundlegender Bedeutung nicht nur für die Grundprinzipien der zellulären Funktionen zu verstehen, sondern auch für verschiedene biomedizinische Anwendungen, wie zB im Tissue Engineering, Anti-Metastasen und entzündungshemmenden Medikamentenentwicklung voranzutreiben. Da in vivo Beobachtung ist technisch anspruchsvoll, hat viele Bemühungen auf die in vitro Rekapitulation Zellmigration konzentriert.
In-vitro-Verfahren zur Zellmigration zu untersuchen sind weitgehend für Assays auf zweidimensionale (2D) Oberflächen der Grund auf neu konzipiert worden, vor allem oder Wundheilungs Test 2. Solche Tests bieten einfachen Versuchsaufbau, einfache Live-Cell Imaging und liefern nützliche Einblicke in verschiedene biochemische Mechanismen der Zellmigration zugrunde liegen. Allerdings sind diese Tests nicht für die extrazelluläre Matrix (ECM) Architektur und Umbau, die kritischen Aspekte im Verständnis in vivo Migration berücksichtigen. Kürzlich wurde zunehmend klar, dass ein 3D-Kulturmodell häufig Kollagen-Matrices auf der Basis 3, eine Plattform, die besser ähnelt der in vivo Situation. Tatsächlich Zellen zeigen Migrations Dynamik, die sich von denen auf 2D Oberflächen sind, insbesondere aufgrund der unterschiedlichen Dimensionalität der Umgebung 4. Darüber hinaus sind die biophysikalischen und mechanischen Eigenschaften der Matrix beeinflussen empfindlich Zellmigration 5, auch im Rahmen der Tumorzellinvasion 6.
Hier präsentieren wir eine Methode zur 3D-Zellmigrationsverhalten in ECM mit biophysikalischen Eigenschaften, die mit Herstellungsbedingungen leicht variiert werden kann studieren. Die Zellen sindin einem "inneren Gel" ausgesät und dürfen sich zu entkommen und dringen in die anfänglich zellfreie "äußeren Gel". Das Verfahren stützt sich auf die Fähigkeit der Zelle, das Vorhandensein von zu erkennen, und die Neigung in der äußeren Gel, das in engem Zusammenhang mit Zell mechanosensing 7 verbunden ist, in zellfreien Bereiche migrieren. In dieser Studie beschäftigen wir Kollagen Netzwerke wie die ECM von hoch invasiven Brustkrebszellen, MD-MBA-231 eingedrungen. Die mechanischen Eigenschaften und die Mikrostruktur sowohl der inneren und äußeren Gele können abgestimmt werden, 8 und 9 dadurch um physiologisch relevante Bedingungen zu erreichen. Rekonstruktion und Analyse der Zellspuren erlauben detaillierte quantitative Untersuchung der raumzeitlichen Wanderungsverhalten sowohl auf Bevölkerungsebene und Einzelzellebene. Wichtig ist, dass der Aufbau des konzentrischen Gelsystem ahmt die in vivo Gewebetopologie durch wandernde Zellen, insbesondere Krebszellen eindringen und bietet somit wichtige Einblicke in konfrontiertan die physikalischen Mechanismen der Zellmigration und Metastasierung.
Protokoll
1. Zellernte
- Erhalten MD-MBA-231-Zellen aus dem 37 ° C, 5% CO 2 -Inkubator. Nehmen Zellen aus Gewebekulturplatte mit 0,5% Trypsin-EDTA-Lösung. Verwenden Sie 1 ml Trypsin-EDTA-Lösung für die Zelle in einem T25-Kolben kultiviert.
- Pellet-Zellen in einem 15 ml konischen Röhrchen durch Zentrifugation bei 200 × g für 4 min, saugt den Überstand, und die Zellen in 5 ml Kulturmedium.
- Zählen der Zelldichte, ρ, unter Verwendung eines Hämocytometers.
Hinweis: Um die Zellaussaat inneren Gel herzustellen, wird die Zellsuspension später verdünnt werden 10 x, um die endgültige Zellaussaatdichte zu erreichen. Daher ist 10 × konzentrierten Zellsuspension benötigt. - Berechnen Sie das Volumen des Mediums erforderlich, um eine 10 x Zellkonzentration zu erreichen:
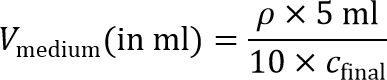
Hinweis: Eine endgültige Zellaussaatdichte, c Finale von etwa 215; 10 6 Zellen / ml für MD-MBA-231-Zellen empfohlen und ist in diesem Protokoll verwendet. Andere Einsaatdichten kann auch für andere Zelltypen untersucht werden. - Pellet-Zellen ein weiteres Mal in einem 15 ml konischen Röhrchen durch Zentrifugation bei 200 × g für 4 min, und Aspiration des Überstands.
- Die Zellen in der erforderlichen Menge (V Medium in Schritt 1.3 berechnet) von serumfreien Zellkulturmedium gründlich Zellverklumpung minimieren.
Anmerkung: Phenolrot Autofluoreszenz und können mit Fluoreszenz / Reflexionsabbildungs stören. Verwendung von phenolrotfreiem Medium kann als die beste Bildqualität zu erzielen.
2. Herstellung von Kollagenlösungen
- Besorgen Sie sich die Lager Kollagenlösung, 10 × PBS-Puffer, Milli-Q H 2 O, 0,1 M NaOH, und mehrere Reaktionsgefäße. Halten Sie alle auf dem Eis zu einer vorzeitigen Kollagen Polymerisation zu verhindern und zu pflegen steril.
- Äquilibrieren einer sterilen GlasBodenschale durch Vorwärmen in der 37 ° C-Inkubator.
Hinweis: Alle Bände in diesem Protokoll sind für die Glasbodenschale mit 12 mm und optimiert. Wenn andere Art von Geschirr verwendet wird, stellen Sie die Lautstärke entsprechend. - Berechnen des erforderlichen Volumens benötigt, um 50 & mgr; l von 2,4 mg / ml Collagenlösung für die innere Gel (Lösung I), bezogen auf das Kollagen Stoffkonzentration herzustellen.
Hinweis: Andere Kollagenkonzentrationen für die innere Gel kann ebenfalls verwendet werden. - In einer sterilen Umgebung (typischerweise ein Biosicherheitshaube), langsam 5 ul 10 × PBS-Puffer auf die gewünschte Menge an Kollagen-Stammlösung (in Schritt 2.3 berechnet) unter leichtem Schwenken. Achten Sie darauf, die Luftblasenbildung zu vermeiden.
- Den pH-Wert der Mischung auf 7,4 unter Verwendung von 0,1 M NaOH unter Verwendung von kalibrierten pH-Meters. Als grobe Richtlinie, verwenden Sie etwa 5 & mgr; der pH-Wert in der Nähe von 7,4 zu bringen (die Menge hängt von der Stoffkonzentration und pH-abhängig).
Hinweis: Beachten Sie, dass die Volumina in diesem beteiligtSchritt zu klein ist für den Einsatz von Standard-pH-Meter. Verwenden Sie eine der folgenden Tricks:- Bereiten Kollagenlösungen für mehrere Proben. Den pH-Wert in Groß mit Standard-pH-Meter und verteilen Sie die Kollagen-Lösungen in den Proben.
- Alternativ kann die pH-Wert einer Kollagenlösung einzustellen in größeren Volumen (i. E., Volumen, das die Verwendung von Standard-pH-Meter zu ermöglichen). Beachten Sie die NaOH-Menge benötigt wird, um den pH-Wert auf den End-pH zu bringen. Verkleinere die Volumina und verwenden Sie die entsprechende Menge an NaOH für das Experiment. Bestätigen Sie den pH-Wert mit Lackmuspapier.
- Andernfalls einen Mikro-pH-Elektrode, um den pH-Wert kleiner Mengen genauer einstellen.
- Man bringt die Lösung auf ein Volumen von 45 & mgr; l mit H 2 O. Führen Sie alle Schritte auf dem Eis zu einer vorzeitigen Kollagen Polymerisation zu verhindern.
3. Bildung von Concentric Gel Kultur
- Nehmen Sie die vorgewärmte Glasbodenschale (siehe Schritt 2.2) von thE-Inkubator.
- In 5 ul des 10 × konzentrierten Zellsuspension (in Schritt 1.5 vorbereitet) zu Lösung I resuspendieren gründlich. Achten Sie darauf, die Luftblasenbildung zu vermeiden. Die Mischung hat jetzt ein Volumen von 50 ul und enthält die endgültige Kollagenkonzentration (2,4 mg / ml) und die Zelldichte (c endgültige = 2 × 10 6 Zellen / ml).
- Mit 20 & mgr; l der Zellen enthaltenden Lösung I langsam auf die Mitte der Vertiefung, so dass es eine kuppelförmige Tröpfchen (1A) bildet. Achten Sie darauf, um eine Blasenbildung bei diesem Schritt zu vermeiden. Wenn eine Blase bildet, vorsichtig, aber schnell versuchen, entweder brechen oder sie saugen es aus mit einer Pipettenspitze. Legen Sie vorsichtig die Schale zurück in den Inkubator der innere Gel-Polymerisation für 45 Minuten zu lassen.
- Bereiten Lösung O (für die äußere, azellulärer Kollagengel) etwa 15 min vor dem Ende dieser Inkubationsstufe.
Anmerkung: Die äußere Gelzustand kann hinsichtlich der Kollagenkonzentration und Polymerisation pH variiert wirderhalten Netzwerken mit unterschiedlichen Mikrostrukturen 10. In diesem Protokoll werden, sich auf eine von 1,5 bis 4,0 mg / ml Kollagen-Gel bei einem pH-Wert von 7,4 polymerisiert.- Basierend auf der Kollagenstoffkonzentration, berechnet das erforderliche Volumen benötigt, um 200 ul Kollagenlösung O in einer Endkonzentration vorzubereiten.
- In 20 ul 10 x PBS Puffer langsam auf die erforderliche Menge an Kollagen-Stammlösung (in Schritt 3.4 berechnet) unter leichtem Schwenken. Man stellt den pH der Mischung auf den End-pH unter Verwendung von 0,1 M NaOH unter Verwendung von kalibrierten pH-Meters. Siehe Hinweis zu Schritt 2.5 in Bezug auf pH-Einstellung.
- Man bringt die Lösung auf ein Endvolumen von 200 & mgr; l mit H 2 O. Führen Sie alle Schritte auf dem Eis zu einer vorzeitigen Kollagen Polymerisation zu verhindern.
- Nehmen Sie die Schale aus dem Brutschrank nach 45 min Polymerisation des inneren Gels (siehe Schritt 3.3). Vorsichtig hinzu 180 ul Lösung A auf der Oberseite des inneren Gels, so dass die Lösung vollständig den innerenGel und füllt den gut (Abbildung 1B).
- Führen Sie diesen Schritt vorsichtig ohne Rühren der Lösung, was zu einer ungleichmäßigen Faserorientierungen in der äußeren Gel führen kann. Achten Sie darauf, nicht zu berühren das innere Gel mit einer Pipettenspitze, und um die Bildung von Blasen oder Lufteinschlüsse zu vermeiden. Wenn eine Blase bildet, vorsichtig, aber schnell versuchen, entweder brechen oder sie saugen es aus mit einer Pipettenspitze. Legen Sie vorsichtig die Schale zurück in den Inkubator, um die äußere Gel polymerisieren lassen.
- Nehmen Sie die Schale aus dem Brutschrank nach 45 min Polymerisation. Das Gel sollte schon ziemlich an dieser Stelle verfestigt werden, obwohl es immer noch von der Bodenfläche zu lösen, wenn grob behandelt.
- Vorsichtig gießen 2 ml erwärmt Zellkulturmedium in die Schale (Abbildung 1C). Achten Sie darauf, dass das Gel vollständig in das Medium eingetaucht ist. Aktualisieren Sie das Medium alle 2 - 3 Tage während der gesamten Dauer der Kultur.
4. Live-Cell-Imaging
- Führen Bildgebung mit einem inversen Konfokalmikroskop mit langfristigen Lebendzell-Imaging-Fähigkeit ausgestattet. Gehören eine eingebaute Inkubationskammer mit einer Temperatur (37 ° C) und CO 2 (5%) Kontrolle. Schalten Sie das Mikroskop und erwärmen die Bühne mindestens 1 Stunde vor dem Start des Experiments.
Hinweis: Verwenden Sie Objektiv mit großem Arbeitsabstand, um die Beobachtung und Lokalisierung von Zellen in den 3D-Gele zu optimieren. - Inkubieren des Gels in Medium, das 5 & mgr; l Zell-Fluoreszenzfarbstoff-Tracker für 30 min, um eine genaue Lokalisierung der Zellen in der 3D-System zu ermöglichen. Anschließend entfernen ungebundenen Farbstoff durch dreimaliges Waschen mit 1 x PBS. Anschließend fügen Zellkulturmedium zu der Schale.
- Nehmen Sie die Schale aus dem Inkubator nehmen und auf dem Mikroskoptisch (1D).
Hinweis: Live-Cell-Imaging kann grundsätzlich direkt nach der Polymerisation der äußeren Gel starten. Jedoch zu diesem Zeitpunkt die Zellen in die inneren Gel noch nicht verbreitet. Um die Migration der ersten Zellen, die eingedrungen sind die äußere Gel kann Abbildung lebender Zellen 24 Stunden zu starten (je nach Zelltyp) nach dem Beginn der Kultur zu untersuchen. Als grobe Richtlinie, in der Umgebung von 12 bis 14 Tagen der Kultur werden für die meisten der Zellen in der inneren Gels erforderlich, um den äußeren Gels geben. - Wählen Sie die Volumes of View (VoV die) in Regionen, in der äußeren Gel um den inneren Gel. Nach 24 Stunden Inkubation wird die Zellpopulation ausgebreitet haben, überquerte die Schnittstelle zwischen den inneren und äußeren Gele, und fing an, die äußere Gel einzudringen.
- Für die VoV ist, sind die Gel-Regionen in unmittelbarer Nähe der Gel-Grenzfläche, Zwischenbereiche und Regionen nahe am Rand der äußeren Gel 7. Regionen nicht näher als 50 & mgr; m von den Boden- und Seitenflächen, als auch von der Oberseite des Gels, um mögliche Randeffekte zu vermeiden. Jedes VoV Regel misst 647 x 647 x 100 um 3 (in x, Y und z-Richtung, jeweils), mit 5 & mgr; m Abstand in der z -stack.
- Stellen Sie sicher, Bildgebungsmodi, Kanäle / Filter, Belichtungszeit und Bildauflösungen sind richtig ausgewählt. Für markierungsfreie Bildgebung des Kollagennetzwerk verwenden konfokalen Reflexionsmikroskopie gleichzeitig während der Zeitraffer-Live-Cell-Imaging.
- Nehmen Sie Beispielbilder, zeichnen Sie die Intensität Histogramme, und passen Sie die Gewinne und Offsets, um eine ausreichende Signal beobachten und Sättigung zu vermeiden, indem sichergestellt wird, dass das Histogramm liegt zwischen Null und der maximalen Intensität. Diese Einstellungen nicht mehr während der gesamten Dauer des Experiments nicht verändern.
- Nehmen Sie Zeitraffer-Aufnahmen der ausgewählten VoV die mit einem Zeitintervall Δ t, von 10 min 8 Stunden (oder länger, falls erforderlich).
5. Zellverfolgung und Datenanalyse
- Führen Sie quantitative Bildnachbearbeitung von der z -stack Bildern mit? Fall te Bildverarbeitungs-Software.
- Segment die Zeitraffer-Aufnahmen, um automatisch die 3D-Zellpositionen (x, y, z, t).
- Für jeden Rahmen manuell überprüfen Sie die Lokalisierungsgenauigkeit und entfernen False Positives durch Zelltrümmer und Zell Vorsprünge, die für Zellen geirrt haben kann. Entfernen proliferativen Zellen von der Analyse und Split Überlappung oder verbundenen Zellen in verschiedene Objekte.
- Generieren 3D Zeitrafferzellspuren aus den Zellkoordinaten (x, y, z, t) in dem vorhergehenden Schritt durch Verknüpfung der Position jeder Zelle in der Zeitsequenz erhalten.
- Beseitigen zufälligen und Systemrauschen durch Entfernen Spuren kürzer als eine Schwellengleislänge (typischerweise 20 min).
- Korrigieren Sie für die Probendrift durch Subtraktion der Gesamtnettoverschiebungen von den Gleisen, wenn nötig.
- Berechnen Zell Verschiebung,iles / ftp_upload / 52.735 / 52735eq2.jpg "width =" 80 "/>, und die Zellwanderung Abstand,
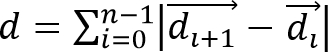 ,, Wobei aus den beobachteten Zell Trajektorien ein Vektor, der die 3D-Position von einer Zelle an Zeit und ist die Gesamtzahl von Zeitpunkten.
,, Wobei aus den beobachteten Zell Trajektorien ein Vektor, der die 3D-Position von einer Zelle an Zeit und ist die Gesamtzahl von Zeitpunkten. - Berechnen Zellengeschwindigkeit S = d / (n • Δ t), wo Δ t ist das Zeitintervall zwischen den Rahmen. Berechnen der Zellmigration Direktionalität (oder Persistenz) mit P = & Dgr; d / d. Durch diese einfache Maßnahme der Persistenz bedeutet, dass für P = 0 ist die Nettoverschiebung Null ist und für P = 1 die Bahn eine gerade Richtungslinie.
Ergebnisse
Die konzentrische Geltest hier vorgestellt wurde unter Verwendung hochinvasiven Brustkrebszellen MDA-MB-231 mit 2,4 mg / ml inneren Kollagen-Gel und einer Zellansiedlungsdichte = 2 x 10 6 Zellen / ml, als Beispiel. Wie in Abbildung 2 dargestellt, in der Regel nach wenigen Tagen Kultur verletzt die Zellen die Innen-Außen-Gel-Grenzfläche und damit begonnen, die äußeren Gel einzudringen. Die Zellpopulation verbreiten überwiegend radial nach außen.
Die Polymeris...
Diskussion
In this protocol we describe an in vitro assay to study the 3D migrational behavior of cells in matrix environments that topologically resemble ECMs encountered in vivo. There are three main strengths of this assay as compared to other currently available methods. First, this assay allows one to simultaneously examine the cell migration mechanisms at both population level and individual cell level. This opens up possibilities of studying collective cell migration13, which has to date been lar...
Offenlegungen
Die Autoren haben nichts zu offenbaren.
Danksagungen
Die Autoren danken W. Sun und K. Jansen für die kritischen Diskussionen und Unterstützung durch die Nano Biomechanics Lab erkennen an der National University of Singapore. NAK dankt für die Unterstützung durch ein Marie-Curie-Stipendium IIF.
Materialien
| Name | Company | Catalog Number | Comments |
| Cell culture incubator | Fisher Scientific Pte Ltd | Model: 371, S/No 318854-6055 | |
| Confocal microscope | Nikon A1R | Inverted confocal laser scanning microscope equipped with incubator chamber | |
| Dulbecco's Modified Eagle's Medium (DMEM) | Life Technologies | 11965-092 | |
| Fetal Bovine Serum (FBS) | Life Technologies | 10082-147 | |
| Fluorescent CellTracker dye CMTMR | Life Technologies | C2927 | |
| Glass-bottom dish | IWAKI Cell Biology | 3931-035 | 35 mm diameter dish with 12 mm diameter glass-bottom well |
| Hemocytometer | iN CYTO | DHC-N01 (Neubauer Improved) | |
| Microprocessor pH meter | Hanna Instruments | pH 211 | |
| Nutragen Collagen | Advanced BioMatrix | #5010-D | Acid-solubilized bovine collagen type I (stock pH ~ 2) |
| Objective lens | Nikon | CFI Super Plan Fluor ELWD ADM 20XC, W.D. 8.2-6.9mm, NA 0.45. | |
| Penicillin-Streptomycin | Life Technologies | 15140-122 | |
| pH meter | Sartorius | S/No 29153352 | Basic pH Meter PB-11 |
| Trypsin-EDTA | Life Technologies | 15400-054 |
Referenzen
- Horwitz, R., Webb, D. Cell migration. Curr Biol. 13 (19), R756-R759 (2003).
- Liang, C. C., Park, A. Y., Guan, J. L. In vitro scratch assay: a convenient and inexpensive method for analysis of cell migration in vitro. Nat. Protoc. 2 (2), 329-333 (2007).
- Provenzano, P. P., Eliceiri, K. W., Inman, D. R., Keely, P. J. Engineering three-dimensional collagen matrices to provide contact guidance during 3D cell migration. Curr. Prot. Cell Biol. 10, 10-17 (2010).
- Friedl, P., Sahai, E., Weiss, S., Yamada, K. M. New dimensions in cell migration. Nat. Rev. Mol. Cell Biol. 13 (11), 743-747 (2012).
- Grinnell, F., Petroll, W. M. Cell motility and mechanics in three-dimensional collagen matrices. Annu. Rev. Cell. Dev. Biol. 26, 335-361 (2010).
- Wirtz, D., Konstantopoulos, K., Searson, P. C. The physics of cancer: the role of physical interactions and mechanical forces in metastasis. Nat. Rev. Cancer. 11 (7), 512-522 (2011).
- Sun, W., Kurniawan, N. A., Kumar, A. P., Rajagopalan, R., Lim, C. T. Effects of migrating cell-induced matrix reorganization on 3D cancer cell migration. Cell. Mol. Bioeng. 7 (2), 205-217 (2014).
- Achilli, M., Mantovani, D. Tailoring mechanical properties of collagen-based scaffolds for vascular tissue engineering: the effects of pH, temperature and ionic strength on gelation. Polymers. 2 (4), 664-680 (2010).
- Kurniawan, N. A., Wong, L. H., Rajagopalan, R. Early stiffening and softening of collagen: interplay of deformation mechanisms in biopolymer networks. Biomacromolecules. 13 (3), 691-698 (2012).
- Sun, W., Lim, C. T., Kurniawan, N. A. Mechanistic adaptability of cancer cells strongly affects anti-migratory drug efficacy. J. R. Soc. Interface. 11 (99), 20140638 (2014).
- Guzman, A., Ziperstein, M. J., Kaufman, L. J. The effect of fibrillar matrix architecture on tumor cell invasion of physically challenging environments. Biomaterials. 35 (25), 6954-6963 (2014).
- Wolf, K., et al. Physical limits of cell migration: control by ECM space and nuclear deformation and tuning by proteolysis and traction force. J. Cell Biol. 201 (7), 1069-1084 (2013).
- Friedl, P., Gilmour, D. Collective cell migration in morphogenesis, regeneration and cancer. Nat. Rev. Mol. Cell Biol. 10 (7), 445-457 (2009).
- Vedula, S. R. K., Ravasio, A., Lim, C. T., Ladoux, B. Collective cell migration: a mechanistic perspective. Physiology. 28 (6), 370-379 (2013).
- Petrie, R. J., Doyle, A. D., Yamada, K. M. Random versus directionally persistent cell migration. Nat. Rev. Mol. Cell Biol. 10 (8), 538-549 (2009).
- Miron-Mendoza, M., Seemann, J., Grinnell, F. The differential regulation of cell motile activity through matrix stiffness and porosity in three dimensional collagen matrices. Biomaterials. 31 (25), 6425-6435 (2010).
- Mouw, J. K., et al. Tissue mechanics modulate microRNA-dependent PTEN expression to regulate malignant progression. Nat. Med. 20 (4), 360-367 (2014).
- Wolf, K., et al. Collagen-based cell migration models in vitro and in vivo. Semin. Cell Dev. Biol. 20 (8), 931-941 (2009).
- Wong, L. H., Kurniawan, N. A., Too, H. -. P., Rajagopalan, R. Spatially resolved microrheology of heterogeneous biopolymer hydrogels using covalently bound microspheres. Biomech. Model. Mechanobiol. 13 (4), 839-849 (2014).
- Gupton, S. L., Waterman-Storer, C. M. Spatiotemporal feedback between actomyosin and focal-adhesion systems optimizes rapid cell migration. Cell. 125 (7), 1361-1374 (2006).
- Provenzano, P. P., Inman, D. R., Eliceiri, K. W., Trier, S. M., Keely, P. J. Contact guidance mediated three-dimensional cell migration is regulated by Rho/ROCK-dependent matrix reorganization. Biophys. J. 95 (11), 5374-5384 (2008).
- Wolf, K., et al. Multi-step pericellular proteolysis controls the transition from individual to collective cancer cell invasion. Nat. Cell Biol. 9 (8), 893-904 (2007).
- Jansen, K. A., Bacabac, R. G., Piechocka, I. K., Koenderink, G. H. Cells actively stiffen fibrin networks by generating contractile stress. Biophys. J. 105 (10), 2240-2251 (2013).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten