É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Sistema de Gel Concentric para estudar o papel Biofísica da Matrix Microenvironment sobre a migração celular 3D
Neste Artigo
Resumo
As propriedades mecânicas e a micro estrutura da matriz extracelular afectar fortemente a migração de células 3D. Um método in vitro para estudar o comportamento de migração celular no espaço-temporal ambientes biofísico variáveis, tanto na população e os níveis de células individuais, é descrito.
Resumo
A capacidade das células para migrar é crucial numa grande variedade de funções celulares ao longo da vida do desenvolvimento embrionário e na cicatrização de tumor e a metástase do cancro. Apesar dos esforços intensos de investigação, os princípios bioquímicos e biofísicos básicos de migração de células ainda não são totalmente compreendidos, especialmente nas três dimensões (3D) microambientes fisiologicamente relevantes. Aqui, nós descrevemos um ensaio in vitro concebido para permitir a análise quantitativa de 3D comportamentos de migração celular. O método explora mechanosensing capacidade da célula e propensão para migrar para dentro da matriz extracelular previamente desocupado (ECM). Usamos a invasão de células do cancro da mama altamente invasivos, MDA-MB-231, em géis de colagénio, tal como um sistema modelo. A disseminação da população de células e as dinâmicas migratórias de células individuais ao longo de semanas de cultura pode ser controlada por meio de imagens ao vivo de células e analisados para extrair dados espaço-temporalmente-resolvidos. Além Do Mais, O método é facilmente adaptável para diversas matrizes extracelulares, oferecendo assim uma maneira simples e poderosa para investigar o papel dos factores biofísicos no microambiente sobre a migração celular.
Introdução
A migração de células desempenha um papel chave em várias respostas fisiológicas, tais como o desenvolvimento embrionário, hemostase, e resposta imunitária, bem como em processos patológicos tais como doenças vasculares, inflamação e cancro 1. Dissecando os factores bioquímicos e biofísicos subjacentes a migração celular é portanto fundamentalmente importante não só para compreender os princípios básicos de funções celulares, mas também para fazer avançar várias aplicações biomédicas, tais como em engenharia de tecidos, anti-metástases e desenvolvimento de drogas anti-inflamatórias. Desde observação in vivo é tecnicamente desafiador, uma série de esforços tem sido focada em in vitro recapitulação da migração celular.
Os métodos in vitro para estudar a migração de células têm sido amplamente concebido para ensaios em duas superfícies (2D) dimensionais, mais notavelmente o arranhão ou ferimento ensaio 2 de cura. Tais ensaios oferecem configuração experimental simples, fácil live-imagens de células, e fornecer informações úteis sobre vários mecanismos bioquímicos subjacentes migração celular. No entanto, estes ensaios não têm em conta a arquitetura da matriz extracelular (ECM) e remodelação, que são aspectos críticos na compreensão da migração vivo. Recentemente, tem sido cada vez mais apreciado que um modelo de cultura 3D, muitas vezes em matrizes à base de colagénio 3, fornece uma plataforma que melhor se assemelha à situação in vivo. De fato, as células apresentam dinâmicas migratórias que são distintas daquelas em superfícies 2D, especialmente devido à diferente dimensionalidade do meio ambiente 4. Além disso, as propriedades biofísicas e mecânicas da matriz afecta sensivelmente a migração celular 5, nomeadamente no contexto de invasão de células tumorais 6.
Aqui, apresentamos um método para estudar o comportamento de migração celular 3D em ECM com propriedades biofísicas que podem ser facilmente variado, com condições de preparação. As células sãosemeadas em um "gel interior" e estão autorizados a escapar para dentro e invadir o "gel exterior" inicialmente acelular. O método baseia-se na capacidade da célula para reconhecer a presença de, e a propensão para migrar para dentro, regiões livres de células no exterior de gel, que está intimamente ligado à célula mechanosensing 7. Neste estudo, utilizamos redes de colágeno que a ECMs invadidas por células de câncer de mama altamente invasivos, MD-MBA-231. As propriedades mecânicas e microestrutura de ambos os géis de interiores e exteriores pode ser sintonizado 8 e 9 caracterizado para atingir as condições f isiologicamente relevantes. Reconstrução e análise das faixas de células permitir o exame quantitativa detalhada do comportamento de migração espaço-temporais, tanto a nível da população e nível de célula individual. É importante notar que a configuração do sistema de gel concêntrico imita a topologia de tecido in vivo enfrentado pelas células migrantes, especialmente células cancerosas invasoras, oferecendo, assim, conhecimentos importantespara os mecanismos físicos de migração celular e metástase.
Protocolo
Colheita 1. celular
- Obter MD-MBA-231 a partir do 37 ° C, 5% de CO2. Separar as células do tecido placa de cultura utilizando-se 0,5% de solução de Tripsina-EDTA. Use 1 ml de solução de tripsina-EDTA para células cultivadas num frasco T25.
- Células de pellets em um tubo de 15 ml por centrifugação a 200 x g durante 4 min, aspirar o sobrenadante e ressuspender as células em 5 ml de meio de cultura.
- Contagem de densidade celular, ρ, utilizando um hemocitómetro.
Nota: Para preparar o gel interior da célula-semeado, a suspensão de células vai ser, mais tarde, diluiu-se 10 × para atingir a densidade final de células de semeadura. Portanto, 10 × suspensão concentrada de célula é necessário. - Calcula-se o volume de meio necessário para atingir uma concentração de 10 × celular:
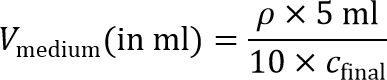
Nota: A densidade final semeadura de células, c final, de cerca de 215; 10 6 células / ml é recomendado para MD-MBA-231 e é utilizado no presente protocolo. Outros densidades de sementeira podem também ser explorado para outros tipos de células. - Células de pellets mais uma vez em um tubo de 15 ml por centrifugação a 200 × g por 4 min, e aspirar o sobrenadante.
- Ressuspender as células na quantidade necessária (V médio calculadas no passo 1.3) de meio de cultura celular livre de soro cuidadosamente para minimizar a aglutinação celular.
Nota: O fenol vermelho é auto-fluorescente, e pode interferir com a imagiologia por fluorescência / reflectância. O uso de meio isento de vermelho de fenol pode ser considerado para alcançar melhor qualidade de imagem.
2. Preparação de Soluções de colágeno
- Obter a solução de colagénio de estoque, 10 × tampão PBS, Milli-Q H 2 O, NaOH a 0,1 M, e vários tubos de microcentrífuga. Mantenha tudo no gelo para evitar a polimerização prematura de colágeno, e manter a condição estéril.
- Equilibrar uma Glass- estérilprato inferior por pré-aquecendo-a no 37 ° C incubadora.
Nota: Todos os volumes neste protocolo foram otimizados para um prato fundo de vidro de 12 mm assim. Se outro tipo prato é usado, ajustar os volumes proporcionalmente. - Calcular o volume requerido necessário para preparar 50 ul de solução de colagénio a 2,4 mg / ml para o gel interior (solução I) com base na concentração de estoque de colagénio.
Nota: Outras concentrações de colagénio para o interior de gel pode também ser usado. - Em um ambiente estéril (tipicamente um capuz biosegurança), adiciona-se lentamente 5 mL de 10 × tampão PBS para a quantidade necessária de solução-mãe de colagénio (calculado no passo 2.3), com agitação suave. Tome cuidado para evitar a formação de bolhas de ar.
- Ajustar o pH da mistura para 7,4 utilizando NaOH 0,1 M calibrado usando medidor de pH. Como orientação geral, utilizar cerca de 5 ul para trazer o pH para perto de 7,4 (a quantidade varia dependendo da concentração estoque e pH).
Nota: Observe que os volumes envolvidos nestepasso é demasiado pequeno para o uso do medidor de pH padrão. Use um dos seguintes truques:- Preparar soluções de colágeno para várias amostras. Ajustar o pH em grandes quantidades usando um medidor de pH padrão e distribuir as soluções de colagénio entre as amostras.
- Em alternativa, ajustar o pH de uma solução de colagénio em maior volume (i. E., O volume que permitem o uso de medidor de pH padrão). Note-se a quantidade de NaOH necessária para trazer o pH para o valor final de pH. Reduza os volumes e usar a quantidade apropriada de NaOH para o experimento. Confirme o valor pH utilizando papel Litmus.
- Caso contrário, utilizar um eléctrodo de pH de micro para ajustar de forma mais precisa o pH de pequenas quantidades.
- Levar a solução para um volume de 45 ul usando H 2 O. Execute todas as etapas no gelo para evitar a polimerização prematura de colágeno.
3. Formação de Concentric Cultura Gel
- Pegue o prato com fundo de vidro pré-aquecido (veja o passo 2.2) a partir de the incubadora.
- Adiciona-se 5 ul da suspensão de células de 10 x concentrado (preparado no passo 1.5) a solução I. Ressuspender completamente. Tome cuidado para evitar a formação de bolhas de ar. A mistura agora tem um volume de 50 ul e contém a concentração final de colagénio (2,4 mg / ml) e a densidade celular (final c = 2 × 10 6 células / ml).
- Adicionar 20 ul da solução contendo as células que se lentamente para o centro do poço, de modo a formar uma gotícula (Figura 1A) em forma de cúpula. Tome cuidado para evitar a formação de bolhas nesta etapa. Caso se forme um bolha, com cuidado, mas rapidamente quer tentar rompê-lo ou sugá-lo para fora usando uma ponteira. Suavemente colocar o prato de volta na incubadora para permitir que o gel interior polimerizar durante 45 minutos.
- Preparar a solução de O (para o, gel de colagénio acelular exterior) cerca de 15 minutos antes do final deste passo de incubação.
Nota: A condição exterior de gel pode ser variada em termos de concentração de colagénio e ao pH de polimerizaçãoobter redes com diferentes microestruturas 10. Neste protocolo, o foco sobre um 1,5 - gel de colagénio a 4,0 mg / ml polimerizada a um pH de 7,4.- Com base na concentração de estoque de colagénio, calcular o volume requerido necessário para preparar 200 mL de solução de colagénio O na concentração final.
- Adicionar 20 ul de tampão PBS 10 × lentamente para a quantidade necessária de solução-mãe de colagénio (calculado no passo 3.4), com agitação suave. Ajustar o pH da mistura para o valor final de pH utilizando NaOH 0,1 M e o uso de medidor de pH calibrado. Veja a nota para o passo 2.5 relativamente ao ajuste de pH.
- Levar a solução para um volume final de 200 ul usando H 2 O. Execute todas as etapas no gelo para evitar a polimerização prematura de colágeno.
- Leve o prato da incubadora após 45 min de polimerização do gel interior (veja o passo 3.3). Adicionar cuidadosamente 180 ul de solução de S no topo do gel interior, de modo que a solução de cobre completamente o interiorgel e enche-se a cavidade (Figura 1B).
- Executar este passo cuidadosamente sem agitar a solução, o que pode levar a orientações de fibras não-uniformes no exterior de gel. Tome cuidado para evitar tocar o gel interno com uma ponteira, e para evitar a formação de bolhas ou bolsas de ar. Caso se forme um bolha, com cuidado, mas rapidamente quer tentar rompê-lo ou sugá-lo para fora usando uma ponteira. Suavemente colocar o prato de volta na incubadora para permitir que a polimerizar exterior de gel.
- Leve o prato da incubadora após 45 min de polimerização. O gel deve ser já bastante solidificado, neste ponto, embora ainda pode destacar da superfície inferior, se manuseada com cuidado.
- Suavemente para 2 ml de meio de cultura celular aquecida ao prato (Figura 1C). Certifique-se de que o gel é completamente submergido no meio. Atualize a médio a cada 2-3 dias ao longo do período de cultura.
4. Imagens ao vivo de células
- Realizar imagem usando um microscópio confocal invertido equipado com capacidade de geração de imagens de células vivas de longo prazo. Incluir um embutido câmara de incubação com uma temperatura (37 ° C) e CO2 (5%) de controlo. Ligue o microscópio e aquecer o palco, pelo menos, 1 hora antes do início do experimento.
Nota: Use lente objetiva com longa distância de trabalho para otimizar a observação e localização das células nos géis 3D. - Incubar o gel em meio contendo 5 ul de corante de rastreador de células fluorescentes durante 30 min para permitir uma localização precisa das células no sistema 3D. Posteriormente, remover o corante não ligado por lavagem três vezes com 1 × PBS. Em seguida, adicionar meio de cultura de células para o prato.
- Leve o prato da incubadora e colocá-lo no palco microscópio (Figura 1D).
Nota: imagens de células vivas pode começar em princípio, logo após a polimerização do gel exterior. No entanto, neste momento, as células in O gel interno não se espalharam ainda. Para examinar a migração das primeiras células que invadiram o exterior de gel, de imagem de células vivas pode começar 24 horas (dependendo do tipo de células) após o inicio da cultura. Como um guia, em torno de 12 - são necessárias mais 14 dias de cultura para a maioria das células no interior do gel para entrar no exterior de gel. - Selecionar volumes de Vista (VOV de) em regiões do gel exterior em torno do gel interior. Após 24 h de incubação, a população de células terá difundido, atravessou a interface entre os geles de interior e exterior, e começou a invadir o exterior de gel.
- Para o VOV de, incluem as regiões de gel imediatamente ao lado da interface de gel, regiões intermédias, e as regiões perto da periferia da gel exterior 7. Excluir regiões mais próximas do que 50 mm a partir das superfícies de fundo e laterais, bem como a partir do topo do gel, a fim de evitar possíveis efeitos de borda. Cada VOV tipicamente mede 647 × 647 × 100 mm 3 (em x, y, z e indicações, respectivamente), com intervalo de 5 mm na -stack z.
- Assegurar modos de imagem, canais / filtros, tempos de exposição, e resoluções de imagem estão corretamente selecionado. Para imagens de livre-label da rede de colágeno, utilize microscopia confocal de reflectância simultaneamente durante o lapso de tempo de imagens ao vivo de células.
- Tome imagens de exemplo, traçar os histogramas de intensidade, e ajustar os ganhos e compensações para observar sinal suficiente e evitar a saturação, assegurando que o histograma fica entre zero e a intensidade máxima. Não altere estas configurações mais durante todo o período do experimento.
- Tome imagens de lapso de tempo dos VOV de selecionados, com um intervalo de tempo, Δ t, de 10 min para 8 horas (ou mais, se necessário).
5. Acompanhamento de celular e Análise de Dados
- Realizar imagem quantitativa pós-processamento das imagens -stack z utilizando appropria software de processamento de imagem te.
- Segmento as imagens de lapso de tempo para selecionar automaticamente as posições celulares 3D em (x, y, z, t).
- Para cada quadro, verificar manualmente a precisão de localização e remover falsos positivos devido a restos de células e saliências celulares que podem ter sido confundido com células. Remover as células de proliferação a partir da análise e as células que se sobrepõem ou ligados divididas em objetos distintos.
- Gerar faixas de células de lapso de tempo em 3D a partir de células (as coordenadas x, y, z, t) obtidos no passo anterior, ligando a localização de cada célula na sequência de tempo.
- Elimine o ruído aleatório e sistema removendo faixas mais curtos do que um comprimento de rasto limiar (tipicamente 20 minutos).
- Corrija para a amostra deriva subtraindo os deslocamentos líquidos globais dos trilhos, se necessário.
- Calcular o deslocamento de células,iles / ftp_upload / 52735 / 52735eq2.jpg "width =" 80 "/>, e distância de migração celular,
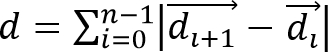 , A partir de células observadas as trajectórias, em que é um vector que representa a localização de uma célula de 3D em tempo e é o número total de pontos de tempo.
, A partir de células observadas as trajectórias, em que é um vector que representa a localização de uma célula de 3D em tempo e é o número total de pontos de tempo. - Calcular a velocidade célula como S = d / (n • Δ t), onde Δ t é o intervalo de tempo entre os quadros. Calcule direcionalidade migração celular (ou persistência) usando P = Δd / d. Esta simples medida de persistência implica que, para P = 0 o deslocamento líquido é zero e para P = 1 a trajetória é uma linha reta direcional.
Resultados
O ensaio de gel concêntrico aqui apresentada foi realizada utilizando células de cancro da mama altamente invasivos, MDA-MB-231, com 2,4 mg / ml de gel de colagénio interior e uma densidade de sementeira de células de = 2 x 10 6 células / ml, como um exemplo. Como mostrado na Figura 2, tipicamente depois de alguns dias de cultura, as células violou a interface gel interior-exterior e começou a invadir o exterior de gel. A população de células predominantemente espalhar radialmente p...
Discussão
In this protocol we describe an in vitro assay to study the 3D migrational behavior of cells in matrix environments that topologically resemble ECMs encountered in vivo. There are three main strengths of this assay as compared to other currently available methods. First, this assay allows one to simultaneously examine the cell migration mechanisms at both population level and individual cell level. This opens up possibilities of studying collective cell migration13, which has to date been lar...
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
Os autores agradecem W. Sun e K. Jansen para as discussões críticas, e reconhecer o apoio pelo Nano Laboratório de Biomecânica da Universidade Nacional de Cingapura. NAK reconhece o apoio por um Marie Curie IIF Fellowship.
Materiais
| Name | Company | Catalog Number | Comments |
| Cell culture incubator | Fisher Scientific Pte Ltd | Model: 371, S/No 318854-6055 | |
| Confocal microscope | Nikon A1R | Inverted confocal laser scanning microscope equipped with incubator chamber | |
| Dulbecco's Modified Eagle's Medium (DMEM) | Life Technologies | 11965-092 | |
| Fetal Bovine Serum (FBS) | Life Technologies | 10082-147 | |
| Fluorescent CellTracker dye CMTMR | Life Technologies | C2927 | |
| Glass-bottom dish | IWAKI Cell Biology | 3931-035 | 35 mm diameter dish with 12 mm diameter glass-bottom well |
| Hemocytometer | iN CYTO | DHC-N01 (Neubauer Improved) | |
| Microprocessor pH meter | Hanna Instruments | pH 211 | |
| Nutragen Collagen | Advanced BioMatrix | #5010-D | Acid-solubilized bovine collagen type I (stock pH ~ 2) |
| Objective lens | Nikon | CFI Super Plan Fluor ELWD ADM 20XC, W.D. 8.2-6.9mm, NA 0.45. | |
| Penicillin-Streptomycin | Life Technologies | 15140-122 | |
| pH meter | Sartorius | S/No 29153352 | Basic pH Meter PB-11 |
| Trypsin-EDTA | Life Technologies | 15400-054 |
Referências
- Horwitz, R., Webb, D. Cell migration. Curr Biol. 13 (19), R756-R759 (2003).
- Liang, C. C., Park, A. Y., Guan, J. L. In vitro scratch assay: a convenient and inexpensive method for analysis of cell migration in vitro. Nat. Protoc. 2 (2), 329-333 (2007).
- Provenzano, P. P., Eliceiri, K. W., Inman, D. R., Keely, P. J. Engineering three-dimensional collagen matrices to provide contact guidance during 3D cell migration. Curr. Prot. Cell Biol. 10, 10-17 (2010).
- Friedl, P., Sahai, E., Weiss, S., Yamada, K. M. New dimensions in cell migration. Nat. Rev. Mol. Cell Biol. 13 (11), 743-747 (2012).
- Grinnell, F., Petroll, W. M. Cell motility and mechanics in three-dimensional collagen matrices. Annu. Rev. Cell. Dev. Biol. 26, 335-361 (2010).
- Wirtz, D., Konstantopoulos, K., Searson, P. C. The physics of cancer: the role of physical interactions and mechanical forces in metastasis. Nat. Rev. Cancer. 11 (7), 512-522 (2011).
- Sun, W., Kurniawan, N. A., Kumar, A. P., Rajagopalan, R., Lim, C. T. Effects of migrating cell-induced matrix reorganization on 3D cancer cell migration. Cell. Mol. Bioeng. 7 (2), 205-217 (2014).
- Achilli, M., Mantovani, D. Tailoring mechanical properties of collagen-based scaffolds for vascular tissue engineering: the effects of pH, temperature and ionic strength on gelation. Polymers. 2 (4), 664-680 (2010).
- Kurniawan, N. A., Wong, L. H., Rajagopalan, R. Early stiffening and softening of collagen: interplay of deformation mechanisms in biopolymer networks. Biomacromolecules. 13 (3), 691-698 (2012).
- Sun, W., Lim, C. T., Kurniawan, N. A. Mechanistic adaptability of cancer cells strongly affects anti-migratory drug efficacy. J. R. Soc. Interface. 11 (99), 20140638 (2014).
- Guzman, A., Ziperstein, M. J., Kaufman, L. J. The effect of fibrillar matrix architecture on tumor cell invasion of physically challenging environments. Biomaterials. 35 (25), 6954-6963 (2014).
- Wolf, K., et al. Physical limits of cell migration: control by ECM space and nuclear deformation and tuning by proteolysis and traction force. J. Cell Biol. 201 (7), 1069-1084 (2013).
- Friedl, P., Gilmour, D. Collective cell migration in morphogenesis, regeneration and cancer. Nat. Rev. Mol. Cell Biol. 10 (7), 445-457 (2009).
- Vedula, S. R. K., Ravasio, A., Lim, C. T., Ladoux, B. Collective cell migration: a mechanistic perspective. Physiology. 28 (6), 370-379 (2013).
- Petrie, R. J., Doyle, A. D., Yamada, K. M. Random versus directionally persistent cell migration. Nat. Rev. Mol. Cell Biol. 10 (8), 538-549 (2009).
- Miron-Mendoza, M., Seemann, J., Grinnell, F. The differential regulation of cell motile activity through matrix stiffness and porosity in three dimensional collagen matrices. Biomaterials. 31 (25), 6425-6435 (2010).
- Mouw, J. K., et al. Tissue mechanics modulate microRNA-dependent PTEN expression to regulate malignant progression. Nat. Med. 20 (4), 360-367 (2014).
- Wolf, K., et al. Collagen-based cell migration models in vitro and in vivo. Semin. Cell Dev. Biol. 20 (8), 931-941 (2009).
- Wong, L. H., Kurniawan, N. A., Too, H. -. P., Rajagopalan, R. Spatially resolved microrheology of heterogeneous biopolymer hydrogels using covalently bound microspheres. Biomech. Model. Mechanobiol. 13 (4), 839-849 (2014).
- Gupton, S. L., Waterman-Storer, C. M. Spatiotemporal feedback between actomyosin and focal-adhesion systems optimizes rapid cell migration. Cell. 125 (7), 1361-1374 (2006).
- Provenzano, P. P., Inman, D. R., Eliceiri, K. W., Trier, S. M., Keely, P. J. Contact guidance mediated three-dimensional cell migration is regulated by Rho/ROCK-dependent matrix reorganization. Biophys. J. 95 (11), 5374-5384 (2008).
- Wolf, K., et al. Multi-step pericellular proteolysis controls the transition from individual to collective cancer cell invasion. Nat. Cell Biol. 9 (8), 893-904 (2007).
- Jansen, K. A., Bacabac, R. G., Piechocka, I. K., Koenderink, G. H. Cells actively stiffen fibrin networks by generating contractile stress. Biophys. J. 105 (10), 2240-2251 (2013).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados