Method Article
Hochdurchsatz-Gene Tagging in
In diesem Artikel
Zusammenfassung
Addition of a tag to a protein is a powerful way of gaining insight into its function. Here, we describe a protocol to endogenously tag hundreds of Trypanosoma brucei proteins in parallel such that genome scale tagging is achievable.
Zusammenfassung
Improvements in mass spectrometry, sequencing and bioinformatics have generated large datasets of potentially interesting genes. Tagging these proteins can give insights into their function by determining their localization within the cell and enabling interaction partner identification. We recently published a fast and scalable method to generate Trypanosoma brucei cell lines that express a tagged protein from the endogenous locus. The method was based on a plasmid we generated that, when coupled with long primer PCR, can be used to modify a gene to encode a protein tagged at either terminus. This allows the tagging of dozens of trypanosome proteins in parallel, facilitating the large-scale validation of candidate genes of interest. This system can be used to tag proteins for localization (using a fluorescent protein, epitope tag or electron microscopy tag) or biochemistry (using tags for purification, such as the TAP (tandem affinity purification) tag). Here, we describe a protocol to perform the long primer PCR and the electroporation in 96-well plates, with the recovery and selection of transgenic trypanosomes occurring in 24-well plates. With this workflow, hundreds of proteins can be tagged in parallel; this is an order of magnitude improvement to our previous protocol and genome scale tagging is now possible.
Einleitung
Trypanosoma brucei ist ein einzelliger Parasit, der bei Rindern menschlichen afrikanischen trypanosomasis und Nagana verursacht. T. brucei ist ein idealer Organismus für die Analyse von Proteinfunktion aufgrund der Kombination von hoher Qualität Genoms, zahlreiche Proteomik und Transkriptomik Datensätze und gut entwickelte molekulare Werkzeuge 1-3. Die Fortschritte in der Proteomik und Sequenzierung haben in großen Datensätzen geführt , die potentiell interessante Gene 4-6 hervorzuheben; Allerdings haben viele Gene minimale Informationen mit ihnen in den vorhandenen Datenbanken verbunden. Es besteht daher ein Bedarf für ein Hochdurchsatzverfahren Protein funktionelle Charakterisierung zu unterstützen.
Expression eines markierten Proteins kann eine Vielzahl von Einsichten in ein Protein der Funktion geben. Zum Beispiel ist ein Protein mit einem fluoreszierenden Protein oder epitopmarkiert kann durch Fluoreszenzmikroskopie lokalisiert werden, die Informationen darüber, wo die prot gibtein könnte seine biologische Wirkung werden ausübt. Alternativ kann ein Protein mit einer TAP getaggt 7, HaloTag 8 oder His - Tag kann für biochemische Assays und Identifizierung seiner Interaktionspartner , gereinigt werden.
Wir entwickelten kürzlich eine robuste Tagging Methodik für T. brucei 9. Benutzten langen Primer PCR die DNA für die Transfektion zu erzeugen und das Tagging von Dutzenden von Proteinen in parallel erlaubt - eine wesentliche Verbesserung bestehender Protokolle. Wir haben nun die Skalierbarkeit dieses Protokolls in prozyklischen Formen durch eine Größenordnung verbessert. Hier stellen wir unsere Methode, wo wir die PCR und Transfektion in 96-Well-Platten durchzuführen, wobei die Rückgewinnung und Auswahl auftretenden in Platten mit 24 Vertiefungen. Wie Hunderte von Proteinen können nun parallel markiert bietet dieses Verfahren eine kostengünstige und praktikable Methode für die gesamte Trypanosomen Genom-Tagging.
Protokoll
1. 96-Well-Lange Primer PCR
- Pre-freeze 96-Well PCR-Kühler in -80 ° C Gefrierschrank.
- In einem 10 - ml - Tube, Vorbereitung Master Mix A: 2.100 ul ddH 2 O, 105 & mgr; l PCR - Klasse DMSO, 105 & mgr; l 10 mM dNTPs, 105 & mgr; l pPot - Vorlage (25 ng / ul) 9, für ein Gesamtvolumen von 2.415 ul pro Platte .
- Aliquot 23 ul in jede Vertiefung einer 96-Well-PCR-Platte.
- In 2 ul 100 uM gepoolt Primer an jeder geladen und ein P20 12-Kanal Mehrkanal-Pipette. Dichtung mit Film, Platz Platte auf der vorgekühlte PCR-Kühler und lassen für mindestens 20 min bei -80 ° C einzufrieren.
- In einem 10 - ml - Tube, Vorbereitung Master Mix B: 2.048 ul ddH 2 O, 525 ul 10x - Puffer, 52,5 ul Polymerase für ein Gesamtvolumen von 2.626 ul pro Platte.
- Entfernen Sie Platte und PCR-Kühler von -80 ° C-Gefrierschrank.
- Mit der Platte noch auf dem PCR-Kühler, fügen Sie 25 ul Master Mix B auf der Oberseite des gefrorn Master Mix A für eine Gesamtreaktionsvolumen von 50 & mgr; l. Dies ist zeitkritisch und sollte abgeschlossen sein, bevor Mix A auftauen kann. Dichtungsplatte mit Folie.
- Eingestellt Thermocycler wie folgt: 94 ° C für 10 min, dann 30 Zyklen von 94 ° C für 15 sec, 65 ° C für 30 sec, 72 ° C für 2 min, gefolgt von einem letzten Verlängerungszeitraum von 72 ° C für 7 min .
- Laden Sie die PCR-Platte, nachdem der Block 94 ° C erreicht hat.
2. Validierung von 96-Well-Lang Primer PCR
- Guss eine große 1% (w / v) Agarosegel in Tris-Acetat-EDTA (TAE) Laufpuffer mit 4 x 28-well Kämme ein Gel mit 4 Reihen von Vertiefungen zu geben. Richten abwechselnde Zähne des Kamms mit den Spitzen eines 12-Kanal-Mehrkanalpipette. versenken Sie vorsichtig das Gel in TAE-Puffer laufen, nachdem die Bahnen geladen werden.
- Last 1 kb DNA - Leiter in der 1. und 28. Vertiefung jeder Reihe.
- Unter Verwendung eines P20 12-Kanal Mehrkanal-Pipette 2 ul PCR-Produkt direkt auf jede Spur des Gels (1A). Tun Sie dies schnell die Möglichkeit einer Verunreinigung der Amplikons zu reduzieren.
- Laden Sie die PCR-Produkte aus der Zeile A der PCR-Platte in die ungeraden Bahnen der ersten Reihe des Gels (A1 - Spur 3, A2 - Bahn 5 ...... A11 - Spur 23, A12 - Spur 25) und laden Sie die PCR-Produkte von Zeile B der PCR - Platte in den geradzahligen Spuren der 1. Zeile des Gels (B1 - Spur 4, A2 - Spur 6 ...... B11 - Spur 24, B12 - Spur 26).
- Laden Sie die PCR-Produkte von Zeile C und D der PCR-Platte das gleiche Muster wie oben beschrieben, mit der Zeile C in die ungeraden Bahnen und Reihe D in den geradzahligen Spuren. Wiederholen Sie diesen Lademuster, bis jede Vertiefung der PCR-Platte auf das Gel geladen wird. DNA-Ladepuffer ist nicht erforderlich, um dieses Gel zu laden.
- Legen Sie das Gel in den Lauftank und füllen Sie den Tank mit TAE-Laufpuffer, bis das Gel eingetaucht ist. Laufen bei 100 V für 30 min undunter Verwendung eines UV-Transilluminator sichtbar zu machen. Siehe 1B für ein Beispiel - Gel.
Hinweis: Die PCR-Produkte können bei -20 ° C für mehrere Wochen vor der Transfektion gelagert werden
3. 96-Well-Transfection
- Verwenden Sie die T. brucei prozyklischen Form Zelllinie SMOXP9 10 für dieses Verfahren und wachsen in SDM-79 - Medium , das 10% FCS 10.
- Verwenden 1 x 10 7 Zellen pro Transfektion (insgesamt 1,1 x 10 9 Zellen) , wenn auf dem N - Terminus des Proteins und 2 x 10 7 Zellen pro Transfektion (insgesamt 2,2 × 10 9 Zellen) etikettieren , wenn am C - Terminus Tagging von das Protein. Aufrechterhaltung der Zellen in mid-log (1,2 x 10 & sup6 ; -1 · 10 & sup7 ; Zellen / ml) für einige Tage vor der Transfektion geerntet und die Zellen für die Transfektion in einer Dichte von 5-8 x 10 6 Zellen / ml.
- Erlauben frozen PCR-Produkte für die Transfektion bei Raumtemperatur auftauen.
- Zählen von Zellen unter Verwendung eines Hämocytometer oder automatische Zellzähler.
- Pellet erforderliche Anzahl von Zellen in mehrere 50 ml-Röhrchen bei 800 xg für 10 min.
- Nach Zentrifugation discard Überstand und Resuspendieren der Zellen in 10 ml modifizierte cytomix (0,8 mM EGTA, 24 mM KCl, 0,15 mM CaCl 2, 10 mM Kaliumphosphatpuffer pH 7,6, 25 mM HEPES-KOH pH 7,6, 2,6 mM MgCl 2, 0,5% (w / v) Glucose, 100 ug / ml BSA, 1 mM Hypoxanthin, 144 mM Saccharose) pro Röhrchen. Übertragen Sie alle Zellenlösungen zu einem einzigen Rohr und drehen wieder bei 800 × g für 10 min.
- Nach dem Zentrifugieren der Überstand verwerfen und die Zellen in 23 ml modifizierter cytomix für eine Endkonzentration von 5 x 10 7 Zellen / ml resuspendieren.
- Während Zellen drehen, 1 ml SDM-79-Medium zu jeder Vertiefung von 4 x 24-well Gewebekulturplatten. Beschriften Sie die Platten AD und zeichnen einen Ring um gut A1 auf jeder Platte in Permanentmarker Plattenorientierung zu helfen.
- Schließen Sie den Plattenhandhabungs zum -Elektroporator. Auf der electroporator Einheit eingestellt, die Spannung auf 1500 V, bis 500 msec die Impulslänge auf 100 & mgr; s und die Anzahl der Impulse bis 12 und des Impulsintervalls. Auf der Platte Handler, stellen Sie die Pulszahl auf 1. Dadurch wird sichergestellt, dass die -Elektroporator einen einzigen Impuls zu jeder der 12 Spalten der Elektroporation Platte gelten.
- Unter Verwendung eines P200 12-Kanal-Mehrkanalpipette, übertragen Sie die PCR-Reaktionen aus der PCR-Platte auf den 96-Loch-Einweg-Elektroporation Platten, 4 mm Spalt.
- Wenn die Zellen versponnen und werden bis zu einer Endkonzentration von 5 x 10 7 Zellen / ml (Schritt 3.7) resuspendiert, Transfer in ein Reaktionsmittelreservoir. Je 200 ul der Zelllösung in jede Vertiefung der Elektroporation Platte mit einer P200 12-Kanal-Multikanal-Pipette, mit PCR-Produkt durch Pipettieren mischen.
- Mit Gewebe entfernen Sie alle Tröpfchen von der Oberseite der Platte um Kurzschlüsse zu vermeiden.
- Tragen Sie die Dichtungsfolie in der Platte Verpackung an die Spitze der Elektroporation Platte, pos bereitgestelltitioning es, um die Löcher für die Elektroden an der Oberseite und Unterseite jeder Spalte freigelegt zu lassen. Vermeiden Sie die angesprochenen Punkte Abdeckung verwendet, um den Film zu führen.
- Legen Sie die Elektroporation Platte in den Plattenhandhabungs und den Deckel schließen. Drücken Sie 'Pulse' auf der -Elektroporator Einheit.
- Nach der Elektroporation übertragen schnell die Zellen von der 96-Well-Platte Elektroporation auf die 4 x 24-well Gewebekulturplatten. Um die Zellen zu übertragen, verwenden Sie einen P200 12-Kanal-Mehrkanalpipette mit jeder anderen Spitze fehlt, so dass die restlichen 6 Tipps Linie mit den sechs Reihen auf einer 24-Well-Gewebekulturplatte. Übertragen Sie die elektroporierten Zellen in den 96-Well - Platten electroporations in vordefinierten Muster auf die 24-Well - Gewebekulturplatten (2A).
- Bereiten Sie die P200 Pipettenspitzen - für jede 96-Well-Transfektion 2 Boxen mit 96 Spitzen mit jeder anderen Spalte entfernt vorzubereiten.
- Transfer Brunnen A1 / A3 / A5 / A7 / A9 / A11 auf der 96-Loch-Elektroporation PlatteEine Reihe A der 24-Well-Gewebekulturplatten Platte, B1 / B3 / B5 / B7 / B9 / B11 in Reihe B, C1 / C3 / C5 / C7 / C9 / C11 in Reihe C und D1 / D3 / D5 / D7 / D9 / D11 in Reihe D. nach der Übertragung eines jeden Satzes von 6 Vertiefungen von der 96-Well-Platte mit der 24-Well-Platte werden die Vertiefungen in der 96-Well-Platte dann mit Medien aus den entsprechenden Vertiefungen in der 24- gewaschen Well-Platte und die Waschlösung wird dann zurück in die Vertiefungen in der Platte mit 24 Vertiefungen überführt.
- Transfer Brunnen E1 / E3 / E5 / E7 / E9 / E11 auf der 96-Loch-Elektroporation Platte bis 24-Well-Platte B Reihe A, F1 / F3 / F5 / F7 / F9 / F11 in Reihe B, G1 / G3 / G5 / G7 / G9 / G11 in Zeile C und H1 / H3 / H5 / H7 / H9 / H11 in Reihe D. nach der Übertragung eines jeden Satzes von 6 Vertiefungen von der 96-Well-Platte mit der 24-Well-Platte, die Vertiefungen in der 96 -Wanne Platte werden dann mit Medien aus den entsprechenden Vertiefungen in der Platte mit 24 Vertiefungen gewaschen und die Waschlösung wird dann zurück in die Vertiefungen in der Platte mit 24 Vertiefungen überführt.
- Transfer Brunnen A2 / A4 / A6 / A8 / A10 / A12 auf der 96-Loch-Elektroporation Platte bis 24-Well-Platte C Reihe A, B2 / B4 / B6 / B8 / B10 / B12 in Reihe B, C2 / C4 / C6 / C8 / C10 / C12 in Reihe C und D2 / D4 / D6 / D8 / D10 / D12 in Reihe D. Nach der Übertragung eines jeden Satzes von sechs Brunnen aus dem 96 -Wanne Platte an der 24-Well-Platte, die Vertiefungen in der Platte mit 96 Vertiefungen werden dann mit Medien aus den entsprechenden Vertiefungen in der Platte mit 24 Vertiefungen gewaschen und die Waschlösung wird dann zurück in die Vertiefungen in der Platte mit 24 Vertiefungen überführt.
- Transfer Brunnen E2 / E4 / E6 / E8 / E10 / E12 auf der 96-Loch-Elektroporation Platte bis 24-Well-Platte D Reihe A, F2 / F4 / F6 / F8 / F10 / F12 in Reihe B, G2 / G4 / G6 / G8 / G10 / G12 in Reihe C und H2 / H4 / H6 / H8 / H10 / H12 in Reihe D. nach der Übertragung eines jeden Satzes von 6 Vertiefungen von der 96-Well-Platte mit der 24-Well-Platte, die Vertiefungen in der 96 -Wanne Platte werden dann mit Medien aus den entsprechenden Vertiefungen in der Platte mit 24 Vertiefungen gewaschen und die Waschlösung wird dann zurück in die Vertiefungen in der Platte mit 24 Vertiefungen überführt.
- Unter Verwendung eines invertierten Phasenkontrastmikroskop untersuchen 1 gut aus jeder Spalte, die Zellen zu überprüfen. Es wird mit einem eine Mischung aus lebenden und toten Zellen seinbeträchtliche Menge an Zelltrümmern. Lebende Zellen können von toten Zellen unterschieden werden, weil sie unter dem Phasenkontrastmikroskopie hell sein wird und sich bewegen.
- Legen Sie die 24-Well-Platten in einem sauberen, Kunststoff-Box mit einem Deckel, der eine dichte Abdichtung bildet. Legen Sie die Box in einem 28 ° C Inkubator.
- Zwischen 6-8 Stunden später 1 ml SDM-79 mit FCS 10% gut 2x selektive Antibiotikum jeweils. In unserer Erfahrung, wo Zellinien die Gene am N-Terminus markiert sind, sind widerstandsfähig gegen höhere Konzentrationen des selektiven Arzneimittels. Daher erfordert / ml auf eine Endkonzentration von 20 ug verwendet Blasticidin als Beispiel am N-Terminus Tagging am C-Terminus Blasticidin und Etikettieren in einer Endkonzentration von 10 ug erfordert / ml Blasticidin.
Hinweis: Transgene Zelllinien 9-10 Tage nach der Transfektion sichtbar werden. - Passage mindestens zweimal in frischem Arzneimittel- und Medien vor der Analyse. 2B zeigt die Ergebnisse aus einem typischen 96-well transfection für 96 verschiedene Proteine am N-Terminus etikettieren. Beurteilen Sie jede Vertiefung durch Phasenkontrastmikroskopie zu bestimmen, ob es gesund enthält, arzneimittelresistenten Zellen. Eine gesunde und enthält überwiegend (<95%) hochaktive Zellen, die in der Phase Vertrag Mikroskopie hell sind. Anschließende Analyse durch Epifluoreszenzmikroskopie oder Western-Blot ist erforderlich, um festzustellen, ob das Protein erfolgreich markiert ist.
Ergebnisse
In dieser repräsentativen Transfektion wurden die Primer mit der TagIt Perl - Skript 9 und synthetisiert kommerziell entwickelt. Die 96-Well - PCR wurde wie beschrieben (1A) durchgeführt und validiert; in diesem Beispiel 95/96 PCRs erfolgreich waren (1B). Nach unserer Erfahrung Wiederholen fehlgeschlagen Reaktionen entweder mit den ursprünglichen Primern oder neu synthetisierten Primer nicht in erfolgreicher PCR führt.
Die Amplikons wurden in 96-well Platten transferiert Elektroporation zur Elektroporation (2A) und dann in 24-Well - Kulturplatten zur Gewinnung und Selektion transferiert. Nach ausreichender Zeit die Auswahl stattfinden zu lassen, wurden die Vertiefungen für das Überleben vor der weiteren Analyse erzielt. In diesem Beispiel 88/96 Brunnen (92%) waren nach 15 Tagen Selektion positiv.

Abbildung 1: Validierung von langer Primer PCR (A) Muster für die Übertragung von PCR - Produkte von 96-Well - PCR - Platte auf dem Agarose - Gel für die Validierung von PCR.. (B) Ein repräsentativer 96-Well - PCR - Validierung Gel für 95 Gene auf dem N - Terminus - Tagging und 1 Amplikon keine 5 'Targeting Homologie enthalten , als negative Kontrolle in die Transfektionseffizienz zu wirken. Die Proben werden geladen auf ein 1% (w / v) Agarose - Gel und für 30 Minuten bei 100 V aufgelöst. Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.
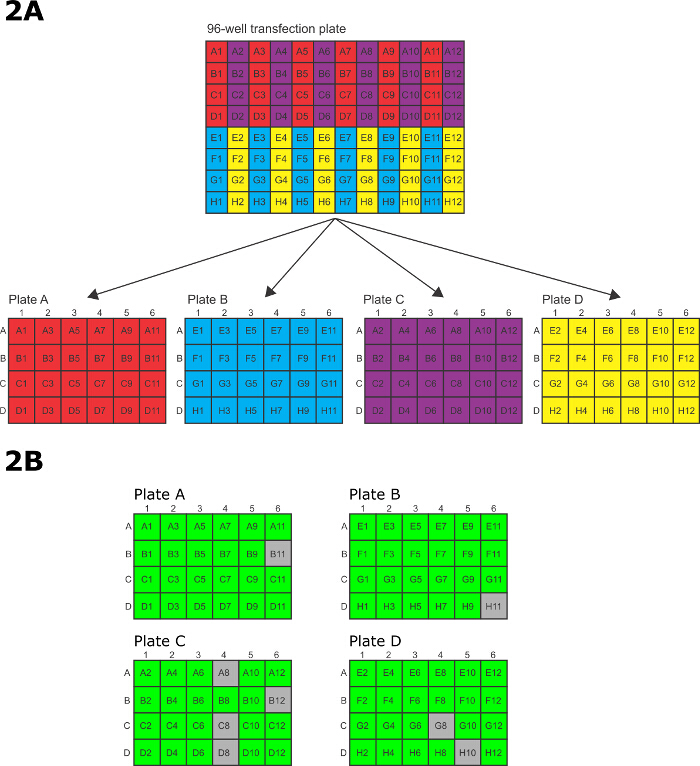
Abbildung 2: 96-Well - Elektroporation und Selektion (A) Muster für elektroporierten Zellen von 96-Well - Elektroporation plat übertragen.E 24-Well-Gewebekulturplatten. (B) Ein Schema , das eine typische Live / Dead - Scoring nach 15 Tagen Selektion in 24-Well - Platten zeigt. Grün steht für eine erfolgreiche Transfektion, grau stellt ein gut mit nur tote Zellen. In diesem Beispiel 88/96 (92%) von Wells enthielten erfolgreich transfizierten Parasiten. Bitte hier klicken , um eine größere Version dieser Figur zu sehen.
Diskussion
Dramatische Verbesserungen in der Empfindlichkeit der Proteomik und Transkriptomik Methoden in den letzten 5-10 Jahren auf Tausenden von Genen und ihren Produkten wertvolle Daten zur Verfügung gestellt. Allerdings sind die Werkzeuge die Funktion dieser Proteine Adresse haben nicht Schritt gehalten.
Tagging ein Protein erleichtert zahlreiche Versuche, seine Funktion zu bestimmen. Zum Beispiel kann ein Protein in einer Vielzahl von verschiedenen Farben zu einem fluoreszierenden Protein fusioniert werden Studien Lokalisation und Co-Lokalisierung zu erleichtern. Stichworte für die Elektronenmikroskopie entwickelt, wie APEX2 oder miniSOG 11,12 erlauben ultrastrukturelle Lokalisierung des markierten Proteins. Tags für die Biochemie, wie dem TAP - Tag und ProtC-TEV-ProtA (PTP) -Tag 7,13 Reinigung von Komplexen ermöglichen , mit dem Protein zur Identifizierung von Bindungspartnern oder in vitro biochemische Assays verbunden.
Die spezifischen Schritte, die für den Erfolg der Protoc kritischol, sind: der Einbau von DMSO in die PCR Master Mix 1, Gefrieren des PCR Master Mix 1 vor der Zugabe des Master Mix 2, die Verwendung des handelsüblichen Polymerase in Master Mix 2 und der Modifikation des cytomix Elektroporationspuffer. Nach unserer Erfahrung ist es notwendig, die doppelte Anzahl von Zellen für die C-terminalen Markierungs Transfektionen wie für N-Terminal-Tagging Transfektionen verwendet werden, um einen ähnlichen Anteil an positiven Vertiefungen zu erreichen. Daher sollten alle Schritte wie beschrieben durchgeführt.
Diese Technik ist wahrscheinlich nur erfolgreich sein, wenn das Insekt prozyklischen Form Trypanosomen transfizieren. Blutstrom Formen haben eine geringere Transfektionseffizienz 14, zudem sind sie wahrscheinlich aufgrund von Dichteabhängigen Toxizität bei der Auswahl zu sterben, die mit dem selektiven Medikament nicht in Beziehung steht. Daher stellt unsere bisherigen Protokoll die aktuelle beste Technologie für 9 von Blutformen Trypanosomen - Tagging. Es ist auch wahrscheinlich, dass die transfection Effizienz hängt von der spezifischen Trypanosomen-Isolat variieren. Dieses Protokoll wurde mit 927 SMOX prozyklischen Formen optimiert - andere Stämme zusätzliche Optimierung erfordern. Maßnahmen, welche die Erfolgswahrscheinlichkeit erhöhen können, umfassen: die Menge des PCR-Amplikons, Erhöhung der Anzahl von Zellen in der Transfektionseffizienz eingeschlossen.
Wir stellen eine Methode, in der Hunderte von Proteinen können parallel markiert werden. Dies wird, groß angelegte Studien über die Lokalisierung und Interaktion zu erleichtern, die Ergänzung bestehender großer Datensätze und wertvolle Informationen für die Allgemeinheit erbringen. Primer-Vorlagen sind auf Anfrage erhältlich und eine Liste von Plasmiden Vorlagen finden Sie unter: http://www.sdeanresearch.com/
Offenlegungen
The authors have nothing to disclose.
Danksagungen
SD was supported by a Sir Henry Wellcome Fellowship [092201/Z/10/Z]. This work was funded by Wellcome Trust grants [WT066839MA][104627/Z/14/Z][108445/Z/15/Z] to Professor Keith Gull. We would like to thank Professor Keith Gull for helpful conversations and insights.
Materialien
| Name | Company | Catalog Number | Comments |
| Oligonucleotide | Life technologies | na | desalt only (do not use additional purification) |
| DMSO | Roche | Included with the Expand HiFi pack | Must be PCR grade |
| dNTP | any reputable | ||
| Expand HiFi polymerase | Roche | 11759078001 | |
| BTX ECM830 | Harvard Apparatus | Electroporator | |
| HT-200 plate handler | Harvard Apparatus | HT-200 | Handles the 96-well eletroplates |
| MOS 96 ELECTROPLATE 4 mm | Harvard Apparatus | 45-0452 | Electroplate for the 96-well electroporation |
| Blasticidin S Hydrochloride | Melford | 3513-03-9 | |
| Hygromycin b Gold | Invivogen | ant-hg-1 | |
| Phleomycin | Melford | P0187 | |
| 225 cm TC Flask, canted neck, phenolic cap | Appletonwoods | BC006 | |
| 24 well culture plates | Appletonwoods | BC017 | |
| Eppendorf® PCR Cooler, iceless cold storage system for 96 well plates and PCR tubes | Sigma Aldrich | Z606634-1EA | |
| 96-well Multiply® PCR plate with lateral skirt | Sarstedt | 72.1979.203 |
Referenzen
- Kelly, S., Reed, J., et al. Functional genomics in Trypanosoma brucei: a collection of vectors for the expression of tagged proteins from endogenous and ectopic gene loci. Mol Biochem Parasitol. 154 (1), 103-109 (2007).
- Wirtz, L. E., Leal, S., Ochatt, C., Cross, G. A. M. A tightly regulated inducible expression system for conditional gene knock-outs and dominant-negative genetics in Trypanosoma brucei. Mol Biochem Parasitol. 99 (1), 89-101 (1999).
- Shi, H., Djikeng, A., Mark, T., Wirtz, L. E., Tschudi, C., Ullu, E. Genetic interference in Trypanosoma brucei by heritable and inducible double-stranded RNA. RNA. 6 (7), New York, NY. 1069-1076 (2000).
- Alsford, S., Turner, D. J., et al. High-throughput phenotyping using parallel sequencing of RNA interference targets in the African trypanosome. Genome Res. 21 (6), 915-924 (2011).
- Alsford, S., Eckert, S., et al. High-throughput decoding of antitrypanosomal drug efficacy and resistance. Nature. 482 (7384), 232-236 (2012).
- Mony, B. M., MacGregor, P., et al. Genome-wide dissection of the quorum sensing signalling pathway in Trypanosoma brucei. Nature. 505 (7485), 681-685 (2014).
- Rigaut, G., Shevchenko, A., Rutz, B., Wilm, M., Mann, M., Séraphin, B. A generic protein purification method for protein complex characterization and proteome exploration : Abstract Nature Biotechnology. Nature Biotechnol. 17 (10), 1030-1032 (1999).
- Los, G. V., Encell, L. P., et al. HaloTag: A Novel Protein Labeling Technology for Cell Imaging and Protein Analysis. ACS Chem Biol. 3 (6), 373-382 (2008).
- Dean, S., Sunter, J. D., Wheeler, R. J., Hodkinson, I., Gluenz, E., Gull, K. A toolkit enabling efficient, scalable and reproducible gene tagging in trypanosomatids. Open biol. 5 (1), 140197(2015).
- Poon, S. K., Peacock, L., Gibson, W., Gull, K., Kelly, S. A modular and optimized single marker system for generating Trypanosoma brucei cell lines expressing T7 RNA polymerase and the tetracycline repressor. Open biol. 2 (2), 110037(2012).
- Lam, S. S., Martell, J. D., et al. Directed evolution of APEX2 for electron microscopy and proximity labeling. Nature meth. 12 (1), 51-54 (2014).
- Shu, X., Lev-Ram, V., Deerinck, T. J., Qi, Y., Ramko, E. B. A genetically encoded tag for correlated light and electron microscopy of intact cells, tissues, and organisms. PLoS biology. , (2011).
- Schimanski, B., Nguyen, T. N., Günzl, A. Highly efficient tandem affinity purification of trypanosome protein complexes based on a novel epitope combination. Eukaryotic Cell. , (2005).
- Burkard, G., Fragoso, C. M., Roditi, I. Highly efficient stable transformation of bloodstream forms of Trypanosoma brucei. Mol Biochem Parasitol. 153 (2), 220-223 (2007).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten