Method Article
High-throughput di Gene Tagging in
In questo articolo
Riepilogo
Addition of a tag to a protein is a powerful way of gaining insight into its function. Here, we describe a protocol to endogenously tag hundreds of Trypanosoma brucei proteins in parallel such that genome scale tagging is achievable.
Abstract
Improvements in mass spectrometry, sequencing and bioinformatics have generated large datasets of potentially interesting genes. Tagging these proteins can give insights into their function by determining their localization within the cell and enabling interaction partner identification. We recently published a fast and scalable method to generate Trypanosoma brucei cell lines that express a tagged protein from the endogenous locus. The method was based on a plasmid we generated that, when coupled with long primer PCR, can be used to modify a gene to encode a protein tagged at either terminus. This allows the tagging of dozens of trypanosome proteins in parallel, facilitating the large-scale validation of candidate genes of interest. This system can be used to tag proteins for localization (using a fluorescent protein, epitope tag or electron microscopy tag) or biochemistry (using tags for purification, such as the TAP (tandem affinity purification) tag). Here, we describe a protocol to perform the long primer PCR and the electroporation in 96-well plates, with the recovery and selection of transgenic trypanosomes occurring in 24-well plates. With this workflow, hundreds of proteins can be tagged in parallel; this is an order of magnitude improvement to our previous protocol and genome scale tagging is now possible.
Introduzione
Trypanosoma brucei è un protozoo parassita che causa trypanosomasis africana umana e nagana nei bovini. T. brucei è un organismo ideale per l'analisi della funzione proteica grazie alla combinazione di un genoma di alta qualità, numerose proteomica e trascrittomica set di dati e strumenti molecolari ben sviluppati 1-3. I progressi nella proteomica e il sequenziamento hanno portato a grandi serie di dati che evidenziano potenzialmente geni interessanti 4-6; Tuttavia, molti geni hanno informazioni minime ad essi associati nei database esistenti. Vi è quindi la necessità di un metodo di alta produttività per aiutare caratterizzazione funzionale della proteina.
Espressione di una proteina marcata può dare una molteplicità di intuizioni in funzione di una proteina. Ad esempio, una proteina marcata con una proteina fluorescente o epitopo può essere localizzato mediante microscopia a fluorescenza, che fornisce informazioni su dove protein potrebbe essere esercitando il suo effetto biologico. In alternativa, una proteina contrassegnati con un TAP 7, 8 o HaloTag sua etichetta può essere purificato per saggi biochimici e l'identificazione dei suoi partner di interazione.
Recentemente abbiamo sviluppato una metodologia di codifica robusta per T. brucei 9. Questo era lungo Primer PCR per generare il DNA per trasfezione e ha permesso la marcatura di decine di proteine in parallelo - un miglioramento importante di protocolli esistenti. Ora abbiamo migliorato la scalabilità di questo protocollo in forme prociclico da un ordine di grandezza. Qui, vi presentiamo il nostro metodo in cui eseguiamo la PCR e trasfezione in piastre a 96 pozzetti, con il recupero e la selezione che si verificano in piastre da 24 pozzetti. Come centinaia di proteine possono essere contrassegnati in parallelo questo metodo fornisce un metodo conveniente e fattibile per la codifica l'intero genoma trypanosome.
Protocollo
1. 96 pozzetti lungo Primer PCR
- Pre-freeze 96 pozzetti PCR più fresco in congelatore -80 ° C.
- In un tubo da 10 ml, preparare Master Mix A: 2.100 ml DDH 2 O, 105 ml PCR grado DMSO, 105 ml 10 mM dNTP, 105 ml modello pPOT (25 ng / ml) 9, per un volume totale di 2.415 ml per piastra .
- Aliquota 23 ml in ciascun pozzetto di una piastra a 96 pozzetti PCR.
- Aggiungere 2 ml di 100 micron pool primer per ogni caricato bene con un P20 12 canali pipetta multicanale. Sigillare con la pellicola, piastra posto sul radiatore PCR pre-raffreddata e permettere di congelare per un minimo di 20 minuti a -80 ° C.
- In un tubo da 10 ml, preparare Master Mix B: 2.048 ml DDH 2 O, 525 microlitri di buffer 10x, 52,5 ml polimerasi per un volume totale di 2.626 ml per piastra.
- Rimuovere la piastra e PCR più fresco dal -80 ° C freezer.
- Con la piastra ancora sul radiatore PCR, aggiungere 25 microlitri Master Mix B in cima alla congelaton Master Mix A per un volume di reazione totale di 50 microlitri. Questo è il tempo critico, e dovrebbe essere completato prima di Mix A può scongelare. Guarnizione della piastra con la pellicola.
- Impostare termociclatore come segue: 94 ° C per 10 min, quindi 30 cicli di 94 ° C per 15 sec, 65 ° C per 30 sec, 72 ° C per 2 min seguita da un periodo di estensione finale di 72 ° C per 7 min .
- Caricare la piastra PCR dopo il blocco ha raggiunto 94 ° C.
2. Convalida dei 96 pozzetti lungo Primer PCR
- Cast un'ampia 1% (w / v) di gel di agarosio in Tris acetato EDTA (TAE) tampone di corsa con 4 x 28 pozzetti pettini per dare un gel con 4 file di pozzi. Allineare i denti supplenti del pettine con le punte di una pipetta multicanale a 12 canali. immergere con attenzione il gel in TAE tampone di corsa dopo le corsie vengono caricati.
- Carico 1 kb scala DNA nel 1 ° e il 28 ° e di ogni riga.
- Utilizzando un P20 12 canali multicanale pipetta, trasferire 2 ml di PCR dir prodottoectly per ogni corsia di gel (Figura 1A). Fare questo rapidamente per ridurre la possibilità di contaminare gli ampliconi.
- Caricare i prodotti di PCR di riga A della piastra PCR nelle corsie numerate dispari della prima fila del gel (A1 - corsia 3, A2 - corsia 5 ...... A11 - corsia 23, A12 - corsia 25) e caricare i prodotti di PCR da riga B della piastra PCR nei vicoli numerazione pari del 1 ° fila del gel (B1 - corsia 4, A2 - corsia 6 ...... B11 - corsia 24, B12 - corsia 26).
- Caricare i prodotti di PCR di fila C e D della piastra PCR utilizzando lo stesso modello come descritto sopra, con fila C in corsie numerate e le fila D dispari nelle corsie pari. Continuare questo modello loading finché ogni pozzetto della piastra PCR viene caricato sul gel. buffer di DNA di caricamento non è tenuto a caricare questo gel.
- Posizionare il gel nel serbatoio esecuzione e riempire il serbatoio con TAE tampone di corsa fino a quando il gel viene sommerso. Eseguire a 100 V per 30 minutivisualizzare utilizzando un transilluminatore UV. Vedi Figura 1B per un gel esempio.
Nota: PCR prodotti possono essere conservati a -20 ° C per diverse settimane prima trasfezione
3. Trasfezione 96 pozzetti
- Utilizzare il T. brucei prociclico linea di cellule forma SMOXP9 10 per questa procedura e crescere in SDM-79 supporti contenenti il 10% di FCS 10.
- Utilizzare 1 x 10 7 cellule per trasfezione (totale di 1,1 x 10 9 cellule) quando codifica sul terminale N della proteina e 2 x 10 7 cellule per trasfezione (totale di 2,2 x 10 9 cellule) quando codifica sul terminale C di la proteina. Mantenere le cellule in mid-log (1,2 x 10 6 -1 x 10 7 cellule / ml) per diversi giorni prima della trasfezione e raccogliere le cellule per trasfezione ad una densità di 5-8 x 10 6 cellule / ml.
- Lasciare prodotti di PCR congelati per trasfezione per scongelare a temperatura ambiente.
- Contare le cellule utilizzando un emeocytometer o contatore di cellule automatica.
- Pellet numero di celle in più provette da 50 ml a 800 xg per 10 min.
- Dopo la centrifugazione Gettare il surnatante e risospendere le cellule in 10 ml cytomix modificato (0,8 mm EGTA, 24 mm KCl, 0,15 mm CaCl 2, 10 mM fosfato di potassio tampone pH7.6, 25 mM HEPES-KOH pH7.6, 2,6 mm MgCl 2, 0.5% (w / v) di glucosio, 100 ug / ml BSA, 1 mM ipoxantina, 144 mM saccarosio) per provetta. Trasferire tutte le soluzioni cellulari ad un singolo tubo e far girare nuovamente a 800 xg per 10 min.
- Dopo centrifugazione, scartare il surnatante e risospendere le cellule in 23 ml di cytomix modificato per una concentrazione finale di 5 x 10 7 cellule / ml.
- Mentre le cellule sono filatura, aggiungere 1 ml di SDM-79 media per ciascun pozzetto di piastre di coltura tissutale 4 x 24 pozzetti. Etichettare il piastre dC e disegnare un anello attorno pozzetto A1 su ogni piatto in pennarello indelebile per aiutare l'orientamento piatto.
- Collegare il gestore piastra al electroporator. Sulla electroporaUnità tor impostare la tensione a 1500 V, la lunghezza dell'impulso di 100 microsec e il numero di impulsi a 12 e l'intervallo di impulso per 500 msec. Sul conduttore piatto, impostare il conteggio degli impulsi a 1. Questo assicura che tale electroporator applicherà un singolo impulso a ciascuna delle colonne 12 della piastra elettroporazione.
- Utilizzando un P200 a 12 canali multicanale pipetta, trasferire le reazioni di PCR dalla piastra PCR per le piastre di elettroporazione 96 pozzetti usa e getta, Gap 4 mm.
- Quando le cellule sono state filate e sono risospesi alla concentrazione finale di 5 x 10 7 cellule / ml (Passo 3.7), trasferire ad un serbatoio di reagenti. Pipettare 200 ml di soluzione di cellule in ciascun pozzetto della piastra elettroporazione utilizzando un P200 a 12 canali pipetta multicanale, mescolare con il prodotto PCR pipettando.
- Usando tessuto rimuovere eventuali goccioline dalla parte superiore della piastra per evitare cortocircuiti.
- Applicare la pellicola sigillante fornito nella confezione piastra alla sommità della piastra elettroporazione, positioning in modo da lasciare i fori per gli elettrodi scoperti nella parte superiore e inferiore di ogni colonna. Evitare di coprire i punti sollevati utilizzati per guidare il film.
- Caricare la piastra di elettroporazione nel gestore piastra e chiudere il coperchio. Premere 'Pulse' sull'unità electroporator.
- Dopo l'elettroporazione, trasferire rapidamente le cellule dalla piastra elettroporazione 96 pozzetti alle piastre di coltura tissutale 4 x 24 pozzetti. Per trasferire le cellule, usare un P200 a 12 canali multicanale pipetta con ogni altro punta mancante in modo tale che i restanti 6 punte allineano con i 6 file su un 24-pozzetti di coltura tissutale. Trasferire le cellule elettroporate nei electroporations piastre a 96 pozzetti nel modello predefinito per le piastre di coltura tissutale 24 pozzetti (Figura 2A).
- Preparare le punte per pipette P200 - per ogni 96 pozzetti trasfezione preparare 2 scatole di 96 punte con ogni altra colonna rimosso.
- pozzi di trasferimento A1 / A3 / A5 / A7 / A9 / A11 sulla piastra elettroporazione 96 pozzetti apiatto Una riga A delle piastre di coltura di tessuto da 24 pozzetti, B1 / B3 / B5 / B7 / B9 / B11 in riga B, C1 / C3 / C5 / C7 / C9 / C11 in riga C e D1 / D3 / D5 / D7 / D9 / D11 in fila D. Dopo il trasferimento di ogni serie di 6 pozzi dalla piastra a 96 pozzetti per la 24-pozzetti, i pozzi della piastra a 96 pozzetti vengono poi lavate con supporti da rispettivi pozzetti in 24- pozzetti e il lavaggio viene poi trasferito di nuovo ai pozzetti nella piastra 24 pozzetti.
- Trasferimento pozzi E1 / E3 / E5 / E7 / E9 / E11 sul piatto elettroporazione 96 pozzetti a 24 pozzetti B riga A, F1 / F3 / F5 / F7 / F9 / F11 in riga B, G1 / G3 / G5 / G7 / G9 / G11 in riga C e H1 / H3 / H5 / H7 / H9 / H11 in fila D. Dopo il trasferimento di ogni serie di 6 pozzi dalla piastra a 96 pozzetti per la 24-pozzetti, i pozzi in 96 piastra -well vengono poi lavati con supporti da rispettivi pozzetti nella piastra 24 pozzetti ed il lavaggio viene poi trasferito nuovamente ai pozzetti nella piastra 24 pozzetti.
- pozzi di trasferimento A2 / A4 / A6 / A8 / A10 / A12 sulla piastra elettroporazione 96 pozzetti a 24 pozzetti C riga A, B2 / B4 / B6 / B8 / B10 / B12 in riga B, C2 / C4 / C6 / C8 / C10 / C12 in riga C e D2 / D4 / D6 / D8 / D10 / D12 in fila D. Dopo il trasferimento di ogni serie di 6 pozzi dal 96 piastra -well alla piastra 24 pozzetti, i pozzi della piastra a 96 pozzetti vengono poi lavati con supporti da rispettivi pozzetti nella piastra 24 pozzetti ed il lavaggio viene poi trasferito nuovamente ai pozzetti nella piastra 24 pozzetti.
- pozzi di trasferimento E2 / E4 / E6 / E8 / E10 / E12 sul piatto elettroporazione 96 pozzetti a 24 pozzetti D riga A, F2 / F4 / F6 / F8 / F10 / F12 in riga B, G2 / G4 / G6 / G8 / G10 / G12 in riga C e H2 / H4 / H6 / H8 / H10 / H12 in fila D. Dopo il trasferimento di ogni serie di 6 pozzi dalla piastra a 96 pozzetti per la 24-pozzetti, i pozzi del 96 piastra -well vengono poi lavati con supporti da rispettivi pozzetti nella piastra 24 pozzetti ed il lavaggio viene poi trasferito nuovamente ai pozzetti nella piastra 24 pozzetti.
- Utilizzando un microscopio a contrasto di fase invertito, esaminare 1 e da ogni colonna per controllare le cellule. Ci sarà una miscela di cellule vive e morte connotevole quantità di detriti cellulari. cellule vive possono essere distinte dalle cellule morte perché saranno luminoso al microscopio a contrasto di fase e saranno in movimento.
- Posizionare le piastre da 24 pozzetti in una scatola pulita, di plastica con un coperchio che forma una tenuta ermetica. Mettete la scatola in un incubatore a 28 °.
- Tra 6-8 ore più tardi, aggiungere 1 ml di SDM-79 con 10% FCS contenente antibiotico selettivo 2x a ciascun pozzetto. Nella nostra esperienza, le linee cellulari in cui i geni sono targhetta su terminale N sono resistenti alle alte concentrazioni di farmaco selettivo. Pertanto, utilizzando blasticidin come esempio, codifica sul terminale N richiede una concentrazione finale di 20 ug / ml blasticidin e codifica il terminale C richiede una concentrazione finale di 10 ug / ml blasticidin.
Nota: linee cellulari transgeniche saranno visibili 9-10 giorni dopo la trasfezione. - Passaggio almeno due volte nel farmaco fresco e supporti prima dell'analisi. La Figura 2B mostra i risultati di un tipico Transfe 96 pozzettiction per la codifica di 96 diverse proteine sul terminale N. Valutare ogni bene al microscopio a contrasto di fase per determinare se contiene cellule sane, resistenti ai farmaci. A ben sano contiene prevalentemente (<95%), le cellule altamente attivi che sono luminose nel campo della microscopia contratto di fase. L'analisi successiva mediante microscopia in epifluorescenza o Western Blot è necessaria per determinare se la proteina è stato etichettato con successo.
Risultati
In questo trasfezione rappresentante, i primer sono stati progettati utilizzando lo script perl Tagit 9 e sintetizzati in commercio. Il 96 pozzetti PCR è stato eseguito e convalidato come descritto (Figura 1A); in questo esempio, 95/96 PCR hanno avuto successo (Figura 1B). Nella nostra esperienza, ripetendo le reazioni fallite sia con i primer originali o primer ri-sintetizzato non si traduca in una PCR di successo.
Gli ampliconi sono state trasferite in piastre da 96 pozzetti elettroporazione per elettroporazione (Figura 2A) e poi trasferiti piastre di coltura da 24 pozzetti per il recupero e la selezione. Dopo un tempo sufficiente a permettere la selezione avvenga, i pozzetti sono stati ottenuti per la sopravvivenza prima ulteriori analisi. In questo esempio, 88/96 pozzi (92%) sono risultati positivi dopo la selezione di 15 giorni.

Figura 1: convalida di lunga Primer PCR (A) del modello per il trasferimento di prodotti di PCR da 96 pozzetti PCR per il gel per la validazione della PCR.. (B) A 96 pozzetti PCR gel rappresentante convalida per la codifica 95 geni sul terminale N e 1 amplicon non contenente 5 'omologia di targeting per fungere da controllo negativo nella trasfezione. I campioni vengono caricati su un 1% (w / v) gel e risolti a 100 V per 30 min. Cliccate qui per vedere una versione più grande di questa figura.
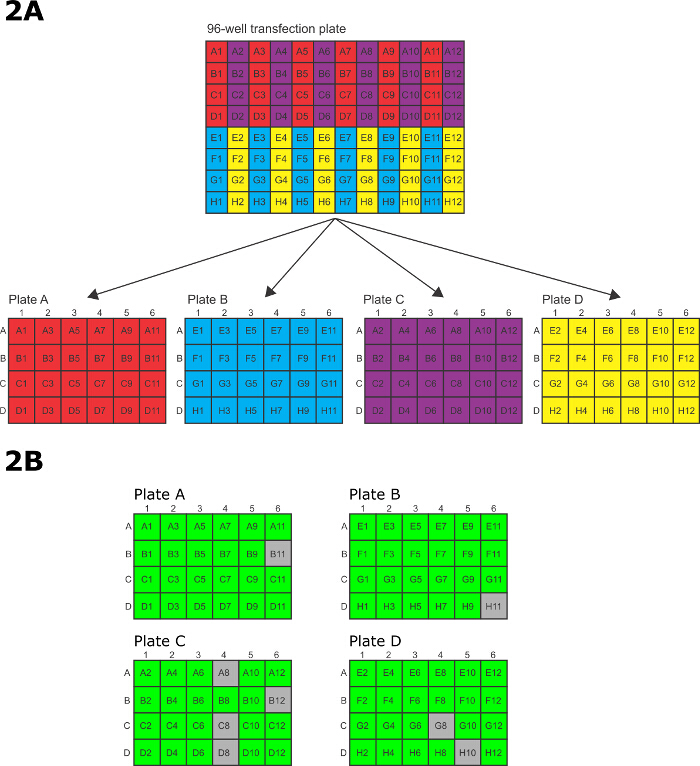
Figura 2: 96 pozzetti elettroporazione e la selezione (A) del modello per il trasferimento di cellule elettroporate da 96 pozzetti elettroporazione plat.e per 24 pozzetti piastre di coltura dei tessuti. (B) Un schematicamente una tipica competenza live / morti dopo 15 giorni di selezione in piastre da 24 pozzetti. Verde rappresenta un trasfezione successo, grigio rappresenta un pozzo con solo cellule morte. In questo esempio, 88/96 (92%) dei pozzi conteneva parassiti transfettate con successo. Si prega di cliccare qui per vedere una versione più grande di questa figura.
Discussione
notevoli miglioramenti nella sensibilità di proteomica e trascrittomica metodi negli ultimi 5-10 anni ha fornito dati preziosi su migliaia di geni e dei loro prodotti. Tuttavia, gli strumenti per affrontare la funzione di queste proteine non hanno tenuto il passo.
Tagging una proteina facilita numerosi esperimenti per determinare la sua funzione. Ad esempio, una proteina può essere fuso ad una proteina fluorescente in una varietà di colori diversi per facilitare gli studi di localizzazione e co-localizzazione. Tag sviluppato per la microscopia elettronica, come ad esempio APEX2 o miniSOG 11,12, consentire la localizzazione ultrastrutturale della proteina codificata. Parole chiave per la biochimica, come ad esempio il tag TAP, e ProtC-TEV-Prota (PTP) Indicatore 7,13 permettere la purificazione di complessi associati con la proteina per l'identificazione di partner di legame o in saggi biochimici in vitro.
I passi specifici che sono cruciali per il successo del ProtoColo sono: l'incorporazione di DMSO nella PCR Master Mix 1, congelamento della PCR Master Mix 1 prima dell'aggiunta del Master Mix 2, l'uso della polimerasi commerciale in Master Mix 2 e la modifica del buffer cytomix elettroporazione. Nella nostra esperienza, è necessario utilizzare il doppio del numero di celle per C trasfezioni terminale di codifica da per N trasfezioni terminale di codifica per ottenere una percentuale simile di pozzetti positivi. Pertanto, tutti i passaggi devono essere eseguiti come descritto.
Questa tecnica è solo probabile che sia successo quando transfecting l'insetto prociclico modulo trypanosome. Forme sangue hanno una bassa efficienza di trasfezione 14, inoltre sono probabilità di morire durante la selezione a causa della tossicità densità-dipendente che non è correlato al farmaco selettivo. Pertanto, il nostro precedente protocollo rappresenta la migliore tecnologia attuale per la codifica di tripanosomi forma sangue 9. E 'anche probabile che il transefficienza perfezione varia dipende dalla specifica isolato trypanosome. Questo protocollo è stato ottimizzato utilizzando 927 SMOX forme prociclico - altri ceppi possono richiedere ulteriore ottimizzazione. Le misure che possono aumentare la probabilità di successo includono: aumentando la quantità di PCR amplicone, aumentando il numero di cellule inclusi nella trasfezione.
Vi presentiamo un metodo in cui centinaia di proteine possono essere contrassegnati in parallelo. Ciò faciliterà studi su larga scala sulla localizzazione e l'interazione, integrando grandi set di dati esistenti e di fornire informazioni preziose per la comunità. modelli Primer sono disponibili su richiesta e un elenco di plasmidi modelli sono disponibili presso: http://www.sdeanresearch.com/
Divulgazioni
The authors have nothing to disclose.
Riconoscimenti
SD was supported by a Sir Henry Wellcome Fellowship [092201/Z/10/Z]. This work was funded by Wellcome Trust grants [WT066839MA][104627/Z/14/Z][108445/Z/15/Z] to Professor Keith Gull. We would like to thank Professor Keith Gull for helpful conversations and insights.
Materiali
| Name | Company | Catalog Number | Comments |
| Oligonucleotide | Life technologies | na | desalt only (do not use additional purification) |
| DMSO | Roche | Included with the Expand HiFi pack | Must be PCR grade |
| dNTP | any reputable | ||
| Expand HiFi polymerase | Roche | 11759078001 | |
| BTX ECM830 | Harvard Apparatus | Electroporator | |
| HT-200 plate handler | Harvard Apparatus | HT-200 | Handles the 96-well eletroplates |
| MOS 96 ELECTROPLATE 4 mm | Harvard Apparatus | 45-0452 | Electroplate for the 96-well electroporation |
| Blasticidin S Hydrochloride | Melford | 3513-03-9 | |
| Hygromycin b Gold | Invivogen | ant-hg-1 | |
| Phleomycin | Melford | P0187 | |
| 225 cm TC Flask, canted neck, phenolic cap | Appletonwoods | BC006 | |
| 24 well culture plates | Appletonwoods | BC017 | |
| Eppendorf® PCR Cooler, iceless cold storage system for 96 well plates and PCR tubes | Sigma Aldrich | Z606634-1EA | |
| 96-well Multiply® PCR plate with lateral skirt | Sarstedt | 72.1979.203 |
Riferimenti
- Kelly, S., Reed, J., et al. Functional genomics in Trypanosoma brucei: a collection of vectors for the expression of tagged proteins from endogenous and ectopic gene loci. Mol Biochem Parasitol. 154 (1), 103-109 (2007).
- Wirtz, L. E., Leal, S., Ochatt, C., Cross, G. A. M. A tightly regulated inducible expression system for conditional gene knock-outs and dominant-negative genetics in Trypanosoma brucei. Mol Biochem Parasitol. 99 (1), 89-101 (1999).
- Shi, H., Djikeng, A., Mark, T., Wirtz, L. E., Tschudi, C., Ullu, E. Genetic interference in Trypanosoma brucei by heritable and inducible double-stranded RNA. RNA. 6 (7), 1069-1076 (2000).
- Alsford, S., Turner, D. J., et al. High-throughput phenotyping using parallel sequencing of RNA interference targets in the African trypanosome. Genome Res. 21 (6), 915-924 (2011).
- Alsford, S., Eckert, S., et al. High-throughput decoding of antitrypanosomal drug efficacy and resistance. Nature. 482 (7384), 232-236 (2012).
- Mony, B. M., MacGregor, P., et al. Genome-wide dissection of the quorum sensing signalling pathway in Trypanosoma brucei. Nature. 505 (7485), 681-685 (2014).
- Rigaut, G., Shevchenko, A., Rutz, B., Wilm, M., Mann, M., Séraphin, B. A generic protein purification method for protein complex characterization and proteome exploration : Abstract Nature Biotechnology. Nature Biotechnol. 17 (10), 1030-1032 (1999).
- Los, G. V., Encell, L. P., et al. HaloTag: A Novel Protein Labeling Technology for Cell Imaging and Protein Analysis. ACS Chem Biol. 3 (6), 373-382 (2008).
- Dean, S., Sunter, J. D., Wheeler, R. J., Hodkinson, I., Gluenz, E., Gull, K. A toolkit enabling efficient, scalable and reproducible gene tagging in trypanosomatids. Open biol. 5 (1), 140197 (2015).
- Poon, S. K., Peacock, L., Gibson, W., Gull, K., Kelly, S. A modular and optimized single marker system for generating Trypanosoma brucei cell lines expressing T7 RNA polymerase and the tetracycline repressor. Open biol. 2 (2), 110037 (2012).
- Lam, S. S., Martell, J. D., et al. Directed evolution of APEX2 for electron microscopy and proximity labeling. Nature meth. 12 (1), 51-54 (2014).
- Shu, X., Lev-Ram, V., Deerinck, T. J., Qi, Y., Ramko, E. B. A genetically encoded tag for correlated light and electron microscopy of intact cells, tissues, and organisms. PLoS biology. , (2011).
- Schimanski, B., Nguyen, T. N., Günzl, A. Highly efficient tandem affinity purification of trypanosome protein complexes based on a novel epitope combination. Eukaryotic Cell. , (2005).
- Burkard, G., Fragoso, C. M., Roditi, I. Highly efficient stable transformation of bloodstream forms of Trypanosoma brucei. Mol Biochem Parasitol. 153 (2), 220-223 (2007).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon