Method Article
Ein Web-Tool für die Erstellung hochwertiger maschinenlesbare biologische Pathways
In diesem Artikel
Zusammenfassung
PathWhiz ist ein umfassendes Online-Pfad Zeichenwerkzeug für die biochemische und biologische Pfade zu erzeugen. Es nutzt öffentlich zugänglichen Datenbanken und leicht erweiterbare Paletten aus vorgezeichneten Weg Komponenten. Dieses Protokoll beschreibt, wie leicht auf neue Wege zu bauen, zu replizieren und bestehende Wege zu bearbeiten, und propagieren zuvor gezeichneten Wege zu verschiedenen Organismen.
Zusammenfassung
PathWhiz ist ein Web-Server die Erstellung von bunten, interaktive, optisch ansprechende Pfaddiagramme zu erleichtern gebaut, die in biologischen Informationen reich sind. Die Wege durch diese Online-Anwendung generiert sind maschinenlesbar und voll kompatibel mit im wesentlichen allen Web-Browsern und Betriebssystemen. Es verwendet einen speziell entwickelten, webfähigen Stoffwechselweg Zeichenoberfläche, die die Auswahl und Platzierung der verschiedenen Kombinationen von vorgezeichneten biologische oder biochemische Einheiten ermöglicht darzustellen Reaktionen, Interaktionen, Transportprozesse und Bindungsereignisse. Diese Palette von Entitäten besteht aus chemischen Verbindungen, Proteine, Nukleinsäuren, Zellmembranen, subzellulären Strukturen, Gewebe und Organe. Alle der visuellen Elemente in interaktiv eingestellt und angepasst werden können. Darüber hinaus, weil dieses Tool ein Web-Server ist, sind alle Wege und Wege Elemente öffentlich zugänglich. Diese Art von Weg "Crowd Sourcing" bedeutet, dass PathWhizenthält bereits eine große und Sammlung von zuvor gezeichneten Wege und Wege Elemente schnell wächst. Hier beschreiben wir ein Protokoll für die schnelle und einfache Erstellung neuer Wege und die Veränderung bestehender Wege. Zur weiteren Weg Bearbeitung und Erstellung zu erleichtern, enthält das Werkzeug Replikation und Vermehrung Funktionen. Die Replikationsfunktion ermöglicht bestehende Wege als Vorlagen verwendet werden, um neue Wege zu erstellen oder zu bearbeiten. Die Ausbreitungsfunktion erlaubt es, eine bestehende Weg zu nehmen und es automatisch auf verschiedene Arten ausbreiten. Pathways mit diesem Tool erstellt werden "re-Stil" in verschiedenen Formaten (KEGG-like oder Text-Buch wie), mit verschiedenen Hintergründen gefärbt, exportiert nach BioPAX, SBGN-ML, SBML oder PWML Datenaustauschformate und heruntergeladen als PNG oder SVG-Bilder. Die Wege lassen sich problemlos in Online-Datenbanken integriert werden, integriert in Präsentationen, Plakate oder Publikationen oder ausschließlich für die Online-Visualisierung und Exploration. Diese protocol wurde über 2.000 Pfaddiagramme erfolgreich angewendet zu erzeugen, die jetzt in vielen Online-Datenbanken einschließlich HMDB, Drugbank, SMPDB und ECMDB zu finden sind.
Einleitung
Biologische Pfaddiagramme sind wie Baupläne für Lebenswissenschaftler. Sie sind vielleicht die prägnant und informativ Routen für die Darstellung von biologischen Prozessen und die kontextuellen Verbindungen zwischen Genen, Proteinen und Metaboliten. Dies liegt daran , Bilder werden wesentlich effizienter verarbeitet und oft viel leichter von den Menschen als Text 1 verstanden. Die Qualität, Detail, und die Inhalte der Pfaddiagramme können sehr unterschiedlich sein. Diese Unterschiede hängen oft von dem beabsichtigten Zweck des Weges und die Fähigkeiten des Weges Künstler. Pathways für pädagogische Zwecke geschaffen wie Wandtafeln oder Lehrbücher werden oft von professionellen Künstlern. Als Ergebnis sind diese Pfaddiagramme mehr viel optisch ansprechend und bieten deutlich mehr biologische Detail mit voller Darstellungen von Metaboliten Strukturen, subzellulären Komponenten, Zellstrukturen, Gewebe und Organe. Diese "Lehrbuch" Darstellungen enthalten häufig detaillierte Notizen undKommentare. Auf der anderen Seite, Pfaddiagramme für Internet-Anwendungen entwickelt haben oft Artistik und visuellen Reichtum zugunsten der vereinfachten maschinenlesbaren "Verdrahtung" Diagramme zu opfern. Diese Drahtmodell-Diagramme sind leichter Bild abgebildet und mittels Hyperlink verwiesen. Vereinfachte Pfaddiagramme sind die Basis für solche populären Online - Weg - Datenbanken wie KEGG 2, MetaCyc 3, Wikipathways 4 und Reactome 5. Das Aufkommen von computerkompatiblen Stoffwechselweg Datenbanken hat auch zum Auftreten von Computer-kompatiblen Stoffwechselweg Ziehwerkzeuge geführt. Mit anderen Worten, hat man keine professionellen Künstler oder einen professionellen Programmierer zu generieren nutzbare Pfaddiagramme zu sein. Zum Beispiel BioCyc der Pathway - Tools 6 und PathVisio Software Wikipathway 7 können Benutzer frei zu generieren und teilen maschinenlesbaren Bahnen in BioPAX 8 and / oder HTML-Format. Darüber hinaus gibt es eine Reihe von anderen Stand-alone - Freeware - Pakete sowie kommerzielle Pakete, die die Erzeugung von verschiedenen maschinenlesbaren, Wire-Frame - Bahnen, wie Cytoscape 9, GenMAPP 10 unterstützen, PathCase 11 und Visant 12.
Die Vereinfachung der Internet-Weg-Diagramme wuchs weitgehend von den historischen in vielen Web-Browsern und Web-basierte Rendering-Tools gefunden Einschränkungen. Jedoch erhebliche Fortschritte in der Web-Technologien wurden in den letzten Jahren gemacht. Dies deutete darauf hin, dass es möglich sein könnte, interaktive, internetfähigen Pfaddiagramme zu erzeugen, die genauso bunt sind, ebenso wie ästhetisch ansprechend und ebenso biologisch vollständig wie diejenigen, die in Lehrbüchern zu finden. Diese Arbeit führte zu der Entwicklung von PathWhiz. PathWhiz wurde mit ein Ruby on Rails implementiert (http://rubyonrails.org, Version 4.2.0) web framework eine MySQL relationalen Datenbank enthält (https://www.mysql.com, Version 5.1.50) alle der Weg Daten zu verwalten, einschließlich Entitätsbeziehungen, externe Referenzen, Beschreibungen, Visualisierung Spezifikationen und chemischen Strukturen. Der Front-End-Web-Client wird von Ruby on Rails mit Backbone.js kombiniert gesteuert (http://backbonejs.org, Version 1.0.0) als Front-End-Web-Framework für den Editor.
Ursprünglich entwickelt für die Kuration der Mensch-nur kleine Molekül - Pathway - Datenbank (SMPDB) 13, hat PathWhiz 14 seit erweitert für viele andere Organismen Weg Generation zu unterstützen und als allgemeine Weg Bild und Wissensdatenbank zu funktionieren. Insbesondere ermöglicht das Web-Tool für die Erstellung des gesamten Spektrums von biochemischen / biologischen Wege einschließlich metabolischen, Protein-Wechselwirkung, molekularen Signal, physiologischen und Drogen / Krankheit Wege. Dieser Weg Zeichenwerkzeug unterscheidet sich von den meisten anderenWeg erzeugende Werkzeuge in drei wesentlichen Punkten: 1) Es ist ein Web-Server und nicht als Stand-alone, installierbare Software-Paket; 2) es unterstützt die einfache Erzeugung und interaktive Visualisierung von chemischen Verbindungen, Proteine, Nukleinsäuren, Zellmembranen, subzellulären Strukturen, Geweben und Organen; und 3) es ermöglicht die einfache leihen, bauen oder auf der Arbeit anderer Benutzer zu verbessern, wodurch "Crowdsourcing" Weg Generation. Als Web-Server, es hat mehrere Vorteile gegenüber herunterladbare, plattformspezifischen Software-Tools. Insbesondere ist es kompatibel mit jeder Plattform, Betriebssystem und moderne Web-Browser. Darüber hinaus ist es nicht erforderlich, die Benutzer registrieren, um einen Weg zu beginnen, zu schaffen (obwohl Benutzer frei ein "privates Konto" um und die Zugänglichkeit der Wege schaffen sie kontrollieren zu verfolgen anlegen zu können). Vielleicht die attraktivste Merkmal dieses Werkzeugs ist die Menge der biologischen und biochemischen Detail die leicht hinzugefügt werden können, injeder Weg durch eine Palette von vorgerenderten Bilder und eine umfangreiche Datenbank von Protein und biochemische Daten. Auf diese Weise können die beiden "Nicht-Künstler" und "Nicht-Programmierer" zu leicht bunt schaffen, ästhetisch ansprechend und detailreiche Wege, die web-kompatibel und vollständig maschinenlesbar sind. Ein detaillierter Vergleich zwischen PathWhiz und anderen Weg Zeichenwerkzeuge ist in Tabelle 1 zur Verfügung gestellt.
Eine Reihe von beliebten Life-Science-Datenbanken bereits diesen Weg Zeichenwerkzeug zum Erstellen datenbankspezifische, interaktive Online-Pfaddiagramme verwendet. Zum Beispiel 15 der Escherichia coli Metabolom - Datenbank (ECMDB) seine Weg - Bibliothek vor kurzem mit mehr als 1.650 Bahnen aktualisiert Web - basierte Werkzeug gezogen werden. Jeder Weg im ECMDB ist nun als reich gefärbt, vollständig verlinkte Bild Karte mit detaillierten Metabolit und Proteinstrukturdarstellungen, sowie eine vereinfachte schwarze und weiße KEGG-l angezeigtike Draht Diagramm. Diese großen Weg Update führte zur Entdeckung vieler intermediäre Metaboliten, die zuvor nicht in anderen Escherichia coli metabolischen Datenbanken enthalten waren. Andere Datenbanken, wie beispielsweise die Human Metabolome Database (HMDB) 15, nicht nur auf PathWhiz Bahnen verlassen , um zu zeigen und zu beschreiben , Stoffwechsel- und Signalwege, sondern auch die metabolische Veränderungen in Krankheiten wie Krebs beteiligt darzustellen. HMDB umfasst derzeit 101 Stoffwechselwege, 376 Drogenaktionswege, 233 krankheitsassoziierte Wege und 16 Signalwege, die alle durch diese Web-Tool generiert.
Das folgende Protokoll beschreibt im Detail, wie PathWhiz leicht verwendet werden kann, zu schaffen, replizieren und biochemischen Wege für eine Vielzahl von Zwecken und Anwendungen fortzupflanzen.
Protokoll
1. Pathway-Generation
- Zum http://smpdb.ca/pathwhiz jedem modernen Web-Browser.
- In der Menüleiste, wählen Sie entweder "Try Me!" das Werkzeug als Gast (Bahnen sind öffentlich gezeichnet), "Sign Up" zu verwenden, für ein Konto (nur Wege gezogen wird vom Benutzer bearbeitet werden) zu registrieren, oder "Anmelden" ein bestehendes Konto zu verwenden. Um ein Konto zu registrieren, gehen Sie zu Schritt 1.2.1, sonst gehen zu Schritt 1.3 umgesetzt.
- Um ein Konto zu registrieren, füllen Sie das Formular mit einer E-Mail, Name, Zugehörigkeit, Stadt, Land und 8 Zeichen Mindest Passwort mit Passwortbestätigung und klicken Sie auf "Anmelden".
- Wenn nicht automatisch auf den Pfad Index umgeleitet, wählen Sie die "Draw" Link in der Menüleiste. Diese Index-Seite wird eine Tabelle der vorhandenen Wege zeigen. Klicken Sie auf den "neuen Weg", um einen neuen Weg zu starten.
- Geben Sie den Namen des Weges zu ziehen (zB TCA - Zyklus).
- Wählen Sie die typ der Weg aus der Dropdown - Liste (zB Krankheit / Drug / Stoffwechsel / Signalisieren / Physiologische) zu zeichnen. Es ist möglich, chemische Wege sowie Protein Signalgebung, DNA / RNA Bahnen oder Protein-Protein-Wechselwirkungswege zu erzeugen.
- Suchen und eine Art wählen , durch den wissenschaftlichen Namen des Organismus in der Auto - Vervollständigen Feld eingeben (zB Typ Escherichia coli). Wenn der Name des Organismus nicht in der Dropdown-Liste gefunden wird, klicken Sie auf die Schaltfläche "Neu" und folgen Sie den Schritten 1.6.1 bis 1.6.5 einen neuen Organismus zu fügen, sonst gehen 1,7 Schritt.
- Geben Sie den wissenschaftlichen Namen der Arten (zB Gorilla gorilla) und dem gemeinsamen Namen (zB Gorilla).
- Wählen Sie die Klassifizierung der Arten aus der Dropdown - Liste als Eukaryoten oder Prokaryoten und geben Sie die Taxonomie - ID aus der NCBI Taxonomie (zB 9593).
- Klicken Sie auf den "Create Species" Taste.
- Geben Sie einen umfassive Beschreibung für den Weg. Siehe Ergänzende Datei 1 für ein Beispiel einer Beschreibung. Je vollständiger die Beschreibung, desto leichter ist es, um den Weg zu suchen und zu finden, und die immer beliebter der Weg unter den Nutzern sein.
- Fügen Sie externe Verweise auf den Weg, indem Sie auf die Schaltfläche "Hinzufügen Reference" und folgende Schritte 1.8.1 bis 1.8.2, sonst gehen 1,9 Schritt.
- Geben Sie die PubMed ID (die automatisch die Zitationstext generiert) oder die Zitation Text manuell hinzuzufügen (und lassen Sie die PubMed ID - Feld leer) (zB PMC545700 oder Kang Y, et al. Genomweite Expressionsanalyse zeigt , dass FNR von Escherichia coli K-12 Regelt eine große Anzahl von Genen mit unbekannter Funktion J. Bacteriology 187 (3):.. 1135-1160 doi:. 10,1128 / JB.187.3.1135-1160 2005).
- Wiederholen Sie diesen Vorgang, bis alle haben die gewünschten Referenzen hinzugefügt.
HINWEIS: In den meisten Fällen nur ein oder zwei Referenzen benötigt. Allerdings, je mehr Meta-Daten ein can hinzufügen, desto beliebter wird der Weg sein.
- Klicken Sie auf den "Create Pathway" -Taste. Ein weißer gridded Leinwand wird mit einem grauen Menüleiste angezeigt. Dies ist, wo die Zeichnung stattfindet.
- Klicken Sie auf die Schaltfläche "Hinzufügen Process" Link und wählen Sie "Add Reaction", um den ersten Prozessvisualisierung hinzufügen. Beispielsweise mit einem Reaktions beginnen die Umwandlung von Oxalessigsäure Zitronensäure, über das Enzym Citratsynthase zeigt.
HINWEIS: Prozesse sind biologische Ereignisse oder Aktivitäten. Reaktionen, Transportereignisse, Interaktionen und Bindungsereignisse: Prozesse lassen sich in vier Kategorien eingeteilt werden. In diesem Beispiel wird eine Reaktions gezeigt, aber die gleichen Prinzipien gelten, jede Art von Prozess zu addieren. - Suchen Sie die vorhandenen Reaktionen von Reaktanden, Produkten oder Enzyme in der Auto - Vervollständigen - Box (z .B. "Oxalessigsäure" oder "Acetyl - CoA") eingeben. Blättern Sie durch die vorhandenen Reaktionen die gewünschte Reaktion zu finden, wählen Sie es und gehen Sie direkt zu step 1,12 oder, falls die gewünschte Reaktion nicht gefunden wird, klicken Sie auf die "New Reaction" und folgen Sie den Schritten 1.11.1 bis 1.11.11 ein neues Reaktionsmodell zur Datenbank hinzuzufügen.
- Auf der New Reaction Formular, klicken Sie auf die Schaltfläche "Hinzufügen linke Element", um einen Reaktanden auf der linken Seite der Reaktion hinzuzufügen. Reaktionen werden als Reaktionsgleichungen mit einer linken Seite geschrieben und eine rechte Seite.
- Wählen Sie die Stöchiometrie und Elementtyp (Verbindung / Protein-Komplex / Element-Sammlung / Nukleinsäure / Bound-Element). Suchen Sie nach dem Element mit Namen in der Auto - Vervollständigen - Box (zB "Oxalessigsäure"). Wählen Sie das gewünschte Element. Gehen 1.11.3 oder zu Schritt, wenn das gewünschte Element nicht gefunden wird, klicken Sie auf die Schaltfläche "Neu", und folgen Sie Schritt 1.11.2.1.
- Auf dem neuen Element Form, füllen Sie die Felder in geeigneter Weise und zu speichern.
- Wiederholen Sie die Schritte 1.11.1 und 1.11.2 für jedes Element in der linken Seite der Reaktion beteiligt sind (zB fügen Sie zwei weitere Verbindungen, "Acetyl-CoA "und" Wasser ").
- Wählen Sie die Richtung des Weges aus der Dropdown-Liste (zB wählen Sie den Pfeil von links gerichtet nach rechts).
HINWEIS: Pfeildarstellungen umfassen links nach rechts, von rechts nach links, und reversibel. - Mit dem linken Element der Reaktion nun abgeschlossen ist, klicken Sie auf die Schaltfläche "Hinzufügen rechtes Element", um ein Produkt hinzuzufügen.
- Wählen Sie die Stöchiometrie und Elementtyp, wie in Schritt 1.11.2. Suchen Sie nach dem Element mit Namen in der Auto - Vervollständigen - Box (zB "Citronensäure") und wählen Sie das gewünschte Element. Wenn das gewünschte Element nicht gefunden wird, klicken Sie auf die Schaltfläche "Neu", und folgen Sie Schritt 1.11.2.1.
- Wiederholen Sie die Schritte 1.11.5 und 1.11.6 für jedes Element in der rechten Seite der Reaktion beteiligt sind (zB add "Hydrogen Ion" und "Coenzym A").
- Nachdem die Reaktanden und Produkte erzeugt wurden, klicken Sie auf die Schaltfläche "Hinzufügen Enzym" -Taste ein Enzym zur Reaktion hinzuzufügen.
- Meerrch für das Enzym durch seinen Namen in der Auto - Vervollständigen Feld eingeben (zB "Citratsynthase") und wählen Sie das gewünschte Enzym. Wenn das Enzym in der Datenbank nicht vorhanden ist, klicken Sie auf die Schaltfläche "Neu" und folgen Sie den Schritten 1.11.9.1 bis 1.11.9.7.
- Wenn Sie ein neues Enzym erstellen, verwenden Sie das neue Enzym Form im Enzym Namen und Arten auf den entsprechenden Registerkarten (zB Name: Citratsynthase, Holzart: Escherichia coli) zu füllen.
- Klicken Sie auf die Schaltfläche "Hinzufügen Protein" , um die Spezies-spezifische Informationen hinzuzufügen (dh die Sequenz und Quartärstruktur Informationen) zu diesem Enzym.
- Geben Sie hier die Stöchiometrie und die Suche nach dem Protein mit Namen (Citratsynthase), Gen-Namen (gltA) oder UniProt-ID in der Auto-Vervollständigen-Box. Wählen Sie das gewünschte Protein oder, wenn das gewünschte Protein nicht gefunden wird, klicken Sie auf die Schaltfläche "Neu", und folgen Sie Schritt 1.11.9.3.1.
- An der New Proteinform, füllen Sie die Felder entsprechend und klicken Sie auf die. 4, Erstellen Protein "Taste Pflichtfelder sind Name und UniProt ID Die restlichen Felder sind optional..
- Verwenden Sie die "Add Modifikationen" und / oder "Add Cofaktoren" Tasten Proteinmodifikation hinzuzufügen oder Kofaktoren Enzym, falls erforderlich. In diesem Fall werden weder erforderlich. Füllen Sie in geeigneter Weise in die Felder ein.
- Klicken Sie auf die Schaltfläche "Hinzufügen Biologischer Zustand", um einen subzellulären Ort an das Enzym hinzuzufügen.
- Suchen Sie nach den gewünschten biologischen Zustand durch Eingabe der Spezies, Zelltyp und / oder subzellulären Ort in der Auto - Vervollständigen - Box (zB "Escherichia coli, Cell, Cytosol"). Wählen Sie den gewünschten Zustand oder, falls der gewünschte Zustand nicht gefunden wird, klicken Sie auf die Schaltfläche "Neu" und gehen Sie 1.11.9.6.1 zu treten.
- Auf der neuen biologischen Staatsform, füllen in geeigneter Weise in die Felder ein. Wenn die Arten, etc., die nicht gefunden wird, verwenden Sie die Schaltfläche "Neu" , wie oben 1.6.1 zu 1.6.5 in Schritten beschrieben. Sobald tunne, klicken Sie auf den "Create Biologischer Zustand" -Taste.
- Klicken Sie auf den "Create Enzym", um das Enzym zu speichern.
- Wiederholen Sie die Schritte 1.11.8 bis 1.11.9, um zusätzliche Enzyme hinzufügen, falls erforderlich.
ANMERKUNG: Eine Reaktion viele verschiedene Enzyme zugeordnet sein können, und die Enzyme müssen nicht auf den gleichen biologischen Zustand oder Arten gehören (so lange, wie die Reaktanden und Produkte gleich sind). Wenn eine Reaktion Zeichnung ist es immer möglich, die seiner zugehörigen Enzyme zu wählen, sollte angezeigt werden. - Klicken Sie auf den "Create Reaction", um die neue Reaktion zu speichern.
- Geben Sie die subzellulären Ort Reaktion (für diesen besonderen Weg) in den biologischen Zustand Feld, falls bekannt.
- Wählen Sie die gewünschte Ausrichtung für die anfängliche Rendern der Reaktion aus dem Drop - Down - Listen ( von links nach rechts, von rechts nach links, Horizontal / Vertikal) (Beispiel : Wählen von links nach rechts und horizontal).
- Klicken Sie auf den "Create Reaction" Taste. Die neu geschaffene Reaktion wird auf der Zeichenfläche angezeigt werden.
- Mit Hilfe einer Maus oder Touchpad, klicken Sie auf und ziehen Sie die Reaktionselemente auf der Leinwand neu zu positionieren, wie gewünscht. Führen Sie die Schritte 1.15.1 bis 1.15.5 zur Repositionierung und Bearbeitung von Reaktionselementen (Verbindungen, Kanten und / oder Enzyme) der neu geschaffenen Reaktion.
- Wählen Sie ein oder mehrere Elemente (Verbindungen, Kanten und / oder Enzyme) in der gleichen Zeit, indem Sie entweder ein einzelner Klick jedes Element, um sie hinzuzufügen one-by-one auf die aktuelle Auswahl, oder wählen Sie alle Elemente, indem Sie den Cursor mit einem zu ziehen Box die entsprechenden Elemente um. Die ausgewählten Elemente werden rot gefärbt werden.
- Ziehen Sie die ausgewählten Elemente auf der Leinwand, sie in den gewünschten Bereich (in der Regel in der Mitte der Leinwand) mit einer Maus oder Touchpad neu zu positionieren. Deaktivieren Sie Elemente, indem Sie auf sie ein zweites Mal klicken, oder auf die leere Leinwand klicken.
- Doppelklicken Sie auf eine chemische Verbindung(Es wird durch eine gestrichelte graue Feld umgeben geworden ist) ein Pop-up Seitenleiste zuzugreifen, die sich auf der rechten Seite des Bildschirms angezeigt. Verwenden Sie diese Sidebar die Vorlage, biologischen Zustand, z-index und vollständige Reaktion Details zu bearbeiten.
- Wählen Sie eine Art der Vorlage aus den verfügbaren Optionen (Groß, Mittel oder Klein Verbindung Visualisierung, Groß, Mittel oder Klein Drug-Visualisierung, Cofaktor-Visualisierung, einfache Unten, Links rechts oder von oben Visualisierung). Fügen Sie ein Biologischer Zustand und bearbeiten Sie den z-index, wenn nötig.
HINWEIS: Am besten ist es die Art der Vorlage konsistent über den gesamten Weg zu halten, außer zwischen Verbindungstypen, dh Medikamente gegen Stoffwechselprodukte zu unterscheiden.
- Wählen Sie eine Art der Vorlage aus den verfügbaren Optionen (Groß, Mittel oder Klein Verbindung Visualisierung, Groß, Mittel oder Klein Drug-Visualisierung, Cofaktor-Visualisierung, einfache Unten, Links rechts oder von oben Visualisierung). Fügen Sie ein Biologischer Zustand und bearbeiten Sie den z-index, wenn nötig.
- Doppelklicken Sie auf ein Protein / Enzym (es wird durch eine gestrichelte graue Feld umgeben geworden ist) ein Popup-Sidebar zuzugreifen (auf der rechten Seite des Bildschirms angezeigt werden). Verwenden Sie diese Sidebar die Vorlage, biologischen Zustand, den Z-Index, Protein-Komplex Details und vollständige Umsetzung Details zu bearbeiten.
- Wählen Sie eine Art der Vorlage aus den verfügbaren Optionen (Enzym-Monomer, Dimer oder Tetramer, kein Label, Protein Etikett oder Subunit Etikett, Transporter, Rezeptor oder Repressor). Fügen Sie ein Biologischer Zustand und bearbeiten Sie den z-index, wenn nötig.
HINWEIS: Verschiedene Farben sind zwischen Proteinen zu unterscheiden; die Standardfarbe auf grün gesetzt wird.
- Wählen Sie eine Art der Vorlage aus den verfügbaren Optionen (Enzym-Monomer, Dimer oder Tetramer, kein Label, Protein Etikett oder Subunit Etikett, Transporter, Rezeptor oder Repressor). Fügen Sie ein Biologischer Zustand und bearbeiten Sie den z-index, wenn nötig.
- Um die Informationen für einen gesamten Prozess bearbeiten, klicken Sie doppelt eines ihrer Elemente (werden sie durch eine gestrichelte graue Feld umgeben geworden ist) und Zugriff auf die "Edit Selected" Link im Sekundärmenüleiste (grau). Zwei Optionen werden angezeigt: "Bearbeiten
" und "Bearbeiten ".
HINWEIS: "Bearbeiten" wird der Benutzer auf die Sidebar für das entsprechende Element. "Bearbeiten " werden Optionen enthüllen auf "Edit Details", die Richtung zu ändern, in dem der Prozess gemacht wird (horizontal / vertikal und links / rechts) oder die Kanten wieder (auchHorizontal / Vertikal und links / rechts). die Details bearbeiten Link klicken, wird auf einem Bildschirm führen, dass Details der Reaktion und alle seine Elemente können gleichzeitig bearbeitet werden, einschließlich biologischer Staaten, Vorlagen und z-Indizes. Enzyme können hinzugefügt oder aus der Anzeige entfernt werden, und die Elemente und Kanten können, wenn gewünscht ausgeblendet werden. Zum Beispiel, doppelklicken Sie auf das Wasserstoff-Ion-Element, und klicken Sie dann auf "Edit Selected". Setzen Sie den Cursor über "Edit-Acetyl-CoA + Oxalessigsäure + Wasser → Citronensäure + Coenzym A + Hydrogen Ion" und wählen Sie "Edit Details". Fügen Sie ein Biologischer Zustand für jede Verbindung (Escherichia coli, Zelle, Zytoplasma). Ist das erledigt, gehen Sie auf die unten auf der Seite und klicken Sie auf die lila "Update Reaction" Taste. Nur Enzyme, die bei dieser Reaktion bereits zugeordnet sind, können in diesem Stadium auf den Weg gegeben werden. Wenn neue Enzyme in das Reaktionsmodell hinzugefügt werden müssen, kehren Sie zu dem Reaktionsindex (unter der Registerkarte "Prozesse"), finden die gewünschte Reaktion,und fügen Sie sie dort. - Bearbeiten Sie die Reaktions Kanten durch einen einzigen Klick oder Doppelklick.
- Wählen Sie die Reaktionsränder sie in der gleichen Weise wie Verbindungen und Proteine zu manipulieren. Klicken und Ziehen am Rand die ganze Kante zu bewegen.
HINWEIS: Die Start- und Endpunkte können auch angeklickt werden, und zog den Kantenlänge zu ändern. Wenn der Start- / Endpunkt einer Flanke ausgewählt wurde, kann der zugehörige "Knopf" eingestellt werden, um die Richtung und die Krümmung der Start- / Endpunkt zu steuern. So fügen Sie zusätzliche Knoten an den Rand, wählen Sie den Rand, und beachten Sie das blaue Rechteck, das erscheint. Klicken Sie auf die obere Hälfte des Rechtecks einen Knoten hinzuzufügen, und klicken Sie auf die untere Hälfte einen Knoten zu entfernen. - Doppelklicken Sie auf den Start- / Endpunkt einer Kante des Start- / Endpunkt durch den spitzen Pfeil Zyklus, Sperrung Pfeil und kein Pfeil Optionen.
- Wählen Sie die Reaktionsränder sie in der gleichen Weise wie Verbindungen und Proteine zu manipulieren. Klicken und Ziehen am Rand die ganze Kante zu bewegen.
- Wählen Sie ein oder mehrere Elemente (Verbindungen, Kanten und / oder Enzyme) in der gleichen Zeit, indem Sie entweder ein einzelner Klick jedes Element, um sie hinzuzufügen one-by-one auf die aktuelle Auswahl, oder wählen Sie alle Elemente, indem Sie den Cursor mit einem zu ziehen Box die entsprechenden Elemente um. Die ausgewählten Elemente werden rot gefärbt werden.
- Nachdem die erste Reaktion wie gewünscht gezogen worden ist , wählen Sie ein Produkt der Reaktion (zB Zitronensäure)mit Doppelklick (beachten Sie die Änderung in der Farbe rot) , um den nächsten Prozess auf dieses Reaktionsprodukt (in diesem Fall Zitronensäure zu cis -Aconitic Säure über Aconitathydratase) hinzuzufügen.
- Einmal ausgewählt, klicken Sie auf die Schaltfläche "Hinzufügen Prozess" und klicken Sie auf "Hinzufügen Reaction" Option.
- Fügen Sie eine weitere Reaktion auf den TCA-Zyklus (in diesem speziellen Beispiel) durch Wiederholen des Verfahrens zum Hinzufügen eines Reaktions (Schritte von 1,11 bis 1,15). Da diese Reaktion wird eine bestehende Reaktionsprodukt aufgebaut aus, nur Reaktionen, die das ausgewählte Element enthalten wird angezeigt.
- Fügen Sie die übrigen Reaktionen für den TCA-Zyklus durch folgende Schritte an 1,11-1,15 für jede Reaktion.
- Nachdem alle Reaktionen hinzugefügt, fügen Sie visuelle Elemente wie Membranen, DNA, tRNAs, subzellulären Organellen, Organe, Gewebe, Zoom-Boxen oder Etiketten, indem Sie auf die Schaltfläche "Hinzufügen von Visual Element" Link und einer der "Add Membrane" Auswahl "hinzufügen Bild "," Zoom-Box "oder"In Etikett "Optionen Führen Sie die Schritte 1.20.1 bis 1.20.4 visuelle Elemente hinzuzufügen;..
- Klicken Sie auf die Schaltfläche "Hinzufügen Membrane" Option, um eine Zellmembran hinzuzufügen. Bearbeiten Sie diese Membran durch einen Doppelklick auf den Seitenleiste zuzugreifen. Wählen Sie die Art der Membran in das Schablonenfeld auf der Seitenleiste befindet. Wählen Sie den "Geschlossenen Membran" Option eine Box-Membran zu machen.
- Klicken Sie auf die "Add Image" Option ein Bild hinzufügen derzeit bestehenden in der PathWhiz Datenbank (Standardbilder umfassen Organe, Organellen und Gewebe). Bearbeiten Sie das Bild mit einem Doppelklick auf eine Sidebar zuzugreifen. Bearbeitungsoptionen sind selbsterklärend und umfassen Tiefe Skalierung mit der z-index, Scale-up / verkleinern und drehen Sie nach links / rechts drehen.
- Klicken Sie auf die Schaltfläche "Hinzufügen Zoom Box" Option ein Zoom-Box zu einem bestimmten Bild hinzuzufügen.
- Klicken Sie auf die Schaltfläche "Hinzufügen Etikett" Option eine Beschriftung hinzuzufügen. Bearbeiten Sie das Etikett durch einen Doppelklick auf sie ihre Sidebar zuzugreifen. Bearbeitungsoptionen umfassen die Etikettenvorlage, Text undz-index.
HINWEIS: Der "Vakuummetallisieren Element" Link bietet Optionen zum Hinzufügen von Fremd Verbindungen, Proteine, Nukleinsäuren, Element Sammlungen oder Kanten auf die Leinwand, die nicht mit einem Prozess verbunden sind. Diese Elemente werden zunächst in der oberen linken Ecke der Leinwand angezeigt. Sie erscheinen als beliebige Elemente, die in der Seitenleiste bearbeitet werden kann, in dem der Benutzer das gewünschte Element auswählen und die Visualisierung ändern Details des Elements. Das Element sollte vor dem Hinzufügen irgendwelcher neuer vacuous Elemente, um in den Signalweg eingebracht werden Stoffwechselweg Neatness aufrechtzuerhalten. Vacuous Elemente sind in visuelles Verständnis zu unterstützen , nur gemeint (dh , welche die Anwesenheit von mehreren tRNAs während der Transkription) und nicht integraler Prozesskomponenten darzustellen. Sie sollten sparsam verwendet werden, da sie nur in der Visualisierung angezeigt wird, und nicht in die maschinenlesbare Formate integriert (BioPAX, SBML, SBGN, PWML).
- Fügen Sie einen Unter pathway durch den "Add-Prozess" Link klicken und dann auf "Hinzufügen Sub-Pathway" Option.
HINWEIS: Sub-Bahnen können auch auf bestehende Reaktionen verkettet werden, in der gleichen Weise wie in den Schritten 1,16-1,18 demonstriert. Die Zugabe von Teilwegen kann die Komplexität von großen oder komplexen Bahnen zu reduzieren. Sie können auch zusätzliche Informationen über die Verbindungen zwischen bekannten Wege zu schaffen, verwendet werden. - Suchen Sie nach dem Unter Weg Name in der Auto-Vervollständigen-Box. Nur Teilwege, die für diesen Weg bereits definiert, erscheint in der Auto-Vervollständigen-Box haben, also, ob dies ein neuer Weg ist, keine Unter Wege erscheint. Wenn der gewünschte Unter Pfad nicht existiert, klicken Sie auf den "New Sub-Pathway" und Schritte 1.22.1 bis 1.22.3 folgen.
- Wählen Sie den Unter Weg Typ (Sub Pathway / inhibitorischen Unter Pathway / Aktivierung Sub Pathway).
- Geben Sie die Sub-Pfad-Namen.
- Hinzufügen Eingangs- und Ausgangselemente mit dem Unterpfad in der gleichen Weise wie das Hinzufügen Reaktanden und proddukte zu einer Reaktion (Schritte 1.11.1 bis 1.11.3 oben).
HINWEIS: Ein Unterpfad sollte mindestens ein Eingangs- oder Ausgangselement haben. Dies ermöglicht es den anderen Prozessen in der Bahn verbunden werden, in der gleichen Weise wie Reaktionen verkettet sind (Schritte 1,16-1,18 oben). - Klicken Sie auf den "Create Unter Pathway" -Taste.
- Passen Sie die Größe der Leinwand eines Weges Diagramm durch das "Andere" Link klicken und die "Leinwandgröße ändern" Option auswählen. Um die Größe der Leinwand zu ändern, folgen Sie den Schritten 1.23.1 bis 1.23.3.
- Füllen Sie das "Neue Größe" -Feld und das "Neue Breite" Feld entsprechend.
- Wählen Sie die gewünschte Richtung, in die die Leinwand in der Größe auf dem Raster die entsprechende Taste erhöhen oder zu verringern sollte im "Anchor" Abschnitt durch einen Klick.
- Klicken Sie auf den "Update Canvas Größe" -Taste.
- Alternativ passen sich automatisch die Größe der Leinwand. Sobald der Weg abgeschlossen ist, clecken "Sonstige" link und wählen Sie die "Fit Canvas Pathway" Option. Dadurch wird automatisch schneiden Sie die Leinwand um den bestehenden Weg Elemente.
- Wenn der Weg abgeschlossen ist, klicken Sie auf den "Pathway" Link und wählen Sie die "Export und Ansicht" Option.
- Verwenden Sie den "Hintergrundfarbe für Bilder" Option entweder blau oder weiß als Hintergrundfarbe für das Bild zu wählen.
- Wählen Sie entweder Ja oder Nein für die "auch vereinfachte Version generieren?" Option. Wenn Ja ausgewählt ist, wird ein KEGG artige Draht Diagramm auch automatisch für den Weg erzeugt werden.
- Klicken Sie auf die "Generieren von Bilddateien" klicken.
HINWEIS: Dies erzeugt die verschiedenen Datenaustauschformate und Bilddateien für den Weg. Die Bilder müssen jedes Mal neu generiert werden, der Weg aktualisiert wird, und dies kann einige Minuten dauern. - Sobald das Bild erzeugt wurde, klicken Sie auf das Feld "Viewer", um eine vollständig verlinkte, hochauflösende Pfad anzuzeigenWeg Bild im Browser.
HINWEIS: Die "Show Viewer" Schaltfläche erscheint nur für Wege, sobald ihre Bilder erzeugt wurden. Diese Ansicht enthält auch Links, um den Weg in den verschiedenen Datenaustausch und Bildformaten zum Download bereit.
2. Pathway-Replikation
HINWEIS: Pathway-Replikation ist eine schnelle und einfache Weg einen vorhandenen Pfad in PathWhiz Bibliothek zu nehmen und es zu duplizieren, so dass es als Vorlage für die weitere Bearbeitung oder Veränderung dienen kann. Um einen Weg zu replizieren, führen Sie die Schritte 1.1 bis 1.3 anzumelden, falls noch nicht geschehen.
- Gehen Sie auf die Pfad-Index, wenn es nicht schon von "Pathways" auf der Hauptmenüleiste klicken. Suchen Sie nach den gewünschten Weg repliziert werden durch seinen Namen in die Suchleiste eingeben und auf die Schaltfläche "Suchen" (zB "TCA - Zyklus").
- Suchen Sie den Pfad repliziert (zB "TCA - Zyklus") werden soll, und klicken Sie auf die grüne "Replizieren" , aberTonne.
- Ändern Sie den Namen des Pfades , falls gewünscht (zB Typ "TCA - Zyklus Practice". Keine zwei Wege den gleichen Namen haben können.
- Geben Sie eine neue oder eine andere Beschreibung für den Weg (siehe Schritt 1.7).
- So fügen Sie neue oder andere Verweise auf den Weg, klicken Sie auf den "Verweis hinzufügen" und folgen Sie Schritt 1.8 oben.
- Klicken Sie auf den "Create Pathway" -Taste. Ein lila Fortschritt Rad wird angezeigt, während der Weg gebaut wird. Dieser Vorgang kann mehrere Minuten oder länger dauern, je nach Größe des Weges.
- Bearbeiten oder fügen Sie alle gewünschten Elemente auf den Weg Schritten 1,10-1,22 verwenden.
- Führen Sie die Schritte 1,23-1,29, um den Pfad vervollständigen und zu exportieren.
3. Pathway Vermehrung
HINWEIS: Pathway Ausbreitung ist eine schnelle und einfache Weg für einen Organismus einen vorhandenen Pfad in PathWhiz der Bibliothek zu übernehmen (zB Escherichia coli) und einen ähnlichen Weg für andere zu schaffenOrganismus (zB Staphylococcus aureus). Dieser Prozess beinhaltet das Finden und S. aureus - Proteine von E. coli - Proteine ersetzen und den gesamten Weg mit S. aureus Proteine oder Gene zu regenerieren. Um einen Weg Start propagieren durch die Schritte 2,1 bis 2,2 über folgende.
- Suchen Sie den Weg (in diesem Fall TCA-Zyklus) propagiert werden und klicken Sie auf den Button "Anzeigen".
- Klicken Sie auf die Schaltfläche "Propagieren" in der rechten oberen Ecke.
- Auf der Ausbreitungs Formular, klicken Sie auf die Schaltfläche "Hinzufügen Species", um die Spezies zu identifizieren umgewandelt, auf die wird der bestehende Weg. Mehrere Arten können hinzugefügt werden, obwohl die mehr Spezies vorhanden sind, desto mehr die Umwandlungszeit.
- Suchen Sie nach einer vorhandenen Spezies oder eine neue Spezies folgenden Schritte 1.6.1 bis 1.6.5 oben hinzuzufügen.
- Falls gewünscht, ändern Sie die E-Wert, der die Ähnlichkeitsschwelle für die Suche nach Protein-Homologe zu bestimmen. Wählen Sie Ja oder Nein für die "Bewertet Proteine Nur"Option (dies zeigt an, welche UniProt Proteine sollten in dem erzeugten Pfad verwendet werden).
- Klicken Sie auf die "Propagieren Pathway" -Taste.
- Klicken Sie auf die Schaltfläche "OK" im Pop-up-Fenster.
HINWEIS: Ein lila Fortschritt Rad wird angezeigt, während der Weg ausbreitet. Dieser Vorgang kann mehrere Minuten oder länger dauern, je nach der Größe des Weges und die Anzahl von Arten, auf die die anfängliche Weg propagiert wird. - Ein zweites Pop-up-Fenster wird angezeigt, wenn die Ausbreitungs abgeschlossen ist. Klicken Sie auf die Schaltfläche "OK". Dies wird einen Index zeigt alle neuen Wege schaffen, die aus dieser Ausbreitung erzeugt wurden.
- Von diesem Index, überprüfen Sie die Bahn Details und klicken Sie auf die "Draw" -Taste jeder der neuen Wege zu betrachten und zu bearbeiten.
- Bearbeiten oder fügen Sie alle gewünschten Elemente auf den Weg Schritten 1,10-1,22 verwenden.
- Führen Sie die Schritte 1,23-1,29, um den Pfad vervollständigen und zu exportieren.
4. BearbeitenVorhandenes Pathway
HINWEIS: In einigen Fällen neue Informationen über einen vorhandenen Pfad hinzugefügt oder falsche Informationen über einen Pfad korrigiert werden muss werden. Um einen vorhandenen Pfad zu bearbeiten, starten durch die Schritte 1.1 bis 1.3 folgende anmelden.
- Gehen Sie auf die Pfad-Index, wenn es nicht schon von "Pathways" auf der Hauptmenüleiste klicken.
- Suchen Sie den Weg zu editierenden (in diesem Fall die "TCA-Zyklus"). Wenn das Bild bearbeitet werden soll, klicken Sie auf die "Draw" -Taste, wenn die Beschreibung oder Verweise zu bearbeitenden, klicken Sie auf die Schaltfläche "Bearbeiten".
- Um das Bild zu bearbeiten, folgen die Schritte 1,10-1,29 oben.
- Um den Weg Beschreibung oder Referenzen zu bearbeiten, wiederholen Sie die Schritte 1,4 bis 1,8 und klicken Sie auf den "Update Pathway" -Taste, wenn Sie fertig, um die Änderungen zu speichern.
- Generieren Sie das Bild, um die Änderungen zu aktualisieren, indem Sie folgende Schritte 1,25-1,28.
5. Pathway Viewing und downloadenIchng
HINWEIS: Diese Web-basierte Tool Tausende von sorgfältig erstellt und bearbeitet Wege enthält, die für verschiedene Anwendungen eingesehen oder heruntergeladen werden können. Zum Anzeigen oder einen Weg herunterzuladen, folgen Sie den Schritten 1.1 bis 1.3 anmelden.
- Gehen Sie auf die Pfad-Index (wenn nicht bereits) von "Pathways" auf der Hauptmenüleiste klicken.
- Suchen Sie den Pfad heruntergeladen werden (in diesem Fall, TCA-Zyklus) und klicken Sie auf den Button "Anzeigen".
- Klicken Sie auf die lila-Taste mit der gewünschten PathWhiz ID neben der "Show Viewer" Etikett (es kann mehr als eine ID sein).
- Klicken Sie auf die Registerkarte "Downloads" in der Seitenleiste.
- Klicken Sie auf die Hyperlinks, den Weg in verschiedenen Dateiformaten zum Download bereit.
Ergebnisse
Die Hauptweg Generierungstool in diesem Manuskript beschrieben Webserver ist in Abbildung 1 und Abbildung 2 dargestellt. Die Menüoptionen von jedem Register zur Verfügung gestellt werden auch gezeigt. Die 3 und 4 stellen eine Reihe von Screenshots des Weges Erstellungsprozess. Abbildung 5 stellt eine Reihe von Screenshots des Reaktionsentstehungsprozess. Abbildung 6 zeigt die Online - Weg - Viewer und sein Menü.
PathWhiz kann verwendet werden Wege mit verschiedenen Content-Arten und Stile zu erzeugen. Dazu gehören "traditionellen" Stoffwechselwege (Abbildung 7), Krankheit und Drogenwege zeigt Nebenwirkungen (Abbildung 8) und Arzneimittelreaktionen (Abbildung 9), sowie Protein - Signalwege (Abbildung 10). Pathways werden reich gefärbt mit erheblichen biologische Detail , oder sie können auf einfache Schwarz-Weiß - Darstellungen (Abbildung 11) umgewandelt werden. Einmal abgeschlossen, können diese Wege in den interaktiven Weg - Viewer angezeigt werden (Abbildung 6), als Bilder heruntergeladen oder in mehreren verschiedenen maschinenlesbaren Datenaustauschformate für die weitere Analyse exportiert. Beachten Sie, dass sich die Qualität der verschiedenen Datenaustauschformaten eingegeben von der Qualität der Daten abhängt, wenn ursprünglich den Weg zu ziehen. Zum Beispiel können mehr Reaktions Detail Hinzufügen (dh Stöchiometrie biologischen Zustände) wird umfassender BioPAX produzieren. Auf der anderen Seite, Wege mit überlappenden Elemente (für optischen Gründen, wie zum Beispiel zeigt gebundene Elemente oder Proteinkomplexe) gezogen können auch überlappende Glyphen in SBGN-ML produzieren.

Abbildung 1: Pathway Editor - Schnittstelle. Der Editor-Schnittstelle ist COMPOsed von 3 Hauptbereiche: eine obere Hauptmenüleiste, eine sekundäre Menü und eine gerasterte Leinwand. Die obere Hauptmenüleiste (lila) enthält Links zu Anzeigen, Bearbeiten und Pathway-Elemente erstellen. Die untere sekundäre Menüleiste (grau) bietet Links hinzufügen und Sehbahn Elemente im aktuellen Weg-Diagramm zu bearbeiten, wie Reaktionen, Interaktionen, Transportprozesse, Unterwege, Verbindungen, Proteine, Nukleinsäuren, sowie Membranen, zelluläre / subzellulärer Bilder, Zoom-Boxen, und Etiketten. Dieses Menü enthält auch zwei Registerkarten, die die Bearbeitung von ausgewählten Elementen oder die Bearbeitung der Leinwand ermöglichen. Die gerasterten weißen Leinwand unter Menüleisten ist, wo die Reaktionswege und Prozesse hinzugefügt werden. Die Zoom-Box funktioniert als visuelle cue die Vergrößerung eines ausgewählten Bereichs in einem Bild anzuzeigen. Es besteht aus einem kleinen Platz, die zu einer Wieder beträchtliche Viereck verbunden ist. Der kleine Platz ist auf der Fläche platziert, die erweitert oder vergrößert werden soll, während das Viereck als Leinwand arbeitet, in dem ein th hinzufügene Reaktionen, die in den ausgewählten Bereich passieren (durch den kleineren Platz). Bearbeiten Sie die Zoom-Box mit einem Doppelklick auf das seine Sidebar zuzugreifen. Bearbeitungsoptionen umfassen für Vorlage Dropdown-Listen, Farbe und z-index. Die Rendering-Orientierung des Zoom-Box kann durch die Auswahl oben, rechts geändert werden, links oder unten in der Vorlage Registerkarte. Wenn die Zoom-Box ausgewählt ist, können die schwarzen Kreise gezogen werden, um die verschiedenen Zoom-Box-Komponenten, um die Größe und formatieren. Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.
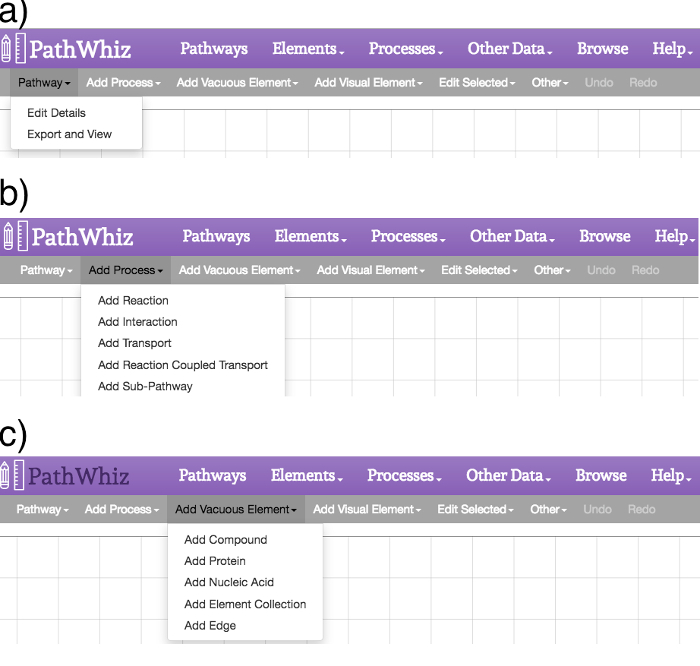
Abbildung 2: Pathway Editor Menüs. Die Editor-Menüs bieten Optionen Prozesse und Elemente hinzuzufügen, sowie die Elemente und die Leinwand zu bearbeiten. (A) Der "Pathway" Link bietet Optionen "Edit Details" und "Export und Ansicht". Die "Edit Details" Option erlaubt die Bearbeitung des Weges Beschreibung und Referenzen, während die "Export und Ansicht" Option, um die Erzeugung oder Regeneration der Bilddateien ermöglicht. (B) Der "Prozess" Link bietet Optionen für das Hinzufügen einer Reaktion, Interaktion, Bindungsereignis, Transportereignis, gekoppelte Reaktion Transport oder Unter Weg auf die Leinwand. (C) Die "Add Vakuummetallisieren Element" Link bietet Optionen für eine Verbindung hinzufügen, ein Protein, eine Nukleinsäure, ein Element Sammlung oder eine Kante an den Leinen. Diese Elemente werden in der oberen linken Ecke der Leinwand angezeigt. Ein beliebiges Element wird neben dem Pop-up Seitenleiste in der Leinwand erscheinen, in dem der Benutzer für das gewünschte Element suchen oder die Details des Elements ändern. Das Element sollte vor der Zugabe keine neuen Elemente in vacuous der Bahn eingebracht werden, um Stoffwechselweg Neatness zu halten._upload / 54869 / 54869fig2large.jpg "target =" _ blank "> Bitte hier klicken, um eine größere Version dieser Figur zu sehen.
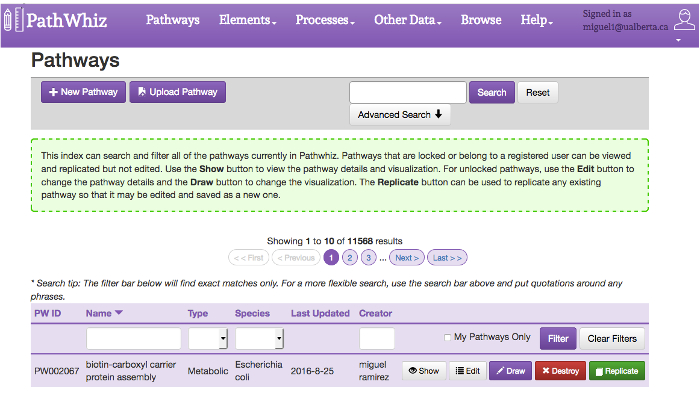
Abbildung 3: Pathway Index bilden. Der Weg Index bietet eine Sammlung von derzeit bestehenden Wege und ein Suchfeld für spezifische Wege abfragen. Pathways können nach Namen gefiltert werden, Typ, Spezies und Schöpfer des Filterleiste oben auf der Indextabelle. Sie können auch durch Namen mit Hilfe der Suchleiste am oberen Rand der Seite gesucht werden. Die Öffnung des "Advanced Search" ermöglicht spezifischere Suche nach Kombinationen von biologischen Zustand, Typ, Spezies, Verbindung und Protein. Die erweiterte Suche ermöglicht die Verwendung von AND, OR und NOT logischen Operatoren komplexe Abfragen zu erstellen. Jeder Weg enthält 5 Tasten: "Show", "Bearbeiten", "Zeichnen", "Destroy" und "Replizieren". Der Button "Show"mit dem Viewer ermöglicht des Weges sehen. Die Schaltfläche "Bearbeiten" ermöglicht die Bearbeitung des Weges Metadaten, einschließlich Name, Typ, Spezies, Beschreibung und Referenz. Die "Zeichnen" Taste ermöglicht die Bearbeitung der Leinwand, um den Pfad enthält. Die "Destroy" Taste ermöglicht das Entfernen des Weges aus der Datenbank (wenn der Benutzer die Berechtigung hat). Die "Replizieren" Taste ermöglicht die Replikation des gewählten Weges. Die "Zurück" und "Weiter" Tasten kann der Benutzer zwischen den Seiten von Pfaden zu navigieren. Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.
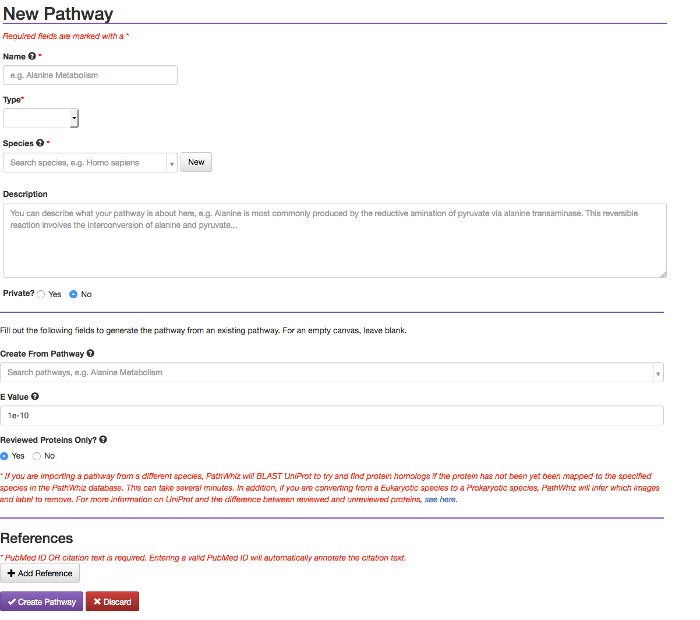
Abbildung 4: Create New Pathway - Formular. Die "New Pathway" -Taste (siehe Abbildung 3) führt zu dem hier gezeigten Weg Form. Dieses Formular enthältFelder für den Namen des Weges, Art, Arten und Beschreibung. Die "New-Weges" Taste ermöglicht auch eine von einem vorhandenen Weg zu starten und Referenzen hinzufügen. Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.
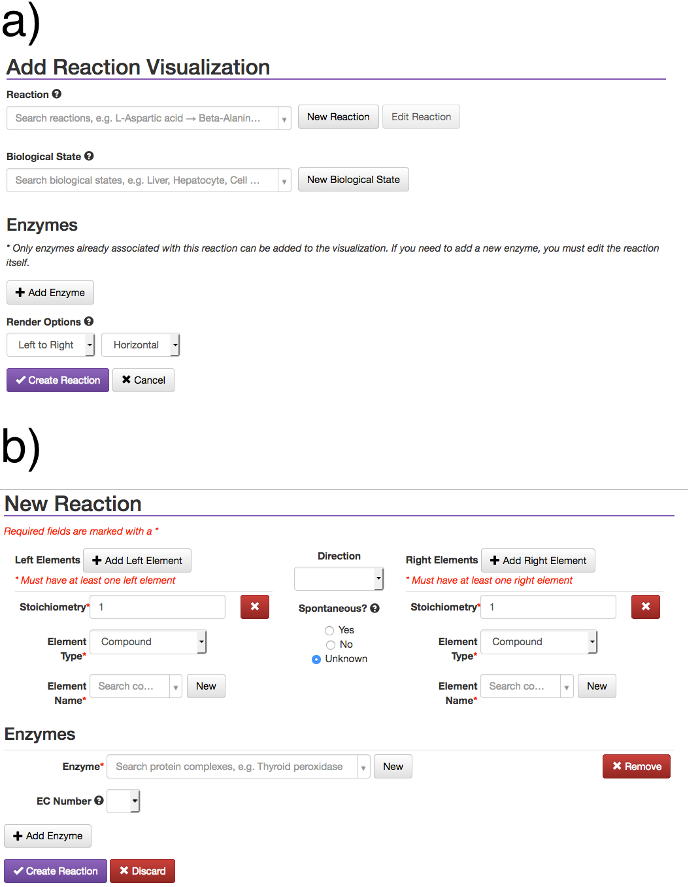
Abbildung 5: Erstellen neuer Reaktions Formular. Der "Prozess" Link ermöglicht es Benutzern, einen neuen Prozess an, wie eine Reaktion oder Bindungsereignis. ein Verfahren Beim Hinzufügen eines Reaktionsmodell, aus dem Reaktions Visualisierungen erzeugt werden kann und hinzugefügt Diagramme Weg. Das Reaktionsmodell und Visualisierung sind separate Einheiten. (A) Die Reaktionsfeld suchen Genehmigungen für eine bestehende Reaktion, durch Reaktanden, Produkt oder Enzym. Die biologischen Zustand Feld ermöglicht die Suche und Auswahl einer bestehenden biological Zustand. Wenn eine Reaktion ausgewählt wird, können die entsprechenden Enzyme zugesetzt werden, mit der "Enzyme Hinzufügen", die ein Enzym zur automatischen Vervollständigung Box bringen wird. Die Render-Optionen ermöglichen es dem Anwender, die Richtung sie die Reaktion wollen wählen gerendert werden. Eine neue Reaktion kann durch das (b) "New Reaction" Taste erstellt werden, die in ein neues Reaktionsform führt , wo Elemente und Enzyme hinzugefügt werden können. Wenn alle Felder ausgefüllt sind, kann die Reaktion durch die "Create Reaction" Taste erstellt werden. Um die zugrunde liegende Reaktionsmodell ändern, bei dem man den Weg Illustrator zu verlassen, gehen Sie auf die Reaktion Index und finden und die Reaktion dort bearbeiten. Um Diskrepanzen zwischen Daten zu verhindern und ungewollt zu verändern bestehenden Bahnen, ist es nicht möglich, die Reaktanden / Produkte oder entfernen Enzyme aus einem Reaktionsmodell zu ändern, wenn sie bereits Visualisierungen in bestehenden Bahnen hat. Somit ist die Bearbeitung des Reaktionsmodell nicht automatisch aktualisiert bestehende ReaktionVisualisierungen. Damit ein Reaktionsmodell zu ändern, muss man das entsprechende Visualisierung auf dem Weg Diagramm erneut hinzufügen, damit die Änderungen angezeigt werden. Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.
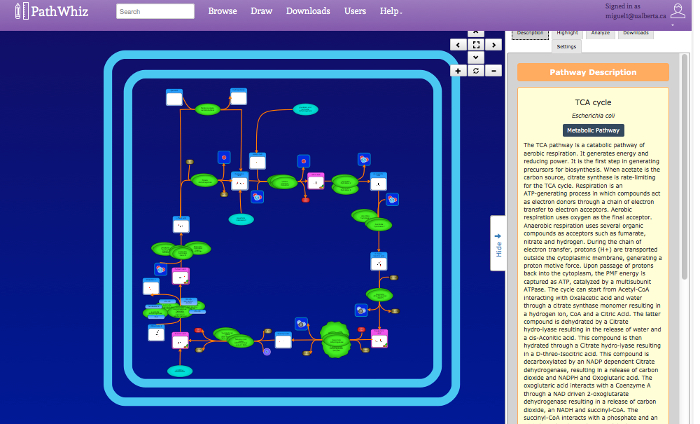
Abbildung 6: Pathway - Viewer. Die oben rechts Viewer-Schnittstelle Tasten bieten grundlegende Navigation, Zoomen und Bildschirm Makeln Aktionen. Das zentrale Ansichtsfenster zeigt den Weg, der durch Klicken und Ziehen navigiert werden kann, oder das Zoomen mit einer Maus. Die Bahn - Elemente angezeigt werden , auf andere Wege mittels Hyperlink verwiesen und Datenbanken (zB HMDB, Drugbank, UniProt). Die seitlichen Menüleiste zeigt eine Beschreibung des Weges mit Hinweisen durch den Benutzer geliefert. Die Seite im Menü zeigt auch die Registerkarten "Highlight", "Analysieren", "Downloads "und" Einstellungen ". Das" Highlight "Tab ermöglicht Verbindungen und Enzyme ausgewählt und rot markiert werden. Die" ermöglicht Registerkarte Analysieren "experimentellen Konzentrationsdaten eingegeben werden, die dann auf den Weg abgebildet wird einen Farbverlauf verwenden. Die Registerkarte "Downloads" bietet Links zu den entsprechenden herunterladbare Bilddateien und Datenaustausch-Dateien. Die PNG-Datei ist eine kleinere nicht-Vektor-image-Datei. Die SVG + BioPAX Links bieten größere Vektor-Bild-Dateien mit eingebetteten BioPAX, für Maschinenlesbarkeit. Die BioPAX, SBML, SBGN und PWML Links bieten verschiedene maschinenlesbare Formate. die Registerkarte "Einstellungen" ermöglicht die visuelle Anpassung des Weges Bild angezeigt. Bitte hier klicken , um eine größere Version dieser Figur zu sehen.
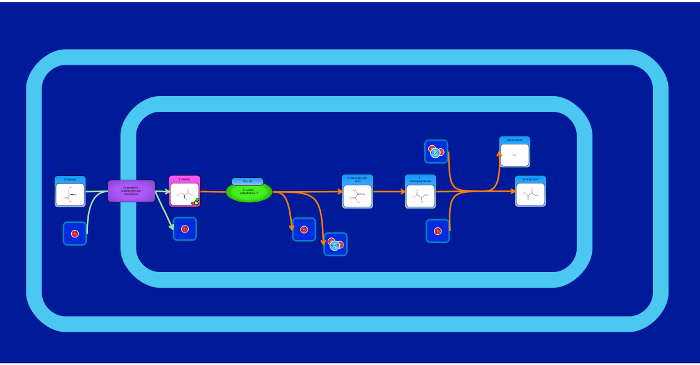
Abbildung 7: Stoffwechselweg Bild. Dies ist ein Beispiel für eine "herkömmliche" Stoffwechselweg, die die Biosynthese und den Abbau einer bestimmten Verbindung (D-Serin) beschreibt. Der Hauptmetabolit ist in der Mitte der Leinwand und die Reaktions- und Transportpfeile (Kanten) zeigen den Ablauf des Weges positioniert. Kanten und Elemente können automatisch sein " , blaffte" zusammen, dh verbunden. Element Fangpunkte durch transparente rote Kreise auf den Elementseiten angezeigt werden, und Kanten Start- / Endpunkte werden durch transparente graue Kreise am Rand Enden vertreten. Fangpunkte drehen ein transparentes grün, wenn schwebte über, und eine solide grün, wenn ausgewählt. Um eine Kante zu einem Element zu schnappen, zuerst auf klicken Sie entweder auf den Rand Start / Ende oder dem Element Punkt schnappen (es wird grün leuchten). Dann klicken Sie auf den Rand Start / Ende oder dem Element Fangpunkt, die verbunden werden müssen. Der Rand wird sich automatisch auf die Fangpunkt verbinden und verbunden bleiben Until es (die Kante mit einem Doppelklick und Ziehen Sie den Endpunkt weg, oder es an einen anderen Fangpunkt verbindet) entfernt ist. Es ist wichtig, versehentlich die Auswahl Fangpunkte aufmerksam zu sein, da dies unerwünschte Folgen haben kann, wenn Sie versuchen, Kanten zu bewegen. Der feste grüne Farbe ausgewählter Fangpunkte soll den Anwender zu alarmieren Punkte schnappen sie sich aktuell befinden. Fangpunkte können durch Klicken auf sie ein zweites Mal abgewählt werden. Kanten können auch auf ihre ursprünglichen Elemente verbunden werden. Wenn eine Kante ausgewählt wird, den Besuch der "Edit Selected" Menü-Link und dann den Link "Edit Edge". Dies wird Optionen bringen, um automatisch die Kante in verschiedenen Richtungen zu verbinden. Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.
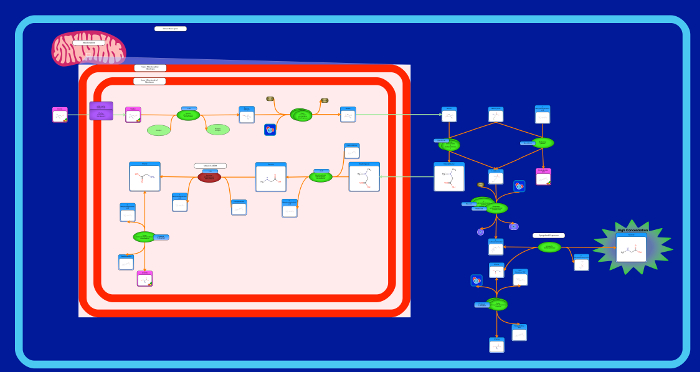
Abbildung 8: Krankheit Pathway Bild. Dies ist ein Beispiel für einen Krankheits Weg, der die Organe zeigt, die von der Krankheit (Sarcosine Oncometabolite Weg). Zusätzliche Bildelemente werden verwendet, um die Zunahme oder Abnahme der Metaboliten-Konzentrationen und deren Akkumulation oder Dissipation darzustellen. Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.
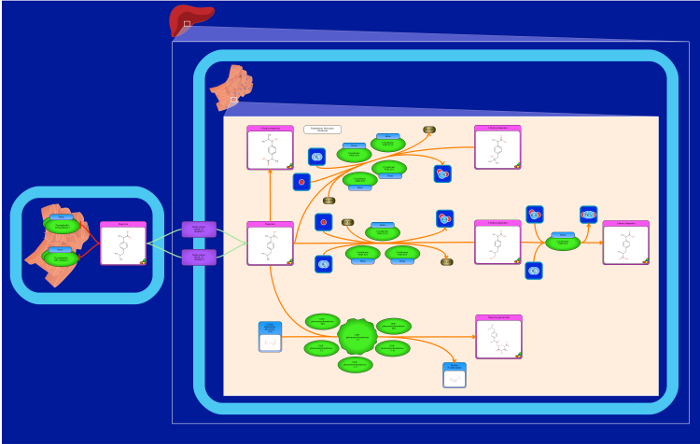
Abbildung 9: Drug Pathway Bild. Dies ist ein Beispiel eines Arzneimittels Stoffwechselweg, dass die Organe zeigt, wo das Medikament verstoffwechselt wird (Ibuprofen Pathway). Die Farbe des Arzneimittels Metabolit umgibt, wird in der Regel als rosa dargestellt. Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.
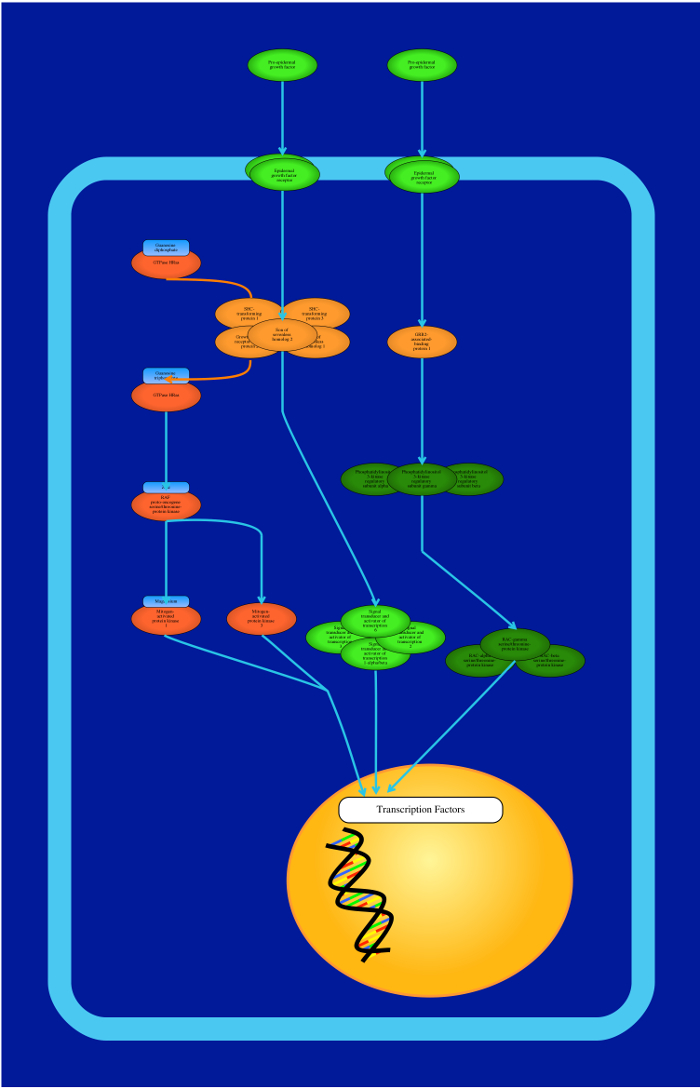
Abbildung 10: Protein Signaling Pathway Bild. Dies ist ein Beispiel eines Signalwegs, der eine Sammlung von Signalreaktionen zwischen verschiedenen Proteinen (EGFR Pathway) zeigt. Proteine können mit mehreren Farben dargestellt werden, und sie kann entweder durch das Protein Namen oder der Untereinheit Namen dargestellt werden. Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.
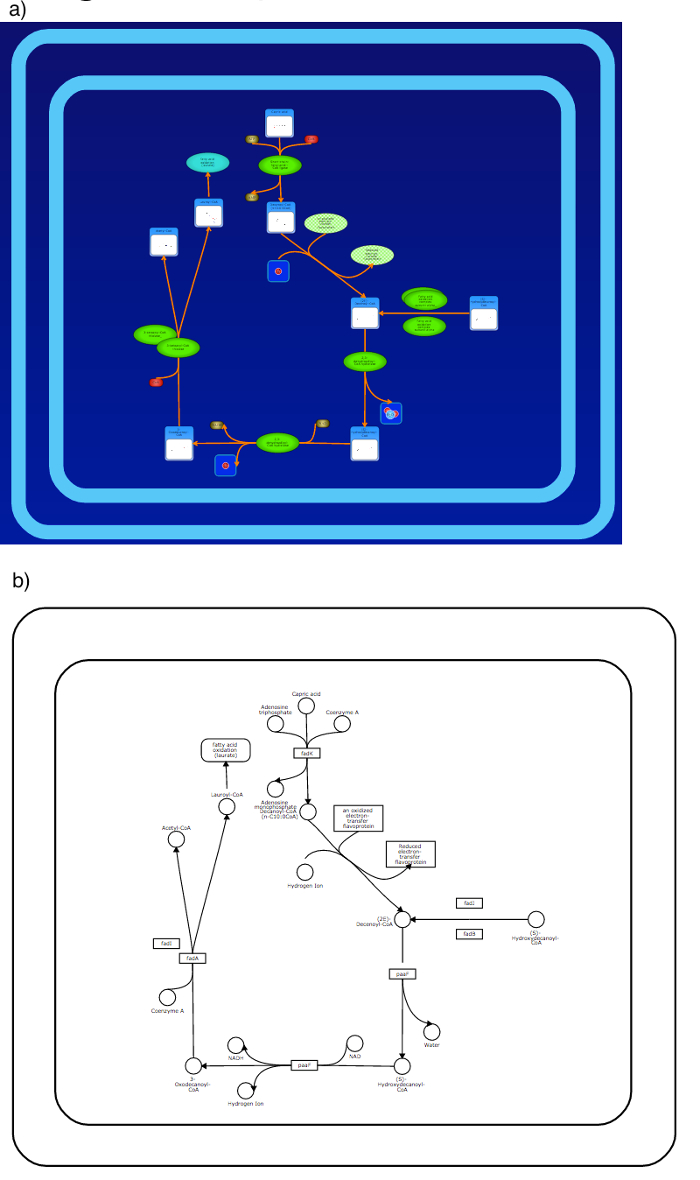
Abbildung 11: Bunte vs einfache Pathway Bilder. Bunte Wege können mit einer reichen biologischen Kontext entweder auf einem weißen oder blauen Hintergrund (a) erzeugt werden. Folat-Stoffwechsel ist hier dargestellt. Einfach, KEGG artige Wege kann auch sein, generated eine einfache Schwarz-Weiß - Darstellung unter Verwendung von (b). Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.
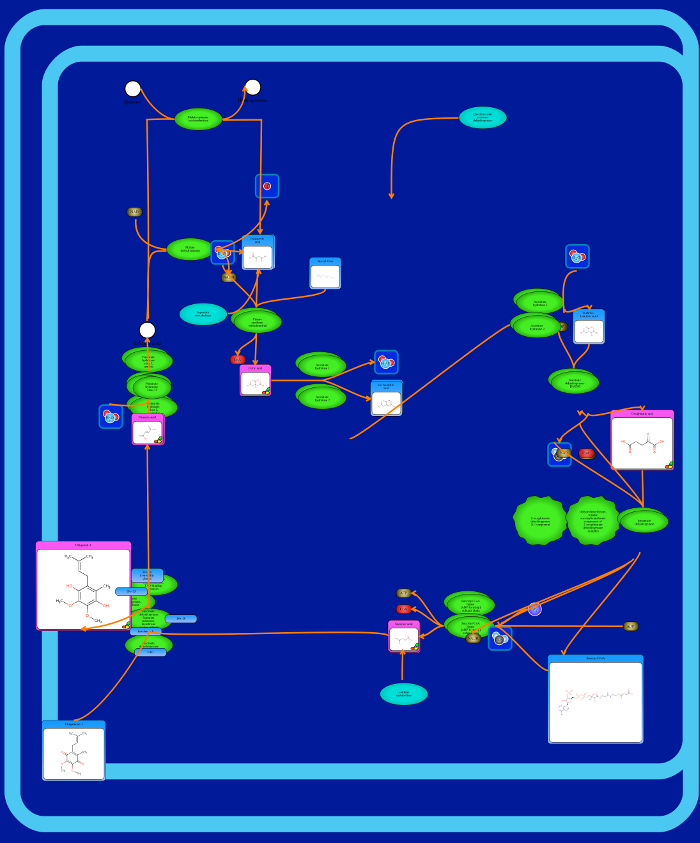
Abbildung 12: Suboptimale Pathway Bild. Ein Bild zeigt, was eine suboptimale Weg (TCA-Zyklus) aussieht. Overlapping Elemente und Kreuzung Kanten machen den Weg unverständlich. Dies kann passieren, wenn die Reaktionselemente sind nicht sorgfältig oder korrekt auf der Leinwand manipuliert. die Elemente Manipulieren für die Verbindungen (Large, Medium, Small Compound Visualisierung oder Drug-Visualisierung, Cofaktor-Visualisierung, einfache Unten, Links rechts oder von oben Visualisierung) führt zu mehreren Bild Inkonsistenzen mehr als zwei verschiedene Vorlagentypen zu haben. Die Template-Typen werden in Schritt 1.15.2 gezeigt. Nicht verbinden the Kanten wirkt sich auf die Strömung des Bildes zu einer schlechten Interpretationen des Weges führt. Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.
| PathWhiz | VANTED | PathVisio | Pathway Werkzeuge | Visant | |
| Web Server | Ja | Nein | Nein | Nein | Nein |
| installierbaren Programm | Nein | Ja | Ja | Ja | Ja |
| Protein Pathways | Ja | Ja | Ja | Nein | Ja |
| Metabolic Pathways | Ja | Ja | Ja | Ja | Ja |
| Speichern als PNG / JPG | Ja | Ja | Ja | Nein | Nein |
| Als HTML speichern | Ja | Nein | Nein | Ja | Nein |
| Speichern als SVG | Ja | Ja | Ja | Nein | Ja |
| Als PDF speichern | Ja | Ja | Ja | Ja | Ja |
| Speichern unter BioPAX | Ja | Ja | Ja | Ja | Ja |
| Speichern unter SBML | Ja | Ja | Ja | Ja | Ja |
| Speichern als SBGN-ML | Ja | Ja | Ja | Nein | Nein |
| Identifier Mapping | Ja | Ja | Ja | Ja | Ja |
| Membran-Rendering | Ja | Nein | Nein | Nein | Nein |
| Organell Rendering | Ja | Nein | Nein | Nein | Nein |
| Organ-Rendering | Ja | Nein | Nein | Nein | Nein |
| Colour Rich Images | Ja | Nein | Nein | Nein | Nein |
| Pathway Beschreibung | Ja | Nein | Nein | Ja | Nein |
| Pathway DB Verbindung | Ja | Nein | Ja | Ja | Nein |
| Pathway Inference | Ja | Nein | Nein | Ja | Nein |
| Expt. Daten-Einblendung | Nein | Ja | Nein | Ja | Ja |
| Pathway Analysis | Nein | Ja | Ja | Ja | Ja |
Tabelle 1: Funktionsvergleich. Ein Merkmal Vergleich von mehreren gemeinsamen Weg Bearbeitung / Rendering-Tools.
Ergänzende Datei 1: Beispiel für TCA Zyklus Beschreibung für PathWhiz Pathway. Klicken Sie hier , um die Ergänzungs Datei zum Download bereit .
Diskussion
Das hier beschriebene Protokoll für eine einfache Stoffwechselweg Schaffung kann (die TCA-Zyklus) eine Vielzahl von maschinenlesbaren, biologisch komplexe Wege für jede Spezies zu schaffen angepasst werden. Darüber hinaus beschreibt dieses Protokoll auch, wie man bestehende Wege von anderen Nutzern zu replizieren oder zu verbreiten. einen Weg Konstruieren dieses Werkzeug erfordert wiederholte Schritt-für-Schritt-Additionen von Reaktionen, Interaktionen Transportprozessen und Teilpfade, die jeweils durch sich überlappenden Elemente verbunden sind. Putting alles zusammen ermöglicht eine bunte, optisch ansprechende Pfaddiagramme zu erstellen, die erhebliche biologische Detail und nützliche biologische Kontext zu machen. Die Schritte in diesem Protokoll beschrieben sind, sind relativ einfach, und die Zeit, die einen Weg Diagramm aufzubauen, hängt von der Größe und Komplexität des Weges. Mit etwas Übung können die meisten Menschen eine hohe Qualität Weg-Diagramm-Darstellung von aus etwa 15-20 Reaktionen oder Verfahren eind mehrere zelluläre Komponenten in etwa 15 min. Ein brandneues Benutzer kann bis zu 30-40 Minuten einen Weg von ähnlicher Größe und Komplexität zu erzeugen. Die benötigte Zeit, einen Weg zu erzeugen, ist in etwa linear mit der Anzahl der Reaktionen / Prozesse, die gerendert werden müssen.
einen qualitativ hochwertigen Weg durch diese Web-basiertes Tool erstellen, hängt von der Qualität und Detail des Ausgangsmaterials (Wege aus Bücher, Online-Datenbanken, experimentelle Daten, von Hand gezeichnete Skizzen) und der Verwöhntheit des Weges "Künstler". Diejenigen, die zu erzeugen höhere Qualität Pfaddiagramme sollte ein besonderes Augenmerk auf die Abschnitte 1.11, 1.15 und 1.20 des Protokolls zahlen, da diese Abschnitte beschreiben die Erstellung und Bearbeitung von Reaktionselementen (Reaktanden / Produkte, Enzyme, Kanten, Bilder, Zoom-Boxen, Etiketten, und Membranen). Die besten Pfaddiagramme werden auf intelligente Weise Informationen aus möglichst vielen vorhandenen Darstellungen des Weges wie möglich einschließlich der in b gefunden amalgamierenooks, Plakate, Zeitungen und Online-Datenbanken. Ein weiterer Schlüssel für qualitativ hochwertige Wege zu erzeugen prüft sorgfältig die Richtigkeit der Reaktionen vor einer Reaktion zu schaffen (durch Abschnitt 1.11 des Protokolls). die Zeit und Mühe nehmen die Reaktanten, Produkte und Enzyme beteiligt sind (von denen viele bereits existieren in PathWhiz der großen Datenbank) sind für jede Art richtig sicherzustellen ist sehr wichtig. Es ist auch wichtig, die zelluläre Lokalisation der Reaktionen zu sein und Schlüssel zellulären oder subzellulären Komponenten einzubeziehen, um den richtigen biologischen Kontext bereitzustellen. Dies kann durch die Überprüfung und bestätigende die Reaktion durch Online-Datenbanken wie UniProt erfolgen. Nachdem alle erforderlichen Informationen zur Hand, zusammen mit einer rauen, von Hand gezeichnete Skizze des Weges stark geschaffen werden, werden Fehler und der Gesamtzeit auf Zeichnung oder Rendering verbrachte reduzieren.
Wie zu erwarten größere und komplexere Wege länger dauern wird, insbeson zu machensonders wenn die gewünschten Elemente und Prozesse sind nicht bereits in PathWhiz Datenbank. Wenn mit größeren Wegen gearbeitet wird, ist es meist sinnvoll, von der Autosave-Modus auf den manuellen Sparmodus zu wechseln, um eine lange Verzögerung zwischen den Aktionen zu verhindern. Wenn eine Bahn replizierende, der Benutzer kann die Menge an Zeit warten, bis der Weg erzeugt auf der Anzahl von Elementen in dem Pfad hängt werden. Die meisten Wege sind in ca. 1-2 min repliziert werden. Wenn Sie einen Weg, erfolgreich Wiedergabe des neu propagierten Weg ausbreitende hängt davon ab , wie ähnlich sich die beiden Arten, wie PathWhiz BLAST 17 - Sequenz verwendet sucht homologe Enzyme zwischen den Arten zu finden. Größere Wege langsamer sein wird fortzupflanzen, da BLAST auf eine größere Anzahl von Enzymen ausgeführt werden müssen. Versuch Wege zwischen signifikant unterschiedlichen Spezies zu propagieren (sagen wir zwischen Hefe und Menschen) führt in Bahnen mit einer Anzahl von unbekannten Proteinen gemacht werden. Diese "weitläufig" propagated Wege werden in der Regel zusätzliche manuelle Bearbeitung erfordern. Aufgrund der sehr visuelle Art von Pfaddiagramme und dem Detail, das auf einen Weg gebracht werden kann, ist es immer eine gute Idee, auf einem Computer mit einem recht großen Bildschirm (> 20 Zoll oder> 50 cm) und eine gute Internetverbindung zu arbeiten (> 5 mbps).
Wenn die Probleme mit Rendering oder einen Neuaufbau des Bildschirms auftreten, kann der Benutzer eine kleine Menge zur Fehlerbehebung zu tun. Wenn ein großer, komplexer Weg zu aktualisieren, zu lange dauert, kann der Benutzer auf die Seite aktualisieren. Wenn ein Weg nicht wie erwartet propagieren, kann der Benutzer eine manuelle Bearbeitung zu tun, um sicherzustellen, dass alle Elemente korrekt angezeigt werden. Auch als ein spezielleres Beispiel, wenn Elemente einer Reaktion nicht angezeigt werden, kann der Benutzer sicherstellen muss, dass alle Elemente oder Enzyme geeignet ausgewählt, und die Kanten werden nicht verborgen. Die "Hilfe" auf der Haupt-Header kann nützlich sein, wenn ein Problem auftritt. Ein Tutorial istunter der "Tutorial" Registerkarte und einer Anleitung ist unter "Benutzerhandbücher" Tab zur Verfügung. Beide erklären viele der Funktionen des Werkzeugs im Detail. Das Benutzerhandbuch verwendet werden können, mögliche Einschränkungen für ein bestimmtes Merkmal, wie zu beheben oder zu erklären, wenn ein Benutzer einen Weg blockiert und später möchte sie zu bearbeiten.
Wie durch dieses Protokoll hervorgehoben und durch die in den beigefügten Figuren bereitgestellten Beispiele, bietet dieses Tool eine Reihe einzigartiger Merkmale gefunden in keiner (oder die meisten) anderen Weg Zeichenwerkzeuge (siehe Tabelle 1). Erstens, es ist vollständig web-basierte und vollständig plattformunabhängig. Zweitens unterstützt es die Darstellung und einfache Erzeugung von bunten, biologisch komplexe, optisch ansprechend, vollständig verlinkte Pfaddiagramme , die auch in maschinenlesbare Formate (BioPAX, SBGN-ML 18, 19 SBML, PWML 14) umgerechnet werden können. Drittens Pfaddiagramme Gattungented von diesem Tool können durchsucht, durchsucht, ausgewählt und leicht durch eine einfach zu bedienende Online-Datenbank und Anzeigen Schnittstelle erkundet werden. Viertens wird die Web-Tool entwickelt Community Weg Beiträge zu unterstützen, so dass für "Weg Crowd Sourcing", die den Austausch und die Erzeugung von neuen Wegen und neuen Weg Elemente fördert.
Pathways von dieser Web-basiertes Tool erzeugt wird, kann für eine Vielzahl von Anwendungen eingesetzt werden. Detailreiche, vollständig mittels Hyperlink verwiesen Wege leicht Organismus spezifische Datenbanken für Proteomik, Metabolomik oder Anwendungen der Systembiologie integriert werden können. Internet zugänglichen Wege sind besonders nützlich für die Bildungs- und Ausbildungszwecken, da die zur Verfügung stehenden Informationen über Web-basierte Bilder sind oft viel größer als das, was über ein statisches Bild oder durch ein einziges Lehrbuch oder Journal-Seite angezeigt werden. Diese Web-basierte Tool unterstützt auch die Erzeugung von Pathway Darstellungen, die besser geeignet für den Druck und die Veröffentlichung sind.viele Bilder erzeugt von dieser Web-basiertes Tool erscheinen in Zeitungen, Poster und Präsentationen Als Folge. Exportieren Wege in textbasierten Datenaustausch Dateiformate (wie BioPAX und SBML) ermöglicht Wege mit dieser Web-Server generiert, um direkt in Computeranalyse für die Systembiologie oder metabolische Modellierungsanwendungen eingesetzt werden. Das Inverkehrbringen von Vermehrungswege zwischen den Arten ermöglichen Aussagen über biologische Prozesse werden, vor allem bei jenen Arten, die erst kürzlich sequenziert wurden. Obwohl nicht alle vorhandenen Wege derzeit in PathWhiz existieren, setzt seine öffentlichen Weg Datenbank zu wachsen, zu der Entstehung neuer, Crowdsourcing Weg Sammlungen führt. Diese Sammlungen werden nicht nur leicht erweiterbar auf neue Arten, sie werden hoffentlich zu einem tieferen Verständnis ihrer einzigartigen Biologie und Biochemie führen.
Offenlegungen
Die Autoren haben nichts zu offenbaren.
Danksagungen
Die Autoren möchten die kanadischen Institutes of Health Research (CIHR) und Genome Alberta, eine Abteilung von Genome Canada, für die finanzielle Unterstützung danken.
Materialien
| Name | Company | Catalog Number | Comments |
| Computer with colour screen | N/A | N/A | >20 inches or >50 cm |
| Internet connection | N/A | N/A | >5 mbps |
| Modern web browser | N/A | N/A | Google Chrome (v. 31 and above), Internet Explorer (v. 9 and above), Safari (v. 7 and above), Opera (v. 15 and above) and Firefox (v. 23 and above) |
Referenzen
- Michal, G. On representation of metabolic pathways. Biosystems. 47 (1-2), 1-7 (1998).
- Kanehisa, M., Sato, Y., Kawashima, M., Furumichi, M., Tanabe, M. KEGG as a reference resource for gene and protein annotation. Nucleic Acids Res. 44 (D1), D457-D462 (2016).
- Karp, P., Riley, M., Paley, S. The MetaCyc Database. Nucleic Acids Res. 30 (1), 59-61 (2002).
- Kelder, T., et al. WikiPathways: building research communities on biological pathways. Nucleic Acids Res. 40 (Database issue), D1301-D1307 (2011).
- Croft, D., et al. The Reactome pathway knowledgebase). Nucleic Acids Res. 42 (Database issue), D472-D477 (2014).
- Karp, P. D., et al. Pathway Tools version 13.0: integrated software for pathway/genome informatics and systems biology. Brief Bioinform. 11 (1), 40-79 (2010).
- Van Iersel, M. P., et al. Presenting and exploring biological pathways with PathVisio. BMC Bioinformatics. 9, 399 (2008).
- Demir, E., et al. The BioPAX community standard for pathway data sharing. Nat. Biotechnol. 28 (9), 935-942 (2010).
- Shannon, P., et al. Cytoscape: A Software Environment for Integrated Models of Biomolecular Interaction Networks. Genome Res. 13 (11), 2498-2504 (2003).
- Salomonis, N., et al. GenMAPP 2: new features and resources for pathway analysis. BMC Bioinformatics. 8, 217 (2007).
- Elliott, B., et al. PathCase: pathways database system. Bioinformatics. 24 (21), 2526-2533 (2008).
- Hu, Z., et al. VisANT 3.0: new modules for pathway visualization, editing, prediction and construction. Nucleic Acids Res. 35 (Web Server), W625-W632 (2007).
- Jewison, T., et al. SMPDB 2.0: Big Improvements to the Small Molecule Pathway Database. Nucleic Acids Res. 42 (D1), D478-D484 (2013).
- Pon, A., et al. Pathways with PathWhiz. Nucleic Acids Res. 43 (W1), W552-W559 (2015).
- Sajed, T., et al. ECMDB 2.0: A richer resource for understanding the biochemistry of E. coli. Nucleic Acids Res. 44 (D1), D495-D501 (2015).
- Wishart, D., Mandal, R., Stanislaus, A., Ramirez-Gaona, M. Cancer Metabolomics and the Human Metabolome Database. Metabolites. 6 (1), 10 (2016).
- Altschul, S. F., Gish, W., Miller, W., Myers, E. W., Lipman, D. J. Basic local alignment search tool. J. Mol. Biol. 215 (3), 403-410 (1990).
- Le Novere, N., et al. The Systems Biology Graphical Notation. Nat. Biotechnol. 27 (8), 735-741 (2009).
- Hucka, M., et al. The systems biology markup language (SBML): a medium for representation and exchange of biochemical network models. Bioinformatics. 19 (4), 524-531 (2003).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten