Method Article
A ferramenta Web para caminhos biológicos que gera alta qualidade de leitura mecânica
Neste Artigo
Resumo
PathWhiz é uma ferramenta de desenho abrangente, via on-line para gerar caminhos bioquímicos e biológicos. Ele usa bases de dados acessíveis ao público e facilmente paletas expansíveis que consistem em componentes pré-desenhados via. Este protocolo descreve como construir facilmente novos caminhos, replicar e editar percursos existentes, e propagar percursos previamente elaborados a diferentes organismos.
Resumo
PathWhiz é um servidor web construído para facilitar a criação de diagramas coloridos e interativos, visualmente agradável via que são ricos em informação biológica. As vias gerados por esta aplicação on-line são legíveis por máquina e totalmente compatível com praticamente todos os navegadores web e sistemas operacionais de computador. Ele usa uma interface de desenho especialmente desenvolvido, web-enabled via que permite a selecção e colocação de diferentes combinações de entidades biológicas e bioquímicas pré-desenhadas para descrever reações, interações, processos de transporte e eventos de ligação. Esta paleta de entidades consiste em compostos químicos, proteínas, ácidos nucleicos, membranas celulares, estruturas subcelulares, tecidos e órgãos. Todos os elementos visuais em que pode ser interativamente ajustada e personalizada. Além disso, porque esta ferramenta é um servidor web, todas as vias e elementos da via são acessíveis ao público. Este tipo de via "crowdsourcing" significa que PathWhizjá contém uma grande e rapidamente crescente coleção de percursos previamente elaborados e elementos da via. Aqui nós descrevemos um protocolo para a criação rápida e fácil de novas vias ea alteração das vias existentes. Para facilitar ainda mais a edição via e criação, a ferramenta contém funções de replicação e propagação. A função de replicação permite vias existentes para serem usados como modelos para criar ou editar novos caminhos. A função de propagação permite tirar uma via existente e automaticamente propagá-la entre espécies diferentes. Pathways criados com esta ferramenta pode ser "re-denominados" em formatos diferentes (KEGG-like ou livro-texto like), colorida com diferentes origens, exportados para BioPAX, SBGN-ML, SBML ou PWML formatos de troca de dados, e baixadas como PNG ou SVG imagens. Os caminhos podem ser facilmente incorporado em bases de dados online, integrados em apresentações, cartazes ou publicações, ou utilizados exclusivamente para visualização on-line e exploração. esta protocol foi aplicado com sucesso para gerar mais de 2.000 diagramas da via, que agora são encontrados em muitos bancos de dados on-line incluindo HMDB, drugbank, SMPDB e ECMDB.
Introdução
diagramas via biológica são como modelos para cientistas da vida. Eles são, talvez, as rotas mais conciso e informativo para descrever processos biológicos e as ligações contextuais entre genes, proteínas e metabolitos. Isso ocorre porque as imagens são muito mais eficientemente processada e frequentemente muito mais facilmente compreendida pelos seres humanos do que de texto 1. A qualidade, detalhe, e o conteúdo de diagramas de vias pode variar consideravelmente. Essas diferenças muitas vezes dependem da finalidade da via e as habilidades do artista percurso. Caminhos criados para fins educacionais, tais como gráficos de parede ou livros são muitas vezes criados por artistas profissionais. Como resultado, estes diagramas via são muito mais agradável visualmente e oferecem detalhes consideravelmente mais biológica com descrições completas de estruturas de metabolito, componentes subcelulares, estruturas de células, tecidos e órgãos. Estas representações "manual" muitas vezes incluem notas detalhadas ecomentários. Por outro lado, os diagramas das vias projetadas para aplicações de internet, muitas vezes tem que sacrificar a arte e riqueza visual em favor de máquinas legível diagramas "fiação" simplificados. Esses diagramas de estrutura de arame são mais facilmente e com hiperlinks mapeados na imagem. Diagramas via simplificados são a base para tais bancos de dados via on-line populares como KEGG 2, metacyc 3, WikiPathways 4 e 5 Reactome. O surgimento de bases de dados da via de computador compatível, também conduziu ao aparecimento de ferramentas de desenho por computador via compatível. Em outras palavras, a pessoa não tem que ser um artista profissional ou um programador profissional para gerar diagramas das vias utilizáveis. Por exemplo, as ferramentas de Caminho de BioCyc 6 e software PathVisio de Wikipathway 7 permitem ao usuário gerar livremente e compartilhar percursos de leitura óptica em BioPAX 8 and / ou formato HTML. Além disso, há uma série de outros pacotes de freeware stand-alone, bem como pacotes comerciais que suportam a geração de várias vias legível por máquina, fio-frame, como Cytoscape 9, GenMAPP 10, PathCase 11 e visant 12.
A simplificação dos diagramas via internet em grande parte, cresceu a partir das limitações históricas encontradas em muitos navegadores da web e ferramentas de renderização baseadas na web. No entanto, avanços significativos em tecnologias web têm sido feitos nos últimos anos. Isto sugere que pode ser possível para gerar diagramas interativos, internet compatível via que são tão colorido, tão esteticamente agradáveis e tão biologicamente completos como aqueles encontrados nos livros didáticos. Esse trabalho levou ao desenvolvimento de PathWhiz. PathWhiz foi implementado usando uma Ruby on Rails (http://rubyonrails.org, versão 4.2.0) web framework incorporando um banco de dados MySQL relacional (https://www.mysql.com, versão 5.1.50) para gerir todos os dados da via, incluindo relações entre entidades, referências externas, descrições, especificações de visualização e estruturas químicas. O cliente web front-end é controlada por Ruby on Rails combinados com Backbone.js (http://backbonejs.org, versão 1.0.0) como o framework web front-end para o editor.
Originalmente desenvolvido para a curadoria da-somente humano pequena molécula Caminho de Dados (SMPDB) 13, PathWhiz 14 já foi estendido para suportar geração caminho para muitos outros organismos e para funcionar como uma imagem Caminho geral e base de conhecimento. Em particular, esta ferramenta de web permite a criação de toda a gama de vias bioquímicas / biológicos incluindo metabólica, a interacção de proteínas, sinalização molecular, fisiológico, e as vias de droga / doença. Esta ferramenta de desenho via diferente da maioria dos outrosferramentas de geração de caminho de três maneiras principais: 1) é um servidor web, em vez de, um pacote de software instalável stand-alone; 2) que suporta a geração e fácil visualização interactiva de compostos químicos, proteínas, ácidos nucleicos, membranas celulares, estruturas subcelulares, tecidos e órgãos; e 3) que permite aos usuários para emprestar facilmente, construir ou melhorar em cima do trabalho de outros usuários, permitindo assim "crowdsourced" geração via. Como um servidor web, ele tem várias vantagens sobre download, ferramentas de software específicos da plataforma. Em particular, é compatível com qualquer plataforma, sistema operacional e navegador moderno. Além disso, ele não requer que o usuário se registre para começar a criar um caminho (embora os usuários podem criar livremente uma "conta privada", a fim de acompanhar e controlar a acessibilidade das vias que criam). Talvez o aspecto mais atraente da presente ferramenta é a quantidade de detalhe biológicos e bioquímicos que podem ser facilmente adicionadas emqualquer via através de uma paleta de imagens pré-renderizados e uma extensa base de dados de proteínas e dados bioquímicos. Isso permite que ambos os "não-artistas" e "não-programadores" para criar facilmente vias coloridas, esteticamente agradável e ricamente detalhados que são na web compatível e totalmente legível por máquina. Uma comparação mais pormenorizada entre PathWhiz e outras ferramentas de desenho via é fornecida na Tabela 1.
Um número de bancos de dados de ciências da vida popular, já usou essa ferramenta de desenho via para criar específicos de banco de dados, on-line, diagramas via interativos. Por exemplo, o banco de dados Escherichia coli Metaboloma (ECMDB) 15 recentemente atualizou sua biblioteca percurso com mais de 1.650 percursos desenhados com a ferramenta baseada na web. Cada caminho no ECMDB é agora apresentado como um mapa ricamente colorido, totalmente hiperligada imagem com metabolitos e estrutura da proteína descrições detalhadas, assim como uma simplificado preto e branco KEGG-like diagrama fio. Esta actualização via de grande escala levou à descoberta de diversos metabolitos intermediários que não tinha sido previamente incluída em outras bases de dados de Escherichia coli metabólicas. Outras bases de dados, tais como a base de dados Metaboloma Humana (HMDB) 15, não só dependem de vias PathWhiz que mostrem e descrevem processos metabólicos e de sinalização, mas também para descrever as alterações metabólicas envolvidas em doenças tais como o cancro. HMDB inclui atualmente 101 vias metabólicas, 376 vias de luta contra a droga, 233 percursos associados à doença e 16 vias de sinalização, todos gerados através desta ferramenta web.
O protocolo que se segue descreve detalhadamente como PathWhiz pode ser usado para criar facilmente, replicar e propagar vias bioquímicas para uma variedade de propósitos e aplicações.
Protocolo
1. Caminho Generation
- Ir para http://smpdb.ca/pathwhiz usando qualquer navegador moderno.
- Na barra de menu, selecione "Try Me!" usar a ferramenta como um convidado (percursos desenhados será público), "Sign Up" para registrar uma conta (percursos desenhados só será editado pelo usuário), ou "Log In", para usar uma conta existente. Para se inscrever para uma conta, vá para o passo 1.2.1, caso contrário, vá para o passo 1.3.
- Para se inscrever para uma conta, preencha o formulário com um e-mail, nome, filiação, cidade, país, e 8 caracteres senha mínimo, com confirmação de senha, clique em "Sign Up".
- Se não for redireccionado automaticamente para o índice de caminho, selecione o link "Empate" na barra de menu. Esta página índice vai mostrar uma tabela de vias existentes. Clique no botão "Novo Caminho" para iniciar um novo caminho.
- Digite o nome da via a ser desenhado (por exemplo Ciclo TCA).
- Escolha o tipo de percurso para desenhar a partir da lista suspensa (por exemplo, Doença / Drogas / Metabólico / Sinalização / fisiológica). É possível gerar vias químicas, bem como proteínas de sinalização, vias de ADN / ARN, ou vias de interacção proteína-proteína.
- Pesquisar e escolher uma espécie, digitando o nome científico do organismo na caixa de preenchimento automático (por exemplo, tipo Escherichia coli). Se o nome do organismo não é encontrado na lista suspensa, clique no botão "Novo" e siga os passos 1.6.1 a 1.6.5 para adicionar um novo organismo, caso contrário, avance para o passo 1.7.
- Digite o nome científico da espécie (por exemplo, Gorilla gorilla) e o nome comum (por exemplo Gorilla).
- Escolha a classificação das espécies da lista como Eucarioto ou Prokaryote para baixo e insira o ID taxonomia da taxonomia NCBI (por exemplo, 9593).
- Clique no botão "Criar Espécies".
- Introduza um compreensíveisA inscrição sive para a via. Ver Documento Suplementar 1 para um exemplo de uma descrição. Quanto mais completa a descrição, o que é mais fácil para pesquisar e encontrar o caminho, ea mais popular o percurso será entre os usuários.
- Adicionar referências externas para o caminho clicando no botão "Add Reference" e seguindo os passos 1.8.1 a 1.8.2, caso contrário, vá para o passo 1.9.
- Digite o ID PubMed (que irá gerar automaticamente o texto da citação) ou adicione o texto da citação manualmente (e deixe o campo PubMed ID vazia) (por exemplo PMC545700 ou Kang Y, et al. Análise da expressão Genome-Wide Indica que FNR de Escherichia coli K-12 Regula um grande número de genes de função desconhecida J. Bacteriology 187 (3):.. 1135-1160 doi:. 10,1128 / JB.187.3.1135-1160 2005).
- Repita este processo até que todas as referências desejadas foram adicionados.
NOTA: Na maioria dos casos, são necessários apenas uma ou duas referências. No entanto, o mais meta-dados de um can adicionar, mais populares da via será.
- Clique no botão "Criar Caminho". Uma tela quadriculada branca aparecerá com uma barra de menu cinza. Este é o lugar onde o desenho ocorre.
- Clique no link "Adicionar processo" e selecione "Adicionar Reaction" para adicionar o primeiro visualização do processo. Por exemplo, começar com uma reacção que mostra a conversão de ácido oxaloacético ao ácido cítrico, através da enzima sintase de citrato.
NOTA: Os processos são eventos ou atividades biológicas. Os processos podem ser divididos em quatro categorias: reacções, eventos de transporte, interações e eventos de ligação. Neste exemplo, uma reacção é mostrada, mas os mesmos princípios aplicam-se a adição de qualquer tipo de processo. - Pesquisar as reações existentes, introduzindo reagentes, produtos ou enzimas na caixa de preenchimento automático (por ex. "Acid oxaloac�ico" ou "Acetil CoA"). Percorra as reações existentes para encontrar a reação desejada, selecione-o e ir direto para step 1,12, ou, se a reação desejada não for encontrado, clique no botão "Novo Reaction" e siga os passos 1.11.1 a 1.11.11 para adicionar um novo modelo de reação ao banco de dados.
- No formulário de nova reação, clique no botão "Adicionar elemento Esquerda" para adicionar um reagente no lado esquerdo da reação. As reacções são escritas como equações de reacção com um lado esquerdo e um lado direito.
- Selecione o tipo de estequiometria e dos elementos (Composto / Protein Complex / elemento de coleção Ácido / Nucleic / Limite Element). Procure o elemento por nome na caixa de preenchimento automático (por exemplo, "Acid oxaloac�ico"). Selecione o elemento desejado. Vá para a etapa 1.11.3 ou, se o elemento desejado não for encontrado, clique no botão "Novo" e siga o passo 1.11.2.1.
- No novo elemento formulário, preencha os campos apropriadamente e salve.
- Repetir passos 1.11.1 e 1.11.2 para cada elemento envolvido no lado esquerdo da reacção (por exemplo, adicionar mais dois compostos, "ÁsTyl-CoA "e" Água ").
- Seleccione a direcção da via a partir da lista drop-down (por exemplo, escolha a seta dirigida da esquerda para a direita).
NOTA: Seta representações incluem esquerda para a direita, direita para a esquerda, e reversível. - Com o elemento esquerdo da reacção agora concluída, clique no botão "Adicionar Elemento Right" para adicionar um produto.
- Selecione a estequiometria e tipo de elemento como no passo 1.11.2. Procure o elemento por nome na caixa de preenchimento automático (por exemplo, "O ácido cítrico") e selecione o elemento desejado. Se o elemento desejado não for encontrado, clique no botão "Novo" e siga o passo 1.11.2.1.
- Repita os passos 1.11.5 e 1.11.6 para cada elemento envolvido no lado direito da reacção (por exemplo, adicionar "de iões de hidrogénio" e "Coenzima A").
- Depois de os reagentes e produtos foram gerados, clique no botão "Adicionar Enzima" para adicionar uma enzima para a reacção.
- MarRCH para a enzima digitando seu nome na caixa de preenchimento automático (por exemplo, "citrato sintase") e selecione a enzima desejada. Se a enzima não existe no banco de dados, clique no botão "Novo" e siga os passos 1.11.9.1 a 1.11.9.7.
- Ao criar uma nova enzima, use o formulário nova enzima para preencher o nome enzima e espécies nas guias correspondentes (por exemplo, Nome: citrato sintase Espécies: Escherichia coli).
- Clique no botão "Adicionar Protein" para adicionar as informações espécie-específico (ou seja, a sequência e estrutura quaternária informação) para esta enzima.
- Preencha a estequiometria e procurar a proteína por nome (citrato sintase), o nome do gene (gltA), ou UniProt ID na caixa de preenchimento automático. Selecione a proteína desejada ou, se a proteína desejada não for encontrado, clique no botão "Novo" e siga o passo 1.11.9.3.1.
- No formulário de nova proteína, preencha os campos apropriadamente e clique no4;. Botão Criar Protein "campos obrigatórios incluem Nome e UniProt ID O resto dos campos são opcionais..
- Use os botões "Adicionar Modificações" e / ou "Adicionar Cofatores" para adicionar modificação da proteína ou enzima cofatores, se necessário. Neste caso, não são necessários. Preencha os campos de forma apropriada.
- Clique no botão "Adicionar Biológica Estadual" para adicionar uma localização subcelular da enzima.
- Procure o estado biológico desejado, digitando o espécies, tipo de célula, e / ou localização subcelular na caixa de preenchimento automático (por exemplo, "Escherichia coli, Cell, citosol"). Selecione o estado desejado ou, se o estado desejado não for encontrado, clique no botão "Novo" e vá para a etapa 1.11.9.6.1.
- No formulário Nova Biológica Estadual, preencher os campos adequadamente. Se as espécies, etc, não é encontrado, usar o botão "Novo" tal como descrito acima nos passos 1.6.1 a 1.6.5. Uma vez que fazerne, clique no botão "Criar Biológica Estadual".
- Clique no botão "Criar Enzyme" para salvar a enzima.
- Repetir passos 1.11.8 a 1.11.9 adicionar enzimas adicionais, se necessário.
NOTA: A reacção pode ter muitas enzimas diferentes associados, e as enzimas não tem de pertencer à mesma espécie ou estado biológico (desde que os reagentes e produtos são os mesmos). Ao desenhar uma reação é sempre possível escolher qual das suas enzimas associadas deve ser exibido. - Clique no botão "Criar Reaction" para salvar a nova reação.
- Especificar localização subcelular da reacção (por esta via particular) no campo Estado biológica, se conhecida.
- Selecione a orientação desejada para a primeira prestação a reação do listas suspensas (esquerda para a direita, direita para a esquerda, Horizontal / Vertical) (por exemplo, selecione esquerda para a direita e horizontal).
- Clique no botão "Criar Reaction". A reação recém-criado será exibido na tela de desenho.
- Usando um mouse ou touch pad, clique e arraste para reposicionar os elementos de reação na tela conforme desejado. Siga passos 1.15.1 a 1.15.5 para reposicionamento e edição de elementos de reacção (compostos, bordas e / ou enzimas) da reacção recentemente criado.
- Selecione um ou mais elementos (compostos, arestas e / ou enzimas) ao mesmo tempo por um ou outro single-clicando em cada elemento de adicioná-los um por um para a seleção atual, ou selecione todos os elementos usando o cursor para arrastar um caixa em torno dos elementos relevantes. Os elementos seleccionados serão de cor vermelha.
- Arraste os elementos selecionados em toda a tela para reposicioná-los na área desejada (geralmente no centro da tela) usando um mouse ou touch pad. Desmarque elementos, clicando sobre eles uma segunda vez, ou clicando sobre a tela em branco.
- Clique duas vezes em um composto químico(Ele vai tornar-se rodeado por uma caixa cinza pontilhada) para acessar uma barra lateral pop-up que irá aparecer no lado direito da tela. Use esta barra lateral para editar os detalhes do modelo, estado biológico, Z-index e reacção completa.
- Escolha um tipo de modelo de entre as opções disponíveis (grande, médio ou pequeno Composto Visualization, grande, médio ou pequeno Drogas Visualization, Cofator Visualization, inferior simples, Esquerda ou Direita Top Visualization). Adicionar um Estado Biológica e editar o índice z, se necessário.
NOTA: É melhor para manter o tipo de modelo consistente ao longo de todo o percurso, exceto para diferenciar entre os tipos de compostos, drogas ou seja vs. metabolitos.
- Escolha um tipo de modelo de entre as opções disponíveis (grande, médio ou pequeno Composto Visualization, grande, médio ou pequeno Drogas Visualization, Cofator Visualization, inferior simples, Esquerda ou Direita Top Visualization). Adicionar um Estado Biológica e editar o índice z, se necessário.
- Clique duas vezes em uma proteína / enzima (que irá tornar-se rodeado por uma caixa cinza pontilhada) para acessar uma barra lateral pop-up (aparecerá no lado direito da tela). Use esta barra lateral para editar o modelo, estado biológico, o índice z, proteína detalhes complexos, e os detalhes completos de reação.
- Escolha um tipo de modelo a partir das opções disponíveis (Enzyme monômero, dímero ou tetrâmero, não exibam rótulo, etiqueta Protein ou o rótulo Subunidade, transportador, receptor ou repressor). Adicionar um Estado Biológica e editar o índice z, se necessário.
NOTA: As cores diferentes são fornecidos para distinguir entre proteínas; a cor padrão é definido como verde.
- Escolha um tipo de modelo a partir das opções disponíveis (Enzyme monômero, dímero ou tetrâmero, não exibam rótulo, etiqueta Protein ou o rótulo Subunidade, transportador, receptor ou repressor). Adicionar um Estado Biológica e editar o índice z, se necessário.
- Para editar as informações de todo o processo, clique duas vezes em qualquer um dos seus elementos (eles vão tornar-se rodeado por uma caixa cinza pontilhada) e acessar o link "Editar selecionado" na barra de menu secundário (cinza). Duas opções serão exibidas: "Editar " e "Editar ".
NOTA: "Editar " irá direcionar os usuários para a barra lateral para o elemento correspondente. "Editar " vai apresentar opções para "Editar Detalhes", mudar a direção em que o processo é processado (horizontal / vertical e esquerda / direita), ou voltar a ligar as bordas (tambémHorizontal / Vertical e Esquerda / Direita). Ao clicar no link Editar Detalhes levará a uma tela onde os detalhes da reação e todos os seus elementos podem ser editadas de uma vez, incluindo os Estados Biológica, modelos, e z-índices. As enzimas podem ser adicionados ou removidos a partir do visor, e os elementos e as bordas podem ser escondidas, se desejar. Por exemplo, clique duas vezes o elemento de iões de hidrogénio, e em seguida, clique em "Editar Selecionados". Coloque o cursor sobre o "ácido Editar acetil-CoA + oxaloac�ico ácido + água → cítrico + Coenzima A + Hydrogen Ion" e selecione "Editar detalhes". Adicionar um Estado Biológica para cada composto (Escherichia coli, celular, citoplasma). Uma vez feito isso, vá até a parte inferior da página e clique no botão roxo "Update Reaction". Só as enzimas que já estão associados com esta reacção pode ser adicionado à trilha nesta fase. Se novas enzimas precisam ser adicionados ao modelo de reação, voltar para o índice de reacção (na guia "Processos"), encontrar a reação desejada,e adicioná-los lá. - Editar as bordas reacção através de um único clique ou um clique duplo.
- Seleccionar as bordas de reacção para manipulá-los na mesma maneira como os compostos e proteínas. Clique e arraste na borda para mover a borda inteira.
NOTA: O início e fim pontos também podem ser clicadas e arrastadas para alterar o comprimento da aresta. Quando o ponto de início / fim de uma borda foi selecionada, o "botão" associado pode ser ajustado para controlar a direção ea curvatura do ponto de início / fim. Para adicionar nós adicionais para a borda, selecione a borda e observe o retângulo azul que aparece. Clique na metade superior do rectângulo para adicionar um nó e clique na metade inferior para remover um nó. - Clique duas vezes no ponto de início / fim de uma vantagem para o ciclo do ponto de início / fim através da seta apontada, seta bloqueio, e há opções de seta.
- Seleccionar as bordas de reacção para manipulá-los na mesma maneira como os compostos e proteínas. Clique e arraste na borda para mover a borda inteira.
- Selecione um ou mais elementos (compostos, arestas e / ou enzimas) ao mesmo tempo por um ou outro single-clicando em cada elemento de adicioná-los um por um para a seleção atual, ou selecione todos os elementos usando o cursor para arrastar um caixa em torno dos elementos relevantes. Os elementos seleccionados serão de cor vermelha.
- Uma vez que a primeira reacção foi desenhado como desejado, seleccionar um produto da reacção (por exemplo, ácido cítrico)clicando duas vezes nele (note a mudança de cor para vermelho), a fim de adicionar o próximo processo sobre este produto de reacção (neste caso ácido cítrico para cis ácido -Aconitic via aconitato hidratase).
- Uma vez selecionado, clique na guia "Adicionar processo" e clique na opção "Adicionar Reaction".
- Adicionar outra reacção para o ciclo do TCA (neste exemplo em particular), repetindo o processo para a adição de uma reacção (passos 1,11-1,15). Desde essa reação está sendo construído fora de um produto de reacção existente, apenas reações que contêm o elemento selecionado irá aparecer.
- Adicione as reações restante do ciclo TCA seguindo os passos 1,11-1,15 para cada reação.
- Uma vez que todas as reações foram adicionados, adicionar elementos visuais, como membranas, DNA, tRNAs, organelas subcelulares, órgãos, tecidos, caixas de zoom ou rótulos clicando no botão "Adicionar Visual Elemento" link e selecionar um dos "Adicionar Membrane", "Adicionar imagem "," Adicionar Zoom Box "ou"; Adicionar etiqueta "opções Siga os passos 1.20.1 a 1.20.4 para adicionar elementos visuais..
- Clique na opção "Adicionar Membrane" para adicionar uma membrana celular. Editar esta membrana clicando duas vezes nele para acessar a barra lateral. Escolha o tipo de membrana no campo do modelo localizado na barra lateral. Selecione a opção "membrana fechado" para renderizar uma membrana in a box.
- Clique na opção "Adicionar Imagem" para adicionar uma imagem actualmente existentes na base de dados PathWhiz (imagens padrão incluem órgãos, organelas, e tecidos). Editar a imagem clicando duas vezes nele para acessar uma barra lateral. Editar opções são auto-explicativos e incluem dimensionamento profundidade com o z-index, ampliar / escalar para baixo, e girar para a esquerda / rodar para a direita.
- Clique na opção "Adicionar Zoom Box" para adicionar uma caixa de zoom para uma determinada imagem.
- Clique na opção "Adicionar rótulo" para adicionar um rótulo de texto. Editar o rótulo clicando duas vezes nele para acessar a sua barra lateral. opções de edição incluem o modelo de etiqueta, texto ez-index.
NOTA: O link "Adicionar vazia Elemento" oferece opções para adicionar compostos estranhos, proteínas, ácidos nucleicos, coleções de elementos, ou arestas para a tela que não estão associadas a qualquer processo. Estes elementos irão aparecer inicialmente no canto superior esquerdo da tela. Eles aparecem como elementos arbitrários que podem ser editados na barra lateral, onde o usuário pode escolher o elemento desejado e alterar os detalhes de visualização do referido elemento. O elemento deve ser incorporada no caminho antes de adicionar quaisquer novos elementos vazios, a fim de manter o asseio via. Elementos vazios são apenas para auxiliar na compreensão visual (ou seja, ilustrando a presença de múltiplas tRNAs durante a transcrição) e não para representar componentes processo integral. Eles devem ser usados com moderação, pois só vai aparecer na visualização, e não são incorporados os formatos legíveis por máquina (BioPAX, SBML, SBGN, PWML).
- Adicione uma sub-pathway, clicando no link "Adicionar processo" e selecionando a opção "Adicionar Sub-Pathway".
NOTA: Sub-vias também podem ser encadeadas para reacções existentes, da mesma maneira como foi demonstrado em passos de 1,16 a 1,18. A adição de sub-trajecto pode reduzir a complexidade das vias grandes ou complexas. Eles também podem ser usados para fornecer informações adicionais sobre ligações entre vias conhecidas. - Procure o nome da sub-caminho na caixa de preenchimento automático. Somente sub-caminhos que já foram definidos para esta via irá aparecer na caixa de autocomplete, portanto, se este é um novo caminho, não há sub-vias aparecerá. Se o sub-percurso pretendido não existir, clique no botão "Novo Sub-Caminho" e siga os passos 1.22.1 a 1.22.3.
- Selecione o tipo de sub-via (Sub Pathway / Inibitória Sub Pathway / Ativação Sub Pathway).
- Digite o nome do sub-via.
- Adicionar elementos de entrada e de saída para o sub-via da mesma forma como a adição de reagentes e prodcado de reacção para um (passos 1.11.1 a 1.11.3 acima).
NOTA: Um sub-via deve ter pelo menos uma entrada ou elemento de saída. Isto permite que seja ligado a outros processos na via, da mesma forma como reacções são encadeadas (passos 1,16-1,18 acima). - Clique no botão "Criar Sub-Pathway".
- Ajuste o tamanho da tela de um diagrama de caminho, clicando no link "Outros" e selecionando a opção "Alterar Canvas Size". Para alterar o tamanho da tela, siga os passos 1.23.1 a 1.23.3.
- Preencha o campo "New Altura" e no campo "New Largura" em conformidade.
- Seleccione a direcção desejada no sentido de que a tela deve aumentar ou diminuir de tamanho, clicando no botão correspondente na grade fornecido na seção "âncora".
- Clique no botão "Atualizar Canvas Size".
- Alternativamente, ajuste o tamanho da tela automaticamente. Uma vez que o percurso está completa, clamber o link "Outros" e selecione a opção "Fit lona com a via" opção. Isto irá cortar automaticamente a tela em torno dos elementos de vias existentes.
- Quando o caminho estiver concluída, clique no link "Caminho" e selecione a opção "Exportar e Vista".
- Use a "cor de fundo para imagens" opção de escolher azul ou branco como a cor de fundo para a imagem.
- Selecione Sim ou Não para o "também geram Versão Simplificada?" opção. Se for selecionado Sim, um diagrama de fio KEGG-like também será gerado automaticamente para a via.
- Clique no botão "Gerar arquivos de imagem".
NOTA: Isto gera os diferentes formatos de troca de dados e arquivos de imagem para a via. As imagens devem ser re-gerado cada vez que o caminho é atualizado, e isso pode levar vários minutos. - Uma vez que a imagem tenha sido gerado, clique no botão "Show em Visualizar" para ver um caminho totalmente hiperlink, de alta resoluçãoimagem Maneira no navegador.
NOTA: O botão "Show in Viewer" aparece apenas em percursos uma vez que suas imagens foram geradas. Este ponto de vista também contém links para baixar o percurso em vários formatos de intercâmbio de dados e imagem.
Replication 2. Pathway
NOTA: A replicação do Caminho é uma rota rápida e fácil de tomar uma via já existente na biblioteca de PathWhiz e duplicá-lo para que ele possa servir como um modelo para posterior edição ou modificação. Para replicar um caminho, siga os passos de 1,1-1,3 para efetuar login, se não tiver feito.
- Ir para o índice via se não já, clicando em "Caminhos" na barra de menu principal. Procure o caminho desejado para ser replicado, inserindo seu nome na barra de pesquisa e clicando no botão "Pesquisar" (por exemplo, "Ciclo TCA").
- Localize o caminho para ser replicado (por exemplo, "Ciclo TCA") e clique no verde "Replicar", mastonelada.
- Edite o nome da via, se desejado (por exemplo, tipo "Ciclo TCA Practice". Não há dois caminhos podem ter o mesmo nome.
- Digite uma descrição nova ou diferente para a via (veja o passo 1.7).
- Para adicionar referências novas ou diferentes para a via, clique no botão "Add Reference" e siga o passo 1.8 acima.
- Clique no botão "Criar Caminho". Uma roda roxo progresso aparecerá enquanto a via está sendo construída. Este processo pode demorar vários minutos ou mais, dependendo do tamanho da via.
- Editar ou acrescentar quaisquer elementos desejados para o caminho usando as etapas 1,10-1,22.
- Siga os passos de 1,23 a 1,29 para completar e exportar o percurso.
Propagação 3. Pathway
NOTA: A propagação Caminho é uma rota rápida e fácil de tomar uma via já existente na biblioteca de PathWhiz para um organismo (por exemplo, Escherichia coli) e criar um caminho semelhante para outraorganismo (por exemplo, Staphylococcus aureus). Este processo envolve a descoberta e substituindo proteínas S. aureus para proteínas de E. coli e a regeneração de todo o percurso com proteínas S. aureus ou genes. Para propagar um começo caminho seguindo os passos 2.1 e 2.2 supra.
- Localize o caminho para ser propagado (nesse ciclo TCA caso) e clique no botão "Show".
- Clique no botão "Propagar" no canto superior direito.
- Sobre a forma de propagação, clique no botão "Adicionar Espécies" para identificar a espécie à qual o caminho existente será convertido. Várias espécies podem ser adicionados, embora os mais espécies existem, quanto maior o tempo de conversão.
- Procurar por uma espécie existente ou adicionar uma nova espécie seguindo os passos 1.6.1 a 1.6.5 supra.
- Se desejar, altere o E-valor para determinar o limiar de similaridade para encontrar homólogos de proteínas. Seleccione Sim ou Não para os "Proteínas Avaliado Só"Opção (isto indica que a proteína de UniProt deve ser usado na via gerado).
- Clique no botão "Propagar Caminho".
- Clique no botão "OK" na janela pop-up.
NOTA: Uma roda roxo progresso aparecerá enquanto a via está propagando. Este processo pode demorar vários minutos ou mais, dependendo do tamanho da via e o número de espécies a que o percurso inicial está a ser propagada. - Uma segunda janela pop-up irá aparecer quando a propagação é completa. Clique no botão "OK". Isto irá criar um índice que mostra todos os novos caminhos que foram gerados a partir desta propagação.
- A partir deste índice, verificar os detalhes da via e clique no botão "Empate" para visualizar e editar cada um dos novos caminhos.
- Editar ou acrescentar quaisquer elementos desejados para o caminho usando as etapas 1,10-1,22.
- Siga os passos de 1,23 a 1,29 para completar e exportar o percurso.
4. ediçãouma via existente
NOTA: Em alguns casos, novas informações sobre uma via já existente tem de ser adicionado ou informações incorretas sobre um caminho precisa ser corrigido. Para editar um caminho existente, começar por seguir os passos 1,1-1,3 fazer login.
- Ir para o índice via se não já, clicando em "Caminhos" na barra de menu principal.
- Localizar o caminho a ser editado (neste caso, o "ciclo de TCA"). Se a imagem é para ser editado, clique no botão "Empate", se a descrição ou referências estão a ser editado, clique no botão "Editar".
- Para editar a imagem, siga os passos 1,10-1,29 acima.
- Para editar a descrição caminho ou referências, siga os passos de 1,4 a 1,8 e clique no botão "Update Pathway" quando terminar para salvar as alterações.
- Regenerar a imagem para atualizar as alterações seguindo os passos 1,25-1,28.
5. Visualizando Caminho e downloadConcordong
NOTA: Esta ferramenta baseada na Web contém milhares de caminhos cuidadosamente desenhados e editados que podem ser visualizados ou transferidos para diferentes aplicações. Para visualizar ou baixar um caminho, siga os passos de 1,1-1,3 para fazer login.
- Ir para o índice via (se já não estiver lá), clicando em "Caminhos" na barra de menu principal.
- Localize o caminho para ser baixado (neste caso, o ciclo TCA) e clique no botão "Show".
- Clique no botão roxo com o desejado PathWhiz ID ao lado do rótulo "Show No Viewer" (pode haver mais de um ID).
- Clique na guia "downloads" na barra lateral.
- Clique nos links para baixar o percurso em vários formatos de arquivo.
Resultados
Principal ferramenta de geração de caminho do servidor Web descritos neste manuscrito é mostrado na Figura 1 e Figura 2. As opções de menu fornecidas por cada um dos separadores também são mostrados. Figuras 3 e 4 fornecem um conjunto de screenshots do processo de criação do caminho. A Figura 5 fornece um conjunto de screenshots do processo de criação de reação. A Figura 6 mostra o espectador via on-line e seu menu.
PathWhiz pode ser usado para gerar vias com diversos tipos e estilos de conteúdo. Estes incluem "tradicionais" vias metabólicas (Figura 7), as doenças e as vias de droga que mostram efeitos colaterais (Figura 8) e de respostas ao fármaco (Figura 9), bem como as vias de sinalização de proteína (Figura 10). Caminhos podem ser ricamente colorido com Biolo consideráveldetalhe gico ou eles podem ser convertidos em representações preto e branco simples (Figura 11). Depois de concluído, estas vias podem ser vistos no visualizador via interativa (Figura 6), baixado como imagens, ou exportados em diversos formatos de troca de dados legíveis por máquina para análise posterior. Note-se que a qualidade dos diferentes formatos de troca de dados depende da qualidade dos dados introduzidos quando originalmente desenho da via. Por exemplo, a adição de mais detalhe reacção (isto é, estequiometria, estados biológicos) irá produzir BioPAX mais abrangente. Por outro lado, vias desenhada com elementos (por razões visuais, tais como mostrando os elementos ligados ou complexos de proteína), podem também produzir glifos sobrepostas em SBGN-ML sobreposição.

Figura 1: Caminho Editor de Interface. A interface do editor é composed de 3 seções principais: uma parte superior da barra de menu principal, um menu secundário e uma lona em grade. A barra superior do menu principal (roxo) fornece links para visualizar, editar e criar elementos da via. A barra de menu secundário inferior (cinza) fornece links para adicionar e editar elementos de vias visuais no diagrama de caminho atual, tais como reações, interações, processos de transporte, sub-vias, compostos, proteínas, ácidos nucleicos, bem como membranas, celular / imagens subcelulares, caixas de zoom, e etiquetas. Este menu também inclui duas guias que permitem a edição de elementos selecionados ou a edição da tela. A lona branca em grade abaixo barras de menus é onde os percursos e processos de reação será adicionado. A caixa de zoom funciona como uma sugestão visual para indicar a ampliação de uma área selecionada de uma imagem. É constituída por um pequeno quadrado que está ligado a um quadrilátero re-considerável. O quadrado pequeno é colocado sobre a área que está a ser expandido ou reduzido, enquanto o quadrilátero funciona como uma tela em que se pode adicionar threações e que acontecem na área selecionada (pelo quadrado menor). Editar caixa de zoom clicando duas vezes nele para acessar a sua barra lateral. opções de edição incluem listas drop-down para o modelo, cor e z-index. A orientação de renderização da caixa de zoom pode ser alterado selecionando topo, direita, esquerda ou inferior na guia do modelo. Quando a caixa de zoom é selecionado, os círculos pretos podem ser arrastadas para redimensionar e reformatar as diferentes componentes da caixa de zoom. Por favor clique aqui para ver uma versão maior desta figura.
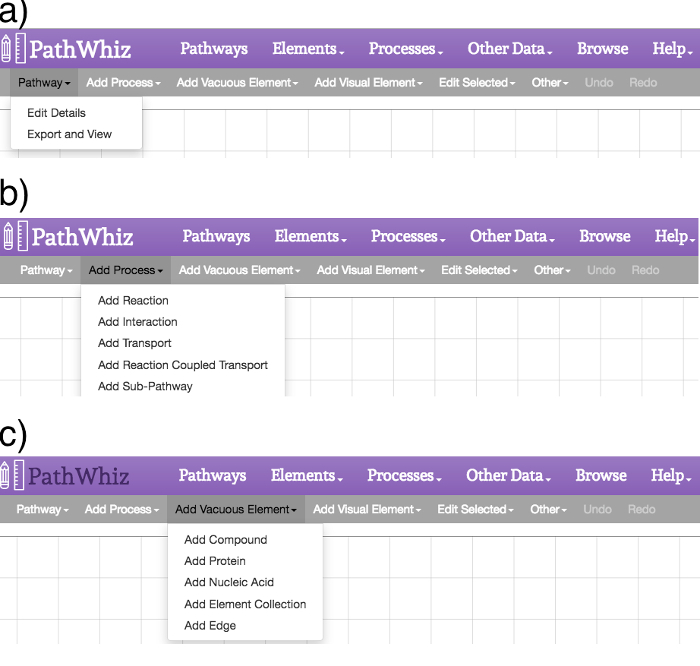
Figura 2: Caminho do Editor de Menus. Os menus de edição fornecer opções para adicionar os processos e elementos, bem como para editar os elementos existentes e a tela. (A) O link "Caminho" oferece opções para "Editar Detalhes" e "Export and View". A opção "Editar Detalhes" permite a edição da descrição do percurso e referências enquanto a opção "Exportar e View" permite a geração ou regeneração dos arquivos de imagem. (B) O link "Adicionar processo" oferece opções para adicionar uma reação, a interação, a ligação evento, evento de transporte, reação acoplada de transporte, ou sub-caminho para a tela. (C) O link "Adicionar vazia Elemento" oferece opções para adicionar um composto, uma proteína, um ácido nucleico, uma coleção elemento, ou uma vantagem para a tela. Estes elementos irão aparecer no canto superior esquerdo da tela. Um elemento arbitrário aparecerá na tela ao lado da barra lateral pop-up, onde o usuário pode procurar o elemento desejado ou alterar os detalhes do referido elemento. O elemento deve ser incorporada no caminho antes de adicionar quaisquer novos elementos vazios, a fim de manter o asseio via._upload / 54869 / 54869fig2large.jpg "target =" _ blank "> Clique aqui para ver uma versão maior desta figura.
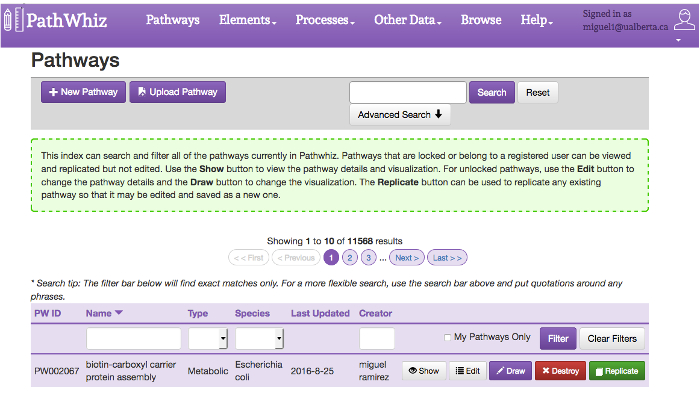
Figura 3: Caminho Índice Form. O índice via oferece uma coleção de vias existentes atualmente e uma caixa de pesquisa para consultar percursos específicos. Caminhos podem ser filtrados por nome, tipo, espécie, e criador usando a barra de filtro no topo da tabela de índice. Eles também podem ser pesquisados por nome usando a barra de pesquisa no topo da página. A abertura do "Advanced Search" permite buscas mais específicas por combinações de biológica estado, tipo, espécie, composto, e proteína. A pesquisa avançada permite o uso de AND, OR, e operadores lógicos para não criar consultas complexas. Cada caminho inclui 5 botões: "Show", "Editar", "Empate", "Destroy" e "Replicar". O botão "Show"permite a visualização da via utilizando o Viewer. O botão "Editar" permite a edição dos metadados caminho, incluindo o nome, tipo, espécie, descrição e referência. O botão "Empate" permite a edição da tela que contém o caminho. O botão de "destruir" permite a remoção da via do banco de dados (se o usuário tem permissão). O botão "Duplicar" permite a replicação do percurso seleccionado. Os botões "Anterior" e "Avançar" permitem ao utilizador navegar entre as páginas de vias. Por favor clique aqui para ver uma versão maior desta figura.
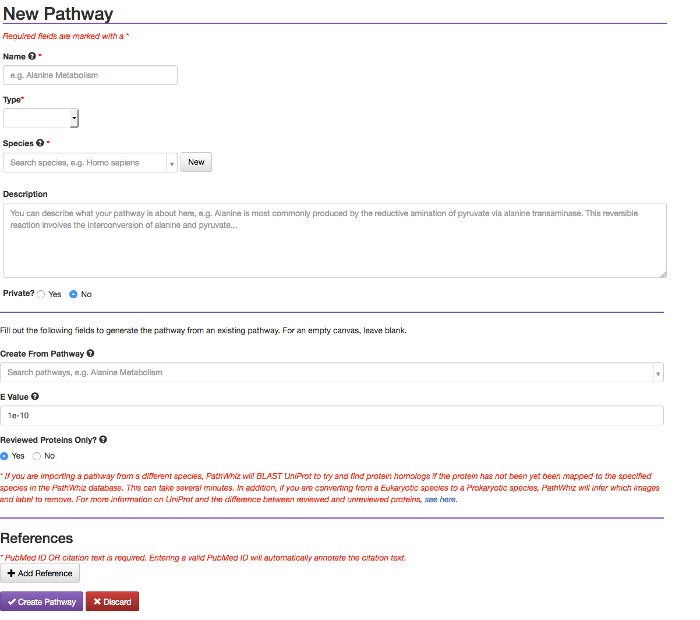
Figura 4: Criar nova via formulário. O botão "Novo Pathway" (ver Figura 3) leva a via a forma mostrada aqui. Este formulário contémcampos para o caminho nome, tipo, espécie, e descrição. O botão "Novo Caminho" também permite começar a partir de uma via existente e adicionar referências. Por favor clique aqui para ver uma versão maior desta figura.
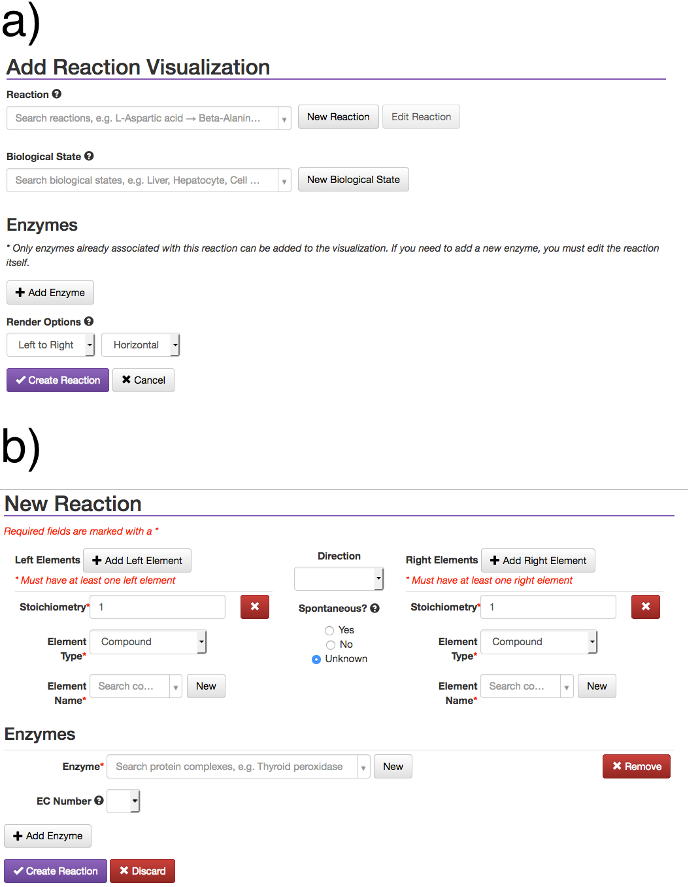
Figura 5: Criar novo formulário Reaction. O link "Adicionar processo" permite aos usuários adicionar um novo processo, como uma reação ou evento obrigatório. Adicionando um processo cria um modelo de reacção, a partir da qual as visualizações de reacção pode ser gerado e adicionado com a via diagramas. O modelo de reacção e visualização são entidades separadas. (A) As autorizações de campo reacção à procura de uma reacção já existente, por reagente, produto, ou enzima. As autorizações de campo estado biológico busca e seleção de um biologica existentel Estado. Uma vez que uma reação é escolhido, as enzimas correspondentes pode ser adicionado usando o botão "Adicionar Enzyme", o que fará com que uma caixa de enzima autocomplete. As opções tornam permitir ao usuário escolher a direção que deseja que a reação a ser processado. Uma nova reacção pode ser criada por meio do botão (b) "New Reaction", o que conduz a uma nova forma de Reacção em que os elementos e as enzimas podem ser adicionadas. Uma vez que todos os campos estiverem preenchidos, a reacção pode ser criada por meio do botão "Criar Reacção". Para editar o modelo de reação subjacente deve sair do ilustrador caminho, vá para o índice de reação, descobrir e editar a reação lá. A fim de evitar que as discrepâncias de dados e alterando involuntariamente vias existentes, não é possível alterar os reagentes / ou remover produtos enzimas a partir de um modelo de reacção se ele já tem visualizações em vias existentes. Assim, a edição do modelo de reacção não atualiza automaticamente reação existentevisualizações. A fim de mudar um modelo de reacção deve-se voltar a adicionar a visualização correspondente ao diagrama de caminho para que as alterações aparecem. Por favor clique aqui para ver uma versão maior desta figura.
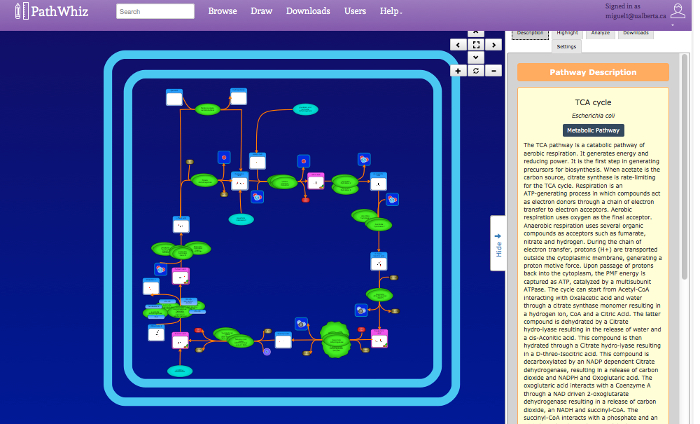
Figura 6: Caminho Viewer. Os primeiros botões da interface visualizador de certas proporcionar ações básicas de navegação, zoom e tela alternando. A janela central, mostra o caminho que pode ser navegado, clicando e arrastando, ou zoom usando um mouse. Os elementos da via exibidos são hiperlinks a outros caminhos e bancos de dados (por exemplo HMDB, drugbank, UniProt). A barra de menu lateral exibe uma descrição da via com referências fornecidas pelo usuário. O menu lateral também exibe as guias "Destaque", "Analisar", "Façawnloads ", e" Configurações ". O" Destaque "guia permite que os compostos e enzimas para ser selecionado e destacado em vermelho. A guia" Analisar "permite que os dados de concentração experimental a ser inserido, que é então mapeado para o caminho usando um gradiente de cor. o separador "Transferências" oferece links para os arquivos de imagem para download correspondentes e trocar arquivos de dados. o arquivo PNG é um arquivo de imagem menor não-vector. os SVG + BioPAX links fornecem arquivos de imagem maior do vetor com BioPAX incorporado, para a máquina-a legibilidade. o BioPAX, SBML, SBGN e PWML links fornecem diferentes formatos legíveis por máquina. o guia "Configurações" permite a personalização visual da imagem caminho exibido. por favor clique aqui para ver uma versão maior desta figura.
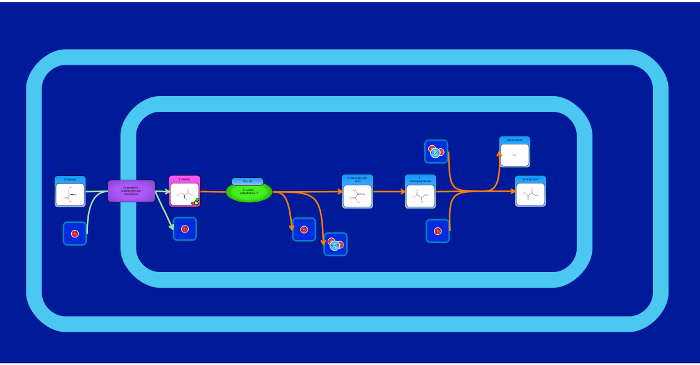
Figura 7: Metabolic imagem Caminho. Este é um exemplo de uma via metabólica "tradicionais", que descreve a biossíntese e degradação de um composto particular (D-serina). O metabolito principal está posicionada no centro da tela e as setas de reacção e de transporte (arestas) mostram o fluxo da via. Bordas e elementos podem ser automaticamente "estalou" em conjunto, ou seja, conectado. Elemento tirar pontos são indicados por círculos vermelhos transparentes sobre os lados do elemento, e os pontos de início edge / finais são representados por círculos cinzentos transparentes sobre as extremidades de ponta. pontos de snap virar um verde transparente quando pairava sobre, e um verde sólido quando selecionado. A fim de tirar uma vantagem para um elemento, clique primeiro em qualquer borda de início / fim ou o elemento de ponto de snap (ele vai ficar verde). Em seguida, clique na borda de início / fim ou no ponto de encaixe elemento que precisa ser conectado. A borda irá conectar-se automaticamente ao ponto de pressão, e permanecer conectado until é removido (clicando duas vezes a borda e arrastando o ponto final de distância, ou conectando-o a um ponto de alinhamento diferente). É importante estar atento a seleção acidentalmente pontos de snap, uma vez que isso pode ter consequências não intencionais ao tentar mover bordas ao redor. A cor verde sólida de pontos de snap selecionados destina-se a alertar o utilizador para tirar pontos eles têm atualmente selecionados. pontos de snap pode ser desmarcado, clicando sobre eles uma segunda vez. Bordas também pode ser reconectado a seus elementos originais. Quando uma aresta é selecionada, visitando o link "Editar selecionado" no menu e, em seguida, no link "Editar Edge". Isso fará com que as opções para se conectar automaticamente a borda em diferentes direções. Por favor clique aqui para ver uma versão maior desta figura.
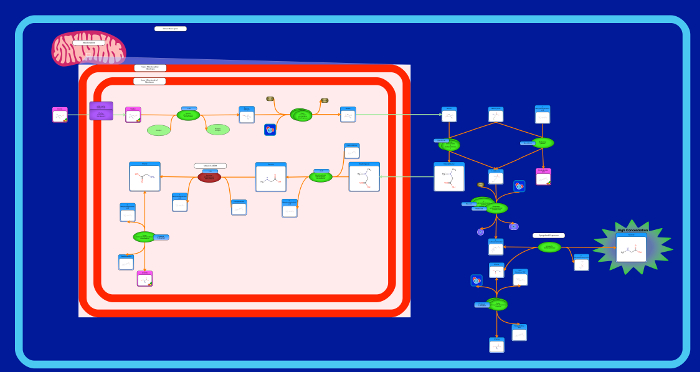
Figura 8: Doenças imagem Caminho. Este é um exemplo de uma via de doença que mostra os órgãos afectados pela doença (Sarcosina Oncometabolite via). elementos de imagem adicionais são usados para descrever o aumento ou a diminuição das concentrações do metabolito ea sua acumulação ou dissipação. Por favor clique aqui para ver uma versão maior desta figura.
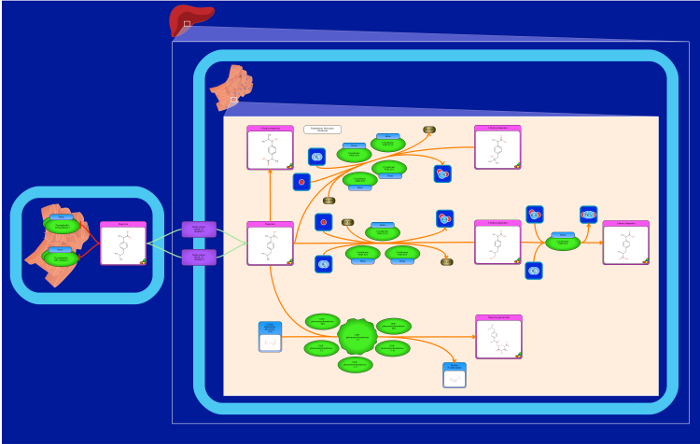
Figura 9: Droga imagem Caminho. Este é um exemplo de uma via de drogas que mostra os órgãos onde a droga é metabolizada (Ibuprofeno) Pathway. A cor em torno do metabólito da droga é geralmente descrito como rosa. Por favor clique aqui para ver uma versão maior desta figura.
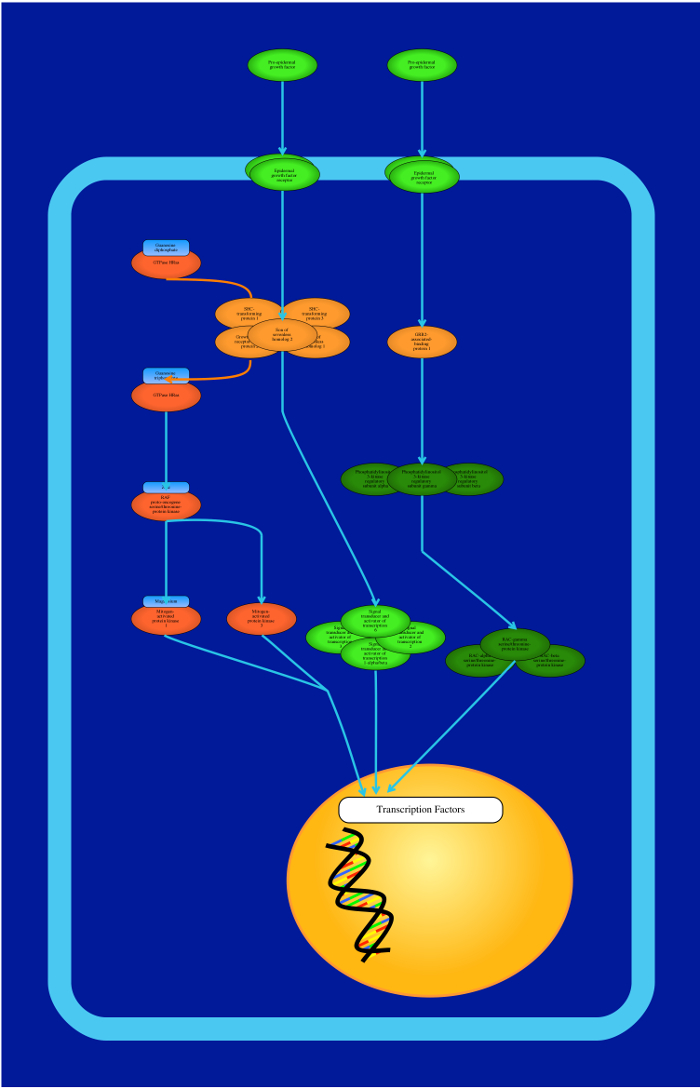
Figura 10: proteína de sinalização Caminho de imagem. Este é um exemplo de uma via de sinalização que mostra um conjunto de sinalização reacções entre diferentes proteínas (EGFR) Pathway. As proteínas podem ser representadas com cores múltiplas e que pode ser representada quer pelo nome proteína ou o nome da subunidade. Por favor clique aqui para ver uma versão maior desta figura.
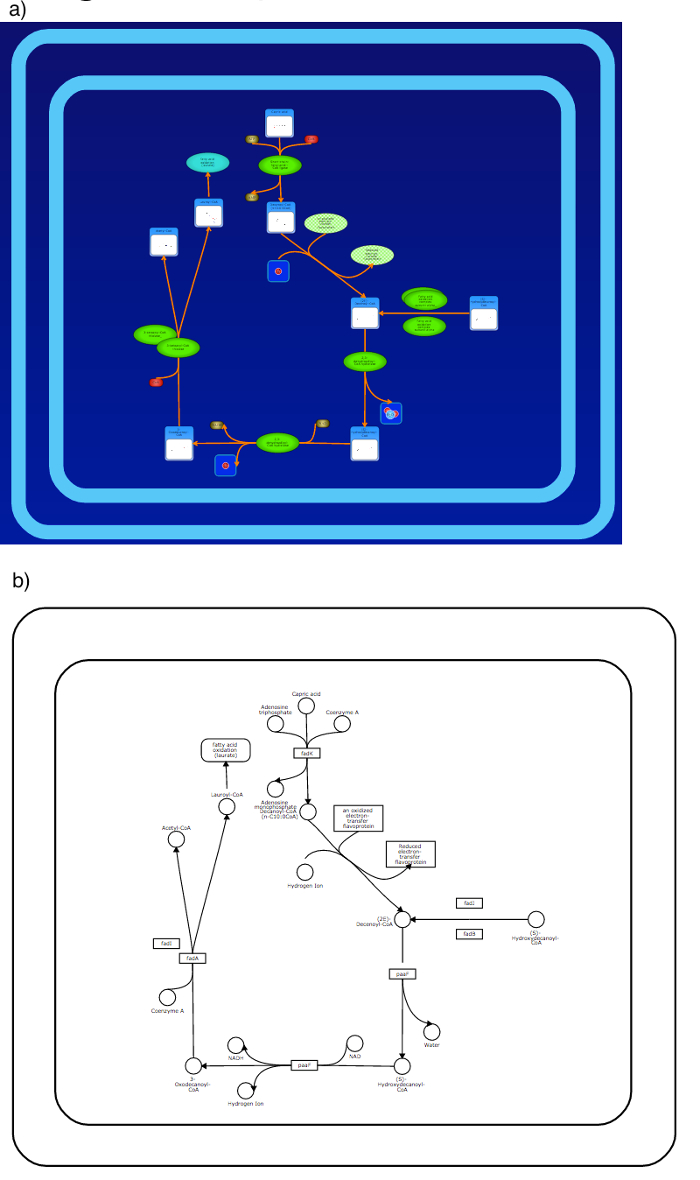
Figura 11: Colorful vs Simples Pathway Imagens. Vias coloridas podem ser gerados com contexto biológico rico em qualquer um fundo branco ou azul (a). metabolismo do folato é descrito aqui. Simples, vias KEGG-como também pode ser generated usando uma representação preto e branco simples (b). Por favor clique aqui para ver uma versão maior desta figura.
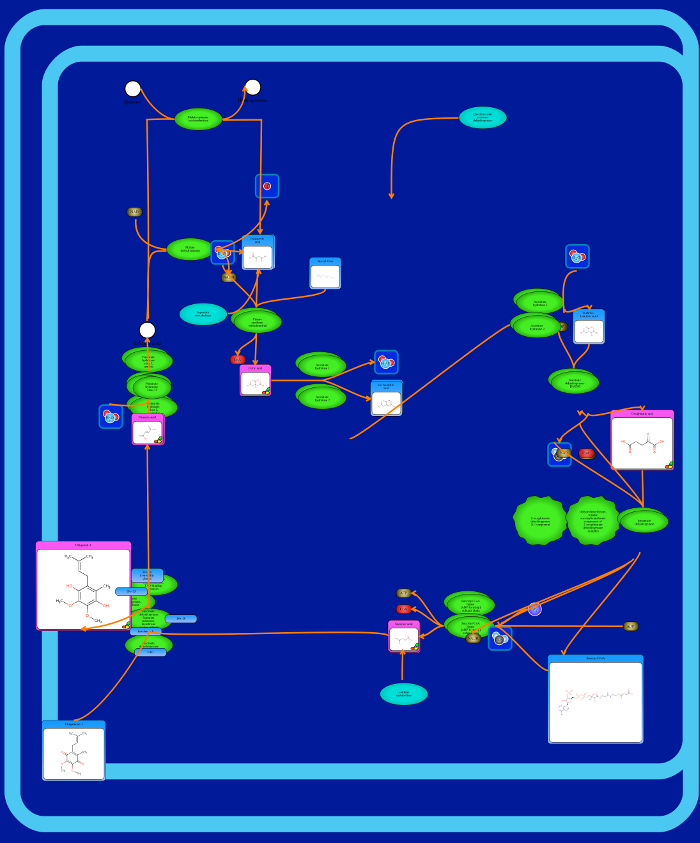
Figura 12: Suboptimal imagem Caminho. Uma imagem que descreve o que um caminho abaixo do ideal (ciclo TCA) parece. elementos sobrepostos e arestas que cruzam fazer o percurso incompreensível. Isso pode acontecer se os elementos de reação não são cuidadosamente ou corretamente manipulada na tela. Manipulando os elementos a ter mais do que dois tipos de modelos diferentes para os compostos (grande, médio, pequeno visualização composto ou fármaco visualização, Cofator visualização, de fundo simples, Esquerda ou Direita Top visualização) leva a várias inconsistências de imagem. Os tipos de modelo são mostrados na etapa 1.15.2. Não conectando the bordas afecta o fluxo da imagem levando a interpretações pobres da via. Por favor clique aqui para ver uma versão maior desta figura.
| PathWhiz | VANTED | PathVisio | Ferramentas pathway | visant | |
| Servidor web | sim | Não | Não | Não | Não |
| Programa instalável | Não | sim | sim | sim | sim |
| Caminhos de proteína | sim | sim | sim | Não | sim |
| Vias metabólicas | sim | sim | sim | sim | sim |
| Salvar como PNG / JPG | sim | sim | sim | Não | Não |
| Salvar como HTML | sim | Não | Não | sim | Não |
| Salvar como SVG | sim | sim | sim | Não | sim |
| Salvar como PDF | sim | sim | sim | sim | sim |
| Salvar como BioPAX | sim | sim | sim | sim | sim |
| Salvar como SBML | sim | sim | sim | sim | sim |
| Salvar como SBGN-ML | sim | sim | sim | Não | Não |
| Mapeamento identificador | sim | sim | sim | sim | sim |
| membrana Rendering | sim | Não | Não | Não | Não |
| organela Rendering | sim | Não | Não | Não | Não |
| órgão Rendering | sim | Não | Não | Não | Não |
| Cores ricas Imagens | sim | Não | Não | Não | Não |
| Caminho Descrição | sim | Não | Não | sim | Não |
| Caminho DB link | sim | Não | sim | sim | Não |
| Caminho Inference | sim | Não | Não | sim | Não |
| Experimento. dados Overlay | Não | sim | Não | sim | sim |
| Análise pathway | Não | sim | sim | Sim | sim |
Tabela 1: Comparação de recursos. Uma comparação de recursos de várias ferramentas de edição / tradução comum da via.
Documento Suplementar 1: Exemplo de Descrição Ciclo TCA para PathWhiz Caminho. Clique aqui para baixar o arquivo suplementar.
Discussão
O protocolo aqui descrito para a criação de uma via metabólica simples (ciclo do TCA) pode ser adaptado para criar uma grande variedade de vias, biologicamente complexas legíveis por máquina para qualquer espécie. Além disso, este protocolo também descreve como se pode replicar ou propagar vias existentes criadas por outros usuários. Construção de um caminho usando esta ferramenta requer adições repetidas passo-a-passo de reações, interações, processos de transporte, e sub-caminhos, cada um dos quais estão ligados por elementos sobrepostos. Colocando tudo isso junto permite criar diagramas via coloridos, visualmente agradável que fornecem detalhes biológica considerável e contexto biológico útil. Os passos descritos neste protocolo são relativamente simples, eo tempo que leva para construir um diagrama de percurso depende do tamanho e da complexidade da via. Com um pouco de prática, a maioria dos indivíduos pode render um diagrama de caminho de alta qualidade que consiste em cerca de 15-20 reações ou processa umd vários componentes celulares em cerca de 15 min. Um novo usuário pode levar até 30-40 min para gerar um caminho de tamanho e complexidade semelhante. O tempo necessário para gerar um caminho é aproximadamente linear com o número de reacções / processos que precisam ser processados.
Criando um caminho de alta qualidade através desta ferramenta baseada na Web depende da qualidade e detalhe do material de origem (vias de livros, bancos de dados on-line, os dados experimentais, esboços desenhados à mão) e a meticulosidade da via "artista". Aqueles que desejam gerar diagramas via qualidade superior devem prestar especial atenção aos pontos 1.11, 1.15 e 1.20 do protocolo, uma vez que estas seções descrevem a criação e edição de elementos de reação (reagentes / produtos, enzimas, bordas, imagens, caixas de zoom, etiquetas, e membranas). Os melhores diagramas via inteligentemente amalgamar informações de tantas representações existentes da via possível, incluindo aqueles encontrados em books, cartazes, documentos e bases de dados on-line. Outra chave para a geração de caminhos de alta qualidade é verificar cuidadosamente a exactidão das reações antes de criar uma reação (a secção 1.11 do protocolo). Tomando o tempo e esforço para garantir que os reagentes, produtos e enzimas envolvidos (muitas das quais já existem no banco de dados grande de PathWhiz) está correto para cada espécie é muito importante. Também é importante estar ciente da localização celular das reacções e para incluir componentes celulares ou subcelulares chave, a fim de proporcionar o contexto biológica correcta. Isto pode ser feito através da verificação e corroborando a reacção através de bases de dados online, como UniProt. Tendo todas as informações necessárias à mão, juntamente com um esboço, desenhado mão da via a ser criada irá reduzir significativamente os erros e o tempo total gasto em desenho ou renderização.
Como seria de esperar, maiores e mais complexos caminhos levará mais tempo para render, particcularmente se os elementos e processos desejados não estiverem no banco de dados do PathWhiz. Quando se trabalha com vias maiores, é geralmente aconselhável para alternar entre o modo de gravação automática com a manual modo de economia, a fim de evitar um longo intervalo de tempo entre as acções. Quando a replicação de uma via, a quantidade de tempo que o utilizador pode esperar para o percurso a ser gerada depende do número de elementos na via. A maioria das vias pode ser replicado em cerca de 1-2 min. Quando uma via de propagação, render bem sucedida da via recém propagadas depende de quão semelhante as duas espécies são, como PathWhiz utiliza BLAST sequência de 17 pesquisa para encontrar enzimas homólogos entre espécies. percursos maiores será mais lento para se propagar porque BLAST terá de ser executado em um maior número de enzimas. A tentativa de propagar vias entre as espécies significativamente diferentes (digamos entre leveduras e humanos) resultará em caminhos que está sendo processado com um número de proteínas desconhecidas. Estes "distante" pvias ropagated normalmente requerem edição manual adicional. Devido à natureza altamente visual de diagramas de percurso e o detalhe que pode ser levado a um caminho, é sempre uma boa idéia para trabalhar em um computador com uma tela razoavelmente grande (> 20 polegadas ou> 50 cm) e uma boa conexão de internet (> 5 Mbps).
Se os problemas com a renderização ou refrescante tela são encontrados, o usuário pode ter que fazer uma pequena quantidade de solução de problemas. Se um grande, complexa via leva muito tempo para atualizar, o usuário pode ter que atualizar a página. Se um caminho não se propaga como se espera, o usuário pode ter que fazer alguma edição manual para garantir que todos os elementos são exibidos corretamente. Além disso, como um exemplo mais específico, se os elementos de uma reação não são exibidos, o usuário pode ter a certeza de que todos os elementos ou enzimas são devidamente selecionados e as bordas não estão escondidos. O link "Ajuda" no cabeçalho principal pode ser útil se for encontrado algum problema. Um tutorial édisponíveis sob a aba "Tutorial" e um manual do usuário está disponível na guia "guias do utilizador". Ambos explicar muitas das características da ferramenta em detalhe. O Manual do Usuário pode ser usado para solucionar ou explicar potenciais limitações para um determinado recurso, tais como quando um usuário bloqueia um caminho e mais tarde pretende editá-lo.
Conforme destacado através deste protocolo e através dos exemplos fornecidos nas figuras anexas, esta ferramenta oferece uma série de características únicas não encontradas em nenhum (ou a maioria) outras ferramentas de desenho via (ver Tabela 1). Primeiro, é totalmente baseado na web e completamente independente de plataforma. Em segundo lugar, ele suporta o processamento e geração de facile da biologicamente complexos diagramas coloridos,, visualmente agradável, totalmente hiperlinks via que também podem ser convertidos em formatos de leitura óptica (BioPAX, SBGN-ML 18, SBML 19, PWML 14). Em terceiro lugar, os diagramas de vias génerosted por esta ferramenta pode ser navegado, procurou, selecionados e facilmente explorada através de uma interface fácil de usar banco de dados on-line e visualização. Em quarto lugar, a ferramenta web é projetado para suportar contribuições via comunidade, permitindo "crowd sourcing via" que incentiva a partilha ea geração de novas vias e novos elementos da via.
Vias gerados por este instrumento baseado na Internet pode ser usada para uma variedade de aplicações. Ricamente detalhados, caminhos totalmente hiperlinks podem ser facilmente integrados em bancos de dados específicos do organismo para proteômica, metabolômica ou aplicações de biologia de sistemas. percursos acessíveis pela Internet são especialmente úteis para fins de educação e de formação, como os detalhes disponíveis através de imagens baseadas na Web são frequentemente muito maior do que o que pode ser exibido através de uma imagem estática ou através de uma única página livro ou revista. Esta ferramenta baseada na Web também suporta a geração de representações da via que são mais adequados para impressão e publicação.Como resultado, muitas imagens geradas por esta ferramenta baseada na Web estão aparecendo em jornais, cartazes e apresentações de slides. Exportadores vias em formatos de arquivo de troca de dados baseados em texto (como BioPAX e SBML) permite percursos gerados usando esse servidor web para ser usado diretamente na análise computacional para biologia de sistemas ou aplicações de modelagem metabólicas. Propagação vias entre as espécies permite inferências a serem feitas sobre os processos biológicos, especialmente entre aquelas espécies que têm sido muito recentemente sequenciados. Embora nem todas as vias existentes existir atualmente no PathWhiz, seu banco de dados via pública continua a crescer, levando ao surgimento de coleções de vias novas, multidão de origem. Essas coleções não só será prontamente extensível a novas espécies, que esperamos levar a uma compreensão mais profunda da sua biologia única e bioquímica.
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
Os autores agradecem a Canadian Institutes of Health Research (CIHR) e Genoma Alberta, uma divisão da Genome Canada, para apoio financeiro.
Materiais
| Name | Company | Catalog Number | Comments |
| Computer with colour screen | N/A | N/A | >20 inches or >50 cm |
| Internet connection | N/A | N/A | >5 mbps |
| Modern web browser | N/A | N/A | Google Chrome (v. 31 and above), Internet Explorer (v. 9 and above), Safari (v. 7 and above), Opera (v. 15 and above) and Firefox (v. 23 and above) |
Referências
- Michal, G. On representation of metabolic pathways. Biosystems. 47 (1-2), 1-7 (1998).
- Kanehisa, M., Sato, Y., Kawashima, M., Furumichi, M., Tanabe, M. KEGG as a reference resource for gene and protein annotation. Nucleic Acids Res. 44 (D1), D457-D462 (2016).
- Karp, P., Riley, M., Paley, S. The MetaCyc Database. Nucleic Acids Res. 30 (1), 59-61 (2002).
- Kelder, T., et al. WikiPathways: building research communities on biological pathways. Nucleic Acids Res. 40 (Database issue), D1301-D1307 (2011).
- Croft, D., et al. The Reactome pathway knowledgebase). Nucleic Acids Res. 42 (Database issue), D472-D477 (2014).
- Karp, P. D., et al. Pathway Tools version 13.0: integrated software for pathway/genome informatics and systems biology. Brief Bioinform. 11 (1), 40-79 (2010).
- Van Iersel, M. P., et al. Presenting and exploring biological pathways with PathVisio. BMC Bioinformatics. 9, 399 (2008).
- Demir, E., et al. The BioPAX community standard for pathway data sharing. Nat. Biotechnol. 28 (9), 935-942 (2010).
- Shannon, P., et al. Cytoscape: A Software Environment for Integrated Models of Biomolecular Interaction Networks. Genome Res. 13 (11), 2498-2504 (2003).
- Salomonis, N., et al. GenMAPP 2: new features and resources for pathway analysis. BMC Bioinformatics. 8, 217 (2007).
- Elliott, B., et al. PathCase: pathways database system. Bioinformatics. 24 (21), 2526-2533 (2008).
- Hu, Z., et al. VisANT 3.0: new modules for pathway visualization, editing, prediction and construction. Nucleic Acids Res. 35 (Web Server), W625-W632 (2007).
- Jewison, T., et al. SMPDB 2.0: Big Improvements to the Small Molecule Pathway Database. Nucleic Acids Res. 42 (D1), D478-D484 (2013).
- Pon, A., et al. Pathways with PathWhiz. Nucleic Acids Res. 43 (W1), W552-W559 (2015).
- Sajed, T., et al. ECMDB 2.0: A richer resource for understanding the biochemistry of E. coli. Nucleic Acids Res. 44 (D1), D495-D501 (2015).
- Wishart, D., Mandal, R., Stanislaus, A., Ramirez-Gaona, M. Cancer Metabolomics and the Human Metabolome Database. Metabolites. 6 (1), 10 (2016).
- Altschul, S. F., Gish, W., Miller, W., Myers, E. W., Lipman, D. J. Basic local alignment search tool. J. Mol. Biol. 215 (3), 403-410 (1990).
- Le Novere, N., et al. The Systems Biology Graphical Notation. Nat. Biotechnol. 27 (8), 735-741 (2009).
- Hucka, M., et al. The systems biology markup language (SBML): a medium for representation and exchange of biochemical network models. Bioinformatics. 19 (4), 524-531 (2003).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados