Method Article
Volumen-Segmentierung und Analyse von biologischen Materialien mit SuRVoS (Super-Region Volumen Segmentierung)-Workbench
In diesem Artikel
Zusammenfassung
Segmentierung der dreidimensionalen Daten aus vielen bildgebenden Verfahren ist ein großer Engpass bei der Analyse komplexer biologischer Systeme. Hier beschreiben wir die Verwendung von SuRVoS Workbench, halbautomatisch Segment volumetrischer Daten auf unterschiedlichen Längenskalen mit Beispiel-Datasets von Cryo-Elektron Tomographie, Cryo weiche Röntgen-Tomographie und Phase Kontrast Röntgen-Tomographie Techniken.
Zusammenfassung
Segmentierung ist der Prozess bestimmte Regionen oder Objekte innerhalb einer abgebildeten Volumens zu isolieren, so dass weiterer Untersuchungen auf diese Bereiche von Interesse durchgeführt werden kann. Bei der Analyse von komplexen biologischen Systemen ist die Segmentierung des dreidimensionalen Bilddaten ein zeitaufwendig und arbeitsintensiv intensive Schritt. Mit der zunehmenden Verfügbarkeit von vielen bildgebender Verfahren und automatisierte Sammelsystemen stellt dies eine erhöhte Herausforderung für modernen experimentellen Biologen aus Daten wissen zu bewegen. Diese Publikation beschreibt die Verwendung der SuRVoS-Workbench, ein Programm entwickelt, um diese Probleme zu lösen, durch die Bereitstellung von Methoden zur semi-automatisch komplexe biologische volumetrische Segmentdaten. Drei Datensätze unterschiedlicher Vergrößerung und bildgebende Modalitäten sind jeweils unterschiedliche Strategien der Segmentierung mit SuRVoS Hervorhebung hier präsentiert. Phase Kontrast Röntgentomographie (MicroCT) der Fruchtkörper einer Pflanze wird zur Segmentierung mit Modelltraining demonstrieren, Cryo-Elektron Tomographie (CryoET) der menschlichen Thrombozyten wird zur Segmentierung mit Super - und Megavoxels und Cryo soft zeigen Röntgen-Computertomographie (CryoSXT) einer Säugerzelle Linie wird verwendet, um das Label teilen Werkzeuge zu demonstrieren. Strategien und Parameter für jeden Datentyp werden ebenfalls vorgestellt. Durch die Mischung einer Auswahl von semi-automatische Prozesse in einem interaktiven Werkzeug, bietet SuRVoS mehrere Vorteile. Insgesamt wird zu Segment volumetrischer Daten um einen Faktor 5 im Vergleich zu manuellen Segmentierung, ein Standbein in vielen Bildfelder Verarbeitung verkürzt. Dies ist eine erhebliche Einsparungen bei voller manueller Segmentierung Wochen Aufwand in Anspruch nehmen kann. Darüber hinaus richtet sich die Subjektivität durch den Einsatz von rechnerisch ermittelten Grenzen und splitting komplexe Sammlungen von Objekten durch ihre berechneten Eigenschaften anstatt auf einer Schachtel-durchschachtel Grundlage.
Einleitung
Die SuRVoS-Workbench ist ein Stück Software, die so konzipiert, dass Forscher wissenschaftlich relevanten Informationsextraktion aus volumetrischer Daten aus verschiedenen Proben, unabhängig von der Struktur von Interesse, Auflösung oder bildgebende Modalität1, 2. Volumetrische datenbasierte wie diese sind oft gesammelt, mit Röntgen- oder Elektron Tomographie-Systemen, routinemäßig bei großen Laboratorien oder zentrale Einrichtungen aufgrund ihrer Komplexität. Beide dieser Methoden und andere Techniken, produzieren große, Informationen reichen Datasets beweisen die anspruchsvollen Segment mit entweder halbautomatischen Methoden oder manuell. Insbesondere erfordern fast nativer Zustand Cryo immobilisiert Datasets Niedrigdosis-Image Bedingungen, was zu einem niedrigen Signal-Rausch-Verhältnis und schlechten Kontrast, vor allem im Cryo Elektron Tomographie (CryoET)3,4,5 . Ein weiterer Faktor in einigen 3D Datensätzen ist das Vorhandensein von Artefakten, die durch die anspruchsvollen experimentellen Bedingungen beteiligten eingeführt, z. B. fehlende Keil Artefakte durch Erhebung von Daten über eine begrenzte Kippweite, was zu fehlenden Informationen und Dehnung in die Richtung des Strahls3,4,5. Auch wenn niedrige Signal-Rausch- oder fehlende Keil Artefakte sind nicht problematisch (z.B. Ionenstrahl SEM6 konzentriert oder serielle Block Gesicht SEM7), der Komplexität und dreidimensionale Art der Probe und die große Menge von Daten bedeutet, dass die Analyse würde noch einen automatisierten Prozess für Datensegmentierung profitieren.
Derzeit bei biologischen Mengen von Zellen gibt es viele Möglichkeiten für sehr spezifische zelluläre Funktionen, wie Actin, Mikrotubuli oder spezielle Proteinkomplexe, Template-basierte Suche automatisch oder halbautomatisch zu identifizieren, oder identifizieren von Features in bestimmte Arten von Datensätzen (z.B. hoher Kontrast, gebeizt, Harz eingebettete Proben)8,9,10,11,12. Allerdings sind in diesen Fällen a-priori Informationen oder bestimmten Beispiel Vorbereitung Protokolle erforderlich, die breite Anwendbarkeit dieser Segmentierung Strategien zu begrenzen. Es gibt auch Tools zur Verfügung, die Modelltraining Voxel Ebene zu lernen, die Darstellung der verschiedenen Strukturen von Interesse wenn gegebene Benutzer eingegebenen13durchführen. Auf dieser Ebene kann die Komplexität der Ausbildung und Prüfung der Modelle jedoch fehleranfällig und rechnerisch teuer sein. Angesichts der anspruchsvollen Bild-Bedingungen und das Fehlen von breit anwendbar, halbautomatische Segmentierungsstrategien, ist manuelle Segmentierung üblich, sogar bei der Arbeit mit komplexen biologischen Materialien14,15, 16 , 17. aber es ist allgemein anerkannt, dass der Prozess der manuellen Segmentierung nicht nur zeitaufwendig, sondern auch fehleranfällig, subjektive und Variable4,5,18,19 ist ,20. Einige Segmentierung Programme bieten Werkzeuge, um die manuelle Segmentierung (d.h. Interpolation, Lasso oder Schlag Tools) erleichtern21,22, jedoch in Fällen von lauten Datasets, sie sind schwer zu erfolgreich, zu bewerben und selbst wenn sie erfolgreich verwendet werden, ist der Prozess noch subjektiv und Variable.
Traditionell werden Segmentierungen auf zwei verschiedene Arten: qualitativ oder quantitativ. Imaging-Technologien und Segmentierungsstrategien verbessern, ist häufiger mit Segmentierung als ein quantitatives Tool, biologische Fragen zu beantworten und als "Ground Truth" Algorithmus Entwicklung8,12geworden, 15,23,24,25. Um dies zu tun, müssen detaillierte Überprüfungen und Balancen Variabilität und Subjektivität im gesamten Prozess26zu verringern. Diese Vorsichtsmaßnahmen weiter erhöhen jedoch die aufwändige Art der Segmentierung. Aus diesem Grund ist es wichtig, eine schnellere und weniger Variable Segmentierungsstrategie bereitzustellen.
Der SuRVoS-Workbench fängt an, diese Probleme anzugehen, indem der Benutzer mit einer Auswahl des maschinellen Lernens und Bildverarbeitungs-Tools, die den Benutzer bei der Segmentierung zu unterstützen, dabei auch den Benutzer durch die erforderlichen Schritte. Um dies zu erreichen, sind zwei wesentliche Innovationen zusammen in SuRVoS implementiert. Zunächst wird eine super-Region-Hierarchie Gruppe ähnlich, nahe gelegenen Regionen der Daten anhand ihrer inhärenten Eigenschaften verwendet. Aller Regionen in der Hierarchie repräsentiert die gleiche Lautstärke mit weniger Elementen, bieten aber trotzdem starke Begrenzung festhalten. So super-Regionen verringern Sie die Komplexität der Segmentierung ein Volumen um mehrere Größenordnungen doch noch stellen die Daten ohne erheblichen Verlust an Informationen27. Zweitens bietet SuRVoS eine halbautomatische Segmentierungsstrategie, die minimale manuelle Segmentierung Eingänge verwendet, um Klassifikatoren, die dann verwendet werden, um die restlichen Volumen28,29segment zu trainieren. Diese Strategie reduziert manuelle Segmentierung, stark verringert die Menge von Benutzer-Zeitaufwand für die Segmentierung und Verwendung von Super-Regionen entfernt manuelle Festlegung der Grenzen, potenziell reduzieren, Variabilität und Subjektivität.
Ein weiteres wesentliches Merkmal des SuRVoS ist das Tool Bezeichnung Splitter, wobei ein Benutzer kann eine Reihe von bereits segmentierten Objekten anhand ihrer inhärenten Eigenschaften klassifizieren. Nach der Aufteilung der verschiedenen Objekte von Interesse dieses Tool kann verwendet werden, um den Satz in Unterklassen basierend auf Maßnahmen wie durchschnittliche Objekt Intensität, Varianz, Größe unterteilen, Standort, etc. Dies ist nützlich, wenn große Gruppen von Objekten mit Klassifizierung hohe Komplexität. Beispielsweise kann eine Gruppe von zellulären Organellen in Mitochondrien, leeren Vesikeln, Lipid-Tröpfchen aufgeteilt werden.; eine Reihe von Material, die Einschlüsse getrennt werden können je nach Größe oder Form oder. Sobald die einzelnen Etiketten segmentiert kann in Gruppen aufteilen einer beliebigen Anzahl von Klassifikatoren, Verringerung der Identifikation Bias verwenden.
Der SuRVoS-Workbench hat Segment Daten aus mehreren bildgebenden Verfahren erfolgreich eingesetzt. Hier Synchrotron Phase Kontrast Röntgentomographie (MicroCT) der Fruchtkörper einer Pflanze verwendet wird, um Segmentati zu zeigenüber die Verwendung der Modelltraining, Cryo-Elektron Tomographie (CryoET) der menschlichen Thrombozyten dient zur Segmentierung mit Super- und Megavoxels zu demonstrieren, und Cryo soft Röntgentomographie (CryoSXT) einer Säugerzelle Linie wird verwendet, um das Label teilen Werkzeuge zeigen
Protokoll
Hinweis: nützliche Bereiche der Parameter für jeden Bearbeitungsschritt und die spezifischen Parameter für jeden Datentyp, die hier gezeigt werden in Tabelle 1 angeboten.
1. Vorbereitung einen Arbeitsbereich und Laden von Daten
- SuRVoS Workbench starten, klicken Sie auf die Schaltfläche "open Dataset" und in der daraus resultierenden Popup, wählen Sie die Datendatei segmentiert werden. Wählen Sie eine entsprechende Ausrichtung des Datasets. Als nächstes wählen Sie oder erstellen Sie einen Ordner, in dem der Arbeitsbereich und die zugehörigen Dateien gespeichert werden. Es wird empfohlen, dass dieser Ordner leer, beim Start einer neuen Segmentierung ist. Nach dem Laden der Daten die Workbench öffnet sich mit dem Bereich "Plugins" auf der linken Seite, der Bereich "Visualisierung" auf der rechten Seite und einen Satz von Tools Verknüpfungen zwischen den beiden Glasscheiben ( Abbildung 1).
2. Vorverarbeitung und Datendarstellung
- In wählen Sie ROI Registerkarte Eingabe Z, y und X Anfang und Ende Koordinaten für die Region von Interesse und klicken Sie auf hinzufügen. Wählen Sie geeignete y und X Maus Koordinaten der über einen Punkt auf dem Bild. Wählen Sie mithilfe des Schiebereglers an der Spitze des Bereichs Visualisierung Z-Koordinaten. Sobald eine Region hinzugefügt wurde, stellen Sie sicher, dass es ausgewählt ist, durch Anklicken des Kästchens auf der rechten Seite. Alle nachfolgende Berechnungen werden auf den ausgewählten Bereich durchgeführt werden. In der Regel, beginnend mit einer kleinen, repräsentativen Region of Interest (ROI), Parameter zu optimieren und dann erneut anwenden dieser Parameter auf das ganze Gebiet segmentiert werden empfohlen ist.
- In the Feature Channels-Registerkarte, verwenden Sie die Drop-Down-Menü am oberen, wählen einen Funktion/Filter und zur Warteschlange hinzufügen (siehe Diskussion für mehr Infos über Feature-Kanäle). Sobald ein Funktion/Filter hinzugefügt und durch Klicken auf den entsprechenden Namen ausgewählt wurde, ändern Sie die Optionen für die Funktion/Filter und wählen Sie das Eingabe-Dataset für die Ausführung der Funktion/Filter. Wenn alle Optionen gewählt haben, aktivieren Sie das Kontrollkästchen auf der rechten Seite mit dem Namen der Funktion/Filter zu berechnen.
- Zur Optimierung der Parameter für ein neues Dataset mehrere Filter/Features hinzufügen und wählen Sie die Parameter für sie, bevor Sie in der Reihenfolge berechnet wird, eins nach dem anderen. Um dies zu tun, fügen Sie jede neue Filterfunktion und wählen Sie die entsprechenden Parameter, aktivieren Sie das Kontrollkästchen links neben jeder Filter/Funktion ausgeführt werden soll und klicken Sie auf Feld berechnen Funktionen an der Spitze des Bereichs. Weitere Informationen finden Sie unter Diskussion.
3. Generierung von entsprechenden Super-Regionen
- In the Super Regionen Registerkarte im Abschnitt Supervoxels verwenden Quelle Drop-Down-Menü, wählen die gefilterten Datensatz aus dem Supervoxels erstellt werden. Geben Sie dann die Form, Abstand und Kompaktheit der Supervoxels (siehe Diskussion und Abbildung 2 für weitere Details). Abschließend klicken Sie auf die übernehmensschaltfläche, um die Supervoxels zu generieren. Sobald die Supervoxels erstellt wurden, können sie im Bereich Visualisierung, ein-oder ausgeschaltet und die Transparenz in der Registerkarte "Darstellung" und Viewer-Fenster Verknüpfung gesteuert eingesehen werden.
- In Super-Regionen ", im Bereich Megavoxels verwenden Quelle Drop-Down-Menü, um das gefilterte Dataset auswählen, aus dem Megavoxels erstellt wird. Geben Sie als nächstes die Lambda, NumBins und Gamma-Parameter von der Megavoxels (siehe Diskussion für weitere Details). Sobald die Megavoxels erstellt wurden, können sie im Bereich Visualisierung, ein-oder ausgeschaltet und die Transparenz in der Registerkarte "Darstellung" und Viewer-Fenster Verknüpfung gesteuert eingesehen werden.
4. Einführung in die Annotation
- In den Anmerkungen Registerkarte auf die Schaltfläche hinzufügen Ebene fügen Sie eine Beschriftung Ebene. Nachdem eine Ebene hinzugefügt wurde, verwenden Sie die Schaltfläche "hinzufügen-Label" in dieser Ebene, eine Bezeichnung für die Beschriftung hinzuzufügen. Sobald hinzugefügt, können den Namen und die Farbe des Etiketts für eine einfache Beschriftung geändert werden.
- Wählen Sie weiter, um beginnen zu kommentieren, das Stift-Symbol aus dem Werkzeug-Verknüpfung-Abschnitt. Bei Auswahl dieser Option wird eine Reihe von Optionen erscheinen an der Spitze des Bereichs Visualisierung. Diese Optionen steuern die Stiftbreite und ob Voxel, Supervoxels oder Megavoxels verwendet werden wird, um zu kommentieren.
- Zum Zwecke der Modelltraining, in der Regel wählen Sie Supervoxels im Feld Dropdown-Annotation-Ebene und eine mittelmäßige zu großen Stiftbreite sollte verwendet werden. Wählen Sie in der Registerkarte "Anmerkungen" die Bezeichnung, die durch Anklicken des Kästchens auf der rechten Seite die Etiketteninformationen beschriftet werden. Klicken Sie im Bereich Visualisierung zu kommentieren ein einzelnes Supervoxel oder klicken und ziehen viele kommentieren.
Hinweis: Voxel und Megavoxels im Feld Dropdown-Annotation Ebene ausgewählt und verwendet werden können auf die gleiche Weise, mit Anmerkungen versehen, die im Falle von Megavoxels, viele tausend ähnliche Voxel segmentiert werden mit einem einzigen Klick der Maus aktivieren können.
- Zum Zwecke der Modelltraining, in der Regel wählen Sie Supervoxels im Feld Dropdown-Annotation-Ebene und eine mittelmäßige zu großen Stiftbreite sollte verwendet werden. Wählen Sie in der Registerkarte "Anmerkungen" die Bezeichnung, die durch Anklicken des Kästchens auf der rechten Seite die Etiketteninformationen beschriftet werden. Klicken Sie im Bereich Visualisierung zu kommentieren ein einzelnes Supervoxel oder klicken und ziehen viele kommentieren.
5. Segmentierung verwenden Modelltraining demonstrierte mit einem Dataset MicroCT.
Hinweis: die erste Segmentierung für viele Datensätze ist, mehrere große Bereiche von einander zu unterscheiden. Z. B. Trennung des Kerns von Zytoplasma oder die Zelle von der externen Unterstützung und Eis Struktur. Für diese Art der Segmentierung, mit klar definierten Grenzen und große Regionen ist Modelltraining nützlich. Um dies zu demonstrieren, werden Phase Kontrast tomographische Röntgendaten des Klettenlabkraut verwendet werden.
- Laden Sie die Daten, vorverarbeiten, die Filter und Feature Suite verwenden und entsprechende Supervoxels bzw. Megavoxels wie in den obigen Abschnitten mit den Parametern in Tabelle 1 als Anleitung beschrieben zu bestimmen. Weiterhin verwenden Sie die Parameter aus Tabelle 1 und den Anweisungen in Abschnitt 4, etwa große Teile des Datasets, beschriften, wie in Abbildung 3 gezeigt.
Hinweis: Das Dataset muss nicht vollständig zu diesem Zeitpunkt segmentiert werden. - In Modelltraining Registerkarte festlegen der Predict-Ebene auf die Ebene, die die manuelle Ausbildung Anmerkungen enthält, und in den Deskriptor Abschnitt die Region auf Supervoxels gesetzt. Wählen Sie als nächstes die Deskriptoren, die Regionen der Daten unterscheiden sich durch einen Klick auf das Dropdown-Menü wählen Sie Quellen und überprüfen die Boxen der Funktionen und Filter Wahl verwendet werden (siehe Tabelle 1 und Diskussion).
- Weiter, klicken Sie auf die Schaltfläche "Predict". Nach Berechnung abgeschlossen ist, wird der Bereich "Visualisierung" aktualisiert mit den Vorhersagen für alle die nicht beschriftet Voxel zeigen, die von der Annotation-Klassen, dass sie voraussichtlich um zu gehören. In der Regel die Standardparameter für jede Klassifizierung Methodik bieten einen guten Ausgangspunkt und die Benutzer müssen nur Umschalten zwischen Klassifikatoren, eine gute Passform zu finden. Jedoch für Experten oder erfahrene Benutzer Optionen für jede Klassifizierung sind verfügbar und können geändert werden.
- Nach Beurteilung der Wirkung der Trainingsmethoden und wählen Sie eine, gelten zusätzliche Verfeinerung durch Klicken auf das verfeinern Dropdown-Liste im Abschnitt Raffinesse. Am unteren Rand das Modelltraining tab, in dem " Update Anmerkungen " Abschnitt, stellen Sie sicher, dass die Visualisierung Drop-Down-Menü auf Predictio eingestellt istNS. Verwenden Sie den Schieberegler Vertrauen Zuweisen von mehr oder weniger von der Genomsequenz Supervoxels der ausgewählten Beschriftung Beschriftungen.
- Nach der Auswahl eines angemessenen Maß an Vertrauen verwenden basierend auf Sichtprüfung, den Speichern-Knopf bei der Etiketten an der Unterseite des Werkzeugs Vertrauen um die Vorhersagen in bestimmten Etiketten speichern. Der Bereich "Visualisierung" wird aktualisiert, um die Änderungen anzuzeigen. Jedes Etikett kann separat gespeichert werden, und in der Tat Etiketten eingespart werden von kleineren Unterregionen durch Eingabe der Werte in die Ausgangs- und Z, y und X-Boxen und klicken Sie auf den speichern-Button für jedes Etikett.
- Adresse kleinere Etikettenschwindel, durch die Bereitstellung weiterer Trainingsdaten, wie in Abschnitt 4 beschrieben. Nachdem entsprechende Vorhersagen Etiketten hinzugefügt werden, wiederholen Sie den Vorgang des Modells training mit Raffinesse und hohes Vertrauen Vorhersagen hinzufügen, bis es nicht mehr gibt stumme Supervoxels. Dies ist effektiv, weil jedes Mal, wenn der Modell Trainingsprozess läuft es mehr Supervoxels mit trainieren zugeordnet sind, und daher wird der Prozess robuster als die Iterationen erhöhen.
6. Segmentierung mit super-Regionen, demonstrierte mit einem CryoET Dataset.
Hinweis: da super-Region Segmentierung eignet sich für kleinere, diskret gebundene Bereiche, der Fokus liegt hier auf Segmentierung der Organellen und Mikrotubuli innerhalb dieses Dataset. Modelltraining wurde verwendet, um schnell die Thrombozyten aus dem Hintergrund Eis und Kohlenstoff-segment; Diese Parameter sind nicht weiter diskutiert, aber sind in Tabelle 1 vorgestellt.
- Laden Sie die Daten, vorverarbeiten, die Filter und Feature Suite verwenden und entsprechende Supervoxels bzw. Megavoxels wie in den obigen Abschnitten mit den Parametern in Tabelle 1 als Anleitung beschrieben zu bestimmen. Angemessene
- hinzufügen und Beschriftungen auf der Registerkarte "Annotation" wählen Sie eine Beschriftung und beginnen Anmerkungen über eine mittelmäßige Stift Breite mit Supervoxels ausgewählt. Eingedenk der Notwendigkeit, unterschiedliche Beschriftungen für Objekte in unmittelbarer Nähe zueinander zu wählen, um zu vermeiden, beschriften sie als ein einzelnes Objekt werden.
- Um die Anmerkungen darüber hinaus bereinigen verwenden morphologische Verfeinerung Methoden (Dilatation, Erosion, öffnen, schließen und füllen Sie Löcher). Diese Optionen finden Sie am unteren Rand der Registerkarte "Anmerkungen". Wenn sie verwenden möchten, wählen Sie die Segmentierung Label und Raffinesse. Geben Sie einen Radiuswert und entscheiden Sie, wie Sie die Verfeinerung gelten. Klicken Sie dann auf verfeinern.
7. Klassifizierung und Analyse von Daten Objekte basierend auf inhärenten Eigenschaften demonstriert mit einem CryoSXT-Dataset
Hinweis: im Allgemeinen ist der nächste Schritt nach der Segmentierung Analyse der Daten. Das Tool Bezeichnung Splitter in SuRVoS kann für die Klassifizierung von segmentierten Objekte mithilfe von Regeln basierend auf Eigenschaften von Objekten wie z. B. durchschnittliche Objekt Intensität, Varianz, Lautstärke oder Position. Die Label-Statistik-Tool ermöglicht die Visualisierung der Beziehungen zwischen diesen Maßnahmen für jede neue Objektklasse. Das sind die leistungsstarken neuen Werkzeugen für die Analyse von komplexen 3D Datensätze nach Segmentierung.
- Laden Sie die Daten, vorverarbeiten, die Filter und Feature Suite verwenden, entsprechende Supervoxels bzw. Megavoxels und Segment, wie in den obigen Abschnitten mit den Parametern in Tabelle 1 als Anleitung beschrieben zu bestimmen.
- Nach Segmentierung, klicken Sie auf der zweiten Registerkarte im Bereich "Visualisierung" Label Splitter genannt. Dadurch wird einen neuen Bereich auf der rechten Seite des Fensters - Bereich Regelerstellung hinzugefügt.
- An der Spitze dieses Gebietes, wählen Sie einen angemessenen und Etiketten für die Beschriftung aufteilen. Dann wählen Sie das Dataset Abfragen und klicken Sie auf Beschriftung. Alle Objekte in den ausgewählten Labels wird jetzt blau umrandet werden als separate Objekte im Bereich Visualisierung und ein Plot zeigt die durchschnittliche Intensität der Objekte im Bereich Regelerstellung angezeigt werden. Um die Maßnahme in die Handlung gezeigt zu ändern, klicken Sie auf das Dropdown-Listenfeld am oberen Rand der rechten Seite.
- Zu beginnen, um die Objekte in relevanten Klassen aufgeteilt klicken Sie auf neues Etikett an der Unterseite des Bereichs Regelerstellung hinzufügen. Den Namen und die Farbe dieser neuen Bezeichnung zugeordnet können geändert werden, wie zuvor beschrieben.
- Hinzufügen, klicken Sie auf neue Regel und über die Dropdown-Liste und Freiform Eintrag Felder definieren die Regel angewendet werden. Klicken Sie auf anwenden, um die Auswirkungen der Neuregelung im Bereich Visualisierung und die Handlung im Bereich Regelerstellung sehen. Mehrere Regeln können auch auf ein einzelnes Etikett und mehrere Labels können innerhalb der gleichen Dataset erstellt werden.
Hinweis: Um alle unmarkierten Objekte zu sammeln, erstellen Sie eine neue Beschriftung und anstatt eine Regel hinzufügen, klicken Sie auf Wählen Sie andere.
- Hinzufügen, klicken Sie auf neue Regel und über die Dropdown-Liste und Freiform Eintrag Felder definieren die Regel angewendet werden. Klicken Sie auf anwenden, um die Auswirkungen der Neuregelung im Bereich Visualisierung und die Handlung im Bereich Regelerstellung sehen. Mehrere Regeln können auch auf ein einzelnes Etikett und mehrere Labels können innerhalb der gleichen Dataset erstellt werden.
- Wenn neue Bezeichnungen der Objekte von Interesse aufgeteilt haben, erstellen Sie eine neue, leere Ebene in der Registerkarte "Anmerkungen". Dann wählen Sie diese Stufe in der Registerkarte "Regel erstellen" und klicken Sie auf Speichern Etiketten. Das spart die neuen Etiketten auf diese leere Ebene.
- Am Rande des Bereichs Visualisierung, klicken Sie auf die Registerkarte Beschriftung Statistik. Dadurch wird einen neue Visualisierung Bereich geöffnet, der verwendet werden können, zu beginnen, um Zusammenhänge zwischen Objektklassen in den Daten zu verstehen. Wählen Sie oben ein angemessenes und Etiketten und das Dataset auf Abfrage.
- Wählen Sie ein paar von den Maßnahmen von Interesse durch Markieren der Kästchen neben ihnen. Klicken Sie dann auf Beschriftung. Dies wird für jeden der ausgewählten Maßnahmen paarweisen Vergleich Grundstücke produzieren. Um die Grundstücke zu aktualisieren, wählen Sie oder deaktivieren Sie geeignete Maßnahmen und klicken Sie auf Update Plot.
8. Export von Daten und Segmentierungen
- exportieren die Grundstücke und die rohen Messdaten durch Anklicken Export Plot und Export von Daten, bzw. in der Registerkarte Beschriftung Statistik des Bereichs Visualisierung.
- Export der Bilddaten und Segmentierungen durch Klicken auf die Registerkarte "exportieren" im Bereich "Plugins". Klicken Sie zunächst, um einen Ordner auszuwählen, wo die Daten gespeichert werden sollen. Wählen Sie als nächstes die Ausgabe (Raw-Daten, Raw Anmerkungen, Segmentierung Masken oder maskierten Daten) und das Format (HDF5, MRC oder TIFF). Schließlich wählen Sie die Annotation Ebene(n) exportiert werden mithilfe der Kontrollkästchen und klicken Sie auf exportieren. Die Daten können skaliert und invertiert vor dem exportieren wie nötig. Wenn die maskierten Daten exportieren, das Dataset, das die Mask(s) angewendet werden kann ausgewählt werden mit einem Dropdown-Menü.
Ergebnisse
Drei Volumen Datensätze gesammelt aus drei verschiedenen Techniken (MicroCT, CryoET und CryoSXT) wurden verwendet, um drei wichtige Merkmale der SuRVoS-Workbench zu demonstrieren: Modell, Ausbildung, super-Region-Segmentierung und Label aufteilen. Die Datasets repräsentieren eine heterogene Gruppe von experimentellen Ergebnissen, von die jede volle Verarbeitungsparameter (Tabelle 1) vorgesehen sind.
Um Modelltraining mit SuRVoS-Workbench zu demonstrieren, wurde ein relativ hohem Kontrast Dataset mit Region Abgrenzung gewählt. Dieses Dataset von der Frucht des Galium Aparineoder Klettenlabkraut, wurde mittels Röntgentomographie Phase Kontrast auf das I13-2 Diamant-Manchester Imaging Strahlrohr auf Diamond Light Source, Chilton, Oxfordshire, Vereinigtes Königreich erhoben. Die frische Probe wurde in Luft auf ein Goniometer Basis auf dem Drehtisch montiert, bei einer Probe-Detektor-Abstand von 30 mm. Belichtung Zeiten waren 0,10 s mit dem rosa Strahl-Spektrum hat eine mittlere Energie von rund 22 keV. Projektionen wurden durch 180° mit einer Schrittweite von 0,1 ° gesammelt. Tomographische Rekonstruktionen wurden mit Savu durchgeführt30,31 mit dem Paganin Filter für Vermehrung-basierte Phasenkontrast Bilder32 gefolgt von gefilterten Rückprojektion Rekonstruktion in der ASTRA-Toolkit-33 , 34. diese Daten wurde dann mit 2 x 2 x 2 binning um die Dateigröße zu reduzieren, bevor Sie in die SuRVoS-Workbench eingespeisten verkleinert.
Erstens war die Eingabedaten (Abbildung 3A) gefiltert und geklemmt (zum oberen und unteren Intensitätswerte in den Daten entfernen) (Abb. 3 b). Auf diese Weise die Hintergrund- und Vordergrundfarben wurden leichter unterscheidbar und die Textur der inneren Struktur der Frucht wurde betont. Als nächstes wurden Supervoxels auf der Oberseite des gefilterten Datasets (Abbildung 3) gebaut. Um die Qualität der Supervoxels zu beurteilen, wurden sie angezeigt, ohne die Daten zu überprüfen, ob die relevanten Details des Datensatzes durch die Supervoxels (Abbildung 3D) gut vertreten waren. Nächste, manuelle Anmerkungen mit Supervoxels wurden als Trainingsdaten auf drei Scheiben des Volumens (Abbildung 3E, dunkle Farben) zur Verfügung gestellt. Diese Trainingsdaten reichte der Sichter (Lichtfarben) die Bereiche entsprechend den Hintergrund (grün), Obst Borste (rot), Saatgut (lila), Vorhersagen zu trainieren und den umliegenden Fleisch (blau). Morphologische Verfeinerungen wurden verwendet, um die Segmentierungen bereinigen durch füllen von Löchern, wachsen oder schrumpfen als benötigt (Abbildung 3F). Die Gesamtzeit ausgegeben, um entsprechende Parameter zu ermitteln und dieses Dataset-segment war 2 h.
Um Super-Region Segmentierung mit SuRVoS-Workbench zu demonstrieren, wurde laut und komplexe Dataset15gewählt. Dieses Dataset wurde mittels CryoET am National Center for Macromolecular Imaging am Baylor College of Medicine, Houston, TX USA erhoben. Thrombozyten waren kurz, Sprung auf Glut entladen gefroren und gold so behandelt löchrigen Kohlenstoff TEM Raster. Tilt-Serie wurden von ±65 ° mit einer 2° Inkrement gesammelt. Der Tilt-Serie wurde dann umgebaut mit gewichteten Rückprojektion in IMOD35.
Nach dem Laden der Daten in SuRVoS (Abb. 4A), eine Region of Interest wurde ausgewählt und ein entsprechenden Filter-Satz angewendet wurde. In diesem Fall wurde ein Glättungsfilter "glockenförmig" gefolgt von einem gesamten Variation-Filter mit dem Kontrast geklemmt zur Akzentuierung der Kanten und Texturen der Daten (Abbildung 4 b). Als nächstes war Modelltraining mit minimalen Supervoxel-basierte Benutzereingaben verwendet, um das Plättchen aus dem Hintergrund Eis und Kohlenstoff-segment. Dann war semi-manueller Segmentierung mit Megavoxels und Supervoxels verwendet, um die Organellen zu segmentieren. Zu guter Letzt der Quellparameter Supervoxel wurde geändert, um eine schwächere Entrauschen Filter und Supervoxel Form wurde kleiner (siehe Tabelle 1) vorgenommen, um die Mikrotubuli-Grenzen für die Segmentierung (Abbildung 4) besser erhalten bleibt. Für die Organellen und der Mikrotubuli, schnelle manuelle Anmerkungen verwendet wurden alle 5-10 Scheiben der Supervoxels auswählen, die die Funktion von Interesse zu beschreiben (Abbildung 4 & 4E). Die Gesamtzeit ausgegeben, um entsprechende Parameter zu ermitteln und zu segmentieren des Interessenbereichs präsentiert war 6 h.
Um Label Aufteilung mit SuRVoS-Workbench zu demonstrieren, wurde ein Dataset mit vielen, abwechslungsreichen Organellen gewählt. Dieser Datensatz wurde mittels CryoSXT am Strahlrohr B24 bei Diamond Light Source, Chilton, Oxfordshire, Vereinigtes Königreich36erhoben. Kurz, HEK293 Zellen auf gold Finder Gitter angebaut wurden, entsprechend dimensionierte gold Fiducials wurden hinzugefügt und das Gitter war Sprung mit ein EM mit Rücken-seitig beflecken eingefroren. Tilt-Serie wurden dann am Mikroskop von ±65 ° mit einer 0,5 ° Inkrement gesammelt. Der Tilt-Serie wurde dann umgebaut mit gewichteten Rückprojektion in IMOD35.
Nach dem Laden der Daten in SuRVoS (Abb. 5A), eine Region of Interest wurde ausgewählt und ein entsprechenden gesamten Variation-Filter wurde verwendet, um die Grenzen der Organellen im gesamten Volumen (Abb. 5 b) zu verbessern. Als nächstes wurden Organellen semi manually segmentiert, mit Megavoxels und Supervoxels, und dann verfeinert mit Löcher füllen, schließen und Dilatation, glatte Kanten (Abbildung 5). Die Gesamtzeit für entsprechende Parameter zu ermitteln und zu segmentieren des Interessenbereichs vorgestellt wurde 4 h. Sobald die Segmentierung abgeschlossen war, wurde das Label Splitter verwendet, um jede Organelle als Objekt in das Dataset (Abbildung 5) und verschiedenen Merkmalen über jedes Objekt in der Daten-Plot (Abb. 5E) zu visualisieren. Die Label-Splitter-Schnittstelle ist interaktiv, Aktualisierung jede neue Label-Klasse in die Visualisierung und die Daten-Handlung zugeordnete Farbe. Dies ermöglicht die Erstellung von verschiedenen Regeln anhand der Merkmale innewohnt, die Daten, die verwendet werden können, um die Objekte in nützliche Klassen (Abb. 5F) zu trennen.

Abbildung 1. Das Layout und die allgemeinen Merkmale der SuRVoS-Workbench.
Die GUI-Schnittstelle ist auf der linken Seite, während der Bereich "Visualisierung" auf der rechten Seite ist. Diese beiden Bereiche sind durch eine Spalte von Werkzeugen und Shortcuts getrennt. Die GUI ist angeordnet, um den Benutzer durch die wichtigsten Schritte in der Vorverarbeitung der Daten Auswahl Supervoxel und/oder Megavoxel Parameter, Segmentierung der Daten gehen und bei Bedarf mit Modelltraining, vor dem Export die Segmentierungen. Der Bereich "Visualisierung" kann in drei Modi verwendet werden: einfache Visualisierung und Segmentierung zum Anzeigen der Datenund irgendwelche Filter angewendet, die Daten zu segmentieren beschriften Splitter um Objekte in neuen Etiketten basierend auf Aspekte auf die Daten zu kategorisieren, und schließlich Statistiken zu messen und zu visualisieren, Merkmale der segmentierten Objekte beschriften. Für jede dieser Modi die Drop-Down-Menü oben links Ecke steuert, welche Daten angezeigt werden, und mithilfe des Schiebereglers an der Spitze können die z-Achse. Die Werkzeug-Verknüpfungen bietet einfachen Zugangskontrolle über Kontrast, Transparenz der Schichten, zoomen, schwenken und Rückkehr zu "Home" im Bereich "Visualisierung" und Öffnungswerkzeuge für Anmerkung im Protokoll beschriebenen. Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 2. Super-Region-Hierarchie verringert die Komplexität des Image Segmentation.
Ein Bild aus dem Berkeley Segmentierung Dataset (BSDS50037) wurde verwendet, um die Eigenschaften und Wirkungen von Super-Regionen zu demonstrieren. Das Originalbild (links) besteht aus Tausenden von Voxeln, die dann in benachbarten, ähnliche Gruppierungen erstelle ich ein paar hundert Supervoxels (Mitte) versammelt sind. Supervoxels können auch in benachbarten, ähnliche Gruppierungen erstelle ich ein paar Dutzend Megavoxels (rechts) gesammelt werden. Mit jeder Gruppierung wird die Komplexität der Aufgabe Segmentierung für Rechen- und manuelle Ressourcen verringert. Wichtig ist, ein 2D Beispiel hier, aber Supervoxels und Megavoxels sind 3D. Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 3. Verarbeitung von ein MicroCT Dataset mithilfe einer Schulungsstrategie Segmentierung Modell.
A. eine einzige 2D Scheibe der Rohdaten. B. anwenden eines Filters eingespannten total Variation auf die raw-Daten erweitert die Grenzen zwischen den verschiedenen Aspekten der Fruchtkörper. C. geeignete Supervoxel Parameter wurden ausgewählt. D. eine Zielregion (rot markiert in C) wird angezeigt, zu zeigen, dass die Grenzen der Daten in die Supervoxels selbst vorhanden sind. E. drei Scheiben des Volumens mit manuellen Beschriftungen der verschiedenen Bereiche des Datasets in dunklen Farben (grün, rot, blau und lila) und nach Modell Lauftraining Prognosen angezeigt in den gleichen hellen Farben. F. die gleichen drei Scheiben mit der endgültigen Segmentierung nach Ausbildung Modellvorhersagen angenommen worden sind. Maßstabsleisten sind 1 mm. Bitte klicken Sie hier für eine größere Version dieser Figur.
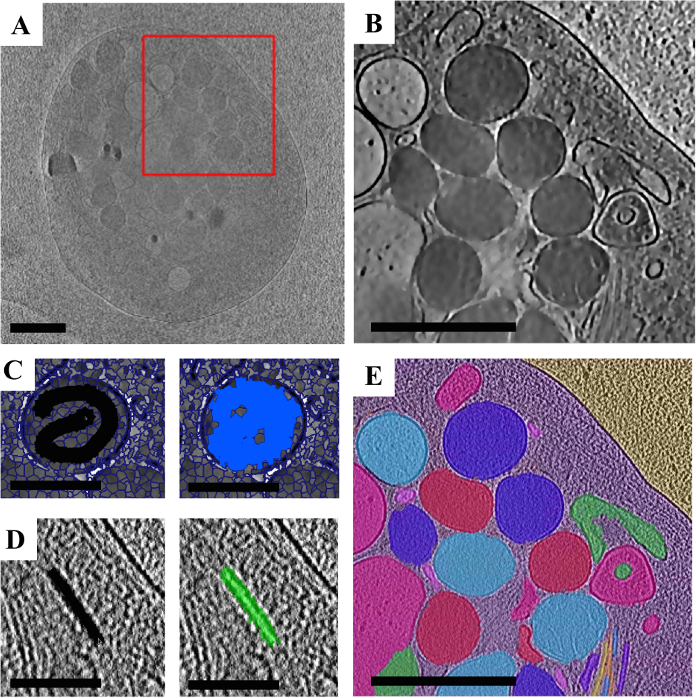
Abbildung 4. Verarbeitung von einem CryoET Dataset mithilfe eine Segmentierungsstrategie Super-Region.
A. eine einzige 2D Scheibe der Rohdaten. B. einer Region of Interest (rot markiert in der A) mit einem mehrschichtigen Filter angewendet, um die Grenzen der Organellen zu akzentuieren. C. Beispiel kommentieren ein Organell mit super-Regionen. Ein einzelnes Organell zeigt mit Supervoxels überlagert die manuelle Benutzer Anmerkung angezeigt in schwarz (links) und die Supervoxels gewählt, mit diesem Vermerk in blau (rechts) dargestellt. D. Beispiel kommentieren ein Mikrotubuli mit super-Regionen. Eine Region der Mikrotubuli zeigt sich mit dem manuellen Benutzer Vermerk angezeigt in schwarz (links) und die Supervoxels gewählt, mit diesem Vermerk in grün (rechts) dargestellt. E. die letzte Segmentierung mit den Thrombozyten aus dem Hintergrund mit Modell (siehe Tabelle 1 für Details), Ausbildung und verschiedenen Organellen und Mikrotubuli segmentiert, mit einem super-Region Segmentierungsstrategie segmentiert. Farben bedeuten nicht bestimmten Organell-Typen, wie sie hier vor der Klassifizierung angezeigt werden. Skalieren, Bars in A, B und E sind 1 μm und in C und D sind 0,5 μm. Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 5. Analyse einer CryoSXT Dataset mit dem Label-Splitter-Tool.
A. eine einzige 2D Scheibe der Rohdaten. B. einer Region of Interest (rot markiert in der A) mit einem gesamten Variation Filter angewendet, um die Organellen zu akzentuieren. C. die endgültige Segmentierung mit Supervoxels überlagert. D. Der Visualisierung Teil der Label-Splitter mit der Organellen, klassifiziert nach den Regeln in Fangezeigt. E. der Handlung Teil der Label-Splitter zeigt die durchschnittliche Intensität innerhalb jedes Objekt mit den Regeln in F angewendet angezeigt. Jede vertikale Linie entlang der x-Achse repräsentiert ein einzelnes Objekt und ist farbkodiert, um die Klasse übereinstimmen, die, der es zugewiesen wurde. F. Beispiel Klassifizierungsregeln, verschiedene Objekte anhand ihrer inhärenten Eigenschaften zu trennen. Maßstabsleisten sind 1 μm. Bitte klicken Sie hier für eine größere Version dieser Figur.
| Name / Dataset | Quelle | P1 | P2 | P3 | P4 |
| Gauß-Filter | Sigma | ||||
| Range / Standard | - | [0.5, 10] / 1 | |||
| (G1) cryoET | RAW-Daten | 1 | |||
| (G2) cryoET | RAW-Daten | 2 | |||
| Gesamten Variation | Lambda | Abstand | # Iter | Klemme | |
| Range / Standard | - | [0,1, 30] / 10 | [0,1, 10] / 1 | [50, 500] / 100 | - |
| (TV1) microCT | RAW-Daten | 10 | 1 | 100 | (1,-) |
| (TV2) cryoET | G1 | 7 | 1 | 200 | - |
| (TV3) cryoET | G2 | 10 | 1 | 100 | - |
| (TV4) cryoSXR | RAW-Daten | 7 | 1 | 100 | - |
| Schnittstellenüberwachung | Vmin | Vmax | |||
| Range / Standard |
Tabelle 1. Optimierte Parameter verwendet, um jedes der drei Datasets (MicroCT, CryoET und CryoSXT) zu verarbeiten.>
Für jeden Parameter eine Allzweck-Range und Standard gegeben. In vielen Fällen dient die gefilterte Daten als Quelle für downstream-Processing. In diesen Fällen ist eine Abkürzung verwendet, um die neue Quelle Dataset zu bezeichnen. Zum Beispiel G1 ("glockenförmig" gefilterte CryoET Rohdaten) diente als Eingang während eines gesamten Variation Filters TV2 erstellen. Informationen werden nur für Aspekte der Workbench dargestellt, die verwendet wurden, um jedes Dataset zu verarbeiten. Zum Beispiel Modelltraining wurde nicht während der Verarbeitung des hier vorgestellten CryoSXT-Datasets verwendet, daher sind keine Parameter für diese gegeben.
Diskussion
SuRVoS-Workbench unterscheidet sich von anderen Programmen Segmentierung Optimierung der Parameter ist ein notwendiger und wichtiger Schritt vor Beginn der eigentlichen Segmentierung. In einigen Programmen manuellen oder halb-manuelle Segmentierung beginnt der Benutzer Segmentierung innerhalb weniger Augenblicke ein neues Projekt zu öffnen. Mit SuRVoS ist, weil große Mengen des Volumens mit sehr wenig Benutzereingaben segmentiert werden und Grenzen vom Programm abgegrenzt sind Optimierung der Parameter entscheidend für eine erfolgreiche Segmentierung. Insbesondere sind Funktion Kanäle und super-Region-Building zwei Bereiche, denen Aufmerksamkeit geschenkt werden sollte.
Feature-Kanäle und Modelltraining
Zusätzlich zu den Rohdaten ermöglicht SuRVoS dem Benutzer zusätzliche Datasets oder Kanäle abgeleitet aus einem vorhandenen Dataset erstellen. Diese Kanäle können mit einer Auswahl von computergestützten Methoden oder Feature Extraktoren erstellt werden. Jede Datendarstellungen stehen parallel und können einzeln angezeigt werden, um Ergebnisse der Funktion oder Filter Anwendung zu beurteilen. Aufgrund dieser Eigenschaften sind sie als Feature Kanäle in SuRVoS bezeichnet. Es gibt viele Feature-Kanal-Optionen innerhalb von SuRVoS. Für Informationen über die Optionen und Parameter, die hier verwendet siehe Tabelle 1, für eine vollständige Liste und Beschreibung der verfügbaren Feature Kanäle https://diamondlightsource.github.io/SuRVoS/ 2besuchen. Erste, laut Datasets profitieren vom denoising mit dem Gauß oder insgesamt Variation Filter. Es wird empfohlen, die weitere Funktion Kanal und Supervoxel/Megavoxel Berechnungen durchgeführt werden, unter Verwendung eines dieser denoised Datensätze als Datenquelle. Im Allgemeinen ist die totale Variation verrauscht Dataset als Quelldaten für Kanal- und Supervoxel/Megavoxel Berechnungen Features verwendet. Es wird empfohlen, führen Sie zunächst mit den Standardwerten, das Ergebnis in 3D zu bewerten und schließlich iterativ optimieren die Parameter für das Dataset. Darüber hinaus Funktion, die Kanäle in "Filter-Sets", speziell aufgebaut werden können isoliert Aspekten des Datasets, und diese kann dann als Datenquellen verwendet werden, um Supervoxels und Megavoxels zu schaffen. Während diese Strategie sehr Dataset angewiesen ist, kann es vorteilhaft sein.
Feature-Kanäle dienen auch als Quellen des Klassifikators in Modelltraining zu trainieren. Bei der Entscheidung, auf welche Funktion Kanäle verwenden, wird empfohlen, dass ein paar robuste Funktion Kanäle (z. B.von Blob Erkennung, Textur und Struktur oder robusten Funktionen Kategorien) beim Arbeiten mit einer kleinen Menge von Anmerkungen verwendet werden, um zu trainieren die Sichter. Beim Arbeiten mit einer großen Menge von Trainingsdaten, es empfiehlt sich, mehr Funktion Kanäle insgesamt, aus einer der Kategorien zu verwenden, solange sie vielfältige Informationen zu den Klassifikator bieten (z.B., hinzu kommen die obige Liste Feature Kanäle von lokalen Funktionen und Gaußsche Funktionen Kategorien).
Es gibt drei Hauptteile, Ausbildung zu modellieren: Bereitstellung von input-Daten-Quellen, die die Daten beschreiben, verwenden diese Eingänge, um eine Klassifizierung zu trainieren und schließlich verfeinern die Ausgang Vorhersagen. Kleinere Bereiche der Daten erfordern in der Regel, mehr Benutzer Anmerkungen genau der Sichter, trainieren, während größere Regionen die Daten weniger Benutzer Anmerkungen benötigen. Modelltraining kann zuerst ohne Auswahl einer Verfeinerung verwendet werden, um die besten Vorhersagen zu finden. Dann gehören Sie die Verfeinerung und optimieren Sie den Lambda-Parameter zu, wie notwendig, um Probleme mit den Vorhersagen wie Löcher oder gezackte Ränder zu beheben.
Supervoxels und megavoxels
Supervoxels sind Gruppen von mehreren in der Nähe, ähnliche Voxel38,39. Supervoxels beginnen als standard 3D Gitter überlagert, die Daten, die dann iterativ verformt werden, um die zugrunde liegenden Grenzen einzuhalten und somit besser darstellen der Daten. Supervoxel Erstellung und Verformung wird durch vier Eingaben des Benutzers gesteuert: Datenquelle, Superpixel Form, Abstand und Kompaktheit. Die Datenquelle enthält die Dateneingaben, die während der Erstellung der Supervoxel abgefragt werden. Jede Quelle kann verwendet werden, einschließlich der gefilterten Datenquellen. Die Superpixel Formparameter bestimmen die Startaufstellung 3D und die ungefähre gewünschte Form des daraus resultierenden Supervoxels. Änderung dieser Parameter erhöhen oder verringern Sie die Größe des Supervoxels vor Verformung. Die Abstände Parameter definieren die Bedeutung von Grenzen in jeder Richtung. Änderung dieser Parameter kann betonen, Grenzen in einer oder zwei Richtungen auf Kosten des anderen (s), d. h. die resultierende Supervoxels werden verformen, um Daten in die angegebene Richtung Grenzen besser zu folgen. Der letzte Parameter, Kompaktheit, steuert, wie viel die Supervoxels verformen kann. Eine Reihe von niedrigen Kompaktheit ermöglicht die Supervoxels um mehr zu verformen. Diese Parameter sollten optimiert werden, um Supervoxels zu bieten, die die Grenzen der relevanten Daten darstellen. Hinweis: Derzeit Supervoxel Formparameter müssen gleich 1024 oder weniger wenn miteinander multipliziert.
In gewisser Weise Supervoxel Parameter ausgleichen können, d. h. es gibt keine "richtige Antwort" bei der Entscheidung über Parameter. Zum Beispiel eine große Startaufstellung (z.B. Superpixel Form: 10 x 10 x 10) und eine niedrige Kompaktheit Nummer (z.B. 20) kann Supervoxels mit ähnlichen Grenze festhalten im Vergleich zu einem kleinen Startaufstellung (z.B. Superpixel Form 5 x 5 x 5) und eine höhere Kompaktheit Zahl (z.B. 50). Da gibt es mehr, kleinere Supervoxels im zweiten Szenario, sie müssen nicht so viel Grenzen darstellen zu verformen. Beide Parameter können für Segmentierung des Datasets geeignet.
Die größte Betrachtung bei der Auswahl Supervoxel Parameter wie gut die Supervoxels die Daten darzustellen. Anzeige der Supervoxels allein, ohne Daten darunter, wie in Abb. 2D, ist eine gute Möglichkeit, Supervoxel Parameter zu bewerten. Wenn auf diese Weise angezeigt, sollten die Kanten und Umrisse von Formen, die in den Daten gefunden noch in der Supervoxels sichtbar.
Megavoxels sind Konglomerate von mehreren benachbarten, ähnlich wie Supervoxels38,39. Sie werden wieder von vier Eingaben des Benutzers gesteuert: Datenquelle, Lambda, Numbins und Gamma. Wie bei Supervoxels, stellt die Datenquelle die Dateneingaben, die während der Erstellung der Megavoxel abgefragt werden. Lambda und Numbins Auswirkungen auf die Größe und Grenze Einhaltung der Megavoxels. Wenn die Megavoxels größer (hohe Lambda, niedrige Numbins) wachsen, nimmt ihr Grenze festhalten. Das Gegenteil trifft, Einhaltung der Grenze erhöht sich mit kleineren Megavoxels (niedriger Lambda, hohe Numbins), aber wie die Megavoxel Größe abnimmt, so ist ihre Nützlichkeit in schnell große Mengen an Voxel Segmentierung. Der optionale Gamma-Parameter steuert die Glätte Faktor im Vergleich zu den beiden Supervoxels zusammenführen. Kleine Werte der Gamma können die Ähnlichkeit zwischen zwei Supervoxels, um den Preis mit weniger Megavoxels insgesamt verbessern.
Wie bei Supervoxels, ist die größte Betrachtung bei der Auswahl und Optimierung Megavoxel Parameter, wie gut die Megavoxels die Daten darzustellen. Anzeige der Megavoxels allein, wie bei Supervoxels beschrieben kann wieder verwendet werden, Parameter zu bewerten. Jedoch weil Megavoxels in der Regel viel größer werden und sind dreidimensional, mit dem die Annotation Werkzeuge to Wählen Sie einzelne Megavoxels um sicherzustellen, dass die Grenze zwischen den Regionen von Interesse eng wird auch empfohlen.
Annotation-Strategie
Zwei allgemeine Anmerkung Strategien beschrieben worden: ein Modellansatz Training eignet sich für große Regionen eines Datasets trennt, ist zwar ein super-Region Segmentierung Ansatz nützlich für kleinere, vielfältiger Funktionen wie einzelnen Organellen. Anmerkungen können in einer hierarchischen Weise organisiert werden, so dass es möglich ist, große Gebiete zuerst kommentieren, dann unterteilen sie in spezifische Bereiche mit einer Parent-Child-Beziehung. Die übergeordnete Bezeichnung für ein Label kann durch Klick auf den Bereich rechts vom Label-Farb-Auswahl und wählen Sie eine entsprechende Elternteil als Beschriftung aus einer vorherigen Ebene zugewiesen werden. In der Praxis verwenden die meisten Datensätze die Modelltraining und super-Region Segmentierungsstrategien Regionen/Besonderheiten des Interesses zu segmentieren.
In der Ausbildung Musterbeispiel hier waren ein paar Trainings-Eingänge (in Form von manuellen Benutzer Supervoxel-basierte Anmerkungen) auf drei gleichmäßig angeordneten Scheiben der Daten verwendet. Auf diese Weise erhöht der Modell-Training-Aspekt des SuRVoS erheblich die Geschwindigkeit, mit der Segmentierung möglich ist, vor allem bei Arbeiten mit großen, differenzierte Regionen wie die Kluft zwischen den Regionen in den Klettenlabkraut Fruchtkörper wie in hervorgehoben Abbildung 3.
Beim Modell kann Ausbildung, wenn die Vorhersagen nicht sichtbar sind, es notwendig sein, gehen Sie auf die Registerkarte "Darstellung" und stellen Sie sicher, dass die Vorhersagen Schicht eingeschaltet ist und auf einen angemessenen Betrag von Transparenz gesetzt. Auch wird ein Vertrauen von 0 zuweisen jeder unbeschriftete Supervoxel ein Label, basierend auf was auch immer das nächste Spiel ist. Vertrauen von 100 weisen nur ein Etikett, wenn nur eine Kategorie von Label proportionale Übereinstimmung hat. Alles dazwischen ist ein Kompromiss zwischen diesen beiden extremen. Bei der Auswahl von einer Konfidenzniveau wird vorgeschlagen, um ein paar Scheiben Sichtprüfung zu überprüfen, die gibt es kein falsch vorhergesagte Voxel vor dem Speichern der Vorhersage auf einem Etikett.
Eine gute Strategie für kommentieren, mit super-Regionen ist die Vergrößerung Tool vergrößern Sie die Daten, mit Anmerkungen versehen ein paar Organellen auf einmal auf eine Scheibe, eine "schnelle, chaotisch" Ansatz zuerst zu verwenden (Abbildung 4). Als nächstes bewegen nach oben oder unten ein paar Scheiben in Z, und wiederholen Sie diesen Vorgang. Weil Supervoxels dreidimensional sind, sind viele der Fehler der "unordentlich" Ansatz von Anmerkungen in die oben oder unten Scheiben fixiert. Auf diese Weise Segmentierung wird beschleunigt und die Grenzen werden durch die Supervoxels anstatt manuell zur Verfügung gestellt.
Um eine Beschriftung zu bereinigen, wurden standard Segmentierung Verfeinerung Optionen bereitgestellt. Dilatation verursacht das ausgewählte Segmentierung Label durch den angegebenen Radius wachsen, Erosion verursacht es zu schrumpfen. Öffnen und schließen sind die Anwendung der ersten Erosion und Dilatation, oder umgekehrt, bzw.. Und Löcher füllen tut genau das. Die Reihenfolge dieser Operationen spielt Rolle. In der Regel Durchführung Löcher füllen, dann öffnen, dann Dilatation funktioniert gut. Jede Raffinesse-Methode kann auf eine einzige Scheibe ("Diese Scheibe"), auf alle Scheiben in 2D ("alle Slices (2D)") oder in 3D ("ganze Volume (3D)") angewendet werden. Alle Scheiben (2D) wird empfohlen.
Bedeutung und zukünftige Richtungen
Effiziente und genaue Segmentierung ist der nächste Engpass bei der Verarbeitung von 3D Datensätzen, vor allem mit der Routine automatisierte Erfassung von Terabyte Bilddaten während langfristig Sitzungen. SuRVoS-Workbench kann um den Faktor 5 im Vergleich zu manuellen Segmentierung den Segmentierung zu beschleunigen. Auch, weil die Grenzen von Supervoxels abgegrenzt werden, sollte die Variabilität der daraus resultierenden Segmentierungen verbessern. In Zukunft wollen wir Möglichkeiten der Verwendung der Segmentierung von einer repräsentativen 3D Region of Interest als Trainingsdaten auf den Rest des Volumens oder sogar einen separaten Datenträger mit hohem Vertrauen anwenden. Dieser Fortschritt würde die Menge der Benutzerzeit und Eingabe notwendig, Segment auch komplexe biologische Volumen, um den Bild-Verarbeitung und Segmentierung Engpass zu lindern helfen weiter zurückgehen. Dies, ermöglicht wiederum den quantitativen Vergleich der biologischen Daten in verschiedenen Zuständen (z. B. nicht-Krankheit, Krankheit, behandelt) mit robusten experimentelle zahlen.
Offenlegungen
Die Autoren erklären, dass sie keine konkurrierenden finanziellen Interessen haben.
Danksagungen
Wir würden gerne anerkennen und vielen Rui Wang und Wah Chiu vom Baylor College of Medicine für die Bereitstellung des CryoET-Datasets und Andrew Bodey von Diamond Light Source für die Unterstützung mit I13 Strahlzeit. Teile dieser Forschung wurden von den National Institutes of Health (NIH) Grant Nr. (P41GM103832) erkennen wir Diamond Light Source gemeinsam Förderanträge Imanol Luengo unter PhD STU0079 unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| computer | n/a | n/a | Must be running Linux operating system and have an NVidia GPU with at least 4 GB of memory |
Referenzen
- DiamondLightSource/SuRVoS: Version 1.0. Zenodo. , Available from: https://zenodo.org/record/247547 (2017).
- SuRVoS Workbench - Super Region Volume Segmentation. , Available from: https://diamondlightsource.github.io/SuRVoS/ (2017).
- Frangakis, A. S., Förster, F. Computational exploration of structural information from cryo-electron tomograms. Curr Opin Struct Biol. 14 (3), 325-331 (2004).
- Lucić, V., Förster, F., Baumeister, W. Structural studies by electron tomography: from cells to molecules. Annu Rev Biochem. 74, 833-865 (2005).
- Lučič, V., Rigort, A., Baumeister, W. Cryo-electron tomography: the challenge of doing structural biology in situ. J Cell Biol. 202 (3), 407-419 (2013).
- Milani, M., Drobne, D. Focused ion beam manipulation and ultramicroscopy of unprepared cells. Scanning. 28 (3), 148-154 (2006).
- Denk, W., Horstmann, H. Serial Block-Face Scanning Electron Microscopy to Reconstruct Three-Dimensional Tissue Nanostructure. PLoS Biol. 2 (11), (2004).
- Rigort, A., et al. Automated segmentation of electron tomograms for a quantitative description of actin filament networks. J Struct Biol. 177 (1), 135-144 (2012).
- Förster, F., Han, B. -G., Beck, M. Visual Proteomics. Meth Enzymolb. 483, 215-243 (2010).
- Liu, Y., Sigworth, F. J. Automatic cryo-EM particle selection for membrane proteins in spherical liposomes. J Struct Biol. 185 (3), 295-302 (2014).
- Asano, S., et al. A molecular census of 26S proteasomes in intact neurons. Science. 347 (6220), 439-442 (2015).
- Garduño, E., Wong-Barnum, M., Volkmann, N., Ellisman, M. H. Segmentation of electron tomographic data sets using fuzzy set theory principles. J Struct Biol. 162 (3), 368-379 (2008).
- Sommer, C., Straehle, C., Köthe, U., Hamprecht, F. A. Ilastik: Interactive learning and segmentation toolkit. 2011 IEEE Int Symp Biomed Imaging: From Nano to Macro. , 230-233 (2011).
- Darrow, M. C., et al. Structural Mechanisms of Mutant Huntingtin Aggregation Suppression by the Synthetic Chaperonin-like CCT5 Complex Explained by Cryoelectron Tomography. J Biol Chem. 290 (28), 17451-17461 (2015).
- Wang, R., et al. Electron cryotomography reveals ultrastructure alterations in platelets from patients with ovarian cancer. Proc Natl Acad Sci USA. 112 (46), 14266-14271 (2015).
- Shahmoradian, S. H., et al. TRiC's tricks inhibit huntingtin aggregation. eLife. 2, (2013).
- Dai, W., et al. Visualizing virus assembly intermediates inside marine cyanobacteria. Nature. 502 (7473), 707-710 (2013).
- Sandberg, K. Methods for Image Segmentation in Cellular Tomography. Methods Cell Biol. 79, 769-798 (2007).
- Volkmann, N. Methods for Segmentation and Interpretation of Electron Tomographic Reconstructions. Methods Enzymol. 483, 31-46 (2010).
- Tsai, W. -T., et al. From Voxels to Knowledge: A Practical Guide to the Segmentation of Complex Electron Microscopy 3D-Data. J Vis Exp. (90), (2014).
- FEI. Amira & Avizo 3D: Software for Scientific and Industrial Data. , Available from: https://www.fei.com/software/amira-avizo/ (2016).
- Kremer, J. R., Mastronarde, D. N., McIntosh, J. R. Computer Visualization of Three-Dimensional Image Data Using IMOD. J Struct Biol. 116 (1), 71-76 (1996).
- Nguyen, H., Ji, Q. Shape-driven three-dimensional watersnake segmentation of biological membranes in electron tomography. IEEE trans med imaging. 27 (5), 616-628 (2008).
- Rusu, M., Starosolski, Z., Wahle, M., Rigort, A., Wriggers, W. Automated tracing of filaments in 3D electron tomography reconstructions using Sculptor and Situs. J Struct Biol. 178 (2), 121-128 (2012).
- Moussavi, F., Heitz, G., Amat, F., Comolli, L. R., Koller, D., Horowitz, M. 3D segmentation of cell boundaries from whole cell cryogenic electron tomography volumes. J Struct Biol. 170 (1), 134-145 (2010).
- Hecksel, C. W., et al. Quantifying Variability of Manual Annotation in Cryo-Electron Tomograms. Microsc Mpicroanal. 22 (3), 487-496 (2016).
- Achanta, R., Shaji, A., Smith, K., Lucchi, A., Fua, P., Süsstrunk, S. SLIC superpixels compared to state-of-the-art superpixel methods. IEEE trans pattern anal mach intell. 34 (11), 2274-2282 (2012).
- Luengo, I., Basham, M., French, A. P. Fast global interactive volume segmentation with regional supervoxel descriptors. Proc. SPIE. 9784, (2016).
- Luengo, I., et al. SuRVoS: Super-Region Volume Segmentation Workbench. J Struct Biol. 198 (1), 43-53 (2017).
- Wadeson, N., Basham, M. Savu: A Python-based, MPI Framework for Simultaneous Processing of Multiple, N-dimensional, Large Tomography Datasets. arXiv:1610.08015 [cs]. , Available from: http://arxiv.org/abs/1610.08015 (2016).
- DiamondLightSource/Savu: Version 1.2. Zenodo. , Available from: https://zenodo.org/record/198726 (2014).
- Paganin, D., Mayo, S. C., Gureyev, T. E., Miller, P. R., Wilkins, S. W. Simultaneous phase and amplitude extraction from a single defocused image of a homogeneous object. J Microsc. 206 (Pt 1), 33-40 (2002).
- van Aarle, W., et al. Fast and flexible X-ray tomography using the ASTRA toolbox. Opt Express. 24 (22), 25129-25147 (2016).
- Palenstijn, W. J., Batenburg, K. J., Sijbers, J. Performance improvements for iterative electron tomography reconstruction using graphics processing units (GPUs). J Struct Biol. 176 (2), 250-253 (2011).
- Mastronarde, D. N. Dual-Axis Tomography: An Approach with Alignment Methods That Preserve Resolution. J Struct Biol. 120 (3), 343-352 (1997).
- Duke, E., Dent, K., Razi, M., Collinson, L. M. Biological applications of cryo-soft X-ray tomography. J Microsc. 255 (2), 65-70 (2014).
- Arbeláez, P., Maire, M., Fowlkes, C., Malik, J. Contour detection and hierarchical image segmentation. IEEE trans pattern anal mach intell. 33 (5), 898-916 (2011).
- Luengo, I., Basham, M., French, A. P. Hierarchical Piecewise-Constant Super-regions. arXiv:1605.05937 [cs]. , Available from: http://arxiv.org/abs/1605.05937 (2016).
- Luengo, I., Basham, M., French, A. P. SMURFS: superpixels from multi-scale refinement of super-regions. , Available from: http://www.bmva.org/bmvc/2016/papers/paper004/index.html 1-12 (2016).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten