Method Article
Volume di segmentazione e analisi dei materiali biologici utilizzando SuRVoS (Super regione Volume segmentazione) Workbench
In questo articolo
Riepilogo
La segmentazione di dati tridimensionali da molte tecniche di imaging è un serio ostacolo nell'analisi dei sistemi biologici complessi. Qui, descriviamo l'uso di SuRVoS Workbench per semi-automaticamente i dati volumetrici del segmento a varie scale di lunghezza utilizzando i set di dati di esempio da cryo-elettrone tomografia, tomografia a raggi x molle cryo e tecniche di tomografia a raggi x di fase contrasto.
Abstract
Segmentazione è il processo di isolare determinate aree o oggetti all'interno di un volume imaged, in modo che ulteriori studi possono essere intrapresa su queste aree di interesse. Quando si considera l'analisi di sistemi biologici complessi, la segmentazione dei dati immagine tridimensionale è un passo che richiede tempo e lavoro intensivo. Con la maggiore disponibilità di molte modalità di imaging e con sistemi di raccolta automatica dei dati, ciò pone una sfida maggiore per il biologo sperimentale moderna spostare dai dati alla conoscenza. Questa pubblicazione descrive l'uso di SuRVoS Workbench, un programma progettato per affrontare questi problemi fornendo metodi a semi-automaticamente segmento biologico volumetrico dati complessi. Tre set di dati di diverse ingrandimento e modalità di formazione immagine sono presentati qui, ciascuno di questi differenti strategie di segmentazione con SuRVoS. Fase contrasto x-ray tomography (microCT) del corpo fruttifero di una pianta viene utilizzato per illustrare utilizzando il training del modello di segmentazione, cryo-tomografia elettronica (cryoET) delle piastrine umane viene utilizzata per illustrare la segmentazione utilizzando super - e megavoxels e cryo morbido Tomografia a raggi x (cryoSXT) di una linea di cellule di mammifero viene utilizzata per illustrare l'etichetta spaccare strumenti. Strategie e i parametri per ogni tipo di dati inoltre sono presentati. Miscelando una selezione dei processi semi-automatici in un unico strumento interattivo, SuRVoS offre diversi vantaggi. Tempo di dati volumetrici segmento complessivo è ridotto di un fattore di cinque rispetto alla segmentazione manuale, un pilastro in molti campi di elaborazione di immagine. Si tratta di un significativo risparmio quando segmentazione manuale completo può richiedere settimane di sforzo. Inoltre, soggettività viene risolto mediante l'utilizzo di limiti informaticamente identificati e scissione complessi insiemi di oggetti di loro proprietà calcolate, piuttosto che su una base del contenitore-da-contenitore.
Introduzione
Il Workbench di SuRVoS è un pezzo di software progettato per consentire ai ricercatori di estrarre informazioni scientificamente rilevanti da dati volumetrici da vari campioni, indipendentemente dalla struttura di interesse, ad alta risoluzione o la modalità imaging1, 2. Dati volumetrici come questi vengono spesso raccolti utilizzando sistemi di tomografia a raggi x o elettroni, ordinariamente in base a grandi laboratori o impianti centralizzati a causa della loro complessità. Entrambi questi metodi e altre tecniche, produrre grande, ricco set di dati informazioni che rivelarsi difficile per segmento con entrambi metodi semi-automatici o manuale. In particolare, quasi nativa stato cryo-immobilizzata DataSet richiedono condizioni imaging della basso-dose, risultante in un rapporto segnale-rumore basso e scarso contrasto, soprattutto in cryo electron tomografia (cryoET)3,4,5 . Un ulteriore fattore in alcuni set di dati 3D è la presenza di artefatti introdotti da difficili condizioni sperimentali coinvolte, ad esempio mancanti artefatti di Cuneo Grazie alla raccolta di dati su un limitato intervallo, informazioni mancanti con conseguente inclinazione e allungamento in direzione del fascio3,4,5. Anche quando basso segnale-rumore o artefatti Cuneo mancanti non sono problematici (ad es. concentrati fascio ionico SEM6 o seriale blocco faccia SEM7), la complessità e la natura tridimensionale del campione e la grande quantità di dati significa analisi sarebbe ancora beneficiare di un processo automatizzato per la segmentazione dei dati.
Attualmente, quando si considera volumi biologici delle cellule, ci sono molte opzioni per automaticamente o semiautomaticamente identificando caratteristiche cellulari molto specifiche, come l'actina, microtubuli o complessi proteici specifici, utilizzando una ricerca basata su modello, o identificazione delle caratteristiche in specifici tipi di set di dati (ad es. ad alto contrasto, macchiato, campioni incastonati resina)8,9,10,11,12. Tuttavia, in questi casi a priori informazioni o protocolli di preparazione del campione specifici sono necessari, limitando l'ampia applicabilità di queste strategie di segmentazione. Sono inoltre disponibili strumenti che eseguire il training del modello a livello di voxel per conoscere l'aspetto di varie strutture di interesse quando dato utente ingresso13. Tuttavia, a questo livello la complessità di addestramento e prove dei modelli può essere soggetta a errori e dispendiosa. Date le difficili condizioni di immagine e la mancanza di strategie di segmentazione ampiamente applicabili, semi-automatica, manuale segmentazione è comune, anche quando si lavora con materiali biologici complessi14,15, 16 , 17. Tuttavia, è generalmente accettato che il processo di segmentazione manuale non è solo che richiede tempo, ma anche errori, soggettiva e variabile4,5,18,19 ,20. Alcuni programmi di segmentazione offrono strumenti per facilitare il processo di segmentazione manuale (cioè interpolazione, lazo o strumenti di colpo)21,22, tuttavia, in caso di set di dati rumorosi, sono difficili da applicare con successo, e anche quando vengono utilizzati con successo, il processo è ancora soggettive e variabili.
Tradizionalmente, le segmentazioni sono stati utilizzati in due modi distinti: qualitativamente o quantitativamente. Come migliorano la segmentazione strategie e tecnologie di imaging, è diventato più comune utilizzare la segmentazione come uno strumento quantitativo per rispondere a domande biologiche e come una "verità a terra" per algoritmo sviluppo8,12, 15,23,24,25. A tal fine sono necessari per diminuire la variabilità e la soggettività in tutto il processo26saldi e controlli dettagliati. Tuttavia, queste precauzioni ulteriormente aumentano la natura richiede tempo di segmentazione. Per questo motivo, è fondamentale per fornire una strategia di segmentazione più veloce e meno variabile.
Il SuRVoS Workbench incomincia ad affrontare questi problemi fornendo all'utente con un'ampia scelta di apprendimento automatico e image processing strumenti che assistono l'utente nel processo di segmentazione, mentre anche guidando l'utente attraverso i passaggi necessari. Per raggiungere questo obiettivo, due innovazioni chiave vengono implementate insieme in SuRVoS. In primo luogo, utilizza una gerarchia di Super regione al gruppo simile, vicine regioni dei dati basato sulla loro proprietà intrinseche. Ciascuna delle regioni nella gerarchia rappresenta il volume stesso utilizzando meno elementi, garantendo comunque l'aderenza forte limite. Così, Super-regioni riducono la complessità di segmentazione di un volume di diversi ordini di grandezza ancora ancora rappresentano i dati senza perdita significativa di informazioni27. In secondo luogo, SuRVoS fornisce una strategia di segmentazione semi-automatizzato che utilizza ingressi di segmentazione manuale minimo addestrare classificatori, che vengono poi utilizzati per segmentare il restante volume28,29. Questa strategia riduce segmentazione manuale, diminuendo notevolmente la quantità di utente tempo trascorso sulla segmentazione e, quando si utilizza Super-regioni, rimuove manuale delineazione dei confini, potenzialmente riducendo la variabilità e la soggettività.
Un ulteriore caratteristica fondamentale del SuRVoS è lo strumento etichetta Splitter, per cui un utente può classificare una serie di oggetti già segmentati in base alla loro proprietà intrinseche. Dopo la segmentazione di vari oggetti di interesse, questo strumento può essere utilizzato per dividere il set in sottoclassi basate su misure come oggetto di media intensità, varianza, dimensione, posizione, ecc questo è utile quando classificare grandi gruppi di oggetti con elevata complessità. Ad esempio, un gruppo di organelli cellulari può essere suddiviso in mitocondri, vescicole vuote, le goccioline del lipido, ecc.; o un set di materiale inclusioni possono essere separate basato su dimensione o forma. Una volta segmentato le singole etichette può essere diviso in gruppi utilizzando un numero qualsiasi di classificatori, riducendo la polarizzazione di identificazione.
Il Workbench di SuRVoS è stato utilizzato con successo per segmento di dati da diverse tecniche di imaging. Qui, tomografia sincrotrone di contrasto di fase dei raggi x (microCT) del corpo fruttifero di una pianta viene utilizzata per illustrare segmentatisull'utilizzo di training del modello, cryo-tomografia elettronica (cryoET) delle piastrine umane viene utilizzata per illustrare la segmentazione utilizzando super - e megavoxels, e tomografia a raggi x molle cryo (cryoSXT) di una linea di cellule di mammifero viene utilizzata per illustrare l'etichetta Divisione strumenti
Protocollo
Nota: generalmente utile gamme di parametri per ogni fase di lavorazione e i parametri specifici per ogni tipo di dati mostrati qui sono fornite nella tabella 1.
1. preparare un Workspace e caricamento di dati
- lanciare SuRVoS Workbench, fare clic sul pulsante open dataset e nel popup risultante, selezionare il file di dati per essere segmentato. Scegliere un orientamento appropriato del dataset. Successivamente, scegliere o creare una cartella in cui verranno memorizzati i file associati e area di lavoro. È consigliabile che questa cartella è vuota quando si avvia una nuova segmentazione. Dopo i dati sono stati caricati, il banco di lavoro si aprirà con il plugin riquadro a sinistra, il riquadro di visualizzazione a destra e un set di scelte rapide di strumento tra i due riquadri ( Figura 1).
2. Pre-elaborazione e rappresentazione dei dati
- In ROI selezionare scheda, input all'inizio x, y e z e fine coordinate per la regione di interesse e fare clic su Aggiungi. Per scegliere l'appropriato y e x coordinate passa il mouse sopra un punto dell'immagine. Scegliere le coordinate z utilizzando il dispositivo di scorrimento nella parte superiore del riquadro di visualizzazione. Una volta che una regione è stato aggiunto, assicurarsi che è selezionata la casella a destra. Tutti i calcoli a valle si articolerà sull'area selezionata. Generalmente, a partire da un piccolo, rappresentante regione di interesse (ROI), ottimizzando i parametri e poi ri-applicazione di questi parametri per l'intera area segmentarli consiglia.
- Scheda in the presentano canali, utilizzare il menu a discesa nella parte superiore per scegliere un caratteristica/filtro e aggiungerlo alla coda (vedi discussione per maggiori informazioni sui canali di funzionalità). Una volta che un filtro/funzione è stato aggiunto e selezionato facendo clic sul suo nome, modificare opzioni specifiche per il filtro/feature e scegliere il set di dati di input su cui eseguire il filtro/feature. Una volta scelte tutte le opzioni, fare clic sulla casella a destra del nome del filtro e funzione per calcolare.
- Al fine di ottimizzare i parametri per un nuovo dataset, aggiungere filtri/caratteristiche multiple e scegliere i parametri per loro, prima di essere calcolata nell'ordine, uno dopo l'altro. A tale scopo, aggiungere ogni nuovo filtro/funzionalità e selezionare i parametri appropriati, selezionare la casella a sinistra di ogni funzionalità di filtro da eseguire e fare clic sulla casella di funzioni di calcolo nella parte superiore del riquadro. Per ulteriori informazioni, vedere discussione.
3. Generazione di Super-regioni Appropriate
- In the Super regioni scheda, nella sezione Supervoxels, utilizzare il menu a discesa origine per selezionare dataset filtrato da cui verrà creato supervoxels. Quindi specificare la forma, la spaziatura e la compattezza della supervoxels (Vedi la discussione e la Figura 2 per maggiori dettagli). Infine, fare clic sul pulsante Applica per generare il supervoxels. Dopo avere creato il supervoxels che possano essere visualizzati nel riquadro di visualizzazione, acceso o spento e la loro trasparenza controllato nella scheda visualizzazione e scelta rapida della finestra del visualizzatore.
- Scheda di in the Super regioni, nella sezione Megavoxels, utilizzare il menu a discesa origine per selezionare dataset filtrato da cui verrà creato megavoxels. Successivamente, specificare la lambda, numBins e parametri di Gamma di megavoxels (per ulteriori dettagli, vedere la discussione). Dopo avere creato il megavoxels che possano essere visualizzati nel riquadro di visualizzazione, acceso o spento e la loro trasparenza controllato nella scheda visualizzazione e scelta rapida della finestra del visualizzatore.
4. Introduzione all'annotazione
- In the annotazioni scheda utilizzare il pulsante Aggiungi livello per aggiungere un livello di annotazione. Dopo che è stato aggiunto un livello, è possibile utilizzare il pulsante Aggiungi etichetta in quel livello per aggiungere un'etichetta per l'annotazione. Una volta aggiunto, il nome e il colore dell'etichetta può essere modificati per la facilità di annotazione.
- Successivamente, al fine di iniziare l'annotazione, selezionare l'icona della penna dalla sezione di scelta rapida di strumento. Quando questa opzione è selezionata una serie di opzioni vengono visualizzate nella parte superiore del riquadro di visualizzazione. Queste opzioni controllano la larghezza della penna e se voxel, supervoxels o megavoxels verrà utilizzato per annotare.
- Ai fini della formazione del modello, in genere, selezionare supervoxels nella casella di elenco a discesa livello di annotazione e un'ampiezza media larghezza di penna di grandi dimensioni deve essere utilizzato. Nella scheda annotazioni, selezionare l'etichetta deve essere annotata selezionando la casella all'estrema destra delle informazioni etichetta. Fare clic nel riquadro di visualizzazione per annotare un singolo supervoxel, o fare clic e trascinare per annotare molti.
Nota: Voxel e megavoxels può essere selezionato nella casella a discesa livello di annotazione e utilizzato per annotare nello stesso modo, che, nel caso di megavoxels, può abilitare molte migliaia di simili voxel per essere segmentato con un solo clic del mouse.
- Ai fini della formazione del modello, in genere, selezionare supervoxels nella casella di elenco a discesa livello di annotazione e un'ampiezza media larghezza di penna di grandi dimensioni deve essere utilizzato. Nella scheda annotazioni, selezionare l'etichetta deve essere annotata selezionando la casella all'estrema destra delle informazioni etichetta. Fare clic nel riquadro di visualizzazione per annotare un singolo supervoxel, o fare clic e trascinare per annotare molti.
5. Training del modello utilizzando segmentazione ha dimostrato con un dataset microCT.
Nota: la segmentazione prima per molti DataSet è di differenziare più grandi regioni da altro. Ad esempio, che separa il nucleo dal citoplasma, o la cella dalla struttura esterna di ghiaccio e supporto. Per questo tipo di segmentazione, con chiari confini delineati e grandi regioni, training del modello è utile. Per dimostrare questo, verranno utilizzati dati tomografici con contrasto di fase dei raggi x di Goosegrass.
- Caricare i dati, utilizzando la suite di filtro e caratteristica della pre-elaborazione e determinare supervoxels appropriato e/o megavoxels come descritto nelle sezioni precedenti utilizzando i parametri nella tabella 1 come guida. Continuando a utilizzare i parametri da tabella 1 e le istruzioni nella sezione 4, approssimativamente annotare alcune grandi aree del dataset, come mostrato nella Figura 3.
Nota: Il set di dati non ha bisogno di essere completamente segmentati a questo punto. - In the Training del modello di scheda, impostare il livello di predire il livello che contiene le annotazioni di formazione manuale e nel descrittore di sezione impostata la regione di Supervoxels. Successivamente, selezionare i descrittori che devono essere utilizzati per differenziare le regioni dei dati cliccando sull'elenco a discesa selezionare fonti e controllando le caselle delle funzioni e filtri di scelta (Vedi tabella 1 e la discussione).
- Fare clic sul pulsante di predict. Al termine del calcolo, il riquadro di visualizzazione verrà aggiornato con le previsioni per tutti i voxel non etichettata mostrando che le classi di annotazione che sono previsti di appartenere a. In genere, i parametri predefiniti per ogni metodologia di classificazione forniscono un buon punto di partenza e l'utente deve solo bisogno di passare tra classificatori per trovare un buon adattamento. Tuttavia, per gli utenti esperti o con esperienza le opzioni per ogni classificatore sono disponibili e possono essere modificate.
- Dopo la valutazione dell'effetto delle metodologie di formazione e scegliendo uno, applicare ulteriore affinamento cliccando su Refine a discesa nella sezione raffinatezza. Alla parte inferiore della formazione modello disponibile nella scheda il " aggiornamento annotazioni " sezione, assicurarsi che il menu a discesa di visualizzazione è impostato su PredictioNS. Utilizzare il dispositivo di scorrimento di fiducia per assegnare più o meno del supervoxels annotate le etichette annotazione selezionata.
- Dopo un adeguato livello di fiducia è stato selezionato basato su controllo visivo, utilizzare i pulsanti di Salva accanto alle etichette di nella parte inferiore dello strumento fiducia per salvare le previsioni in etichette specifiche. Il riquadro di visualizzazione verrà aggiornata per mostrare le modifiche. Ogni etichetta può essere salvato separatamente, e infatti, le etichette possono essere salvate da piccole sub-regioni immettendo valori in da e per caselle x, y e z e facendo clic sul pulsante Salva per ogni etichetta.
- Indirizzo minori erronea fornendo ulteriori dati di training, come descritto nella sezione 4. Dopo previsioni adeguate vengono aggiunti alle etichette, ripetere il processo del modello di formazione con raffinatezza e aggiungendo le previsioni alta fiducia fino a quando non ci sono più senza etichetta supervoxels. Questo è efficace, perché ogni volta che viene eseguito il processo di formazione di modello ci sono assegnate più supervoxels ad allenarsi con, e quindi il processo diventa più robusto come aumentano le iterazioni.
6. Segmentazione utilizzando Super-regioni, ha dimostrato con un Dataset di CryoET.
Nota: poiché Super regione segmentazione è utile per zone più piccole, discretamente rilegate, qui l'attenzione sarà sulla segmentazione del organelli e microtubuli all'interno di questo set di dati. Training del modello è stato usato per segmentare rapidamente la piastrina dal fondo ghiaccio e carbonio; questi parametri non sono oggetto di ulteriori discussioni, ma sono presentati nella tabella 1.
- Caricare i dati, utilizzando la suite di filtro e caratteristica della pre-elaborazione e determinare supervoxels appropriato e/o megavoxels come descritto nelle sezioni precedenti utilizzando i parametri nella tabella 1 come guida.
- Aggiungi livelli appropriati ed etichette nella scheda di annotazione, selezionare un'etichetta e iniziare annotazione utilizzando un'ampiezza media larghezza della penna con la supervoxels selezionata. Essere consapevoli della necessità di scegliere etichette diverse per oggetti nella prossimità vicina a vicenda al fine di evitare di etichettarli come oggetto singolo.
- Al fine di ripulire le annotazioni ulteriormente, utilizzare metodi morfologici raffinatezza (dilatazione, erosione, apertura, chiusura e riempimento fori). Queste opzioni possono essere trovate nella parte inferiore della scheda annotazioni. Per utilizzarli, è necessario selezionare il metodo di etichetta e la raffinatezza di segmentazione. Immettere un valore di raggio e scegliere come applicare la raffinatezza. Quindi fare clic su Ottimizza.
7. Classificazione e analisi di dati oggetti basati su caratteristiche intrinseche, ha dimostrato con un Dataset di CryoSXT
Nota: generalmente, il passo successivo dopo la segmentazione è l'analisi dei dati. Lo strumento di divisione di etichetta in SuRVoS consente la classificazione di oggetti segmentati utilizzando regole basate su caratteristiche intrinseche degli oggetti come intensità media degli oggetti, varianza, il volume o posizione. Lo strumento di statistiche di etichetta consente la visualizzazione di relazioni tra queste misure per ogni nuova classe di oggetti. Questi sono nuovi potenti strumenti per l'analisi di set di dati 3D complessi dopo segmentazione.
- Caricare i dati, utilizzando la suite di filtro e caratteristica della pre-elaborazione, determinare supervoxels appropriato e/o megavoxels e segmento come descritto nelle sezioni precedenti utilizzando i parametri nella tabella 1 come guida.
- Dopo la segmentazione, clicca sulla seconda scheda del riquadro di visualizzazione, chiamata etichetta Splitter. Verrà aggiunta una nuova area sul lato destro della finestra - riquadro creazione regola.
- Nella parte superiore di questa zona, selezionare un livello adeguato ed etichette per la divisione di etichetta. Quindi selezionare il set di dati per eseguire query e fare clic su etichetta. Ora tutti gli oggetti nelle etichette selezionate saranno delineati in blu come oggetti separati nel riquadro di visualizzazione e un grafico che mostra l'intensità media degli oggetti verranno visualizzati nel riquadro di creazione di regole. Per modificare la misura viene mostrata nella trama, fare clic sulla casella a discesa nella parte superiore del lato destro.
- Di cominciare a dividere gli oggetti in classi rilevanti, fare clic su Aggiungi nuova etichetta nella parte inferiore del riquadro di creazione di regole. Il nome e il colore associato con questa nuova etichetta può essere modificati come descritto in precedenza.
- Fare clic su Aggiungi nuova regola e utilizzando le caselle a discesa e forma libera voce definire la regola da applicare. Fare clic su Applica per vedere gli effetti della nuova regola nel riquadro di visualizzazione e la trama nel riquadro creazione di regole. Più regole possono essere applicate a una singola etichetta e più etichette possono essere create all'interno del dataset stesso.
Nota: Al fine di raccogliere tutti gli oggetti senza etichetta, creare una nuova etichetta e invece di aggiungere una regola ad esso, fare clic su selezionare altri.
- Fare clic su Aggiungi nuova regola e utilizzando le caselle a discesa e forma libera voce definire la regola da applicare. Fare clic su Applica per vedere gli effetti della nuova regola nel riquadro di visualizzazione e la trama nel riquadro creazione di regole. Più regole possono essere applicate a una singola etichetta e più etichette possono essere create all'interno del dataset stesso.
- Quando gli oggetti di interesse sono stati suddivisi in nuove etichette, è possibile creare un nuovo livello vuoto nella scheda annotazioni. Scegliere questo livello nella scheda Creazione regola, quindi fare clic su Salva etichette. Questo salverà le nuove etichette a questo livello vuoto.
- Sul bordo del riquadro di visualizzazione, fare clic sulla scheda etichetta statistiche. Verrà aperto un nuovo riquadro di visualizzazione che può essere utilizzato per cominciare a capire le relazioni tra classi di oggetti in dati. Nella parte superiore, selezionare un livello adeguato ed etichette e il set di dati per eseguire una query.
- Selezionare un paio delle misure di interesse selezionando le caselle accanto a loro. Fare clic su etichetta. Questo produrrà pairwise confronto trame per ciascuna delle misure selezionate. Per aggiornare i grafici, selezionare o deselezionare misure appropriate quindi fare clic su aggiornamento trama.
8. Esportazione di dati e segmentazioni
- esportare le trame e i dati di misurazione crudo cliccando su trama di esportazione e di esportazione dati, rispettivamente, nella scheda etichetta statistiche del riquadro di visualizzazione.
- Esportare i dati di immagine e segmentazioni cliccando sulla scheda Esporta nel riquadro plugin. In primo luogo, fare clic per selezionare una cartella dove verranno salvati i dati. Avanti, selezionare l'output (dati Raw, Raw annotazioni, maschere di segmentazione o dati mascherati) e il formato (HDF5, MRC o TIFF). Infine, selezionate i livelli di annotazione per essere esportati utilizzando le caselle di controllo e fare clic su Esporta. I dati possono essere ridimensionati e invertiti prima di esportare come necessario. Quando si esportano i dati mascherati, il set di dati che la mask(s) verrà applicato a può essere selezionato tramite un menu a discesa.
Risultati
Tre set di volume dati raccolti da tre diverse tecniche (microCT, cryoET e cryoSXT) sono stati utilizzati per dimostrare tre importanti caratteristiche del banco da lavoro SuRVoS: modello di formazione, Super-regione segmentazione ed etichetta spaccare. I set di dati rappresentano un gruppo eterogeneo di risultati sperimentali, per ognuno dei quali parametri di elaborazione completo sono forniti (tabella 1).
Per dimostrare il training del modello utilizzando SuRVoS Workbench, è stato scelto un set di dati relativamente ad alto contrasto con la definizione dei limiti di regione. Questo set di dati del frutto di Galium aparine, o goosegrass, sono stati raccolti usando la tomografia a raggi x fase contrasto sulla I13-2 Diamond-Manchester Imaging Beamline presso Diamond Light Source, Chilton, Oxfordshire, Regno Unito. Il campione fresco è stato montato in aria su un goniometro base sopra la fase di rotazione, ad una distanza di campione-rivelatore di 30 mm. esposizione volte erano 0.10 s con lo spettro di raggio rosa che ha un'energia media di circa 22 keV. Le proiezioni sono stati raccolti attraverso 180° con un passo di 0,1 °. Tomografiche ricostruzioni sono state eseguite utilizzando Savu30,31 con il filtro di Paganin per contrasto di fase di propagazione-basato immagini32 seguita da ricostruzione di proiezione posteriore filtrata in ASTRA toolkit33 , 34. questi dati e ' stato poi ridimensionati con 2 x 2 x 2 binning per ridurre la dimensione del file prima in ingresso in SuRVoS Workbench.
In primo luogo, i dati di input (Figura 3A) è stati filtrati e bloccati (per rimuovere i valori di intensità superiore ed inferiore nei dati) (Figura 3B). In questo modo, lo sfondo e primo piano sono state fatte più facilmente distinguibili e la consistenza della struttura interna del frutto è stata accentuata. Successivamente, supervoxels sono stati costruiti sulla cima di dataset filtrato (Figura 3). Per valutare la qualità della supervoxels, sono stati visualizzati senza i dati per verificare che le informazioni pertinenti del dataset erano ben rappresentate da supervoxels (Figura 3D). Successiva, manuale annotazioni utilizzando supervoxels sono state fornite come dati di training su tre fette del volume (Figura 3E, colori scuri). Questi dati di formazione erano sufficienti a formare il classificatore per predire (colori chiari) aree corrispondenti al fondo (verde), setola di frutta (rosso), materiale di seme (viola) e che circonda la carne (blu). Perfezionamenti morfologici sono stati usati per ripulire le segmentazioni di fori di riempimento, aumento o la riduzione come necessari (Figura 3F). Il tempo totale trascorso per determinare i parametri appropriati e segmentare questo dataset era 2 h.
Per dimostrare la segmentazione Super regione utilizzando SuRVoS Workbench, un set di dati complessi e rumoroso è stato scelto15. Questo dataset è stato raccolto utilizzando cryoET presso il centro nazionale per l'Imaging macromolecolare presso il Baylor College of Medicine, Houston, TX, Stati Uniti d'America. Brevemente, le piastrine erano tuffo congelato il bagliore scaricata e griglie TEM oro carbonio holey fiducial-trattati. Serie di inclinazione sono stati raccolti da ± 65 ° con un incremento di 2°. La serie di inclinazione allora è stata ricostruita utilizzando la proiezione posteriore ponderata in IMOD35.
Dopo il caricamento dei dati in SuRVoS (Figura 4A), è stata selezionata un'area di interesse e un set di filtro appropriato è stato applicato. In questo caso, un filtro gaussiano seguito da un filtro di variazione totale con il contrasto bloccato è stato utilizzato per accentuare i bordi e le texture dei dati (Figura 4B). Successivamente, training del modello con input dell'utente minima basata su supervoxel è stato utilizzato per segmentare la piastrina dal fondo ghiaccio e carbonio. Quindi, segmentazione semi-manuale con megavoxels e supervoxels è stata utilizzata per segmentare gli organelli. Infine, il parametro di origine supervoxel è stato cambiato in un più debole filtro denoising e supervoxel forma è stata fatta più piccolo (Vedi tabella 1) al fine di preservare meglio i confini di microtubuli per segmentazione (Figura 4). Per entrambi gli organelli e i microtubuli, sono state utilizzate annotazioni manuale rapide ogni 5-10 fette di selezionare il supervoxels che descrivono la funzionalità di interesse (Figura 4 & 4E). Il tempo totale trascorso per determinare i parametri appropriati e segmentare la regione di interesse presentato era di 6 h.
Per dimostrare l'etichetta spaccare utilizzando SuRVoS Workbench, è stato scelto un set di dati con molti, diversi organelli. Questo set di dati raccolti utilizzando cryoSXT su beamline B24 a Diamond Light Source, Chilton, Oxfordshire, UK,36. Brevemente, cellule HEK293 sono state coltivate sulle griglie di cercatore d'oro, dimensioni appropriate fiducials oro sono stati aggiunti e la griglia è stato congelato utilizzando un EM con retro-retro macchiare con trampolino. Serie di inclinazione sono stati poi raccolti su un microscopio da ± 65 ° con un incremento di 0,5 °. La serie di inclinazione allora è stata ricostruita utilizzando la proiezione posteriore ponderata in IMOD35.
Dopo il caricamento dei dati in SuRVoS (Figura 5A), è stata selezionata un'area di interesse e un filtro appropriato variazione totale è stato utilizzato per migliorare i confini degli organelli in tutto il volume (figura 5B). Successivamente, organelli erano semi-manually segmentato utilizzando megavoxels e supervoxels e poi raffinato con fori di riempimento, chiusura e dilatazione per smussare gli angoli (Figura 5). Il tempo totale per determinare i parametri appropriati e segmentare la regione di interesse presentato era di 4 h. Una volta che la segmentazione è stata finalizzata, lo Splitter di etichetta è stato utilizzato per visualizzare ogni organello come oggetto nel dataset (Figura 5) e varie caratteristiche su ogni oggetto nella trama dei dati (Figura 5E). L'interfaccia di etichetta Splitter è interattiva, aggiornando il colore associato con ogni nuova classe di etichetta sia la visualizzazione che la trama di dati. Questo permette la creazione di varie regole in base alle caratteristiche intrinseche nei dati che possono essere utilizzati per separare gli oggetti in classi utili (Figura 5F).

Figura 1. Il Layout e le caratteristiche generali del banco da lavoro SuRVoS.
L'interfaccia GUI è sulla sinistra, mentre il riquadro di visualizzazione è sulla destra. Queste due zone sono separate da una colonna di strumenti e scorciatoie. La GUI è predisposta per guidare l'utente attraverso le fasi principali nella pre-elaborazione dei dati, scelta dei parametri di supervoxel e/o megavoxel, segmentare i dati e, se necessario utilizzando il training del modello, prima di esportare le segmentazioni. Il riquadro di visualizzazione può essere utilizzato in tre modalità: Visualizzazione base e segmentazione per visualizzare i datie qualsiasi filtri applicati e segmentare i dati, etichetta splitter per classificare gli oggetti in nuove etichette basate su aspetti inerenti ai dati e infine etichetta statistiche per misurare e visualizzare le caratteristiche degli oggetti segmentati. Per ognuna di queste modalità, il menu a discesa in alto a sinistra quali dati vengono visualizzati i controlli di angolo, e il dispositivo di scorrimento nella parte superiore controlla l'asse z. Le scorciatoie di strumento permette facile accesso controllo contrasto, trasparenza dei livelli, lo zoom, panning e tornare a "casa" nel riquadro di visualizzazione e strumenti di apertura per l'annotazione come descritto nel protocollo. Clicca qui per visualizzare una versione più grande di questa figura.

Nella figura 2. Super-regione gerarchia riduce la complessità di segmentazione di immagini.
Un'immagine del set di dati per la segmentazione della Berkeley (BSDS50037) è stata utilizzata per dimostrare le proprietà e gli effetti delle Super-regioni. L'immagine originale (a sinistra) è composto da migliaia di voxel, che sono poi riuniti in raggruppamenti adiacente, simili per creare qualche centinaio supervoxels (centro). Supervoxels possono essere raccolte anche in raggruppamenti adiacente, simili per creare alcune decine di megavoxels (a destra). Con ogni raggruppamento, la complessità del compito di segmentazione è diminuita, per risorse computazionali e di manuale. Cosa importante, ecco un esempio 2D, tuttavia, sia supervoxels che megavoxels sono 3D. Clicca qui per visualizzare una versione più grande di questa figura.

Nella figura 3. Elaborazione di un oggetto Dataset utilizzando una strategia di segmentazione di formazione modello microCT.
R. una sola fetta 2D dei dati grezzi. B. applicazione di un filtro bloccato variazione totale ai dati grezzi migliorato i confini tra i vari aspetti del corpo fruttifero. C. sono stati scelti i parametri appropriati supervoxel. D. una regione di interesse (scatola rossa in C) è indicata per dimostrare che i confini dei dati sono presenti nel supervoxels essi stessi. E. tre fette del volume con le annotazioni manuali delle varie aree del set di dati visualizzati in colori scuri (verde, rossi, blu e viola) e previsioni dopo aver eseguito il training del modello visualizzati negli stessi colori luce. F. la stesse tre fette con la segmentazione finale dopo le previsioni di formazione del modello sono state accettate. Barre della scala sono 1 mm. per favore clicca qui per visualizzare una versione più grande di questa figura.
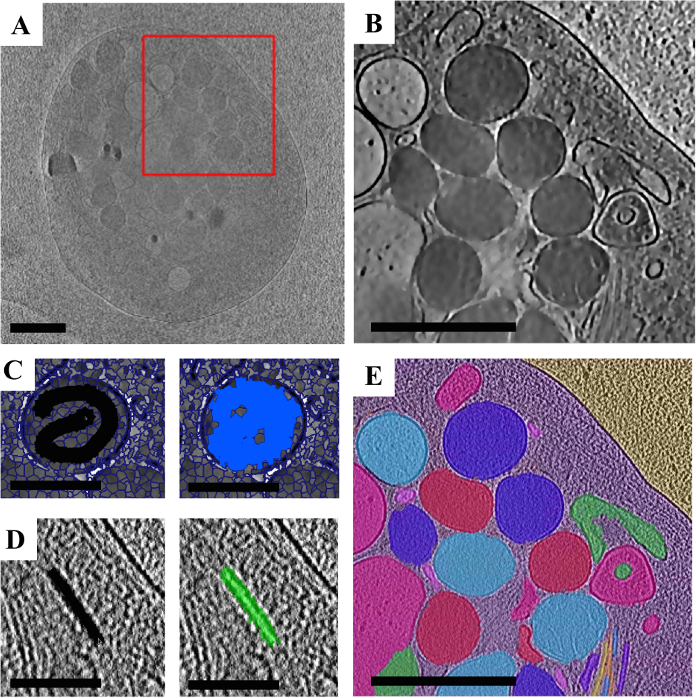
Nella figura 4. Elaborazione di un oggetto Dataset utilizzando una strategia di segmentazione della Super-regione di cryoET.
R. una sola fetta 2D dei dati grezzi. B. una regione di interesse (riquadro rosso in A) con un insieme di strati filtro applicato per accentuare i confini degli organelli. C. esempio di annotazione di un organello utilizzando Super-regioni. Un singolo organello è mostrato con supervoxels sovrapposto con l'annotazione manuale utente visualizzato in nero (a sinistra) e il supervoxels scelto con tale annotazione in blu (a destra). D. esempio di annotazione di un microtubulo utilizzando Super-regioni. Una singola regione di microtubuli è mostrata con l'annotazione manuale utente visualizzato in nero (a sinistra) e il supervoxels scelto con tale annotazione in verde (a destra). E. la segmentazione finale con la piastrina segmentato da sfondo utilizzando il modello di formazione (Vedi tabella 1 per i dettagli) e vari organelli e microtubuli segmentati utilizzando una strategia di segmentazione della Super-regione. Colori non indicano tipi specifici organelli che sono mostrati qui prima classificazione. Scala bar in A, B ed E sono 1 μm e in C e D sono 0,5 μm. Clicca qui per visualizzare una versione più grande di questa figura.

Nella figura 5. Analisi di un Dataset utilizzando lo strumento di Splitter etichetta cryoSXT.
R. una sola fetta 2D dei dati grezzi. B. una regione di interesse (scatola rossa nella A) con un filtro di variazione totale applicato per accentuare gli organelli. C. la segmentazione finale con supervoxels sovrapposti. D. La parte di visualizzazione della barra di divisione etichetta con gli organelli classificati utilizzando le regole visualizzate in F. E. la parte di trama dello splitter etichetta visualizzare l'intensità media all'interno di ogni oggetto, con le regole visualizzate in F applicata. Ogni linea verticale lungo l'asse x rappresenta un unico oggetto ed è con codifica a colori per abbinare la classe a che è stato assegnato. F. esempi di regole di classificazione per separare i vari oggetti in base alle loro proprietà intrinseche. Barre della scala sono 1 μm. Clicca qui per visualizzare una versione più grande di questa figura.
| Nome / Dataset | Fonte | P1 | P2 | P3 | P4 |
| Filtro gaussiano | Sigma | ||||
| Gamma / predefinito | - | [0,5, 10] / 1 | |||
| CryoET (G1) | Dati grezzi | 1 | |||
| CryoET (G2) | Dati grezzi | 2 | |||
| Variazione totale | Lambda | Spaziatura | # Iter | Morsetto | |
| Gamma / predefinito | - | [0.1, 30] / 10 | [0.1, 10] / 1 | [50, 500] / 100 | - |
| MicroCT (TV1) | Dati grezzi | 10 | 1 | 100 | (1,-) |
| CryoET (TV2) | G1 | 7 | 1 | 200 | - |
| CryoET (TV3) | G2 | 10 | 1 | 100 | - |
| CryoSXR (TV4) | Dati grezzi | 7 | 1 | 100 | - |
| Sogliatura | Vmin | Vmax | |||
| Gamma / predefinito |
Tabella 1. Parametri ottimizzati utilizzato per elaborare tutte le tre serie di dati (microCT, cryoET e cryoSXT).>
Per ogni parametro predefinito e una gamma di uso generale vengono fornite. In molti casi, i dati filtrati viene utilizzati come fonte per l'elaborazione a valle. In questi casi, l'abbreviazione è usata per denotare il nuovo dataset di origine. Ad esempio, G1 (i dati grezzi cryoET filtrata gaussiana) utilizzati come input durante un filtro di variazione totale per creare TV2. Informazioni viene presentate solo per aspetti del banco di lavoro che sono stati utilizzati per ogni set di dati di processo. Ad esempio, Training del modello non è stato utilizzato durante l'elaborazione di dataset cryoSXT presentato qui, quindi parametri non sono dato per questo.
Discussione
SuRVoS Workbench si differenzia da altri programmi di segmentazione in ottimizzazione dei parametri è un passo necessario e importante prima di iniziare la segmentazione effettiva. In alcuni programmi di segmentazione manuale o semi-manuale, l'utente inizia la segmentazione in pochi istanti di apertura di un nuovo progetto. Con SuRVoS, perché grandi quantità del volume vengono segmentati con l'input dell'utente molto poco e i confini sono delineati dal programma, l'ottimizzazione dei parametri è fondamentale per una segmentazione di successo. In particolare, dispongono di canali e costruzione Super-regione sono due aree dove occorre prestare attenzione.
Dispongono di canali e training del modello
Oltre ai dati grezzi, SuRVoS permette all'utente di creare set di dati aggiuntivi o canali derivati da un dataset esistente. Questi canali possono essere creati utilizzando una selezione di metodologie computazionali o estrattori di funzionalità. Ognuna delle rappresentazioni dei dati sono disponibile in parallelo e possono essere visualizzati singolarmente per valutare i risultati dell'applicazione caratteristica o filtro. A causa di queste caratteristiche, si riferiscono a come presentano canali in SuRVoS. Ci sono molte opzioni di canale di funzionalità fornite all'interno di SuRVoS. Per informazioni sulle opzioni e parametri utilizzati qui, Vedi tabella 1, per un elenco completo e una descrizione dei canali disponibili funzionalità visitare https://diamondlightsource.github.io/SuRVoS/ 2. Primo, rumoroso DataSet potranno beneficiare di denoising con il totale o gaussiana filtro di variazione. È consigliabile che ulteriori funzionalità canale e supervoxel/megavoxel di calcoli eseguiti utilizzando uno di questi set di dati denoised come origine dati. Generalmente, il dataset di variazione totale denoised viene utilizzato come origine dati per i calcoli di canale e supervoxel/megavoxel di funzionalità. Si consiglia di eseguire con i valori predefiniti in primo luogo, valutare il risultato in 3D e infine iterativamente ottimizzare i parametri per l'oggetto dataset. Inoltre, funzionalità di canali possono essere costruite in "set di filtri" per specificamente isolare aspetti del set di dati e questi possono quindi essere utilizzati come origini dati per creare supervoxels e megavoxels. Sebbene questa strategia sia altamente dataset dipendente, può essere utile.
Dispongono di canali è anche utilizzati come fonti di addestrare classificatore nel training del modello. Quando si decide su quali canali di funzionalità da utilizzare, è consigliabile che alcuni canali di robusta funzionalità (ad esempio, dal rilevamento di blob, trama e struttura o categorie di caratteristiche di robustezza) vengono utilizzati quando si lavora con una piccola quantità di annotazioni per addestrare il classificatore. Quando si lavora con una grande quantità di dati di training, si raccomanda di utilizzare più canali di funzionalità complessive, da una qualsiasi delle categorie purché essi forniscono varie informazioni al classificatore (ad esempio, aggiungere ai canali funzione elenco sopra dal locale caratteristiche e categorie di caratteristiche gaussiana).
Ci sono tre parti principali per modellare formazione: fornendo fonti di dati di input che descrivono i dati, utilizzando questi ingressi addestrare un classificatore e raffinazione infine le previsioni di uscita. Generalmente, le regioni più piccole dei dati richiederà più le annotazioni dell'utente addestrare accuratamente il classificatore, mentre più grandi regioni dei dati richiederà meno le annotazioni dell'utente. Training del modello prima senza selezionare una raffinatezza può essere utilizzato per trovare i migliori pronostici. Quindi includere la raffinatezza e ottimizzare il parametro lambda come necessario per risolvere problemi con le previsioni quali fori o bordi frastagliati.
Supervoxels e megavoxels
Supervoxels sono raggruppamenti di più interesse, simili voxel38,39. Supervoxels iniziare come una griglia 3D standard sovrapposta sui dati che poi sono deformati in modo iterativo allo scopo di rispettare i confini sottostanti e così meglio rappresentare i dati. Creazione di Supervoxel e deformazione è controllato da quattro ingressi utente: origine dei dati, superpixel forma, spaziatura e compattezza. L'origine dati fornisce gli input di dati che vengono interrogati durante la creazione del supervoxel. Può essere utilizzata qualsiasi fonte comprese le fonti di dati filtrati. I parametri di forma superpixel determinano la griglia di partenza 3D e la forma desiderata approssimativa del supervoxels risultante. Cambiare questi parametri può aumentare o diminuire la dimensione del supervoxels prima deformazione. I parametri di spaziatura definiscono l'importanza dei confini in ogni direzione. Cambiare questi parametri può enfatizzare i confini in una o due direzioni a spese di altro (s), che significa il supervoxels risultante si deformano per seguire meglio i limiti dati nella direzione specificata. L'ultimo parametro, compattezza, controlla quanto il supervoxels può deformare. Un numero basso compattezza permette la supervoxels deformare più. Questi parametri dovrebbero essere ottimizzati per fornire supervoxels che rappresentano i limiti dei dati di interesse. Nota: Attualmente, i parametri di forma supervoxel sono pari a 1024 o meno quando moltiplicati tra loro.
In qualche modo, supervoxel parametri possono compensare per l'altro, significato non c'è nessuno "risposta giusta" al momento di decidere sui parametri. Ad esempio, una grande griglia di partenza (ad es. forma di superpixel: 10 x 10 x 10) e un numero basso di compattezza (es. 20) può dare supervoxels con simili contorno aderenza rispetto ad una piccola griglia di partenza (ad es. forma di superpixel 5 x 5 x 5) e un più alto numero di compattezza (ad es. 50). Perché ci sono più, più piccolo supervoxels nel secondo scenario, non hanno deformare tanto per rappresentare i confini. Entrambi gli insiemi di parametri potrebbero essere appropriati per la segmentazione del dataset.
La più grande considerazione quando si sceglie supervoxel parametri è quanto bene il supervoxels rappresentano i dati. Il supervoxels da solo, senza dati sotto di loro, come in Figura 2D, la visualizzazione è un buon modo per valutare i parametri supervoxel. Quando visualizzato in questo modo, i bordi e contorni di forme trovate nei dati devono ancora essere visibile nella supervoxels.
Megavoxels sono conglomerati dei più vicini, simile supervoxels38,39. Essi sono ancora controllati da quattro ingressi utente: origine dei dati, lambda, numbins e gamma. Come con supervoxels, l'origine dati fornisce gli input di dati che vengono interrogati durante la creazione del megavoxel. Lambda e numbins un impatto l'aderenza di dimensione e contorno della megavoxels. Mentre il megavoxels si sviluppano più grande (lambda alta, bassa numbins), loro aderenza limite diminuisce. È vero anche il contrario, l'aderenza limite aumenterà con più piccolo megavoxels (lambda basso, alta numbins), tuttavia come la dimensione megavoxel diminuisce, così la loro utilità nella segmentazione rapidamente grandi quantità di voxel. Il parametro opzionale gamma controlla il fattore di scorrevolezza rispetto al costo della fusione di due supervoxels insieme. Piccoli valori di gamma possono migliorare la somiglianza tra due supervoxels, al costo di avere meno megavoxels globale.
Come con supervoxels, la più grande considerazione quando si sceglie e ottimizzazione dei parametri di megavoxel è quanto bene il megavoxels rappresentano i dati. Visualizzati dalla megavoxels da sola come descritto per il supervoxels può essere utilizzato nuovamente per valutare i parametri. Tuttavia, perché megavoxels sarà generalmente molto più grandi e sono tridimensionali, utilizzando la t di strumenti di annotazioneo scegliere singola megavoxels per assicurare il confine tra le regioni di interesse è stretto è anche raccomandato.
Strategia di annotazione
Sono state descritte due strategie di annotazione generale: un approccio di formazione del modello è utile per separare grandi regioni di un dataset, mentre un approccio di segmentazione Super regione è utile per le caratteristiche più piccole, più diversificate come singoli organelli. Annotazioni possono essere organizzate in modo gerarchico, in modo che è possibile annotare grandi regioni in primo luogo, quindi li suddividere in regioni più specifiche utilizzando una relazione padre-figlio. L'etichetta di padre per un'etichetta può essere assegnato facendo clic sull'area a destra della selezione colore etichetta e scegliendo un'etichetta di padre appropriato da un livello precedente. In pratica, maggior parte dei set di dati utilizzare il training del modello e la strategie di segmentazione Super regione per segmentare specificità regioni di interesse.
Nell'esempio del modello formazione qui, pochi ingressi di formazione (sotto forma di annotazioni di base di supervoxel manuale utente) sono stati utilizzati tre fette equidistanti dei dati. In questo modo, l'aspetto della formazione di modello di SuRVoS aumenta notevolmente la velocità con cui la segmentazione è possibile soprattutto quando lavora con grande, differenziato regioni come il divario tra le regioni del corpo fruttifero goosegrass come evidenziato in Figura 3.
Quando modello di formazione, se le previsioni non è visibile, potrebbe essere necessario andare sul tab Visualizzazione e assicurarsi che il livello di previsioni sia acceso e impostato su una quantità appropriata di trasparenza. Inoltre, una fiducia 0 assegnerà ogni supervoxel senza etichetta a un'etichetta, basata su qualunque sia la corrispondenza più vicina è. Fiducia dei 100 solo assegnerà un'etichetta se solo una categoria dell'etichetta ha qualsiasi corrispondenza proporzionale. Tutto il resto è un compromesso di questi due estremi. Quando si seleziona un livello di confidenza che è suggerito per controllare alcune fette controllare visivamente che non ci sono nessun voxel erroneamente previsto prima di salvare la previsione di un'etichetta.
Una buona strategia per l'annotazione utilizzando Super-regioni è quello di utilizzare lo strumento di ingrandimento per ingrandire i dati, annotare pochi organelli contemporaneamente su una fetta, prima tramite un approccio disordinato e "veloce" (Figura 4). Successivamente, spostare su o giù un paio di fette a Z e ripetere questo processo. Perché supervoxels sono tridimensionali, molti dei difetti dell'approccio "disordinato" vengono fissati mediante annotazioni fatte in precedenza o sotto fette. In questo modo, segmentazione è velocizzato e i confini sono forniti dal supervoxels piuttosto che manualmente.
Per eliminare un'etichetta, sono state fornite opzioni di raffinatezza di segmentazione standard. La dilatazione provoca l'etichetta selezionata segmentazione a crescere di dato raggio, erosione e a ridursi. Apertura e chiusura sono l'applicazione di erosione prima e quindi la dilatazione o vice-versa, rispettivamente. E fori di riempimento fa esattamente questo. L'ordine di queste operazioni è importante. In genere, eseguire fori di riempimento, quindi apertura, poi dilatazione funziona bene. Ogni metodo di raffinatezza può essere applicato su una sola fetta ("questa fetta"), su tutte le fette in 2D ("tutte le sezioni (2D)") o in 3D ("volume intero (3D)"). Tutte le sezioni (2D) è raccomandato.
Significato e direzioni future
Efficiente e accurata segmentazione è il collo di bottiglia successiva nell'elaborazione dei dataset 3D, soprattutto con la sistematica raccolta automatizzata di terabyte di dati immagine durante le sessioni di lungo periodo. SuRVoS Workbench può accelerare il processo di segmentazione di un fattore 5 rispetto alla segmentazione manuale. Inoltre, perché i confini sono delineati dal supervoxels, dovrebbe migliorare la variabilità delle segmentazioni risultante. In futuro, speriamo di esplorare modi di utilizzare la segmentazione di una rappresentanza regione 3D di interesse come dati di training da applicare al resto del volume, o anche un volume separato, con elevata fiducia. Questo progresso sarebbe riducono ulteriormente la quantità di tempo utente e input necessario anche complessi volumi biologici segmento, contribuendo ad per alleviare l'ingorgo di segmentazione e di elaborazione di immagine. Ciò, a sua volta, consentirà il confronto quantitativo di dati biologici in vari Stati (ad es. non-malattia, malattia, trattati) con numeri sperimentali robusti.
Divulgazioni
Gli autori dichiarano che non hanno nessun concorrenti interessi finanziari.
Riconoscimenti
Vorremmo citare e ringraziare Rui Wang e Wah Chiu da Baylor College of Medicine per fornire il set di dati di cryoET e Andrew Bodey da Diamond Light Source per assistere con la valutazione di I13. Parti di questa ricerca sono stati sostenuti dalla National Institutes of Health (NIH) concessione n. (P41GM103832) riconosciamo Diamond Light Source per finanziamento congiuntamente Imanol Luengo sotto PhD STU0079.
Materiali
| Name | Company | Catalog Number | Comments |
| computer | n/a | n/a | Must be running Linux operating system and have an NVidia GPU with at least 4 GB of memory |
Riferimenti
- DiamondLightSource/SuRVoS: Version 1.0. Zenodo. , Available from: https://zenodo.org/record/247547 (2017).
- SuRVoS Workbench - Super Region Volume Segmentation. , Available from: https://diamondlightsource.github.io/SuRVoS/ (2017).
- Frangakis, A. S., Förster, F. Computational exploration of structural information from cryo-electron tomograms. Curr Opin Struct Biol. 14 (3), 325-331 (2004).
- Lucić, V., Förster, F., Baumeister, W. Structural studies by electron tomography: from cells to molecules. Annu Rev Biochem. 74, 833-865 (2005).
- Lučič, V., Rigort, A., Baumeister, W. Cryo-electron tomography: the challenge of doing structural biology in situ. J Cell Biol. 202 (3), 407-419 (2013).
- Milani, M., Drobne, D. Focused ion beam manipulation and ultramicroscopy of unprepared cells. Scanning. 28 (3), 148-154 (2006).
- Denk, W., Horstmann, H. Serial Block-Face Scanning Electron Microscopy to Reconstruct Three-Dimensional Tissue Nanostructure. PLoS Biol. 2 (11), (2004).
- Rigort, A., et al. Automated segmentation of electron tomograms for a quantitative description of actin filament networks. J Struct Biol. 177 (1), 135-144 (2012).
- Förster, F., Han, B. -G., Beck, M. Visual Proteomics. Meth Enzymolb. 483, 215-243 (2010).
- Liu, Y., Sigworth, F. J. Automatic cryo-EM particle selection for membrane proteins in spherical liposomes. J Struct Biol. 185 (3), 295-302 (2014).
- Asano, S., et al. A molecular census of 26S proteasomes in intact neurons. Science. 347 (6220), 439-442 (2015).
- Garduño, E., Wong-Barnum, M., Volkmann, N., Ellisman, M. H. Segmentation of electron tomographic data sets using fuzzy set theory principles. J Struct Biol. 162 (3), 368-379 (2008).
- Sommer, C., Straehle, C., Köthe, U., Hamprecht, F. A. Ilastik: Interactive learning and segmentation toolkit. 2011 IEEE Int Symp Biomed Imaging: From Nano to Macro. , 230-233 (2011).
- Darrow, M. C., et al. Structural Mechanisms of Mutant Huntingtin Aggregation Suppression by the Synthetic Chaperonin-like CCT5 Complex Explained by Cryoelectron Tomography. J Biol Chem. 290 (28), 17451-17461 (2015).
- Wang, R., et al. Electron cryotomography reveals ultrastructure alterations in platelets from patients with ovarian cancer. Proc Natl Acad Sci USA. 112 (46), 14266-14271 (2015).
- Shahmoradian, S. H., et al. TRiC's tricks inhibit huntingtin aggregation. eLife. 2, (2013).
- Dai, W., et al. Visualizing virus assembly intermediates inside marine cyanobacteria. Nature. 502 (7473), 707-710 (2013).
- Sandberg, K. Methods for Image Segmentation in Cellular Tomography. Methods Cell Biol. 79, 769-798 (2007).
- Volkmann, N. Methods for Segmentation and Interpretation of Electron Tomographic Reconstructions. Methods Enzymol. 483, 31-46 (2010).
- Tsai, W. -T., et al. From Voxels to Knowledge: A Practical Guide to the Segmentation of Complex Electron Microscopy 3D-Data. J Vis Exp. (90), (2014).
- FEI. Amira & Avizo 3D: Software for Scientific and Industrial Data. , Available from: https://www.fei.com/software/amira-avizo/ (2016).
- Kremer, J. R., Mastronarde, D. N., McIntosh, J. R. Computer Visualization of Three-Dimensional Image Data Using IMOD. J Struct Biol. 116 (1), 71-76 (1996).
- Nguyen, H., Ji, Q. Shape-driven three-dimensional watersnake segmentation of biological membranes in electron tomography. IEEE trans med imaging. 27 (5), 616-628 (2008).
- Rusu, M., Starosolski, Z., Wahle, M., Rigort, A., Wriggers, W. Automated tracing of filaments in 3D electron tomography reconstructions using Sculptor and Situs. J Struct Biol. 178 (2), 121-128 (2012).
- Moussavi, F., Heitz, G., Amat, F., Comolli, L. R., Koller, D., Horowitz, M. 3D segmentation of cell boundaries from whole cell cryogenic electron tomography volumes. J Struct Biol. 170 (1), 134-145 (2010).
- Hecksel, C. W., et al. Quantifying Variability of Manual Annotation in Cryo-Electron Tomograms. Microsc Mpicroanal. 22 (3), 487-496 (2016).
- Achanta, R., Shaji, A., Smith, K., Lucchi, A., Fua, P., Süsstrunk, S. SLIC superpixels compared to state-of-the-art superpixel methods. IEEE trans pattern anal mach intell. 34 (11), 2274-2282 (2012).
- Luengo, I., Basham, M., French, A. P. Fast global interactive volume segmentation with regional supervoxel descriptors. Proc. SPIE. 9784, (2016).
- Luengo, I., et al. SuRVoS: Super-Region Volume Segmentation Workbench. J Struct Biol. 198 (1), 43-53 (2017).
- Wadeson, N., Basham, M. Savu: A Python-based, MPI Framework for Simultaneous Processing of Multiple, N-dimensional, Large Tomography Datasets. arXiv:1610.08015 [cs]. , Available from: http://arxiv.org/abs/1610.08015 (2016).
- DiamondLightSource/Savu: Version 1.2. Zenodo. , Available from: https://zenodo.org/record/198726 (2014).
- Paganin, D., Mayo, S. C., Gureyev, T. E., Miller, P. R., Wilkins, S. W. Simultaneous phase and amplitude extraction from a single defocused image of a homogeneous object. J Microsc. 206 (Pt 1), 33-40 (2002).
- van Aarle, W., et al. Fast and flexible X-ray tomography using the ASTRA toolbox. Opt Express. 24 (22), 25129-25147 (2016).
- Palenstijn, W. J., Batenburg, K. J., Sijbers, J. Performance improvements for iterative electron tomography reconstruction using graphics processing units (GPUs). J Struct Biol. 176 (2), 250-253 (2011).
- Mastronarde, D. N. Dual-Axis Tomography: An Approach with Alignment Methods That Preserve Resolution. J Struct Biol. 120 (3), 343-352 (1997).
- Duke, E., Dent, K., Razi, M., Collinson, L. M. Biological applications of cryo-soft X-ray tomography. J Microsc. 255 (2), 65-70 (2014).
- Arbeláez, P., Maire, M., Fowlkes, C., Malik, J. Contour detection and hierarchical image segmentation. IEEE trans pattern anal mach intell. 33 (5), 898-916 (2011).
- Luengo, I., Basham, M., French, A. P. Hierarchical Piecewise-Constant Super-regions. arXiv:1605.05937 [cs]. , Available from: http://arxiv.org/abs/1605.05937 (2016).
- Luengo, I., Basham, M., French, A. P. SMURFS: superpixels from multi-scale refinement of super-regions. , Available from: http://www.bmva.org/bmvc/2016/papers/paper004/index.html 1-12 (2016).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon