Method Article
Multimodale hierarchische Darstellung der Schnittserien für die Suche nach spezifischen zellulären Ziele innerhalb großer Mengen
In diesem Artikel
Zusammenfassung
Dieses Protokoll zielt auf bestimmte Zellen im Gewebe für die Bildgebung mit nanoskaligen Auflösung mit einem Rasterelektronenmikroskop (REM). Große Anzahl von Schnittserien aus biologischem Material Harz eingebettet werden zuerst in einem Lichtmikroskop, das Ziel zu identifizieren und dann hierarchisch im SEM abgebildet.
Zusammenfassung
Ausrichtung auf bestimmte Zellen in Ultrastrukturforschung Auflösung innerhalb einer gemischten Zellpopulation oder ein Gewebe kann durch hierarchische Bildgebung mit einer Kombination aus Licht- und Elektronenmikroskopie erreicht werden. Proben in Harz eingebettet sind in Arrays bestehend aus Bändern von Hunderten von ultradünnen Abschnitte geschnitten und auf Silizium-Wafer oder leitend beschichtetes Deckgläsern hinterlegt. Arrays werden mit einer niedrigen Auflösung mit einem digitalen Verbraucher wie Smartphone-Kamera oder Lichtmikroskop (LM) für eine rasche großflächige Übersicht oder ein weites Feld Fluoreszenzmikroskop (Fluoreszenz Lichtmikroskopie (FLM)) nach Markierung mit Fluorophore abgebildet. Nach der Post-Färbung mit Schwermetallen, werden Arrays in einem Rasterelektronenmikroskop (REM) abgebildet. Auswahl der Ziele ist möglich von 3D-Rekonstruktionen von FLM generiert oder von 3D-Rekonstruktionen von den SEM Bild Stapeln mit mittlerer Auflösung gemacht, wenn keine fluoreszierenden Marker zur Verfügung stehen. Für Ultrastrukturforschung Analyse ausgewählten Ziele sind schließlich im SEM bei hoher Auflösung aufgezeichnet (wenige Nanometer Bildpixel). Ein Band-Umgang mit Werkzeug, das bei jedem regungsloses nachgerüstet werden kann beweist. Es hilft bei Array-Produktion und Substrat Entfernung vom Boot aus speziellen Messer. Eine Softwareplattform, die ermöglicht die automatisierte Bildverarbeitung von Arrays in die SEM wird diskutiert. Im Vergleich zu anderen großvolumigen EM Datengenerierung, wie z. B. serielle Block-Gesicht SEM (SBF-SEM) oder fokussierten Ionenstrahl SEM (FIB-SEM), dieser Ansatz hat zwei wesentliche Vorteile: (1) die Harz eingebettet Probe konserviert, wenn auch in einer in Scheiben geschnittenen Up-Version. Es kann auf unterschiedliche Weise eingefärbt und mit unterschiedlichen Auflösungen abgebildet. (2) wie in den Abschnitten Post-befleckt werden können, ist es nicht notwendig, Proben stark Block befleckt mit Schwermetallen einzuführen Kontrast für SEM Bildgebung oder machen das Gewebe Blöcke leitfähigen zu verwenden. Dies macht die Methode für eine Vielzahl von Materialien und biologische Fragestellungen. Besonders vorangestellten Materialien z.B.aus Biopsie Banken und Pathologie Labs können direkt eingebettet und in 3D rekonstruiert.
Einleitung
Zur Rekonstruktion großer Mengen von Gewebe bei Ultrastrukturforschung Auflösung wurden eine Reihe von verschiedenen bildgebenden Ansätzen basierend auf SEM verwendet1: umfassende Bewertungen sind erhältlich zB. für SBF-SEM2, FIB-SEM3und Array Tomographie (AT)4. Während das Probenmaterial für die letztere Methode als ein Array von Serienschnitte auf einem Substrat erhalten bleibt, sind SBF-SEM und FIB-SEM destruktiven Methoden auf die probenblock arbeiten und konsumieren sie während der Aufnahme. Durch die Erhebung des Harzes in die SEM hängt auch stark metallisierte Probe Blöcke5.
Auf der anderen Seite profitieren Identifizierung bestimmter Zellen oder Strukturen von Interesse in eine Gewebeprobe vor allem von korrelative Licht- und Elektronenmikroskopie (CLEM)6,7,8. Mit FLM für targeting schließt die Anwendung von großen Mengen an Schwermetallen da das Fluoreszenz-Signal9stillen würde. Für solche nur leicht metallisierten Proben ist AT die Methode der Wahl, da Arrays nach dem LM Imaging leicht Post-gebeizt mit Heavy Metal sein können. Darüber hinaus fast jede Probenart verwendet werden, für AT, sogar Routineproben aus der Pathologe Schatz Brust10.
Ein weiterer großer Vorteil der AT ist das Potenzial für hierarchische11 oder Multi-Resolution imaging12: Es ist nicht notwendig, alles im hochauflösenden Bild, als Ziele können, in einer anderen Modalität (z.B.FLM) oder in ausgewählt werden SEM-Bilder mit niedriger Auflösung. Nur die interessanten Regionen einer Gewebe- oder Bevölkerung an hochauflösenden Bildgebung digitale Daten-Speicherplatz spart und produziert kleinere Bild-Datensätzen, die einfacher zu handhaben sind. Hier die AT-Workflow zeigt sich mit einer eher schwach metallisierte Stichprobe: Hochdruck eingefroren Pflanzenwurzeln (Arabidopsis Thaliana) in hydrophilen Harz eingebettet.
Wie die Arrays sind vorbereitet, gebeizt und im FLM und SEM abgebildet, und wie die Bild-Stacks registriert sind, werden erläutert. Auch, wie der 3D-Rekonstruktion des FLM Volumes verwendet werden kann, bestimmte Zellen für die Bildgebung in der SEM mit nanoskaligen Auflösung auswählen wird dargestellt.
Protokoll
Hinweis: Beispielblöcke polymerisiert werden sollte und einige Schwermetalle enthalten. Fixierung und Einbettung Protokolle für die beiden Stichproben in Figur 1A-B gezeigt wurden beschrieben an anderer Stelle,11. Kurz gesagt, war die Probe in Figur 1A chemisch fixiert, gebeizt en bloc mit 1 % OsO4, dann mit 1 % Uranyl Acetat, erste und in Spurrs Harz eingebettet. Die Probe in Figur 1 b Hochdruck gefroren war, frieren substituierte mit 0,4 % Uranyl Acetat in Aceton und in Lowicryl HM20 Harz eingebettet. Verwenden Sie Handschuhe Pulver für die nächsten Vorbereitungsschritte.
1. Erstellen von Arrays
- Trimmen des Probe-Blocks
Hinweis: Immer Schrauben Sie, auch wenn Teile einfügen.- Fügen Sie den probenblock in der Probenhalter der regungsloses, bringen Sie die Halterung in den Block trimmen und schieben Sie sie in der unteren Stufe der die regungsloses.
- Trim entfernt Harz rund um den eingebetteten Gewebe mit einer Rasierklinge, die nur einen kleinen Rand des Harzes um die Probe verlassen. Schneiden Sie von oben, bis das Ziel erreicht ist.
Hinweis: Die Form des Block-Gesichts möglicherweise trapezförmig oder rechteckig (wir haben beide Formen erfolgreich eingesetzt). Es ist wichtig, die längeren Seiten des Rechtecks als führende und Hinterkante zu verwenden, um sicherzustellen, dass eine große Fläche durch die Stabilisierung der Bänder Leim-Mischung bedeckt ist. - Einsetzen Sie der Probenhalter in den Arm von der regungsloses und trimmen-Block in der unteren Stufe durch den Messerhalter. Legen Sie ein Diamant Trimm-Messer (normalerweise 45°), in den Messerhalter. Richten Sie die Block-Gesicht genau parallel zum Trimmen Messerschneide.
Hinweis: Um genau parallel führende (unten) und nachfolgende (obere) Kanten erzeugen, drehen Sie oder verschieben Sie nur das Messer auf gegenüberliegende Seiten zu trimmen. Für große Mengen (mehr als ein paar hundert Abschnitte) ist ein 90 °-Trimm-Messer vorteilhaft. - Glätten Sie alle vier Seiten zu, dann drehen Sie der Probenhalter, so dass die führenden und Hinterkante jetzt in die horizontale Position sind.
- Bestreichen Sie sorgfältig die führenden und nachfolgenden Seiten des Blocks mit Klebstoff Mischung. Verwenden Sie einen kleinen Pinsel aus ein paar Haare fest mit einem Zahnstocher13gebildet. Führen Sie diesen Schritt schnell, da das Lösungsmittel dieser Mischung innerhalb von Sekunden verdampft. Verunreinigen Sie das Block-Gesicht mit dieser Mischung nicht. Lassen Sie die beschichtete Probe-Block für 5-10 min trocknen.
Hinweis: für größere Anzahl von Abschnitten (> 200), es kann besser sein, nur, die Vorderkante zu beschichten, weil mit der Zeit, eine Ausbuchtung des Klebers an der Hinterkante aufbauen könnte, und potenziell Abschnitte auf des Messers Schneide zurückziehen.
- Vorbereitung des Untergrunds
- Schneiden Sie Stücke von Silizium-Wafern auf eine Größe, die in der Messer-Boot (ca. 2 x 2,5 cm2 für das Jumbo-Messer) passt. Bei Bedarf markieren Sie die Wafer-Stücke (Zahlen oder Buchstaben) mit einer Diamant-Scriber vor der Reinigung oder mit permanent-Marker nach der Reinigung. Reinigen Sie die Silizium-Wafer manuell mit Isopropanol und fusselfreien Gewebe.
Hinweis: Verwenden Sie für die korrelative Bildgebung Indium-Zinn-Oxid (ITO)-beschichtet Glasdeckgläser. Sie müssen sehr vorsichtig behandelt werden, und zusätzliche Reinigung ist nicht notwendig. - Befestigen Sie das Substrat an einem Ende der Trägerplatte mit einem abnehmbaren Klebstoff.
Hinweis: Alternativ können die Substrat Kanten nach unten Parallel auf des Messers Schneide, mit zwei Streifen Klebeband fixiert werden. - Plasma aktivieren (Glimmentladung) das Substrat mit Luft eine hydrophile Oberfläche zu erhalten. Dies sollte so erfolgen, dass ein Tropfen Wasser auf das Substrat breitet sich auf einer sehr dünnen Folie (niedrige Kontaktwinkel, Abbildung 2, D) platziert. Hydrophilie Parameter hängen das Plasma-Gerät verwendet; die hier verwendeten Parameter finden Sie unter Tabelle der Materialien.
Hinweis: Plasmaaktivierung ist sehr volatil, also unmittelbar vor der Substrat-Nutzung führen. - Legen Sie die Träger in die Klammer aus einem Substrat-Halter mit dem montierten Substrat näher auf des Messers Schneide, optimale Benetzung des Substrats in der Messer-Boot zu erreichen.
Hinweis: Eine ausführliche Beschreibung der Substrat-Halter, einschließlich Computer aided Design (CAD) Zeichnungen, erhält in Wacker Et Al. 11
- Schneiden Sie Stücke von Silizium-Wafern auf eine Größe, die in der Messer-Boot (ca. 2 x 2,5 cm2 für das Jumbo-Messer) passt. Bei Bedarf markieren Sie die Wafer-Stücke (Zahlen oder Buchstaben) mit einer Diamant-Scriber vor der Reinigung oder mit permanent-Marker nach der Reinigung. Reinigen Sie die Silizium-Wafer manuell mit Isopropanol und fusselfreien Gewebe.
- Vorbereitung-Schnitt
- Setzen Sie ein Jumbo Diamant Messer in den Messerhalter, festlegen Sie der Freiwinkel (0° für Jumbo-Messer) und füllen Sie das Messer Boot mit destilliertem Wasser. Ansatz die Messer bis zu einer Entfernung von 1 – 2 mm auf die Probe.
- Senken Sie das Substrat in das Wasser mit Schrauben 1 – 3 (Abbildung 2A) des Inhabers Substrat. Überprüfen Sie, dass die Wasserlinie sich im oberen Drittel des Substrats befindet.
Hinweis: Überprüfen Sie, dass der Kleber zwischen Substrat und Träger Platte gut ausgehärtet ist, durch das Substrat mit sauberen Pinzette stieß. Es sollte nicht bewegen. - Weil es schwer, die Bodenfreiheit zu sehen, bei einem Silizium-Wafer, senken das Substrat, bis du es fühlen den Boden berühren. Heben Sie nun das Substrat eine kleine Menge. Stellen Sie sicher, dass das Substrat weder den Träger das Messer Boot während des Schneidens berührt.
- Verwenden Sie eine Spritze oder Pipette, um den Wasserstand im Boot anzupassen. Beim betrachten durch das Fernglas, hinzufügen oder Entfernen von Wasser, bis die gesamte Fläche der Wasseroberfläche eine homogene Reflexion der oberen Beleuchtung von der regungsloses zeigt.
- Wechseln Sie auf dem Boden der regungsloses. Stellen Sie sicher, dass der Arm in der mittleren Position und nicht einziehen mit dem Handrad der regungsloses ist. Das Messer zur Probe zu nähern, bis die Reflexion des Messers auf dem Block Gesicht sichtbar ist.
- Verwenden Sie die Einstellmöglichkeiten der regungsloses um die Probe mit dem Messer auszurichten. Zuerst drehen Sie das Messer dann drehen und Kippen der Probenmaterials.
Hinweis: Es ist richtig ausgerichtet, wenn der helle Streifen, die in die Lücke zwischen der Probe und das Messer zu sehen, parallele Kanten (gerade parallele Linien und nicht keilförmig zeigt). - Überprüfen Sie die Neigung der Probe: Stellen Sie sicher, dass die helle Streifen nicht zu dicker oder dünner während die Probe auf und ab bewegt. Verwenden Sie bei Bedarf die Arc-Einstellschraube, um dies zu korrigieren. Bewegen Sie das Messer näher zur Probe, bis es befindet sich direkt über dem Block-Gesicht (aber nicht berühren).
- Schnittdicke (Futter), schneiden, Geschwindigkeit und Schneidefenster am Steuergerät eingestellt.
- Starten Sie schneiden. Falls erforderlich, warten Sie, bis die ersten Vollschnitt geschnitten wird. Schneiden Sie einige Abschnitte um sicher zu sein, dass sie zusammen an Form Bänder halten (sonst muss der Kleber wieder angewendet werden). Beginnen Sie mit einer hohen Futterwert (maximal 200 nm für das Ultra Messer) bis die ersten Vollschnitt geschnitten wird. Legen Sie den gewünschten feed Wert. Für ausreichende Band Stabilität, eine Dicke von 100 nm und einer Schnittgeschwindigkeit von 1 mm/s ist ein guter Ausgangspunkt. Die niedrigsten erreichbaren Schnittdicke beträgt rund 60 nm, je nach Probenqualität.
- Schnitt zu stoppen. Entfernen Sie alle nicht benötigten (teilweise) Zuschnitte aus der Messers Schneide und Boot mit einer Wimper/Katzenhaare. Gibt es eine Menge kleine Trümmer im Umlauf, das Wasser mit einer Pipette vollständig zu entfernen und das Boot mit frischem Wasser füllen. Jetzt ist der Prozess für das erste produktive Band bereit.
- -Schnitt
- Starten Sie schneiden. Sobald eine Reihe von Abschnitten (die tatsächliche Anzahl hängt von der Größe der Abschnitte und Substrat) geschnitten wurde, beenden Sie den speziellen Prozess und Freigabe die Multifunktionsleiste aus der Messers Schneide durch sanft streichelte auf des Messers Schneide mit einer Wimper14 oder noch besser mit einem sehr weiches Haar von einer Katze das Fell.
- Manipulieren (Gegentakt) das Band mit der Wimpern auf das Substrat und befestigen Sie den ersten Teil auf das Substrat.
Hinweis: Es ist notwendig, das Band schieben, bis es an den trockenen Teil des Substrats klebt. - Weiter Schnitt und befestigen die Bänder auf dem Untergrund. Auf der einen Seite beginnen und allmählich über auf die andere mit jedem neuen Band bewegen.
Hinweis: Vermeiden Sie massive Wasserbewegungen um zu vermeiden, lösen die bereits angeschlossenen Bänder. Das gleiche gilt für Luftströmungen. Verwenden Sie den Atem-Schutzschild mit der regungsloses geliefert. Ungünstige Umweltbedingungen empfiehlt sich ein Gehege für die regungsloses15. - Wann ist das Substrat vollständig bedeckt mit Bändern (in der Regel 4 – 5 Bänder sind möglich), sanft Lift-out-das Substrat vom Messer Boot mit den Mikromanipulator Schrauben des Inhabers Substrat.
Hinweis: Geeignete Bewegungen sind: vertikal anheben (1 Schraube) und drehen/kippen (Schraube 3) oder eine Kombination aus beidem. - Lassen Sie das Band-Array trocknen vor der Lagerung in einer staubfreien Umgebung. Nach dem Trocknen entfernen der klebenden montierten Substrats so schnell wie möglich vom Träger (am selben Tag, ansonsten, den das Substrat wäre zu schwer, zu entfernen oder kann sogar während der Demontage brechen).
2. Färbung für die LM-Bildgebung
Hinweis: Unterschiedliche Färbung/Kennzeichnung Methoden sind möglich, einschließlich Immunfluoreszenz-Protokolle. Hier ist ein direkter, eher unspezifischen Fleck gewählt, die Zellwände zu skizzieren.
-
Propidium Jodid Färbung
- Bedecken Sie den Boden eines großen Glases Petrischale (30 cm Durchmesser) mit Parafilm und säumen Sie den Rand des Tellers mit feuchten Tuch, eine feuchte Kammer zu bauen.
- Verwenden Sie ca. 300-500 µL Lösung pro Deckglas. Einen Tropfen für jede Deckglas auf der Parafilm legen Sie und das Glas kopfüber auf den Tropfen damit die Abschnitte in Kontakt mit der Färbung Flüssigkeit sind. Bedecken Sie die Schüssel und wickeln Sie es mit Alufolie zu die Proben vor Licht zu schützen. Inkubieren Sie die Proben für 16 h bei 4 ° c
- Entfernen Sie das Deckglas mit Pinzette und waschen Sie ihn durch bewegen nach oben und unten in einen 100-mL-Becherglas mit 80 mL destilliertem Wasser gefüllt. Wiederholen Sie diesen Schritt in ein weiteres Becherglas mit frischem Wasser. Trocknen Sie das Deckglas vorsichtig mit Druckluft.
3. Aufnahme der Bildstapel in die FLM
- Platzieren Sie das Deckglas auf der Bühne von einem gemeinsamen Weitfeld-FLM.
- Wählen Sie die entsprechende Filter-Set (Table of Materials) für die Fluoreszenz zu beachten.
- Mit einem geeigneten Objektiv nehmen Sie ein Bild des Objekts in den einzelnen Abschnitten: versuchen Sie, füllen das Sichtfeld und die Ausrichtung konstant zu halten. Für die Wurzelspitze wurde ein 40 X Luft Ziel verwendet.
Hinweis: Wenn die Bänder nicht ganz gerade sind, helfen eine rotierenden Bühne um die Abschnitte neu zu orientieren, während der Einnahme von Bildern. Wenn möglich, zentrieren Sie das Bild auf eine bestimmte Funktion oder halten ein Feature wie die Kante des Abschnitts im gleichen Abstand zum Rand des aufgenommenen Bilds. - Wenn möglich, verwenden Sie 16-Bit gesättigten Pixel zu beschränken und die Belichtungszeit konstant zu halten.
4. Anmeldung des Stapels FLM Bild
- Importieren Sie die Bildserien in Fidschi16 als virtuellen Stapel.
- Öffnen Sie eine neue TrakEM17 (leer) aus dem Dateimenü.
- Rechtsklick in das Bildfeld, und importieren Sie den Stapel in TrakEM als "Eine Scheibe pro Schicht".
- Richten Sie Schichten (Rechtsklick ins Bildfeld), legen Sie den Bereich (erstes Bild für die Ewigkeit), und wählen Sie als Referenz. Verwenden Sie für alle Einstellungen die Standardwerte, und wählen Sie steif wie die gewünschte Transformation.
- Wenn die Registrierung fertig und zufrieden stellend ist, sparen die ausgerichteten Dataset durch Rechtsklick und wählen Sie exportieren. Machen Sie flache Bild zu, legen Sie den Bereich vom ersten zum letzten Bild und lassen Sie die Software, die daraus resultierende Stapel zeigen. Speichern Sie den Stapel im Tif-Format.
5. Färbung und Montage für die SEM-Bildgebung
Hinweis: Vorbereitung befleckenden Lösungen finden Sie unter Tabelle der Materialien. Lösungen können bei 4 ° C für bis zu 12 Monate gespeichert werden, geschützt vor Licht und Luft.
Achtung: Citrat und Uranyl Bleiacetat enthalten Schwermetalle, die giftig sind. Tragen Sie Handschuhe und entsorgen Sie die Abfälle gemäß den Anweisungen der örtlichen Behörden.
- Bedecken Sie den Boden eines großen Glases Petrischale (30 cm Durchmesser) mit Parafilm und säumen Sie den Rand des Tellers mit feuchten Tuch, eine feuchte Kammer zu bauen.
Hinweis: Es ist wichtig, dass mehrere Pellets von NaOH in der Petrischale in der Nähe der Färbung Tropfen gegen übermäßige Ausscheidung von Blei Citrat positioniert sind. -
Uranyl Acetat Färbung
- Zentrifugieren Sie die Uranyl-Acetat-Lösung bei 2.680 x g für ein paar Sekunden, um kleine Trubstoffe.
- Verwenden Sie ca. 300 – 500 µL Lösung pro Deckglas. Einen Tropfen für jede Deckglas auf der Parafilm legen Sie und das Glas kopfüber auf den Tropfen damit die Abschnitte in Kontakt mit der Färbung Flüssigkeit sind.
- 10 min bei Raumtemperatur inkubieren Sie, und bedecken Sie die Schüssel während der Färbung.
- Entfernen Sie das Deckglas mit Zange und waschen durch bewegen nach oben und unten in einen Becher gefüllt mit destilliertem Wasser (siehe Punkt 2.1.3).
-
Führen Citrat Färbung
- Während die Uranyl-Acetat-Inkubation bereiten Sie die Blei-Citrat-Lösung.
Hinweis: Blei Citrat sollten immer unmittelbar vor dem Gebrauch entfernen Ausscheidungen gefiltert werden. Auch Zentrifugieren Sie die Blei-Citrat-Lösung bei 2.680 x g für ein paar Sekunden wie in Schritt 5.2.1. - Verwenden Sie ca. 300 – 500 µL Lösung pro Deckglas. Legen Sie einen Tropfen für jede Deckglas auf der Parafilm unmittelbar vor dem Waschen der Deckgläsern nach Uranyl Acetat Färbung, und bringe das Glas kopfüber Tropfen, so dass die Teile in Kontakt mit der Färbung Flüssigkeit sind. Legen Sie die Tropfen (300 – 500 µL) auf die Parafilm unmittelbar vor dem Waschen der Deckgläsern nach Uranyl Acetat Färbung.
Hinweis: Zur Vermeidung von Bildung von Ausscheidungen nicht atmen Sie auf die Blei-Citrat-Tröpfchen ein. - Legen Sie die gewaschenen Deckglas kopfüber auf den Tropfen (es gibt keine Notwendigkeit, es zu trocknen).
- 5 min bei Raumtemperatur inkubieren Sie, und bedecken Sie die Schüssel während der Färbung.
- Entfernen Sie das Deckglas mit Pinzette und waschen Sie es wie oben beschrieben in einen Becher mit frischem Wasser (Schritt 5.2.4).
- Während die Uranyl-Acetat-Inkubation bereiten Sie die Blei-Citrat-Lösung.
- Trocknen Sie das Deckglas vorsichtig mit Druckluft.
-
Montage-Proben für die SEM-Bildgebung
- Montieren Sie die Silizium-Wafer auf Aluminium Baumstümpfen mit einem klebrigen Kohlenstoff-Pad.
Hinweis: ITO-beschichtete Deckgläsern können entweder montiert werden mit Silberfarbe und Cu-Band – sicherstellen, dass die leitende Oberfläche ist an den Stub verbunden – oder mit Carbon-Pads wie oben beschrieben. In diesem Fall kann eine leitende Verbindung von der ITO-Oberfläche an den Stub mit einem Tropfen Silberfarbe erfolgen.
- Montieren Sie die Silizium-Wafer auf Aluminium Baumstümpfen mit einem klebrigen Kohlenstoff-Pad.
(6) hierarchischen Bildgebung im SEM
Hinweis: In einem Feldemission SEM, wählen Sie eine niedrige Primärenergie (3 kV oder niedriger), einen Strahlstrom in einem Bereich von 50 bis 800 pA zu vermeiden, laden und einer geeigneten Arbeitsabstand für effiziente Sammlung von sekundären und/oder zurückgestreute Elektronen. Auswahl an den Strahlstrom richtet sich nach den Eigenschaften der Probe (zB., Einbettung Harz); die Elektron-Dosis werden auch einen Kompromiss zwischen ein kleiner Strom (weniger schädlich für die Probe) und ein hoher Strom, das ist vorteilhaft für imaging-Geschwindigkeit und senkt somit das Gesamtbild Erfassungszeit. Spezielle Detektoren für zurückgestreute Elektronen guten Kontrast bieten, sind weniger anfällig für die Aufladung der Probe und zeigen weniger die Probe obertägigen Artefakte (Falten, Messer Marken). Kontrast und Helligkeit sollten angepasst werden, so dass das Histogramm zentriert ist.
-
SEM-Bildgebung
- Definieren Sie zunächst die vier Ecken des Arrays durch ein Bild von jeder Ecke bei kleiner Vergrößerung über 100 X greifen. Erstellen einer Region of Interest (ROI) umschließt das gesamte Array. Zuweisen einer bildgebenden Protokoll mit den folgenden Parametern: verwenden Sie einen Sekundär-Elektronen (SE)-Detektor, der high-Speed-Bildgebung ein großes Bild-Pixel-Größe (zum Beispiel 1.000 nm) und eine kurze Verweilzeit (z. B. 0,2 µs) ermöglicht.
Hinweis: Um Elektron optischen Einschränkungen bei einem großen Scan Gesichtsfelder (FOV), zu überwinden, die Verzerrungen an der Peripherie der Bilder führen könnte, verwenden Sie dedizierte geringer Vergrößerung Modi (bereitgestellt durch die meisten Hersteller von SEM) oder verwenden Sie Medium, 1 bis 2 k Scan Felder für einzelne Bilder. - Generieren Sie einen Abschnitt durch die Schaffung eines ROI nur das Gewebe im ersten Abschnitt skizziert. Klonen Sie es auf alle nachfolgenden Abschnitten mit dem Stempel-Werkzeug. Drehen des ROIs als gebogene Bänder aufnehmen musste.
- Aufzeichnen der Bildserie mit einer mittleren Pixelgröße (etwa 50 nm) und eine Zeit lang genug, um zu identifizieren und erkennen die Zielstruktur wohnen. Verwenden Sie eine FOV für einzelne Bilder in einer Reihe von 6-10 k Pixel.
Hinweis: Die Atlas 5-Software kann automatisch Mosaiken bestehend aus angrenzenden Bilder ROI/Abschnitt großflächig über die Schnittserien decken sammeln. - Erstellen Sie eine Website, die inmitten dieser Abschnitt-Set, mit der Zielstruktur für die höhere Auflösung SEM Bildgebung. Machen Sie den ROI groß genug ist, entfallen Bühne Präzision. Überprüfen Sie und stellen Sie die Positionen der Seiten.
Hinweis: Es ist wichtig, ROIs so platzieren, dass das Zentrum, wo der Autofokus und die Autostigmation wird, nicht auf "leer" Material ohne strukturelle Details, z.B.sitzt., Vakuolen. - Die Autofokus-Einstellungen definieren und überprüfen Sie die Leistung über mindestens die Länge eines Bandes (d. h., die längste Strecke, die die Bühne zu reisen hat) auf eine kleine ROI nah an dem Aufstellungsort, die abgebildet wird.
- Definieren Sie ein imaging-Protokoll für den hochauflösenden SEM-Erwerb. Membran-Fächer, 3 – 5 nm Pixel Bildgröße wählen Sie sehen. Wählen Sie eine Verweilzeit je nach den Detektor, so dass das Bild nicht zu laut ist.
- Vor dem Start der Akquisition, definieren Sie die Fokus-Werte zumindest im ersten Abschnitt jedes Band über die Check-Protokoll-Option.
- Starten Sie die automatisierte SEM Imaging über die gesamte Serie des Ziels ROIs.
- Exportieren Sie die erfassten Daten als Bildserie, vorzugsweise im Tif-Format.
- Definieren Sie zunächst die vier Ecken des Arrays durch ein Bild von jeder Ecke bei kleiner Vergrößerung über 100 X greifen. Erstellen einer Region of Interest (ROI) umschließt das gesamte Array. Zuweisen einer bildgebenden Protokoll mit den folgenden Parametern: verwenden Sie einen Sekundär-Elektronen (SE)-Detektor, der high-Speed-Bildgebung ein großes Bild-Pixel-Größe (zum Beispiel 1.000 nm) und eine kurze Verweilzeit (z. B. 0,2 µs) ermöglicht.
7. Anmeldung des Stapels SEM Bild
- Importieren Sie Bildserien in Fidschi als virtuellen Stapel.
Hinweis: Diese werden große Datendateien im Bereich von ein paar GB je nach der Anzahl der Abschnitte und die Größe des ROI. - Ernte der REM-Aufnahme für die Weiterverarbeitung zu einem Gebiet möglichst nahe an die Struktur des Interesses wie möglich zu stapeln und passen Sie die Helligkeit und den Kontrast.
- Öffnen Sie eine neue TrakEM17 (leer) aus dem Dateimenü.
- Rechtsklick in das Bildfeld, und importieren Sie den Stapel in TrakEM als "Eine Scheibe pro Schicht".
- Richten Sie die Schichten (rechter Mausklick in das Bildfeld), kleinste Quadrate als Modus wählen, legen Sie den Bereich (erstes Bild für die Ewigkeit), und wählen Sie als Referenz. Die Einstellungen verwenden Sie die Standardwerte und wählen Sie steif wie die gewünschte Transformation.
- Wenn die Registrierung abgeschlossen und zufriedenstellend ist, speichern die ausgerichteten Dataset durch Rechtsklick und wählen Sie exportieren. Machen Sie ein flaches Bild zu, legen Sie den Bereich vom ersten zum letzten Bild und lassen Sie die Software, die daraus resultierende Stapel zeigen. Speichern Sie den Stapel im Tif-Format.
Ergebnisse
Der Workflow beschrieben hier (Abbildung 1) startet mit einer Probe in einem Block Harz eingebettet. Während der Probenvorbereitung einige Schwermetalle in das Gewebe eingeführt werden sollten, aber es ist nicht notwendig, optimiert für ziemlich starke Metallisierung Protokolle zu verwenden. Abbildung 1A zeigt eine Pflanzenwurzel (Kresse) Block-gefärbten konventionell mit 1 % OsO4 und 1 % Uranyl Acetat, während die Arabidopsis Wurzel in Figur 1 b nur schwach ist metallisiert mit 0,5 % Uranyl Acetat. Die letzteren Probentyp eignet sich am besten für korrelative Ansätze wie einige Schwermetalle sind in der Regel Fluoreszenz zu stillen. Mit einem speziellen Substrat Halter (Abbildung 2), können Arrays von mehreren hundert Abschnitte werden hergestellt (Abbildung 1). Nach fluoreszierende Kennzeichnung, sind solche Arrays abgebildet in einem standard Weitfeld-FLM (Abbildung 1), dann gefärbt mit Heavy-Metal-Lösungen und abgebildet in einem SEM in verschiedenen Auflösungen (Abb. 1E–G).
Wichtige Werkzeuge für die reproduzierbare Erzeugung von Arrays, sind vor allem, wenn mehrere Bänder aus dem Mikrotom Messer Boot auf einem Substrat, Platzierung der Substrat-Halter (Abb. 2A, speziell in der Autoren-Labor) und ein Jumbo-Diamant Messer mit einem Boot groß genug für Objektträger (Abbildung 2 b). Eine flache Meniskus, so dass gute Beobachtung der Bänder, ist notwendig und kann durch Plasmareinigung des Substrats erreicht werden: ein kleiner Tropfen destilliertes Wasser sollte keine Objektiv-artige Struktur bilden, auf dem Substrat wie in Abbildung 2 (unbehandeltes Substrat), aber eine dünne Folie (Abbildung 2D, Plasma aktiviert Substrat). Unter diesen Bedingungen Bänder befestigt auf den trockenen Teil des eine ITO-beschichtete Deckglas sind leicht (Abb. 2E) visualisiert und können beobachtet und kontrolliert während der Lift-out-das Substrat aus dem Wasser.
Als Beispiel wurden Arrays mit Propidium Jodid, beschriften Sie die pflanzlichen Zellwände befleckt mit einem breiten Standardfeld FLM (Abbildung 3A) abgebildet. Da die Abschnitte nur 100 sind nm dick, noch übermäßig Färbung, wie hier gezeigt führt wenig verwischen. Nach der Registrierung wurden die beiden Zellen vollständig umschlossen in der rekonstruierten Volumen aus den Bildstapel (Abb. 3 b) für die hochauflösende Bildgebung in 3D ausgewählt (siehe auch Ergänzende Film S1). Im Anschluss an weitere Färbung mit Uranyl-Acetat und Lead-Citrat, wurden die Arrays abgebildet im SEM Abbildung 3 C zeigt eine Übersicht, aufgenommen mit 60 nm Bildpixel; Das dunkle Quadrat in der Mitte des Bildes zeigt die Position, wo die Autofokus-Funktionen wurden hingerichtet, und die zusätzliche Dosis führte zu leichten Verschmutzungen. Geeignete ROIs in diesen Serienschnitte (Scheiben 51 bis 248 435 Scheiben insgesamt) mit den zwei Zielzellen im FLM Stapel ausgewählt wurden dann mit einer 5 nm Bildgröße Pixel aufgezeichnet (Abbildung 3D; siehe auch Ergänzende Film S2).
Automatisierte hierarchische Darstellung der Arrays im hier beschriebenen SEM erfolgte mit der Software/Hardware-Plattform-Lösung ZEISS Atlas 5. Zuerst ein Überblick über das gesamte Array wurde erstellt mit der SE-Detektor mit sehr großen (1.000 nm) Bild Pixel und sehr geringe Verweilzeit (Abb. 4A). Ein ROI umreißen nur das Gewebe wurde in der ersten Sektion und vermehrt auf andere Abschnitte des Arrays. Dieser Abschnitt Satz wurde dann mit 60 nm Bildpixel über eine längere Verweilzeit (Abbildung 4 b) aufgenommen. Schließlich wurde ein Website-Set, mit den zwei Zielzellen plus eins "Schicht" der umgebenden Zellen für Bühne Ungenauigkeit, Konto mit den folgenden Parametern eingerichtet: ESB (Energie selektiv Backscatter) Detektor, 5 nm Bild Pixel, sehr lang (40 µs) wohnen ( Zeit Abbildung 4). Einzoomen auf solch ein Bild zeigt subzelluläre Detail (Abbildung 4) z. B. Mitochondrien (M), Vakuolen (V), Kern (N) und endoplasmatische Retikulum (Pfeile). Siehe auch Ergänzende Film S3 für Zoomen aus einen Überblick über das gesamte Array auf der subzellulären Detail einer Zielzelle.
Das Array abgebildet (200 Teile) plus eine zusätzliche nahm einer der 250 Abschnitte etwa 8 h zu produzieren, eine Nacht, für LM, und eines Tages (manuell) auf die FLM aufzeichnen zu beflecken. Post-Färbung dauert ca. 1-2 h insgesamt, abhängig von der Anzahl der einzelnen Arrays. Für SEM-Aufnahme, ein paar Stunden sind erforderlich, um den Atlas-Lauf einzurichten, und automatisierte Aufnahme war 3 – 4 h für die mittlere Auflösung (60 nm Pixelgröße) Abschnitt Set (200 Abschnitte, 450 x 200 µm2) und etwa 5 Tage für die hochauflösende (5 nm Pixelgröße) ROI enthält die beiden Zielzellen (200 Abschnitte, 55 x 30 µm2). Beachten Sie, dass eine sehr langsame Scan-Geschwindigkeit wegen der geringen Metallgehalt des hier gezeigten Beispiels verwendet werden musste, um ein gutes Signal-Rausch-Erkennung, die (für den derzeit verfügbaren Detektor) eine Verweilzeit von 40 µs für die hochauflösende ROI impliziert zu erreichen.
Anfällig für Fallstricke im gesamten Workflow gibt es mehrere Schritte: im Idealfall sollte die Bänder werden mehr oder weniger gerade und in der richtigen Reihenfolge (Abbildung 5A). Allerdings sind oft gebogen (Abb. 5 b), gebogene (Abbildung 5) oder sogar gebrochen Bänder produziert. Dies kann durch falsche trimmen (führenden und Hinterkanten nicht genau parallel) oder ungleichmäßig angewandten Klebstoff, sondern auch aus einer asymmetrischen oder ungleichmäßig infiltrierten Probe führen. Besonders lästig sind Proben, weiche und sehr harte Komponenten enthält. Die letztgenannten Komponenten möglicherweise schwer, wie die Zellwand der Pflanzenwurzeln hier gezeigt (Abbildung 5) zu infiltrieren. In diesem Fall können Falten (Pfeilspitzen) durch Variable Kompression und Entspannung beim Schneiden leicht verursacht werden. Für die automatisierte Bildverarbeitung in der SEM, sind geschwungene Bänder kein großes Problem, da ROIs gedreht werden können, um die Krümmung des Menübands unterzubringen.
Ein weiterer wichtiger Schritt im Protokoll Färbung: unzureichende waschen kann Rückstände auf dem Abschnitt (Abb. 5E, F) führen und im schlimmsten Fall, decken die interessanteste Gegend (Kreis auf eines der beiden Zielzellen in Abbildung 5F). Auch Staub (Abbildung 5, stark Licht streuenden Partikeln) eingeführt in das Messer Schiff, z.B.mit einem schmutzigen Substrat Träger, kann schwerwiegende Probleme verursachen: In der FLM Staub stark fluoreszierende werden kann (vgl. einige Scheiben in ergänzende Film S1) zu einem Ausmaß, die einige Registrierung Algorithmen nicht funktionieren. Die "align" Funktion in TrakEM17 verträgt jedoch solche Stapel wie in Ergänzenden Film S1gezeigt.
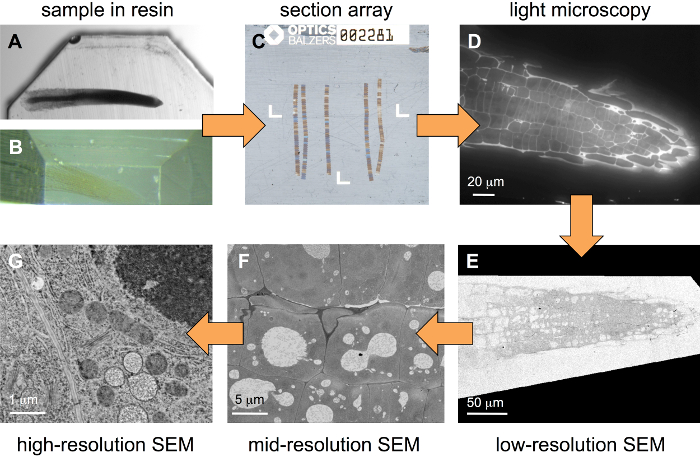
Abbildung 1: Workflow für die korrelative hierarchische Darstellung. Ausgehend von einer Stichprobe in einem Harz-Block (A, stark metallisierte Probe) eingebettet, die Probe zuerst getrimmt (B, schwach metallisierte Probe) und dann arrays bestehend aus mehreren Bändern von Schnittserien (C), hier platziert auf einer ITO-beschichtete Deckglas ein regungsloses hergestellt werden. Nach der Färbung mit einem Fluoreszenzfarbstoff werden Stapel von Bildern in einer Weitfeld-FLM (D) aufgezeichnet. Nachdem weitere Färbung Runden mit Heavy Metal Salze, werden Stapel in einem SEM (E–G) in verschiedenen Auflösungen (Bild Pixelgrößen) abgebildet. Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 2: Werkzeuge für die Erstellung des Arrays. Substrat-Halter montiert von Mikromanipulatoren mit sieben Achsen der Bewegung mit einem standard regungsloses (A) verbunden: die Schrauben, mit Kreisen hervorgehoben sind für vertikale (1) und (2) Horizontalbewegung und zum Kippen (3) des Substratträgers . Das Jumbo Diamant Messer mit einem übergroßen Boot, großer Substrate (Pfeil), hier mit einem Stück Plasma aktiviert Silizium-Wafer, montiert auf einer Folie Größe Aluminium-Träger (B) unterzubringen. 20 µL Tropfen destilliertes Wasser platziert auf einer unbehandelten Silizium-Wafer-Substrat (C) oder auf einem Plasma aktiviert Substrat (D). Vier Bänder schweben in der Messer-Boot, ein Deckglas ITO beschichtet mit ihren unteren Enden befestigt. Bitte klicken Sie hier für eine größere Version dieser Figur.
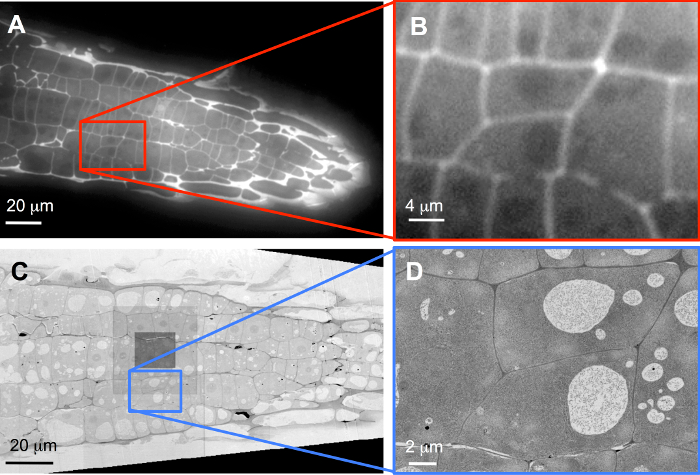
Abbildung 3: Korrelation der LM-Daten mit SEM. Übersichten (A, C) und Zielzellen (B, D) erfasst mit FLM (A, B) und SEM (C, D). (B) ist ein Software-Zoom, und die ursprünglichen Daten wurden aufgenommen mit einem 40 X Objektiv auf einem 1.388 x 1.040 Pixel-Kamera-Chip, während (C) mit 60 nm Pixel Bildgröße und (D) aufgezeichnet wird mit 5 nm Pixel Bildgröße, illustrieren die wahre Erhöhung der Auflösung im SEM Klicken Sie bitte hier, um eine größere Version dieser Figur.

Abbildung 4: hierarchische imaging im SEM mit ZEISS Atlas 5 at Überblick über ein Array mit 1.000 nm Bildpixel mit der SE-Detektor (A) aufgenommen. Abschnitt enthält mit den ROI auf Gewebe in jedem Abschnitt gelegt und mit 60 nm Bildpixel (B) aufgenommen. Seite setzt mit einem seriellen ROI auf Zielzellen platziert und mit 5 nm Bildpixel (C) aufgenommen. Beim Zoomen in solchen hochauflösende Bilder (D) sichtbar intrazellulären Membran Fächer wie Vakuolen (V), Kern (N) und Mitochondrien (M) endoplasmatische Retikulum (Pfeile). Bitte klicken Sie hier für eine größere Version dieser Figur.
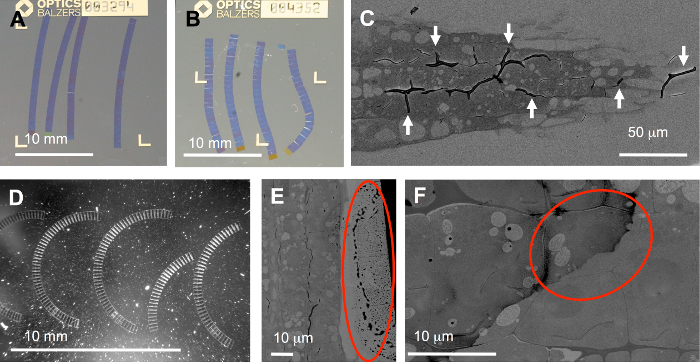
Abbildung 5: typische Probleme. (1) die aus den speziellen Prozess: Bänder gelegt auf die ITO-beschichtete Deckgläsern sind im Idealfall gerade (A), aber unregelmäßig Kompression beim Schneiden verursachen Bent (B) oder gekrümmte Bänder (D) oder sogar Falten (C). (2) verursacht durch den Umgang mit Substrat und Bändern in Wasser, z.B.beim Schneiden und färben: Lichtstreuung Partikel auf Substrat (D), Rand der Tröpfchen auf Abschnitt (Kreis in E) oder Schmutz heraus über Gewebe aufgrund unzureichender verschmiert Waschen nach der Färbung (F). Bitte klicken Sie hier für eine größere Version dieser Figur.
Ergänzende Film S1: FLM Bildstapel. 435 Bilder in Fidschi16 mit TrakEM17 ausgerichtet und als Filmdatei (.avi) gespeichert. Bitte klicken Sie hier, um diese Datei herunterladen.
Ergänzende Film S2: SEM Bildstapel. 210 Bilder ausgerichtet in Fidschi16 mit TrakEM17. Der ursprüngliche Stapel (300 Bilder) dieses Datensatzes wurde 15 GB. Um den Stapel von 3,3 GB (nach Ausrichtung und beschneiden auf nur zwei Zellen gezielt) zu verkleinern, wurde es so skaliert, in x und y um den Faktor 0,2 mit Fidschi und dann als AVI-Film gespeichert. Bitte klicken Sie hier, um diese Datei herunterladen.
Ergänzende Film S3: Zoomen mit verschiedenen Auflösungsstufen im SEM Film erstellt und von der Atlas 5-Software im MP4-Format exportiert. Bitte klicken Sie hier, um diese Datei herunterladen.
Diskussion
Ein Workflow für die Ausrichtung auf bestimmte Zellen in einem Gewebe von Multi-modale hierarchische AT zeigte: eine Probe Harz eingebettet in Arrays von Schnittserien, die auf einem leitfähigen Substrat mit einem maßgeschneiderten Substrat Halter platziert werden zerschnitten ist. Nach Markierung mit einem Fluorophor und Bildgebung in der FLM, dient das rekonstruierte Volumen für die Auswahl der Zielzellen. Nach zusätzlichen Färbung mit Schwermetallen runden, Kontrast einzuführen, sind diese Ziele über mehrere hundert Abschnitte mit nanoskaligen Auflösung in einem SEM mit einer automatisierten Software-Plattform abgebildet.
Für die Herstellung von dicht gepackten Arrays mit mehreren langen Bändern, ist Inhaber eines Substrat ähnlich der hier beschriebenen notwendig. Eine qualifizierte und geduldige Person möglicherweise mehrere Bänder an Silizium-Substrat, halb eingetaucht in das Messer Boot anhängen und das Array abrufen, indem Sie nach und nach Senkung des Wasserspiegels bis die Bänder auf dem Substrat sitzen. Jedoch in der nach unserer Erfahrung gibt es eine Tendenz zur Bildung zu zerbrechen, wenn das Substrat Teil des Bootes Messer (vgl. Note 1.3.2 im Protokoll) berührt. Darüber hinaus ist dieses Verfahren sehr viel schwieriger mit ITO-beschichtete Substrate: (1) aufgrund der Transparenz der ITO-Glas, ist es schwierig, den Rand des Wassers zu sehen, wo die Enden der Bänder müssen befestigt sein; (2) da die ITO-beschichtete Oberfläche viel rauer als die Hochglanz polierte Siliziumwafer ist, die Bänder sind in der Regel, während Lift-out- und kleinere Fragmente, bestehend aus ein paar teilen können schweben, so zerstören die Reihenfolge der Abschnitte zu brechen.
Der gesamte Workflow ist auch möglich ohne Korrelation zur FLM Daten. In diesem Fall möglicherweise Datenerhebung im SEM in mehreren Sitzungen durchgeführt werden. Eine erste 3D Rekonstruktion oder zumindest Auswertung der Daten mit geringer oder mittlerer Auflösung kann erforderlich sein, Ziele zu identifizieren. Darüber hinaus können konventionelle histologische Flecken für Hellfeld LM (nicht erfordern FLM) angewendet werden. Natürlich sind andere Optionen6,7,8 Antikörper Kennzeichnung in den Arrays, wie bereits in dem ersten Dokument über18, oder genetisch codiert fluoreszierende Proteine (XFPs) oder Pre-einbetten Kennzeichnung mit der Erhaltung der Fluoreszenz während der Probenvorbereitung.
Eine allgemeine Einschränkung der diskutierte Methode ist die Verwendung von Abschnitten einer bestimmten Dicke und die daraus resultierende diskrete Probenahme das 3D-Volumen: Auflösung in Z kann nur so gut sein wie die Dicke der Abschnitte sein, da das SEM nur Daten aus der Abschnitt Oberfläche (d sammelt Epending auf die primäre Energie/Landung Energie ausgewählt). Dies bedeutet, dass die daraus resultierenden 3D-Volumen anisotropen Voxel, zB., 5 x 5 x 100 nm3 wenn 100 nm Abschnitte und eine Bild-Pixel-Größe von 5 nm verwendet. Für sehr kleine Unternehmen in einem Größenbereich unter 1 µm kann dies nicht für eine wahre Ultrastrukturforschung Beschreibung ausreichen. Eine weitere technische Einschränkung ist die Genauigkeit der Bühne verwendet im SEM für automatisierte imaging. Aus diesem Grund ist es notwendig, wählen einen ROI größer als die Spezifikationen der Bühne Genauigkeit zu gewährleisten, dass das gesamte Zielgebiet abgebildet wird.
Im Vergleich zum SBF-SEM und FIB-SEM als Block-Gesicht bildgebenden Verfahren, hat korrelative AT den endgültigen Nachteil anisotropen Voxel, wie oben beschrieben. Mit FIB-REM isotrope Voxel von 5 x 5 x 5 nm3 erhalten Sie bei eine richtigen Drift-Korrektur.
Lücken in der rekonstruierten Volumen durch den Verlust der Abschnitte während der Vorbereitung des Arrays könnten auch ein Anliegen, die nicht mit SBF-SEM oder FIB-SEM begegnet ist Mit einer guten Band Stabilisierung durch Kleber, ist dies in der Regel nur ein Problem für den letzten Abschnitt eines Bandes: beschädigt werden, wenn es aus der Messers Schneide mit der Wimper zu veröffentlichen. Allerdings beeinflusst der Verlust eines Abschnitts in jeder 20 – 50 Abschnitten nach unserer Erfahrung nicht Bild-Registrierung.
Auf der anderen Seite verleiht die Möglichkeit, Arrays nach dem Färben gutes Signal und Kontrast für SEM Bildgebung auch auf schwach metallisierte Proben wie der Hochdruck Wurzelspitzen, die hier gezeigte eingefroren. Daher ist es nicht optimale Ultrastrukturforschung Konservierung durch zahlreiche chemische Fixierung und Metallisierung Schritte Kompromisse notwendig. Darüber hinaus liefern Routineproben aus der Pathologie-Labor mit einem mittleren Grad an Metallisierung hervorragende Daten10. Ein Post-Einbettung Kontrastverstärkung ist generell nicht möglich für SBF-SEM und FIB-SEM. Außerdem, da diese Methoden destruktiv sind, ist d.h., verbrauchen die Probe während der Bildgebung, hierarchische imaging bei verschiedenen Auflösungen und Websites oder wiederholte Bildgebung zu späteren Zeitpunkten in der Zeit unmöglich. Im Prinzip unbegrenzte Mengen, bestehend aus großen FOVs (z. B.bis zu mehreren Millimetern für ganze Mäusegehirnen in Connectomics) erstellt durch Nähte Mosaiken und eine Vielzahl von Abschnitten von AT, während im FIB-SEM, FOVs über 100 µm X 100 µm erworben werden können sind schwer zu erreichen mit Routine Instrumente.
Weiteren Automatisierung des beschriebenen AT-Workflows wäre ein klarer Vorteil, da die oben genannten Methoden SBF-SEM und FIB-SEM sowohl schneiden und Bildgebung innerhalb desselben Instruments in einer vollständig automatisierten Weise durchführen. Gibt es eine Art der Automatisierung der Schneiden: The ATUMtome12 können generieren und Tausende von Abschnitten zu sammeln, aber die Verwendung von Kapton Klebeband wie ein Substrat solche macht arrays schwierig, Bild in ein FLM. Auf die ITO-beschichtete Deckgläsern hier verwendet sollte auch super-Auflösung möglich sein. Eine weitere, sehr wünschenswerte Ziel für Automatisierung wäre die Aufnahme der FLM-Daten-Stacks. Auf der anderen Seite Automatisierung kann teuer sein und mit Ausnahme des Substrat-Inhabers der hier vorgestellten Workflow hängt (in Bezug auf die Hardware) nur Instrumentierung in der Regel verfügbar in einer Routine EM Labor oder Core Einrichtung, so dass es niedrige Ebene Zugriff.
Offenlegungen
KIT hat Rückzahlungen durch Boeckeler Instrumente zur Versorgung der ein funktionierendes Modell der Substrat-Inhaber erhalten. Marlene Thaler ist Mitarbeiter des ZEISS Microscopy GmbH, Hersteller von Mikroskopsystemen, die in diesem Artikel erwähnt. Darüber hinaus bietet ZEISS bestimmte Lösungen wie ZEISS Atlas Lösungspakete für ein breites Anwendungsspektrum in der großflächigen, 3D Bildgebung für SEM und FIB-SEM Instrumente. Andere Autoren haben nichts preisgeben.
Danksagungen
Diese Arbeit wurde von Grant FKZ 13GW0044 vom Bundesministerium für Bildung und Forschung, Projekt MorphiQuant-3D unterstützt. Wir danken Carolin Bartels für den technischen Support.
Materialien
| Name | Company | Catalog Number | Comments |
| Instrumentation | |||
| Ultramicrotome | RMC | PT-PC | Alternative: Leica UC7 |
| Substrate holder | RMC | ASH-100 | Alternative: home built |
| Plasma cleaner | Diener | Zepto 40kHz | Alternatives: Ted Pella Pelco or other benchtop plasma cleaner Example Parameters for Diener Zepto with 40kHz generator (0-100W); 0.5 mbar, 5 sccm (Air), 10% performance |
| Widefield fluorescence light microscope | Zeiss | Axio Observer.Z1 | Alternatives: Leica, Nikon, Olympus |
| Fluorescence filter set | Zeiss | 43 HE (Cy3/DsRed) | |
| Objective lens | Zeiss | Zeiss Neofluar 40x | 0.75 NA |
| Decent workstation able to handle GB-sized image data | |||
| FESEM | Zeiss | Ultra 55 | Alternatives: FEI, Jeol, Hitachi, TESCAN |
| Name | Company | Catalog Number | Comments |
| Sectioning | |||
| Razor blades | Plano | T585-V | |
| Diamond knife for trimming 45° | Diatome | DTB45 | |
| Diamond knife for trimming 90° | Diatome | DTB90 | |
| Jumbo diamond knife for sectioning | Diatome | DUJ3530 | |
| Silicon wafer (pieces) | Si-Mat | Custom Made | Doping: P/Bor, orientation: <100>, thickness: 525 ± 25 µm, resistivity: 1-30 Ω-cm http://si-mat.com/silicon-wafers.html |
| ITO-coated coverslips | Balzers | Type Z | 22 × 22 × 0.17 mm https://www.opticsbalzers.com/de/produkte/deckglas-fenster/corrslide.html |
| Aluminium carrier | Custom Made | 76 × 26 mm | |
| Wafer forceps | Ideal-tek | 34A.SA | |
| Stubs forceps | Dumont | 0103-2E1/2-PO-1 | Dumoxel-H 2E 1/2 |
| Diamond scriber | Plano | T5448 | |
| Eyelash/very soft cat's hair | Selfmade | Alternative: Plano | |
| Brush | Selfmade | ||
| Pattex contact adhesive | Pattex | PCL3C | Kraftkleber Classic (the yellowish one) |
| Fixogum | Marabu | 290110001 | for fixing substrate to carrier |
| Adhesive tape | 3M | 851 | for fixing substrate to carrier |
| Isopropanol | Bernd Kraft | 07029.4000 | |
| Xylene | Carl Roth | 4436 | thinner for glue mixture |
| Rotihistol | Carl Roth | 6640 | alternative, limonene based thinner |
| Name | Company | Catalog Number | Comments |
| Software | |||
| Image processing | Open source | Fiji (http://fiji.sc/#download) | |
| Image acquisition | Zeiss | Atlas 5 AT (module for Zeiss SEM) | Alternative for automated image acquisition: WaferMapper: https://software.rc.fas.harvard.edu/lichtman/LGN/WaferMapper.html |
| Name | Company | Catalog Number | Comments |
| Staining | |||
| Propidiumiodide | Sigma-Aldrich | P4170 | Stock solution: 1.5 mM in 0.1 % sodium azide |
| Uranylacetate | Science Services | E22400 | |
| Lead(II) Nitrate | Merck | 107398 | |
| Tri Sodium Citrate Dihydrate | Merck | 106448 | |
| NaOH pellets | Merck | 106469 | |
| 1M NaOH solution | Bernd Kraft | 01030.3000 | |
| Glass petri dish | Duran | 23 755 56 | |
| Name | Company | Catalog Number | Comments |
| Mounting | |||
| Stubs | Plano | G301F | |
| Carbon pads | Plano | G3347 | |
| Copper tape | Plano | G3397 | double-sided adhesive, conductive |
| Silver paint | Plano | G3692 | Acheson Elektrodag 1415M |
| Name | Company | Catalog Number | Comments |
| Solutions/mixtures | |||
| Adhesive mixture for coating blocks | Pattex contact adhesive /xylene as thinner, ratio 1:3. (Alternative for xylene: Rotihistol) | ||
| Reynolds lead citrate | 50 mL: Dissolve 1.33 g of lead(II) nitrate in 10 mL of dH2O. Dissolve 1.76 g of tri-sodium citrate dihydrate in 10 ml dH2O. Mix both and add 1 M sodium hydroxide until the solution is clear. Fill up with dH2O to 50 mL. | ||
| Propidium iodide staining solution | Prepare 1:1500 dilution from stock in dH2O. Vortex for adequate mixing. | ||
| Aqueous uranyl acetate | Dissolve 3 % uranyl acetate in dH2O (mix thoroughly). |
Referenzen
- Peddie, C. J., Collinson, L. M. Exploring the third dimension: Volume electron microscopy comes of age. Micron. 61, 9-19 (2014).
- Wanner, A. A., Kirschmann, M. A., Genoud, C. Challenges of microtome-based serial block-face scanning electron microscopy in neuroscience. J Microsc. 259 (2), 137-142 (2015).
- Kizilyaprak, C., Daraspe, J., Humbel, B. Focused ion beam scanning electron microscopy in biology. J Microsc. 254 (3), 109-114 (2014).
- Wacker, I., Schröder, R. R. Array tomography. J Microsc. 252 (2), 93-99 (2013).
- Tapia, J. C., Kasthuri, N., Hayworth, K. J., Schalek, R., Lichtman, J. W., Smith, S. J., Buchanan, J. High-contrast en bloc staining of neuronal tissue for field emission scanning electron microscopy. Nat protoc. 7 (2), 193-206 (2012).
- Lucas, M. S., Günthert, M., Gasser, P., Lucas, F., Wepf, R. Bridging Microscopes: 3D Correlative Light and Scanning Electron Microscopy of Complex Biological Structures. Methods Cell Biol. 111, 325-356 (2012).
- De Boer, P., Hoogenboom, J. P., Giepmans, B. N. Correlated light and electron microscopy: ultrasructure lights up!. Nat Methods. 12 (6), 503-513 (2015).
- Verkade, P., Müller-Reichert, T. . Methods in Cell Biology Correlative Light and Electron Microscopy III. 140, 1-352 (2017).
- Gibson, K. H., Vorkel, D., Meissner, J., Verbavatz, J. -. M. Fluorescing the Electron: Strategies in Correlative Experimental Design. Methods Cell Biol. 124, 23-54 (2014).
- Wacker, I., Schröder, R. R., Schroeder, J. A. Pathology goes 3D: Exploring the potential of array tomography versus FIB nanotomography for a CADASIL sample. Ultrastruct Pathol. 41 (1), 114-115 (2017).
- Wacker, I., Spomer, W., Hofmann, A., Thaler, M., Hillmer, S., Gengenbach, U., Schröder, R. R. Hierarchical imaging: a new concept for targeted imaging of large volumes from cells to tissues. BMC Cell Biol. 17 (1), 38 (2016).
- Hayworth, K. J., Morgan, J. L., Schalek, R., Berger, D. R., Hildebrand, D. G. C., Lichtman, J. W. Imaging ATUM ultrathin section libraries with WaferMapper: a multi-scale approach to EM reconstruction of neural circuits. Front Neural Circuits. 8, (2014).
- Micheva, K. D., O'Rourke, N., Busse, B., Smith, S. J. Array Tomography: Production of Arrays. Cold Spring Harb Protoc. 11, 1267-1269 (2010).
- Fahrenbach, W. H. Continuous serial thin sectioning for electron microscopy. J. Elec. Microsc. Tech. 1 (4), 387-398 (1984).
- Harris, K. M., et al. Uniform serial sectioning for transmission electron microscopy. J Neurosci. 26 (47), 12101-12103 (2006).
- Schindelin, J., Arganda-Carreras, I., Frise, E., Kaynig, V., Longair, M., Pietzsch, T., Preibisch, S., Rueden, C., Saalfeld, S., Schmid, B., Tinevez, J. -. Y., White, D. J., Hartenstein, V., Eliceiri, K., Tomancak, P., Cardona, A. Fiji: an open-source platform for biological-image analysis. Nat Methods. 9 (7), 676-682 (2012).
- Cardona, A., Saalfeld, S., Schindelin, J., Arganda-Carreras, I., Preibisch, S., Longair, M., Tomancak, P., Hartenstein, V., Douglas, R. J. TrakEM2 software for neural circuit reconstruction. PLoS ONE. 7 (6), e38011 (2012).
- Micheva, K. D., Smith, S. J. Array Tomography: A new tool for imaging the molecular architecture and ultrastructure of neural circuits. Neuron. 55 (1), 25-36 (2007).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten