Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Funktionelle Charakterisierung von Carboxylesterases in Insektizid gegen Stubenfliegen, Musca Domestica
In diesem Artikel
Zusammenfassung
Hier präsentieren wir Ihnen ein Protokoll zur Produktion Haus fliegen Carboxylesterase Proteine in Vitro mit einem Baculovirus vermittelte Insekt Ausdruck Zellsystem und später funktionell charakterisieren ihre Rolle bei der Metabolisierung Permethrin, dadurch konferieren pyrethroid Widerstand von zellbasierten MTT-Assay und in-vitro- metabolische Studien.
Zusammenfassung
Carboxylesterase vermittelte Stoffwechsel wird gedacht, um in Insektizid Widerstand bei verschiedenen Insekten eine wichtige Rolle spielen. Mehrere Carboxylesterase Gene wurden bis in die resistenten Haus fliegen Belastung geregelt gefunden, während ihre Rolle bei der Übertragung der Insektizid Widerstand blieb um erkundet zu werden. Hier haben wir ein Protokoll für die funktionelle Charakterisierung von Carboxylesterases entwickelt. Drei Beispiel-Experimenten werden dargestellt: (1) Ausdruck und Isolation von Carboxylesterase Proteinen durch ein Insekt Baculovirus-vermittelten Zellsystem Ausdruck Spodoptera Frugiperda (Sf9); (2) eine zellbasierte MTT (3-[4, 5-Dimethykthiazol-2-Yl] -2, 5-Diphenyltetrazolium-Bromid) Zytotoxizität Assay, Messen Sie die Toleranz von Insektenzellen gegenüber verschiedenen Permethrin Behandlungen; und (3) in-vitro- metabolische Studien, die metabolische Funktionen des Carboxylesterases in Richtung Permethrin zu erkunden. Die Carboxylesterase gen, die MdαE7 aus einem beständigen Haus geklont wurde Belastung ALHF fliegen und verwendet, um eine rekombinante Baculovirus für Sf9 Zellen Infektion zu konstruieren. Die Zelle künstliche gegen verschiedene Permethrin Behandlungen wurden mit dem MTT-Assay gemessen. Die verbesserte Zell Toleranz der Versuchsgruppe (MdαE7-rekombinante Baculovirus infizierte Zellen) im Vergleich zu den Kontrollgruppen (CAT-rekombinante Baculovirus infiziert und GFP-rekombinante Baculovirus infizierte Zellen), permethrin in metabolisierenden Insektizide, dadurch Schutz der Zellen vor chemischen Schäden vorgeschlagenen Behandlungen die Funktionen des MdαE7. Darüber hinaus wurden Carboxylesterase Proteine in Sf9 Insektenzellen ausgedrückt und isoliert, um eine in-vitro- metabolische Studie durchzuführen. Unsere Ergebnisse zeigten eine signifikante in Vitro stoffwechseleffizienz des MdαE7 in Richtung Permethrin, direkt auf die Einbeziehung der Carboxylesterases in metabolisierenden Insektizide und verleiht somit Insektizid Widerstand im Haus fliegt.
Einleitung
Insektizid-Widerstand ist derzeit ein wichtiges Thema der Haus Fliegenbekämpfung weltweit1,2. Bemühungen um den Mechanismus der Insektizid Widerstand bestimmen erleichtert dieses Problem besseren zu verstehen und somit für neue Strategien, um effektiv zu verhindern oder zu minimieren die Ausbreitung der Widerstand Entwicklung3. Carboxylesterases, haben eine Menge Aufmerksamkeit für ihre Rollen in Sequestrierung und metabolisierenden Insektizide in verschiedenen Insekten4,5,6als eines der wichtigsten Entgiftung Enzyme angezogen. Unsere früheren Studie identifizierte mehrere Carboxylesterases in Stubenfliegen und ihren Ausdruck Ebenen wurden nicht nur in der resistenten Stamm ALHF konstitutiv geregelt sondern können auch induzierte zu höheren Ebenen in Reaktion auf Permethrin Behandlungen7 . Allerdings müssen die funktionale Charakterisierungen dieser Carboxylesterase Gene in metabolisierenden Insektizide noch erkundet werden.
Seit dem ersten Bericht in den frühen 1980er Jahren8wurde eine Baculovirus-vermittelten Fremdgen Expressionssystems weithin wegen seiner hohen Protein-Produktions-Leistungsfähigkeit und eukaryotic Protein Verarbeitung Funktionen9eingesetzt. Diese binären System besteht aus zwei wesentlichen Elementen: die konstruierte rekombinante Baculovirus Bereitstellung fremder Gene in den Wirtszellen und die groß angelegte Expression interessiert Proteine von Zellen durch rekombinante Baculovirus infiziert. In den letzten Jahrzehnten das Baculovirus vermittelte Zelle Ausdruck System ist weit verbreitet, Tausende von rekombinanten Proteinen, Membrane-springen Proteine im Insekt von zytosolischen Enzyme bis zu produzieren und Säugetier-Zellen10. Unsere früheren Studie äußerte erfolgreich mehrere CYP450 Enzymen in Sf9 Insektenzellen mit diesem System11. In dieser Studie wir konstruiert eine Carboxylesterase-rekombinante Baculovirus Sf9 Insektenzellen infizieren, erkundet die Zelle Toleranz gegenüber unterschiedlichen Permethrin Behandlungen und groß angelegte ausgedrückt Carboxylesterase Proteine in Vitro für funktionale Exploration. Anstatt mehrere Carboxylesterase isozymen Mischungen von Insekten Homogenates wie von früheren Studien12,13angenommen, dieses Insekt Zelle Baculovirus-vermittelten Ausdruck System ermöglicht die Konkretisierung und Isolierung von gezielten Proteine für bessere Charakterisierung ihrer biochemischen und strukturellen Eigenschaften.
Tetrazoliumsalz Salz basierende Assays (MTT) ist ein Hochdurchsatz-kolorimetrischen Verfahren entwickelt und optimiert, um die Zellviabilität messen. Dieser Test basiert auf den Mechanismus, die nur lebende Zellen in der Lage metabolisierenden gelb gefärbten MTT Reagenz zu einem dunklen lila farbigen Formazan Niederschlag, farbmetrisch analysiert werden kann sind, nachdem in organischen Lösungsmitteln14aufgelöst, 15. Einige genauere aber zeitaufwändige Methoden, z. B. Trypan blau Ausgrenzung und das Thymidin Titration Assay16,17, wurden in den letzten Jahren entwickelt. Die zellbasierte MTT-Assay wird jedoch derzeit noch als die schnellste und einfach zu bedienen-Methode anerkannt, Zellviabilität schnell erkennen. Hier verwenden wir die MTT-Assay, um Zell Toleranz gegen insektizidbehandlungen zu erkunden. Die verbesserte Toleranz der Zellen wenn mit rekombinanten Baculovirus Carboxylesterase stark infiziert unterstützt die metabolische Rollen des Carboxylesterases gegen Insektizide, die wiederum ihre Beteiligung in Insektizid Widerstand schlägt.
Darüber hinaus wurde ein in-vitro- metabolische Test auch in dieser Studie durchgeführt. Im Vergleich mit allgemeinen Carboxylesterase Tests, mit denen gemeinsame Substrate wie α-Napthyl-Acetat (α-NA) und β-Naphthyl Acetat (β-NA) hydrolytische Tätigkeit der Carboxylesterases widerspiegeln, ist die in-vitro- metabolische Studie eine genaue Weg angesehen Aktivitäten der Carboxylesterases in Richtung Insektizide18direkt zu messen. Diese Methode wurde bereits erfolgreich in verschiedene Insekten eingesetzt, mehrere Cytochrom P450s in Verbindung mit Insektizid Widerstand11,19,20zu charakterisieren. Allerdings hat diese Methode noch nicht in Carboxylesterase Studien angewendet. Mit der Verfügbarkeit von Carboxylesterase Proteine produziert durch Baculovirus-vermittelten Expressionssystems kann eine in-vitro- metabolische Untersuchung der Carboxylesterases in Richtung Permethrin, führen wir die weitere starke Beweise für die Beteiligung bieten können der Carboxylesterases in Übertragung Pyrethroid-Resistenz im Haus fliegt.
Protokoll
1. Ausdruck und Isolation von Zielproteinen mit einem Insekt Zelle Baculovirus-vermittelten Expressionssystem
- Direktional Klonen Sie PCR-Produkte von Zielproteinen von Stubenfliegen Blunt beendet.
- Design-PCR Primer grün fluoreszierenden Proteins (GFP) und das Haus fliegen MdαE7 gen basierend auf ihre Sequenzen und die besonderen Anforderungen des gewählten Vektors (Tabelle 1).
- Verwenden Sie ein Lektorat, thermostabile DNA-Polymerase und Primer aus Schritt 1.1.1 150 µL-PCR-Reaktion (30 µL Puffer Reaktion, 3 µL 10 mM dNTPs, 1,5 µL DNA-Polymerase, 6 µL Haus fliegen Schablone DNA, 7,5 µL vorwärts Grundierung durchführen 7,5 µL des rückwärts-Primer, mit Wasser zu einem Endvolumen 150 µL). Erhitzen die PCR-Reaktion auf 98 ° C für 30 s, gefolgt von 35 Zyklen von 98 ° C für 10 s, 53 ° C für 30 s und 72 ° C für 60 s, und dann eine letzte Verlängerung bei 72 ° C für 2 min).
- Führen Sie eine 1 % Agarose-Gel mit 150 µL des PCR-Produktes.
- Die Ziel DNA-Fragmente aus dem Agarose-mit einem scharfen, sauberen Skalpell Gel Verbrauchsteuern: 1317 bp für MdαE7 und 858 bp für GFP. Reinigen Sie die DNA mit im Handel erhältlichen Gel Extraktion Kit nach Herstellerangaben-Protokolls (Table of Materials). Die gereinigte DNA in 15 µL destilliertes Wasser auflösen.
- Führen Sie eine 1 % Agarose-Gel mit 1 µL gereinigte DNA-Integrität zu überprüfen. Verwenden Sie ein anderes 1 µL gereinigte DNA-Konzentrationen mit dem Spektralphotometer messen.
- Ein Eintrag Plasmid für Zielproteine zu konstruieren
- Richten Sie das Klonen Reaktion. Mix frisch gereinigte DNA-Produkte aus Schritt 1.1.3 und handelsüblichen Eintrag Vektoren mit AttL-Websites (Table of Materials) in einem Molverhältnis von 1:1 (0,5-2 µL frische PCR-Produktes bei 70-200 ng/µL Konzentrationen: 0,5 µL Vektor). Dann fügen Sie 1 µL einer handelsüblichen Salzlösung (1,2 M NaCl2 und 0,06 M MgCl2) und fügen Sie Wasser zu einem Endvolumen von 6 µL. vorsichtig mischen und in 1 h bei Raumtemperatur inkubieren.
- Übertragen 4 µL des Klonens Reaktionsprodukte in Schritt 1.2.1 in 50 µL chemisch kompetenten E. Coli Zellen. Inkubation auf Eis für 30 min. Hitzeschock Zellen für 30 s im Wasserbad 42 ° C ohne schütteln.
- Setzen Sie den Schlauch wieder in Eis für eine weitere 2 min. Fügen Sie 250 µL Raumtemperatur S.O.C. Medium (2 % Tryptone; 0,5 % Hefeextrakt; 10 mM NaCl; 2,5 mM KCl, 10 mM MgCl2; 10 mM MgSO4; 20 mM Glukose). Inkubation bei 37 ° C für 1 h mit sanft schütteln.
- Verbreiten von 50-200 µL der Bakterienkultur im Schritt 1.2.3 auf den selektiven LB-Platten (1 g Tryptone; 1 g NaCl; 0,5 g Hefeextrakt 1,5 g Agar in 100 mL destilliertem Wasser gelöst. Autoklaven und 0,1 % von 50 mg/mL Kanamycin hinzufügen). Brüten Sie LB-Platten für 16 h bei 37 ° C für das koloniewachstum.
- Wählen Sie 5 bis 10 Kolonien und wieder auszusetzen Sie einzeln in 5 µL destilliertes Wasser.
- Führen Sie PCR durch Zugabe von 5 µL Reaktion Puffer, 0,5 µL 10 mM dNTPs, 0,25 µL DNA-Polymerase, 1 µL der suspendierten Kolonien, 1,25 µL 10 µM M13 vorwärts Grundierung, 1,25 µL 10 µM-rückwärts-Primer des Zielgens und Wasser zu Endvolumen von 25 µL. Erhitzen der PCR Reaktion auf 98 ° C für 30 s, gefolgt von 35 Zyklen von 98° C für 10 s, 53 ° C für 30 s und 72 ° C für 60 s, und dann eine letzte Verlängerung bei 72 ° C für 2 min (Tabelle 1).
- Re Kultur 3 µL der suspendierten Kolonien aus Schritt 1.2.5 in TB Medien (100 mL Phosphatpuffer mit 0,17 M KH2PO4 und 0,72 M K2HPO4 mit 450 mL Basis Brühe 6 g Tryptone, 12 g Hefe und 2 mL-haltigem medium Glycerin, sterilisiert, mit 0,1 % 50 mg/mL Kanamycin) für 16 h extrahieren hochreine Plasmid-DNA nach Protokoll des Herstellers.
- Einsatz 200 ng/µL hochreine Plasmid aus Schritt 1.2.7 für kommerzielle Sanger Sequenzierung mit vorwärts- und Primern M13. Überprüfen Sie die korrekte Einführung des Zielgens in Vektor basierend auf der Reihenfolge Karte vom Hersteller bereitgestellt.
- Speichern Sie die Sequenz verifiziert hochreine Plasmid DNA-Proben von MdαE7 und GFP bei-20 ° C.
Hinweis: Dies sind die Eintrag Plasmid DNA-MdαE7 und GLP.
- Erstellen eines rekombinanten Baculovirus Durchführung Lambda Rekombination (LR) Reaktion.
- Mischen Sie jeweils 1 µL von 300 ng/µL Eintrag Plasmid DNA der GFP, MdαE7 aus Schritt 1.2.7 oder Eintrag Plasmid DNA von Chloramphenicol-Acetyltransferase-gen (CAT) durch die Zelle Transfektion Kit mit 5 µL der im Handel erhältlichen (mit AttL Seiten) zur Verfügung gestellt C-Begriff lineare DNA (mit AttR Seiten) in 250 µL PCR-Röhrchen. Fügen Sie zu einem Gesamtvolumen von 8 µL TE-Puffer.
Hinweis: Die Reaktionen der GFP und Katze dienten als Kontrollgruppe. - Fügen Sie 2 µL der handelsüblichen Lambda Rekombination (LR) Enzym-Mix (Table of Materials) in jeder Mischung aus Schritt 1.3.1, die LR-Reaktion zu katalysieren. Vorsichtig mischen Sie und inkubieren Sie bei 25 ° C über Nacht (≈16 h).
Hinweis: Die LR-Reaktion erleichtert die Rekombination eine AttL-haltigen Eintrag Plasmid DNA mit einem AttR-haltigen C-Begriff lineare DNA, eine AttB-haltigen mit dem Ausdruck ihrer Virus zu generieren. Dieser Schritt liefert der rekombinanten Baculovirus für jedes Zielprotein. - Verdünnen Sie 1 µL LR Reaktionsprodukte aus Schritt 1.3.2 20 X. Führen Sie PCR unter Verwendung eines Polyhedrin vorwärts Grundierung und V5-rückwärts-Primer (Tabelle 1). Verwenden Sie 5 µL des PCR-Produkte auf einem 1 % Agarosegel um die Qualität zu überprüfen. Abbildung 1 zeigt ein Beispiel für die LR Reaktionsprodukt von MdαE7 gen.
- Mischen Sie jeweils 1 µL von 300 ng/µL Eintrag Plasmid DNA der GFP, MdαE7 aus Schritt 1.2.7 oder Eintrag Plasmid DNA von Chloramphenicol-Acetyltransferase-gen (CAT) durch die Zelle Transfektion Kit mit 5 µL der im Handel erhältlichen (mit AttL Seiten) zur Verfügung gestellt C-Begriff lineare DNA (mit AttR Seiten) in 250 µL PCR-Röhrchen. Fügen Sie zu einem Gesamtvolumen von 8 µL TE-Puffer.
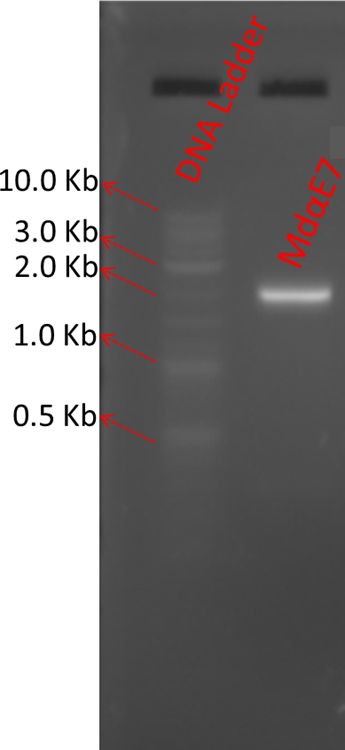
Abbildung 1: Beispiel-Ergebnisse der MdαE7 LR Reaktion durch PCR-Analyse. Verdünnen Sie 2 µL der LR Reaktion 200-fach und verwenden Sie 2 µL der Verdünnung mit Polyhedrin forward Primer und V5-rückwärts-Primer, um eine 25 µL PCR durchzuführen. Verwenden Sie 5 µL des PCR-Produkte um 1 % Agarosegel überprüfen die Qualität der LR Reaktion auszuführen. ein
- Transfizieren Sf9 Insektenzellen
- Kultur Sf9 Insektenzellen mit 5 mL der vollständige Zelle Wachstum mittlerer (serumfreien Medium mit 10 % fetalen bovine Serum (FBS)) T25 behandelt Fläschchen bei 27 ° c.
- Entfernen Sie den überstand und bündig unten angebracht Zellen mit 3 mL komplette Frischzellen Wachstumsmedium. Übertragen Sie 0,5 mL neu suspendierten Zellen in einem neuen T25 behandelt Kolben. 4,5 mL komplette Wachstumsmedium hinzugeben. 3-4 Tage bis zur nächsten Übertragung bei 27 ° C inkubieren.
- Samen 2 mL der Log-Phase Wachstum Insekt Sf9 Zellen Kultur (≈3.0-5.0 × 106 Zellen) gleichmäßig auf die Zellkultur gut. Können Sie Zellen für mindestens 3 h bei Raumtemperatur in der Haube zu befestigen.
- Überprüfen Sie die Zellhaftung durch die Beobachtung mit einem inversen Phase Mikroskop bei 250 X. Das Zellkulturmedium entfernen und ersetzen mit 2 mL Graces Insekt Medium.
- Transfektion Mischung A Lösung (8 µL der im Handel erhältlichen Zelle Infektion Reagenz (Table of Materials) mit 100 µL Graces Insekt Medium) und Transfektion Mischung B Lösung von jedem Zielgen (9 µL LR Reaktionsprodukte aus Schritt 1.3 vorbereiten mit 100 µL unsupplemented Grace Insekt Medium) in 1,5 mL Zentrifugieren Röhren, beziehungsweise.
- Mischen Sie vorsichtig Transfektion Mischung A und B zusammen durch Tippen auf die Rohre. Inkubation bei Raumtemperatur für 35 min in der Kapuze.
- Fügen Sie die Mischung gleichmäßig aus Schritt 1.4.6 tropfenweise auf die gesetzten Zellen aus Schritt 1.4.4. Brunnen mit Bändern verschließen und über Nacht bei 27 ° C inkubieren.
- 2 mL Graces Insekt Medium mit 2 mL komplette Wachstumsmedium zu ersetzen. Fügen Sie 100 µM Ganciclovir in jedem Bohrloch negativ gegen nicht-rekombinante Baculovirus auswählen. Versiegeln Sie Brunnen mit Klebeband und inkubieren Sie bei 27 ° C für 72 h.
- Sammeln Sie 72 h Post Infektion Zellkulturmedium aus jedem Brunnen und übertragen Sie auf 1,5 mL Zentrifuge Röhren. Zentrifugieren Sie bei 1.500 x g für 5 min bei 4 ° C, Zellen oder großen Schmutz zu entfernen.
- Übertragen Sie den überstand in neuen 1,5 mL Zentrifuge Röhren. Bei 4 ° C mit vor Licht geschützt aufbewahren.
Hinweis: Dies sind P1 Virus auf lagerlösungen für jedes Zielgen. - Verstärken Sie den niedrigen Titer P1 virale bestand (1 × 105-1 × 106 Pfu/mL), eine hohe Titer P2 virale Aktie (5 × 107-1 × 108 Pfu/mL).
- Samen 2 mL der Log-Phase Wachstum Insekt Sf9 Zellen Kultur (≈3.0-5.0 × 106 Zellen) gleichmäßig auf die Zellkultur gut. Können Sie Zellen für mindestens 3 h bei Raumtemperatur in der Haube zu befestigen.
- Impfen Sie 5 µL P1 Virus Aktien erwarb Schritt 1.4.10 in der Zelle bzw. auch der Schritt 1.4.12, ausgesät. Fügen Sie 100 µM Ganciclovir in jede Vertiefung. Versiegeln Sie Brunnen zu und inkubieren Sie bei 27 ° C für 72 h.
- P2-Virus Stammlösungen der 72 h Post Infektion zu sammeln. Bei 4 ° C mit vor Licht geschützt aufbewahren.
Hinweis: Dies sind P2 Virus auf lagerlösungen für jedes Zielgen. - (Optional) Wiederholen Sie die Schritte 1.4.12-1.4.14 mit 5 µL P2 Virus Lager P3 Virus Stammlösungen zu sammeln.
Hinweis: Dies sind P3 Virus auf lagerlösungen für jedes Zielgen. - Speichern Sie alle konstruierten Baculovirus bei 4 ° C mit Lichtschutz.
Hinweis: Die GFP und CAT-gen wurden diente als Kontrollgruppe. Abbildung 2 zeigten die Zellen wenn in verschiedenen Verstärkung Phasen von GFP-rekombinante Baculovirus infiziert.
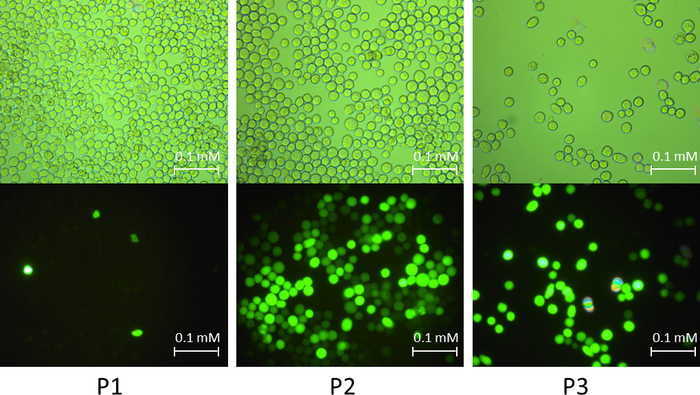
Abbildung 2: Beispiel für infizierte Anzeichen für Sf9 Zellen in verschiedenen Baculovirus Verstärkung Phasen. Die Abbildung zeigt GFP-rekombinante Baculovirus Zellen unter Tageslicht und fluoreszierende Licht. In der Phase der P1-Infektion ist das Verhältnis der Infektion gering. In der Phase der Infektion der P2 war das Verhältnis der Infektion deutlich verbessert. In der Phase der P3-Infektion fast alle Zellen zeigten Symptome der Loslösung von Kultur Zellplatte, erhöht der Zelle Durchmesser sowie die Einstellung des Zellwachstums. Bitte klicken Sie hier für eine größere Version dieser Figur.
- Groß angelegte Ausdruck der Zielproteine in Sf9 Insektenzellen
- Kultur 10 mL Log Phase Wachstum Sf9 Insektenzellen mit vollständigen Wachstumsmedium in T25 unbehandelten Flaschen.
- Fügen Sie 200 µL P2 Virus-Stammlösung (erhältlich im Schritt 1.4.14) um Zellkulturen im Schritt 1.5.1 für 72 h zu infizieren.
- 72 h Post Infektion Zellkulturmedium zu sammeln. Zentrifuge bei 1.500 x g für 5 min bei 4 ° C.
- Überstand und waschen Pellets zweimal mit 1 mL 0,1 M-PBS-Puffer (pH 7,5) zu verwerfen.
- Zelle Pellets mit 1 mL Insekt Zelle Proteingewinnung & Lyse Puffer (Table of Materials) vollständig zu lösen. Fügen Sie 10 % Glycerin in Zelle Lysis. Sofort bei-80 ° c Lagern
- Wiederholen Sie die Schritte 1.5.1-1.5.5 mit P2 Virus Stammlösung CAT Proteine als die Kontrollgruppe dienen.
Hinweis: Wiederholen Sie Schritt 1.5 in dreifacher Ausführung für unterschiedliche Protein Präparate.
(2) eine zellbasierte MTT Zytotoxizität Assay
- Kultur 5 mL Log Phase Wachstum (1,5 bis 2,5 × 106 Zellen/mL) Sf9 Insektenzellen mit vollständigen Wachstumsmedium in T25 unbehandelten Flaschen.
- Fügen Sie 25 µL P1 Virus-Stammlösung (erhältlich im Schritt 1.4.14) um Zellkulturen in Schritt 2.1 für 48 h mit sanft schütteln bei 27 ° c zu infizieren.
- Bereiten Sie 100 mM standard Stammlösungen Permethrin in Acetonitril. Verwenden Sie Acetonitril, bis 6,25 mM, 50 mM, 25 mM und 12,5 mM durch Zugabe von 500 µL, 250 µL, 125 µL und 62,5 µL Permethrin Stammlösung in einem Gesamtvolumen von 1.000 µL bzw. zu verdünnen.
- Samen Sie gleichmäßig 200 µL des infizierten Zellkulturen aus Schritt 2.2 mit 300 µL kompletten Wachstumsmedium in 24-Well-Platte bei einer Dichte von 2 × 105 Zellen/mL ergänzt.
- Fügen Sie 4 µL Permethrin Standardlösungen (6,25 mM, 12,5 mM, 25 mM und 50 mM) in jede Vertiefung 400 µM und 100 µM, eine Endkonzentration 50 µM und 200 µM bzw. zu machen. Fügen Sie nur 4 µL Acetonitril in Vertiefungen für Zelle Lebensfähigkeit Berechnungen (Abbildung 3).
- Versiegeln Sie Platten mit Bändern zu und inkubieren Sie bei 27 ° C für 48 h mit Lichtschutz.
- 5 mg/mL MTT (Thiazolyl blau Tetrazolium Bromid) Reagenz gelöst in Puffer vorzubereiten (2,0 g NaCl, KCl 0,05 g, 0,36 g NaH2PO4∙2H2O, 0,28 g NaH2PO4, 0,05 g KH2PO4 in 200 mL aufgelöst destilliertem Wasser, pH 7,5).
- 2.6 Schritt nach 48 h Inkubation entfernen Sie Zellkulturmedium ohne Berührung der unteren fraktionslosen Zellschicht. Fügen Sie 200 µL MTT Reagenz in Schritt 2.7 in jede Vertiefung. Inkubation bei 37 ° C für 4 h bis dunkel violette Formazan Ausscheidungen in jede Vertiefung (Abbildung 3) gebildet.
- Inkubation bei 37 ° C für 4 h bis dunkel violette Formazan Form in jedes gut ausfällt. Fügen Sie 500 µL von DMSO, Ausscheidungen (Abbildung 3) vollständig zu lösen.
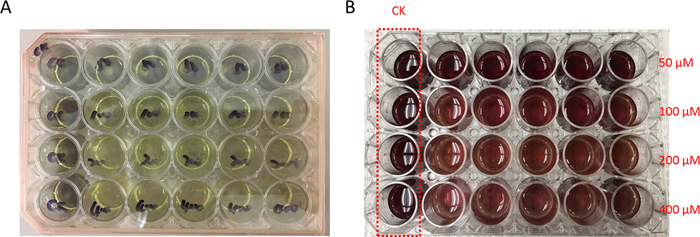
Abbildung 3: Beispiel für MTT Ergebnisse. Nach 48 h post Infektion mit Virus-Stammlösung P1 CAT rekombinante Baculovirus Lösung gleichmäßig 500 µL Samenzellen CAT-gen in einer 24-Well Zellplatte Kultur zum Ausdruck zu bringen. Jeweils fügen Sie 4 µL Permethrin an eine andere Dosis (6,25 mM, 12,5 mM, 25 mM und 50 mM hinzu) in jeder Zeile die Endkonzentration an 400 µM und 100 µM, 50 µM und 200 µM vornehmen. Die Steuerelement-Reihe wurde mit Acetonitril nur behandelt und gekennzeichnet als CK in der Platte. (A) nach 48 h Inkubation bei 27 ° C verwerfen das Zellkulturmedium auf der oberen Ebene und ersetzen mit 200 µL gelb gefärbten MTT Reagenzien. Dann bei 37 ° C für 48 h dunkel lila farbigen Reduzierung Form ausfällt in jedes gut darauf hinweist, dass die Überlebens-Zellen in der Lage, die gelbe Farbe metabolisierenden inkubieren Sie MTT Reagenzien in den dunklen lila farbigen Niederschlag. (B) 500 µL DMSO Lösungsmittel wurde hinzugefügt, in jede Vertiefung den gebildeten Niederschlag aufzulösen. Der Extinktion Wert jedes gut war röntgenologisch Spektralphotometer bei 540 nm. Wie Permethrin Konzentrationen zu erhöhen, wird die Farbe allmählich ändern von dunkelrot, Hellrot, was darauf hindeutet, dass die Zelle künstliche schrittweise verringert wurden. Bitte klicken Sie hier für eine größere Version dieser Figur.
- Übertragen Sie 200 µL des gelösten Lösung in 96-Well-Platte. Messwerte der Extinktion bei 540 nm mit einer Mikrotestplatte Reader (Table of Materials).
- Berechnen Sie Zellviabilität durch Vergleichen der Werte der Extinktion von Permethrin behandelt Zellen mit denen der nur Acetonitril behandelt Zellen.
- Wiederholen Sie für die Kontrollgruppe alle Schritte mit P2-Virus-Stammlösung von Chloramphenicol-Acetyltransferase (CAT) rekombinante Baculovirus erwarb Schritt 1.4.
- Wiederholen Sie 4 Mal für verschiedene Virus-Vorbereitungen.
3. in-vitro- metabolische Assay
- Bereiten Sie 100 mM standard Stammlösungen Permethrin in Acetonitril. Verwenden Sie Acetonitril, um 50 mM, 25 mM, 12,5 mM und 6,25 mM zu verdünnen. Erkennen Sie die entsprechenden Peakfläche unter jede Konzentration mittels HPLC. Erstellen und Berechnen der Standardkurve Permethrin anhand der Peakfläche mit verschiedenen Permethrin Konzentrationen.
- Messen der proteinkonzentrationen in Schritt 1.5 mit Bradford Methoden21.
- Bereiten Sie 700 µL Stoffwechselreaktion mit 40 µM Permethrin Standard und 1 mg von Proteinen, die in Schritt 1.5 in 0,2 M Tris-HCl-Puffer (pH 7,4) aufgelöst. Inkubation bei 30 ° C für 2 h mit sanft schütteln. Vor Licht schützen.
- Die Reaktion durch Zugabe von 700 µL eiskaltem Acetonitril zu stillen. Inkubation bei 30 ° c für weitere 30 min. mit sanft schütteln. Vor Licht schützen.
- Zentrifugieren der Reaktionsmischung bei 16.000 x g für 2 min bei Raumtemperatur. Sammeln Sie die überstand und Filter durch einen 0,45 µm-Membran. Übertragen Sie die Filtration in ultraclean braunen Glasfläschchen für HPLC-Analytik.
- Ausführen der HPLC unter optimalen chromatographischen Bedingungen (Mobile Phase A: 90 % Acetonitril und 10 % Wasser; Die Mobile Phase B: 5 % Acetonitril angepasst auf pH 2,3 mit 85 % Phosphorsäure). Farbverlauf eluieren mit einer Durchflussmenge von 1 mL/min und Maßnahme bei einer Wellenlänge von 232 nm.
- Berechnen Sie die Erschöpfung Prozentsatz der Permethrin durch den Vergleich mit Reaktionen ohne Protein Probe hinzugefügt.
- Verwenden Sie CAT-Protein in Schritt 1.5.6 erhaltenen als Kontrolle dienen.
- Wiederholen Sie die Schritte mit verschiedenen Protein-Präparaten im Schritt 1.5.7.
Ergebnisse
Die Zellviabilität gegenüber verschiedenen Permethrin Behandlungen (MTT-Assay)
Die Zytotoxizität von Permethrin wurde im MdαE7-rekombinante Baculovirus infiziert Sf9 (Versuchsgruppe) und CAT-rekombinante Baculovirus (bereitgestellt von Baculovirus infiziert Kit) infizierte Zellen (Kontrollgruppe) untersucht. Die verstärkte Zelle Toleranzen zu Permethrin in MdαE7 mit dem Ausdruck Zellen stark unterstützen die metabolische Rol...
Diskussion
In den letzten Jahrzehnten haben heterologen Expressionssysteme verbreitet zu äußern und große Mengen von Proteinen, so dass Biochemische und funktionelle Bestimmung und Charakterisierung der Enzyme in Vitrozu isolieren. Bisher wurden mehrere verschiedene Modellsysteme wie Escherichia coli, Pichia Pastoris, Sacccharomyces Cerevisiaeund Spodoptera Frugiperda angepasst für rekombinante Proteinexpression und die Wahl des der in-vitro- System ist entscheidend für gro...
Materialien
| Name | Company | Catalog Number | Comments |
| Q5 High-Fidelity DNA Polymerase | New England Biolabs inc. | M0491L | |
| QIAquick Gel Extraction Kit | QIAGEN | 28704 | |
| pENTR/D-TOPO Cloning Kit, with One Shot TOP10 Chemically Competent E. coli | Invitrogen by life technology | K240020 | S.O.C medium and universal M13 sequence primers were included in this kit. |
| PureLink HiPure Plasmid Miniprep Kit | Invitrogen by life technology | K210002 | |
| Gateway LR Clonase II Enzyme mix for BaculoDirectTM Kits | Invitrogen by life technology | 11791-023 | |
| BaculoDirect C-Term Linear DNA Transfection Kit | Invitrogen by life technology | 12562-019 | Cellfectin transfection reagent and ganciclovir were included in this kit |
| pENTR-CAT plasmid | Invitrogen by life technology | Included in BaculoDirect C-Term Linear DNA Transfection Kit, concentration: 0.5 ug/uL | |
| Heat inactivated Fetal Bovine Serum, Certified | Gibco by Life Technologies | 10082-139 | |
| Sf9 cells in Sf-900 III SFM | Gibco by Life Technologies | 12659017 | |
| Insect Cell-PE LB Insect Cell Protein Extraction & Lysis Buffer | G Biosciences by A Geno Technology Inc | 786-411 | |
| Sf-900 III SFM (1×) Serum Free Medium Complete | Gibco by Life Technologies | 12658-019 | |
| Grace's Insect Medium, unsupplemented | Gibco by Life Technologies | 11595030 | |
| Permethrin (isomers) analytical standard | SUPELCO by Solutions WithinTM | 442748 | |
| Methanol (analytical graded) | Sigma-Aldrich | 67-56-1 | |
| Acetonitrile (analytical graded) | Sigma-Aldrich | 75-05-8 | |
| GHP Acrodisc 25 mm Syringe Filters with 0.45 μm GHP Membrane (HPLC Certified) | Pall Life Sciences | 21890388 | |
| Alliance Waters 2695 HPLC System | Waters | ||
| T100 Thermal Cycle | Bio-Rad Laboratories Inc. | 1861096 | |
| Nanodrop 2000/2000c Spectrophotometers | ThermoFisher Scientific | ND2000CLAPTOP | |
| Cytation 5 Cell Imaging Multi-Mode Reader | BioTek |
Referenzen
- Scott, J. G., et al. Insecticide resistance in house flies from the United States: Resistance levels and frequency of pyrethroid resistance alleles. Pesticide Biochemistry and Physiology. 107 (3), 377-384 (2013).
- Li, M., et al. A whole transcriptomal linkage analysis of gene co-regulation in insecticide resistant house flies, Musca domestica. BMC Genomics. 14, 803 (2013).
- Liu, N. Insecticide resistance in mosquitoes: impact, mechanisms, and research directions. Annual Review of Entomology. 60, 537-559 (2015).
- Grigoraki, L., et al. Transcriptome profiling and genetic study reveal amplified carboxylesterase genes implicated in temephos resistance, in the Asian tiger mosquito Aedes albopictus. e0003771. 9, e0003771 (2015).
- Grigoraki, L., et al. Carboxylesterase gene amplifications associated with insecticide resistance in Aedes albopictus: Geographical distribution and evolutionary origin. PLOS Neglected Tropical Diseases. 11, e0005533 (2017).
- Wheelock, C., Shan, G., Ottea, J. Overview of carboxylesterases and their role in the metabolism of insecticides. Journal of Pesticide Science. 30, 75-83 (2005).
- Feng, X., Li, M., Liu, N. Carboxylesterase genes in pyrethroid resistant house flies, Musca domestica. Insect Biochemistry and Molecular Biology. 92, 30-39 (2018).
- Mosmann, T. Rapid colorimetric assay for cellular growth and survival: application to proliferation and cytotoxicity assays. J Immunol Methods. 65 (1-2), 55-63 (1983).
- Jarvis, D. L. Baculovirus-insect cell expression systems. Methods in Enzymology. 463, 191-222 (2009).
- Berger, I., Fitzgerald, D. J., Richmond, T. J. Baculovirus expression system for heterologous multiprotein complexes. Nature Biotechnology. 22 (12), 1583 (2004).
- Gong, Y., Li, T., Feng, Y., Liu, N. The function of two P450s, CYP9M10 and CYP6AA7, in the permethrin resistance of Culex quinquefasciatus. Scientific Reports. 7 (1), 587 (2017).
- Cao, C. W., Zhang, J., Gao, X. W., Liang, P., Guo, H. L. Overexpression of carboxylesterase gene associated with organophosphorous insecticide resistance in cotton aphids, Aphis gossypii (Glover). Pesticide Biochemistry and Physiology. 90 (3), 175-180 (2008).
- Zhang, L., Gao, X., Liang, P. Beta-cypermethrin resistance associated with high carboxylesterase activities in a strain of house fly, Musca domestica (Diptera: Muscidae). Pesticide Biochemistry and Physiology. 89, 65-72 (2007).
- Van Meerloo, J., Kaspers, G. J., Cloos, J. Cell sensitivity assays: the MTT assay. Cancer cell culture. , 237-245 (2011).
- Stockert, J. C., Blázquez-Castro, A., Cañete, M., Horobin, R. W., Villanueva, &. #. 1. 9. 3. ;. MTT assay for cell viability: Intracellular localization of the formazan product is in lipid droplets. Acta Histochemica. 114 (8), 785-796 (2012).
- Strober, W. Trypan blue exclusion test of cell viability. Current Protocols in Immunology. , (2001).
- Riss, T. L., Moravec, R. A., Niles, A. L., Duellman, S., Benink, H. A., Worzella, T. J., Minor, L. Cell viability assays. Assay Guidance Manual. , (2013).
- Wheelock, C. E., Shan, G., Ottea, J. Overview of carboxylesterases and their role in the metabolism of insecticides. Journal of Pesticide Science. 30 (2), 75-83 (2005).
- Li, X., Schuler, M. A., Berenbaum, M. R. Molecular mechanisms of metabolic resistance to synthetic and natural xenobiotics. Annual Review of Entomology. 52, 231-253 (2007).
- Nakamura, Y., et al. The in vitro metabolism of a pyrethroid insecticide, permethrin, and its hydrolysis products in rats. Toxicology. 235 (3), 176-184 (2007).
- Kruger, N. J. The Bradford method for protein quantitation. The protein protocols handbook. , 15-21 (2002).
- Macauley-Patrick, S., Fazenda, M. L., McNeil, B., Harvey, L. M. Heterologous protein production using the Pichia pastoris expression system. Yeast. 22 (4), 249-270 (2005).
- Berger, I., Fitzgerald, D. J., Richmond, T. J. Baculovirus expression system for heterologous multiprotein complexes. Nature Biotechnology. 22 (12), 1583 (2004).
- Terpe, K. Overview of bacterial expression systems for heterologous protein production: from molecular and biochemical fundamentals to commercial systems. Applied Microbiology and Biotechnology. 72 (2), 211 (2006).
- Bulter, T., et al. Functional expression of a fungal laccase in Saccharomyces cerevisiae by directed evolution. Applied Microbiology and Biotechnology. 69 (2), 987-995 (2003).
- Stepanenko, A. A., Dmitrenko, V. V. Pitfalls of the MTT assay: Direct and off-target effects of inhibitors can result in over/underestimation of cell viability. Gene. 574 (2), 193-203 (2015).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten