Method Article
Einsatz von rekombinanten Fusionsproteinen in eine fluoreszierende Protease-Assay-Plattform und ihre In-Gel-Renaturierung
In diesem Artikel
Zusammenfassung
Hier präsentieren wir Ihnen die Einzelheiten des Verfahrens einer neu entwickelten Protease Assays Plattform nutzen N-terminalen Hexahistidine/Maltose-bindendes Protein und fluoreszierende Protein fusioniert rekombinante Substrate an der Oberfläche der Nickel-Nitrilotriacetic befestigt saure magnetische Agarose Korne. Eine anschließende Gelanalyse der Assay-Proben durch Natrium-Dodecyl-Sulfat-Polyacrylamid-Gelelektrophorese getrennt wird ebenfalls dargestellt.
Zusammenfassung
Proteasen sind intensiv untersuchten Enzyme aufgrund ihrer wesentlichen Aufgaben in mehreren biologischer Signalwege von lebenden Organismen und Pathogenese; Sie sind daher wichtige Drogeziele. Wir haben eine magnetische-Agarose Bead-basierte Test-Plattform für die Untersuchung der proteolytischen Aktivität entwickelt, auf den Einsatz von rekombinanten Fusion Protein Substrate basiert. Um die Verwendung von diesem Testsystem zu zeigen, wird ein Protokoll am Beispiel des humanen Immundefizienz-Virustyp 1 (HIV-1) Protease vorgestellt. Die eingeführten Assay-Plattform genutzt werden effizient in die biochemische Charakterisierung der Proteasen, einschließlich der Enzym-Aktivität-Messungen in Mutagenese, kinetische, Hemmung oder Spezifität Studien, und es möglicherweise für Hochdurchsatz-geeignet Substrat-Screening oder anderen proteolytischen Enzymen angepasst werden kann.
In diesem Testsystem Spaltstellen für Tabak Ätzen Virus (TEV) und HIV-1-Proteasen und eine C-terminale fluoreszierenden Proteins, der eingesetzten Substrate enthalten N-terminalen Hexahistidine (seine6) und Maltose-bindende Protein (MBP) Tags. Die Substrate in Escherichia coli Zellen effizient herstellbar und leicht gereinigt mit Nickel (Ni) - Chelat - beschichtete Perlen. Während der Test führt der proteolytische Spaltung von Perle befestigt Substraten zur Freisetzung fluoreszierende Spaltung Fragmente, die durch Fluorimetry gemessen werden kann. Darüber hinaus können die Spaltung Reaktionen von Sodium Dodecyl Sulfat-Polyacrylamid-Gelelektrophorese (SDS-PAGE) analysiert werden. Ein Protokoll für die in-Gel-Renaturierung von Assay Komponenten ist auch beschrieben, wie teilweise Renaturierung von fluoreszierenden Proteinen ihre Erkennung anhand Molekulargewicht und Fluoreszenz ermöglicht.
Einleitung
Proteolytische Enzyme gehören zu den am intensivsten untersuchten Enzym Gruppen aufgrund ihrer Bedeutung in Stoffwechselwege und in industriellen Anwendungen sowie. Ihre Schlüsselrolle bei Viruserkrankungen, die Regulierung der Blutgerinnung, Krebs und Herz-Kreislauf- und neurodegenerativen Erkrankungen macht Proteasen wichtige Ziele auf dem Gebiet der Wirkstoffforschung. Daher die detaillierte Charakterisierung der Substratspezifität und Inhibitor Profilierung der Protease (PR) von Interesse ist von zentraler Bedeutung und erfolgt vorzugsweise durch schnelle, kostengünstige und robuste biochemischen Assays1,2, 3.
Heutzutage sind die überwiegende Mehrheit der in-vitro-Protease Assays angewendet im Bereich der Arzneimittelforschung für zusammengesetzte Profilerstellung homogene, fluoreszierende Peptid-basierte und Hochdurchsatz-Screening (HTS)-kompatiblen Plattformen4. Darüber hinaus beschriftete Peptide sind nicht nur für Bibliothek Screening geeignet, aber sie bieten auch große Werkzeuge für die Bestimmung des Enzyms kinetische Parameter der ausgewählten Substrate. In anderen Fällen, wo Kennzeichnung des Substrates nicht möglich ist, können die Trennung basierende Assays eine mögliche Lösung für die kinetischen Eigenschaften der proteolytischen Reaktionen3bewerten.
In der Regel in-vitro-Protease Assays basieren auf der Verwendung von zwei Arten von Substrat: kurze Peptide oder ganze Proteine. In den Fällen, wo die Spaltung von kurzen Peptid-Sequenzen entsprechen die Spaltung Eigenschaften ausreichend, gelten die folgenden Standardansätze: (i) standard Protein Substrate wie oxidierte Insulin B-Kette, (Ii) Prüfung Prüfung im Handel erhältlichen Substrate von anderen Proteasen, (Iii) screening synthetischer und Gewebekulturen beschriftet Peptid Bibliotheken erstellt von kombinatorischen Chemie oder (iv) mit genetischen Methoden, zum Beispiel biologische anzeigen Technologien5, 6. Neben der konventionellen Klassifikation, andere neuartigen Plattformen sind erhältlich für Substrat Generation (z. B. die Bildung von Proteom-abgeleitete Peptid Bibliotheken7 oder spezielle Subtypen von genetischen Methoden, wie die rekombinante Fusion Protein-basierten Substrate8,9,10,11,12).
Alle der oben genannten Substrat Typen und Tests haben ihre eigenen Vorzüge und Grenzen, und die Entwicklung von Assays Formaten kombiniert und/oder die Vorteile der bekannten Plattformen zu verbessern ist nach wie vor gefragt. Hier beschreiben wir ein Protokoll für eine Trennung-basierte fluoreszierende Protease-Assay, der rekombinante Substrate verwendet. Diese Schmelzverfahren Proteine bestehen aus seinen6 und MBP Tags verschmolzen zu einem Steuerelement Spaltstelle von TEV PR, gefolgt von der Substrat-Reihenfolge des Interesses, die direkt mit einer C-terminalen fluoreszierenden Proteins (FP) (Abbildung 1A) verbunden ist. Das Klonen einer DNA-Sequenz, die Codierung für eine Spaltstelle von Interesse in der "Klonen Kassette" kann durch einen einzigen Ligatur Reaktion in das Expressionsplasmid durchgeführt werden, die zuvor durch Beschränkung Endonucleases linearisiert worden.
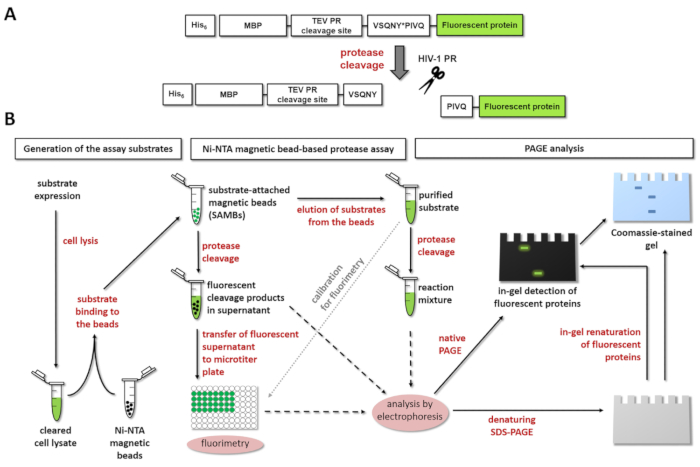
Abbildung 1: Prinzip des fluoreszierenden Protease Assays. (A) die schematische Darstellung von einem fluoreszierenden Substrat und seiner Spaltung durch humane Immundefizienz-Virustyp 1 (HIV-1) Protease wird angezeigt. Der Pfeil zeigt die Position der Spaltung innerhalb der Matrix/Kapsid Spaltung Website Sequenz von HIV-1 Protease (VSQNY * PIVQ). (B) die fluoreszierenden Substrate können verwendet werden, Enzymreaktionen analysieren durch Ni-NTA magnetischer Wulst-basierende Assays und Polyacrylamid Gelelektrophorese, ebenso, wie in der Workflow-Abbildung dargestellt ist. Bitte klicken Sie hier für eine größere Version dieser Figur.
Obwohl proteolytischen Assays mit ähnlichen rekombinanten Proteins Substrate-mit einer Affinität-Tags, eine proteolytische Spaltung-Website und ein fluoreszierendes Protein-Have bereits beschrieben wurden8,9,10, das System präsentiert hier beabsichtigt, zu integrieren und auf die Vorteile dieser Methoden zu verbessern. Ein wichtiger Unterschied ist, dass die Fusion Protein Substrate in diesem Assay-Plattform mit MBP Protein Löslichkeit13 und enthalten ein Steuerelement Spaltstelle TEV PRS ausgestattet sind. Darüber hinaus enthalten die Substrate neue Generation fluoreszierende Proteine, die sind sehr stabil und haben eine Monomere Form Substrat Aggregation zu verhindern. Neben den zuvor veröffentlichten Anwendung des mTurquoise2 und mApple verschmolzen Formen14zeigen hier wir auch, dass Ergebnisse durch den Einsatz eines rekombinanten Substrats mit einem Monomeren fluoreszierende Tag gelb fluoreszierende Protein (mEYFP) erweitert. Hiermit haben wir die Kompatibilität des Systems mit anderen fluoreszierende Proteine zu demonstrieren und vertreten einige allgemeinen Arten von Ergebnissen, die durch die Protease Assays erworben werden können.
Die rekombinanten Fusionsproteinen in E. Coli BL21(DE3) Zellen exprimiert werden und dienen als Substrate für die Probe in einer Nickel-Nitrilotriacetic Säure (Ni-NTA)-magnetisch-Agarose Bead-attached Form beschichtet. Die C-terminale Spaltprodukte werden von der Oberfläche der Perle in der Überstand nach Spaltung durch die Protease des Interesses befreit. Nach der Trennung des Überstands (mit dem Enzym und die Spaltprodukte) von der magnetischen Beads kann die Spaltung Eigenschaften des Enzyms bestimmen die Fluoreszenz gemessen werden. Im Gegensatz zu den zuvor beschriebenen Methoden in das hier vorgestellte System basierend auf einer detaillierten Substrat Kalibrierverfahren eindeutig die Mengen an Substrat und C-terminalen Spaltprodukte quantifiziert. Das Testsystem kann durch eine SDS-PAGE-Analyse der Probe Proben unterstützt werden; eine anschließende fluoreszierende in Gel-Visualisierung kann unmittelbar nach der Elektrophorese oder nach in-Gel Renaturierung der nondenatured und denaturierten fluoreszierenden Komponenten, bzw.14angewendet werden.
Die Flexibilität und die Struktur der "Klonen Kassette" erlauben eine Zeit- und kosteneffiziente Einfügung einer Vielzahl von Sequenzen in das Konstrukt und fördert somit die Generation der Substrat-Bibliotheken. Da alle Test-Schritte Automatisierung und HTS-kompatibel sind, kann das System besonders attraktiv für, zum Beispiel werden Protease Spezifität Messungen und Mutagenese Studien oder es auch effektiv für industrielle Protease-Inhibitor-Screening genutzt werden kann und/oder antivirale Medikamentenentwicklung.
Enzym kinetische Parameter (kKatze, Km) können durch die entwickelten Trennung basierende Assays bestimmt werden; Daher kann es einzelne Enzym kinetische Messungen, z. B. den Zeitverlauf, Substrat-abhängige und Hemmung Studien geeignet sein. Dies beweist, dass die rekombinante Fusion Protein Substrate gute Alternativen für die häufig eingesetzten synthetischen Oligopeptid-Substrate bieten, und aufgrund ihrer hohen Ähnlichkeit mit der funkionalen Substrate, sie repräsentieren die natürlich vorkommenden Enzym-Substrat-Interaktionen genauer.
Protokoll
1. Generation der Substrat-Codierung Ausdruck Plasmide
- Linearisieren Sie das pDest, seine6- MBP-FP-Expressionsplasmid von PacI und NheI Beschränkung Endonucleases. Für die Erzeugung von pDest, seine6- MBP-FP siehe Bozóki Et Al.14.
- Hinzufügen von 1.500-2.000 µg des pDest, seine6- MBP-FP Expressionsplasmid, 2 µL der PacI und NheI Beschränkung Endonucleases, 10 µL 10 X Puffer (siehe Tabelle der Materialien), und Nuklease-freies Wasser (NFW) bis 100 µL in einem Microcentrifuge Schlauch.
- Inkubieren Sie die Reaktionsmischung bei 37 ° C für 1 h.
- Fügen Sie 20 µL 6 x DNA lila laden Farbstoff zum Reaktionsgemisch und trennen Sie die Spaltprodukte durch Elektrophorese, mit 1 % Agarose-Gel. Gelten Sie eine 1 kB DNA-Leiter als Standard.
- Spülen Sie das Gel für 15 min in 20 mL TAE Puffer (40 mM Tris, Essigsäure 20 mM, 1 mM EDTA, pH 8,5) mit 20 µL SYBR grün-Lösung und Verbrauchsteuern Sie die Band des linearisierten Plasmids aus dem Agarosegel, mit einem spitzen Werkzeug.
Hinweis: Beim beleuchten des Gels durch eine dunkel-Lesung blau Transilluminator (DRBT), erscheint der linearisierten pDest, seine6- MBP-FP Plasmid als diskrete und helle Band bei ca. 7-8 kB. - Reinigen Sie das linearisierte Expressionsplasmid von der Gel-Scheibe, indem mit einem Gel Extraction Kit gemäß den Anweisungen des Herstellers.
- Die Substrat-Sequenz in der linearisierten pDest, seine6- MBP-FP Expressionsplasmid einsetzen.
- Tempern Sie vorwärts (FWD) und rückwärts (REV) E. Coli Codon-optimierte Oligonukleotid Primer Codierung für die Substrat-Reihenfolge des Interesses.
Hinweis: Die geglühten Primer werden durch die kohäsiven enden entsprechend PacI und NheI Einschränkung Endonuklease Spaltstellen (Abbildung 2) flankiert werden.- Mix-150 ng der linearisierten Expressionsplasmid mit 200 ng FWD und 200 ng REV Oligonucleotide Zündkapseln in einer 0,2 mL Polymerase-Kettenreaktion (PCR) Rohr und stellen Sie die Lautstärke auf 17 µL durch Zugabe von NFW.
- Inkubieren Sie die Mischung bei 65 ° C für 2 min und dann bei 4 ° C für mindestens 2 min.
- Durchführen Sie das Einfügen der geglühten Primer in der linearisierten Plasmid durch Unterbindung.
- Die Mischung mit dem linearisierten Plasmid und die geglühten Primer fügen Sie 2 µL T4-Ligase Puffer (10 X) und 1 µL T4-Ligase hinzu.
- Inkubieren Sie die Ligatur-Mischung bei 16 ° C 16 h.
- Tempern Sie vorwärts (FWD) und rückwärts (REV) E. Coli Codon-optimierte Oligonukleotid Primer Codierung für die Substrat-Reihenfolge des Interesses.

Abbildung 2 : Oligonucleotide Zündkapseln Codierung für eine proteolytische Spaltung Website Sequenz. Forward und reverse Primer kodieren die VSQNY * PIVQ Spaltung Website Abfolge von HIV-1 PR. Nach Glühen die komplementären Oligonukleotids Primer enthält kurze doppelsträngige DNA klebrige enden, entsprechend der PacI und NheI Beschränkung Endonucleases. Bitte klicken Sie hier für eine größere Version dieser Figur.
- 100 µL kompetente Zellen BL21(DE3) von 5 µL der Ligatur Mischung zu verwandeln und die Zellen auf Lysogeny Brühe (LB) Agarplatten mit Ampicillin verteilt.
Hinweis: Die fluoreszierende Proteine werden im gleichen offenen Leseraster mit der N-terminalen Fusion Tags erst nach einer erfolgreichen Verbindung. Ein paar Tage nach der Transformation, zeigen die Kolonien (mit dem Ausdruck Plasmid Codierung für die eingefügten Spaltstelle von Interesse) sichtbare Fluoreszenz mit oder auch ohne Verwendung eines DRBT. - Bereiten Sie Glycerin bestand aus den abgebildeten Kolonien.
- Waschen Sie eine diskrete Kolonie in ein 50 mL Zentrifugenröhrchen mit 5 µL LB-Medium mit Ampicillin (bei einer Endkonzentration von 100 µg/mL).
- Inkubieren sie bei 37 ° C für 8 h unter Schütteln kontinuierlich bei 220 u/min; dann ernten Sie die Zellen durch Zentrifugation bei 1.000 X g für 5 min bei Raumtemperatur.
- Sanft auszusetzen Sie, die Zellen in 1 mL 80 % Glycerin-Lösung (mit destilliertem Wasser verdünnt) und die Federung fügen Sie 500 µL 10 mM MgCl2 Lösung hinzu.
- Übertragen Sie die Aussetzung zu einem eiskalten Schlauch und speichern Sie die Bestände bei-70 ° C.
- Überprüfen Sie die Reihenfolge der generierten Plasmid durch DNA-Sequenzierung.
- 5 mL LB-Medium mit 100 µg/mL Ampicillin in ein 50 mL Zentrifugenröhrchen fügen Sie 10 µL der Glycerin-bestand (vorbereitet in Schritt 1,7 hinzu).
- Inkubieren Sie die Federung bei 37 ° C 16 h unter Schütteln kontinuierlich bei 220 u/min; dann ernten der Zellen durch Zentrifugation bei 2.000 X g für 10 min bei 4 ° C.
- Isolieren der Expressionsplasmid aus der Zelle-Pellets durch ein Plasmid Miniprep kit (siehe Tabelle der Materialien) gemäß den Anweisungen des Herstellers, und verwenden Sie die gereinigte Plasmid für DNA-Sequenzierung.
Hinweis: für die Sequenzierung, 5'-GATGAAGCCCTGAAAGACGCGCAG-3 "(vorwärts) und 5'-GCAAGGCGATTAAGTTGGGTAACGC-3" (rückwärts) Oligonukleotid Primer verwendet werden.
(2) Ausdruck der fluoreszierenden Substrate
- Bereiten Sie die Starterkultur.
- 5 mL LB-Medium mit 100 µg/mL Ampicillin in ein 50 mL Zentrifugenröhrchen fügen Sie 10 µL der Glycerin-bestand (vorbereitet in Schritt 1,7 hinzu).
- Inkubieren Sie die Federung bei 37 ° C für 15 h unter Schütteln kontinuierlich bei 220 u/min.
- Übertragen Sie die Bakterienkultur (5 mL) auf 50 mL frisches LB-Medium mit 100 µg/mL Ampicillin in einem sterilen Erlenmeyerkolben 500 mL.
- Wachsen Sie die Zellen bei 37 ° C, eine Extinktion von 0,6-0,8 bei einer 600 nm Wellenlänge, während kontinuierlich bei 220 u/min schütteln.
Hinweis: Wenn eine Tetracyclin-Behandlung bei Schritt 2.5 angewendet werden, es empfiehlt sich nicht die Zellen wachsen, eine Extinktion von mehr als 0,6 bei 600 nm. - Die Endkonzentration von 1 mM induzieren Proteinexpression fügen Sie Isopropyl β-D-1-Thiogalactopyranoside (IPTG hinzu).
- Wenn eine Tetracyclin-Behandlung nicht angewendet wird, inkubieren Sie die Kultur für 3 h bei 37 ° C unter Schütteln kontinuierlich bei 220 u/min und das Protokoll mit Schritt 2,6. Wenn eine Tetracyclin-Behandlung angewendet wird, fahren Sie das Protokoll mit Schritte 2.5.1-2.5.3.
Hinweis: Einige FPs von E. Coli Zellen produziert möglicherweise eine längere Reifezeit (siehe vorherigen arbeiten)16,17; in diesen Fällen kann die Protein-Übersetzung optional durch die Tetracyclin-Behandlung verhaftet werden, um die fluoreszierenden Ausbeute an die Substratlösung zu erhöhen.- Inkubieren Sie die Zellsuspension für 2 h bei 37 ° C unter Schütteln kontinuierlich bei 220 u/min; Fügen Sie dann eine Tetracyclin-Lösung (auf eine Endkonzentration von 200 µg/mL).
- Inkubieren Sie die Zellkultur nach der Reifezeit des fluoreszierenden Proteins Wahl bei 37 ° C unter Schütteln kontinuierlich bei 220 u/min.
- 2 x 25 mL der Kultur zu 50 mL Zentrifuge Rohre reinigen und Ernten der Zellen durch Zentrifugation bei 4.000 X g für 15 min bei 4 ° c zu übertragen
- Verwerfen Sie den Überstand und speichern Sie die Bakterienzelle Pellets bei-70 ° C für mindestens 1 h.
Hinweis: Zellen, die zum Ausdruck gebrachten fluoreszierende Substrate zeigen sichtbare Fluoreszenz mit oder auch ohne Verwendung eines DRBT.
(3) Zellaufschluss
- Die gefrorenen Zellen Pellet auf Eis und lassen Sie es für 15 Minuten Auftauen.
- Das Pellet 2 mL Lyse-Puffer (50 mM NaH2PO4, 300 mM NaCl, 10 mM Imidazol, 0,05 % Tween 20, pH 8) hinzu und auszusetzen Sie die Zellen.
- Die Suspension fügen Sie 10 µL frisch zubereiteten Phenylmethanesulfonyl-Fluorid (PMSF) Protease-Inhibitor-Lösung (8,7 mg/mL, gelöst in Ethanol hinzu).
- Die Suspension fügen Sie 2 mg Lysozym und 20 Einheiten der DNase hinzu und hängen Sie es.
- Inkubieren Sie die Suspension für 15 min und Wirbel auf Eis es gelegentlich.
- 2 x 1 mL der Suspension auf 1,5 mL Mikrozentrifugenröhrchen übertragen und beschallen die Suspensionen für 3 min in Runden von 10 s von Beschallung und 5 s ruhen.
- Zentrifugieren Sie die Rohre bei 10.000 X g für 20 min bei Raumtemperatur; dann den fluoreszierenden überstand (geräumte bakterielle Zelle lysate) aus jedem Röhrchen vorsichtig entfernen und auf neue Mikrozentrifugenröhrchen übertragen.
Hinweis: Geräumte Lysates mit fluoreszierenden Substrat zeigen sichtbare Fluoreszenz mit oder ohne Verwendung einer DRBT und kann bis zu 2 Wochen bei 4 ° C gelagert werden. NICHT einfrieren. Geräumte Lysates können direkt für die Probenvorbereitung in der Protease-Assay (siehe Abschnitt 4.1) genutzt werden oder auch zur Reinigung von Substrat eingesetzt werden (siehe Punkt 4.5.1).
(4) Ni-NTA magnetische-Perle-basierte Protease Assays
Hinweis: Aufgrund der Flexibilität der Assay-Plattform, können sie auf viele verschiedene Arten von Studien optimiert werden. Aus diesem Grund und aufgrund der Differenz in der Erwerbsquote der Enzyme der Wahl einige der Assay-Parameter (wo es angegeben ist) nicht explizit beschrieben werden aber zu den einzelnen Zielen und experimentelles Design optimiert werden müssen. Als Orientierungshilfe werden Parameter für einige Arten von Studien an den bestimmten Stufen bezeichnet.
- Vorbereitung der Probe
- Generation von Substrat befestigt magnetische beads
- Legen Sie einen geschlossenen 2 mL niedrig-Protein-Bindung (siehe Tabelle der Materialien) Microcentrifuge Schlauch enthalten neu oder recycelt (siehe Abschnitt 4.7) Ni-NTA Agarose magnetische Perlen in einem Magnetpartikel Konzentrator (MPC).
Hinweis: Die Höhe der angewandten Wulst Suspension soll basierend auf den experimentellen Design festgelegt werden. Wir haben 1 mL magnetischer Wulst-Lösung (5 %, V/V) bei jedem Experiment. - Perlen können an der Wand und/oder in den Deckel der Microcentrifuge Schlauch kleben; Daher umdrehen Sie das MPC in alle Richtungen, um sicherzustellen, dass alle die Perlen gesammelt werden.
- Den Überstand zu entfernen und entsorgen.
- Waschen Sie die Perlen durch Lyse Puffer.
- Die Perlen 1,8 mL Lyse Puffer hinzu und entfernen Sie die geschlossene Röhre aus der MPC.
- Unterbrechen Sie die Perlen in der Röhre durch Schütteln und/oder die Rohre Kopf drehen, bis die Probe völlig homogen ist.
- Setzen Sie den Schlauch wieder in die MPC und schalten Sie ihn kopfüber, die Perlen zu sammeln.
- Öffnen Sie der Tube und verwerfen Sie den überstand.
- Die Perlen 1,0-1,8 mL die geräumten lysate (vorbereitet in Schritt 3,7) hinzu und entfernen Sie den Schlauch aus der MPC.
- Umdrehen der geschlossenen Rohr bis die Perlen vollkommen homogen sind und drehen Sie langsam den Schlauch durch ein Rotator bei Raumtemperatur für 30 Minuten.
- Legen Sie es in den MPC und entfernen Sie die geräumte Zelle lysate aus die Perlen und den Deckel.
Hinweis: Die gelöschte Zelle lysate kann verworfen oder für die weitere Verwendung gespeichert (siehe Hinweis nach Schritt 3,7). - Fügen Sie 1 % Tween 20 (pH 7) auf dem Substrat befestigt magnetische Beads (SAMBs).
Hinweis: SAMBs zeigen sichtbare Fluoreszenz mit oder auch ohne Verwendung eines DRBT.
- Legen Sie einen geschlossenen 2 mL niedrig-Protein-Bindung (siehe Tabelle der Materialien) Microcentrifuge Schlauch enthalten neu oder recycelt (siehe Abschnitt 4.7) Ni-NTA Agarose magnetische Perlen in einem Magnetpartikel Konzentrator (MPC).
- Waschen von der SAMBs
- Legen Sie das Rohr mit der SAMB Suspension in der MPC und verwerfen Sie den überstand.
- Waschen Sie die SAMBs 3 X mit jeder Puffer: i) 1,8 mL 1 % Tween 20 (pH 7); (II) 1,8 mL waschen Puffer (50 mM NaH2PO4, 300 mM NaCl, 5 mM Imidazol, 0,05 % Tween 20, pH 7); (III) 1,8 mL Spaltung Puffer (50 mM NaH2PO4, 300 mM NaCl, 0,05 % Tween 20, pH 7).
Hinweis: Für den Waschvorgang, siehe Schritt 4.1.1.4. Die Spaltung Puffer nach den experimentellen Bedürfnissen geändert werden, aber es wird empfohlen, das Handbuch der Ni-NTA magnetische Beads auf Kompatibilität zu überprüfen.
- Vorbereitung der Vorratslösung SAMB
- Die gewaschenen SAMBs erstelle ich eine Stammlösung SAMB fügen Sie Spaltung Puffer hinzu.
Hinweis: Nach der Zugabe des Puffers nicht schütteln oder das Rohr umdrehen. Das Volumen des Puffers Spaltung richtet sich nach den einzelnen Versuchsdesign und muss basierend auf der Anzahl der magnetischen Beads (siehe Schritt 4.1.1.1) und auf den Datenträgern im Schritt 4.1.4.2 zu verwendende berechnet werden. Für 2 mL Röhrchen, das angewandte Volumen beträgt bis zu 1.900 µL (siehe Tabelle 1). Die empfohlene magnetische Wulst Dichte der Vorratslösung SAMB ist 2 % - 10 % (V/V).Studientyp Volumen der Spaltung Puffer (µL) S-abhängige Messungen (Abb. 4) 1600 Zeitverlauf Messungen (Abb. 5A) 1600 Hemmung-Studie (Abb. 5 b) 1900 pH-Abhängigkeit-Studie (Abb. 6) 1400
Tabelle 1: Volumen des Puffers Spaltung zur Vorbereitung der SAMB Stammlösung in die verschiedenen Arten von Messungen verwendet. - Entfernen Sie die geschlossene Röhre aus MPC. Benutzen Sie SAMB Vorratslösung sofort oder lagern Sie es bei 4 ° C für bis zu 24 h.
- Die gewaschenen SAMBs erstelle ich eine Stammlösung SAMB fügen Sie Spaltung Puffer hinzu.
- Generation der Assay-Proben mit der Stammlösung SAMB
Hinweis: Die Details dieses Teils des Assays sind stark abhängig von der individuellen experimentelles Design (Beispiel Arten sind in Tabelle 2dargestellt).Probenart Notizen Reaktion Probe (R) -verwendet für die Beurteilung der Spaltung Eigenschaften
-enthält das Enzym und Substrat im Dekolleté PufferSubstrat leer Probe (B) -verwendet für die Beurteilung der spontanen Substrat Dissoziation (siehe Schritt 4.6.2)
-enthält nur das Substrat im Dekolleté PufferSubstrat-Kontrollprobe (C) -für Detemining Substratkonzentration (siehe Punkt 4.6.3)
-enthält nur das Substrat im Elution Buffer
Tabelle 2: Probentypen des Ni-NTA magnetische-Perle-basierte Protease Assays.- 2 mL Mikrozentrifugenröhrchen niedrig-Protein-Bindung für die Test-Proben vorbereiten.
Hinweis: Andere eiweißarme verbindlich Kunststoff waren kann auch verwendet werden. Verwenden Sie Runde oder flache Unterseite Röhren um den freien Warenverkehr SAMBs sicherzustellen. Die empfohlene Anzahl von Rohren in Tabelle 3zu sehen.Studientyp R B C S-abhängige Messungen (Abb. 4) 5 5 2 Zeitverlauf Messungen (Abb. 5A) 6 6 2 Hemmung-Studie (Abb. 5 b) 7 7 1 pH-Abhängigkeit-Studie (Abb. 6) 5 5 1
Tabelle 3: Anzahl der erforderlichen 2 mL Mikrozentrifugenröhrchen für jede Probe in den Studien gezeigten. - Aussetzen Sie der SAMB Stammlösung bis Homogenität und überweisen Sie den Betrag von Substrat in den Reaktionen sofort in das Probenfläschchen analysiert werden. Die empfohlene Menge ist 25-300 µL, aber es ist anhand der individuellen experimentelles Design (Tabelle 4) festgelegt werden.
Hinweis: Überprüfen Sie, ob alle SAMBs in den Boden der Röhrchen gemessen wurden. SAMBs können an der Wand des Rohres, haften die Untersuchungsergebnisse verfälschen können. Wenn unterschiedliche Volumina sind nacheinander gemessen werden, starten Sie Aliquotierung mit dem höchsten Volumen zu und versuchen Sie, die Änderung der Pipetten und Pipettenspitzen zu minimieren.Studientyp R B C S-abhängige Messungen (Abb. 4) 25 – 50 – 100 – 150 – 250 25 – 50 – 100 – 150 – 250 25 Zeitverlauf Messungen (Abb. 5A) 25 25 25 Hemmung-Studie (Abb. 5 b) 120 120 120 pH-Abhängigkeit-Studie (Abb. 6) 100 100 100
Tabelle 4: Volumen der SAMB Lösung in das Probenfläschchen der einzelnen Muster in den aufgezeigten Studien gemessen. - Legen Sie die Probenröhrchen mit regelmÄÑig SAMB Aussetzung in der MPC und bewegen Sie MPC leicht hin und her.
- Sorgfältig die SAMBs überstand entfernen und entsorgen.
- Entfernen der Röhren von MPC und das berechnete Volumen der Reaktion Puffer (Spaltung oder Elution Buffer [100 mM EDTA, 0,05 % Tween 20, pH 7]) sorgfältig in die SAMBs.
Hinweis: Berechnen Sie das Puffervolumen entsprechend den einzelnen Versuchsplan (Tabelle 5). Für 2 mL-Tuben ist das empfohlene Endvolumen des Reaktionsgemisches (das Volumen des Puffers Reaktion in diesem Schritt hinzugefügt werden + das Volumen der Lösung im Schritt 4.2.3 hinzugefügt werden), 50-150 µL. Stellen Sie sicher, dass alle die SAMBs im zusätzlichen Puffer gewaschen werden. Elution Buffer wird statt Spaltung Puffer in den Fällen von Substrat Kontrollproben (C) verwendet. Für eine Hemmung-Studie wird die Inhibitor der Wahl empfohlen, bei diesem Schritt hinzugefügt werden.Studientyp Volumen des Puffers Reaktion (µL) S-abhängige Messungen (Abb. 4) 68 µL Spaltung Puffer Zeitverlauf Messungen (Abb. 5A) 68 µL Spaltung Puffer Hemmung-Studie (Abb. 5 b) 67,3 µL Spaltung Puffer + 0,7 µL Inhibitor Lager Lösung * pH-Abhängigkeit-Studie (Abb. 6) 69,5 µL Spaltung Puffer **
Tabelle 5: Volumen der Reaktion Puffer in den Studien nachgewiesenen. * Amprenavir wurde in Dimethyl Sulfoxid gelöst; Amprenavir Stammlösungen (von 1 nM bis 1 µM Konzentrationen) wurden für hemmende Studie angewendet (siehe Abbildung 5 b). ** Der pH-Wert des Puffers angewandte Spaltung reichten von pH 6,0-8,5. - Schließen Sie die Abdeckungen der Rohre. Jetzt sind die Proben zum Test bereit.
Hinweis: Die Proben bei 4 ° C bis zu 24 Stunden gelagert werden können, aber der Speicher ist nur anwendbar, wenn der SAMB Stammlösung sofort nach der Zubereitung verwendet wurde (siehe Schritt 4.1.3.2).
- 2 mL Mikrozentrifugenröhrchen niedrig-Protein-Bindung für die Test-Proben vorbereiten.
- Generation von Substrat befestigt magnetische beads
- Einleitung der proteolytischen Reaktionen
- Bereiten Sie die proteolytischen Enzymlösung nach den experimentellen Bedürfnissen.
Hinweis: Es wird empfohlen, mithilfe einen Spaltung Puffer auflösen und/oder verdünnen Sie das Enzym. Protokolle für die Reinigung von HIV-1-14 und TEV PRs18 wurden bisher veröffentlicht. - Legen Sie die Thermoshaker Agitation Rate (600 u/min) und Inkubationstemperatur (Tabelle 6).
Studientyp Inkubationstemperatur (° C) S-abhängige Messungen (Abb. 4) 37 Zeitverlauf Messungen (Abb. 5A) 37 Hemmung-Studie (Abb. 5 b) 37 pH-Abhängigkeit-Studie (Abb. 6) 30
Tabelle 6: Inkubation Temperaturen angewendet in verschiedenen Studientypen. Für HIV-1 PR empfiehlt 37 ° C während 30 ° C für TEV PR. empfohlen wird, - Die Reaktion Proben für die Initialisierung proteolytischen Reaktionen der Enzymlösung hinzugefügt werden.
Hinweis: Bei Substrat leer (B) und C Proben fügen Sie Spaltung Puffer (Enzym-Puffer) und Elution Buffer hinzu, beziehungsweise. Das Volumen beträgt je nach experimentellen Bedarf (Tabelle 7) berechnet werden. Für 2 mL-Tuben ist die empfohlene Endvolumen des Reaktionsgemisches (das Volumen des Puffers Reaktion in Schritt 4.1.4.5 hinzugefügt + das Volumen der Lösung in diesem Schritt hinzugefügt werden) 50-150 µL.Studientyp Volumen des Enzyms Lösung/Enzym Puffer/Elution Puffers (µL) S-abhängige Messungen (Abb. 4) 2 Zeitverlauf Messungen (Abb. 5A) 2 Hemmung-Studie (Abb. 5 b) 2 pH-Abhängigkeit-Studie (Abb. 6) 0,5
Tabelle 7: Volumen des Enzyms Lösung/Enzym Puffer/Elution Puffers hinzugefügt während der Initialisierung der Assay-Proben bei der nachgewiesenen Studien. - Schüren Sie die Perlen sorgfältig durch sanft bewegen die Rohre, und legen Sie die Rohre sofort in die bereits schütteln Thermoshaker.
Hinweis: Manuelle Probe Kündigung (siehe Ziffer 4.3) dauert länger als die Einleitung; Daher empfiehlt sich eine registrierte Verzögerung von mindestens 2 min zwischen den Reaktionen Einweihungen. - Inkubieren Sie die Proben nach den Versuchsplan (Tabelle 8).
Studientyp Inkubation Zeit (min) S-abhängige Messungen (Abb. 4A) 7 S-abhängige Messungen (Abb. 4 b) 120 Zeitverlauf Messungen (Abb. 5A) 0 – 2,5 – 5 – 10 – 15 – 20 Hemmung-Studie (Abb. 5 b) 10 pH-Abhängigkeit-Studie (Abb. 6) 60
Tabelle 8: Inkubationszeiten angewendet an der Assay-Proben in verschiedenen Maßeinheiten.
- Bereiten Sie die proteolytischen Enzymlösung nach den experimentellen Bedürfnissen.
- HinterAtion der proteolytischen Reaktionen
- Nehmen Sie das Beispiel aus Shaker, 30 s vor Ablauf der Inkubation, und drehen Sie es umgehend.
- Legen Sie das Rohr auf die MPC, 15 stehen lassen s, und die MPC leicht hin und her bewegen.
- Öffnen Sie den Deckel und übertragen Sie überstand vorsichtig auf eine Platte oder einen neuen Schlauch.
Hinweis: Berühren Sie die konzentrierten Perlen mit der Pipettenspitze nicht. Die gesammelten Überstand von C und R Proben mit einem hohen Maß an Dekolleté kann sichtbare Fluoreszenz mit oder auch ohne die Verwendung einer DRBT zeigen.
- Fluoreszenz-Detektion
- Übertragen Sie 2 x 30 µL der getrennten Probe Überstände auf eine schwarze Hälfte-Bereich Mikrotestplatte.
- Messung der Fluoreszenz entsprechende Anregung und Emission Filter verwenden.
Hinweis: Messen Sie die grundlegenden Fluoreszenz der Spaltung Puffer und Elution-Puffer. Filter-Kombinationen müssen gewählt werden anhand der gemessenen fluoreszierenden Proteins (Tabelle 9).Fluoreszierendes protein Erregung Filter (nm) Emission Filter (nm) mTurqiouse2 355/40 460/25 mEYFP 544/15 590/10 mApple 544/15 590/10
Tabelle 9: Anregung und Emission Filter verwendet, um verschiedene fluoreszierende Proteine zu erkennen.
- Kalibrierung
Hinweis: Um Kalibrierkurven im Schritt 4.6.1, Fluoreszenz Intensitätswerte der Spaltung-Elution-Puffer-gelöst oder generieren gereinigten Substrate in verschiedenen Konzentrationen gemessen werden müssen.- Reinigen Sie die fluoreszierenden Substrate.
Hinweis: Für die Reinigung, SAMBs Substrat leer (B) Proben nach der Protease Assays gesammelt werden kann oder eine neue SAMB kann Aussetzung auch (siehe Abschnitte 4.1.1 und 4.1.2) vorbereitet werden.- Legen Sie einen Schlauch mit SAMBs ausgesetzt in 1 mL der Spaltung Puffer (2 % - 10 %; V/V), MPC und sammeln Sie die magnetische Beads durch drehen das MPC Kopf in alle Richtungen.
- Öffnen Sie der Tube und entfernen Sie den Spaltung Puffer, sowohl aus dem Rohr und den Deckel zu.
- Entfernen Sie den Schlauch aus der MPC und die SAMBs fügen Sie 400-600 µL Elution Puffer hinzu.
- Die geschlossene Röhre mit einem Rotator bei Raumtemperatur für 5 min langsam zu drehen.
- Legen Sie das Rohr auf die MPC und sammeln Sie die Perlen durch drehen das MPC Kopf.
- Den Überstand mit dem gereinigten intakt fluoreszierende Substrat (Eluat) zu entfernen und auf einen neuen Low-Protein-Bindung Microcentrifuge Schlauch übertragen.
Hinweis: Eluat zeigt deutlich sichtbare Fluoreszenz mit oder auch ohne Verwendung eines DRBT.
- Mit zwei 0,5 mL 10K Ultrafiltration Geräte führen Sie paralleler Buffer Tausch aus.
- Messen Sie die Hälfte das vorbereitete Eluat (200-300 µL) in jedes Ultrafiltration Gerät.
- Nach bei jedem Zentrifugationsschritt verdünnen Sie das konzentrierte Eluat in der ersten und der zweiten Ultrafiltration Geräte durch Elution Buffer und Spaltung Puffer, beziehungsweise.
- Stellen Sie nach der Wiederherstellung der konzentrierten Proben in den verschiedenen Puffern auf derselben Festplatte, zwischen 120-200 µL gelöst.
Hinweis: Jetzt ist der Eiweißgehalt der Spaltung Puffer gelöst Substrat identisch mit der Elution Puffer gelöst Substrat; Daher ist es nicht zu prüfen, der Eiweißgehalt des letzteren bei Schritt 4.5.3, wenn die Methode zur Messung der Proteinkonzentration mit EDTA stört.
- Bestimmen der Proteingehalt der Substrate aufgelöst entweder in Elution oder das Dekolleté Puffer durch Messung der Absorption bei 280 nm.
Hinweis: Andere Methoden (z. B. Bradford oder Bicinchoninic Säure (BCA) Assays) können auch zur Proteinkonzentration zu messen, aber mögliche Störungen mit EDTA (vorhanden in der Elution Buffer) oder die Extinktion des fluoreszierenden Substrates muss sein in Betracht gezogen. Der erste Proteingehalt der Substrat-Lösung im Schritt 4.5.4 angewendet werden wird empfohlen, zwischen 0,4-2,0 mg/mL um eine Kalibrierung generieren Kurve in einem entsprechenden Angebot. Siehe Tabelle 10 für Aussterben Koeffizienten.Substrat Molekulargewicht
(Da)Vom Aussterben bedroht-Koeffizient
(M-1 cm-1, bei 280 nm gemessen in Wasser)Seine6- MBP-VSQNY * PIVQ-mTurquoise2 72101.7 96845 Seine6- MBP-KARVL * AEAM mTurquoise2 72042.7 95355 Seine6- MBP-VSQNY * PIVQ-mEYFP 72367.1 94325 Seine6- MBP-VSQNY * PIVQ-mApple 72145.9 105200
Tabelle 10: Molekulargewichte und die vom Aussterben bedroht-Koeffizienten der verschiedenen rekombinanten fluoreszierende Fusion Protein Substrate. - Bereiten Sie eine zweifache serielle Verdünnung in mindestens acht Stufen, sowohl aus der Elution - Spaltung-Puffer gelöst Substrat-Lösungen, mit Elution oder Spaltung Puffer für die Verdünnung bzw..
- Übertragen Sie 30 µL jeder Verdünnung Punkt auf eine schwarze Hälfte-Bereich Mikrotestplatte.
- Messung der Fluoreszenz mit einem Fluorimeter, mit der Einstellung im Schritt 4.4.2 angewendet.
Hinweis: Messen Sie die grundlegenden Fluoreszenz der Spaltung und der Elution Buffer.
- Reinigen Sie die fluoreszierenden Substrate.
- Auswertung des Tests
- Zeichnen Sie die Kalibrierkurven.
- Berechnen Sie die Konzentration (in mM) der gereinigten Untergrund Lösungen (verwendet in Schritt 4.5.4), basierend auf der Eiweißgehalt im Schritt 4.5.3 bestimmt.
- Korrigieren Sie die relative Fluoreszenz Intensitätswerte (RFU) der serielle Verdünnung Punkte, indem RFU Grundwerte der angewandten Verdünnungspuffer (Spaltung Puffer oder der Elution Buffer).
- Plot-Korrekturwerte RFU gegen die molare Konzentration der Spaltung-Elution-Puffer-gelöst oder gereinigte Substrate und führen eine lineare Regression (Kraft der Schnittpunkt auf Null).
Hinweis: Ein hoher Wert für R2 (˃0.97) zeigt eine gute lineare Korrelation zwischen der Fluoreszenz und der Konzentration des fluoreszierenden Proteins. Die Steigung der Regressionsgeraden ist in diesem Fall lässt sich die Konzentration der Assay-Komponenten im untersuchten Bereich in den Schritten 4.6.2 und 4.6.3 zu beurteilen. Experimentellen Fehler und Daten Punkteverteilung kann Auswirkungen auf die Zuverlässigkeit der Kalibrierung; Somit kann eine grafische Auswertung mit Hilfe der Zoom-in Grafiken (wie in Abbildung 3dargestellt), durchgeführt werden, um festzustellen, ob die Daten R2 und die Neigungswerte beeinflusst werden.
- Berechnen Sie die Höhe der C-terminalen fluoreszierende Spaltprodukt in die Reaktion Proben.
- Korrigieren Sie die RFU Werte jeder R-Probe mit den RFU-Werten der entsprechenden B-Probe.
- Berechnen Sie die Spaltung Produkt-Konzentration (in mM) in die Reaktion Proben dividiert den korrigierten RFU Werte durch die Steigung der Spaltung-Puffer-basierte Kalibrierung Kurve (siehe Schritt 4.6.1.3).
- Berechnen Sie die angewandte Substratkonzentration in die Reaktion Proben.
- Korrigieren Sie die RFU-Werte der Probe C mit der RFU-Wert von der grundlegenden Elution Buffer.
- Die Konzentration der eluierten Substrat (in mM) in die Überstände der C-Probe zu berechnen, indem man ihre korrigierten RFU Werte durch die Steigung der Elution-Puffer-basierte Kalibrierung Kurve (siehe Schritt 4.6.1.3).
- Bestimmen Sie die Substratkonzentration (in mM) der SAMB Vorratslösung für die Erstellung der Assay-Proben im Schritt 4.1.4.2, basierend auf die folgende Gleichung verwendet:
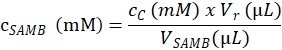
Hier ist cSAMB die molare Konzentration der SAMB Vorratslösung in Abschnitt 4.1.3 zubereitet; cC ist die molare Konzentration der eluierten Substrat in die C-Probe bei Schritt 4.6.3.3 berechnet; VR ist das Volumen des Reaktionsgemisches erstellt durch die Zugabe des Puffers Reaktion in Schritt 4.1.4.5 und der Enzym-Puffer im Schritt 4.2.3.; und VSAMB ist das Volumen der SAMB Vorratslösung in der C-Probe (Schritt 4.1.4.2). - Berechnen Sie die molare Konzentration der Substrate in jeder R Probe der molaren Konzentration der SAMB Vorratslösung (in mM) entsprechend dem Volumen (in µL) in jeder Probe Reaktionsgefäß bei Schritt 4.1.4.2 gemessen.
- Durchführen Sie Verarbeitung der Daten.
Hinweis: Analyse der Daten richtet sich nach dem Ziel des Experiments. Das Video zeigt ein Beispiel für die Datenverarbeitung für eine Substrat-abhängige kinetische Studie auf HIV-1-PR mit seinem6- MBP-VSQNY * PIVQ-mTurquoise2 Substrat. Die Anfangsgeschwindigkeit Werte errechnen sich aus der Anzahl der C-terminalen Spaltung Fragmente und sind gegen die angewandte Substratkonzentration aufgetragen. Die kinetischen Parameter werden durch eine nicht-lineare Regressionsanalyse Michaelis-Menten bestimmt.
- Zeichnen Sie die Kalibrierkurven.
- Recycling von magnetischen beads
Hinweis: Nach der Durchführung eines Tests, die magnetische Agarose Korne können gesammelt und recycelt werden.- Sammeln Sie die verwendeten magnetischen Kügelchen mit dem MPC zu und entsorgen Sie überstand.
- Waschen Sie die Perlen mit 1,8 mL der folgenden Puffer in der angegebenen Reihenfolge: Regeneration Puffer A (0,05 % Tween 20, 0,5 M NaOH), Regeneration Puffer B (0,05 % Tween 20), Regeneration Puffer C (0,05 % Tween 20, 100 mM EDTA, pH 8), Regeneration Puffer B, Regeneration Puffer D ( 0,05 % Tween 20, 100 mM NiSO4, pH 8), Regeneration Puffer B und Regeneration Puffer E (0,5 % Tween 20, 30 % Ethanol, pH 7).
Hinweis: Für den Waschvorgang, siehe Schritt 4.1.1.4.
- Waschen Sie die Perlen mit 1,8 mL der folgenden Puffer in der angegebenen Reihenfolge: Regeneration Puffer A (0,05 % Tween 20, 0,5 M NaOH), Regeneration Puffer B (0,05 % Tween 20), Regeneration Puffer C (0,05 % Tween 20, 100 mM EDTA, pH 8), Regeneration Puffer B, Regeneration Puffer D ( 0,05 % Tween 20, 100 mM NiSO4, pH 8), Regeneration Puffer B und Regeneration Puffer E (0,5 % Tween 20, 30 % Ethanol, pH 7).
- Speichern Sie die recycelten Perlen in Regeneration Puffer E bei 4 ° c
- Sammeln Sie die verwendeten magnetischen Kügelchen mit dem MPC zu und entsorgen Sie überstand.
(5) Seiten-Analyse
-
Vorbereitung der Probe
Hinweis: Nach dem Ausführen des Ni-NTA magnetische-Perle-basierte Tests, können der Assay-Überstände seitenweise analysiert werden. In diesem Fall überspringen Sie Schritte 5.1.1 und 5.1.2. Es ist jedoch auch möglich, die gereinigten fluoreszierende Substrate-Lösung und/oder deren Spaltung Bruchstücke nach der Verdauung-Lösung mit der Protease von Interesse zu analysieren. In diesem Fall weiterhin das Protokoll mit Schritt 5.1.1.- Bereiten Sie die gereinigten fluoreszierende Substratlösung gemäß Schritt 4.5.1.
- Führen Sie in Lösung Verdauung.
- Austausch von Elution Buffer mit Dekolleté-Puffer in der 0,5 mL 10K Ultrafiltration Gerät und Aliquot der Proben in 1,5 mL Mikrozentrifugenröhrchen verdaut werden.
Hinweis: für die Seitenanalyse, wir regelmÄÑig 68 µL jedes Substrat, sondern die Anzahl der Probenröhrchen und das Volumen der Substratlösung regelmÄÑig werden nach den einzelnen experimentelles Design optimiert werden kann. - Die Proben der Enzymlösung hinzugefügt werden.
Hinweis: Für die Seitenanalyse wir 2 µL von HIV-1-PR, wie beschrieben von Bozóki Et Al.14, aber die Lautstärke kann entsprechend der individuellen experimentelles Design optimiert werden. Es wird empfohlen, Spaltung Puffer zu lösen oder verdünnen Sie das Enzym zu verwenden. - Inkubieren Sie die Proben nach den Versuchsplan.
Hinweis: Für die Seitenanalyse inkubiert wir das Reaktionsgemisch für 45 min bei 37 ° C, aber die Inkubationszeit und Temperatur entsprechend der Versuchsplanung eingestellt werden müssen. - Kündigen Sie die Reaktion durch Schritt 5.1.3.
- Austausch von Elution Buffer mit Dekolleté-Puffer in der 0,5 mL 10K Ultrafiltration Gerät und Aliquot der Proben in 1,5 mL Mikrozentrifugenröhrchen verdaut werden.
- Seite die Probe vorbereiten.
Hinweis: Die Leuchtstoff-Substrat-haltigen Proben können für die Seite durch eine nondenaturing oder eine denaturierenden vorbereitet werden. Folgen Sie für die Verwendung von nondenaturing oder denaturierenden Bedingungen Schritt 5.1.3.1 oder 5.1.3.2, beziehungsweise.- Bereiten Sie eine nondenatured Probe: Mischen Sie 30 µL der Probe mit 6 µL 6 x nondenaturing Probenbeladung Puffer (300 mM Tris, 20 % Glycerin, 0,05 % Bromophenol blue, pH 6.8).
- Bereiten Sie eine denaturierte Probe: Mischen Sie 30 µL der Probe mit 6 µL 6 x denaturierenden Probenbeladung Puffer (300 mM Tris, 20 % Glycerin, 0,05 % Bromophenol blue, 12 % SDS, 100 mM β-Mercaptoethanol, pH 6.8), und die Proben bei 95 ° C für 10 min erwärmen.
-
SDS-PAGE-Analyse
Hinweis: Optional, nur nondenatured (vorbereitet in Schritt 5.1.3.1) Proben analysiert werden, kann eine native Seite auch durchgeführt werden. In diesem Fall überspringen Sie Abschnitt 5.3.- Bereiten Sie eine SDS-Polyacrylamid-Gel (Einsatz 14 % trennen und 4 % Stapeln Gel) und füllen Sie den Tank mit Elektrophorese Puffer (2,5 mM Tris, 19,2 mM Glycin, 0,01 % SDS).
- Fügen Sie die Proben (in Schritt 5.1.3.1 oder 5.1.3.2) in die Vertiefungen ein Polyacrylamid-Gel und führen Sie Elektrophorese bei 120 V Spannung.
- Entfernen Sie die Gel-Kassette aus dem laufenden Modul und legen Sie das Gel in einem Waschmaschinenbottich.
Hinweis: Nondenatured Proben sind bereits in das Gel, sogar mit dem bloßen Auge oder mit einem DRBT sichtbar.
-
In-Gel-Renaturierung und Erkennung von fluoreszierenden Proteinen
Hinweis: Um die fluoreszierende Proteine in denaturierten Proben (in Schritt 5.1.3.2) auf die DRBT zu erkennen, muss SDS aus dem Gel, teilweise Renaturierung der Proteine ausgewaschen werden.- Das Gel ~ 100 mL destilliertes Wasser hinzu und spülen Sie das Gel mindestens 30 Minuten.
Hinweis: Um die SDS-Entfernung zu verbessern, erneuern Sie das Wasser alle 10 Minuten oder Spülen Sie bis zu 60 min. - Visualisieren Sie die fluoreszierende Proteine, mithilfe einer DRBT oder durch UV-Bildgebung.
- Das Gel ~ 100 mL destilliertes Wasser hinzu und spülen Sie das Gel mindestens 30 Minuten.
-
Konventionelle Coomassie-Färbung des Gels
- Färben Sie das Gel mit Coomassie Brilliant blauen Farbstoff, nonfluorescent Proteine zu visualisieren.
Ergebnisse
Abbildung 1A zeigt den schematischen Aufbau der ein repräsentatives Leuchtstoffröhren rekombinantes Protein-Substrat, das auf seine spezifische Spaltung Website Abfolge von HIV-1 PR verarbeitet werden kann. Abbildung 1 b stellt die Substrat-Produktion und ihre Einsatzmöglichkeiten in Protease Assays, einschließlich Ni-NTA magnetischer Wulst-basierende Assays und/oder Seite.
Um zuverlässige Daten von Fluorimetry zu erhalten, ist eine Kalibrierung erforderlich, um die Mengen an fluoreszierenden Substrate und Spaltprodukte zu bestimmen. Hierzu die Fluoreszenz Intensitätswerte der verschiedenen Substrate in den verschiedenen Pufferbedingungen gemessen werden müssen und ihre Konzentrationen im definierten Konzentrationsbereich (Abbildung 3) korreliert werden müssen. Die Neigungswerte die Kalibrierkurven können angewendet werden, um die Mengen an Substraten und Spaltprodukte in der Assay-Proben zu bestimmen. Die Pisten der Kalibrierkurven sind unabhängig von der Spaltung Website Sequenzen in den Substraten (Tabelle 11) eingefügt und können für eine Reihe von Substraten verschmolzen auf die gleiche Art von fluoreszierenden Proteins genutzt werden. Zoom-in Graphen sind für alle lineare Regressionen, die unteren Konzentrationsbereichen sowie zu vergrößern (Abbildung 3) dargestellt. Es ist wichtig zu beachten, dass die Kalibrierung muss sorgfältig durchgeführt werden, da eine ordnungsgemäße Verteilung der Datenpunkte für eine zuverlässige Kalibrierung erforderlich ist. Aus diesem Grund wird zweifach serielle Verdünnung angewendet, um Vorbereitung der Proben für die Kalibrierung, weil der R-2 -Wert zeigt eine gute Korrelation zwischen der Konzentration des fluoreszierenden Proteins und Fluoreszenz nur dann, wenn eine ausreichende Anzahl von Datenpunkten wurden zur Deckung des gesamten Konzentrationsbereichs. Darüber hinaus können experimentellen Fehler sehr beeinflussen die Genauigkeit der Kalibrierung; so kann auch eine grafische Auswertung der Regression Linien erforderlich sein.
Eine Vielzahl von enzymatischen Messungen erfolgt durch die Protease Assays, einschließlich einer Untersuchung der Wirkung von der Substratkonzentration auf die Geschwindigkeit der Reaktion (Abb. 4A). Durch nicht-lineare Regression können die Daten zur Bestimmung Enzym kinetische Parameter (z. B. Vmax und Km) verwendet werden. Eine unzureichende Wulst Federung und Streuung und eine falsche Reaktion Kündigung verursachen suboptimale Ergebnisse (Abbildung 4 b), die sich nicht für die Berechnung der kinetischen Werte zuverlässig Enzym eignen.
Eine Abhängigkeit von der Produktbildung pünktlich kann (zum Beispiel bei der Optimierung der Spaltung Reaktionsparameter) Assay (Abb. 5A) bestimmt. Enzym-Aktivität im Beisein eines Inhibitors kann auch untersucht (Abb. 5 b) für die Bestimmung der aktiven Enzym Konzentration und hemmenden konstant. Mit der gleichen Methode, können Effekte von anderen Inhibitoren auch von den Assay untersucht.
Die Protease-Assay ist nützlich, wenn untersuchen die Auswirkungen des pH-Wertes auf Enzym-Aktivität. Abbildung 6A stellt die Abhängigkeit der Enzymaktivität von pH am Beispiel des TEV PR, hat einen breiten optimale pH-Bereich (pH 6-9). Wenn die pH-Abhängigkeit der Enzymaktivität studiert (oder Enzyme mit einer optimalen sauren pH-Wert gemessen werden müssen), ist es notwendig zu berücksichtigen, dass die Affinität Bindung des rekombinanten Substrate, die Perlen im leicht sauren pH-Wert eingeschränkt sein kann. Eine erhöhte Dissoziation der Substrate aus Perlen (Abb. 6 b) verursachen eine Verzerrung der Untersuchungsergebnisse. Um die spontane Substrat Dissoziation von Perlen zu betrachten, die Messwerte für Reaktion Proben von denen B-Proben korrigiert werden müssen.
Abbildung 7 zeigt, dass die nondenatured fluoreszierende Proteine im Gel anhand ihrer Farbe unterschieden werden können mit blauem Licht Durchleuchtung (Abbildung 7A). Wenn die Bestimmung der Molmassen der Substrate/Spaltung Fragmente notwendig ist, kann denaturierenden Bedingungen auch für die Probenvorbereitung, verwendet werden, da fluoreszierende Proteine können teilweise renaturierte im Gel, und durch UV-Beleuchtung erfasst werden können (Abb. 7 b) oder durch Coomassie-Färbung (Abbildung 7). Wenn die R-Proben analysiert werden, sind nur die C-terminale Spaltprodukte sichtbar (Abbildung 7), während die N-terminale Spaltung Fragmente und die uncleaved Substrate die Perlen befestigt bleiben. Gelegentlich, Proteine können teilweise denaturiert werden, trotz Verwendung von nondenaturing Bedingungen (Abbildung 7), und zwar die nondenatured Proteine reichlicher, denaturierte Formen sind auch in der Probe nachweisbar. Dieses Phänomen hat keinen Einfluss auf die Erkennung der proteolytischen Spaltung aber muss bei quantitativen Densitometrie nondenatured Proben in Betracht kommen.
Obwohl die detaillierte Beschreibung nur für einen 2-mL-Tube-basierte Assay angezeigt wird, kann der Assay angepasst für eine 96-Well-Platte-basiertes System (Abbildung 8), werden in unserem Labor (nicht dargestellt) bereits erfolgreich getestet wurde. Die 96-Well-Platte angepasst-Format ist voll kompatibel mit der fluorimetrischen und elektrophoretischen Analysen, sowie, und die gewonnenen Daten können auch basierend auf den in diesem Artikel beschriebenen Methoden ausgewertet werden.
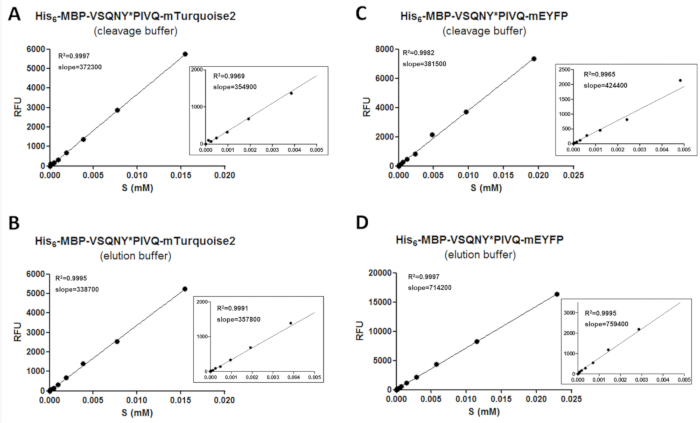
Abbildung 3 : Kalibrierkurven. Repräsentative Substrat Kalibrierkurven werden demonstriert am Beispiel der beiden rekombinanten Substrate verschmolzen zu verschiedenen C-terminale fluoreszierende Tags: (A und B) seine6- MBP-VSQNY * PIVQ-mTurquoise2 und (C und D ) Seine6- MBP-VSQNY * PIVQ-mEYFP. Zoom-in Zahlen sind auch gezeigt, dar, die lineare Regression der Datenpunkte in der 0-0,005 mM Substrat Konzentrationsbereich. Bitte klicken Sie hier für eine größere Version dieser Figur.
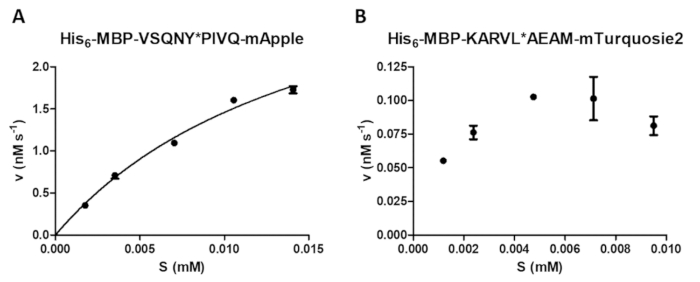
Abbildung 4 : Ermittlung der kinetischen Parameter Enzym. Substrat-abhängige kinetische Messungen wurden von HIV-1-PR (an eine aktive Endkonzentration von 41,2 nM). Die Anfangsgeschwindigkeit Werte wurden gegen die Substratkonzentration aufgetragen und eine nicht-lineare Regressionsanalyse Michaelis-Menten wurde durchgeführt. Die Fehlerbalken darzustellen SD (n = 2). (A) A repräsentatives optimales Ergebnis zeigt sich am Beispiel von seinem6- MBP-VSQNY * PIVQ-mApple Fusion Protein Substrat. (B) ein repräsentatives suboptimales Ergebnis zeigt sich auch für seine6- MBP-KARVL * AEAM mTurquoise2 Substrat, wo die Einstellung der richtigen Substrat-Konzentrationen problematisch wegen eine ungenügende Homogenisierung der Vorratslösung SAMB war , während relativ hohe Fehler durch falsche Reaktion Terminierung verursacht wurden. Bitte klicken Sie hier für eine größere Version dieser Figur.
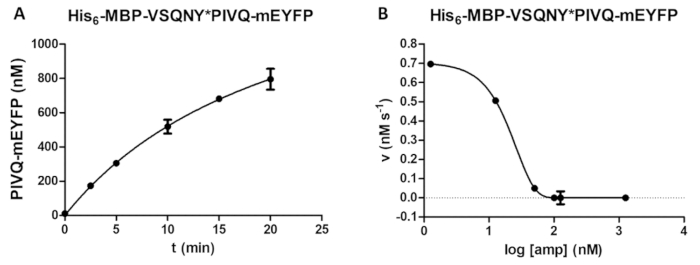
Abbildung 5 : Zeitverlauf und hemmenden Studie. (A) Seine 6- MBP-VSQNY * PIVQ-mEYFP rekombinanten Fusion Protein Substrat (auf eine Endkonzentration von 0,00326 mM) wurde von HIV-1 PR gespalten (an eine aktive Endkonzentration von 41,2 nM), und die Freisetzung von fluoreszierenden PIVQ mEYFP proteolytischen Fragmente wurde gemessen führen Sie eine Analyse der zeitlichen Verlauf. Die Messungen wurden an fünf verschiedenen Zeitpunkten durchgeführt. Die Fehlerbalken darzustellen SD (n = 2). (B) seine6- MBP-VSQNY * PIVQ-mEYFP wurde als Substrat (bei 0,0015 mM) verwendet, um festzustellen, die hemmende Wirkung von Amprenavir auf die Aktivität des HIV-1-PR (bei einer totalen Konzentration von 163,8 nM). Plotten, die Daten, die halbe maximale inhibitorische Konzentration (IC50) beurteilt werden konnte, und die aktive Enzym-Konzentration (eine aktive Endkonzentration von 41,2 nM) der angewandten HIV-1 PR könnte auch werden berechnet auf der Grundlage der Hemmung-Kurve. Die Fehlerbalken darzustellen SD (n = 3). Bitte klicken Sie hier für eine größere Version dieser Figur.
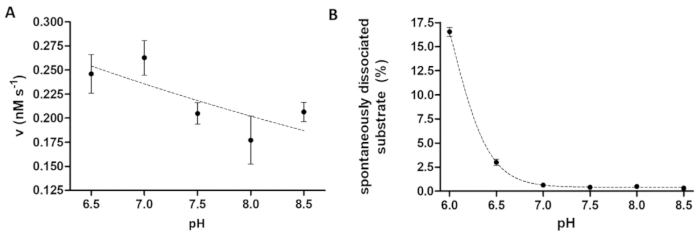
Abbildung 6 : Studium der Abhängigkeit der Enzymaktivität und spontane Substrat Dissoziation auf pH. (A) die seine6- MBP-VSQNY * PIVQ-mTurquoise2 Substrat (in 0,033 mM) wurde verwendet, um die Enzymaktivität der TEV PR messen (an eine totale Endkonzentration von 91.42 nM) im Dekolleté Polsterset einem anders pH-Wert zwischen 6,5-8,5-Bereich. Die Fehlerbalken darzustellen SD (n = 2). Die gezeichneten Daten wurde vorher14veröffentlicht. (B) anhand der relativen fluoreszierende Intensitätswerte der Substrat leer Proben, die spontane Spaltung seiner6- MBP-VSQNY * PIVQ-mTurquoise2 Substrat (0,033 mM) von der magnetischen Beads wurde mithilfe von Spaltung Puffer untersucht mit einem anders pH-Wert zwischen 6,0-8,5. Die gezeichneten Daten wurde vorher14veröffentlicht. Bitte klicken Sie hier für eine größere Version dieser Figur.
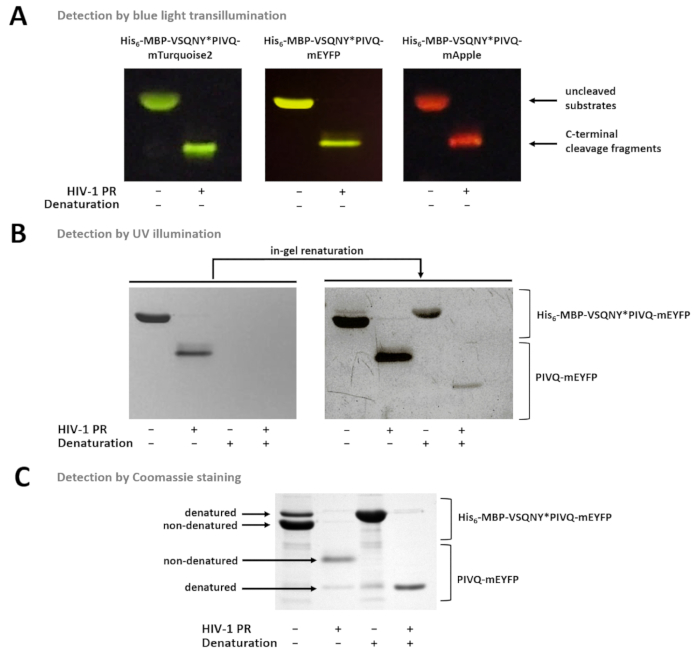
Abbildung 7 : Erkennung von Proteinen im Gel mit verschiedenen Methoden. (A) Uncleaved und HIV-1 PR verdaut Fusion Protein Substrate nach nondenaturing Probenvorbereitung wurden durch blaues Licht durchleuchtbaren nach SDS-PAGE visualisiert. Die Spaltung Reaktion erfolgte durch in-Lösung Verdauung. (B) unmittelbar nach der Seite nur nondenatured Proteine nachgewiesen werden konnte im Gel durch UV-Beleuchtung, während nach der Entfernung der SDS, der zuvor denaturiert fluoreszierende Proteine wurde teilweise renaturierte und nachweisbar. Die Proben wurden von der Überstände des Ni-NTA magnetische-Perle-basierte Tests vorbereitet. (C) Coomassie-Färbung kann auch für die Erkennung von Proteinen nach der Renaturierung in Gel verwendet werden. Die SDS-Gegenwart in der Gel-Mai der partiellen Denaturierung des nativen Proteins verursachen, aber in nativen Proben, die nondenatured Formen sind häufiger. Die Proben wurden von der Überstände des Ni-NTA magnetische-Perle-basierte Tests vorbereitet. Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 8 : 96-Well-Platte-basierte Anpassung der Assay-Plattform. (A) der Test durchgeführt werden kann, nicht nur in 2 mL-Tuben, sondern in den Vertiefungen einer 96-Well-Platte. Hier zeigen wir die schematische Darstellung für die Anwendung des Tests zu studieren die Spezifität des fiktiven Protease mithilfe einer Reihe von fluoreszierenden Substrate, enthalten Wildtyp (wt) oder mutierte (Mut-1-Mut-4) Spaltung Website Sequenzen. Für den Umgang mit magnetischen Beads, soll ein 96-Well kompatibel Magnetpartikel Konzentrator (MPC) in den Experimenten verwendet werden. Die angegebenen Mengen beziehen sich auf einen einzigen Brunnen. Um die Effizienz der Spaltung der verschiedenen Substrate zu vergleichen, kann Substrat Konvertierung aus dem Prozentsatz der Substrat-Blank-korrigierte RFU Werte der Reaktion Proben, wenn man bedenkt das Substrat-Blank-korrigierte RFU Werte der damit zusammenhängenden beurteilt werden Substrat Kontrollproben als 100. (B) nach Fluorimetry, die getrennten Überstände des Assays Proben auch von PAGE und die fluoreszierende Protein-Komponenten analysiert werden können direkt oder nach-Gel Renaturierung bei nondenaturing und Geruchsstoffen Probe analysiert werden können Vorbereitung, beziehungsweise. Die drei verschiedenen Assay Probentypen sind auch in jeder Abbildung illustriert: C = Substrat Control, B = Substrat leer und R = Reaktion. Substrat Kontrollproben sind im Elution Buffer, während das Substrat leer und die Reaktion Proben im Dekolleté Puffer. Bitte klicken Sie hier für eine größere Version dieser Figur.
| Puffer | Fluoreszierendes protein | CV % der Pisten (%) |
| Elution | mTurquoise2 | 6.04 |
| Dekolleté | 9.11 | |
| Elution | mApple | 10,92 |
| Dekolleté | 12.68 |
Tabelle 11: Koeffizient der Varianz (CV %) Werte von den Hängen des Substrat-Kalibrierkurven. Um zu testen ob die Fluoreszenz des rekombinanten Proteins Substrate der eingefügten Spaltstelle abhängt, wurden Kalibrierungen durch Reihe von mApple und mTurquoise2 verschmolzen Substrate (sechs Varianten für die einzelnen, mit verschiedenen Spaltstelle durchgeführt. Sequenzen von HIV-1 Protease), beide in Elutions- und Spaltung Puffern. Wir fanden, dass CV %-Werte der Piste unter 15 % auf jeden Fall, was bedeutet, dass eine einzelnes Substrat-Kalibrierung für die Bewertung der verschiedenen Messungen von Substrat-Varianten mit der gleichen fluoreszierende Beschriftung genutzt werden kann.
Diskussion
Aufgrund der intensiven industriellen und akademischen Untersuchungen von proteolytischen Enzymen und die ständige Nachfrage für rasche und kostengünstige HTS-kompatiblen Protease Assays Plattformen entsprechend, entwickelten eine magnetische-Perle-basierte fluoreszierende Protease wir Assay. Der Test basiert auf den Einsatz von rekombinanten Fusionsproteinen neuartige Alternativen zu den allgemein verwendeten synthetischen Peptid-Substrate werden können.
Im entwickelten Assay-Format sind die Fusion Protein Substrate an den Oberflächen der Ni-Chelat-beschichtete magnetische Agarose Korne immobilisiert. Die Substrat-Anlage ist durch die N-terminale seine6 Affinität Tag des Fusionsproteins, vorgesehen, die direkt an ein MBP-Tag zur Erleichterung der Faltung und erhöhen die Wasserlöslichkeit der Substrat-13verschmolzen wird. Das MBP folgt Spaltstellen TEV PR und eine Protease von Interesse. Ersteres kann dienen als eine Kontrolle Spaltstelle in der Probe, während letztere durch die Protease untersucht werden verarbeitet werden kann. Die Spaltstelle ist austauschbar; eine kurze DsDNA Sequenz Codierung für die Spaltstelle von Interesse kann in der flexiblen Klonen Kassette des Plasmids Ausdruck durch Unterbindung eingefügt werden. Die rekombinanten Fusionsproteinen enthalten einen hochstabile, Monomere fluoreszierendes Protein-Tag an die C-terminale, wodurch die Endpunkterkennung von dem Enzym befreit, fluoreszierende C-terminale Spaltprodukte frei, worauf proteolytische Spaltung ( Abbildung 1A). Gereinigten fluoreszierende intakt Substrate in verschiedenen Puffer gelöst sind auch für die Kalibrierung verwendet, um die Molaren Konzentrationen der Substrate und Spaltprodukte zu beurteilen. Darüber hinaus können nach Fluorimetry, der Assay-Komponenten durch SDS-PAGE, sowie analysiert werden. Beide Native (nondenatured) und denaturierte fluoreszierende Proteine visualisiert werden im Gel, unmittelbar nach der Elektrophorese oder nach nachfolgenden in Gel Renaturierung bzw.. Diese zusätzliche Verfahren in Kombination mit einer konventionellen Coomassie Brilliant Blue Färbung-Mai werden für die Überprüfung der Untersuchungsergebnisse (Abbildung 1 b) effizient genutzt.
Die Testverfahren besteht aus einfachen, leicht ausführen Schritte in einem Low-Volume-Format, die eine automatische Umgebung mit hohem Durchsatz vollständig angepasst werden können. Allerdings gelten unabhängig von der Durchführung des Tests, entweder manuell oder mit einem Automatisierungssystem, folgende Teile des Tests als entscheidend sein und benötigen besondere Aufmerksamkeit bei der Durchführung des Verfahrens. (i) Homogenität der magnetischen Wulst Lösung. Eine homogene magnetische Wulst-Lösung muss in der gesamten Assay, sowohl in der Reinigung und Waschschritte verwendet werden. Insbesondere hängt die Zuverlässigkeit der Protease Assays richtig Aliquotierung der Stammlösungen Substrat befestigt magnetischer Wulst (SAMB). Um die Wirksamkeit der Aufhängung und Dispersion zu erhöhen, empfiehlt es sich, die Perle-Konzentration zwischen 2 % und 10 % (V/V) festgelegt. Während der Probenvorbereitung, die Verwendung von Puffern ergänzt mit nichtionische Reinigungsmittel (z.B. Triton x-100 oder Tween 20) bis zu 2 % kann auch die Einhaltung der magnetische Beads Kunststoffoberflächen vermindern. Die Einhaltung der Perlen an den Wänden der Probenfläschchen kann vermieden werden, wenn die Perle Suspensionen sorgfältig auf die Böden der Durchstechflaschen statt an den Wänden der Probenröhrchen angewendet werden. Die Homogenität des magnetischen Kügelchen während der enzymatischen Reaktion ist auch kritisch und durch Schütteln kontinuierlich die Proben bei 600 u/min während der Inkubation gewährleistet werden kann. Perlen sind in runden oder flachen Kunststoff waren, richtig verteilt, während der Verwendung von V-Boden-Fläschchen nicht empfohlen wird. Ein suboptimales Ergebnis verursacht durch unsachgemäße Wulst Homogenisierung wird in Abbildung 4 bdargestellt. (II) Beendigung der Reaktion Proben. Ein weiterer Vorteil der Methode ist, dass die enzymatische Reaktion ohne den Einsatz von Denaturierung Wärmebehandlung oder jede potentiell störende chemische Arbeitsstoffe15beendet werden kann. Die Kündigung kann einfach durch die Trennung der magnetischen Beads aus dem Reaktionsgemisch, mit einem herkömmlichen Magnetpartikel Konzentrator erfolgen. Während der entfernten Reaktion Puffer das aktive Enzym und die generierten C-terminale fluoreszierende Spaltprodukte enthält, bleiben die uncleaved Substrate mit Perlen verbunden. Aufgrund des Vorhandenseins von das aktive Enzym im Puffer Reaktion muss das Trennung Verfahren für zuverlässige Endpunkterkennung sorgfältig durchgeführt werden. Vor der Platzierung der Probenfläschchen in der Konzentrator, empfiehlt es sich, eine kurze Drehung Zentrifugation gelten. Nach der Platzierung der Rohre in der Konzentrator bieten mindestens 15 s für die Perlen gesammelt werden. Leichte Bewegung des Separators hin- und Herbewegung kann die Sammlung der Perlen erleichtern. Bitte beachten Sie, dass während eine manuell durchgeführte Trennung der Kündigung in der Regel mehr Zeit als die Einleitung der Reaktionen dauert. Daher wird ein ca. 2 min registriert Verzögerung zwischen den Einweihungen empfohlen, wenn die gleiche Inkubationszeit auf alle Proben angewendet werden muss.
Das Prinzip des beschriebenen proteolytischen Assays ist relativ einfach; die Vielseitigkeit des Systems ist jedoch durch die flexiblen und stabilen Untergrund-Struktur gewährleistet. Individuelle Optimierung des Assays kann nur durch die Kompatibilität der Affinität Perlen mit angewandten Bedingungen, Reagenzien und Zusatzstoffe beschränkt werden. Im Einvernehmen mit der Hersteller-Protokoll fanden wir auch, dass die Affinität Bindung von Substraten, die Ni-NTA-Perle-Oberflächen bei pH ≤ 6,515erheblich schwächt. Daher, es wird empfohlen, Substrat leer Proben parallel zur Reaktion Proben gelten, und die Rate der spontanen Substrat Dissoziation muss bei der Auswertung der Ergebnisse berücksichtigt werden.
In den Fällen, wo magnetische-Perle-basierte Tests durch den Einsatz von Perle-inkompatible Komponenten oder einen niedrigen pH-Wert durchgeführt werden kann, kann auch in Lösung Verdauung der gereinigten rekombinante Substrate angewendet werden. In diesen Fällen die Reaktionsgemischen durch Elektrophorese analysiert werden können, und die Proteine im Gel basierend auf dem beschriebenen Protokoll visualisiert werden können. Um proteolytische Aktivität zu untersuchen, möglicherweise in Lösung Verdauung und in Gel Detektion der Proteine auch alternative Werkzeuge des Fluorimetry. Eine Neuheit des Systems entwickelten Substrat ist die Anwendung eines Schritts in Gel Renaturierung nach denaturierenden SDS-PAGE. Native (nondenatured) fluoreszierende Proteine ihre Fluoreszenz während der Elektrophorese behalten, ist die fluoreszierende Eigenschaft auf Denaturierung (Abb. 7 b) abgeschafft. Die Fluoreszenz von denaturierten Proteinen kann jedoch teilweise durch die Beseitigung der SDS aus dem Gel wiederhergestellt werden. So macht eine Trennung der Reaktionskomponenten mit denaturierenden Bedingungen nicht nur die Fluoreszenz-basierte, sondern die molekularen Gewicht basierende Identifizierung möglich. Ein weiterer Vorteil der Fluoreszenz in-Gel-Detektion im Vergleich zur Analyse eines Gels Coomassie gefärbt ist, dass die (Native oder renaturierte) fluoreszierende Proteine im Gel basierend auf ihre Fluoreszenz leicht identifiziert werden können (siehe Abbildung 7). Dies kann wichtig sein, wenn Spaltung Reaktionen in Proben mit nonfluorescent Verunreinigungen durchgeführt werden oder Proteine die Molmassen von einander sehr ähnlich.
Protease Assays mit ähnlich gestalteten Substrate wurden bereits vorher veröffentlicht8,9,10, und obwohl die Spaltstelle von Interesse in diesen Fällen auch zwischen einer Affinität Tag befand und eine fluoreszierenden Proteins, das Testsystem präsentiert hier wiederholt nicht nur die beschriebenen Ideen aber verbindet die unterschiedlichen Vorteile der bisherigen Plattformen und schließt sie auch mit weiteren Verbesserungen: (i) die Nutzung von einem MBP Fusion Partner, (Ii) die Präsenz ein TEV PR Kontrolle Spaltstelle, Iii) die Nutzung der neu entwickelte Monomeren FPs und iv) die Anwendung eines einzigartigen Substrat Kalibrierung. Der Test selbst wurde speziell für Enzym Spezifität und kinetische Untersuchungen an einem sicheren, Zeit- und kosteneffiziente Art und Weise, ohne die Notwendigkeit für teure Instrumente nützlich. Die Methode zielt darauf ab, einen geeigneten und erschwinglichen Werkzeug für beide industriellen und akademischen Forschungszwecke sein. Aufgrund der Flexibilität der "Klonen Kassette" des Plasmids Ausdruck möglicherweise das System für die schnelle und kostengünstige Erzeugung von rekombinanten Substrat Bibliotheken geeignet. Der hierin beschriebene Test eignet sich für die Umsetzung der Substratspezifität, Enzym Mutagenese, machbar und Hemmung studiert und bieten auch ein alternatives Instrument, um Enzym Kinetik durchführen. Die Assay-Plattform (aus Bakterienzelle Unterbrechungen für die Ermittlung der kinetischen Parameter) kann zu einer HTS - und Automatisierung-basierte Umgebung angepasst werden und gegebenenfalls im industriellen Protease-Inhibitor-Screening und/oder antivirale Medikament angewandt werden Entwicklung. Darüber hinaus ist die Anpassung des Assays für wettbewerbsfähige Proteolyse auch zukünftig Umfang unseres Labors. In solch einem wettbewerbsfähigen Assay verschmolzen zwei verschiedene Substrate-jeder mit einer unterschiedlichen Spaltstelle, a verschiedene C-terminale fluoreszierende Tag-werden gleichzeitig in die gleiche Reaktion der Spaltung um zu untersuchen, die Bevorzugung von den untersuchten verwendet werden sollen Enzym für die gegebenen Ziel-Sequenzen. Darüber hinaus wird die Verwendung einer 96-Well-Platte angepasst-Assay-Form (Abbildung 8) auch für Mutation Screening optimiert durch eine Reihe von Substraten mit modifizierten Spaltung Website Sequenzen bei Cystein-Proteasen.
Offenlegungen
Die Autoren haben nichts preisgeben.
Danksagungen
Diese Arbeit wurde teilweise vom GINOP-2.3.2-15-2016-00044 "teaming PHARMPROT" Project unterstützt und auch, finanziert durch die institutionelle Excellence Hochschulprogramm des Ministeriums der menschlichen Fähigkeiten in Ungarn, im Rahmen der Biotechnologie-thematischen Programm der Universität Debrecen. Die Autoren sind dankbar für die Mitglieder des Labor für Retroviren Biochemie für ihre wissenschaftliche Hilfe während der Assay-Entwicklung und auch für ihre Geduld während der Dreharbeiten der Assay (insbesondere an Norbert Kassay, Krisztina Joóné Matúz und Vanda Toldi Wer im Hintergrund des Videos angezeigt werden). Die Autoren möchten auch besonderer Dank Gedeon Richter Plc., vor allem Dr. Zoltán Urbányi dafür, dass Beáta Bozókis Arbeit in der Abteilung für Biochemie und Molekularbiologie als Gastwissenschaftler sagen. Die Autoren möchten auch ihre Dankbarkeit György Zsadányi, Balázs Tőgyi, Balázs Pöstényi und Zoltán Király aus dem Multimedia und E-Learning Technical Center der Universität Debrecen für die professionelle Unterstützung im Bereich Audio und Video zu verlängern Produktion.
Materialien
| Name | Company | Catalog Number | Comments |
| 10K Amicon tubes | Merck-Millipore | UFC501096 | |
| 2-Mercaptoethanol (β-ME) | Sigma-Aldrich (St Louis, MO, USA) | M6250 | |
| 40% Acrylamide/Bis solution 37.5:1 | Bio-Rad | 1610148 | |
| Acetic acid | Merck | 100063 | |
| Agarose | SERVA | 11404.04 | |
| Alpha Imager HP gel documentation system | ProteinSimple | ||
| Ammonium persulfate (APS) | Sigma-Aldrich (St Louis, MO, USA) | A3678 | |
| Ampicillin sodium salt | Sigma-Aldrich (St Louis, MO, USA) | A9518 | |
| Beckman Coulter Allegra X-22 centrifuge | Beckman Coulter | 392185 | |
| Black half-area plates | Greiner bio-One | 675086 | |
| Bromophenol blue | Sigma-Aldrich (St Louis, MO, USA) | B0126 | |
| CutSmart buffer (10x) | New England Biolabs | B7204S | For plasmid linearization (step 1.1.1) |
| Dark Reader transilluminator | Clare Chemical Research | DR-45M | |
| DNase I | New England Biolabs | M0303L | |
| Dynamag-2 magnetic particle concentrator | Thermo Fischer Scientific | 12321D | |
| Escherichia coli BL21(DE3) competent cells | Thermo Fischer Scientific (Invitrogen) | C600003 | |
| Ethanol | Merc-Millipore | 100983 | |
| Ethylenediaminetetraacetic acid (EDTA) | Sigma-Aldrich (St Louis, MO, USA) | 798681 | |
| Gel Loading Dye, Purple (6X) | New England Biolabs | B7024S | |
| Glycerol | Merck | 356350 | |
| Glycine | Sigma-Aldrich (St Louis, MO, USA) | G7126 | |
| High-Speed Plasmid Mini Kit | GeneAid | PD300 | |
| Imidazole | Sigma-Aldrich (St Louis, MO, USA) | 56750 | |
| Isopropyl β-D-1-thiogalactopyranoside (IPTG) | Thermo Fischer Scientific (Invitrogen) | AM9464 | |
| Jouan CR 412 centrifuge | Jouan | CR412 | |
| Labinco LD-76 Rotator | Labinco | 7600 | |
| Luria-Bertani (LB) broth | Sigma-Aldrich (St Louis, MO, USA) | L3022 | |
| Lysozyme | Sigma-Aldrich (St Louis, MO, USA) | L6876 | |
| Magnesium chloride | Scharlau | MA0036 | |
| MERCK eurolab ultrasonic bath | MERCK | USR54H | |
| Millifuge Eppendorf spin centrifuge | Millipore | CT10 | |
| Mini-PROTEAN 3 Electrophoresis Cell | Bio-Rad | ||
| N,N,N′,N′-Tetramethylethylenediamine (TEMED) | Sigma-Aldrich (St Louis, MO, USA) | T9281 | |
| NanoDrop 2000 | Thermo Fischer Scientific | ||
| NheI-HF restriction endonuclease | New England Biolabs | R3131L | |
| Nickel(II) sulfate (NiSO4) | Sigma-Aldrich (St Louis, MO, USA) | 656895 | |
| Ni-NTA magnetic agarose beads | Qiagen | 36113 | |
| Orbital shaker | Biosan | OS-20 | |
| PacI restriction endonuclease | New England Biolabs | R0547L | |
| PageBlue Protein Staining Solution | Thermo Fischer Scientific | 24620 | |
| Phenylmethanesulfonyl-fluoride (PMSF) | Sigma-Aldrich (St Louis, MO, USA) | P7626 | |
| Protein Lobind Micro-centrifuge tubes | Eppendorf | 22431102 | |
| QIAquick Gel Extraction Kit | QIAGEN | 28704 | |
| Snijders Press-to-Mix shaker | Gemini | 34524 | |
| Sodium-acetate trihydrate | Sigma-Aldrich (St Louis, MO, USA) | S7670 | |
| Sodium-chloride | Sigma-Aldrich (St Louis, MO, USA) | S9888 | |
| Sodium-hydroxide | Sigma-Aldrich (St Louis, MO, USA) | S5881 | |
| SYBR Green I Nucleic Acid Gel Stain | Thermo Fischer Scientific | S7563 | |
| T4 DNA ligase | Promega | M180A | |
| Thermo shaker | Biosan | TS-100 | with SC-24 accessory block |
| Tris | Sigma-Aldrich (St Louis, MO, USA) | T1503 | |
| Tween 20 | Sigma-Aldrich (St Louis, MO, USA) | P2287 | |
| WALLAC VICTOR2 1420 multilabel counter | Wallac Oy, Turku, Finland |
Referenzen
- Meldal, M. Smart Combinatorial Assays for the Determination of Protease Activity and Inhibition. Molecular Informatics. 24 (10), 1141-1148 (2005).
- Rao, M. B., Tanksale, A. M., Ghatge, M. S., Deshpande, V. V. Molecular and biotechnological aspects of microbial proteases. Microbiology and Molecular Biology Reviews. 62 (3), 597-635 (1998).
- Zhang, G., Sittampalam, G. S., et al. Protease assays. The Assay Guidance Manual. , (2012).
- Woelcke, J., Hassiepen, U., Chen, T. Fluorescence-based biochemical assay formats. A Practical Guide to Assay Development and High-Throughput Screening in Drug Discovery. , (2010).
- Richardson, P. L. The determination and use of optimized protease substrates in drug discovery and development. Current Pharmaceutical Design. 8 (28), 2559-2581 (2002).
- Diamond, S. L. Methods for mapping protease specificity. Current Opinion in Chemical Biology. 11 (1), 46-51 (2007).
- Schilling, O., Overall, C. M. Proteome-derived, database-searchable peptide libraries for identifying protease cleavage sites. Nature Biotechnology. 26 (6), 685-694 (2008).
- Askin, S. P., Morin, I., Schaeffer, P. M. Development of a protease activity assay using heat-sensitive Tus-GFP fusion protein substrates. Analytical Biochemistry. 415 (2), 126-133 (2011).
- Chaparro-Riggers, J. F., Breves, R., Michels, A., Maurer, K. H., Bornscheuer, U. A GFP based assay for the determination of hydrolytic activity and substrate specificity of subtilisins under washing conditions. Journal of Molecular Catalysis B: Enzymatic. 35, 74-77 (2005).
- Patel, D., Frelinger, J., Goudsmit, J., Kim, B. In vitro assay for site-specific proteases using bead-attached GFP substrate. Biotechniques. 31 (5), 1194-1198 (2001).
- Branchini, B. R., et al. Sequential bioluminescence resonance energy transfer-fluorescence resonance energy transfer-based ratiometric protease assays with fusion proteins of firefly luciferase and red fluorescent protein. Analytical Biochemistry. 414 (2), 239-245 (2011).
- Zhou, C., Yan, Y., Fang, J., Cheng, B., Fan, J. A new fusion protein platform for quantitatively measuring activity of multiple proteases. Microbial Cell Factories. 13 (1), 44 (2014).
- Fox, J. D., Waugh, D. S. Maltose-binding protein as a solubility enhancer. Methods in Molecular Biology. 205, 99-117 (2003).
- Bozóki, B., et al. A recombinant fusion protein-based, fluorescent protease assay for high throughput-compatible substrate screening. Analytical Biochemistry. 540-541, 52-63 (2018).
- Mótyán, J. A., Miczi, M., Bozóki, B., Tözsér, J. Data supporting Ni-NTA magnetic bead-based fluorescent protease assay using recombinant fusion protein substrates. Data in Brief. 18, 203-208 (2018).
- Balleza, E., Kim, J. M., Cluzel, P. Systematic characterization of maturation time of fluorescent proteins in living cells. Nature Methods. 15, 47-51 (2018).
- Hebisch, E., Knebel, J., Landsberg, J., Frey, E., Leisner, M. High variation of fluorescence protein maturation times in closely related Escherichia coli strains. PLoS One. 8, e75991 (2013).
- Kapust, R. B., et al. Tobacco etch virus protease: mechanism of autolysis and rational design of stable mutants with wild-type catalytic proficiency. Protein Engineering, Design and Selection. 14, 993-1000 (2001).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten