Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Automatisierte 3D-Optische Kohärenztomographie zur Aufklärung der Biofilmmorphogenese über große räumliche Skalen
In diesem Artikel
Zusammenfassung
Mikrobielle Biofilme bilden komplexe Architekturen an Interphasen und entwickeln sich zu stark maßstabsabhängigen Raummustern. Hier stellen wir ein experimentelles System (Hard- und Software) zur automatisierten Erfassung von 3D-Datensätzen zur optischen Kohärenztomographie (OCT) vor. Dieses Toolset ermöglicht die nichtinvasive und multiskalige Charakterisierung der Biofilmmorphogenese in Raum und Zeit.
Zusammenfassung
Biofilme sind ein sehr erfolgreicher mikrobieller Lebensstil und setzen sich in einer Vielzahl von Umwelt- und Ingenieurumgebungen durch. Das Verständnis der Biofilmmorphogenese, d. h. der strukturellen Diversifikation von Biofilmen während der Gemeinschaftsversammlung, stellt eine bemerkenswerte Herausforderung über räumliche und zeitliche Skalen hinweg dar. Hier präsentieren wir ein automatisiertes Biofilm-Bildgebungssystem auf Basis der optischen Kohärenztomographie (OCT). OCT ist eine neue bildgebende Technik in der Biofilmforschung. Die Datenmenge, die derzeit erfasst und verarbeitet werden kann, behindert jedoch die statistische Schlussfolgerung großer Muster in der Biofilmmorphologie. Das automatisierte OCT-Bildgebungssystem ermöglicht die Abdeckung großer räumlicher und erweiterter zeitlicher Skalen des Biofilmwachstums. Es kombiniert ein kommerziell erhältliches OCT-System mit einer Robotik-Positionierungsplattform und einer Reihe von Softwarelösungen zur Steuerung der Positionierung der OCT-Scansonde sowie zur Erfassung und Verarbeitung von 3D-Biofilm-Imaging-Datensätzen. Dieses Setup ermöglicht die in-situ und nicht-invasive automatisierte Überwachung der Biofilmentwicklung und kann weiterentwickelt werden, um DIE OCT-Bildgebung mit Makrofotografie und Mikrosensorprofilierung zu koppeln.
Einleitung
Biofilme sind eine sehr erfolgreiche mikrobielle Lebensstilanpassung und diese interphasenassoziierten und matrixumschlossenen Gemeinschaften von Mikroorganismen dominieren das mikrobielle Leben in natürlichen und industriellen Umgebungen1,2. Dort bilden Biofilme komplexe Architekturen, wie längliche Streamer3, Ripples4 oder pilzartige Kappen5 mit wichtigen Folgen für Biofilmwachstum, strukturelle Stabilität und Stressresistenz6. Während viel über die strukturelle Differenzierung von Biofilmen aus der Arbeit an Mono-Arten-Kulturen gelernt wurde, die in Miniatur-Flusskammern angebaut werden, sind die meisten Biofilme hochkomplexe Gemeinschaften, die oft Mitglieder aller Lebensbereiche umfassen6. Diese komplexen Biofilme als mikrobielle Landschaften zu würdigen7 und zu verstehen, wie Biofilmstruktur und -funktion in komplexen Gemeinschaften interagieren, steht damit an vorderster Front der Biofilmforschung.
Ein mechanistisches Verständnis der Morphogenese komplexer Biofilme als Reaktion auf Umwelthinweise erfordert sorgfältig konzipierte Experimente in Verbindung mit räumlich und zeitlich aufgelösten Beobachtungen der physikalischen Struktur des Biofilms über relevante Skalen8. Die zerstörungsfreie Beobachtung des Biofilmwachstums in experimentellen Systemen wurde jedoch durch logistische Zwänge wie die Notwendigkeit, Proben (z. B. in ein Mikroskop) zu verschieben, die häufig die empfindliche Biofilmstruktur schädigen, stark eingeschränkt.
Das hier vorgestellte Protokoll führt ein vollautomatisiertes System auf der Grundlage der optischen Kohärenztomographie (OCT) ein, das die in situ, nicht-invasive Überwachung der Biofilmmorphogenese im Mesoscale (mm-Bereich) ermöglicht. OCT ist eine neue bildgebende Technik in der Biofilmforschung mit Anwendungen in der Wasseraufbereitung und Biofouling-Forschung, Medizin9 und Strömungsökologie10. In OCT wird eine lichtarme Lichtquelle in einen Proben- und Referenzarm aufgeteilt; die Interferenz des vom Biofilm reflektierten und gestreuten Lichts (Probenarm) und das Licht des Referenzarms wird analysiert. Eine Reihe von axialen Intensitätsprofilen (A-Scans), die tief aufgelöste Strukturinformationen enthält, wird erfasst und zu einem B-Scan (einem Querschnitt) zusammengeführt. Eine Reihe benachbarter B-Scans bildet den endgültigen 3D-Volume-Scan10. OCT bietet eine seitliche optische Auflösung im Bereich von ca. 10 m und eignet sich daher gut zur Untersuchung der meskopischen strukturellen Differenzierung von Biofilmen10,12. Eine ausführlichere Beschreibung des ÜLG finden Sie unter Drexler und Fujimoto13und Fercher und Kollegen14. Obwohl das Sichtfeld eines einzelnen OCT-Xy-Scans bis zu Hunderte von Quadratischen Mikrometern erreicht, können größere Muster nicht mit Hilfe von OCT in einem einzigen Scan quantifiziert werden. In Bezug auf Biofilme in natürlichen Lebensräumen wie Bächen und Flüssen schränkt dies derzeit unsere Fähigkeit ein, die Biofilmmorphogenese in Schuppen zu bewerten, die der physikalischen und hydraulischen Vorlage des Lebensraums entsprechen.
Um diese räumlichen Grenzen zu überschreiten und OCT-Scans automatisch zu erfassen, wurde eine Spektral-Domain-OCT-Bildsonde auf einem 3-Achsen-Positionierungssystem montiert. Die Installation ermöglicht die Erfassung mehrerer OCT-Scans in einem überlappenden Mosaikmuster (Kachelscan), wodurch die tomographische Abbildung von Oberflächen bis 100 cm2effektiv erreicht wird. Darüber hinaus ermöglicht die hohe Positionierpräzision dieses Systems eine zuverlässige Überwachung des Wachstums und der Entwicklung von Biofilmmerkmalen an bestimmten Standorten bei Langzeitexperimenten. Das System ist modular aufgebaut und einzelne Komponenten (z.B. Positioniergerät und OCT) der Anlage können als Standalone-Lösungen eingesetzt oder flexibel kombiniert werden. Abbildung 1 gibt einen Überblick über die Hard- und Softwarekomponenten der Installation.
Das System wurde mit einer handelsüblichen GRBL-gesteuerten CNC-Positioniervorrichtung (Materialtabelle )getestet. Die Betriebsabstände dieser speziellen Positionierplattform betragen 600 x 840 x 140 mm, mit einer herstellergenauen Genauigkeit von +/- 0,05 mm und einer programmierbaren Auflösung von 0,005 mm. GRBL ist eine Open-Source-(GPLv3-Lizenz), eine leistungsstarke Bewegungssteuerung für CNC Geräte. Daher sollte jedes GRBL-basierte (Version > 1.1) Positionierungsgerät mit den hier vorgestellten Richtlinien und Softwarepaketen kompatibel sein. Darüber hinaus konnte die Software mit wenigen Modifikationen an andere Schrittmotorregler mit STEP-DIR-Eingangstyp angepasst werden.
Das OCT-Gerät zur Beurteilung der Leistung des Systems (Tabelle der Materialien) verfügt über eine lichtarme Lichtquelle mit einer mittleren Wellenlänge von 930 nm (Bandbreite = 160 nm) und einstellbarer Referenzarmlänge und -intensität. Im hier vorgestellten Beispiel wurde auch ein Tauchadapter zum Eintauchen der OCT-Sonde in fließendes Wasser verwendet (Tabelle der Materialien). Das hier für die automatisierte OCT-Scanerfassung entwickelte Softwarepaket hängt entscheidend vom SDK zusammen mit dem spezifischen OCT-System ab, jedoch sollten OCT-Systeme desselben Herstellers mit unterschiedlichen Scanlinsen und zentralen Wellenlängen leicht kompatibel.
Das GRBL-Gerät wird von einem Webserver gesteuert, der auf einem Einplatinencomputer installiert ist (Abbildung 1). Dadurch wird die Fernsteuerung des Geräts von jedem Computer mit lokalem Netzwerk oder Internetzugang gewährt. Das OCT-Gerät wird von einem separaten Computer gesteuert, so dass der Betrieb des OCT-Systems neben dem automatisierten Versuchsaufbau möglich ist. Schließlich enthalten die Softwarepakete Bibliotheken zum Synchronisieren der OCT-Sondenpositionierung und der OCT-Scanerfassung (d. h. zum automatischen Erfassen von 3D-Bilddatensätzen in einem Mosaikmuster oder in einer Reihe definierterPositionen). Die Definition der Position der OCT-Sonde in 3D ermöglicht effektiv die Einstellung der Brennebene speziell für (regionale) Scans. Insbesondere können auf unebenen Flächen für jeden OCT-Scan unterschiedliche Brennebenen (d.h. unterschiedliche Positionen in z-Richtung) angegeben werden.
Eine Reihe von Softwarepaketen wurde entwickelt, um rohe OCT-Scans zu verarbeiten (Tabelle 1). Die Navigation des Positioniergeräts, die OCT-Scanerfassung und die Datensatzverarbeitung werden mit Python-codierten Jupyter-Notebooks durchgeführt, die eine bemerkenswerte Flexibilität bei der Entwicklung und Optimierung der Software ermöglichen. Zwei bearbeitete und mit Anmerkungen bezeichnete Beispiele solcher Notizbücher (für die Bildaufnahme bzw. -verarbeitung) sind ab https://gitlab.com/FlumeAutomation/automated-oct-scans-acquisition.git Sie sind als Ausgangspunkte für die Anpassung gedacht. der Methode. Ein Jupyter-Notebook ist eine Webbrowser-basierte Anwendung, die Zellen mit mit Anmerkungen python-Code enthält. Jeder Schritt ist in einer Zelle des Notizbuchs enthalten, die separat ausgeführt werden kann. Aufgrund der unterschiedlichen Länge des Lichtpfades durch die Scanlinse (sphärische Aberration)15erscheinen die rohen OCT-Scans verzerrt (Abbildung 2A). Wir haben einen Algorithmus entwickelt, um diese Verzerrung in erfassten OCT-Scans automatisch zu korrigieren (enthalten in ImageProcessing.ipynb, Supplementary File 1). Darüber hinaus kann die Biofilmmorphologie als 2D-Höhenkarte visualisiert werden, wie sie zuvor in Membransystemen16verwendet wurde, und wir veranschaulichen, wie Höhenkarten aus Scans, die in einem Kachelarray aufgenommen wurden, genäht werden können.
Schließlich wird die Funktionalität der beschriebenen Laborinstallation anhand eines Flume-Experiments veranschaulicht, bei dem phototropher Strombiofilm einem Gradienten der Strömungsgeschwindigkeit ausgesetzt ist.
Access restricted. Please log in or start a trial to view this content.
Protokoll
1. Einrichtung des Positioniergeräts
- Verdrahten Sie das Positioniergerät mit einer Mikrocontrollerplatine, die den Anweisungen in https://github.com/grbl/grbl/wiki/Connecting-Grbl folgt.
- Schließen Sie den Mikrocontroller über ein USB-Kabel an einen Single-Board-Computer mit Internetverbindung an und installieren Sie den GRBL-Server wie in https://gitlab.com/FlumeAutomation/GRBL_Server.git beschrieben. Nun sollte das Positionierungsgerät über eine Webseite, die in http://IP:5020/ gehostet wird, navigierbar sein. Alternativ kann das Positionierungsgerät mit einem Python-Skript navigiert werden, wie im ersten Teil des bearbeiteten Beispiels ImagesAcquisition.ipynb (Ergänzende Datei 2) gezeigt wird.
2. OCT-Einrichtung
- Montieren Sie die OCT-Sonde mit einem kompatiblen Taubenschwanzhalter an der Positioniervorrichtung. Installieren Sie bei Bedarf einen Tauchadapter auf der Objektivlinse.
- Positionieren Sie den Computer und die OCT-Basiseinheit auf einer Bank neben dem Experiment (z. B. mikrofluidische Geräte, Durchflusskammern, Flumen, Filtersysteme). Stellen Sie sicher, dass sich das optische Kabel (maximale Länge ca. 1,8 m) frei bewegt, lang genug, um alle vorgesehenen Stellen zu erreichen und den Versuchsaufbau nicht beeinträchtigt.
- Installieren Sie das OCT-System zusammen mit der vom Hersteller beschriebenen verfügbaren Software.
- Installieren Sie die Softwarepakete für die automatische OCT-Scanerfassung, wie in https://gitlab.com/FlumeAutomation/automated-oct-scans-acquisition.git beschrieben.
3. Bildaufnahme
- Schalten Sie das OCT-System und das Positioniergerät ein. Stellen Sie sicher, dass sich das Gerät frei bewegen kann.
- Öffnen Sie die Datei config.json in einem Texteditor. Bearbeiten Sie die Datei config.json, um den Standardparameter für die Bildaufnahme (Tabelle 2) anzupassen, z. B. den Brechungsindex (1,33 für Wasser bei 20 °C, 1,00 für Luft) und den Zielordner für abgerufene Daten und Metadaten.
- Definieren Sie die Größe des Feldes (FOV) und die Anzahl der A-Scans pro B-Scan in config.json.
HINWEIS: Diese beiden Parameter bestimmen die Größe der Voxel des endgültigen Datasets und die Größe der Ausgabedatei und sollten mit der optischen Auflösung der Sonde übereinstimmen (x-y voxel-Größe sollte nicht kleiner als die Hälfte der optischen Auflösung sein). Die Anzahl der A- und B-Scans wirkt sich auf das zu überfliegende räumliche Ausmaß aus, das gegen verfügbaren Speicherplatz und Die Rechenleistung ausgetradet wird. - Definieren Sie die Signalgrenzen des Ausgangs-OCT-Scans in config.json. Diese hängen vom Typ der Probe ab. Es wird daher empfohlen, diese Parameter auf der Grundlage von Intensitätshistogrammen einer Reihe von Vorläufigen zu bestimmen. Speichern Sie die Änderungen in config.json.
- Navigieren Sie mit der OCT-Sonde zu einer Sehenswürdigkeit. Fokussieren Sie die Probe und passen Sie die Referenzarm- und Lichtquellenintensität für eine optimale Bildqualität an. Wiederholen Sie diesen Vorgang für eine Reihe von Positionen, und notieren Sie sich die Koordinaten.
HINWEIS: Dies ermöglicht die anschließende automatische OCT-Scanerfassung um diese Referenzpunkte. Beachten Sie, dass die Länge und Intensität der Referenzarmarmwährend während der automatisierten Bildaufnahme nicht geändert werden kann. - Öffnen Sie die Datei ImageAcquisition.ipynb (Ergänzende Datei 2) in Juypter Notebook. Jede Zelle enthält Code, um bestimmte Aufgaben auszuführen und kann separat über drücken von Zelle | ausgeführt werden. Ausführenoder Strg + Eingabe oder Umschalt + Eingabe.
- Legen Sie den Pfad auf die erforderlichen Bibliotheken und die Standardkonfigurationsparameter fest. Alternativ können Sie einen neuen Satz temporärer Parameter definieren.
- Verbinden Sie sich mit dem Positioniergerät und initialisieren Sie das OCT.
- Kalibrieren Sie das Positioniergerät (d.h., führen Sie ein "Homing" durch).
- Erfassen Sie die Datensätze, die die Positionen der Interessen in Einzelscan- oder Mosaikmustern abdecken, und geben Sie die Zahl und die Überlappung an (z.B. 30 %) benachbarter Fliesen.
HINWEIS: Der Speicher wird vor dem Scan zugewiesen, wodurch die Computerressourcennutzung optimiert wird. Die Daten werden im 8-Bit-Format gespeichert, um Speicherplatz zu sparen, in dem in config.jsondefinierten Zielordner unter Verwendung des Zeitstempels und der Position als Namenskonvention (d. h. %Y%m%d_%H%M%S_). Metadaten, einschließlich der OCT-Einstellungen und Koordinaten, werden im selben Ordner in einer *.srm-Datei mit derselben Namenskonvention gespeichert. Abhängig von Einstellungen wie FOV und Auflösung kann die Dateigröße bis zu 1,5 GB pro OCT-Scan erreichen.
- Um eine Abtreibung der Datenerfassung zu vermeiden, stellen Sie sicher, dass genügend freier Speicherplatz vorhanden ist, oder verschieben Sie OCT-Datasets kontinuierlich auf eine externe Festplatte.
4. Bildkorrektur und Anzeige
- Öffnen Sie das Jupyter-Notebook ImageProcessing.ipynb (Ergänzende Datei 1) für ein bearbeitetes Beispiel der OCT-Bildverarbeitung (Korrektur von Verzerrungen, Hintergrundsubtraktion, Berechnung von Höhenkarten, Höhenkartenheft).
- Bei Bedarf schneiden DIE OCT-Scans, um falsche Signale auszuschließen und den Datensatz neu auszurichten (Biofilm sollte über dem Substrat umstrarangen).
- Korrekt für sphärische Aberration. Dies wird durch einen Korrekturalgorithmus erreicht, der eine hochreflektierende Referenzoberfläche verwendet, von der bekannt ist, dass sie flach ist (z. B. Unterseite des Flumes, Substratum). Zunächst definiert der Algorithmus ein Raster von 20 x 20 vertikalen Linien, die regelmäßig über die xy-Ebene des OCT-Scans verteilt werden. Anschließend wählt er einen kreisförmigen Bereich um jeden Punkt aus und durchschnittlich die Signalintensitäten entlang des vertikalen Profils (Abbildung 2B). Die vertikalen Profile werden mit einem modifizierten Gaußschen Filter verarbeitet:
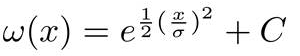
wobei x das Eingangssignal und seine Standardabweichung ist, während C bestimmt wird, z. B.: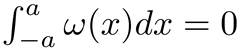
Die Referenzfläche ist in jedem dieser Profile als lokale Maxima lokalisiert. Falsch identifizierte Punkte werden basierend auf den Positionen ihrer Nachbarn in drei Dimensionen gefiltert (Abbildung 2C). Schließlich wird eine Polynomoberfläche in 2 nd-Reihenfolge, die die durch die Scanlinse eingeführte Verzerrung widerspiegelt, über diese Punkte hinweg angebracht (Abbildung2C). Die angepasste Fläche wird dann verwendet, um jedes Pixel in Z-Richtung zu verschieben und so ein abgeflachtes Bild zu erhalten. Die Parameter dieses Algorithmus sollten an die Eigenschaften des OCT-Scans angepasst werden. - Korrekt für Hintergrundgeräusche. Identifizieren Sie einen leeren Bereich des Bildes (typischerweise über dem Biofilm) und verwenden Sie den Korrekturalgorithmus, um die durchschnittliche Hintergrundintensität von den Intensitätswerten des Bildes zu subtrahieren, um ein endgültiges korrigiertes OCT-Bild zu erzeugen (Abbildung 2D).
- Berechnen Sie eine Höhenkarte aus dem 3D-OCT-Dataset. Definieren Sie in diesem Schritt eine Referenzfläche von Interesse für das spezifische Experiment (z. B. das Substratum) und eine geeignete Schwellenwertintensität. Verwenden Sie dann den Berechnungsalgorithmus für die Höhenkarte, um die Dicke des Biofilms für jede Koordinate (x,y) der Binärmaske zu berechnen und einer neuen 2D-Matrix zuzuweisen (Abbildung 3A). Dicke Werte werden dann einer 2D-Matrix der Größe des Originalbildes in x- und y-Richtung zugewiesen. Es wird ein Bild gerendert, in dem die Höhe der Oberfläche als Graustufenwert gemeldet wird (Abbildung 3B).
- Falls mehrere OCT-Scans in einem Mosaikmuster durchgeführt werden, definieren Sie die Anzahl der Zeilen und Spalten und heften Sie die jeweiligen Höhenkarten. Abbildung 5 zeigt Beispiele für genähte Höhenkarten, die die breite Palette von räumlichen Skalen und Auflösungen abdecken, die mit den beschriebenen Einstellungen erreichbar sind.
Access restricted. Please log in or start a trial to view this content.
Ergebnisse
Wir demonstrieren die Funktionalität des automatisierten OCT-Bildgebungssystems mit einem Flume-Experiment zur Untersuchung der räumlich-zeitlichen Morphogenese von phototrophen Strombiofilmen. Eine allmählich emittierende Geometrie der Flumen induzierte Gradienten in der Strömungsgeschwindigkeit entlang der Mitte des Flumes (siehe Referenz17). Die zeitliche Entwicklung und strukturelle Differenzierung des Biofilms wurde über 18 Tage überwacht, um die Auswirkungen hydrodynamischer Bedingunge...
Access restricted. Please log in or start a trial to view this content.
Diskussion
Die OCT-Bildgebung eignet sich gut, um Strukturen im Mikrometerbereich mit einem FOV von mehreren Quadratmillimetern aufzulösen. Es ist somit ein leistungsfähiges Werkzeug für die Biofilmforschung10,18. Allerdings ist OCT derzeit auf eine maximale Scanfläche von 100 - 256 mm2beschränkt, während Biofilmstrukturmuster diese räumliche Skala oft übersteigen19, insbesondere wenn die morphologische Differenzierung durch große...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
Sebastian Schäfer ist bei Thorlabs Inc. angestellt.
Danksagungen
Wir danken Mauricio Aguirre Morales für seinen Beitrag zur Entwicklung dieses Systems. Finanzielle Unterstützung kam vom Schweizerischen Nationalfonds an T.J.B.
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| OCT Probe | Thorlabs | GAN210C1 | OCT imaging device |
| OCT scan lens | Thorlabs | OCT-LK3-BB | |
| Immersion adapter | Thorlabs | OCT-IMM3-SP1 | |
| Stepcraft 840 CK | STEPCRAFT | NA | positioning device |
| microcontroller | Arduino Uno R3 | NA | |
| Single-board computer | Raspberry PI | NA | |
| camera | Canon EOS 7D Mark II | NA | |
| camera lens | Canon MACRO EFS 35 mm | NA |
Referenzen
- Flemming, H. C., Wingender, J. The biofilm matrix. Nature reviews. Microbiology. 8, 623-633 (2010).
- Flemming, H. -C., et al. Biofilms: an emergent form of bacterial life. Nature reviews. Microbiology. 14, 563(2016).
- Stoodley, P., Lewandowski, Z., Boyle, J. D., Lappin-Scott, H. M. Oscillation characteristics of biofilm streamers in turbulent flowing water as related to drag and pressure drop. Biotechnology and Bioengineering. 57, 536-544 (1998).
- Stoodley, P., Lewandowski, Z., Boyle, J. D., Lappin-Scott, H. M. The formation of migratory ripples in a mixed species bacterial biofilm growing in turbulent flow. Environmental microbiology. 1, 447-455 (1999).
- Banin, E., Vasil, M. L., Greenberg, E. P. Iron and Pseudomonas aeruginosa biofilm formation. Proceedings of the Natural Academy of Sciences U.S.A. 102, 11076-11081 (2005).
- Battin, T. J., Besemer, K., Bengtsson, M. M., Romani, A. M., Packmann, A. I. The ecology and biogeochemistry of stream biofilms. Microbiology. 14, 251-263 (2016).
- Battin, T. J., et al. Microbial landscapes: new paths to biofilm research. Nature Reviews. Microbiology. 5, 76-81 (2007).
- Neu, T. R., Lawrence, J. R. Innovative techniques, sensors, and approaches for imaging biofilms at different scales. Trends in Microbiology. 23, 233-242 (2015).
- Meleppat, R. K., Shearwood, C., Seah, L. K., Matham, M. V. Quantitative optical coherence microscopy for the in situ investigation of the biofilm. J. of Biomedical Optics. 21 (12), 127002(2016).
- Wagner, M., Horn, H. Optical coherence tomography in biofilm research: A comprehensive review. Biotechnology and Bioengineering. 114, 1386-1402 (2017).
- Huang, D., et al. Optical coherence tomography. Science. 254, 1178-1181 (1991).
- Haisch, C., Niessner, R. Visualisation of transient processes in biofilms by optical coherence tomography. Water Resources. 41, 2467-2472 (2007).
- Drexler, W., Fujimoto, J. G. Optical Coherence Tomography: Technology and Applications. , Springer Verlag. (2008).
- Fercher, A. F. Optical coherence tomography – development, principles, applications. Zeitschrift für Medizinische Physik. 20, 251-276 (2010).
- Lee, H. -C., Liu, J. J., Sheikine, Y., Aguirre, A. D., Connolly, J. L., Fujimoto, J. G. Ultrahigh speed spectral-domain optical coherence microscopy. Biomedical Optics Express. , 41236-41254 (2013).
- Fortunato, L., Leiknes, T. In-situ biofouling assessment in spacer filled channels using optical coherence tomography (OCT): 3D biofilm thickness mapping. Bioresource Technology. 229, 231-235 (2017).
- Niederdorfer, R., Peter, H., Battin, T. J. Attached biofilms and suspended aggregates are distinct microbial lifestyles emanating from differing hydraulics. Nature Microbiology. 1, 16178(2016).
- Roche, K. R., et al. Benthic biofilm controls on fine particle dynamics in streams. Water Resources. 53, 222-236 (2016).
- Fortunato, L., Jeong, S., Wang, Y., Behzad, A. R., Leiknes, T. Integrated approach to characterize fouling on a flat sheet membrane gravity driven submerged membrane bioreactor. Bioresource Technology. 222, 335-343 (2016).
- Morgenroth, E., Milferstedt, K. Biofilm engineering: linking biofilm development at different length and time scales. Reviews in Environmental Science and Bio/Technology. 8, 203-208 (2009).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten