Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Excitonic Hamiltonians for Calculating Optical Absorption Spectra and Optoelectronic Properties of Molecular Aggregates and Solids
In diesem Artikel
Zusammenfassung
Hier stellen wir ein Protokoll zur Parametrisierung eines eng bindenden exzitonischen Hamiltonian zur Berechnung optischer Absorptionsspektren und optoelektronischer Eigenschaften molekularer Materialien aus quantenchemischen Berechnungen erster Prinzipien vor.
Zusammenfassung
Die rationelle Konstruktion ungeordneter molekularer Aggregate und Feststoffe für optoelektronische Anwendungen beruht auf unserer Fähigkeit, die Eigenschaften solcher Materialien mit theoretischen und rechnerischen Methoden vorherzusagen. Große molekulare Systeme, bei denen eine Störung zu signifikant ist, um in der Störgrenze berücksichtigt zu werden, können jedoch weder mit den ersten Prinzipien der Quantenchemie noch mit der Bandtheorie beschrieben werden. Multiscale-Modellierung ist ein vielversprechender Ansatz, um die optoelektronischen Eigenschaften solcher Systeme zu verstehen und zu optimieren. Es verwendet erste Prinzipien quantenchemische Methoden, um die Eigenschaften einzelner Moleküle zu berechnen, dann konstruiert Modell Hamiltonians von molekularen Aggregaten oder Schüttgütern auf der Grundlage dieser Berechnungen. In diesem Beitrag stellen wir ein Protokoll zur Konstruktion eines eng bindenden Hamiltonian vor, das die angeregten Zustände eines molekularen Materials auf der Basis von Frenckel-Exziten darstellt: Elektronen-Loch-Paare, die auf einzelnen Molekülen lokalisiert sind, aus denen das Material besteht. Die hier vorgeschlagene Hamiltonsche Parametrisierung berücksichtigt exzitonische Kopplungen zwischen Molekülen sowie eine elektrostatische Polarisation der Elektronendichte auf einem Molekül durch die Ladungsverteilung auf umgebenden Molekülen. Ein solches Modell Hamiltonians kann verwendet werden, um optische Absorptionsspektren und andere optoelektronische Eigenschaften von molekularen Aggregaten und Feststoffen zu berechnen.
Einleitung
In den letzten zwei Jahrzehnten haben Feststoffe und Folien, die aus aggregierten organischen Molekülen hergestellt werden, mehrere Anwendungen in optoelektronischen Geräten gefunden. Geräte, die auf solchen Materialien basieren, haben viele attraktive Eigenschaften, einschließlich geringem Gewicht, Flexibilität, geringem Stromverbrauch und Potenzial für eine billige Produktion mit Inkjet-Druck. Displays, die auf organischen Leuchtdioden (OLEDs) basieren, ersetzen flüssige kristalline Displays als State-of-the-Art für Mobiltelefone, Laptops, Fernsehgeräte und andere elektronische Geräte1,2,3,4. Die Bedeutung von OLEDs für Beleuchtungsanwendungen wird in den kommenden Jahren voraussichtlich zunehmen4. Die Leistung von organischen Photovoltaik-Geräten verbessert sich stetig, mit Leistungsumwandlungseffizienzen von über 16% kürzlich für Ein-Kreuzung organische Solarzellen5gemeldet. Organische Materialien haben auch das Potenzial, andere Technologien zu stören, wie glasfaserkommunikation, wo ihre Verwendung die Entwicklung von elektro-optischen Modulatoren mit extrem hohen Bandbreiten von 15 THz und mehr6,7ermöglicht.
Eine große Herausforderung bei der Optimierung von Festkörper-Molekularmaterialien für Anwendungen in der Optoelektronik besteht darin, dass ihre Eigenschaften in der Regel stark von der nanoskaligen Struktur des Materials abhängen. Der Produktionsprozess ermöglicht die Definition der Nanostruktur eines Materials bis zu einem gewissen Grad durch den Einsatz kontrollierter Wachstumstechniken, wie chemische Dampfabscheidung,8 Verformung von optisch aktiven Molekülen auf ein anderes Material (d.h. eine Polymermatrix9,10), thermisches Glühen11,12usw. Nanoskalenstörungen sind jedoch für die meisten molekularen Materialien intrinsisch und können in der Regel nicht vollständig beseitigt werden. Daher ist es für die rationale Gestaltung organischer optoelektronischer Materialien unerlässlich, zu verstehen, wie sich Unordnung auf die Eigenschaften eines Materials auswirkt, und Wege zu finden, es für eine optimale Leistung zu entwickeln.
Der Grad der Störung in molekularen Materialien ist in der Regel zu groß, um es als Störung einer periodischen kristallinen Struktur mit einer elektronischen Struktur zu behandeln, die durch Bandtheorie beschrieben werden kann. Andererseits ist die Anzahl der Moleküle, die in eine Simulation einbezogen werden müssen, um die Eigenschaften eines Schüttguts oder eines Films zu reproduzieren, zu groß, um erste Prinzipien quantenchemischer Methoden wie Dichtefunktionstheorie (DFT)13,14 und zeitabhängige Dichtefunktionstheorie (TD-DFT)15,16zu verwenden. Organische Moleküle mit Anwendungen in der Optoelektronik haben in der Regel relativ große , konjugierte Systeme; viele haben auch Spender- und Akzeptorgruppen. Die Erfassung des korrekten Ladungsübertragungsverhaltens in solchen Molekülen ist für die Berechnung ihrer optoelektronischen Eigenschaften unerlässlich, kann aber nur mit fernkorrigierten Hybridfunktionen in TD-DFT17,18,19,20erreicht werden. Berechnungen, die solche Funktionen verwenden, skalieren super linear mit der Größe des Systems und sind derzeit nur praktisch, um die optoelektronischen Eigenschaften einzelner organischer Moleküle oder kleiner molekularer Aggregate zu modellieren, die mit nicht mehr als 104 atomaren Basisfunktionen beschrieben werden können. Eine Simulationsmethode, die ungeordnete Materialien beschreiben könnte, die aus einer großen Anzahl von Chromophoren bestehen, wäre sehr nützlich für die Modellierung dieser Systeme.
Die Größe intermolekularer Wechselwirkungen in molekularen Materialien ist oft vergleichbar oder kleiner als die Reihenfolge der Variation der energetischen Parameter (wie die Eigenstate-Energien oder Anregungsenergien) zwischen einzelnen Molekülen, aus denen das Material besteht. In solchen Fällen ist Multiskalenmodellierung der vielversprechendste Ansatz, um die optoelektronischen Eigenschaften großer ungeordneter molekularer Systeme zu verstehen und zu optimieren21,22,23. Dieser Ansatz verwendet quantenchemische Methoden erster Prinzipien (in der Regel DFT und TD-DFT), um die Eigenschaften einzelner Moleküle, aus denen das Material besteht, genau zu berechnen. Der Hamiltonian einer Materialprobe, die groß genug ist, um das Massenmolekülmaterial darzustellen (vielleicht durch die Verwendung periodischer Randbedingungen), wird dann mit den Parametern konstruiert, die für einzelne Moleküle berechnet wurden. Dieser Hamiltonian kann dann verwendet werden, um die optoelektronischen Parameter eines großen molekularen Aggregats, eines dünner Films oder eines Massenmolekülmaterials zu berechnen.
Exciton-Modelle sind eine Unterklasse von Multiskalenmodellen, in denen angeregte Zustände eines molekularen Materials auf der Basis von Exzitendargestellt werden: Elektronenlochpaare, die durch Die Coulomb-Attraktion24,25gebunden sind. Für die Modellierung vieler angeregter Zustandsprozesse reicht es aus, nur Frenkel-Exzitonen26einzubeziehen, bei denen das Elektron und das Loch auf demselben Molekül lokalisiert sind. Ladungsübertragungsexztonen, bei denen das Elektron und das Loch auf verschiedenen Molekülen lokalisiert sind, müssen in einigen Fällen (z. B. bei der Modellierung der Ladungstrennung in Spender-Akzeptor-Systemen)27,28enthalten sein. Obwohl Exciton-Modelle Multiskalenmodelle sind, die nur mit Berechnungen des ersten Prinzips an einzelnen Molekülen parametrisiert werden können, berücksichtigen sie immer noch intermolekulare Wechselwirkungen. Die beiden primären Wechselwirkungstypen, die sie berücksichtigen können, sind (a) exzitonische Kopplungen zwischen Molekülen, die die Fähigkeit von Exziten charakterisieren, sich zwischen Molekülen zu delokalisieren oder zwischen Molekülen zu übertragen, und (b) elektrostatische Polarisation der Elektronendichte auf einem Molekül durch die Ladungsverteilung auf umgebenden Molekülen. Wir haben bereits gezeigt, dass beide Faktoren wichtig für die Modellierung der optischen und elektrooptischen Eigenschaften molekularer Aggregate sind, wie z. B. die optischen Absorptionsspektren29 und die ersten hyperpolarisierbaren30.
In diesem Beitrag stellen wir ein Protokoll zur Parametrisierung von Exzitonmodellen vor, mit dem die optischen Spektren und andere optoelektronische Eigenschaften großer molekularer Aggregate und Massenmolekülmaterialien berechnet werden können. Der excitonic Hamiltonian wird angenommen, dass ein eng bindender Hamiltonian24,25,
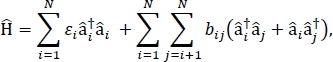
wobei i die Anregungsenergie des i.th Moleküls im Material, bij, die exzitationische Kopplung zwischen den ith- und den jj-th-Molekülen ist, sind i iund ii die Erzeugungs- bzw. Vernichtungsoperatoren für einen angeregten Zustand auf dem ith Molekül im Material.th Die exzitonic Hamiltonian Parameter werden mit TD-DFT-Berechnungen gefunden, die auf einzelnen Molekülen durchgeführt werden, aus denen das Material besteht. In diesen TD-DFT-Berechnungen wird die Ladungsverteilung auf alle anderen Moleküle im Material durch elektrostatische Einbettung von Atompunktladungen dargestellt, um die elektrostatische Polarisation der elektronischen Dichte eines Moleküls zu berücksichtigen. Die Anregungsenergien, ii, für einzelne Moleküle werden direkt aus dem TD-DFT-Berechnungsausgang entnommen. Die excitonischen Kopplungen, bij, zwischen Molekülen werden mit der Transition Density Cube Methode31berechnet, wobei die boden-zu-erregten Zustandsübergangsdichten für die interagierenden Moleküle aus der Ausgabe einer TD-DFT-Berechnung in Gauß-3232 entnommen und mit dem Multiwfn-Multifunktions-Wellenfunktionsanalysator33nachbearbeitet werden. Multiwfn Zur Simulation der Eigenschaften von Massenmolekülfeststoffen können periodische Randbedingungen auf den Hamiltonian angewendet werden.
Das aktuelle Protokoll erfordert, dass der Benutzer Zugriff auf die Programme Gaussian32 und Multiwfn33 hat. Das Protokoll wurde mit Gauß'sche 16, Revision B1 und Multiwfn Version 3.3.8 getestet, sollte aber auch für andere aktuelle Versionen dieser Programme funktionieren. Darüber hinaus verwendet das Protokoll ein benutzerdefiniertes C++-Dienstprogramm und eine Reihe von benutzerdefinierten Python 2.7- und Bash-Skripten, deren Quellcode unter der GNU General Public License (Version 3) zur https://github.com/kocherzhenko/ExcitonicHamiltonian bereitgestellt wird. Die Berechnungen sollen auf einem Computer ausgeführt werden, auf dem ein Betriebssystem aus der Unix/Linux-Familie ausgeführt wird.
Access restricted. Please log in or start a trial to view this content.
Protokoll
1. Aufteilen des multimolekularen Systems in einzelne Moleküle
- Generieren Sie die Struktur des Systems, für das der excitonic Hamiltonian im Molekulardateiformat Tripos MOL2 konstruiert werden muss. Diese Struktur kann eine Momentaufnahme einer molekularen Dynamik oder Monte-Carlo-Simulation des Systems sein.
- Wenn alle Moleküle im System aus der gleichen Anzahl von Atomen bestehen, verwenden Sie das Python 2.7-Skript getMonomers.py, um Dateien zu generieren, die die kartesischen Koordinaten für Atome in einzelnen Molekülen enthalten, aus denen das System besteht. Moleküle, aus denen das System besteht, müssen nicht identisch sein (z. B. können sie Isomere sein). Dieses Skript verwendet zwei Eingabeparameter.
- Geben Sie den Namen der Tripos MOL2-Datei an, die die Geometrie des Systems aus Schritt 1.1 (einer Zeichenfolge) enthält.
- Geben Sie die Anzahl der Atome in jedem einzelnen Molekül an, aus dem das System besteht (eine ganze Zahl). Verwenden Sie den Befehl:
./getMonomers.py YLD124-300K_0-210000.mol2 125
Nimmt die Struktur, die in der Beispieldatei YLD124-300K_0-210000.mol2 auf Github enthalten ist; schreibt kartesische Koordinaten einzelner Moleküle in Dateien monomer_N.com im XYZ-Format, wobei N eine vierstellige Zahl ist, die das Molekül im System identifiziert.
- Wenn ein System aus Molekülen mit unterschiedlicher Anzahl von Atomen besteht, erzeugen Sie die Strukturen für einzelne Moleküle mit einem alternativen Skript oder manuell. Das in den nachfolgenden Schritten beschriebene Verfahren kann ohne Änderungen verwendet werden.
2. Erzeugung von Bodenzustandspunktladungen für Atome in einzelnen Molekülen
- Richten Sie eine Nur-Text-Datei, chargeOptions.txt, mit den Optionen für eine Gaußsche DFT-Berechnung der Atompunktladungen im Bodenzustand eines elektrisch neutralen Moleküls ein. Um eine einigermaßen genaue Ladungsverteilung für Übergänge mit Ladungsübertragungscharakter zu erhalten, wird empfohlen, die Parameter wie folgt anzugeben.
- Verwenden Sie eine langräumige korrigierte Dichtefunktion (z. B. B97X)34.
- Verwenden Sie einen ausreichend großen Basissatz, der mindestens d Polarisationsfunktionen auf Nicht-Wasserstoffatomen (wie 6-31G*)35,36enthält.
- Verwenden Sie ein superfeines Integrationsraster.
- Verwenden Sie ein sehr enges selbstkonsistentes Feldkonvergenzkriterium (Energiekonvergenz auf 10–10 Hartree).
- Verwenden Sie Atompunktladungen, die geeignet sind, um das elektrostatische Potenzial in der Nähe des Moleküls zu reproduzieren, wie dies bei der CHelpG-Methode37geschieht, da die berechneten Atomladungen verwendet werden, um die elektrostatische Umgebung darzustellen.
HINWEIS: In typischen molekularen Aggregaten und Feststoffen sind die Abstände zwischen Molekülen relativ groß, so dass es oft akzeptabel ist, andere Atompunktladungsdefinitionen zu verwenden, wie z. B. Mulliken-Ladungen38. - Fügen Sie das NoSymm-Schlüsselwort in die Eingabedatei ein, um sicherzustellen, dass die atomaren Koordinaten in der Gaußschen Ausgabedatei in Eingabeausrichtung und nicht in Standardausrichtung geschrieben werden.
- Geben Sie den Namen der Berechnung in der Kommentarzeile der Gaußschen Eingabedatei an.
- Geben Sie die Ladung und Multiplizität des Moleküls (0 bzw. 1) in einer separaten Zeile an. Beispielberechnungsoptionen, die in der Datei chargeOptions.txt angegeben sind, können sein:
#p wB97X/6-31G* Integral(Grid=SuperFineGrid) NoSymm SCF(Conver=10) Pop=CHelpG
Monomer-Gebühren
0 1
- Richten Sie die Gaußschen Eingabedateien für alle einzelnen Moleküle ein, aus denen das System besteht, mit den Parametern in der Datei chargeOptions.txt. Dieser Schritt kann mit dem folgenden Bash-Skript effizient durchgeführt werden:
für f in monomer_*.xyz
tun
Cat chargeOptions.txt > s.f%xyz-com
tail -n +3 $f >>
Echo "" >> s.f%xyz-com
fertig
HINWEIS: Das Skript erzeugt Gaußsche Eingabedateien mit den gleichen Namen wie die XYZ-Dateien in Schritt 1.2 generiert, aber mit der Erweiterung .com. Diese Dateien enthalten die in chargeOptions.txt angegebenen Berechnungsoptionen und die atomaren Koordinaten aus den jeweiligen .xyz-Dateien, die durch eine leere Zeile beendet werden. - Führen Sie die Gaußschen Berechnungen aus und geben Sie an, dass der Name der Ausgabedatei mit dem Dateinamen .com identisch ist, jedoch mit der Erweiterung .log.
- Extrahieren Sie die CHelpG-Atompunktladungen aus den Gaußschen Ausgabedateien mit der Erweiterung .log, mit dem Python 2.7-Skript getCHelpG.py. Das Skript nimmt 2 Eingabeparameter: den Namen der Gaußschen Ausgabedatei mit der Erweiterung .log und die Anzahl der Atome in einem einzelnen Molekül.
HINWEIS: Das Skript getCHelpG.py schreibt eine Datei mit demselben Namen wie die Gaußsche Ausgabedatei, jedoch mit der Erweiterung .chg. Es gibt 4 Spalten in dieser Datei: die ersten drei mit den kartesischen Koordinaten der Atome in den Molekülen, die letzte mit den CHelpG-Atompunktladungen. Das folgende Bash-Skript kann Kosten effizient aus allen Dateien extrahieren:
für f in monomer_*.log; tun ./getCHelpG.py $f N; fertig
(N ist die Anzahl der Atome in einem Molekül.) - Wenn in Schritt 2.1 eine andere Definition der Atompunktladungen als CHelpG verwendet wurde, extrahieren Sie die Gebühren aus der Gaußschen Ausgabedatei mit einem alternativen Skript oder manuell.
3. Berechnung der Anregungsenergien und Übergangsdichten einzelner Moleküle im Material in Gegenwart einer elektrostatischen Umgebung
- Richten Sie eine Nur-Text-Datei, monomerOptions.txt, mit den Optionen für eine Gaußsche TD-DFT-Berechnung der angeregten Zustandsenergien und von Boden zu Angerstung seramerten Zustandsübergangsdichtematrizen für einzelne Monomere ein. Die vorgeschlagenen Parameter entsprechen denen, die für die Berechnung der Atompunktladungen in Schritt 2.1 verwendet werden.
- Verwenden Sie eine bereichsgetrennte Dichtefunktion, wie z. B. B97X34.
- Verwenden Sie einen ausreichend großen Basissatz, der mindestens d Polarisationsfunktionen auf Nicht-Wasserstoffatomen (wie 6-31G*)35,36enthält.
- Verwenden Sie ein superfeines Integrationsraster.
- Verwenden Sie ein sehr enges selbstkonsistentes Feldkonvergenzkriterium (Energiekonvergenz auf 10–10 Hartree).
- Fügen Sie das NoSymm-Schlüsselwort ein, um sicherzustellen, dass die atomaren Koordinaten in der Gaußschen Ausgabedatei in Eingabeausrichtung und nicht in Standardausrichtung geschrieben werden.
- Um eine einigermaßen genaue Übergangsdichten zu erhalten, sollten sie einen niedrigen Schwellenwert für den Druck eigenvektorischer Komponenten festlegen (d.h. sogar Koeffizienten für Basisfunktionen mit sehr geringen Beiträgen zu einem Eigenvektor – idealerweise zumindest in der Größenordnung von 10–5 – sollten in die Ausgabedatei gedruckt werden).
ANMERKUNG: Um diese Bedingung festzulegen, muss man gausssche interne Optionen verwenden: Setzen Sie Overlay 9, Option 40 auf den absoluten Wert des Exponenten für den Schwellenwert für den Druck von Eigenvektorkomponenten (z. B. IOp(9/40) = 5 setzt denCutoff-Schwellenwertauf 10 –5 ). - Geben Sie den Namen der Berechnung in der Kommentarzeile der Gaußschen Eingabedatei an.
- Geben Sie die Ladung und Multiplizität des Moleküls (0 bzw. 1) in einer separaten Zeile an. Beispielberechnungsoptionen, die in der Datei monomerOptions.txt angegeben sind, können sein:
#p tda(NStates=10) wB97X/6-31G* Integral(Grid=SuperFineGrid) NoSymm SCF(Conver=10) iop(9/40=5)
Monomer mit Gebühren
0 1
- Richten Sie die Gaußschen Eingabedateien für die Berechnung der Anregungsenergien und Übergangsdichten aller einzelnen Moleküle im Material in Gegenwart einer elektrostatischen Umgebung ein, die durch die Punktladungen auf alle anderen Moleküle im Material dargestellt wird, mit dem Dateinamen monomer_N_wCh.com.
- Fügen Sie eine Anforderung zum Speichern einer Gaußschen Prüfpunktdatei mit Dateiname monomer_N_wCh.chk in die Gaußsche Eingabedatei ein.
HINWEIS: Für die in diesem Protokoll beschriebene Namenskonvention kann dieser Schritt mit dem Python 2.7-Skript getMonomerWCh.py durchgeführt werden, der in den in der Datei monomerOptions.txtangegebenen Berechnungsoptionen die atomaren Koordinaten für einzelne Moleküle im System liest, die in Dateien monomer_N.xyzgespeichert sind, und Atompunktladungen für alle einzelnen Moleküle, die in Dateien gespeichert sind monomer_N.chg (N ist die Nummer des Monomers).
- Fügen Sie eine Anforderung zum Speichern einer Gaußschen Prüfpunktdatei mit Dateiname monomer_N_wCh.chk in die Gaußsche Eingabedatei ein.
- Führen Sie die Gaußschen Berechnungen aus und geben Sie an, dass der Name der Ausgabedatei mit dem Dateinamen .com identisch ist, jedoch mit der Erweiterung .log. Die Berechnung speichert auch eine Prüfpunktdatei mit dem gleichen Dateinamen, jedoch mit der Erweiterung .chk.
4. Extraktion von Anregungsenergien für helle Zustände einzelner Moleküle, aus denen das System besteht, aus den Gaußschen Ausgabedateien
- Kopieren Sie die Anregungsenergien für die hell angeregten Zustände einzelner Monomere aus den Gaußschen Ausgabedateien mit der Erweiterung .log in eine Nur-Text-Datei namens all_energies.txt.
HINWEIS: Wenn es nur einen hell angeregten Zustand gibt und sich alle Gaußschen Ausgabedateien im selben Verzeichnis befinden, kann dieser Schritt effizient ausgeführt werden, indem eine leere Nur-Text-Datei all_energies.txterstellt wird, und dann die Zeile in der Ausgabedatei anhängt, die die Anregungsenergie des hellen Zustands für jedes Monomer mit dem folgenden Bash-Skript enthält:
für f in monomer*WCh.log; do grep "Excited State M" $f >> all_energies.txt; fertig
(M ist die Nummer des angeregten hellen Zustands; die Anzahl der Leerzeichen zwischen dem Wort State und der Zahl M sollte die gleiche sein wie in den Gaußschen Ausgabedateien.) - Bewahren Sie in der Datei all_energies.txt nur die Spalte auf, die die numerischen Werte der Anregungsenergien (in eV) enthält. alle anderen Spalten löschen.
5. Berechnung der exzitonischen Kopplungen für alle Molekülpaare, aus denen das molekulare System besteht
- Konvertieren Sie die Checkpoint-Dateien mithilfe des Dienstprogramms formchk, das Teil von Gaussian ist, mithilfe des folgenden Bash-Skripts in ein lesbares Format:
für f in monomer_*.chk; tun formchk $f; fertig
HINWEIS: Die vom Menschen lesbaren formatierten Checkpoint-Dateien haben den gleichen Namen wie die ursprünglichen Checkpoint-Dateien, jedoch mit der Erweiterung .fchk. - Verwenden Sie das Python 2.7-Skript switchSign.py, das den Namen der Gaußschen Ausgabedatei mit der Erweiterung .log und der Anzahl der angeregten Zustände N, die in die Berechnung einbezogen sind, als Eingabeparameter annimmt.
HINWEIS: Dieses Skript druckt den Inhalt der .log-Datei aus, die die Richtung der Übergangsdipolmomentvektoren vom Boden in alle angeregten Zustände umstellt, wenn der Winkel zwischen dem permanenten Dipolmomentvektor des Bodenzustands und dem Übergangsdipolmomentvektor für den Boden zum ersten angeregten Zustandsübergang stumpf ist. - Speichern Sie die vom Skript erzeugte Ausgabe switchSign.py in einer Datei, die denselben Namen wie die .log-Datei hat, jedoch mit der Erweiterung .log2.
- Für Moleküle, bei denen der Winkel zwischen dem permanenten Dipolmomentvektor des Bodenzustands und dem Übergangsdipolmomentvektor für den Boden zum ersten angeregten Zustandsübergang akut ist, kopieren Sie die .log-Datei in eine Datei mit dem gleichen Namen und der gleichen Erweiterung .log2.
HINWEIS: Wenn die in diesem Protokoll empfohlene Namenskonvention verwendet wird, können die Schritte 5.2–5.4 mithilfe des folgenden Bash-Skripts effizient für alle einzelnen Monomere ausgeführt werden:
für f in monomer*_wCh.log
tun
./switchSign.py $f 10 >
wenn [ -s 'f'2 ]
Dann
echo 'Übergangsdipol-Momentzeichen für' $f
oder
Echo 'Kopieren' $f 'to'
cp -f-F-Nr. 2
Fi
fertig - Verwenden Sie den Multiwfn Multifunktions-Wellenfunktionsanalysator, um die Übergangsdichte-Cubedatei basierend auf der gaußsianformatierten Prüfpunktdatei mit der Erweiterung .fchk und der verarbeiteten Gaußschen Ausgabedatei mit der Erweiterung .log2zu schreiben.
HINWEIS: Gaußian unterstützt nur nativ das Speichern der räumlichen Dichteverteilung in einer Dichtecubedatei für Beobachtbare, die Hermitischen Operatoren entsprechen. Da der Transition Density Operator nicht Hermitisch ist, ist ein Nachbearbeitungsprogramm erforderlich, um eine Dichtecubedatei zu erhalten.- Starten Sie das Multiwfn-Programm.
- Senden Sie die Gaussian formatierte Prüfpunktdatei (die Datei mit der Erweiterung .fchk in Schritt 5.1) als Eingabedatei.
- Wählen Sie Option 18, Elektronenanregungsanalyse, aus dem Hauptfunktionsmenü.
- Wählen Sie Option 1, Analysieren und Visualisieren der Loch-Elektronen-Verteilung, des Übergangsdipolmoments undder Übergangsdichte aus dem Menü Elektronenerregungsanalysen.
- Senden Sie die Gaußsche Ausgabedatei mit angepassten Vorzeichen für die Übergangsdipolvektoren (die Datei mit der Erweiterung .log2 in Schritt 5.2 gespeichert), wenn Sie aufgefordert werden, den Pfad der Gaußschen Ausgabedatei oder Klartextdatei mit Anregungsdaten einzugeben.
- Geben Sie den Übergang an, für den die Cubedatei der Übergangsdichte generiert werden soll (wenn es nur einen hellen Zustand gibt, ist es der Übergang vom Bodenzustand in diesen Zustand).
- Wählen Sie Option 1, Visualisieren und Analysieren von Bohrung, Elektronen- und Übergangsdichte usw.im nächsten Menü.
- Wählen Sie die Anzahl der Punkte im Raster aus, für die die Übergangsdichte-Würfeldatei generiert wird: Eine größere Anzahl von Punkten führt zu genaueren exzitonischen Kopplungen, erhöht aber die Berechnungszeit erheblich, in den meisten Fällen ist Option 1, Raster mit geringer Qualität, das das gesamte System abdeckt, insgesamt etwa 125000 Punkte,ausreichend.
- Wählen Sie im folgenden Menü Option 13, Ausgabecubedatei mit Übergangsdichte inden aktuellen Ordner aus. Transition Density Cube Datei transdens.cub wird geschrieben, benennen Sie diese Datei um, um den gleichen Namen wie die .log2 und .fchk Dateien zu haben, mit der Erweiterung .cub.
HINWEIS: Multiwfn soll interaktiv ausgeführt werden, wobei Berechnungsoptionen von der Tastatur als Reaktion auf Eingabeaufforderungen eingegeben werden. Es ist jedoch bequemer, eine Datei mit Verarbeitungsoptionen einzurichten und dann Von Multiwfn aus dieser Datei lesen zu lassen.
- Um Eingerichtete Dateien mit Multiwfn-Verarbeitungsoptionen für alle .fchk-Dateien im aktuellen Verzeichnis effizient zu generieren, verwenden Sie das Bash-Skript makeOpt.sh. Die vom Skript geschriebenen Dateien makeOpt.sh haben die gleichen Namen wie die .fchk Dateien mit der Erweiterung .opt.
- Generieren Sie die Cubedateien mit der Übergangsdichte in einem einzelnen Batch mit dem folgenden Bash-Skript:
für f in monomer*_wCh.fchk
tun
Multiwfn $f < sf%fchk-opt
mv transdens.cub
fertig
ANMERKUNG: Abbildung 1 zeigt die Übergangsdichte für ein Molekül von 2-[4-[(E,3E)-3-[3-[(E)-2-[4-[bis[2-[tert-butyl-(dimethyl)silyl]oxyethyl]amino]phenyl]ethenyl]-5,5-dimethylcyclohex-2-en -1-yliden]prop-1-enyl]-3-cyano-5-phenyl-5-(trifluormethyl)furan-2-yliden]propanedinitrile (allgemein als YLD 124 [Jen2005] bezeichnet)39 in Gegenwart von Punktladungen umgebender Moleküle. - Konvertieren Sie .cub-Dateien in Dateien, die explizit die Koordinaten der Mittelpunkte aller Cubes im Raster angeben, die in Schritt 5.5.8 (erste 3 Spalten) verwendet werden, und die Werte der Übergangsdichte innerhalb des Cubes (letzte Spalte) mit dem Python 2.7-Skript cubeFormat.py. Das Skript nimmt den Namen einer .cub-Datei als Eingabe an. Um alle .cub-Dateien in einem Verzeichnis zu konvertieren, verwenden Sie das Bash-Skript:
für f in monomer_0*.cub; echo $f; ./cubeFormat.py $f; fertig
HINWEIS: Das Skript cubeFormat.py eine formatierte Dichte-Cube-Datei mit dem gleichen Namen wie die Eingabedatei schreibt, die es verwendet, aber mit der Erweiterung .fcub. - Verwenden Sie die in Schritt 5.8 generierten .fcub-Dateien, um die exzitonischen Kopplungen zwischen allen Molekülpaaren im System mit der Transition Density Cube-Methode31zu berechnen. Dieser Schritt kann mit dem cubePairGen-Programm durchgeführt werden, das zwei .fcub-Dateien für verschiedene Moleküle als Eingabe verwendet. Verwenden Sie zum Ausführen den Befehl:
./cubePairGen monomer_N_wCh.fcub monomer_M_wCh.fcub
HINWEIS: Das Programm gibt eine Datei mit dem Namen coup_N_M mit einer einzigen Zeile zurück, die drei Zahlen enthält: die Molekülnummern N und M, gefolgt von der excitonischen Kopplung zwischen diesen beiden Molekülen in eV. Der Programmquellcode in der Datei CubePairGen.cpp kann mit dem C++-Compiler in der GNU Compiler Collection mit dem Befehl kompiliert werden:
g++ CubePairGen.cpp –o cubePairGen - Wenn die in diesem Protokoll vorgeschlagene Dateibenennungskonvention verwendet wird, können die Berechnungen als Batch für alle Paare einzelner Moleküle ausgeführt werden, aus denen das untersuchte System besteht. Verwenden Sie zum Ausführen dieser Berechnungen das folgende Bash-Skript:
für f in *.fcub
tun
für g in *.fcub
tun
ff= . . . monomer_ . . . . . . . . . . . . . . . . . . . . .
gg= .g'monomer_'
fff= sff%_wCh *
ggg= "gg%_wCh"*
wenn [ "$fff" -gt "$ggg" ]
Dann
(*) ./cubePairgen' $f $g '> coup_'''_'_'''ggg'
Fi
fertig
fertig
ANMERKUNG: Abbildung 2 zeigt die Übergangsdichten für zwei Moleküle von YLD 124, die zur Berechnung der exzitonischen Kopplung zwischen diesen Molekülen verwendet werden. Bei großen Systemen, bei denen die Gesamtzahl der Molekülpaare groß ist, kann die Zeile, die durch ein Sternchen im Bash-Skript markiert ist, geändert werden, um Berechnungen an das Warteschlangensystem eines Supercomputing-Clusters zu übermitteln. - Sobald die Berechnungen in Schritt 5.8 abgeschlossen sind, erstellen Sie eine leere Datei all_couplings.txt und kombinieren Sie alle excitonic Kopplungen in einer einzigen Datei mit dem folgenden Bash-Skript:
für f in coup_0*; tun Katze $f >> all_couplings.txt; fertig
6. Einrichten des excitonic Hamiltonian
- Kombinieren Sie die angeregten Zustandsenergien in der Datei all_energies.txt, die in Schritt 4.2 generiert wurde, und die excitonischen Kopplungen in der Datei all_couplings.txt, die in Schritt 5.9 generiert wurde, in einer einzigen Datei, die die vollständige erregonische Hamiltonsche Matrix mit dem Python 2.7-Skript SetupHam.py mit dem Terminalbefehl enthält:
./Setup_Ham.py all_energies.txt all_couplings.txt N >Hamiltonian.txt
HINWEIS: Das Programm schreibt eine Datei Hamiltonian.txt mit drei Spalten: die Zeilennummer, Spaltennummer und wert in eV für jedes Matrixelement, mit Zeilen durch leere Zeilen getrennt.- Geben Sie den Namen der Datei an, die erregende Energien enthält.
- Geben Sie den Namen der Datei an, die exzitonische Kopplungen enthält.
- Geben Sie die Dimension N der Hamiltonschen Matrix (die Anzahl der Moleküle im System) an.
Access restricted. Please log in or start a trial to view this content.
Ergebnisse
In diesem Abschnitt stellen wir repräsentative Ergebnisse für die Berechnung des optischen Absorptionsspektrums eines Aggregats von sechs YLD 124 Molekülen vor, die in Abbildung 3adargestellt sind, wo die Struktur des Aggregats aus einer grobkörnigen Monte-Carlo-Simulation gewonnen wurde. YLD 124 ist ein prototypisches Ladungstransfer-Chromophor, das aus einer elektronen-fördernden Gruppe von Diethylamin mit Tert-Butyldimethylsilyl-Schutzgruppen besteht, die über eine -konjugierte<...
Access restricted. Please log in or start a trial to view this content.
Diskussion
Die hier vorgestellte Methode ermöglicht mehrere Anpassungen. Beispielsweise ist es möglich, die Parameter der DFT- und TD-DFT-Berechnungen zu ändern, einschließlich der Dichtefunktion, des Basissatzes und der spezifischen Definition der Atompunktladungen.
Es wird empfohlen, über weite Strecken korrigierte Funktionen wie z. B97X, B97XD oder PBE zu verwenden, um angemessene Übergangsdichten für Übergänge mit Ladungsübertragungscharakter zu erhalten. Es kann interessant sein zu untersu...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
Die Autoren geben keine Interessenkonflikte preis.
Danksagungen
Wir danken Dr. Andreas Tillack (Oak Ridge National Laboratory), Dr. Lewis Johnson (University of Washington) und Dr. Bruce Robinson (University of Washington) für die Entwicklung des Programms für grobkörnige Monte-Carlo-Simulationen, das verwendet wurde, um die Struktur des molekularen Systems zu erzeugen, das im Abschnitt Repräsentative Ergebnisse vorgestellt wurde. A.A.K. und P.F.G. werden durch einen Collaborative Research Award des College of Science, CSU East Bay, unterstützt. M.H. wird von einem Forever Pioneer Stipendium des Center for Student Research, CSU East Bay, unterstützt. C.M.I. und S.S. werden vom US-Verteidigungsministerium (Vorschlag 67310-CH-REP) im Rahmen der Air Force Office of Scientific Research Organic Materials Division unterstützt.
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| Gaussian 16, revision B1 | |||
| Multiwfn version 3.3.8 | |||
| GNU compiler collection version 9.2 | |||
| python 2.7.0 |
Referenzen
- Tsujimura, T. OLED Display Fundamentals and Applications, 2nd Ed. , Wiley. Hoboken, NJ. (2017).
- Barnes, D. LCD or OLED: Who Wins. SID Symposium Digest of Technical Papers. 44 (1), 26-27 (2013).
- Mizukami, M., et al. Flexible Organic Light-Emitting Diode Displays Driven by Inkjet-Printed High-Mobility Organic Thin-Film Transistors. IEEE Electron Device Letters. 39 (1), 39-42 (2018).
- Koden, M. OLED Displays and Lighting. , Wiley. Chichester, U.K. (2017).
- Fan, B., et al. Achieving Over 16% Efficiency for Single-Junction Organic Solar Cells. Science China Chemistry. 62 (6), 746-752 (2018).
- Dalton, L. R., Gunter, P., Jazbinsek, M., Kwon, O. P., Sullivan, P. A. Organic Electro-Optics and Photonics: Molecules, Polymers, and Crystals. , Cambridge University Press. Cambridge, U.K. (2015).
- Robinson, B. H., et al. Optimization of Plasmonic-Organic Hybrid Electro-Optics. Journal of Lightwave Technology. 36 (21), 5036-5047 (2018).
- Yu, D., Yang, Y. Q., Chen, Z., Tao, Y., Liu, Y. F. Recent Progress on Thin-Film Encapsulation Technologies for Organic Electronic Devices. Optics Communications. 362 (1), 43-49 (2016).
- Wanapun, D., Hall, V. J., Begue, N. J., Grote, J. G., Simpson, G. J. DNA-Based Polymers as Chiral Templates for Second-Order Nonlinear Optical Materials. ChemPhysChem. 10 (15), 2674-2678 (2009).
- Siao, Y. Y., et al. Orderly Arranged NLO Materials on Exfoliated Layered Templates Based on Dendrons with Alternating Moieties at the Periphery. Polymer Chemistry. 4 (9), 2747-2759 (2013).
- Sepeai, S., Salleh, M. M., Yahaya, M., Umar, A. A. Improvement of White Organic Light Emitting Diode Performances by an Annealing Process. Thin Solid Films. 517 (16), 4679-4683 (2009).
- Mao, G., et al. Considerable Improvement in the Stability of Solution Processed Small Molecule OLED by Annealing. Applied Surface Science. 257 (17), 7394-7398 (2011).
- Parr, R. G., Yang, W. Density Functional Theory of Atoms and Molecules. , University Press. Oxford, U.K. (1989).
- Dreizlerm, R. M., Gross, E. K. U. Density Functional Theory: An Approach to the Quantum Many-body Problem. , Springer. Berlin, Germany. (1990).
- Burke, K., Werschnik, J., Gross, E. K. U. Time-Dependent Density Functional Theory: Past, Present, and Future. Journal of Chemical Physics. 123 (6), 062206(2005).
- Ullrich, C. Time-Dependent Density-Functional Theory: Concepts and Applications. , Oxford University Press. Oxford, U.K. (2011).
- Vydrov, O. A., Scuseria, G. E. Assessment of a Long-Range Corrected Hybrid Functional. Journal of Chemical Physics. 125 (23), 234109(2006).
- Tawada, Y., Tsuneda, T., Yanagisawa, S. A Long-Range-Corrected Time-Dependent Density Functional Theory. Journal of Chemical Physics. 120 (18), 5425(2004).
- Rohrdanz, M. A., Herbert, J. M. Simultaneous Benchmarking of Ground- and Excited-State Properties with Long-Range-Corrected Density Functional Theory. Journal of Chemical Physics. 129 (3), 034107(2008).
- Autschbach, J. Charge-Transfer Excitations and Time-Dependent Density Functional Theory: Problems and Some Proposed Solutions. ChemPhysChem. 10 (11), 1757-1760 (2008).
- Nelson, J., Kwiatkowski, J. J., Kirkpatrick, J., Frost, J. M. Modeling Charge Transport in Organic Photovoltaic Materials. Accounts of Chemical Research. 42 (11), 1768-1778 (2009).
- Walker, A. B. Multiscale Modeling of Charge and Energy Transport in Organic Light-Emitting Diodes and Photovoltaics. Proceedings of the IEEE. 97 (9), 1587-1596 (2009).
- Wang, L., Li, Q., Shuai, Z., Chenc, L., Shic, Q. Multiscale Study of Charge Mobility of Organic Semiconductor with Dynamic Disorders. Physical Chemistry Chemical Physics. 12 (13), 3309-3314 (2010).
- Davydov, A. S. Theory of Molecular Excitons. , Plenum Press. New York, NY. (1971).
- Agranovich, V. M. Excitations in Organic Solids. International Series of Monographs on Physics. 142, Oxford University Press. Oxford, U.K. (2008).
- Frenkel, J. On the Transformation of Light into Heat in Solids. I. Physical Review. 37 (1), 17-44 (1931).
- Kocherzhenko, A. A., Lee, D., Forsuelo, M. A., Whaley, K. B. Coherent and Incoherent Contributions to Charge Separation in Multichromophore Systems. Journal of Physical Chemistry C. 119 (14), 7590-7603 (2015).
- Lee, D., Forsuelo, M. A., Kocherzhenko, A. A., Whaley, K. B. Higher-Energy Charge Transfer States Facilitate Charge Separation in Donor-Acceptor Molecular Dyads. Journal of Physical Chemistry C. 121 (24), 13043-13051 (2017).
- Kocherzhenko, A. A., Sosa Vazquez, X. A., Milanese, J. M., Isborn, C. M. Absorption Spectra for Disordered Aggregates of Chromophores Using the Exciton Model. Journal of Chemical Theory and Computation. 13 (8), 3787-3801 (2017).
- Kocherzhenko, A. A., et al. Unraveling Excitonic Effects for the First Hyperpolarizabilities of Chromophore Aggregates. Journal of Physical Chemistry C. 123 (22), 13818-13836 (2019).
- Krueger, B., Scholes, G., Fleming, G. Calculation of Couplings and Energy-Transfer Pathways between the Pigments of LH2 by the ab Initio Transition Density Cube Method. Journal of Physical Chemistry B. 102 (27), 5378-5386 (1998).
- Frisch, M. J., et al. Gaussian 16, Revision B.01. Gaussian, Inc. , Wallingford, CT. (2016).
- Lu, T., Chen, F. Multiwfn: A Multifunctional Wavefunction Analyzer. Journal of Computational Chemistry. 33 (5), 580-592 (2012).
- Chai, J. D., Head-Gordon, M. Systematic Optimization of Long-Range Corrected Hybrid Density Functionals. Journal of Chemical Physics. 128 (8), 084106(2008).
- Hehre, W., Ditchfield, R., Pople, J. Self-Consistent Molecular Orbital Methods. XII. Further Extensions of Gaussian-Type Basis Sets for Use in Molecular Orbital Studies of Organic Molecules. Journal of Chemical Physics. 56 (5), 2257-2261 (1972).
- Hariharan, P., Pople, J. The Influence of Polarization Functions on Molecular Orbital Hydrogenation Energies. Theoretica chimica acta. 28 (3), 213-222 (1973).
- Breneman, C. M., Wiberg, K. B. Determining Atom-Centered Monopoles from Molecular Electrostatic Potentials. The Need for High Sampling Density in Formamide Conformational Analysis. Journal of Computational Chemistry. 11 (3), 361-373 (1990).
- Mulliken, R. S. Electronic Population Analysis on LCAO-MO MolecularWave Functions. I. Journal of Chemical Physics. 23 (10), 1833-1840 (1955).
- Jen, A., et al. Exceptional Electro-Optic Properties through Molecular Design and Controlled Self-Assembly. Proceedings of SPIE. 5935, 593506(2005).
- Hirata, S., Head-Gordon, M. Time-Dependent Density Functional Theory Within the Tamm-Dancoff Approximation. Chemical Physics Letters. 314 (3-4), 291-299 (1999).
- Randolph, K. A., Myers, L. L. Basic Statistics in Multivariate Analysis. , Oxford University Press. Oxford, U.K. Chapter 2 11-34 (2013).
- Garrett, K., et al. Optimum Exchange for Calculation of Excitation Energies and Hyperpolarizabilities of Organic Electro-optic Chromophores. Journal of Chemical Theory and Computation. 10 (9), 3821-3831 (2014).
- Sekino, H., Maeda, Y. Polarizability and Second Hyperpolarizability Evaluation of Long Molecules by the Density Functional Theory with Long-Range Correction. Journal of Chemical Physics. 126 (1), 014107(2007).
- Johnson, L. E., Dalton, L. R., Robinson, B. H. Optimizing Calculations of Electronic Excitations and Relative Hyperpolarizabilities of Electrooptic Chromophores. Accounts of Chemical Research. 47 (11), 3258-3265 (2014).
- Lee, J., et al. Molecular Mechanics Simulations and Improved Tight-Binding Hamiltonians for Artificial Light Harvesting Systems: Predicting Geometric Distributions, Disorder, and Spectroscopy of Chromophores in a Protein Environment. Journal of Physical Chemistry B. 122 (51), 12292-12301 (2018).
- Bellinger, D., Pflaum, J., Brüning, C., Engel, V., Engels, B. The Electronic Character of PTCDA Thin Films in Comparison to Other Perylene-Based Organic Semi-conductors: Ab Initio-, TD-DFT and Semi-Empirical Computations of the Opto-Electronic Properties of Large Aggregates. Physical Chemistry Chemical Physics. 19 (3), 2434(2017).
- Zuehlsdorff, T. J., Isborn, C. M. Combining the Ensemble and Franck-Condon Approaches for Calculating Spectral Shapes of Molecules in Solution. The Journal of Chemical Physics. 148 (2), 024110(2018).
- Zuehlsdorff, T. J., Isborn, C. M. Modeling Absorption Spectra of Molecules in Solution. International Journal of Quantum Chemistry. 119 (1), 25719(2019).
- Plötz, P. A., Megow, J., Niehaus, T., Kühn, O. All-DFTB Approach to the Parametrization of the System-Bath Hamiltonian Describing Exciton-Vibrational Dynamics of Molecular Assemblies. Journal of Chemical Theory and Computation. 14 (10), 5001-5010 (2018).
- Tillack, A., Johnson, L., Eichinger, B., Robinson, B. H. Systematic Generation of Anisotropic Coarse-Grained Lennard-Jones Potentials and Their Application to Ordered Soft Matter. Journal of Chemical Theory and Computation. 12 (9), 4362-4374 (2016).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten