Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Kernmigration in der Drosophila-Eizelle
In diesem Artikel
Zusammenfassung
Bei Drosophiladurchläuft der Eizellkern während der Oogenese eine mikrotubuliabhängige Migration. Hier beschreiben wir ein Protokoll, das entwickelt wurde, um die Migration durch Live-Bildgebung an Eierkammern ex-vivozu verfolgen. Unser Verfahren hält Eierkammern für 12 Stunden am Leben, um multipositionale 3D-Zeitrafferfilme mit konfokaler Spinning-Disk-Mikroskopie zu erfassen.
Zusammenfassung
Die Bildgebung von Lebendzellen ist besonders notwendig, um die zellulären und molekularen Mechanismen zu verstehen, die Organellenbewegungen, Zytoskelettumlagerungen oder Polaritätsmuster innerhalb der Zellen regulieren. Bei der Untersuchung der Positionierung des Eizellkerns sind Live-Imaging-Verfahren unerlässlich, um die dynamischen Ereignisse dieses Prozesses zu erfassen. Die Drosophila-Eizelle ist eine mehrzellige Struktur und ein ausgezeichnetes Modellsystem, um dieses Phänomen aufgrund ihrer Größe und Verfügbarkeit zahlreicher genetischer Werkzeuge zu untersuchen. Während der Drosophila-Mid-Oogenese wandert der Kern von einer zentralen Position innerhalb der Eizelle, um eine asymmetrische Position einzunehmen, die durch Mikrotubuli erzeugte Kräfte vermittelt wird. Diese Migration und Positionierung des Kerns sind notwendig, um die Polaritätsachsen des Embryos und der nachfolgenden erwachsenen Fliege zu bestimmen. Ein Merkmal dieser Migration ist, dass sie in drei Dimensionen (3D) stattfindet, was eine Notwendigkeit für Live-Imaging schafft. Um die Mechanismen zu untersuchen, die die Kernmigration regulieren, haben wir ein Protokoll entwickelt, um die sezierten Eierkammern zu kultivieren und Live-Bildgebung für 12 Stunden durch Zeitrafferaufnahmen mit konfokale Spinning-Disk-Mikroskopie durchzuführen. Insgesamt erlauben uns unsere Bedingungen, Drosophila-Eierkammern über einen langen Zeitraum am Leben zu erhalten, wodurch der Abschluss der Nuklearen Migration in einer Großen Anzahl von Proben in 3D visualisiert werden kann.
Einleitung
Seit einigen Jahren hat sich die Drosophila-Eizelle als Modellsystem zur Untersuchung der Kernmigration herauskristallisiert. Die Drosophila-Eizelle entwickelt sich in einer mehrzelligen Struktur, die als Eizelle bezeichnet wird. Eizellen umfassen 16 Keimzellen (15 Ammenzellen und die Eizelle), die von einer Epithelschicht aus follikulären somatischen Zellen umgeben sind. Die Entwicklung der Eikammer wurde in 14 Stadien unterteilt (Abbildung 1A), in denen die Eizelle wächst und Reserven ansammelt, die für die frühe Entwicklung des Embryos erforderlich sind. Während der Entwicklung, bei Mikrotubuli-Reorganisation und asymmetrischem Transport mütterlicher Determinanten, polarisiert die Eizelle entlang der antero-dorsalen und dorso-ventralen Achsen. Diese Achsen bestimmen die nachfolgenden Polaritätsachsen des Embryos und des Erwachsenen, die aus der Befruchtung dieser Eizelleentstehen 1. Während der Oogenese nimmt der Kern eine asymmetrische Position in der Eizelle ein. Im Stadium 6 ist der Kern in der Zelle zentriert. Nach einem noch zu identifizierenden Signal, das von den hinteren Follikelzellen emittiert wird, das von der Eizelle empfangen wird, wandert der Kern in Richtung des Schnittpunkts zwischen den vorderen und lateralen Plasmamembranen in Stadium 7 (Abbildung 1B)2,3. Diese asymmetrische Position ist erforderlich, um die Bestimmung der dorso-ventralen Achse zu induzieren.
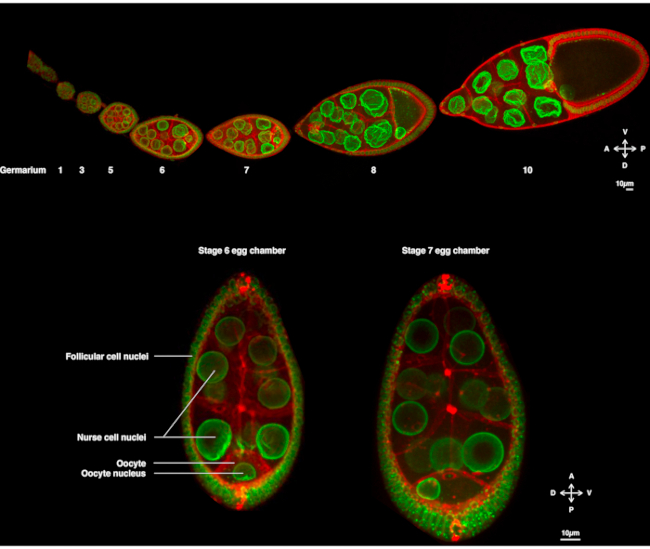
Abbildung 1: Drosophila melanogaster Eierkammern. (A) Festes Ovariol von transgenen Fliegen, die Fs(2)Ket-GFP exprimieren, das die Kernhüllen kennzeichnet, und ubi-PH-RFP, das die Plasmamembranen kennzeichnet. Das Ovariol besteht aus sich entwickelnden Eierkammern in verschiedenen Stadien. Die Reifung nimmt entlang der antero-posterioren Achse mit dem Keimarium an der vorderen Spitze (links), wo sich die Keimstammzelle befindet, und dem älteren Stadium an der hinteren Spitze (rechts) zu. (B) Z-Projektion der lebenden Eikammer durch Spinnscheibenkonfekularmikroskopie im Stadium 6 der Oogenese (links), bei der der Kern in der Eizelle zentriert ist. Der Zellkern wandert, um im Stadium 7 (rechts) in Kontakt mit der vorderen Plasmamembran (zwischen der Eizelle und der Ammenzelle) und der lateralen Plasmamembran (zwischen der Eizelle und den Follikelzellen) eine asymmetrische Position einzunehmen. Diese Position induziert die Bestimmung der dorsalen Seite und damit der dorso-ventralen Achse der Eikammer. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Seit vielen Jahrzehnten wird diese Kernmigration an fixierten Geweben durch Immunfärbung untersucht. Dieser Ansatz hat es insbesondere ermöglicht zu zeigen, dass dieser Prozess von einem dichten Netzwerk von Mikrotubuliabhängt 4,5. In jüngerer Zeit haben wir ein Protokoll entwickelt, das Bedingungen bietet, die mit der Live-Bildgebung der Eizelle während mehrerer Stunden kompatibel sind, so dass es möglich ist, diesen Prozess dynamisch zu untersuchen6.
So konnten wir erstmals beschreiben, dass der Zellkern während seiner Migration bevorzugte und charakteristische Trajektorien aufweist, eine entlang der vorderen Plasmamembran (APM) und eine entlang der lateralen Plasmamembran (LPM) der Eizelle (Abbildung 2). Diese neuesten Ergebnisse unterstreichen die Bedeutung von Live-Imaging-Protokollen bei der Untersuchung dynamischer Prozesse wie der nuklearen Migration.
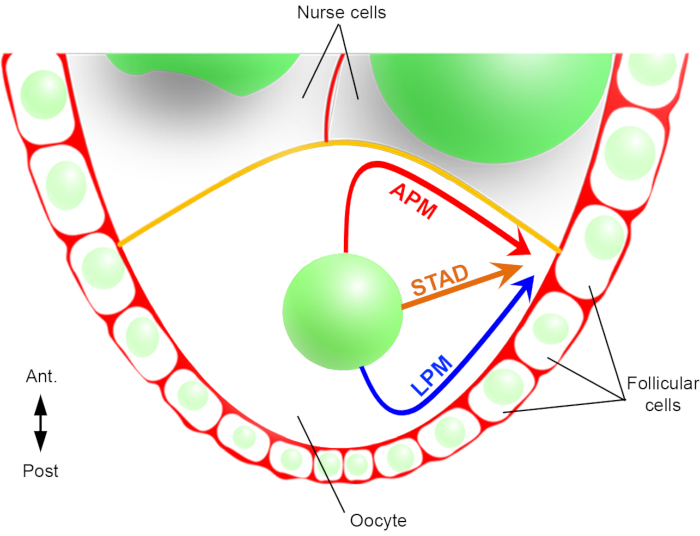
Abbildung 2: Schematische Darstellung der verschiedenen Migrationspfade des Kerns. Im Stadium 6 der Oogenese ist die Eizelle eine große Zelle mit einem zentralen Kern. In diesem Stadium wird die antero-posteriore Polaritätsachse mit einer posterioren/lateralen Plasmamembran der Eizelle in Kontakt mit den Follikelzellen eingestellt und die vordere Plasmamembran (in gelb) ist in Kontakt mit den Ammenzellen2. Wir haben bereits berichtet, dass der Zellkern entweder entlang der vorderen Plasmamembran (APM), entlang der lateralen Plasmamembran (LPM) oder durch das Zytoplasma (STAD, direkt zum antero-dorsalen Kortex) wandern könnte6. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Die Migration des Eizellkerns ist ein Phänomen von etwa 3 h6, und bisher ist das Ereignis, das den Beginn der tatsächlichen Migration auslöst, unbekannt. Der Beginn der Migration kann auch durch Proteinmutanten verzögert werden, die zur Untersuchung dieses Mechanismus verwendet werden. Diese unbekannten Variablen motivierten uns, Bilder über lange Zeiträume (10-12 h) zu erfassen. Es ist daher wichtig sicherzustellen, dass die Eizellen am Leben bleiben. Während sich die Eierkammer entwickelt, verlängert sie sich entlang der antero-posteriären Achse von einer kugelförmigen zu einer elliptischen Form. Diese Dehnung wird durch die Rotation von Follikelzellen angetrieben, die von Stadium 1 bis Stadium 8 senkrecht zur antero-posteriolenAchse 7auftritt. Darüber hinaus umgibt eine röhrenförmige Muskelscheide mit pulsierender Eigenschaft die Eierkammern. Seine physiologische Funktion besteht darin, die sich entwickelnden Follikel kontinuierlich in Richtung Eileiter zu drücken8. Um die Bewegungen zu begrenzen, die nach ihrer Dissektion Schwingungen der Eikammern auslösen, haben wir eine Beobachtungsmikrokammer mit einer Höhe von 150 μm entworfen (Abbildung 3A). Diese Höhe ist geringfügig höher als die Größe eines Follikels in den Stadien 10 und 11. Es schränkt die vertikalen Bewegungen der Probe erheblich ein und bewahrt gleichzeitig die Rotation der Eikammer, was zu begrenzten Defekten in der Follikelentwicklung führt. Wir führen dann Live-Bildgebung für 12 h auf sezierten Eierkammern durch Multi-Position-Zeitraffer-Erfassungen mit einem Spinnscheiben-Konfokalmikroskop durch. Hier beschreiben wir unser Protokoll zur Untersuchung der Eizellkernmigration zwischen den Stadien 6 und 7.
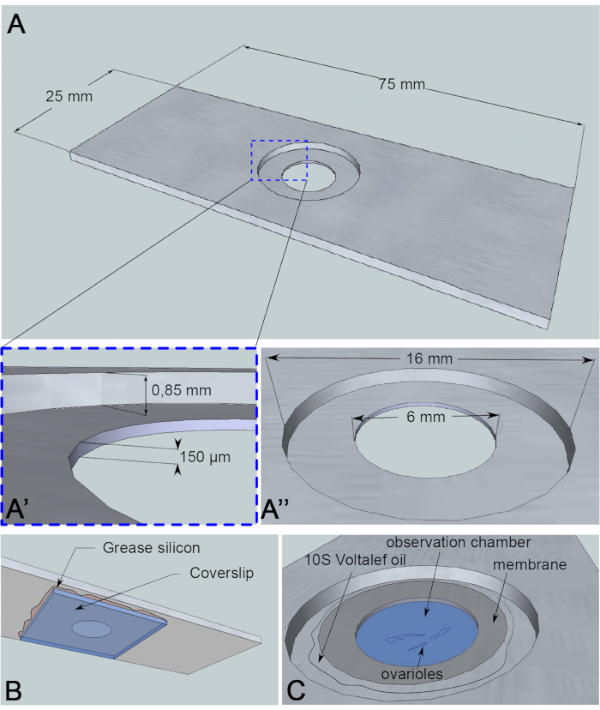
Abbildung 3: Schematische Darstellung der Beobachtungskammer. (A) (Drehe) Genaue Abmessungen des Aluminiumschlittens mit den Höhen (A') und Dem Umfang (A'') des in der Mitte des Schlittens gebohrten Brunnens. (B) (Unteransicht) Ein Abdeckr, der den Brunnen blockiert, wird mit Silikonfett zum Schlitten abgedichtet. (C) (Dranansicht) Sezierte Ovariolen entwickeln sich in einem bildgebenden Medium, das von einer gasdurchlässigen Membran bedeckt ist. Halogenkohlenstofföl wird verwendet, um die Membran zu stabilisieren. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Um die Kernmigration zu verfolgen und die Trajektorien in der Eizelle genau beurteilen zu können, werden Marker sowohl für die Kernhülle als auch für die Plasmamembran benötigt. Zu diesem Zweck wurden zwei Transgene ausgewählt, die ein hohes Signal-Rausch-Verhältnis aufweisen und im Verlauf der Live-Bildgebung nicht verblassen. Zur Kennzeichnung der Plasmamembran wird die Verwendung eines P[ubi-PH-RFP] empfohlen, der die Pleckstrin Homology (PH)-Domäne der humanen Phospholipase C ∂1 (PLC∂1) kodiert, die mit RFP verschmolzen ist. Diese PH-Domäne bindet an das Phosphoinositid PI(4,5)P2, das entlang der Plasmamembran der Eizelle9verteilt ist. Für die Kernhülle zeigt der P[PPT-un1]Fs(2)Ket-GFP-Proteinfallenstamm, bei dem GFP in das Gen eingefügt wird, das für das Drosophila ß-Importin kodiert, ein homogenes und intensives Signal10. Junge Fliegen (1-2 Tage alt) werden 24-48 h vor der Eierstockdissektion in frische Fläschchen mit Trockenhefe gelegt.
Für diesen Live-Imaging-Assay wurde ein 1 mm dickes Stück Aluminium, das für die Probe nicht reaktiv ist, in die Abmessungen eines Objektträgers geschnitten. Es hat ein Loch mit einem Durchmesser von 16 mm in der Mitte des Schlittens, das auf 0,85 mm gegenbohrt wurde. Diese Gegenbohrung hat eine zusätzliche Bohrung mit 6 mm Durchmesser und einer Tiefe von 150 μm (Abbildung 3A). Ein Abdeckrlip wird mit Silikonfett (inert für die Probe) am Boden der Aluminiumkammer verklebt (Abbildung 3B). Nach dem Einbringen der Proben in die mediumgefüllte Vertiefung wird eine Membran, die durchlässig fürO2/CO2-Austausch ist, über das Medium gelegt und von Halogenkohlenstofföl umgeben (Abbildung 3C).
Für die Dissektion wird empfohlen, eine Edelstahlzette mit einem Spitzenmaß von 0,05 x 0,02 mm und Nadeln mit einem Durchmesser von 0,20 mm zur Trennung der Ovariolen zu verwenden (Abbildung 4B,C). Die wandernden Kerne werden auf einem konfokalen invertierten Spinning-Disk-Mikroskop CSU-X1 abgebildet, das mit einer Kamera ausgestattet ist. Multi-Position-Bilder wurden im Zeitraffer alle 15 minuten bei 24 °C aufgenommen. Ein Intervall von 15 Minuten ermöglicht die Durchführung von Multi-Position-Erfassungen mit begrenztem Photobleaching der fluoreszierenden Proteine und Phototoxizität für die Proben. Darüber hinaus würde ein kürzeres Intervall keine viel aussagekräftigeren Daten liefern, um die nuklearen Trajektorien zu verfolgen. Die Filme werden über die Fidschi-Software11verarbeitet und analysiert.
Protokoll
1. Vorbereitung des Bildmediums
- Bereiten Sie frische Medien am Tag der Verwendung vor. Pipette 200 μL Schneider Medium (mit L-Glutamin und 0,40 g/L NaHCO3, ergänzt mit 10% hitzeinaktiviertem fetalem Kälberserum, 100 U/ml Penicillin und 100 mg/ml Streptomycin).
- Ergänzung mit 30 μL Insulin 10 mg / ml.
- Fügen Sie 4 μL hitzeinaktiviertes fetales Wadenserum hinzu.
2. Vorbereitung der Beobachtungskammer
- Tragen Sie mit einer Pipettenspitze eine kleine Menge Silikonfett um das Loch auf der Unterseite des punktierten Schlittens auf (Abbildung 4D).
- Positionieren Sie einen 24 x 50 mm dicken Abdeckrlip von 0,13-0,16 mm.
- Mit dem breiten Ende einer Pipettenspitze druckten Sie auf den Abdeckripp, um das Silikon abzuflachen, um den Abdeckr abzudichten und einen Silikonring im Inneren des Schlittens zu erzeugen (Abbildung 4F,G).
3. Eierstockdissektion
- Betäuben Sie eine weibliche Fliege des gewünschten Phänotyps aufeinem CO 2-Pad.
- Übertragen Sie das Weibchen in 150 μL des Bildmediums in eine Sezierschacht (Abbildung 4H).
- Öffnen Sie ein Weibchen, indem Sie seinen Thorax mit einer Zange greifen und die dorsale Bauchkutikula mit einer zweiten Zange einklemmen.
- Isolieren und lösen Sie das Paar Eierstöcke, das beim Öffnen der Kutikula leicht sichtbar sein sollte.
- Entfernen Sie vorsichtig die Gebärmutter, den Eileiter und die Muskelscheide (Abbildung 4I).
- Legen Sie einen Tropfen von 10-15 μL des Bildmediums und übertragen Sie einen Eierstock in die Bildgebungskammer (Abbildung 4J, K).
4. Isolierung der Eierkammer
- Um die Ovariolen zu trennen, halten Sie das hintere Ende des Eierstocks (in Richtung der älteren Stadien) mit der Nadel. Die Ovariolen zerteinsieren, indem Sie vorsichtig mit einer anderen Nadel am Keimarium ziehen.
- Entfernen Sie die verbleibende Muskelscheide auf den Eierkammern; eine Nadel hält die Scheide und die andere zieht an der Ovariole durch die größeren Kammern (Stadium 9 oder älter).
- Lassen Sie die ungehüllte Ovariole sinken und kontaktieren Sie den Abdeckungsrutsch.
- Entfernen Sie späte Stadien und den Rest der Eierstöcke aus der Mikrokammer mit Hilfe einer Zette. Distanzieren Sie die Ovariolen vorsichtig mit Nadeln von den anderen, um die Erfassung zu erleichtern (Abbildung 4L).
5. Schließen der Beobachtungskammer
- Schneiden Sie ein kleines Quadrat (10 x 10 mm) einer durchlässigen Membran (Abbildung 4M, N).
- Tragen Sie die Membran vorsichtig auf das Bildmedium auf, um Luftblasen auszustoßen (Abbildung 4O).
- Versiegeln Sie die Kammer hermetisch mit einer dünnen Schicht Halogenkohlenstofföl um die Vertiefung auf der Kontur der Membran (Abbildung 4P, Q).
6. Bildgebung
- Platzieren Sie das Bildgebungsset-up mit einem 40-fachen Objektiv (HCX PL Apo, 1.25NA, Ölimmersion) auf dem Objektträgerhalter des invertierten Mikroskops.
- Lokalisieren und speichern Sie Positionen verschiedener Eizellen im Stadium 6, in denen der Kern zur Migration bereit ist.
- Richten Sie die Laser 488 und 561 nm ein. Bei einer gemessenen Ausgangslaserleistung von 150 mW nutzen Sie 30% der Laserleistung bzw. 300 ms bzw. 500 ms Belichtung.
- Richten Sie das Experiment ein. Nehmen Sie einen Zeitraffer von 12 h mit dem Intervall von 15 min-41 Abschnitten mit einem Intervall von 1 μm zentriert auf den Kern.
HINWEIS: Entsprechend der oben beschriebenen Belichtungszeit ermöglicht diese Einstellung die Erfassung einer Position in etwa 45 s. Da es aufgrund von Positionswechsel zu einer Verzögerung kommt, empfiehlt es sich, unter diesen Bedingungen maximal 12 Positionen einzustellen.
7. Bildanalyse
- Verarbeiten Sie die Filme auf der Software Fiji mit der Plug-in-Ansicht Orthogonal und verfolgen Sie die Kerne manuell.

Abbildung 4:Schritt für Schritt Bilder zur Mikrokammermontage. (A,B,C) Vorbereitung der benötigten Werkzeuge: Sezierplatte, Zette, Nadeln, Bildmedien, Silikonfett, durchlässige Membran und der Aluminiumträger. (D) Auftragen des Silikonfetts auf der Rückseite des Aluminiumschlittens mit einer Pipettenspitze. (E) Ein Glasdeckel wird auf das Silikonfett geklebt, um den Boden der Kammer zu erzeugen. (F,G) Druckanwendung auf den Abdeckrlip mit der breiteren Extremität einer Pipettenspitze, um eine Verbindung innerhalb der Kammer zu erzeugen. (H,I) Dissektion der Fliegenovare in den bildgebenden Medien. (J) Pipettieren eines Tropfens bildgebender Medien in der Mikrokammer. (K,L) Trennung der Ovariolen in der Mikrokammer mit Nadeln. (M,N,O) Permeable Membran in ein 10 x 10 mm großes Quadrat geschnitten und über den Tropfen des Mediums in der Mikrokammer gelegt. (P,Q) Abdichtung der Mikrokammer mit Halogenkohlenstofföl. Die Proben sind bereit, abgebildet zu werden. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ergebnisse
Vor der Migration ist der Kern dynamisch und oszilliert um eine zentrale Position während einer Periode, die als Vormigration definiert ist. Diese kleinen Bewegungen spiegeln ein Gleichgewicht von Drücken und Ziehen wider, die das Gleichgewicht in der Mitte der Eizelle aufrechterhalten. Durch die Quantifizierung der Trajektorien der Kerne haben wir gezeigt, dass die APM- und LPM-Trajektorien ähnliche Anteile auf hatten. Wir definieren die Art der Flugbahn durch den ersten Kontakt zwischen dem Kern und der Plasmamembra...
Diskussion
Andere Protokolle beschreiben, wie Drosophila-Eierkammern ex vivo für den Live-Imaging-Assay vorbereitet und kultiviert werden12,13. Die Neuheit dieses Protokolls ist die Verwendung einer Bildgebungskammer, die aus einem ausgehöhlten Aluminiumschlitten, einem Abdeckrutsch und einerO2/ CO 2-permeablenMembran besteht. Der Hauptvorteil dieses Aufbaus besteht darin, die Bewegung in Z zu begrenzen, ohne Druck auf die Probe auszuüben. ...
Offenlegungen
Die Autoren erklären keine Interessenkonflikte.
Danksagungen
Wir sind Jean-Antoine Lepesant und Nicolas Tissot sehr dankbar, die ursprünglich das Protokoll entwickelt und einige grafische Elemente von Abbildung 3 mit uns geteilt haben. Wir danken Fanny Roland-Gosselin, die die Fotos von Abbildung 4 gemacht hat. Wir danken auch anderen Labormitgliedern für hilfreiche Diskussionen, die zur Verbesserung dieser Technik beigetragen haben, und Nathaniel Henneman für seine Kommentare, die zur Verbesserung dieses Manuskripts beigetragen haben. Wir erkennen die ImagoSeine-Kerneinrichtung des Instituts Jacques Monod, Mitglied von France-BioImaging (ANR-10-INBS-04), an. Maëlys Loh wird durch ein Promotionsstipendium des französischen Forschungsministeriums (MESRI) unterstützt. Antoine Guichet und Fred Bernard wurden vom ARC (Grant PJA20181208148), der Association des Entreprises contre le Cancer (Grant Gefluc 2020 #221366) und einem Emergence Grant der IdEx Université de Paris (ANR-18-IDEX-0001) unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| Anesthetize CO2 pad | Dutscher | 789060 | Anesthetize flies |
| Coverslip (24x50 mm) | Knittel Glass | VD12450Y100A | Observation-chamber preparation |
| Forceps Dumont #5 | Carl Roth | K342.1 | Dissection |
| Stainless steel needles | Entosphinx | 20 | Dissection |
| Heat-inactivated fetal calf serum | SIGMA-ALDRICH | F7524 | Imaging medium |
| Insulin solution bovine pancreas | SIGMA-ALDRICH | 10516 - 5ml | Imaging medium |
| Penicilin/Streptomycin solution | SIGMA-ALDRICH | P0781 | Imaging medium |
| Permeable membrane | Leica | 11521746 | Observation-chamber preparation |
| Schneider Medium | Pan Biotech | P04-91500 | Imaging medium |
| Silicon grease | BECKMAN COULTER | 335148 | Observation-chamber preparation |
| Spinning disk confocal | Zeiss | CSU-X1 | Nuclear migration observation |
| Voltalef oil 10S | VWR | 24627 - 188 | Observation-chamber preparation |
Referenzen
- Merkle, J. A., Wittes, J., Schüpbach, T. Signaling between somatic follicle cells and the germline patterns the egg and embryo of Drosophila. Current Topics in Developmental Biology. 140, 55-86 (2020).
- Roth, S., Lynch, J. A. Symmetry breaking during drosophila oogenesis. Cold Spring Harbor Perspectives in Biology. 1 (2), 001891 (2009).
- Bernard, F., Lepesant, J. -. A., Guichet, A. Nucleus positioning within Drosophila egg chamber. Seminars in Cell and Developmental Biology. 82, 25-33 (2017).
- Koch, E. A., Spitzer, R. H. Multiple effects of colchicine on oogenesis in Drosophila: Induced sterility and switch of potential oocyte to nurse-cell developmental pathway. Cell and Tissue Research. 228 (1), 21-32 (1983).
- Januschke, J., et al. The centrosome-nucleus complex and microtubule organization in the Drosophila oocyte. Development. 133, 129-139 (2006).
- Tissot, N., et al. Distinct molecular cues ensure a robust microtubule-dependent nuclear positioning in the Drosophila oocyte. Nature Communications. 8, 15168 (2017).
- Cetera, M., Horne-Badovinac, S. Round and round gets you somewhere: collective cell migration and planar polarity in elongating Drosophila egg chambers. Current Opinion in Genetics & Development. 32, 10-15 (2015).
- Hudson, A. M., Petrella, L. N., Tanaka, A. J., Cooley, L. Mononuclear muscle cells in Drosophila ovaries revealed by GFP protein traps. Developmental Biology. 314, 329-340 (2008).
- Gervais, L., Claret, S., Januschke, J., Roth, S., Guichet, A. PIP5K-dependent production of PIP2 sustains microtubule organization to establish polarized transport in the Drosophila oocyte. Development. 135 (23), 3829-3838 (2008).
- Villányi, Z., Debec, A., Timinszky, G., Tirián, L., Szabad, J. Long persistence of importin- b explains extended survival of cells and zygotes that lack the encoding gene ' n Villa. Mechanisms of Development. 3-4 (125), 196-206 (2008).
- Schindelin, J., et al. Fiji: An open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Prasad, M., Jang, A. C. C., Starz-Gaiano, M., Melani, M., Montell, D. J. A protocol for culturing drosophila melanogaster stage 9 egg chambers for live imaging. Nature Protocols. 2 (10), 2467-2473 (2007).
- Weil, T. T., Parton, R. M., Davis, I. Preparing individual Drosophila egg chambers for live imaging. Journal of Visualized Experiments: JoVE. (60), (2012).
- Chanet, S., Huynh, J. R. Collective cell sorting requires contractile cortical waves in germline cells. Current Biology. 30 (21), 4213-4226 (2020).
- Zhao, T., Graham, O. S., Raposo, A., St Johnston, D. Growing microtubules push the oocyte nucleus to polarize the drosophila dorsal-ventral axis. Science. 336 (6084), 999-1003 (2012).
- Legent, K., Tissot, N., Guichet, A. Chapter 7 Oogenesis using fixed and live imaging. Drosophila Oogenesis: Methods and Protocols. 1328, 99-112 (2015).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten