Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Polymerase-Kettenreaktion und Dot-Blot-Hybridisierung zum Nachweis von Leptospiren in Wasserproben
In diesem Artikel
Zusammenfassung
In dieser Studie wurde eine Dot-Blot-Anwendung entwickelt, um Leptospira aus den drei Hauptkladen in Wasserproben nachzuweisen. Diese Methode ermöglicht die Identifizierung minimaler DNA-Mengen, die spezifisch auf eine Digoxigenin-markierte Sonde abzielen und von einem Anti-Digoxigenin-Antikörper leicht nachgewiesen werden können. Dieser Ansatz ist ein wertvolles und zufriedenstellendes Instrument für Screening-Zwecke.
Zusammenfassung
Der Dot-Blot ist eine einfache, schnelle, empfindliche und vielseitige Technik, die die Identifizierung minimaler DNA-Mengen ermöglicht, die spezifisch durch Sondenhybridisierung in Gegenwart von Träger-DNA anvisiert werden. Es basiert auf der Übertragung einer bekannten Menge an DNA auf einen inerten festen Träger, wie z. B. eine Nylonmembran, unter Verwendung der Dot-Blot-Apparatur und ohne elektrophoretische Trennung. Nylonmembranen haben den Vorteil einer hohen Nukleinsäurebindungskapazität (400 μg/cm2), einer hohen Festigkeit und sind positiv oder neutral geladen. Bei der verwendeten Sonde handelt es sich um ein hochspezifisches ssDNA-Fragment von 18 bis 20 Basen mit einer Länge, das mit Digoxigenin (DIG) markiert ist. Die Sonde wird mit der Leptospira-DNA konjugieren. Sobald die Sonde mit der Ziel-DNA hybridisiert ist, wird sie von einem Anti-Digoxigenin-Antikörper detektiert, was einen einfachen Nachweis durch ihre Emissionen ermöglicht, die in einem Röntgenfilm sichtbar sind. Die Punkte mit einer Emission entsprechen den interessierenden DNA-Fragmenten. Bei dieser Methode wird die nicht-isotopische Markierung der Sonde verwendet, die eine sehr lange Halbwertszeit haben kann. Der Nachteil dieser Standard-Immunmarkierung ist eine geringere Empfindlichkeit als Isotopensonden. Dennoch wird sie durch die Kopplung der Polymerase-Kettenreaktion (PCR) und Dot-Blot-Assays gemildert. Dieser Ansatz ermöglicht die Anreicherung der Zielsequenz und deren Detektion. Darüber hinaus kann es als quantitative Anwendung im Vergleich zu einer seriellen Verdünnung eines bekannten Standards verwendet werden. Eine Dot-Blot-Anwendung zum Nachweis von Leptospira aus den drei Hauptkladen in Wasserproben wird hier vorgestellt. Diese Methode kann auf große Wassermengen angewendet werden, sobald sie durch Zentrifugation konzentriert wurden, um den Nachweis des Vorhandenseins von leptospiraler DNA zu erbringen. Dies ist ein wertvolles und zufriedenstellendes Instrument für allgemeine Screening-Zwecke und kann für andere nicht kultivierbare Bakterien verwendet werden, die im Wasser vorhanden sein können, um das Verständnis des Ökosystems zu verbessern.
Einleitung
Die Leptospirose beim Menschen hat hauptsächlich ihren Ursprung in der Umwelt 1,2. Das Vorkommen von Leptospira in Seen, Flüssen und Bächen ist ein Indikator für die Übertragung von Leptospirose unter Wildtieren sowie Haus- und Nutztieren, die schließlich mit diesen Gewässern in Kontakt kommen können 1,3,4. Darüber hinaus wurde Leptospira in nicht-natürlichen Quellen wie Abwasser, stehendem Wasser und Leitungswasser nachgewiesen 5,6.
Leptospira ist ein weltweit verbreitetes Bakterium 7,8, und die Rolle der Umwelt bei ihrer Erhaltung und Übertragung ist allgemein anerkannt. Leptospira kann im Trinkwasser bei variablem pH-Wert und unterschiedlichen Mineralienüberleben 9 und in natürlichen Gewässern1. Es kann auch über lange Zeiträume in destilliertem Wasserüberleben 10, und bei konstantem pH-Wert (7,8) kann es bis zu 152 Tage überleben11. Darüber hinaus können Leptospiren in bakteriellen Konsortien interagieren, um unter rauen Bedingungen zu überleben12,13. Es kann Teil von Biofilmen im Süßwasser mit Azospirillum und Sphingomonas sein und ist sogar in der Lage, zu wachsen und Temperaturen von über 49 °C zu ertragen14,15. Sie kann sich auch in durchnässten Böden vermehren und bis zu 379 Tage lang lebensfähig bleiben16, wobei ihre Fähigkeit, die Krankheit zu verursachen, bis zu einem Jahr lang erhalten bleibt17,18. Über die Ökologie in Gewässern und deren Verteilung in den Gewässern ist jedoch nur wenig bekannt.
Seit ihrer Entdeckung basierte die Erforschung der Gattung Leptospira auf serologischen Untersuchungen. Erst in diesem Jahrhundert wurden molekulare Techniken bei der Erforschung dieser Spirochäten immer häufiger eingesetzt. Der Dot-Blot wurde bisher kaum für seine Identifizierung verwendet, wobei (1) eine Isotopensonde auf der Basis der 16S rRNA und ein Inter-Simple Sequence Repeat (ISSR)19,20, (2) als Nanogold-basierter Immunoassay für humane Leptospirose, der auf Urin angewendet wurde21, oder (3) als Antikörper-basierter Assay für Rinderurinprobenverwendet wurde 22. Die Technik geriet in Vergessenheit, da sie ursprünglich auf Isotopensonden basierte. Es handelt sich jedoch um eine bekannte Technik, die in Verbindung mit der PCR verbesserte Ergebnisse liefert und aufgrund der Verwendung von nicht-isotopischen Sonden als sicher gilt. Die PCR spielt eine entscheidende Rolle bei der Anreicherung der Leptospira-DNA, indem ein spezifisches DNA-Fragment amplifiziert wird, das in Spuren in einer Probe vorkommen kann. Während jedes PCR-Zyklus wird die Menge des Ziel-DNA-Fragments in der Reaktion verdoppelt. Am Ende der Reaktion wurde das Amplikon mit dem Faktor von mehr als einer Millionmultipliziert 23. Das durch PCR amplifizierte Produkt, das in der Agaroseelektrophorese oft nicht sichtbar ist, wird durch spezifische Hybridisierung mit einer DIG-markierten Sonde im Dot-Blot 24,25,26 sichtbar.
Die Dot-Blot-Technik ist einfach, robust und für zahlreiche Proben geeignet, so dass sie auch für Labore mit begrenzten Ressourcen zugänglich ist. Es wurde in einer Vielzahl von Bakterienstudien eingesetzt, einschließlich (1) oraler Bakterien27, (2) anderer Probentypen wie Lebensmittel und Kot28 und (3) der Identifizierung von nicht kultivierbaren Bakterien29, oft in Übereinstimmung mit anderen molekularen Techniken. Zu den Vorteilen der Dot-Blot-Technik gehören: (1) Die Membran hat eine hohe Bindungskapazität und kann über 200 μg/cm2 Nukleinsäuren und bis zu 400 μg/cm2 binden; (2) Dot-Blot-Ergebnisse können visuell interpretiert werden, ohne dass spezielle Geräte erforderlich sind, und (3) sie können bequem jahrelang bei Raumtemperatur (RT) gelagert werden.
Die Gattung Leptospira wurde in pathogene, intermediäre und saprophytische Kladen eingeteilt 30,31. Die Unterscheidung zwischen diesen Kladen kann anhand spezifischer Gene wie lipL41, lipL32 und der 16S rRNA erreicht werden. LipL32 ist in den pathogenen Kladen vorhanden und zeigt eine hohe Sensitivität in verschiedenen serologischen und molekularen Werkzeugen, während es in Saprophytenspezies21 nicht vorhanden ist. Das Housekeeping-Gen lipL41 ist für seine stabile Expression bekannt und wird in molekularen Verfahren32 verwendet, während das16 S rRNA-Gen für ihre Klassifizierung verwendet wird.
Diese Methode kann auf große Wassermengen angewendet werden, sobald sie durch Zentrifugation konzentriert wurden. Es ermöglicht die Beurteilung verschiedener Punkte und Tiefen innerhalb eines Gewässers, um das Vorhandensein von leptospiraliger DNA und der Klade, zu der sie gehört, nachzuweisen. Dieses Instrument ist sowohl für ökologische als auch für allgemeine Screening-Zwecke wertvoll und kann auch zum Nachweis anderer nicht kultivierbarer Bakterien eingesetzt werden, die möglicherweise im Wasser vorhanden sind.
Darüber hinaus sind PCR- und Dot-Blot-Assays für eine Vielzahl von Laboren technisch und wirtschaftlich erschwinglich, selbst für solche, die nicht über hochentwickelte oder teure Geräte verfügen. Ziel dieser Studie ist es, den Digoxigenin-basierten Dot-Blot zur Identifizierung der drei Leptospira-Kladen in Wasserproben einzusetzen, die aus natürlichen Gewässern entnommen wurden.
Bakterienstämme
Zwölf Leptospira-Serovare (Autumnalis, Bataviae, Bratislava, Canicola, Celledoni, Grippothyphosa, Hardjoprajitno, Icterohaemorrhagiae, Pomona, Pyrogenes, Tarassovi und Wolffi) wurden in diese Studie eingeschlossen. Diese Serovare sind Teil der Sammlung der Abteilung für Mikrobiologie und Immunologie der Fakultät für Veterinärmedizin und Tierzucht der Nationalen Autonomen Universität von Mexiko und werden derzeit im Mikroagglutinationstest (MAT) verwendet.
Alle Leptospira-Serovare wurden in EMJH kultiviert, und ihre DNA wurde mit einem kommerziellen DNA-Extraktionskit extrahiert (siehe Materialtabelle). Ein genomischer DNA-Mix der zwölf Serovare wurde als Positivkontrolle für die pathogene Leptospira-Klade verwendet. Als Positivkontrolle der Leptospira-Intermediärklade wurde die genomische DNA von Leptospira fainei Serovar Hurstbridge Stamm BUT6 eingeschlossen, und als Positivkontrolle für die Leptospira Saprophytenklade wurde auch die genomische DNA von Leptospira biflexa serovar Patoc Stamm Patoc I eingeschlossen.
Die Negativkontrollen bestanden aus einem leeren Plasmid, DNA von nicht verwandten Bakterien (Ureaplasma urealyticum, Staphylococcus aureus, Brucella abortus, Salmonella typhimurium, Shigella boydii, Klebsiella pneumoniae, Acinetobacter baumannii und Escherichia coli) und Wasser in PCR-Qualität, das als Nicht-Template-Kontrolle diente.
Wasserproben
Zwölf Versuchsproben wurden unter Verwendung einer geschichtet-zufälligen Probenahmemethode vom Cuemanco Biological and Aquaculture Research Center (CIBAC) (19° 16' 54" N 99° 6' 11" W) entnommen. Diese Proben wurden in drei Tiefen entnommen: oberflächlich, 10 und 30 cm (Abbildung 1A, B). Die Wasserentnahmeverfahren hatten keine Auswirkungen auf gefährdete oder geschützte Arten. Jede Probe wurde in einem sterilen 15-ml-Mikrozentrifugenröhrchen entnommen. Um die Probe zu entnehmen, wurde jedes Röhrchen vorsichtig in das Wasser getaucht, in der gewählten Tiefe gefüllt und dann verschlossen. Die Proben wurden bei 22 °C gehalten und umgehend zur Verarbeitung ins Labor transportiert.
Jede Probe wurde durch Zentrifugation in sterilen 1,5 mL Mikrozentrifugenröhrchen bei 8000 x g für 20 min bei Raumtemperatur konzentriert. Dieser Schritt wurde wiederholt, bis alle Proben in einem Röhrchen konzentriert waren, das dann für die DNA-Extraktion verwendet wurde (Abbildung 1C).
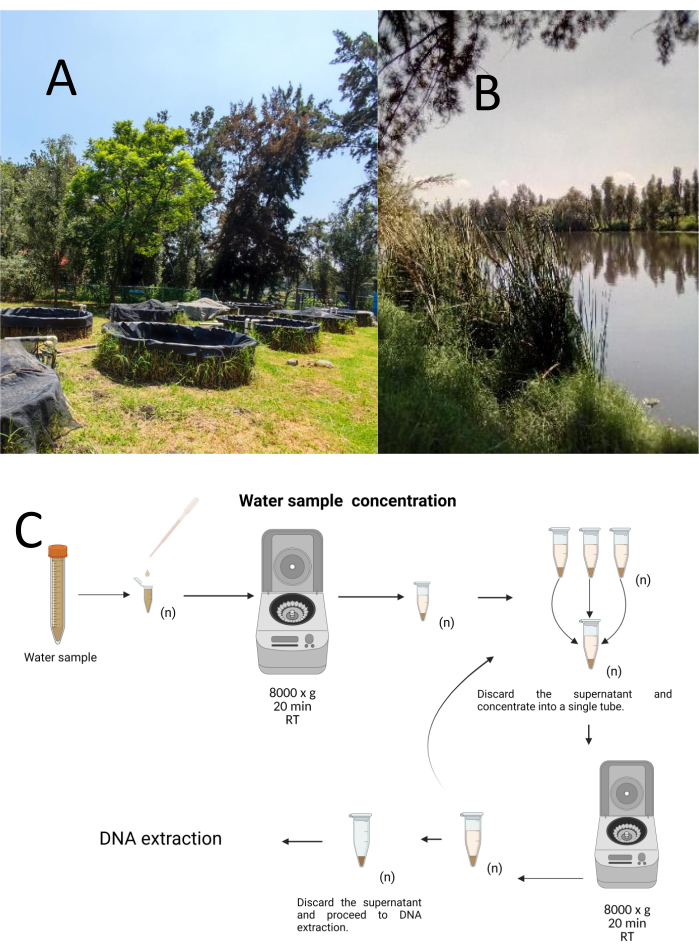
Abbildung 1: Konzentration der Wasserproben durch Zentrifugation. (A) Wasserprobenteiche und (B) Natürliche Bäche. (C) Zentrifugationsbasierte Verarbeitung von Wasserproben in wiederholten Schritten so oft wie nötig (n). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
DNA-Extraktion
Die Gesamt-DNA wurde mit einem handelsüblichen genomischen DNA-Kit gemäß den Anweisungen des Herstellers isoliert (siehe Materialtabelle). DNA-Extraktionen wurden in 20 μl Elutionspuffer eluiert, und die DNA-Konzentration wurde mit einem UV-Spektrophotometer bei 260-280 nm bestimmt und bis zur Verwendung bei 4 °C gelagert.
PCR-Amplifikation
Die PCR-Ziele waren die 16Gene S rRNA, lipL41 und lipL32, die DNA der Gattung Leptospira identifizieren und die Unterscheidung zwischen den drei Kladen pathogen, saprophytisch und intermediär ermöglichen. Sowohl die Primer als auch die Sondendesigns basierten auf den früheren Arbeiten von Ahmed et al., Azali et al., Bourhy et al., Weiss et al. und Branger et al.33,34,35,36,37. Die Sequenz jeder Sonde, jedes Primers und jedes amplifizierten Fragments ist in Tabelle 1 beschrieben, und ihre Ausrichtung mit den Referenzsequenzen ist in der Zusatzdatei 1, der Zusatzdatei 2, der Zusatzdatei 3, der Zusatzdatei 4 und der Zusatzdatei 5 angegeben. Die PCR-Reagenzien und Thermocycling-Bedingungen werden im Abschnitt Protokoll beschrieben.
Die Amplifikationsprodukte wurden durch elektrophoretische Trennung an einem 1%igen Agarosegel in TAE (40 mM Trisbase, 20 mM Essigsäure und 1 mM EDTA; pH 8,3) bei 60 V für 45 min mit Ethidium-Bromid-Detektion visualisiert, wie in der ergänzenden Abbildung 1 gezeigt. Genomische DNA, die aus jedem Serovar gewonnen wurde, wurde mit Konzentrationen von 6 x 106 bis 1 x 104 genomischen Äquivalentkopien (GEq) in jeder PCR-Reaktion verwendet, basierend auf der Genomgröße von L. interrogans (4, 691, 184 bp)38 für pathogene Leptospira, der Genomgröße von L. biflexa (3, 956, 088 bp)39 für saprophytische Leptospira, und die Genomgröße des L . fainei Serovars Hurstbridge Stamm BUT6 (4, 267, 324 bp) mit der Zugangsnummer AKWZ00000000.2.
Die Sensitivität der Sonden wurde in jedem Experiment mit DNA aus jedem pathogenen Serovar, dem L . biflexa Serovar Patoc Stamm Patoc I und dem L. fainei Serovar Hurstbridge Stamm BUT6 bewertet. Um die Spezifität des PCR- und Dot-Blot-Hybridisierungsassays zu beurteilen, wurde DNA von nicht verwandten Bakterien einbezogen.
Tabelle 1: PCR-Primer und -Sonden zur Amplifikation von Produkten zur Identifizierung der pathogenen, Saprophyten- und intermediären Kladen von Leptospira. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Dot-Blot-Hybridisierungs-Assay
Die Technik wird als Dot-Blot bezeichnet, da die Löcher, in die die DNA-Probe gelegt wird, eine Punktform haben, und wenn sie angesaugt werden, um durch Vakuumsaugen fixiert zu werden, erhalten sie diese Form. Diese Technik wurde von Kafatos et al.40 entwickelt. Die Technik ermöglicht die Semi-Quantifizierung von Leptospiren in jeder PCR-positiven Probe. Das Protokoll besteht aus einer Denaturierung mit NaOH 0,4 M bei Raumtemperatur, Proben mit Leptospira-DNA von 30 ng bis 0,05 ng, entsprechend 6 x 106 bis 1 x 104 Leptospiren, werden mit einem 96-Well-Dot-Blot-Gerät auf eine Nylonmembran blott. Nach der Immobilisierung wird die DNA durch Einwirkung von 120 mJ UV-Licht an die Membran gebunden. Jede DNA-Sonde wird durch einen terminalen Transferase-Katalyseschritt am 3'-Ende mit Digoxigenin-11 dUTP konjugiert (Digoxigenin ist ein pflanzliches Steroid, das aus Digitalis purpurea gewonnen wird und als Reporter41 verwendet wird). Nach der stringenten Hybridisierung der markierten DNA-Sonde (50 pmol) bei der spezifischen Temperatur auf die Ziel-DNA werden die DNA-Hybride durch die Chemilumineszenzreaktion mit dem anti-Digoxigenin-alkalischen Phosphatase-Antikörper, der kovalent mit seinem Substrat CSPD konjugiert ist, sichtbar gemacht. Die Lumineszenz wird durch Belichtung mit einem Röntgenfilm erfasst (Abbildung 2).
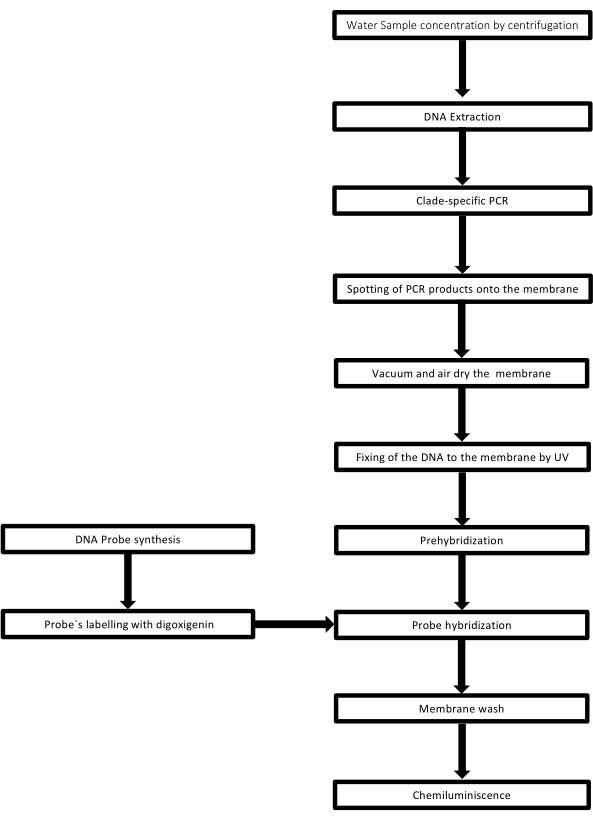
Abbildung 2: Schritte des Ablaufs für den PCR-Dot-Blot-Assay. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Protokoll
1. Vorbereitung der Probe
- Jede Wasserprobe wird in 1,5 mL Mikrozentrifugenröhrchen durch Zentrifugation bei 8.000 x g für 10 min bei 4 °C konzentriert. Wiederholen Sie diesen Schritt so oft wie nötig, um die Probe auf ein Volumen von 250 μl zu konzentrieren.
- Verwenden Sie das DNA-Extraktionskit gemäß den Anweisungen des Herstellers (siehe Materialtabelle).
- Führen Sie die spezifische PCR entsprechend der zu verwendenden Dot-Blot-Sonde durch (Tabelle 1).
- Führen Sie die Amplifikationen in PCR-Röhrchen mit einem Endvolumen von 25 μl durch, das 1 X Puffer, 2,5 Einheiten Taq-Polymerase, 1 μM jedes Primers, 0,2 mM jedes dNTP, 1,5 mM MgCl2 und 100 ng Ziel-DNA aus jeder Wasserprobe oder Referenz-Genom-DNA enthält.
- Programmieren Sie die PCR-Reaktion in einem Thermocycler entsprechend den Thermocycling-Bedingungen (Tabelle 2).
- Lagern Sie die Reaktionen bis zur Verwendung bei 4 °C.
- Geben Sie 10 μl jedes zu tupfenden PCR-Produkts in eine separate Vertiefung einer 96-Well-Platte.
- Geben Sie 40 μl TE in jede Vertiefung und mischen Sie durch Pipettieren.
HINWEIS: Die 96-Well-Platte kann versiegelt und über Nacht bei 4 °C gelagert werden. Befolgen Sie die Anweisungen in der Zusatzdatei 6 , um Puffer und Lösungen für das Protokoll vorzubereiten. Abbildung 3 zeigt die Schritte der Probenvorbereitung.
Tabelle 2: PCR-Thermocycling-Bedingungen für die Gene 16S, lipL41 und lipL32. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
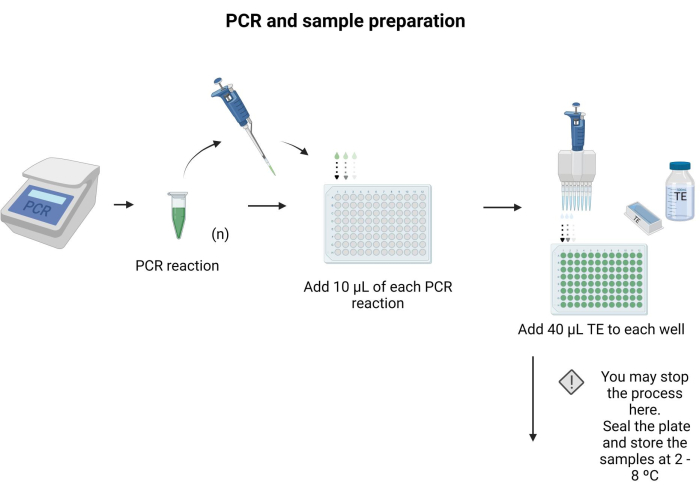
Abbildung 3: PCR und Probenvorbereitung. Unter Anwendung des spezifischen PCR-Protokolls wurde das PCR-Produkt auf eine Mikrotiterplatte überführt und 40 μl TE in jede Vertiefung gegeben. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
2. Zusammenbau des Dot-Blot-Geräts
HINWEIS: Der Zusammenbau des Dot-Blot-Geräts ist in Abbildung 4 dargestellt. Tragen Sie während des Eingriffs Handschuhe, um die Alkalilösungen zu handhaben und die Nylonmembran vor Verunreinigungen zu schützen.
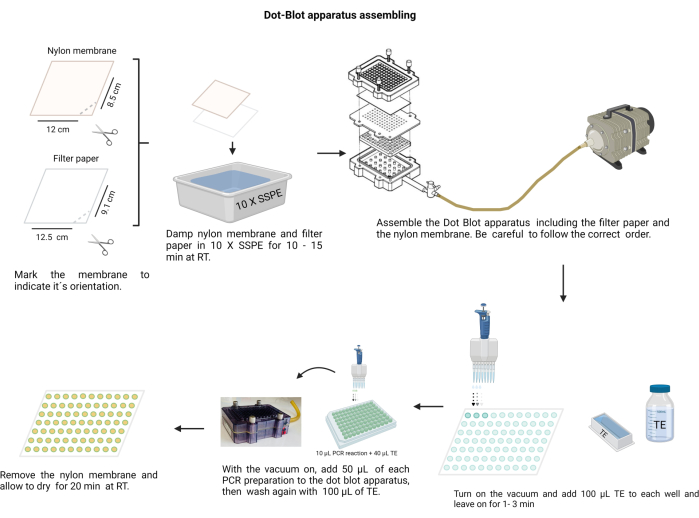
Abbildung 4: Zusammenbau des Dot-Blot-Geräts. Das Filterpapier und die Nylonmembran (zuvor in 10 X SSPE angefeuchtet) müssen in der richtigen Reihenfolge angeordnet werden. Die Baugruppe muss vor dem Anlegen des Vakuums mit den Schrauben fest befestigt werden. Jede Vertiefung muss mit TE gewaschen werden, und die PCR-Produkte werden in die jeweiligen Vertiefungen geladen. Nach dem Transfer des PCR-Produkts durch die Membran wird jede Vertiefung erneut mit TE gewaschen und trocknen gelassen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
- Schneiden Sie die Nylonmembran (siehe Materialtabelle) und das Filterpapier in Blätter mit der Größe 12 x 8,5 cm zu.
- Markieren Sie die Membran mit einem Permanentmarker.
- Machen Sie mit einer Schere an einer Kante eine Kerbe, um die richtige Ausrichtung anzuzeigen. Verwenden Sie die Markierung, um sich die Musterreihenfolge zu merken.
- Befeuchten Sie die Membran und das Filterpapier mit 10 x Kochsalzlösung-Natriumphosphat-EDTA-Puffer (SSPE; 3 M NaCl, 0,2 M NaH2PO4 und 0,02 M EDTA, pH 7,4) (Zusatzdatei 6). Fassen Sie die Membran mit einer sauberen, stumpfen Pinzette an.
- Bauen Sie die Dot-Blot-Kammer zusammen; Zuerst das Filterpapier, dann die Membran über der Kunststoffdichtung. Befestigen Sie die Abdeckung mit den Schrauben quer.
- Schließen Sie die Kammer an das Vakuum an, geben Sie 100 μl TE in jede Vertiefung, halten Sie das Vakuum 1 Minute lang und stoppen Sie es dann.
- Schalten Sie das Vakuum bei niedriger Geschwindigkeit ein und laden Sie 50 μl jeder Probe in die entsprechende Vertiefung auf der Membran des Dot-Blot-Apparats (gemäß der vorgegebenen Membranverteilung).
HINWEIS: Homogenisieren Sie jede Probe in der Hämagglutinationsplatte mit 96 Vertiefungen, bevor Sie sie in die Dot-Blot-Kammer geben. - Lassen Sie das Vakuum die Membran trocknen. Schlagen Sie bei Bedarf vorsichtig auf die Kammer, um die Blasen in der Probe freizusetzen.
- Waschen Sie jede Vertiefung, indem Sie 100 μl TE mit einem kontinuierlichen Vakuum hineingeben und trocknen lassen.
HINWEIS: Nach Abschluss des Transfers ist es wichtig, die Pumpe zuerst auszuschalten und abzunehmen. Andernfalls kann Rückfluss in das Gerät gelangen. Bei der Verwendung einer positiv geladenen Nylonmembran ist kein Denaturierungsschritt erforderlich, bei Verwendung eines anderen inerten Trägers kann jedoch ein Vordenaturierungsschritt und eine alkalibasierte Denaturierung erforderlich sein41.
3. DNA-Denaturierung und -Fixierung
HINWEIS: Abbildung 5 veranschaulicht das Verfahren zur DNA-Membranfixierung.
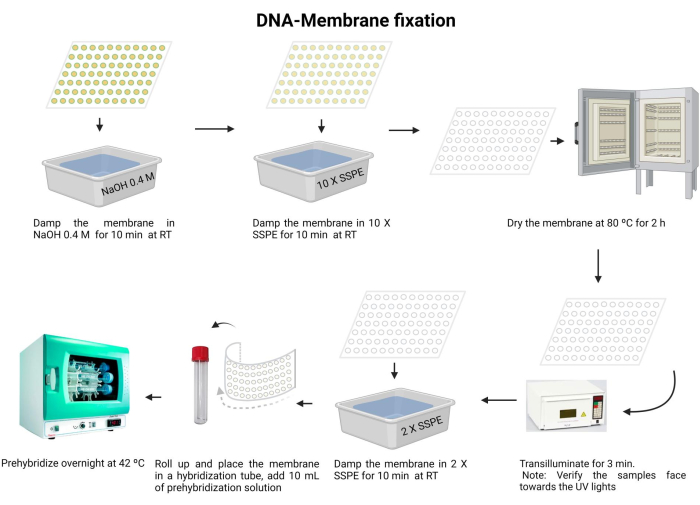
Abbildung 5: Verfahren zur DNA-Membran-Fixierung. Die DNA wird in einer alkalischen Lösung denaturiert. Anschließend wird es mit 10 X SSPE neutralisiert und die Membran getrocknet. Als nächstes wird die Membran durchleuchtet. Die Membran wird mit 2 X SSPE rehydriert und über Nacht vorhybridisiert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
- Inkubieren Sie die Membran mit 0,4 M NaOH für 10 min bei Raumtemperatur.
- Äquilibrieren Sie die Membran mit 10 X SSPE für 10 min bei Raumtemperatur.
- Trocknen Sie die Membran bei 80 °C für 2 h.
- Transilluminate mit 120 mJ UV-Licht für 3 min. Stellen Sie sicher, dass die Proben mit der Vorderseite nach unten auf die UV-Lichtquelle ausgerichtet sind. Wenn Sie einen UV-Vernetzer verwenden, vergewissern Sie sich, dass die Proben nach oben zeigen, und wiederholen Sie diesen Schritt zweimal.
HINWEIS: Die Membran muss vor der UV-Vernetzung vollständig trocken sein. Die DNA wird durch eine kovalente Bindung an die Nylonmembran immobilisiert. Jede Vernetzung dauert etwa 18'' bis 1' mit einer optimalen UV-Bestrahlungsdosis von etwa 0,6-0,8 kJ/m2 für die meisten Hybridisierungsexperimente.
Die Exposition gegenüber UV-Strahlung ist schädlich für Augen und Haut. Tragen Sie geeignete Schutzausrüstung und vermeiden Sie den Kontakt mit nackter Haut. - Waschen Sie die Membran mit 2 X SSPE für 10 min.
- Falten Sie die Membran vorsichtig und führen Sie sie in das Hybridisierungsröhrchen ein (verwenden Sie ein 15-ml-Mikrozentrifugenröhrchen).
- Geben Sie 10 mL der Prähybridisierungslösung (Ergänzende Datei 6) in das Röhrchen und inkubieren Sie sie über Nacht bei 42 °C. Stellen Sie sicher, dass der Schlauch ordnungsgemäß abgedichtet wurde, um ein Auslaufen zu verhindern.
4. Hybridisierung
- Entfernen Sie 5 mL der Prähybridisierungslösung aus dem Hybridisierungsröhrchen. Um die 5 mL ein zweites Mal zu verwenden, bewahren Sie sie in einem anderen Röhrchen auf und lagern Sie sie bei -4 °C.
- Geben Sie 15 μl der markierten Sonde (Tabelle 1) in das Hybridisierungsröhrchen. Spülen Sie die Spitze in die Lösung, um sicherzustellen, dass die markierte Sonde gründlich in die Verdünnung eingeträufelt wurde.
HINWEIS: Die Bindung der DNA-Sonde ist nicht so stark wie die der Antigen-Antikörper-Bindung. Daher können Änderungen der Konzentration der Sonde oder DNA in der Probe die Intensität der emittierten Fluoreszenz beeinflussen. DNA-Sonden sind stöchiometrisch, was bedeutet, dass die Anzahl der an die DNA gebundenen Sondenmoleküle der Anzahl der in der Lösung vorhandenen DNA-Moleküle entspricht. Abbildung 6 zeigt die Sondenhybridisierung. - Über Nacht bei 42 °C inkubieren.
- Führen Sie eine DNA-Sonden-DIG-Markierung durch. Befolgen Sie die Anweisungen in Tabelle 3 , um die Reagenzien zu mischen, und inkubieren Sie die Mischung dann 1 h lang bei 37 °C. Fügen Sie anschließend 80 μl destilliertes Wasser hinzu und lagern Sie die Schwanzsonde bei 4 °C.
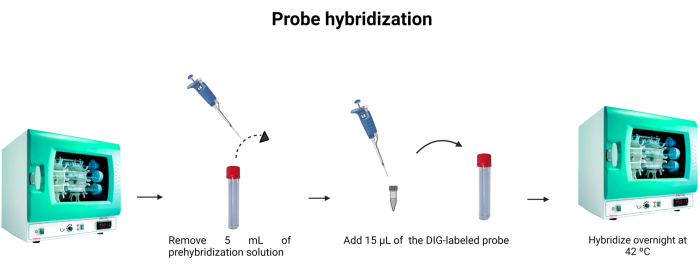
Abbildung 6: Sonden-Hybridisierung. Das Volumen des Hybridisierungspuffers wird angepasst und die mit Digoxigenin markierte Sonde wird eingebaut, um die Hybridisierung der Sonde über Nacht zu ermöglichen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Tabelle 3: Reagenzien für die Sondenmarkierung mit Digoxigenin (DIG). Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
5. Chemilumineszenz (Anti-DIG-Tagging)
- Waschen Sie die Membran zweimal mit 2 X SSPE/0,1% SDS bei Raumtemperatur für 10 min.
- Inkubieren Sie die Membran mit 5 X SSPE/0,1 % SDS bei der Glühtemperatur der Sonde für 15 min.
HINWEIS: Verwenden Sie in diesem Schritt einen Behälter mit Deckel, um die Temperatur so lange wie möglich zu halten. Sorgen Sie für eine gleichmäßige und konstante Temperatur in der gesamten Membran. Das ungefähre Volumen in jeder Wäsche beträgt 100 ml der angegebenen Lösung. - Waschen Sie die Membran einmal mit Puffer 1 (Ergänzungsdatei 6) bei Raumtemperatur für 5 min.
- Waschen Sie die Membran einmal mit Puffer 2 (Zusatzdatei 6) bei Raumtemperatur für 30 min.
- Waschen Sie die Membran mit Puffer 2 und fügen Sie den Anti-Digoxigenin-Antikörper (15 μl) hinzu (siehe Materialtabelle), dann inkubieren Sie 45 Minuten lang (die Inkubationszeit kann zwischen 30 und 60 Minuten variieren). Dieser Sekundärantikörper markiert die Sonde für den Nachweis.
HINWEIS: Die Membran kann in Puffer 2 mit dem Anti-Digoxigenin-Antikörper über Nacht bei 4-8 °C gelagert werden. Das Anti-DIG-Tagging des Chemilumineszenzprozesses ist in Abbildung 7 dargestellt.
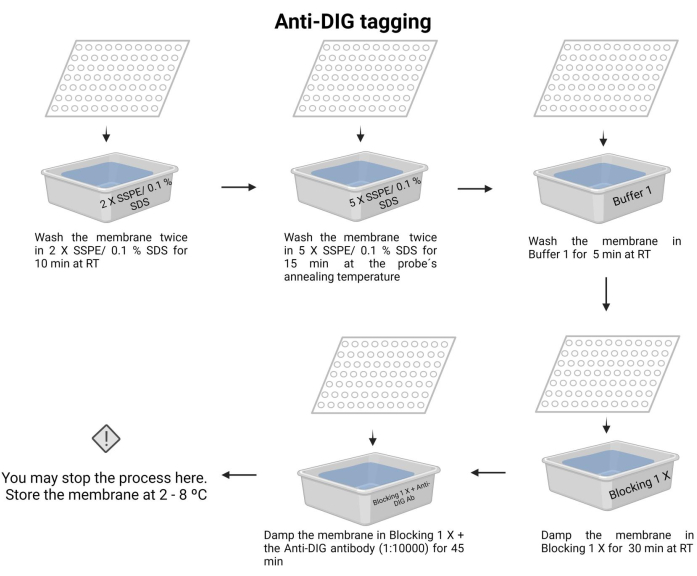
Abbildung 7: Anti-DIG-Tagging des Chemilumineszenzprozesses. Die ungebundenen Nukleinsäuren werden mit Pufferlösungen entfernt. Die Sonde wird auf die Ziel-DNA ausgerichtet und der Überschuss wird entfernt. Die Membran wird mit dem blockierenden 1 X-Puffer blockiert und der Anti-DIG-Antikörper (1:10000) hinzugefügt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
6. Chemilumineszenz (Substratanwendung)
- Waschen Sie die Membran zweimal in Puffer 1 bei Raumtemperatur für 15 min.
- Einmal in Puffer 3 (siehe Zusatzdatei 6) bei Raumtemperatur 5 Minuten waschen, damit die Membran den größten Teil des Puffers ablassen kann und fast trocken bleibt.
- Geben Sie CSPD gebrauchsfertig (Dinatrium-3-(4-methoxyspiro {1,2-dioxetan-3,2′-(5′-chlor)tricyclo [3.3.1.13,7] decan}-4-yl)phenylphosphat, siehe Materialtabelle) hinzu und lassen Sie es 5 min ruhen (mit angefeuchteten Fingern das CSPD von einer Seite zur anderen homogenisieren).
- Legen Sie die Membran in eine durchsichtige Plastiktüte (12,5 x 9 cm), die dem Membranmaß entspricht. Entfernen Sie vorsichtig Luftblasen, verteilen Sie den CSPD gleichmäßig und versiegeln Sie den Beutel mit heißem Siegel.
- Inkubieren Sie die Membran 15 Minuten lang in einem Wasserbad bei 37 °C (es kann bis zu 30 Minuten dauern) und stellen Sie sicher, dass die Membran mit den Proben nach unten gelegt wird, so dass sie gut untergetaucht ist. Stellen Sie sicher, dass die Temperatur konstant bleibt und gut über die Membran verteilt wird.
- Trocknen Sie die Plastiktüte und befestigen Sie sie mit Klebeband auf einer bereits verwendeten Röntgenfolie. Dies ermöglicht eine einfache Handhabung während des Belichtungsvorgangs.
HINWEIS: Abbildung 8 zeigt die Substratanwendung des Chemilumineszenzprozesses.

Abbildung 8: Substratapplikation des Chemilumineszenzprozesses. Der freie Antikörper wird entfernt und das Substrat CSPD (1:250) wird der Membran zugesetzt. Die Reaktion wird durch Inkubation bei 37 °C aktiviert und die Membran so angeordnet, dass sie die Chemilumineszenz in einem Röntgenfilm aufzeichnet. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
7. Chemilumineszenz (Detektion)
- Führen Sie das Belichtungsverfahren durch. Bereiten Sie in der Dunkelkammer die Entwickler- und Fixierlösungen (siehe Materialtabelle) in den jeweiligen Fächern vor.
- Legen Sie die fixierte Membran im Dunkeln vorsichtig auf einen neuen Röntgenfilm und legen Sie ihn in eine Röntgenkassette ein (siehe Materialtabelle).
- Registrieren Sie die Expositionszeit. Die Expositionszeit kann zwischen 1 Minute und 30 Minuten oder mehr variieren. Beginnen Sie mit 5 min und passen Sie die Zeit entsprechend an.
- Den Röntgenfilm in der Entwicklerlösung 1-3 min anfeuchten und vorsichtig mit Leitungswasser (15-45 s) nachspülen.
- Den Röntgenfilm 1-3 min in Fixierlösung anfeuchten und vorsichtig mit Leitungswasser (45 s) nachspülen.
- Lassen Sie den Röntgenfilm an der Luft trocknen und dokumentieren Sie den Assay in einem sichtbaren weißen Leuchtkasten.
HINWEIS: Vermeiden Sie es, die Membran während der Röntgenbelichtungszeit zu schütteln. Der Erkennungsprozess ist in Abbildung 9 dargestellt. - Fahren Sie mit der Ergebnisinterpretation fort. Im belichteten Röntgenfilm können die Punkte mit Emission visuell lokalisiert werden und entsprechen den DNA-Fragmenten mit der Hybridisierung der Sonde. Die Intensität des ausgesendeten Signals hängt von der Lumineszenz und der Dauer der Belichtung ab.
HINWEIS: Membranen können zwischen Filterpapier getrocknet mehrere Monate bei Raumtemperatur gelagert werden.
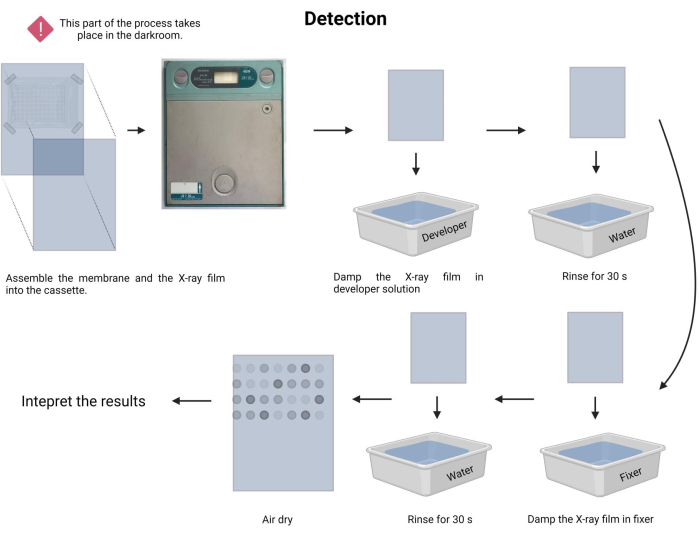
Abbildung 9: Detektion des Chemilumineszenzprozesses. Unter dunklen Bedingungen wird die Membran einem Röntgenfilm in einer Röntgenkassette ausgesetzt. Anschließend ließ man ihn während der Belichtungszeit stehen, dann wurde der Röntgenfilm entwickelt und fixiert. Zum Schluss wurde es an der Luft getrocknet und interpretiert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
8. Verfahren zur Dehybridisierung der Membran
- Waschen Sie die Membran zweimal für 10 Minuten mit destilliertem Wasser.
- Waschen Sie die Membran 20 min lang mit 0,4 M NaOH bei 53 °C (zweimal).
- Waschen Sie die Membran zweimal 10 Minuten lang mit 2 x SSPE.
- Lassen Sie die Membran bei Raumtemperatur trocknen.
- Bewahren Sie die Membran auf und kehren Sie zu Schritt 3.5 des Protokolls zurück.
HINWEIS: Die Membran kann über Nacht bei 4-8 °C im Prähybridisierungspuffer gelagert werden. Am nächsten Tag wird die Inkubation bei 42 °C für 1 h neu gestartet, damit der Vorhybridisierungspuffer die Temperatur erreichen kann. Fahren Sie dann mit Schritt 3.5 des Protokolls fort.
Ergebnisse
Um die Wirksamkeit der Technik zu bewerten, wurde genomische DNA aus Reinkulturen jedes Leptospira-Serovars zusammen mit der kladespezifischen Sonde verwendet. Die Membranen wurden mit 100 ng genomischer DNA pro PCR-Reaktion für jedes Serovar hergestellt, gefolgt von acht genomischen DNA-Partikeln von nicht verwandten Bakterien und variablen Konzentrationen genomischer DNA der Ad-hoc-Leptospira-Serovare. Jeder Assay umfasste Positiv-, Negativ- und Nicht-Template-Kontrollen. Diese nicht verwandte genomi...
Diskussion
Zu den kritischen Schritten der Dot-Blot-Technik gehören (1) DNA-Immobilisierung, (2) Blockierung der freien Bindungsstellen auf der Membran mit nicht-homologer DNA, (3) die Komplementarität zwischen der Sonde und dem Zielfragment unter Annealing-Bedingungen, (4) die Entfernung der nicht hybridisierten Sonde und (5) der Nachweis des Reportermoleküls41.
Der PCR-Dot-Blot weist bestimmte Einschränkungen auf, wie z.B. dass die Technik keine Informationen über die Grö?...
Offenlegungen
Die Autoren erklären, dass kein Interessenkonflikt besteht.
Danksagungen
Wir sind der Leptospira-Sammlung der Abteilung für Mikrobiologie und Immunologie der Fakultät für Veterinärmedizin und Tierzucht der Nationalen Autonomen Universität von Mexiko zu Dank verpflichtet. Wir danken für die großzügige Spende der Referenz-Leptospira-Stämme ; Leptospira fainei Serovar Hurstbridge Stamm BUT6 und Leptospira biflexa Serovar Patoc Stamm Patoc I an Dr. Alejandro de la Peña Moctezuma. Wir danken Dr. José Antonio Ocampo Cervantes, dem CIBAC-Koordinator, und dem Personal für ihre logistische Unterstützung. EDT war Teil des Terminal-Projektprogramms für Studenten im Grundstudium der Metropolitan Autonomous University-Campus Cuajimalpa. Wir weisen auf die Biorender.com Software für die Erstellung der Abbildungen 1 und 3 bis 9 hin.
Materialien
| Name | Company | Catalog Number | Comments |
| REAGENTS | |||
| Purelink DNA extraction kit | Invitrogen | K182002 | |
| Gotaq Flexi DNA Polimerase (End-Point PCR Taq polymerase kit) | Promega | M3001 | |
| Whatman filter paper, grade 1, | Merk | WHA1001325 | |
| Nylon Membranes, positively charged Roll 30cm x 3 m | Roche | 11417240001 | |
| Anti-Digoxigenin-AP, Fab fragments Sheep Polyclonal Primary-antibody | Roche | 11093274910 | |
| Medium Base EMJH | Difco | S1368JAA | |
| Leptospira Enrichment EMJH | Difco | BD 279510 | |
| Blocking Reagent | Roche | 11096176001 | |
| CSPD ready to use Disodium 3-(4-methoxyspiro {1,2-dioxetane-3,2′-(5′-chloro) tricyclo [3.3.1.13,7] decan}8-4-yl) phenyl phosphate | Merk | 11755633001 | |
| Deoxyribonucleic acid from herring sperm | Sigma Aldrich | D3159 | |
| Developer Carestream | Carestream Health Inc | GBX5158621 | |
| Digoxigenin-11-ddUTP | Roche | 11363905910 | |
| EDTA, Disodium Salt (Dihydrate) | Promega | H5032 | |
| Ficoll 400 | Sigma Aldrich | F8016 | |
| Fixer Carestream | Carestream Health Inc | GBX 5158605 | |
| Lauryl sulfate Sodium Salt (Sodium dodecyl sulfate; SDS) C12H2504SNa | Sigma Aldrich | L5750 | |
| N- Lauroylsarcosine sodium salt CH3(CH2)10CON(CH3) CH2COONa | Sigma Aldrich | L-9150 | It is an anionic surfactant |
| Polivinylpyrrolidone (PVP-40) | Sigma Aldrich | PVP40 | |
| Polyethylene glycol Sorbitan monolaurate (Tween 20) | Sigma Aldrich | 9005-64-5 | |
| Sodium Chloride (NaCl) | Sigma Aldrich | 7647-14-5 | |
| Sodium dodecyl sulfate (SDS) | Sigma Aldrich | 151-21-3 | |
| Sodium hydroxide (NaOH) | Sigma Aldrich | 1310-73-2 | |
| Sodium phosphate dibasic (NaH2PO4) | Sigma-Aldrich | 7558-79-4 | |
| Terminal transferase, recombinant | Roche | 3289869103 | |
| Tris hydrochloride (Tris HCl) | Sigma-Aldrich | 1185-53-1 | |
| SSPE 20X | Sigma-Aldrich | S2015-1L | It can be Home-made following Supplementary File 6 |
| Primers | Sigma-Aldrich | On demand | Follow table 1 |
| Probes | Sigma-Aldrich | On demand | Follow table 1 |
| Equipment | |||
| Nanodrop™ One Spectrophotometer | Thermo-Scientific | ND-ONE-W | |
| Refrigerated microcentrifuge Sigma 1-14K, suitable for centrifugation of 1.5 ml microcentrifuge tubes at 14,000 rpm | Sigma-Aldrich | 1-14K | |
| Disinfected adjustable pipettes, range 2-20 µl, 20-200 µl | Gilson | SKU:F167360 | |
| Disposable 1.5 ml microcentrifuge tubes (autoclaved) | Axygen | MCT-150-SP | |
| Disposable 600 µl microcentrifuge tubes (autoclaved) | Axygen | 3208 | |
| Disposable Pipette tips 1-10 µl | Axygen | T-300 | |
| Disposable Pipette tips 1-200 µl | Axygen | TR-222-Y | |
| Dot-Blot apparatus Bio-Dot | BIORAD | 1706545 | |
| Portable Hergom Suction | Hergom | 7E-A | |
| Scientific Light Box (Visible-light PH90-115V) | Hoefer | PH90-115V | |
| UV Crosslinker | Hoefer | UVC-500 | |
| Thermo Hybaid PCR Express Thermocycler | Hybaid | HBPX110 | |
| Radiographic cassette with IP Plate14 X 17 | Fuji |
Referenzen
- Bierque, E., Thibeaux, R., Girault, D., Soupé-Gilbert, M. E., Goarant, C. A systematic review of Leptospira in water and soil environments. PLOS One. 15 (1), e0227055 (2020).
- Haake, D. A., Levett, P. N. Leptospirosis in humans. Current Topics in Microbiology and Immunology. 387, 65-97 (2015).
- Tripathy, D. N., Hanson, L. E. Leptospires from water sources at Dixon Springs Agricultural Center. Journal of Wildlife Diseases. 9 (3), 209-212 (1973).
- Smith, D. J., Self, H. R. Observations on the survival of Leptospira australis A in soil and water. The Journal of Hygiene. 53 (4), 436-444 (1955).
- Karpagam, K. B., Ganesh, B. Leptospirosis: a neglected tropical zoonotic infection of public health importance-an updated review. European Journal of Clinical Microbiology & Infectious Diseases: Official Publication of the European Society of Clinical Microbiology. 39 (5), 835-846 (2020).
- Casanovas-Massana, A., et al. Spatial and temporal dynamics of pathogenic Leptospira in surface waters from the urban slum environment. Water Research. 130, 176-184 (2018).
- Costa, F., et al. Global morbidity and mortality of Leptospirosis: A systematic review. PLOS Neglected Tropical Diseases. 9 (9), e0003898 (2015).
- Mwachui, M. A., Crump, L., Hartskeerl, R., Zinsstag, J., Hattendorf, J. Environmental and behavioural determinants of Leptospirosis transmission: A systematic review. PLOS Neglected Tropical Diseases. 9 (9), e0003843 (2015).
- Andre-Fontaine, G., Aviat, F., Thorin, C. Waterborne Leptospirosis: Survival and preservation of the virulence of pathogenic Leptospira spp. in fresh water. Current Microbiology. 71 (1), 136-142 (2015).
- Trueba, G., Zapata, S., Madrid, K., Cullen, P., Haake, D. Cell aggregation: A mechanism of pathogenic Leptospira to survive in freshwater. International Microbiology: the Official Journal of the Spanish Society for Microbiology. 7 (1), 35-40 (2004).
- Smith, C. E., Turner, L. H. The effect of pH on the survival of leptospires in water. Bulletin of the World Health Organization. 24 (1), 35-43 (1961).
- Barragan, V. A., et al. Interactions of Leptospira with environmental bacteria from surface water. Current Microbiology. 62 (6), 1802-1806 (2011).
- Abdoelrachman, R. Comparative investigations into the influence of the presence of bacteria on the life of pathogenic and apathogenic leptospirae. Antonie van Leeuwenhoek. 13 (1), 21-32 (1947).
- Singh, R., et al. Microbial diversity of biofilms in dental unit water systems. Applied and Environmental Microbiology. 69 (6), 3412-3420 (2003).
- Kumar, K. V., Lall, C., Raj, R. V., Vedhagiri, K., Vijayachari, P. Coexistence and survival of pathogenic leptospires by formation of biofilm with Azospirillum. FEMS Microbiology Ecology. 91 (6), 051 (2015).
- Yanagihara, Y., et al. Leptospira Is an environmental bacterium that grows in waterlogged soil. Microbiology Spectrum. 10 (2), 0215721 (2022).
- Gillespie, R. W., Ryno, J. Epidemiology of leptospirosis. American Journal of Public Health and Nation’s Health. 53 (6), 950-955 (1963).
- Bierque, E., et al. Leptospira interrogans retains direct virulence after long starvation in water. Current Microbiology. 77 (10), 3035-3043 (2020).
- Zhang, Y., Dai, B. Marking and detection of DNA of leptospires in the dot-blot and situ hybridization with digoxigenin-labeled probes. Journal of West China University of Medical Sciences. 23 (4), 353-435 (1992).
- Mérien, F., Amouriaux, P., Perolat, P., Baranton, G., Saint Girons, I. Polymerase chain reaction for detection of Leptospira spp. in clinical samples. Journal of Clinical Microbiology. 30 (9), 2219-2224 (1992).
- Veerapandian, R., et al. Silver enhanced nano-gold dot-blot immunoassay for leptospirosis. Journal of Microbiological Methods. 156, 20-22 (2019).
- Junpen, S., et al. Evaluation of a monoclonal antibody-based dot-blot ELISA for detection of Leptospira spp in bovine urine samples. American Journal of Veterinary Research. 66 (5), 762-766 (2005).
- Ishmael, F. T., Stellato, C. Principles and applications of polymerase chain reaction: basic science for the practicing physician. Annals of Allergy, Asthma & Immunology: Official Publication of the American College of Allergy, Asthma, & Immunology. 101 (4), 437-443 (2008).
- Boerner, B., Weigelt, W., Buhk, H. J., Castrucci, G., Ludwig, H. A sensitive and specific PCR/Southern blot assay for detection of bovine herpesvirus 4 in calves infected experimentally. Journal of Virological Methods. 83 (1-2), 169-180 (1999).
- Curry, E., Pratt, S. L., Kelley, D. E., Lapin, D. R., Gibbons, J. R. Use of a Combined duplex PCR/Dot-blot assay for more sensitive genetic characterization. Biochemistry Insights. 1, 35-39 (2008).
- Pilatti, M. M., Ferreira, S. d. e. A., de Melo, M. N., de Andrade, A. S. Comparison of PCR methods for diagnosis of canine visceral leishmaniasis in conjunctival swab samples. Research in Veterinary Science. 87 (2), 255-257 (2009).
- Conrads, G., et al. PCR reaction and dot-blot hybridization to monitor the distribution of oral pathogens within plaque samples of periodontally healthy individuals. Journal of Periodontology. 67 (10), 994-1003 (1996).
- Langa, S., et al. Differentiation of Enterococcus faecium from Lactobacillus delbrueckii subsp. bulgaricus and Streptococcus thermophilus strains by PCR and dot-blot hybridisation. International Journal of Food Microbiology. 88 (2-3), 197-200 (2003).
- Francesca, C., Lucilla, I., Marco, F., Giuseppe, C., Marisa, M. Identification of the unculturable bacteria Candidatus arthromitus in the intestinal content of trouts using dot-blot and Southern blot techniques. Veterinary Microbiology. 156 (3-4), 389-394 (2012).
- Arent, Z., Pardyak, L., Dubniewicz, K., Plachno, B. J., Kotula-Balak, M. Leptospira taxonomy: then and now. Medycyna Weterynaryjna. 78 (10), 489-496 (2022).
- Thibeaux, R., et al. Biodiversity of environmental Leptospira: Improving identification and revisiting the diagnosis. Frontiers in Microbiology. 9, 816 (2018).
- Carrillo-Casas, E. M., Hernández-Castro, R., Suárez-Güemes, F., de la Peña-Moctezuma, A. Selection of the internal control gene for real-time quantitative RT-PCR assays in temperature treated Leptospira. Current Microbiology. 56 (6), 539-546 (2008).
- Azali, M. A., Yean Yean, C., Harun, A., Aminuddin Baki A, N. N., Ismail, N. Molecular characterization of Leptospira spp. in environmental samples from North-Eastern Malaysia revealed a pathogenic strain, Leptospira alstonii. Journal of Tropical Medicine. 2016, 2060241 (2016).
- Ahmed, N., et al. Multilocus sequence typing method for identification and genotypic classification of pathogenic Leptospira species. Annals of Clinical Microbiology and Antimicrobials. 5, 28 (2006).
- Bourhy, P., Collet, L., Brisse, S., Picardeau, M. Leptospira mayottensis sp. nov., a pathogenic species of the genus Leptospira isolated from humans. International Journal of Systematic and Evolutionary Microbiology. 64, 4061-4067 (2014).
- Weiss, S., et al. An extended Multilocus Sequence Typing (MLST) scheme for rapid direct typing of Leptospira from clinical samples. PLOS Neglected Tropical Diseases. 10 (9), e0004996 (2016).
- Branger, C., et al. Polymerase chain reaction assay specific for pathogenic Leptospira based on the gene hap1 encoding the hemolysis-associated protein-1. FEMS Microbiology Letters. 243 (2), 437-445 (2005).
- Ren, S. X., et al. Unique physiological and pathogenic features of Leptospira interrogans revealed by whole-genome sequencing. Nature. 422 (6934), 888-893 (2003).
- Picardeau, M., et al. Genome sequence of the saprophyte Leptospira biflexa provides insights into the evolution of Leptospira and the pathogenesis of leptospirosis. PLOS One. 3 (2), e1607 (2008).
- Kafatos, F. C., Jones, C. W., Efstratiadis, A. Determination of nucleic acid sequence homologies and relative concentrations by a dot hybridization procedure. Nucleic Acids Research. 7 (6), 1541-1552 (1979).
- Bhat, A. I., Rao, G. P. Dot-blot hybridization technique. Characterization of Plant Viruses. , 303-321 (2020).
- Yadav, J. P., Batra, K., Singh, Y., Singh, M. Comparative evaluation of indirect-ELISA and Dot-blot assay for serodetection of Mycoplasma gallisepticum and Mycoplasma synoviae antibodies in poultry. Journal of Microbiological Methods. 189, 106317 (2021).
- Malinen, E., Kassinen, A., Rinttilä, T., Palva, A. Comparison of real-time PCR with SYBR Green I or 5'-nuclease assays and dot-blot hybridization with rDNA-targeted oligonucleotide probes in quantification of selected faecal bacteria. Microbiology. 149, 269-277 (2003).
- Wyss, C., et al. Treponema lecithinolyticum sp. nov., a small saccharolytic spirochaete with phospholipase A and C activities associated with periodontal diseases. International Journal of Systematic Bacteriology. 49, 1329-1339 (1999).
- Shah, J. S., I, D. C., Ward, S., Harris, N. S., Ramasamy, R. Development of a sensitive PCR-dot-blot assay to supplement serological tests for diagnosing Lyme disease. European Journal of Clinical Microbiology & Infectious Diseases: Official Publication of the European Society of Clinical Microbiology. 37 (4), 701-709 (2018).
- Niu, C., Wang, S., Lu, C. Development and evaluation of a dot-blot assay for rapid determination of invasion-associated gene ibeA directly in fresh bacteria cultures of E. coli. Folia microbiologica. 57 (6), 557-561 (2012).
- Wetherall, B. L., McDonald, P. J., Johnson, A. M. Detection of Campylobacter pylori DNA by hybridization with non-radioactive probes in comparison with a 32P-labeled probe. Journal of Medical Microbiology. 26 (4), 257-263 (1988).
- Kolk, A. H., et al. Detection of Mycobacterium tuberculosis in clinical samples by using polymerase chain reaction and a nonradioactive detection system. Journal of Clinical Microbiology. 30 (10), 2567-2575 (1992).
- Scherer, L. C., et al. PCR colorimetric dot-blot assay and clinical pretest probability for diagnosis of Pulmonary Tuberculosis in smear-negative patients. BMC Public Health. 7, 356 (2007).
- Armbruster, D. A., Pry, T. Limit of blank, limit of detection and limit of quantitation. The Clinical Biochemist Reviews. 29, S49-S52 (2008).
- Zhang, Y., Dai, B. Detection of Leptospira by dot-blot hybridization with photobiotin- and 32P-labeled DNA. Journal of West China University of Medical Sciences = Huaxi like daxue xuebao. 23 (2), 130-132 (1992).
- Terpstra, W. J., Schoone, G. J., ter Schegget, J. Detection of leptospiral DNA by nucleic acid hybridization with 32P- and biotin-labeled probes. Journal of Medical Microbiology. 22 (1), 23-28 (1986).
- Shukla, J., Tuteja, U., Batra, H. V. DNA probes for identification of leptospires and disease diagnosis. The Southeast Asian Journal of Tropical Medicine and Public Health. 35 (2), 346-352 (2004).
- Jiang, N., Jin, B., Dai, B., Zhang, Y. Identification of pathogenic and nonpathogenic leptospires by recombinant probes. Journal of West China University of Medical Sciences = Huaxi like daxue xuebao. 26 (1), 1-5 (1995).
- Fach, P., Trap, D., Guillou, J. P. Biotinylated probes to detect Leptospira interrogans on dot-blot hybridization or by in situ hybridization. Letters in Applied Microbiology. 12 (5), 171-176 (1991).
- Huang, N., Dai, B. Assay of genomic DNA homology among strains of different virulent leptospira by DNA hybridization. Journal of West China University of Medical Sciences = Huaxi like daxue xuebao. 23 (2), 122-125 (1992).
- Dong, X., Dai, B., Chai, J. Homology study of leptospires by molecular hybridization. Journal of West China University of Medical Sciences = Huaxi like daxue xuebao. 23 (1), 1-4 (1992).
- Komminoth, P. Digoxigenin as an alternative probe labeling for in situ hybridization. Diagnostic Molecular Pathology: The American Journal of Surgical Pathology, part B. 1 (2), 142-150 (1992).
- Saengjaruk, P., et al. Diagnosis of human leptospirosis by monoclonal antibody-based antigen detection in urine. Journal of Clinical Microbiology. 40 (2), 480-489 (2002).
- Okuda, M., et al. Enzyme-linked immunosorbent assay for the detection of canine Leptospira antibodies using recombinant OmpL1 protein. The Journal of Veterinary Medical Science. 67 (3), 249-254 (2005).
- Suwimonteerabutr, J., et al. Evaluation of a monoclonal antibody-based dot-blot ELISA for detection of Leptospira spp in bovine urine samples. American Journal of Veterinary Research. 66 (5), 762-766 (2005).
- Kanagavel, M., et al. Peptide-specific monoclonal antibodies of Leptospiral LigA for acute diagnosis of leptospirosis. Scientific reports. 7 (1), 3250 (2017).
- Levett, P. N. Leptospirosis. Clinical Microbiology Reviews. 14 (2), 296-326 (2001).
- Monahan, A. M., Callanan, J. J., Nally, J. E. Proteomic analysis of Leptospira interrogans shed in urine of chronically infected hosts. Infection and Immunity. 76 (11), 4952-4958 (2008).
- Rojas, P., et al. Detection and quantification of leptospires in urine of dogs: a maintenance host for the zoonotic disease leptospirosis. European Journal of Clinical Microbiology & Infectious Diseases: Official Publication of the European Society of Clinical Microbiology. 29 (10), 1305-1309 (2010).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten