Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Pseudotypisierte Viren als molekulares Werkzeug zur Überwachung humoraler Immunantworten gegen SARS-CoV-2 mittels Neutralisationsassay
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Pseudotypisierte Viren (PVs) sind replikationsdefekte Virionen, die verwendet werden, um Wirt-Virus-Interaktionen unter sichereren Bedingungen zu untersuchen als mit authentischen Viren. Hier wird ein detailliertes Protokoll vorgestellt, das zeigt, wie SARS-CoV-2-PVs verwendet werden können, um die neutralisierende Fähigkeit des Serums von Patienten nach einer COVID-19-Impfung zu testen.
Zusammenfassung
Pseudotypisierte Viren (PVs) sind molekulare Werkzeuge, die verwendet werden können, um Wirt-Virus-Interaktionen zu untersuchen und die neutralisierende Fähigkeit von Serumproben zu testen, zusätzlich zu ihrer bekannteren Verwendung in der Gentherapie für die Verabreichung eines Gens von Interesse. PVs sind replikationsfehlerhaft, weil das virale Genom in verschiedene Plasmide unterteilt ist, die nicht in die PVs eingebaut sind. Dieses sichere und vielseitige System ermöglicht den Einsatz von PVs in Laboren der Biosicherheitsstufe 2. Hier stellen wir eine allgemeine Methodik zur Herstellung von lentiviralen PVs vor, die auf drei Plasmiden basiert, wie hier erwähnt: (1) das Rückgratplasmid, das das Reportergen trägt, das zur Überwachung der Infektion benötigt wird; (2) das Verpackungsplasmid, das die Gene für alle Strukturproteine enthält, die zur Erzeugung der PVs benötigt werden; (3) Das Expressionsplasmid der Hülloberfläche, das den Virustropismus bestimmt und den Eintritt des Virus in die Wirtszelle vermittelt. In dieser Arbeit ist SARS-CoV-2 Spike das Hüllglykoprotein, das für die Produktion von nicht-replikativen SARS-CoV-2-pseudotypisierten Lentiviren verwendet wird.
Kurz gesagt, Verpackungszellen (HEK293T) wurden mit den drei verschiedenen Plasmiden unter Verwendung von Standardmethoden co-transfiziert. Nach 48 h wurde der Überstand, der die PVs enthielt, geerntet, filtriert und bei -80 °C gelagert. Die Infektiosität von SARS-CoV-2-PVs wurde getestet, indem die Expression des Reportergens (Luciferase) in einer Zielzelllinie 48 h nach der Infektion untersucht wurde. Je höher der Wert für relative Lumineszenzeinheiten (RLUs) ist, desto höher ist die Infektions-/Transduktionsrate. Darüber hinaus wurden die infektiösen PVs zu den seriell verdünnten Serumproben hinzugefügt, um den Neutralisationsprozess des Eintritts von Pseudoviren in die Zielzellen zu untersuchen, gemessen als Verringerung der RLU-Intensität: niedrigere Werte entsprechen einer hohen neutralisierenden Aktivität.
Einleitung
Pseudotypisierte Viren (PVs) sind molekulare Werkzeuge, die in der Mikrobiologie verwendet werden, um Wirt-Virus- und Pathogen-Pathogen-Interaktionen zu untersuchen 1,2,3,4. PVs bestehen aus einem inneren Teil, dem viralen Kern, der das virale Genom schützt, und einem äußeren Teil, den Hüllglykoproteinen auf der Oberfläche des Virus, die den Tropismus definieren5. Ein Pseudovirus ist in der Zielzelle replikationsunfähig, da es nicht die gesamte genetische Information enthält, um neue Viruspartikel zu erzeugen. Diese Kombination von besonderen Eigenschaften macht PVs zu einer sicheren Alternative zu einem Wildtyp-Virus. Wildtypviren hingegen sind hochpathogen und können in BSL 2-Laboren nicht zur Analyse verwendet werden6.
Die Infektiosität von PVs kann durch ein Reportergen überwacht werden, das in der Regel für ein fluoreszierendes Protein (GFP, RFP, YFP) oder ein Enzym kodiert, das chemilumineszierende Produkte (Luciferase) produziert. Dieses ist in einem der Plasmide enthalten, die für die PV-Produktion verwendet werden, und wird in das Genom des Pseudoviruseingebaut 7.
Derzeit gibt es verschiedene Arten von PV-Kernen, darunter lentivirale Partikel, die auf dem Genom von HIV-1 basieren. Der große Vorteil von HIV-1-basierten PVs gegenüber anderen Plattformen ist ihr intrinsischer Integrationsprozess im Genom der Zielzelle8. Obwohl HIV-1 ein hochansteckendes Virus ist und der Erreger von AIDS ist, sind diese lentiviralen Vektoren aufgrund der umfangreichen Optimierungsschritte im Laufe der Jahre sicher in der Anwendung. Optimale Sicherheitsbedingungen wurden mit der Einführung von lentiviralen Vektoren der 2. Generation erreicht, bei denen virale Gene depleutet wurden, ohne die Transduktionsfähigkeit zu beeinflussen9. Die 3. und 4. Generation trugen mit der weiteren Aufspaltung des viralen Genoms in separate Plasmide zur erhöhten Sicherheit des lentiviralen Vektorhandlings bei10,11. Die neuesten Generationen von PVs werden in der Regel zur Herstellung von lentiviralen Vektoren für die Gentherapie eingesetzt.
PVs können verwendet werden, um Interaktionen zwischen Viren und Wirtszellen sowohl während der Produktions- als auch während der Infektionsphase zu untersuchen. PVs werden insbesondere in Pseudovirus-Neutralisationsassays (PVNA) eingesetzt. PVNAs werden umfassend validiert, um das Neutralisationspotenzial von Serum oder Plasma zu bewerten, indem sie auf das virale Glykoprotein auf der Hülle des PV abzielen12,13. Die Neutralisationsaktivität, ausgedrückt als Hemmkonzentration 50 (IC50), ist definiert als die Verdünnung von Serum/Plasma, die 50 % des Eintritts von Viruspartikeln blockiert14. In diesem Protokoll beschrieben wir den Aufbau einer PVNA, um die Antikörperaktivität gegen das Severe Acute Respiratory Syndrome - Coronavirus 2 (SARS-CoV-2) in Seren zu testen, die vor und nach Erhalt einer Auffrischungsimpfung gesammelt wurden.
Protokoll
Das vorliegende Protokoll wurde von der Ethikkommission der Universität Verona genehmigt und folgt den Richtlinien dieser Kommission (Zulassungsprotokoll Nr. 1538). Von den an der Studie teilnehmenden Probanden wurde eine schriftliche Einwilligung nach Aufklärung eingeholt. Vollblutproben wurden von Freiwilligen des Gesundheitspersonals (HCW) gesammelt, die gerade dabei waren, Anti-SARS-CoV-2-Impfstoffe zu erhalten. Diese Proben wurden in Kunststoffröhrchen mit Antikoagulanzien für die anschließende Isolierung von Serum15 gesammelt.
Alle folgenden Prozesse müssen in einer biologischen Haube der Klasse 2 unter sterilen Bedingungen durchgeführt werden. Der Umgang mit Viren muss mit Vorsicht durchgeführt werden, und alle Abfallprodukte müssen in einer verdünnten Bleichlösung neutralisiert werden. Eine Übersicht über das Protokoll ist in Abbildung 1 dargestellt.
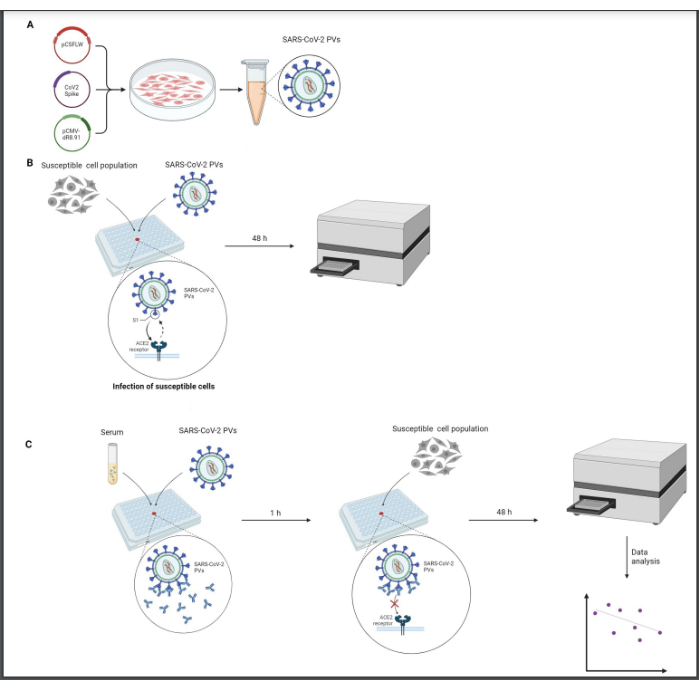
Abbildung 1: Grafische Darstellung eines Neutralisationsassays. (A) PV-Produktion, (B) PV-Titration und (C) Neutralisations-Assay. Alle Eingriffe werden in einer biologischen Haube der Klasse 2 unter sterilen Bedingungen durchgeführt. Der Titrationsschritt (B) muss durchgeführt werden, um die Infektiosität von PVs vor der Verwendung im Neutralisationsassay (C) zu standardisieren. Diese Figur wurde mit BioRender erstellt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
1. SARS-CoV-2-PV-Produktions- und Infektiositätstest
- Säen Sie 5 x 105 HEK293T Zellen in vollständigem Dulbecco's Modified Eagle Medium (DMEM, hoher Glukosegehalt, 10 % fötales Kälberserum (FBS), 1 % L-Glutamin, 1 % Penicillin/Streptomycin) in einer 6-Well-Platte (6WP), um eine geeignete Zelldichte zu erreichen, die mit dem verwendeten Transfektionsreagenz kompatibel ist. Bei der Transfektion mit Polyehtylenimin (PEI) (Reagenz gemäß den Anweisungen des Herstellers vorbereiten) ist darauf zu achten, dass die Zellen am Tag der Transfektion eine Dichte von 40-60 % erreichen (Schritt 1.3). Bewahren Sie die Zellen in einem befeuchteten Inkubator bei 37 °C und 5 % CO2 auf.
- Ersetzen Sie vor der Transfektion das verbrauchte Zellmedium durch ein frisches Medium ohne Antibiotika (DMEM, hohe Glukose, 10 % FBS, 1 % L-Glutamin), um eine höhere Transfektionseffizienz zu erreichen.
HINWEIS: Am Tag nach der Aussaat sind HEK293T Zellen bereit für die Transfizierung. - Transfektieren Sie adhärente HEK293T Zellen mit einem geeigneten Transfektionsreagenz gemäß den Anweisungen des Herstellers. Wenn Sie PEI verwenden, bereiten Sie zwei Mischungen vor und führen Sie die folgenden Schritte aus.
- Zur Herstellung von Mischung A werden 500 ng pCMV-dR8.91-Verpackungsplasmid16, 750 ng pCSFLW-Reporterplasmid16 und 450 ng SARS-CoV-2-Spike-exprimierendes Plasmid in 100 μl reduziertem Serummedium zugegeben.
- Zur Herstellung von Mischung B werden 17,5 μl PEI (Konzentration: 1 mg/ml) zu 100 μl des reduzierten Serummediums gegeben.
- Lassen Sie beide Mischungen 5 Minuten lang bei Raumtemperatur (RT) inkubieren. Als nächstes mischst du den Inhalt beider Röhrchen miteinander, indem du die PEI-Mischung B zu der DNA-Mischung A hinzufügst.
- Inkubieren Sie das Röhrchen für 20-30 Minuten bei RT. Schnippen Sie das Röhrchen vorsichtig alle 3-4 Minuten, um das Mischen zu verbessern. Zum Schluss die Mischung in die HEK293T Zellen geben.
- 16-20 h nach der Transfektion das Nährmedium durch frisches, vollständiges DMEM ersetzen. Inkubation bei 37 °C und 5 % CO2, um die Herstellung von PVs durch transfizierte Zellen zu ermöglichen.
- 72 Stunden nach der Transfektion wird der PV-haltige Überstand geerntet. Dann bei 1600 x g für 7 min bei Raumtemperatur zentrifugieren, um Zelltrümmer und abgestorbene Zellen zu entfernen, und durch einen 0,45 μm Celluloseacetatfilter filtern.
- OPTIONALER SCHRITT: Um die endgültige Ausbeute an PV-Titer zu erhöhen, führen Sie mehrere Transfektionen durch, poolen Sie die PV-haltigen Zellmedien und konzentrieren Sie sie mit Konzentrationsröhrchen.
- Fahren Sie direkt mit den nächsten Schritten fort ("PVs-Titration", Abschnitt 2) oder aliquotieren Sie das PV-haltige Medium in geeigneten Röhrchen, um es bis zur Verwendung bei -80 °C zu lagern. Bereiten Sie ein zusätzliches Aliquot (400-500 μl) vor, das für die Titration verwendet wird.
HINWEIS: Die Herstellung mehrerer Aliquots garantiert die Reproduzierbarkeit zwischen den Experimenten, indem übermäßige Auftau-Gefrier-Zyklen vermieden werden.
2. PVs-Titration
- Verwenden Sie das frische PV-haltige Medium für die nächsten Schritte oder tauen Sie das Testaliquot (Schritt 1.7) auf, um die Titration des neuen Virusbestands durchzuführen. Das Einfrieren von Aliquoten desselben PV-Bestands garantiert die Reproduzierbarkeit.
- Geben Sie 50 μl vollständiges DMEM (oder ein komplettes Medium, das mit der verwendeten Zielzelllinie kompatibel ist) in alle Vertiefungen einer 96-Well-Platte (96 WP), die erforderlich sind, um den PV-Bestand doppelt zu testen, und lassen Sie die Zeile "A" leer. 100 μl PV-Brühe in die Zeile "A" geben. Basierend auf der Anzahl der zu testenden Präparate wird eine Spalte ohne das Virus als "Nur-Zell"-Kontrolle belassen (Abbildung 2).
- Pipettieren Sie 50 μl von Reihe A in Reihe B und wiederholen Sie diesen Vorgang bis Reihe G, um serielle Verdünnungen des Ausgangsmaterials zu erhalten. Verwerfen Sie das überschüssige Volumen aus der letzten Reihe.
- Trennen Sie die Zellen mit Trypsin/Ethylendiamintetraessigsäure 1x (EDTA) in Dulbeccos Phosphatpuffersalzlösung 1x (DPBS 1x), nachdem Sie das verbrauchte Medium entfernt und die Zellen zweimal mit DPBS 1x gewaschen haben. Bereiten Sie die Zellen auf eine Dichte von 4 x 105 Zellen/ml vor.
HINWEIS: In diesem Protokoll wurde die PV-Infektion an der anfälligen Zelllinie HEK293T/ACE2 getestet; Solche Zellen wurden aus HEK293T gewonnen, die mit einem lentiviralen Vektor transduziert wurden, um den ACE2-Rezeptor zu exprimieren. - Geben Sie 50 μl der Zellsuspension in jede Vertiefung, um eine Zellzahl von 2 x 104 Zellen pro Vertiefung zu gewährleisten.
- Bei 37 °C und 5 % CO2 48 h inkubieren.
- Führen Sie nach der Inkubation den Luciferase-Assay durch, um den Messwert gemäß den Anweisungen des Herstellers zu erhalten. Geben Sie 100 μl des Luciferase-Reagenzes in die Vertiefungen und inkubieren Sie im Dunkeln bei RT für 2 Minuten. Verschieben Sie den Inhalt jeder Vertiefung auf eine schwarze 96-Well-Platte (kompatibel mit dem verfügbaren Plattenleser) und lesen Sie die Platten in einem 96-Well-Plattenleser ab.
HINWEIS: Das Luminometer, das für die Luziferase-Auslesung verwendet wird, erstellt eine Tabellenkalkulationsdatei mit den rohen, unverarbeiteten Daten, die für die nachgelagerte Analyse verwendet werden (in diesem Fall eine Excel-Datei). Die Infektiosität des Virus wird als relative Lumineszenzeinheiten (RLU) ausgedrückt (siehe Abschnitt 4.1).
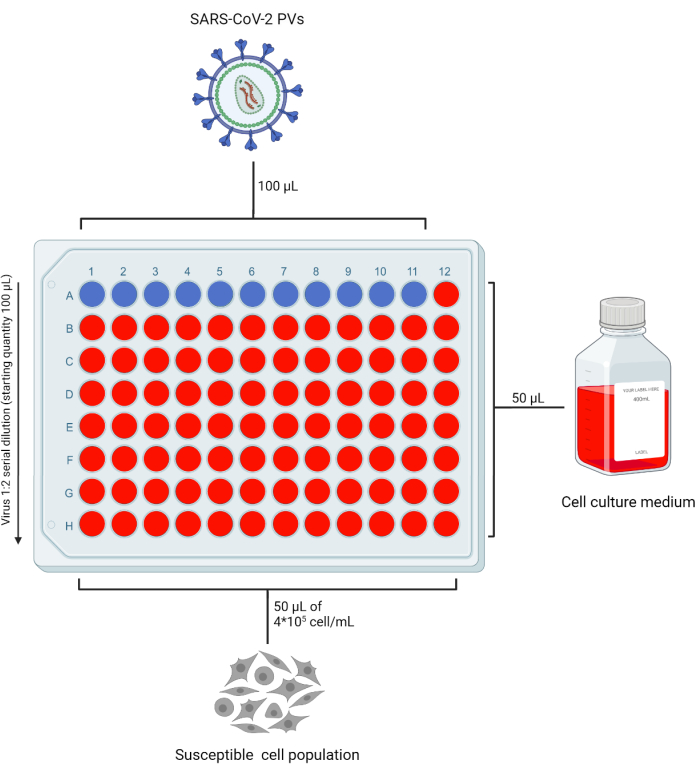
Abbildung 2: Repräsentatives Layout einer 96-Well-Platte für die PV-Titration. Ein festes Volumen des PV-haltigen Überstandes wird in Zeile A, Spalten 1-11 zugegeben und seriell verdünnt. Die letzte Spalte wird als "Nur Zelle"-Steuerelement belassen. Diese Figur wurde mit BioRender erstellt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
3. Neutralisations-Assay
- Tauen Sie die Seren der Patienten auf Eis auf. Inaktivieren Sie Serumproben, indem Sie sie 30 Minuten lang bei 56 °C inkubieren.
- Geben Sie 50 μl des frischen, vollständigen DMEM (oder eines vollständigen Mediums, das mit der verwendeten Zielzelllinie kompatibel ist) in jede der folgenden Vertiefungen: von Reihe B (Spalten 1-10) bis Reihe H (Spalten 1-10). Geben Sie 95 μl des frischen, vollständigen DMEM in Reihe A (Spalten 1-10). Geben Sie 50 μl und 100 μl vollständiges DMEM in die Vertiefungen der Säulen 11 bzw. 12. Dabei handelt es sich um die infizierten (Virus Control oder VC) bzw. nicht infizierten (cell only oder CC) Kontrollen (Abbildung 3).
- 5 μl hitzeinaktivierte Serum-/Plasmaproben in Zeile A (Spalten 1-10) geben. Jede Probe wird in zweifacher Ausfertigung vorliegen. Mischen Sie die Proben mit einer Mehrkanalpipette in der ersten Reihe und bewegen Sie 50 μl serumhaltiges Medium von Reihe A nach Reihe B. Wiederholen Sie diesen Vorgang bis zur letzten Reihe (Abbildung 3). Die restlichen 50 μl verwerfen.
- Die erforderliche Anzahl von Aliquoten der PV auftauen und auf ≥10 4 RLU/ml verdünnen. Geben Sie 50 μl des verdünnten PV-haltigen Mediums in jede Vertiefung (von Spalte 1 bis Spalte 11) mit einer Mehrkanalpipette, um eine 1:1-Verdünnung des hitzeinaktivierten Serums/Plasmas mit dem Virus zu erreichen. Bei 37 °C und 5 % CO2 für 1 h inkubieren, damit die Antikörper in den Serumproben an das SARS-CoV-2-Spike-Protein auf den PVs binden können.
- Mindestens 5 ml Suspension anfälliger Zellen (HEK293T/ACE2) bei einer Zelldichte von 4 x 105 Zellen/ml herstellen. 50 μl der Zellsuspension in jede Vertiefung geben und bei 37 °C und 5 %CO2 48 h lang inkubieren.
- Nach der Inkubation ist die Luziferase-Assay-Messung gemäß den Anweisungen des Herstellers durchzuführen, wie in Schritt 2.7 beschrieben.
HINWEIS: Das Luminometer, das für die Luciferase-Auslesung verwendet wird, erzeugt eine Tabellenkalkulationsdatei (in diesem Fall .xlsx) mit den rohen, unverarbeiteten Daten, die für die nachgelagerte Analyse verwendet werden (die Luciferase-Assay-Datei).
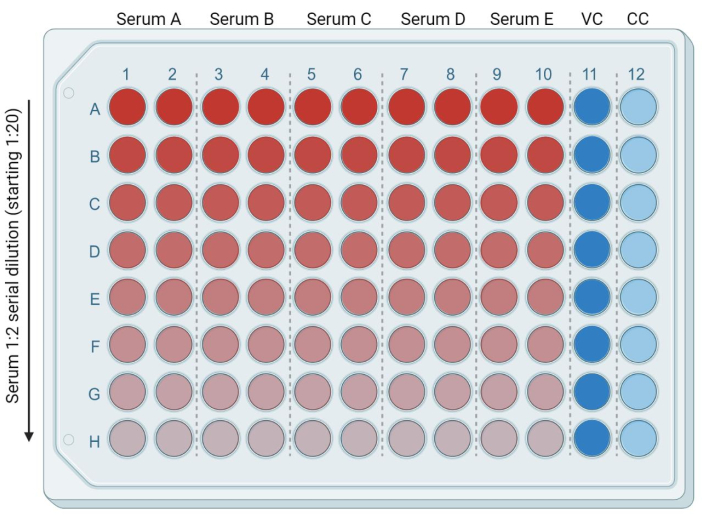
Abbildung 3: Plattendarstellung basierend auf der Serumverdünnung. Helles Rot entspricht einer höheren Serummenge und hellblaue Spur (Spalte 11) entspricht der Kontrolle infizierter Zellen (VC, Viruskontrolle). Die hellblaue Spur (Spalte 12) entspricht nicht infizierten Zellen (CC, Zellkontrolle). Diese Figur wurde mit BioRender erstellt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
4. Titrationsanalyse
- Weisen Sie in der Luciferase-Assay-Datei den entsprechenden Proben die Namen/Titel zu.
- Multiplizieren Sie die RLU-Messung mit den Verdünnungsfaktoren (von oben nach unten im Raster: 20x, 40x, 80x, 160x, 320x, 640x, 1.280x, 2.560x), um RLU/ml zu erhalten. Wenn unterschiedliche Verdünnungsfaktoren verwendet werden, ändern Sie die Multiplikationsfaktoren entsprechend.
- Berechnen Sie die durchschnittliche RLU/ml für jede PV-Vorbereitung.
5. Analyse des Neutralisations-Assays von PVs
- Weisen Sie in der Luciferase-Assay-Tabellenkalkulationsdatei (in diesem Fall .xlsx) den getesteten Proben die entsprechenden Titel zu. Geben Sie den Verdünnungsfaktor der Probe ein (40s, 80x, 160x, 320x, 640x, 1.280x, 2.560x, 5.120x). Berechnen Sie den Log10 der Verdünnungsfaktoren.
- Berechnen Sie die durchschnittliche RLU der nicht infizierten und infizierten Kontrollgruppe (Abbildung 3, Spalten 11 bzw. 12). Diese Werte sind für die Normalisierung in Schritt 5.5 nützlich.
- Öffnen Sie ein neues Dokument für die Datenanalyse. Wählen Sie X/Y-Analyse aus, geben Sie X als Zahlen und Y als Geben Sie 2 ein, um Werte in nebeneinander liegenden Unterspalten zu replizieren.
- Geben Sie Log10-Werte (Verdünnung) als X-Zahlen ein. Geben Sie das Duplikat der RLU der Proben ein.
- Gehen Sie zu Analysieren > Normalisieren > Kennzeichnen Sie alle Proben auf demselben Blatt. Geben Sie die durchschnittlichen VC- und CC-Werte in Wie ist 0 % definiert? bzw. Wie ist 100 % definiert? ein. Klicken Sie auf OK.
- Navigieren Sie auf dem normalisierten Datenblatt zu Analysieren > XY-Analysen > Nichtlineare Analysen (Kurvenanpassung). Markieren Sie alle Beispiele, und klicken Sie auf OK. Wählen Sie für die Dosis-Wirkungs-Hemmung log(inhibitor) vs. normalisierte Reaktion - variable Steigung.
- Ändern Sie unter Einschränken HillSlope in Muss kleiner als 0 sein.
- Markieren Sie unter Ausgabe das Kennzeichen Übersichtstabelle und Diagramm erstellen. Klicken Sie auf OK , um die abschließenden Analysen zu erhalten. Ein Arbeitsblatt mit einer Vorlage für die Analyse finden Sie in der Zusatzdatei 1.
Ergebnisse
Dieses Protokoll beschreibt die Produktion von SARS-CoV-2-PVs und eine nachgelagerte Anwendung dieser PVs zur Analyse der Neutralisationsaktivität von Serum/Plasma von Probanden, die eine Anti-COVID-19-Impfung erhalten17. Darüber hinaus kann dieses Protokoll angewendet werden, um Pseudotypen jeder besorgniserregenden SARS-CoV-2-Variante (VOC) herzustellen, um die Entwicklung der neutralisierenden Reaktion zu testen. Obwohl dieses Protokoll die Untersuchung der humoralen Immunantwort nach einer C...
Diskussion
Obwohl die Verwendung eines Wildtyp-Virus die tatsächliche Infektion simuliert, sind lentivirale PVs eine sicherere Option, um die Mechanismen zu untersuchen, die mit dem Eindringen und der Infektion von Viren verbunden sind, ohne die strengen Sicherheitsanforderungen, die für die Arbeit mit pathogenen Viren erforderlich sind 4,20,21. PVs bestehen aus einem replikationsdefekten Viruskern, der von der Oberflächenhülle eines p...
Offenlegungen
Die Autoren erklären, dass sie keinen Interessenkonflikt haben.
Danksagungen
Wir würdigen den Beitrag der freiwilligen Helfer im Gesundheitswesen. Dieses Projekt wurde vom Department of Excellence 2023/2027, MUR, Italien, unterstützt. AR und DZ wurden durch PRIN2022 (EU-Fördermittel; NextGenerationEU)
Materialien
| Name | Company | Catalog Number | Comments |
| 0.45 μm filter | SARSTEDT | 83 1826 | |
| 6-well plate | SARSTEDT | 83 3920 | |
| 96-well plate | SARSTEDT | 8,33,924 | |
| Amicon Ultra-15 Centrifugal Filter Units | Merck | 10403892 | |
| Black Opaque 96-well Microplate | Perkin Elmer | 60005270 | |
| Dulbecco's Modified Eagle Medium | SIGMA-ALDRICH | D6546 - 500ML | |
| Dulbecco's phosphate buffered saline (PBS 1x) | AUROGENE | AU-L0615-500 | |
| Foetal Bovine Serum | AUROGENE | AU-S1810-500 | |
| Graphpad Prism version 7 | graphpad dotmatics | NA | In the manuscript, we replace the commercial name with 'data analysis program' |
| HEK293T cells | ATCC | CRL-3216 | |
| HEK293T/ACE2 cells | ATCC | CRL-3216 | HEK293T has been transduced to overexpress ACE2 with a lentiviral vector. |
| L-glutamine | AUROGENE | AU-X0550-100 | |
| Luminometer - Victor3 | Perkin Elmer | HH35000500 | In the manuscript, we replace the commercial name with 'luminometer' |
| Opti-MEM | Thermo Fisher Scientific | 11058021 | In the manuscript, we replace the commercial name with 'reduced serum medium' |
| p8.91 packaging plasmid | Di Genova et al., 2021 | A kind gift from Prof. Nigel Temperton (ref 16.) | |
| pCSFLW reporter plasmid | Di Genova et al., 2021 | A kind gift from Prof. Nigel Temperton (ref 16.) | |
| Penicillin/streptomycin | AUROGENE | AU-L0022-100 | |
| Polyethylenimine, branched (PEI) (25 kDa) | SIGMA-ALDRICH | 408727 | |
| RRL.sin.cPPT.SFFV/Ace2.IRES-puro.WPRE (MT126) | Addgene | 145839 | This plasmid was used to generate HEK293Tcells/ACE2 |
| SARS-CoV-2 Spike expressing plasmid | Addgene | pGBW-m4137382 | |
| steadylite plus Reporter Gene Assay System | Perkin Elmer | 6066759 | In the manuscript, we replaced the commercial name with 'luciferase reading reagent' |
| Trypsin EDTA 1x | AUROGENE | AU-L0949-100 |
Referenzen
- Ozaki, D. A., et al. International technology transfer of a GCLP-compliant HIV-1 neutralizing antibody assay for human clinical trials. Plos One. 7 (1), e30963 (2012).
- Pouget, M., et al. Generation of liposomes to study the effect of Mycobacterium tuberculosis lipids on HIV-1 cis- and trans-infections. International Journal of Molecular Sciences. 22 (4), 1945 (2021).
- McKay, L. G. A., et al. The HCV envelope glycoprotein down-modulates NF-κB signalling and associates with stimulation of the host endoplasmic reticulum stress pathway. Frontiers in Immunology. 13, 831695 (2022).
- Xiang, Q., Li, L., Wu, J., Tian, M., Fu, Y. Application of pseudovirus system in the development of vaccine, antiviral-drugs, and neutralizing antibodies. Microbiological Research. 258, 126993 (2022).
- Li, Q., Liu, Q., Huang, W., Li, X., Wang, Y. Current status on the development of pseudoviruses for enveloped viruses. Reviews in Medical Virology. 28, e1963 (2018).
- D'Apice, L., et al. Comparative analysis of the neutralizing activity against SARS-CoV-2 Wuhan-Hu-1 strain and variants of concern: Performance evaluation of a pseudovirus-based neutralization assay. Frontiers in Immunology. 13, 981693 (2022).
- Falzarano, D., Groseth, A., Hoenen, T. Development and application of reporter-expressing mononegaviruses: current challenges and perspectives. Antiviral Research. 103, 78-87 (2014).
- Gutierrez-Guerrero, A., Cosset, F. -. L., Verhoeyen, E. Lentiviral vector pseudotypes: Precious tools to improve gene modification of hematopoietic cells for research and gene therapy. Viruses. 12, 1016 (2020).
- Zufferey, R., Nagy, D., Mandel, R. J., Naldini, L., Trono, D. Multiply attenuated lentiviral vector achieves efficient gene delivery in vivo. Nature Biotechnology. 15 (9), 871-875 (1997).
- Dull, T. A third-generation lentivirus vector with a conditional packaging system. Journal of Virology. 72 (11), 8463-8471 (1998).
- Berkhout, B. A Fourth generation lentiviral Vector: Simplifying genomic gymnastics. Molecular Therapy. 25 (8), 1741-1743 (2017).
- Wu, X. Development and evaluation of a pseudovirus-luciferase assay for rapid and quantitative detection of neutralizing antibodies against Enterovirus 71. Plos One. 8 (6), e64116 (2013).
- Ferrara, F., et al. Development of lentiviral vectors pseudotyped with Influenza B hemagglutinins: application in vaccine immunogenicity, mAb potency, and sero-surveillance studies. Frontiers in Immunology. 12, 661379 (2021).
- Hu, J., et al. Development of cell-based pseudovirus entry assay to identify potential viral entry inhibitors and neutralizing antibodies against SARS-CoV-2. Genes & Diseases. 7 (4), 551-557 (2020).
- Dalle Carbonare, L., et al. Serology study after BTN162b2 vaccination in participants previously infected with SARS-CoV-2 in two different waves versus naïve. Communications Medicine. 1 (1), 38 (2021).
- Di Genova, C., et al. Production, titration, neutralisation, storage and lyophilisation of severe acute respiratory syndrome coronavirus 2 (SARS-CoV-2) lentiviral pseudotypes. Bio-protocol. 11 (21), e4236 (2021).
- Chmielewska, A. M., Czarnota, A., Bieńkowska-Szewczyk, K., Grzyb, K. Immune response against SARS-CoV-2 variants: The role of neutralization assays. NPJ Vaccines. 6 (1), 1-8 (2021).
- Chen, Q., et al. Development and optimization of a sensitive pseudovirus-based assay for HIV-1 neutralizing antibodies detection using A3R5 cells. Human Vaccines & Immunotherapeutics. 14 (1), 199-208 (2018).
- Gauger, P. C., Vincent, A. L. Serum virus neutralization assay for detection and quantitation of serum neutralizing antibodies to influenza A virus in swine. Methods in Molecular Biology (Clifton, N.J). 2123, 321-333 (2020).
- Miglietta, R., Pastori, C., Venuti, A., Ochsenbauer, C., Lopalco, L. Synergy in monoclonal antibody neutralization of HIV-1 pseudoviruses and infectious molecular clones. Journal of Translational Medicine. 12 (1), 346 (2014).
- Chen, M., Zhang, X. -. E. Construction and applications of SARS-CoV-2 pseudoviruses: A mini review. International Journal of Biological Sciences. 17 (6), 1574-1580 (2021).
- Zipeto, D., et al. Induction of human immunodeficiency virus neutralizing antibodies using fusion complexes. Microbes and Infection. 8 (6), 1424-1433 (2006).
- WHO Coronavirus (COVID-19) Dashboard. Available from: https://covid19.who.int (2022)
- Zhou, P. A pneumonia outbreak associated with a new coronavirus of probable bat origin. Nature. 579 (7798), 270-273 (2020).
- Chen, X., Huang, H., Ju, J., Sun, R., Zhang, J. Impact of vaccination on the COVID-19 pandemic in U.S. states. Scientific Reports. 12 (1), 1554 (2022).
- Stefani, C., Fantoni, T., Bissoli, M., Thomas, J., Ruggiero, A. HIV and SARS-CoV-2 Co-Infection: From Population Study Evidence to In Vitro Studies. Life. 12 (12), 2089 (2022).
- Watson, O. J., et al. Global impact of the first year of COVID-19 vaccination: a mathematical modelling study. The Lancet Infectious Diseases. 22 (9), 1293-1302 (2022).
- Cantoni, D. Analysis of antibody neutralisation activity against SARS-CoV-2 variants and seasonal human coronaviruses NL63, HKU1, and 229E induced by three different COVID-19 vaccine olatforms. Vaccines. 11 (1), 58 (2023).
- Siracusano, G., et al. Different decay of antibody response and VOC sensitivity in naïve and previously infected subjects at 15 weeks following vaccination with BNT162b2. Journal of Translational Medicine. 20 (1), 22 (2022).
- Ruggiero, A. SARS-CoV-2 vaccination elicits unconventional IgM specific responses in naïve and previously COVID-19-infected individuals. eBioMedicine. 77, (2022).
- Piubelli, C. Subjects who developed SARS-CoV-2 specific IgM after vaccination show a longer humoral immunity and a lower frequency of infection. eBioMedicine. 89, 104471 (2023).
- Zhang, G. F. Infectivity of pseudotyped SARS-CoV-2 variants of concern in different human cell types and inhibitory effects of recombinant spike protein and entry-related cellular factors. Journal of Medical Virology. 95 (1), e28437 (2023).
- da Costa, K. A. S. Influenza A (N1-N9) and Influenza B (B/Victoria and B/Yamagata) neuraminidase pseudotypes as tools for pandemic preparedness and improved influenza vaccine design. Vaccines. 10 (9), 1520 (2022).
- Condor Capcha, J. M. Generation of SARS-CoV-2 spike pseudotyped virus for viral entry and neutralization assays: a 1-week protocol. Frontiers in Cardiovascular Medicine. 7, 618651 (2021).
- Diomede, L., et al. Doxycycline inhibition of a pseudotyped virus transduction does not translate to inhibition of SARS-CoV-2 infectivity. Viruses. 13 (9), 1745 (2021).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten